沙门氏菌是一种导致人类食源性疾病的重要病原体[1]。由沙门氏菌引起的沙门氏菌病,已经成为全球严重的公共卫生问题,并导致沉重的经济负担[2]。沙门氏菌的感染常由食用被沙门氏菌污染的食品所引起,常见的污染食品有禽类、肉类与蛋制品等[3],肠炎沙门氏菌是引起沙门氏菌感染的主要血清型[4]。临床针对沙门氏菌的感染通常使用抗生素进行治疗,然而由于抗生素的广泛使用甚至是滥用,沙门氏菌对抗生素的耐药性逐渐增强,甚至有多重耐药菌株的出现[5],这对人类的健康构成威胁。在众多抗生素中,多黏菌素被认为是治疗多重耐药革兰氏阴性菌的最后手段,近些年来多黏菌素耐药沙门氏菌菌株也在逐渐出现,这对临床治疗提出了极其严重的挑战[6]。
全基因组测序(whole genome sequencing, WGS)能够获得一个物种完整详细的遗传信息,已经逐渐成为公共卫生监测的一种常用方法,利用WGS数据可以更加详细的了解菌株的遗传信息。通过构建菌株间的系统发育树,探讨不同来源沙门氏菌之间的遗传进化关系,可以对临床感染菌株进行溯源追踪,明晰致病菌经种、养殖农产品—食品—病人链条的传播路径。传统的分子分型技术脉冲场凝胶电泳虽然被誉为金标准,但其溯源分辨能力有限。基于WGS数据分型方法,如cgSNP、cgMLST、wgSNP和wgMLST等,有着更高更精确的分辨率,在实际应用中的范围也更广[7-8]。
沙门氏菌具有入侵宿主肠道、在宿主细胞中存活和繁殖并引发炎症的能力。沙门氏菌是否引起宿主病变以及引起宿主病变的程度是由其致病性所决定的,而其致病性是由大量毒力相关因子和适应性相互作用的结果。沙门氏菌通过食物被摄入人体后,会利用其鞭毛在宿主环境中移动,泳动和群集运动是其主要的运动方式[9]。当沙门氏菌到达肠道附近时,可能会黏附在肠道黏膜上并侵袭肠道上皮细胞从而引起炎症反应。这是沙门氏菌的表面结构(如菌毛)和多种毒力因子相互作用的过程,从而促进黏附和侵入[10]。此外沙门氏菌黏附在肠道上皮细胞的过程中还有可能会形成生物被膜,这有助于它们在宿主中定植并抵抗宿主的免疫反应,提高了菌体的整体耐受性[11]。由于大蜡螟幼虫的先天免疫系统和哺乳动物的免疫系统具有高度的相似性,它已经作为模式物种广泛应用于沙门氏菌的毒力研究中[12-13],这种模型的应用对于研究沙门氏菌的毒力机制十分重要。因此,本研究探讨临床沙门氏菌的耐药状况,不同菌株来源之间的遗传进化关系,测定了沙门氏菌的生物被膜形成能力、运动能力、黏附侵袭能力和对大蜡螟幼虫的致死率。以期探讨临床耐药和高致病性肠炎沙门氏菌的进化来源,从而为食品安全和公共卫生保障提供参考。
1 材料与方法
1.1 菌株、昆虫与试剂
本研究的5株肠炎沙门氏菌,分离自临床腹泻病人的粪便样本;参考菌株为标准菌株ATCC 14028,中国工业微生物菌种保藏管理中心。此外,质控菌株为大肠埃希氏菌ATCC 25922。所有的菌株保存于50%(体积分数)甘油中,并置于-80 ℃冰箱中保存。
人结直肠癌细胞(Caco-2),上海恒生物科技有限公司;6周龄左右的大蜡螟幼虫,体长2 cm左右,体重约300 mg,淘宝店铺上海爬缘。
主要试剂:LB肉汤、LB营养琼脂、细菌琼脂粉,青岛海博有限公司;D-葡萄糖,上海阿拉丁生化科技股份有限公司;MHB(Muller-Hinton broth)培养基、MHA(Muller-Hinton agar)培养基,北京陆桥技术股份有限公司。曲拉通(Triton-X-100),飞净生物科技有限公司;PBS(20 ×),上海源叶生物科技有限公司;DMEM(Dulbecco’s modified eagle medium)培养基、青霉素-链霉素溶液(双抗)、0.25% Tripsin-EDTA(胰酶)、胎牛血清、非必需氨基酸添加剂,上海富恒生物科技有限公司;GN4F革兰氏阴性菌药敏板,美国赛默飞世尔科技有限公司;Ezup柱式细菌DNA提取试剂盒,上海生工生物工程有限公司。
1.2 仪器与设备
SW-CJ-IFD超净工作台,江苏苏州泰安空气技术有限公司;HVE-50立式压力灭菌锅,华粤行仪器有限公司;THZ-1038恒温培养摇床、HWS-26水浴锅、BPN-80CH(UV)二氧化碳培养箱,上海一恒科学仪器有限公司;HWS-150恒温恒湿细菌培养箱,上海比朗仪器有限公司;PF-10D纯水仪,上海和泰仪器有限公司;Fresco 17高速冷冻离心机,赛默飞世尔(中国)科技有限公司;LBZ-XT细菌浊度仪,青岛路博建业环保科技有限公司;50 μL微量注射器,上海玻利鸽工贸有限公司。
1.3 实验方法
1.3.1 WGS与分析
使用DNA提取试剂盒提取沙门氏菌基因组,全基因组序列使用DNBSEQ-G400和PacBio RS II进行短读和长读混合测序;沙门氏菌基因组序列经Prokka软件进行注释[14];使用SeqSero2软件和pubMLST网站分别对5株沙门氏菌的血清型和MLST型进行鉴定[15-16];菌株之间的进化关系使用Parsnp软件进行构建[17],以LT2(登录号:GCA_000006945.2)为参考菌株,并结合从国家生物技术中心(National Center for Biotechnology Information, NCBI)数据库获得的70株肠炎沙门氏菌构建cgSNP进化树。
1.3.2 抗生素耐药表型测定
使用革兰氏阴性药敏版测定32种抗生素的最小抑菌浓度(minimum inhibitory concentration, MIC)。药敏板上包被有非荧光底物和适当稀释的抗生素。从MHA平板上挑取3~5个生长正常的菌落,在无菌水中吹打混匀,调节成0.5麦氏浊度(McFarland)。取配制好的菌液10 μL加入到10 mL MHB中充分混匀,分别取30 μL加入到药敏板的微孔中。将药敏板置于37 ℃恒温恒湿培养箱中静置18~24 h,观察微孔中细菌生长情况。参考美国临床实验室标准化委员会(Clinical and Laboratory Standards Institute,CLSI)所制定的标准中规定的MIC折点对实验结果进行判断[18]。本实验以大肠埃希氏菌ATCC 25922作为质控菌株。
1.3.3 生物被膜形成能力测定
采用钢片法对沙门氏菌的生物被膜形成能力进行测定[19]。对过夜培养至稳定期的菌液梯度稀释后进行初始计数,将无菌不锈钢片(1.4 cm×1.2 cm×0.1 cm,304食品级)置于24孔板中,向每个孔中分别加入1.8 mL的LB液体培养基和200 μL稀释至106 CFU/mL的菌液,在37 ℃静置培养24 h。随后取出培养完成的24孔板,使用一次性无菌镊子取出不锈钢片,用无菌生理盐水反复冲洗3次,去除不锈钢片表面未黏附的菌体,随后将不锈钢片置于装有无菌玻璃珠和5 mL无菌生理盐水的15 mL离心管中,使用涡旋仪振荡2 min后吸取上清液,梯度稀释,并在LB琼脂平板上进行计数。生物被膜的形成量用公式(1)计算:
(1)
式中:N,生物被膜形成量,lg CFU/cm2;N0,不锈钢片上黏附的细胞量,CFU;S,不锈钢片两面的面积,cm2。
1.3.4 运动能力测定
对5株肠炎沙门氏菌分别进行泳动(swimming)和群集运动(swarming)能力的检测,以运动直径作为表征参数。称取25 g的LB液体培养基,加入3 g的细菌琼脂粉和1 L去离子水,配制成泳动实验所需的0.3%半固体培养基。称取25 g的LB肉汤培养基,加入5 g的细菌琼脂粉和5 g葡萄糖,加入1 L去离子水,配制成群集运动实验所需的0.5%半固体培养基[20]。将过夜培养至稳定期的菌液梯度稀释至106 CFU/mL,取1.5 μL上述稀释后的菌液插入0.3%半固体培养基的内部,静置5~10 min后,将平板转移至37 ℃恒温培养箱中培养9 h后取出,测量泳动直径。取1.5 μL稀释后的菌液滴加在0.5%半固体培养基的表面,静置5~10 min后,将平板转移至37 ℃恒温培养箱中培养11 h后取出,测量群集运动直径。
1.3.5 黏附与侵袭能力测定
测定沙门氏菌对Caco-2细胞的黏附和侵袭能力[21]。将约1×105个Caco-2细胞接种在细胞培养瓶中,加入含有10%胎牛血清、1%链霉素-青霉素溶液和1%非必需氨基酸的DMEM培养基中。在37 ℃、5% CO2的培养箱中培养约48 h。使用胰蛋白酶消化4 min,转移到12孔板中,再培养约48 h。随后,去除每个孔中的旧培养基,用1% PBS洗涤3次,每孔再加入1 mL的DMEM培养基。取1 mL的培养至稳定期的菌液,离心后用1 mL的DMEM重悬,取10 μL上述重悬菌液加入到12孔板中并充分混匀。在37 ℃、5% CO2条件下感染1 h。随后,去除每个孔中的旧培养基,并使用1% PBS洗涤3次以去除未黏附的细菌。随后,使用0.1% Triton X-100裂解细胞5 min,并快速收集裂解液制备稀释梯度,在LB平板上计数。对于侵袭实验,在感染1 h后,用1% PBS洗涤3次,然后用1 mL含有100 μg/mL链霉素-青霉素的DMEM培养1 h,以杀灭游离及黏附在细胞表面的细菌。结果以沙门氏菌黏附和侵袭的细胞占初始接菌量的比例来表示。
1.3.6 大蜡螟幼虫存活率测定
使用沙门氏菌感染大蜡螟幼虫,通过其存活率评估沙门氏菌的毒力[22]。将购买的大蜡螟幼虫储存在15 ℃黑暗环境中,实验之前在37 ℃下复苏4 h。将过夜培养至稳定期的菌液用PBS进行梯度稀释至106 CFU/mL备用,在培养皿中将幼虫分成12只一组,从幼虫的血腔(第4对腹足右足处)注射10 μL 106 CFU/mL菌液,注射PBS作为对照。在37 ℃下培养48 h,每隔12 h观察一次幼虫存活情况,当幼虫身体呈黑色且对外界刺激无反应或翻转虫体后无法恢复正常体位,则认定幼虫死亡,计算大蜡螟幼虫的死亡率。
1.3.7 统计分析
每组实验重复不少于3次,使用单因素方差分析(one-way analysis of variance, ANOVA)和邓肯检验(Duncan’s)对5株肠炎沙门氏菌和标准菌株之间的生物被膜形成能力、黏附与侵袭能力和运动能力的差异性进行显著性分析。实验采用95%置信限,P<0.05则差异有统计学意义。所有数据均通过SPSS 27进行分析。
2 结果与分析
2.1 基因组信息及分子分型结果
数据经过质控后,将拼接完成的全基因组序列进行基因组功能元件的注释,结果如表1所示。基因组大小约在4.8 Mb,菌株44A的基因组最大,平均GC含量为52.2%或52.1%;44A的蛋白质编码区(coding sequences, CDS)数量也最多,有4 523个CDS;5株菌拥有相同数量的rRNA,tRNA的数量也接近,在86~88之间。
表1 肠炎沙门氏菌基因组的基本特征
Table 1 Basic characterization of the Salmonella Enteritidis genomes

菌株总长度/bpCDStRNA5S rRNA16S rRNA23S rRNAGC含量/%21A4 807 7504 5068887752.233A4 761 0864 4528787752.138A4 774 7774 4658687752.144A4 827 2784 5238887752.248A4 739 3344 4278787752.2
5株沙门氏菌的血清型通过SeqSero2软件进行鉴定,ST型通过pubMLST网站进行鉴定。5株肠炎沙门氏菌的亚型信息一致,O抗原为O-9,H抗原为H-g, m,血清型为肠炎,ST型为ST11型。
2.2 cgSNP进化分析结果
使用Parsnp软件构建了5株肠炎沙门氏菌以及从NCBI数据库获得的70株肠炎沙门氏菌之间的系统进化树,探究了不同来源肠炎沙门氏菌的进化关系,包括食品源、临床源和环境源等,结果如图1所示。菌株33A、38A和44A进化关系较近,并且和一株来自中国台湾的临床分离株聚类在一起(登录号:GCA_024266125.1)。而菌株21A和一株来自中国浙江省的临床分离株聚类在一起(登录号:GCA_033340735.1),说明可能存在相同的感染源。然而,菌株48A和一株分离自食用动物(鸡)的菌株聚类在一起(登录号:GCA_020410865.1),这很有可能是一起由于食用被污染的鸡肉所引起的感染,并且这是一株高致病性菌株,在对雏鸡急性感染实验中致死率达到了80%,基因组分析显示其携带着大量毒力因子和可移动元件[4]。
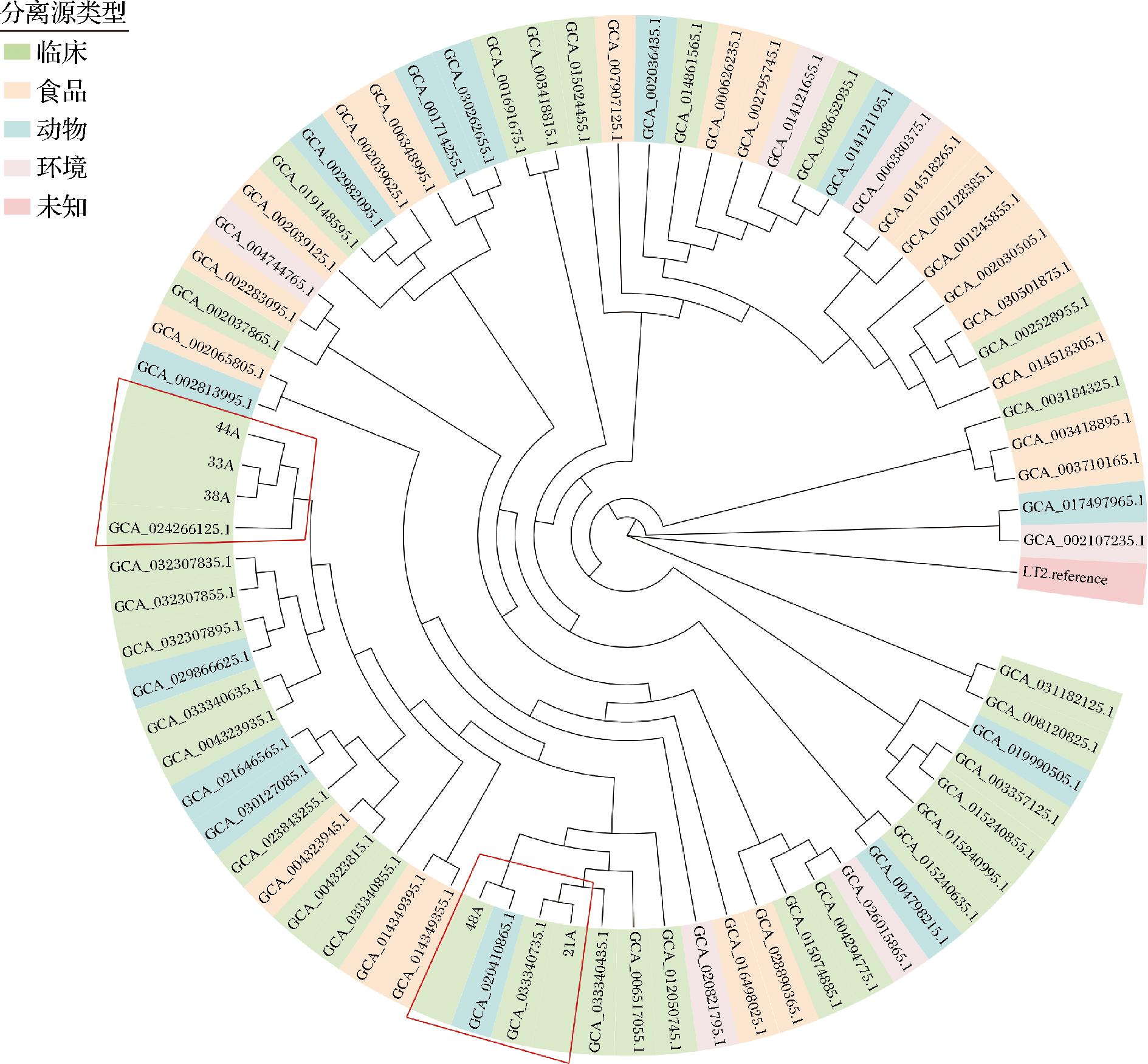
图1 肠炎沙门氏菌系统发育分析
Fig.1 Phylogenetic analysis of Salmonella Enteritidis
2.3 抗生素耐药结果
测定了5株肠炎沙门氏菌对32种抗生素的耐药性。结果如图2所示,所有的菌株都是耐药菌,都对萘啶酸具有耐药性。除菌株48A外,其余菌对氨苄西林具有耐药性,且都对三类及三类以上的抗生素具有耐药性,被定义为多重耐药菌。值得注意的是只有菌株44A对氯霉素、四环素和多西环素具有耐药性,并且33A对黏菌素和多黏菌素B具有耐药性,而多黏菌素是治疗革兰氏阴性多重耐药菌的最后手段,多黏菌素耐药菌株的出现值得我们更加重视。
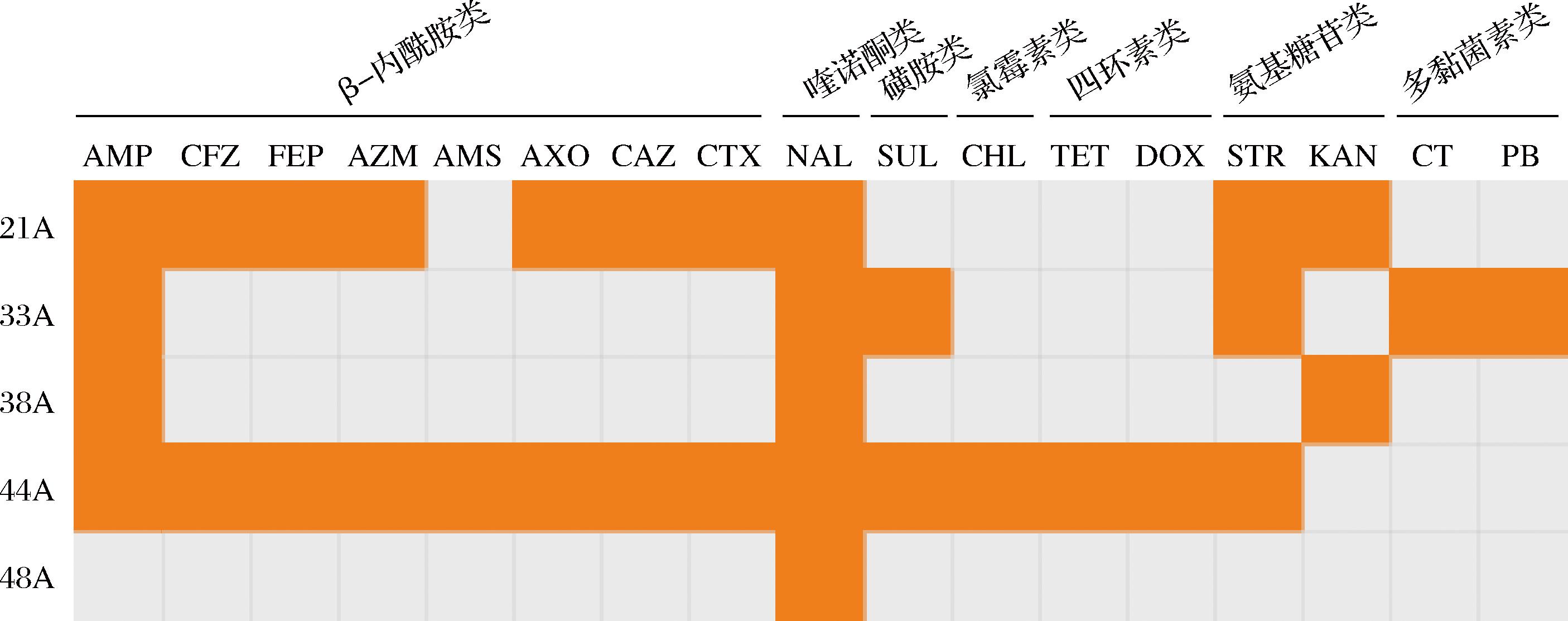
AMP-氨苄西林;CFZ-头孢唑林;FEP-头孢吡肟;AZM-阿奇霉素;AMS-氨苄西林/舒巴坦;AXO-头孢曲松;CAZ-头孢他啶;CTX-头孢噻肟;NAL-萘啶酸;SUL-磺胺异恶唑;CHL-氯霉素;TET-四环素;DOX-多西环素;STR-链霉素;KAN-卡那霉素;CT-黏菌素;PB-多黏菌素B
图2 肠炎沙门氏菌抗生素耐药谱
Fig.2 Antibiotic resistance profile of Salmonella Enteritidis
注:橙色表示耐药。
2.4 生物被膜形成能力分析
生物被膜在沙门氏菌的致病过程中起到了关键的作用,沙门氏菌通过形成生物被膜增强了其在宿主内的生存和致病能力。采用钢片法测定了5株肠炎沙门氏菌及标准菌株ATCC 14028的生物被膜形成能力,结果如图3所示。5株分离株中菌株48A的生物被膜形成能量最高(5.57 lg CFU/cm2),和21A、38A之间无显著性差异(P>0.05),却显著高于33A和44A,33A的生物被膜形成量最低(4.81 lg CFU/cm2)。标准菌株ATCC 14028的生物被膜形成量为5.8 lg CFU/cm2,显著高于33A、38A和48A,和其余菌株无明显差异(P>0.05)。
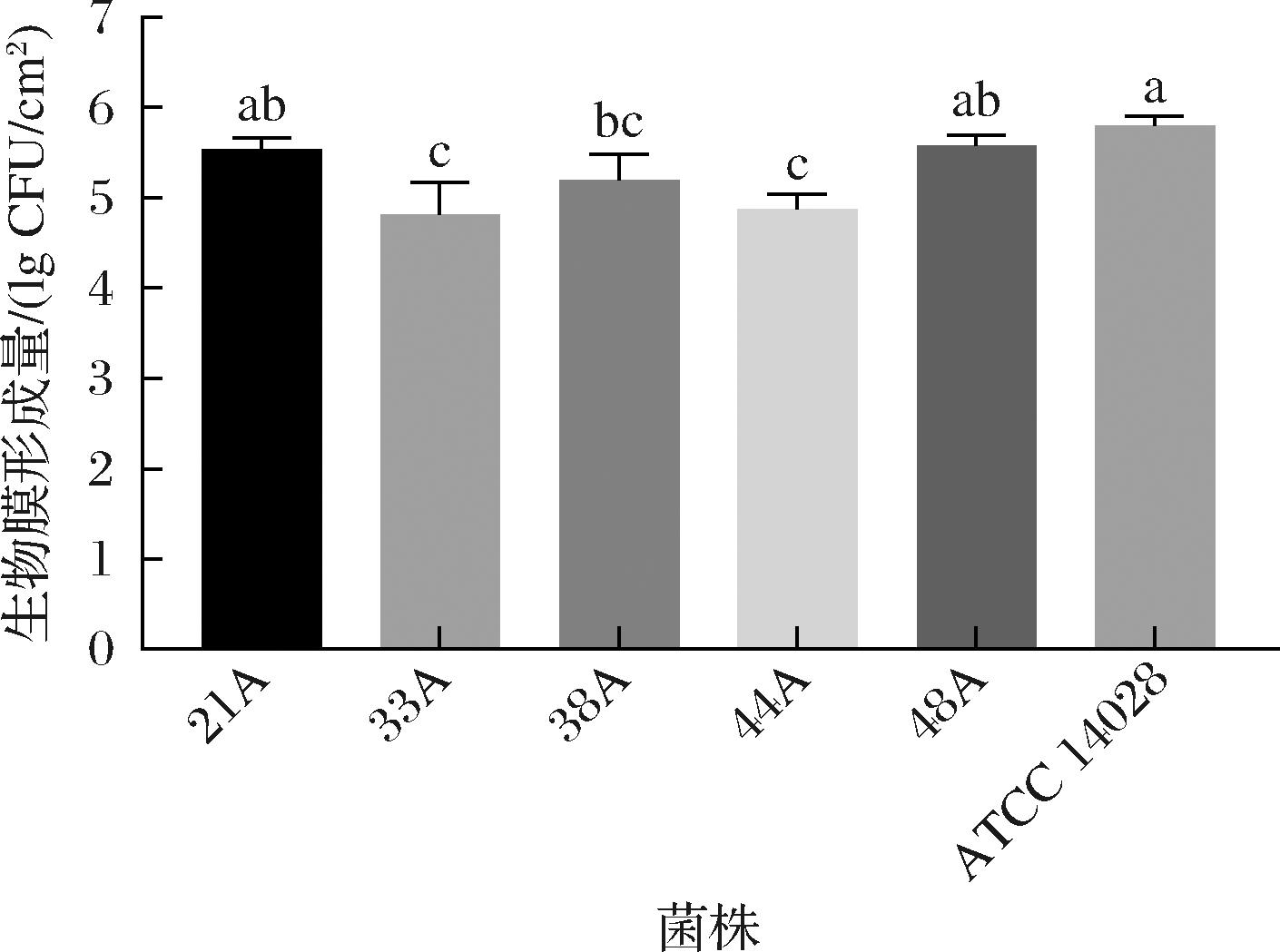
图3 肠炎沙门氏菌生物被膜形成能力
Fig.3 Biofilm-forming capacity of Salmonella Enteritidis
2.5 运动能力分析
细菌的运动性与其侵袭性有关,沙门氏菌的运动方式以泳动和群集运动为主。分别用0.3%和0.5%的半固体培养基测定沙门氏菌的泳动和群集运动能力,根据半固体培养基上菌圈的大小可以很直观的看出菌株运动能力的大小(图4)。沙门氏菌分离株以及标准菌株ATCC 14028的运动能力如图5所示。5株分离株中泳动能力最强的菌株为48A,平均运动直径为29.85 mm,显著高于33A和44A。运动直径最小的是菌株44A(18.38 mm),显著低于另外4株菌。对于群集运动能力,与泳动能力一致,菌株48A的运动直径最大(14.17 mm),33A的运动直径最小(11.82 mm),但是5株菌之间并没有显著性差异(P>0.05)。标准菌株ATCC 14028的泳动和群集运动直径分别为34.13 mm和15.19 mm,均高于本研究中的5株菌。
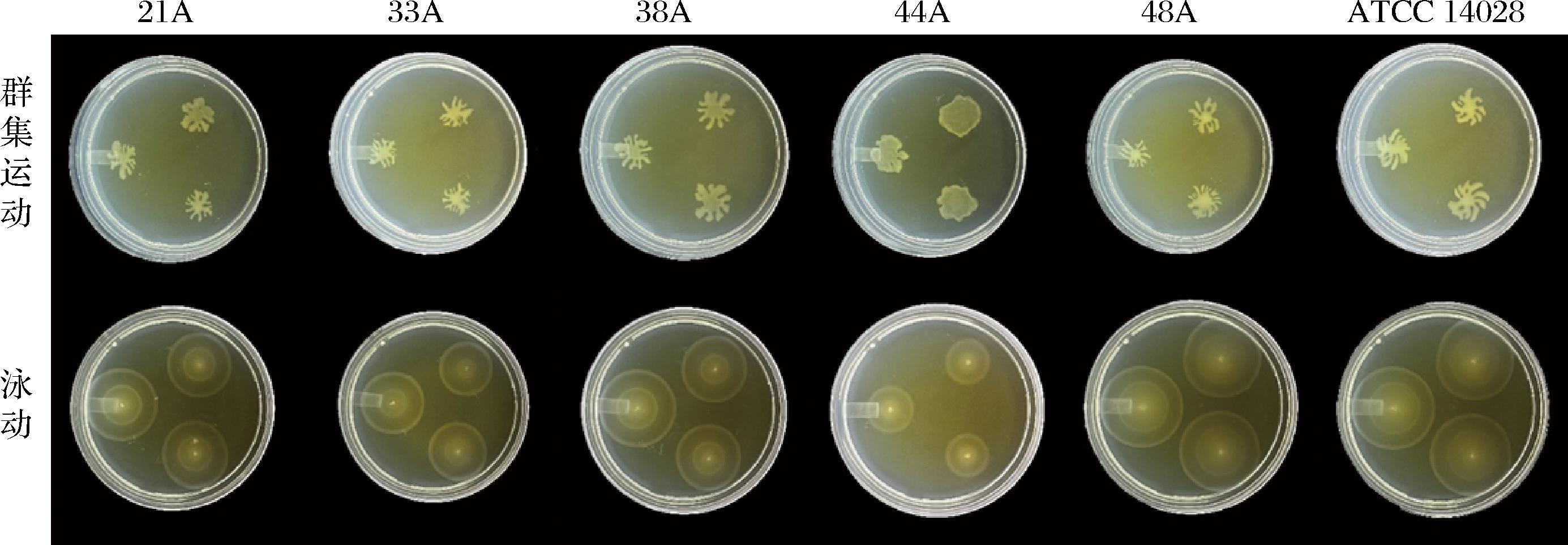
图4 肠炎沙门氏菌泳动和群集运动能力
Fig.4 Swimming and swarming motility of Salmonella Enteritidis
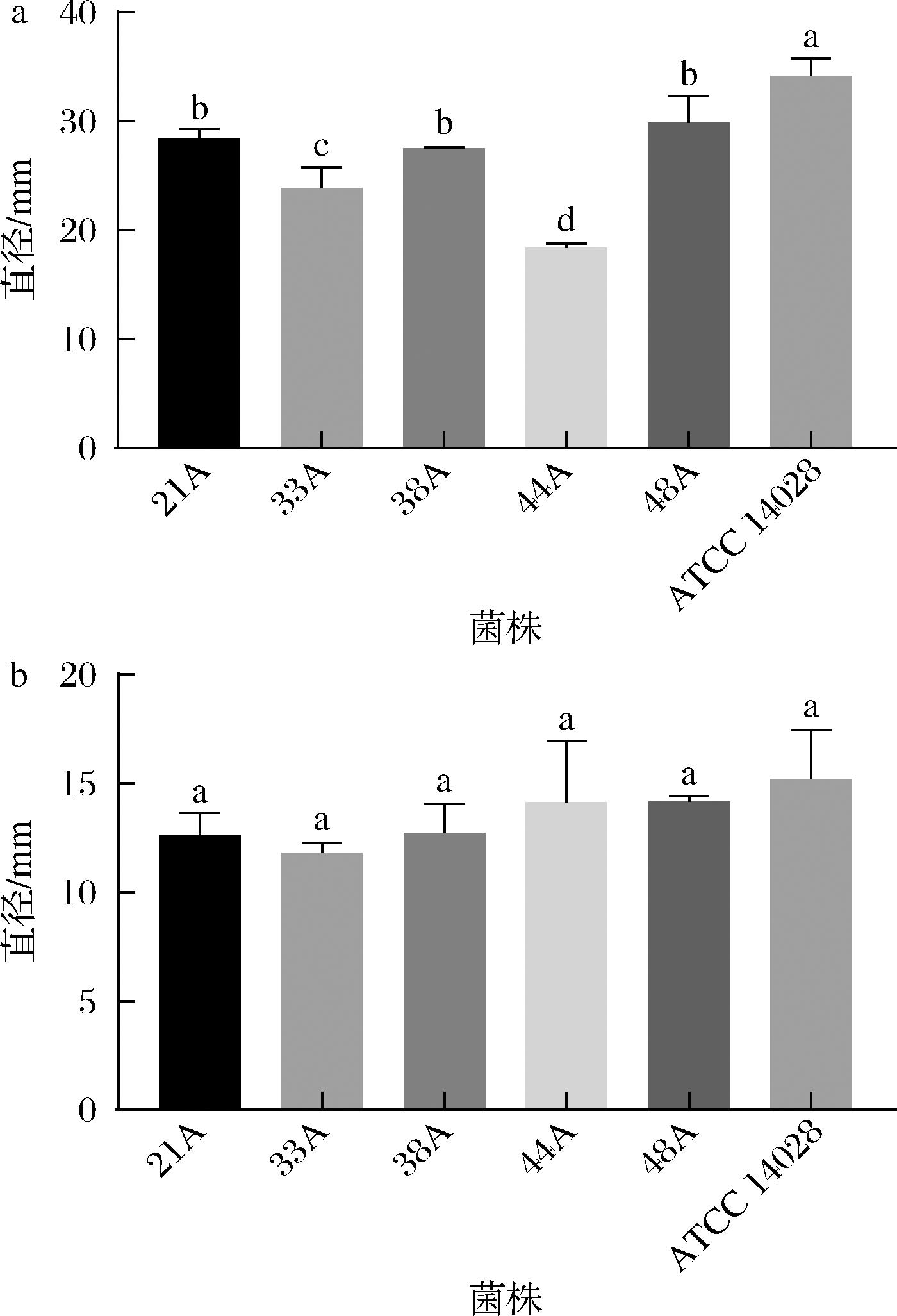
a-泳动;b-群集运动
图5 肠炎沙门氏菌泳动和群集运动直径
Fig.5 Motility diameter of Salmonella Enteritidis swimming and swarming
2.6 黏附与侵袭能力分析
黏附和侵袭是沙门氏菌感染宿主的两个关键步骤,一旦黏附成功,沙门氏菌会通过许多复杂的机制入侵宿主细胞,在宿主细胞内生存并繁殖,引起一系列的病理学变化。以Caco-2细胞作为肠道细胞模型,5株肠炎沙门氏菌及标准菌株对Caco-2细胞的黏附与侵袭结果如图6所示。
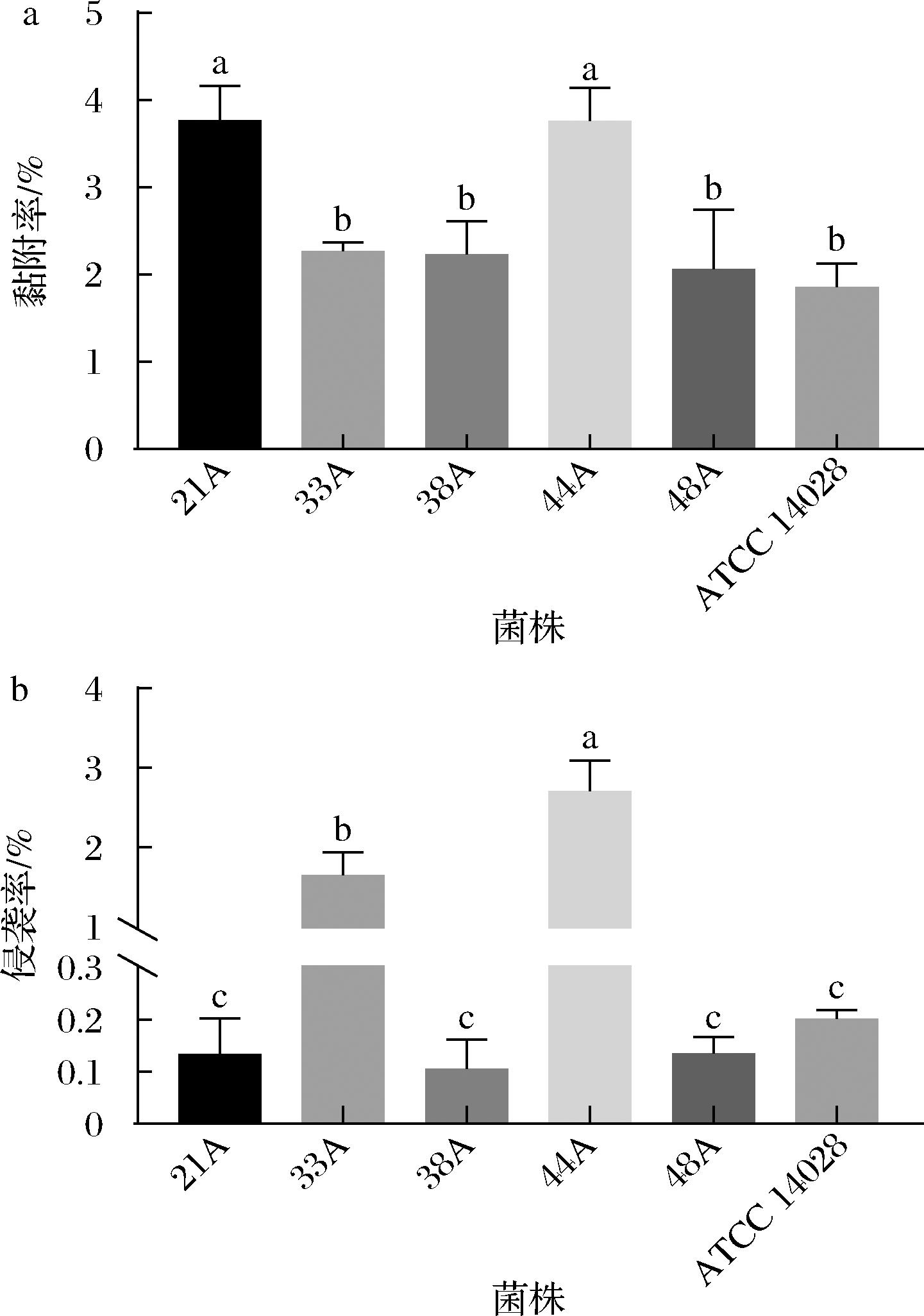
a-黏附;b-侵袭率
图6 肠炎沙门氏菌对Caco-2细胞的黏附与侵袭率
Fig.6 Adhesion and invasion of SalmonellaEnteritidis to Caco-2 cells
5株分离株中,菌株21A与44A的黏附率较强(3.77%),显著高于另外3株菌(P<0.05)。肠炎沙门氏菌侵入Caco-2细胞的能力具有菌株异质性,菌株44A的侵袭能力最强(侵袭率为2.71%),显著高于其余菌株,侵袭率约为菌株38A的26倍(0.11%)。标准菌株ATCC 14028的平均黏附率为1.81%,低于5株肠炎沙门氏菌的平均值(2.93%);平均侵袭率为0.2%,同样低于5株肠炎沙门氏菌的平均值(0.95%)。
2.7 大蜡螟幼虫存活率分析
大蜡螟的免疫系统与哺乳动物的免疫系统相似,尤其是在炎症和免疫反应方面。因此,以大蜡螟作为昆虫模型在微生物感染的致病机制研究中得到了广泛应用。本研究中,以接种106 CFU/mL细菌浓度48 h后的大蜡螟幼虫存活率作为观测指标,结果如图7所示。48 h后菌株48A对大蜡螟幼虫的致死率最高,为44.44%(16/36),其次是菌株44A(41.67%,15/36),菌株38A的致死率最低(36.11%);菌株21A和33A具有相同的致死率(38.89%)。标准菌株ATCC 14028的致死率为19.44%,低于本研究中的所有菌株,此标准菌株是从鸡中分离出来的,这表明临床分离株的致病性是高于食用动物分离源。
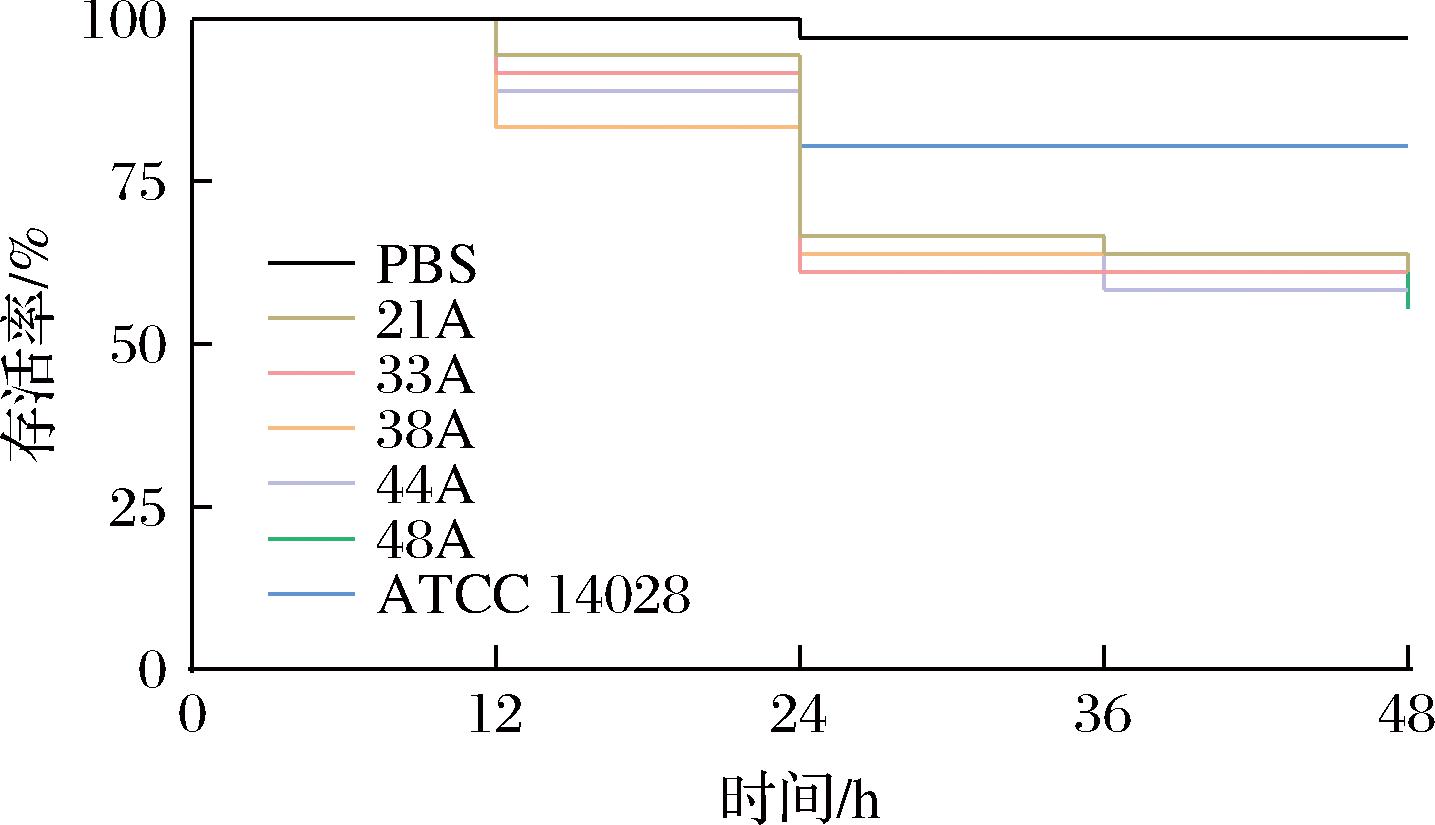
图7 大蜡螟幼虫感染肠炎沙门氏菌后的存活率
Fig.7 Survival rate of Galleria mellonella larvae after infection with Salmonella Enteritidis
3 讨论与结论
沙门氏菌是一种重要的食源性致病菌,传播的主要途径为受污染的禽肉类和蛋类食品。在沙门氏菌众多血清型当中,以肠炎沙门氏菌和鼠伤寒沙门氏菌最为常见。本研究将5株ST11型临床肠炎沙门氏菌与另外70株肠炎沙门氏菌共同构建系统发育树,除菌株48A之外的4株菌和公共数据库中的临床分离株聚类在一起,而48A和分离自食用动物鸡的一株菌具有较高的同源性,并且这是一株高致病性菌株,在对雏鸡急性感染实验中致死率达到了80%,基因组分析显示其携带着大量毒力因子和可移动元件。并且,在本研究的大蜡螟幼虫的在致死率实验中,48A的致死率最高。这两株高致病性菌株之间可能存在着未知的传播途径,需要进一步研究探索。2021年,北京一家食堂发生多例由沙门氏菌引起的腹泻病例,经全基因组测序并使用Parsnp软件构建了cgSNP系统发育树,最终确定肠炎沙门氏菌的感染与受污染的蛋炒饭有关[23]。余树坤等[24]使用Snippy软件构建了wgSNP,确认了一起鼠伤寒沙门氏菌的爆发。利用WGS数据分型方法追踪耐药或高致病性菌株的传播情况,并确定潜在的储存库和传播途径,可以向公共卫生提供更有效的控制策略。
沙门氏菌的耐药性与抗生素药物长期使用导致的选择性压力有关,临床中大量使用抗生素药物治疗沙门氏菌感染,沙门氏菌耐药和多重耐药呈明显上升趋势。本研究中的肠炎沙门氏菌全部是耐药菌株,并且除了菌株48A之外,其余菌株全是多重耐药菌株。近些年来,我国食品和临床沙门氏菌多重耐药率在逐年递增[25-26]。值得注意的是,菌株33A对多黏菌素类的2种抗生素具有耐药性。多黏菌素被誉为治疗革兰氏阴性多重耐药菌感染的最后一道防线,已广泛应用于动物和人类的临床治疗中。芦晓萍[27]研究了江西省南昌市2018年和2020—2021年沙门氏菌对多黏菌素B的耐药性变化,2018年分离株的耐药率为5.4%,2020—2021年分离株的耐药率为14.7%,多黏菌素耐药性呈逐年递增趋势。因此,有必要持续监测沙门氏菌多重耐药及多黏菌素的耐药性流行趋势。
沙门氏菌的致病性与其生存适应相关的毒力因子有关,包括运动能力、生物被膜形成能力、黏附与侵袭等。沙门氏菌的运动能力可以使其黏附在生物表面,在生物被膜的形成中起着关键作用。本研究中菌株48A的泳动能力最强,但群集运动能力和其余菌株无显著性差异。菌株48A的生物被膜形成能力也是最强的,与运动性变化趋势一致。而生物被膜是黏附在胞外物质表面的细胞聚集体,是细菌的一道防御网,可以保护细胞免受某些环境压力的影响。黏附与侵袭主要涉及的是细菌对细胞的黏附、入侵过程,菌株44A对Caco-2细胞黏附侵袭能力最强,然而其余菌株的黏附侵袭能力各不相同,体现了菌株间的异质性。然而,这些都是沙门氏菌感染宿主致病的一个过程。使用大蜡螟幼虫作为模式物种,探究其致病性的强弱。菌株48A的致死率最高,这它们的运动能力和生物被膜形成能力有着较高的一致性。美国国立卫生研究院的观测结果显示约 80%的细菌感染与生物被膜相关[28]。SOLANO等[29]研究表明,在204株不同来源的肠炎沙门菌形成生物被膜的能力存在差异,而在溶菌肉汤培养基中生物被膜形成率约为71%。如何采取有效的措施去除生物被膜态的沙门氏菌是一个亟待解决的问题。
综上所述,本研究使用WGS数据对不同来源的肠炎沙门氏菌进行分析,确定了食用动物源和临床源高致病性菌株之间的相关性,深度揭示了细菌传染病爆发的可能来源。本次分离株存在多重耐药和黏菌素耐药情况,提示我们应该加强对致病菌识别监测和细菌耐药性的监测,对临床选用合适抗生素治疗提供参考依据。同时发现运动能力和生物被膜形成能力在肠炎沙门氏菌的传播及致病过程中起着关键作用,探究沙门氏菌生物被膜形成能力对其致病性的影响具有重要意义。
[1] YANG X J, HUANG J H, WU Q P, et al.Prevalence, antimicrobial resistance and genetic diversity of Salmonella isolated from retail ready-to-eat foods in China[J].Food Control, 2016, 60:50-56.
[2] ADHIKARI B, ANGULO F, MELTZER M.Economic burden of Salmonella infections in the United States[C].American Agricultural Economics Association (New Name 2008:Agricultural and Applied Economics Association), 2004 Annual meeting, August 1-4, Denver, CO, 2004.
[3] EFSA,ECDC.The European Union one health 2018 zoonoses report [J].EFSA Journal, 2019, 17(12):5926.
[4] CUI L Q, WANG X R, ZHAO Y, et al.Virulence comparison of Salmonella enterica subsp.enterica isolates from chicken and whole genome analysis of the high virulent strain S.Enteritidis 211[J].Microorganisms, 2021, 9(11):2239.
[5] WEI X Y, LONG L, YOU L, et al.Serotype distribution, trend of multidrug resistance and prevalence of β-lactamase resistance genes in human Salmonella isolates from clinical specimens in Guizhou, China[J].PLoS One, 2023, 18(4):e0282254.
[6] LU X P, ZHAI P P, LIU Z H, et al.Comparison of antibiotic resistance profiles of Salmonella isolates from retail meats in Nanchang, China, in two periods[J].Foodborne Pathogens and Disease, 2023, 20(6):237-243.
[7] LA T M, KIM T, LEE H J, et al.Whole-genome analysis of multidrug-resistant Salmonella Enteritidis strains isolated from poultry sources in Korea[J].Pathogens, 2021, 10(12):1615.
[8] ZHOU H J, LIU W B, QIN T, et al.Defining and evaluating a core genome multilocus sequence typing scheme for whole-genome sequence-based typing of Klebsiella pneumoniae[J].Frontiers in Microbiology, 2017, 8:371.
[9] BRUNELLE B W, BEARSON B L, BEARSON S M D, et al.Multidrug-resistant Salmonella enterica serovar Typhimurium isolates are resistant to antibiotics that influence their swimming and swarming motility[J].mSphere, 2017, 2(6):e00306-e00317.[10] F BREGA A, VILA J.Salmonella enterica serovar Typhimurium skills to succeed in the host:Virulence and regulation[J].Clinical Microbiology Reviews, 2013, 26(2):308-341.
BREGA A, VILA J.Salmonella enterica serovar Typhimurium skills to succeed in the host:Virulence and regulation[J].Clinical Microbiology Reviews, 2013, 26(2):308-341.
[11] 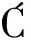 WIEK K, BUGLA-P
WIEK K, BUGLA-P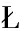 OSKO
OSKO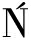 SKA G, WIELICZKO A.Salmonella biofilm development:Structure and significance[J].Postępy Higieny i Medycyny Do
SKA G, WIELICZKO A.Salmonella biofilm development:Structure and significance[J].Postępy Higieny i Medycyny Do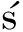 wiadczalnej, 2019, 73:937-943.
wiadczalnej, 2019, 73:937-943.
[12] VILELA F P, GOMES C N, PAZIANI M H, et al.Virulence traits and expression of bstA, fliC and sopE2 in Salmonella Dublin strains isolated from humans and animals in Brazil[J].Infection, Genetics and Evolution:Journal of Molecular Epidemiology and Evolutionary Genetics in Infectious Diseases, 2020, 80:104193.
[13] BISMUTH H D, BRASSEUR G, EZRATY B, et al.Bacterial genetic approach to the study of reactive oxygen species production in Galleria mellonella during Salmonella infection[J].Frontiers in Cellular and Infection Microbiology, 2021, 11:640112.
[14] SEEMANN T.Prokka:Rapid prokaryotic genome annotation[J].Bioinformatics, 2014, 30(14):2068-2069.
[15] JOLLEY K A, BRAY J E, MAIDEN M C J.Open-access bacterial population genomics:BIGSdb software, the PubMLST.org website and their applications[J].Wellcome Open Research, 2018, 3:124.
[16] ZHANG S K, DEN BAKKER H C, LI S T, et al.SeqSero2:Rapid and improved Salmonella serotype determination using whole-genome sequencing data[J].Applied and Environmental Microbiology, 2019, 85(23):e01746-e01719.
[17] TREANGEN T J, ONDOV B D, KOREN S, et al.The Harvest suite for rapid core-genome alignment and visualization of thousands of intraspecific microbial genomes[J].Genome Biology, 2014, 15(11):524.
[18] CLSI M100.Performance standards for antimicrobial susceptibility testing[S].CLSI, 2011.
[19] WANG Y, SUN L J, HU L L, et al.Adhesion and kinetics of biofilm formation and related gene expression of Listeria monocytogenes in response to nutritional stress[J].Food Research International, 2022, 156:111143.
[20] 魏思羽. 舍曲林对沙门氏菌四环素耐药性及毒力作用的研究[D].广州:华南理工大学,2020.WEI S Y.Investigation of the impact of sertraline on tetracycline resistance and virulence of Salmonella[D].Guangzhou:South China University of Technology, 2020.
[21] WANG X, ZHUO Q Y, HONG Y, et al.Correlation between multilocus sequence typing and antibiotic resistance, virulence potential of Campylobacter jejuni isolates from poultry meat[J].Foods, 2022, 11(12):1768.
[22] HONG Y, WU Y F, XIE Y N, et al.Effects of antibiotic-induced resistance on the growth, survival ability and virulence of Salmonella enterica[J].Food Microbiology, 2023, 115:104331.
[23] ZHANG Y W, LIU K K, ZHANG Z B, et al.A severe gastroenteritis outbreak of Salmonella enterica serovar Enteritidis linked to contaminated egg fried rice, China, 2021[J].Frontiers in Microbiology, 2021, 12:779749.
[24] 余树坤, 刘浪, 谭雅心, 等.一起鼠伤寒沙门氏菌ST19引起的食物中毒调查、溯源分析与探讨[J].中国人兽共患病学报,2024,40(1):82-89.YU S K, LIU L, TAN Y X, et al.Investigation, traceability analysis, and discussion of food poisoning caused by Salmonella typhimurium ST19[J].Chinese Journal of Zoonoses,2024,40(1):82-89.
[25] TANG B, ELBEDIWI M, NAMBIAR R B, et al.Genomic characterization of antimicrobial-resistant Salmonella enterica in duck, chicken, and pig farms and retail markets in eastern China[J].Microbiology Spectrum, 2022, 10(5):e0125722.
[26] FANG L, LIN G K, LI Y, et al.Genomic characterization of Salmonella enterica serovar Kentucky and London recovered from food and human salmonellosis in Zhejiang Province, China (2016-2021)[J].Frontiers in Microbiology, 2022, 13:961739.
[27] 芦晓萍. 沙门氏菌分离株对多粘菌素B的耐药特征及耐药机制研究[D].武汉:武汉轻工大学,2023.LU X P.The characteristics and mechanism of polymyxin B resistance in Salmonella isolates[D].Wuhan:Wuhan Polytechnic University, 2023.
[28] WORTHINGTON R J, RICHARDS J J, MELANDER C.Small molecule control of bacterial biofilms[J].Organic &Biomolecular Chemistry, 2012, 10(37):7457-7474.
[29] SOLANO C, GARC A B, VALLE J, et al.Genetic analysis of Salmonella Enteritidis biofilm formation:Critical role of cellulose[J].Molecular Microbiology, 2002, 43(3):793-808.
A B, VALLE J, et al.Genetic analysis of Salmonella Enteritidis biofilm formation:Critical role of cellulose[J].Molecular Microbiology, 2002, 43(3):793-808.