咖啡酸(caffeic acid),又称3, 4-二羟基肉桂酸,是存在于多种植物中的一种天然酚酸类化合物,具有抗氧化、抗炎、抗肿瘤等多种生物学活性[1-3]。此外,咖啡酸是绿原酸[4]、迷迭香酸[5]、丹酚酸B[6]、咖啡酸苯乙酯[7]等高附加值化合物的前体,在食品、药品和化妆品等领域具有广泛的应用[8]。由于咖啡酸存在于咖啡以及茵陈、菜蓟、金银花等多种植物中,因此,可以从这些植物中直接提取来制备。但是植物生长周期长、产物积累量低,且在植物提取过程中需要用到多种溶媒,限制了其大规模的生产。咖啡酸也可以由苯酚或取代苯酚经过化学催化剂反应生成,但化学合成过程中反应条件要求高、制备工艺复杂、成本高,并不是一条绿色经济的生产路线。随着合成生物技术的发展,代谢改造微生物合成咖啡酸成为一种有效的替代方法。
在植物中,苯丙氨酸解氨酶(phenylalamine ammonia lyase,PAL)催化L-苯丙氨酸脱氨生成反式肉桂酸,反式肉桂酸在肉桂酸-4-羟化酶(4H)催化下生成对香豆酸,最后对香豆酸在对香豆酸-3-羟化酶(C3H)催化下生成咖啡酸。植物来源的C3H和C4H属于P450酶,在微生物中难以高效表达[9]。BERNER等[10]首次报告了西班牙糖丝菌(Saccharothrix espanaensis)来源的4-香豆酸-3-羟基化酶(由Sam5编码)和酪氨酸解氨酶(由Sam8编码),在弗氏链霉菌中表达可以将L-酪氨酸转化为咖啡酸。CHOI等[11]将这两个来源于西班牙糖丝菌的Sam8和Sam5基因在大肠杆菌表达,成功将对香豆酸转化为咖啡酸。LIN等[12]发现来自大肠杆菌自身的4-羟基苯乙酸3-加氧酶(HpaB)和4-羟基苯乙酸3-还原酶(HpaC)可以替换微生物中难表达的C3H。通过将大肠杆菌来源的HpaBC与荚膜红细菌(Rhodobacter capsulatus)来源的酪氨酸解氨酶(tyrosine ammonia-lyase,TAL)在大肠杆菌中共表达,成功构建出了一条咖啡酸异源合成途径,并通过解除酪氨酸的反馈抑制步骤,获得了50.2 mg/L的咖啡酸。进一步敲除大肠杆菌中苯丙氨酸合成途径以及系统优化酪氨酸合成途径等,使得咖啡酸的产量达到了766.7 mg/L[13]。2022年,研究人员利用大肠杆菌为出发菌株,表达了约氏黄杆菌(Flavobacterium johnsoniaeu)来源的FjTAL和大肠杆菌内源的HpaBC,并通过优化基因拷贝数、敲除酪氨酸合成的竞争途径、强化辅因子(flavin adenine dinucleotide,FAD )的合成以及过表达转运蛋白ycjP,使得咖啡酸摇瓶产量达到了775.7 mg/L,通过在5 L发酵罐进行发酵条件优化后,发酵66 h咖啡酸产量达到了7.9 g/L[14],这是目前报道的微生物合成咖啡酸的最高产量。在上述构建的大肠杆菌工程菌中,代谢途径中的基因常常通过多个游离质粒进行表达,菌株稳定性差,在培养过程中需要添加多种抗生素,且培养过程中需要添加异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)进行诱导,影响了后期的提取工艺,不具备食品安全性和生产经济性。
酿酒酵母作为基因操作简单的真核微生物,与大肠杆菌相比,不易染噬菌体,且在生物安全性方面和植物基因的功能表达方面有其独特的优势,是植物天然产物异源合成的极佳宿主。在酿酒酵母中,LIU等[15]筛选了不同物种来源的HpaB和HpaC,并通过组合表达发现铜绿假单胞菌的HpaB和肠沙门氏菌的HpaC的组合效果最佳,可以有效地将对香豆酸转化到咖啡酸。并结合圆红冬孢酵母(Rhodotorula toruloides)来源的RtTAL表达,在酿酒酵母中实现了咖啡酸的从头合成,产量达到289.4 mg/L。2022年,CHEN等[16]通过改造中心代谢提高NADPH供应,通过构建胞浆FAD(H2)合成途径以及将线粒体FAD(H2)导到胞浆以提高FAD(H2)供应,显著提高了咖啡酸生物合成效率,摇瓶产量达到了1.6 g/L,在5 L发酵罐中补料分批发酵产量达到了5.5 g/L。
2021年,本课题组以酿酒酵母内源L-酪氨酸为前体,将粘红酵母(Rhodotorula glutinis)来源的RgTAL基因、铜绿假单胞菌来源的HpaB基因和沙门氏菌来源的HpaC基因组合,在酿酒酵母中构建了一条咖啡酸生物合成途径[17],并通过敲除苯丙氨酸反馈抑制的Aro3、过表达酪氨酸反馈抑制不敏感突变体Aro4K229L和Aro7G141S、敲除丙酮酸脱羧酶Aro10和表达运动发酵单胞菌来源的环己二烯脱氢酶TyrC(图1),构建的酿酒酵母菌株YCA113-8B,其咖啡酸产量提高了约2.6倍,摇瓶产量达到了568.5 mg/L。为进一步提高咖啡酸合成产量,提高前体供应至关重要。在上述合成过程中,前体分支酸会在邻氨基苯甲酸合酶(anthranilate synthase,TRP2)催化下生成邻氨基苯甲酸,即进入色氨酸支路,而预苯酸则在预苯酸脱水酶(prephenate dehydratase,PHA2)的催化下合成苯丙酮酸,而苯丙酮酸会进一步经酶催化转化生成苯丙氨酸,即进入苯丙氨酸途径(图1)。因此,为使更多的碳代谢流流向咖啡酸合成途径,本研究在前期构建的咖啡酸合成菌株基础上,利用CRISPR/Cas9编辑系统敲除PHA2和TRP2基因,考察了其对酿酒酵母咖啡酸合成的影响。并对工程菌株中导致氨基酸营养缺陷的URA3、HIS3、MET15标记基因进行了回补。最后将工程菌株在5 L发酵罐中进行了补料分批发酵,以期提高咖啡酸的产量。本研究为发酵生产咖啡酸奠定了基础,也为其他天然酚酸类化合物的生物合成提供了方法参考。
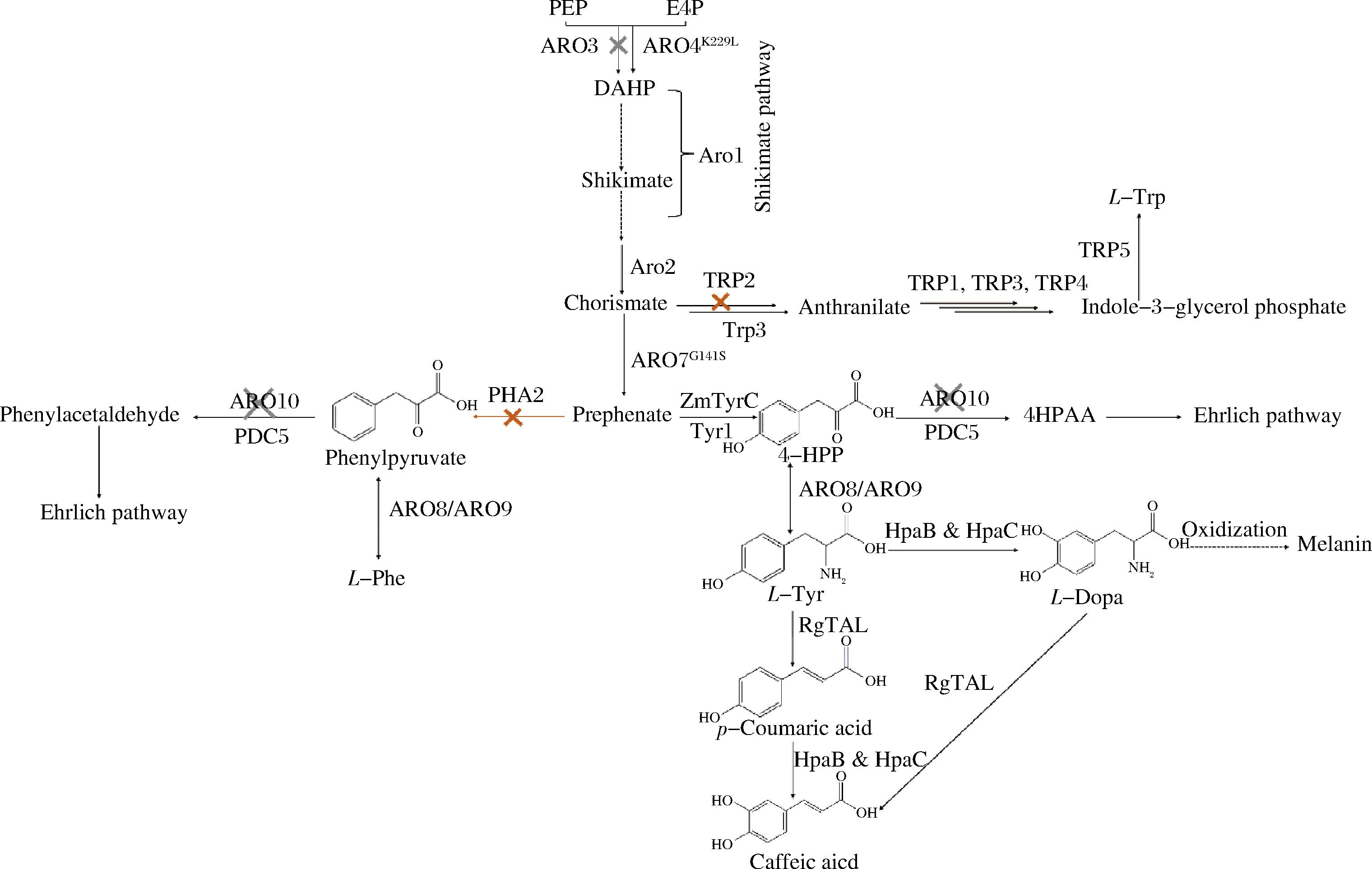
图1 酿酒酵母中咖啡酸生物合成途径
Fig.1 The biosynthetic pathway of caffeic acid in Saccharomyces cerevisiae
1 材料与方法
1.1 实验材料
1.1.1 菌株与质粒
研究使用的引物、质粒和菌株分别见表1~表3。大肠杆菌(Escherichia coli)DH5α用于重组质粒的克隆和扩增实验。前期构建的酿酒酵母咖啡酸合成菌株YCA113-8B为本实验的出发菌株。菌株、质粒全部保藏在本实验室。
表1 本研究所使用的引物
Table 1 Primers used in this study

引物引物序列(5′→3′)gPHA2-F(Esp3I)ATCGCGTCTCTGCGCAAGATTATTCAGTGGTACCGTGTTTTAGAGCTAGAAATAGCAAgTRP2-F(Esp3I)ATCGCGTCTCTGCGCAAGATGTCCTAAGACTTCCAGGTTTTAGAGCTAGAAATAGCAAtRNA-R(Esp3 I)ATCGCGTCTCAAGCTTTGCGCAAGCCCGGAATCGPHA2-FATGGCCAGCAAGACTTTGHAPHA2-FTTTTAACCAATTGGAGAACGACACTAGTATAGATTATTCAGTGGTACCGTTGAAAATTCCACCAATGGAHAPHA2-RTTAAGTAACATTCAACCTGGHATRP2-FTTTCGAGCCAAAAACAAGAAGGCCTTTGAAAGATGTCCTAAGACTTCCAGAGGCATATTTAATGCTTTGTGATHATRP2-RTGCTTCTTCAAAGTGGAGACTRP2-FATGACCGCTTCCATCAAAATMet15-F1AAGTTCTCGTCGAATGCTAGGTCMet15-R1GGTGTTGACACCTTCTCCGCHis3-F1CGTTTTAAGAGCTTGGTGAGCHis3-R1CGCCTCGTTCAGAATGACA
表2 本研究所用的质粒
Table 2 Plasmids used in this study

质粒基因型来源M3-TgRNA scaffold-tRNA, 2 μ ori, PtrunURA3-URA3[18]TYPE3-MPTEF1-CAS9-tRNA-MCS-TCYC1, 2 μ ori, PtrunURA3-URA3[18]TYPE3-M-gPHA2PTEF1-CAS9-tRNA-gPHA2-gRNA scaffold-tRNA-TCYC1, 2 μ ori (for editing PHA2), PtrunURA3-URA3本研究TYPE3-M-gTRP2PTEF1-CAS9-tRNA-gTRP2-gRNA scaffold-tRNA-TCYC1, 2 μ ori (for editing TRP2), PtrunURA3-URA3 本研究pUMRI-ΔPdc5loxp-KanMX-URA3-pbr322ori-loxp, TADH1-MCS1-PGAL10-PGAL1-MCS2-TCYC1, Pdc5 homologous arm[17]
表3 本研究所用菌株
Table 3 Strains used in this study

菌株基因型来源BY4741MATa, his3Δ1,leu2Δ0, met15Δ0, ura3Δ0实验室保藏YCA113-8BBY4741, Δgal80::LEU2, Δgal1-7::TADH1-HpaC-PGAL10-PGAL1-OHpaB-TCYC1, Δdpp1::TADH1-MCS1-PGAL10-PGAL1-ORgTAL-TCYC1, ΔAro3::TADH1-Aro7G141S-PGAL10-PGAL1- Aro4K229L-TCYC1, ΔAro10::TADH1-OZmTyrC-PGAL10-PGAL1-MCS2-TCYC1-KanMX-URA3-PBR322ori[17]YCA113-16BYCA113-8B, Δpha2本研究YCA113-20BYCA113-16 B, Δtrp2本研究YCA113-23BYCA113-16B, ΔPdc5::TADH1-MCS1-PGAL10-PGAL1-MCS2-TCYC1本研究YCA113-23BHYCA113-23B, his3::HIS3本研究YCA113-23BHMYCA113-23B, his3::HIS3, met15::MET15本研究
1.1.2 试剂
T4 DNA连接酶、Primer STARTM HS DNA高保真聚合酶、QuickCut限制性内切酶,大连宝生物公司;KOD OneTM PCR Master Mix高保真DNA聚合酶,上海东洋纺生物科技有限公司;DNA Marker、10×DNA Loading Buffer、2×Rapid Taq Master Mix,南京诺唯赞生物科技有限公司;质粒提取制备试剂盒、DNA凝胶回收试剂盒、PCR清洗试剂盒,康宁生命科学(吴江)有限公司;FM803酵母粉、FP410大豆蛋白胨,安琪酵母股份有限公司;LB肉汤(琼脂)培养基、50×TAE buffer,上海生物工程有限公司;咖啡酸标品,阿拉丁试剂上海有限公司。
1.1.3 母液与培养基
100×氨基酸母液:L-甲硫氨酸、L-组氨酸、L-色氨酸、L-苯丙氨酸、尿嘧啶质量浓度分别按照2L、2、2、5、2 mg/mL配制,添加于培养基时按照1%的体积比。
遗传霉素(G418)母液:按照20 mg/mL配制,添加于培养基时按照1%的体积比。
氨苄青霉素母液:按照100 mg/mL配制,添加于培养基时按照0.1%的体积比。
卡那霉素母液:按照50 mg/mL配制,添加于培养基时按照0.1%的体积比。
Single-straided carrier DNA(ssDNA):20 mg溶于10 mL TE Buffer中,0.22 μm微孔滤膜过滤除菌,分装后-20 ℃保存。
1 mol/L醋酸锂溶液:10.2 g乙酸锂二水定容于100 mL蒸馏水中,121 ℃湿热灭菌15 min,冷却后于4 ℃保存。
PEG MV3 350(500 g/L):50 g PEG MV3 350定容于100 mL蒸馏水中,121 ℃湿热灭菌15 min,冷却后于4 ℃保存。
Luria-Bertani(LB)液体培养基(g/L):LB肉汤培养基25,115 ℃灭菌21 min。
Luria-Bertani(LB)固体培养基(g/L):LB肉汤琼脂40,115 ℃灭菌21 min。
Yeast Extract Peptone Dextrose(YPD)液体培养基(g/L):酵母蛋白胨20,无水葡萄糖20,酵母浸粉10,115 ℃灭菌21 min,在配制固体培养基时,添加1.5%~2%的琼脂。
10×Yeast nitrogen base(10×YNB)母液:YNB 17 g, (NH4)2SO4 50 g,溶于1 L蒸馏水中,过滤除菌,添加于培养基时按照10%的体积比。
Synthetic Defined(SD)固体培养基:葡萄糖2 g,琼脂粉1.5 g,蒸馏水90 mL,115 ℃灭菌21 min, 使用时应加入1 mL 100×氨基酸母液和10 mL 10×YNB母液。
5-氟乳清酸(5-fluoroorotic acid,FOA)固体培养基:称取0.1 g FOA固体溶于1 mL二甲基亚砜中,100 mL SD固体培养基融化降温至50 ℃左右,加入1 mL 100×氨基酸母液、0.1 g/mL FOA溶液混匀。
维生素溶液(g/L):生物素0.05,泛酸钙1,烟酸1,肌醇25,盐酸硫胺素1,吡哆醇1,对氨基苯甲酸0.2,0.22 μm微孔滤膜过滤除菌,分装后4 ℃保存。
微量元素溶液(g/L):EDTA 15,ZnSO4·7H2O 10.2,MnCl2·4H2O 0.5,CuSO40.5,CoCl2·6H2O 0.86,Na2MoO4·2H2O 0.56,CaCl2·2H2O 3.84,FeSO4·7H2O 5.12,115 ℃灭菌21 min。
分批发酵补料浓缩液Ⅰ(g/L):葡萄糖500,KH2PO4 9,MgSO4 2.5,K2SO4 3.5,Na2SO4 0.28,115 ℃灭菌21 min。
分批发酵补料浓缩液Ⅱ(g/L):酵母浸粉250, 115 ℃灭菌21 min。
2.5 mol/L NaOH溶液:30 g NaOH溶于300 mL的灭菌水中。
1.2 实验方法
1.2.1 重组质粒的构建
以质粒M3-T[18]为模板,分别以gPHA2-F(Esp3 I)和tRNA-R(Esp3 I)、gTRP2-F(Esp3 I)和tRNA-R(Esp3 I)为引物,分别扩增得到PHA2 gRNA-scaffold-tRNA序列、TRP2 gRNA-scaffold-tRNA序列。使用限制性核酸内切酶Esp3 I将扩增所得的基因片段与TYPE3-M质粒[18]分别进行酶切,使用T4 DNA连接酶将酶切后的基因片段与质粒分别进行连接,导入E.coli DH5α感受态细胞中,筛选阳性转化子,分别得到TYPE3-M-gPHA2和TYPE3-M-gTRP2载体。
1.2.2 酿酒酵母重组菌株的构建
以酿酒酵母BY4741的基因组为模板,以HAPHA2-F/HAPHA2-R为引物,用高保真的Prime STARTM HS DNA聚合酶进行PCR扩增,获得同源臂HAPHA2;同理,用HATRP2-F和HATRP2-R引物进行PCR扩增,获得同源臂HATRP2。将构建的质粒载体TYPE3-M- gPHA2与同源臂HAPHA2通过醋酸锂转化法导入YCA113-8B酵母菌株中,均匀涂布在SD+Ura-平板上。培养3~4 d后,挑取酵母转化菌株于10 μL无菌水中,从中取2 μL至另一含有8 μL无菌水的PCR管中,100 ℃加热5 min后直接用作PCR模板。以PHA2-F和HAPHA2-R引物,利用KOD OneTM PCR Master Mix聚合酶进行PCR扩增,将扩增得到的PCR产物进行测序验证。将测序正确的菌株,命名为YCA113-16B菌株。为了进行下一轮基因编辑,需要将菌株YCA113-16B中含Cas9蛋白和gRNA的质粒去除。利用在没有筛选压力的培养基中,通过传代,质粒可自然丢失这一原理,将菌株YCA113-16B接种于5 mL YPD试管中,30 ℃下培养过夜后,再转接1次到YPD试管培养过夜。取1 mL菌液无菌水水洗两次后稀释10 000倍,取100 μL涂于FOA平板上。若FOA板上的单菌落在SD不加URA试管中不能生长,而在加了URA的SD试管中能生长,则说明TYPE3-M- gPHA2质粒已经去除,菌株命名为YCA113-16B(-)。同理,将TYPE3-M-gTRP2与同源臂HATRP2,通过醋酸锂转化法导入YCA113-16B (-)酵母菌株中,获得YCA113-20B菌株。
将pUMRI-ΔPdc5用Sfi I进行酶切线性化后,通过醋酸锂转化法导入YCA113-16B (-)酵母菌株中,构建得到菌株YCA113-23B。为了回补YCA113-23B中HIS3和MET15营养缺陷型基因,以酿酒酵母BY4742-MO4-His[19]基因组为模板,用His3-F1/His3-R1为引物,用高保真的Prime STARTMHS DNA聚合酶进行PCR扩增,获得HIS3营养缺陷型基因。通过醋酸锂转化法,将HIS3片段导入到YCA113-23B菌株中,并将转化细胞涂布在不添加组氨酸而添加了甲硫氨酸和苯丙氨酸的SD固体平板上,从而构建出YCA113-23BH菌株。以酿酒酵母BY4742基因组为模板,用Met15-F1/Met15-R1为引物,用高保真的Prime STARTMHS DNA聚合酶进行PCR扩增,获得MET15营养缺陷型筛选标记的基因片段,并将片段导入到YCA113-23BH酵母菌株中,将转化细胞涂布在不添加甲硫氨酸但添加了苯丙氨酸的SD平板上。从而构建出YCA113-23BHM菌株。
1.2.3 重组菌株发酵实验
在摇瓶发酵实验中,将菌株在YPD固体培养基中划线,于30 ℃恒温培养箱中培养72 h。挑取平板上的菌落接种到含5 mL YPD培养基的试管中,30 ℃,220 r/min培养15 h,取一定量的种子液接至含50 mL YPD培养基的250 mL摇瓶中,使初始OD600为0.05,30 ℃,220 r/min培养72 h。
在补料分批发酵试验中,将菌株YCA113-23BHM在YPD固体培养基中划线,于30 ℃恒温培养箱中培养72 h。挑取平板上的菌落接种到含5 mL YPD培养基的试管中,30 ℃,220 r/min培养15 h,转接1 mL菌液至含有125 mL YPD 培养基的500 mL摇瓶中,30 ℃,220 r/min培养20 h。将两瓶250 mL种子液全部接种到含有2.25 L YPD培养基的5 L发酵罐中,发酵过程初始通气量设定为2.5 L/min,搅拌速率设定为300 r/min,此时相对溶氧值设定为100。发酵过程中通过调节搅拌转速和空气流量,将溶氧维持在20左右。培养过程中通过自动补加2.5 mol/L的NaOH溶液,维持pH 5.5,30 ℃。发酵初期,每隔12 h,补250 g/L酵母粉溶液50 mL,直到菌株生长进入平稳期。葡萄糖流加速率在0~20 mL/h,使发酵液中乙醇含量控制在5 g/L以下。
1.2.4 发酵样品处理
发酵结束后,取600 μL发酵液,与600 μL甲醇超声15 min,充分混匀。然后,12 000 r/min离心1 min,取上清液100 μL,与900 μL 50%甲醇-水混匀,用0.22 μm有机系针式过滤器过滤,用于后续的液相分析。
1.2.5 检测方法
实验中使用Agilent 1200高效液相色谱仪,Pntulips QS-C18 plus(4.6 mm×250 mm,5 μm,Puningtech)液相色谱柱,所用流动相为0.1%甲酸(A泵)和乙腈(B泵),流速1 mL/min,梯度洗脱程序设定如下:0~30 min,A溶剂90%~50%,B溶剂10%~50%。柱温35 ℃。每次进样20 μL,每个样品运行15 min。咖啡酸的检测波长为320 nm,出峰时间约为11 min。
2 结果与分析
2.1 敲除PHA2对咖啡酸合成的影响
在前期的研究中,以L-酪氨酸为前体,通过引入咖啡酸合成所需的基因以及敲除苯丙氨酸反馈抑制的Aro3、过表达酪氨酸反馈抑制不敏感突变体Aro4K229L和Aro7G141S、敲除丙酮酸脱羧酶Aro10和表达运动发酵单胞菌来源的环己二烯脱氢酶TyrC,构建了一株产咖啡酸的酿酒酵母菌株YCA113-8B[17]。在该菌株中,L-苯丙氨酸和L-酪氨酸都是由预苯酸转化形成,L-苯丙氨酸途径是L-酪氨酸衍生物的主要竞争途径。阻断这条竞争途径可以使得碳代谢流转向L-酪氨酸衍生物的生物合成。在苯丙氨酸合成途径中,PHA2可以催化预苯酸生成苯丙酮酸,苯丙酮酸在芳香族氨基酸转移酶(Aro8/Aro9)作用下合成苯丙氨酸。为了进一步提高咖啡酸产量,本研究利用CRISPR/Cas9基因编辑系统敲除了YCA113-8B菌株基因组中的PHA2基因,构建了YCA113-16B菌株。经摇瓶培养72 h后,结果如图2所示,相比于YCA113-8B菌株,咖啡酸的产量提高了42%,达到了807.3 mg/L。此外,敲除PHA2后,细胞的生物量没有明显变化。结果表明敲除PHA2基因能有效地降低前体向苯丙氨酸支路途径的转化,使碳代谢更多地流向咖啡酸合成途径。
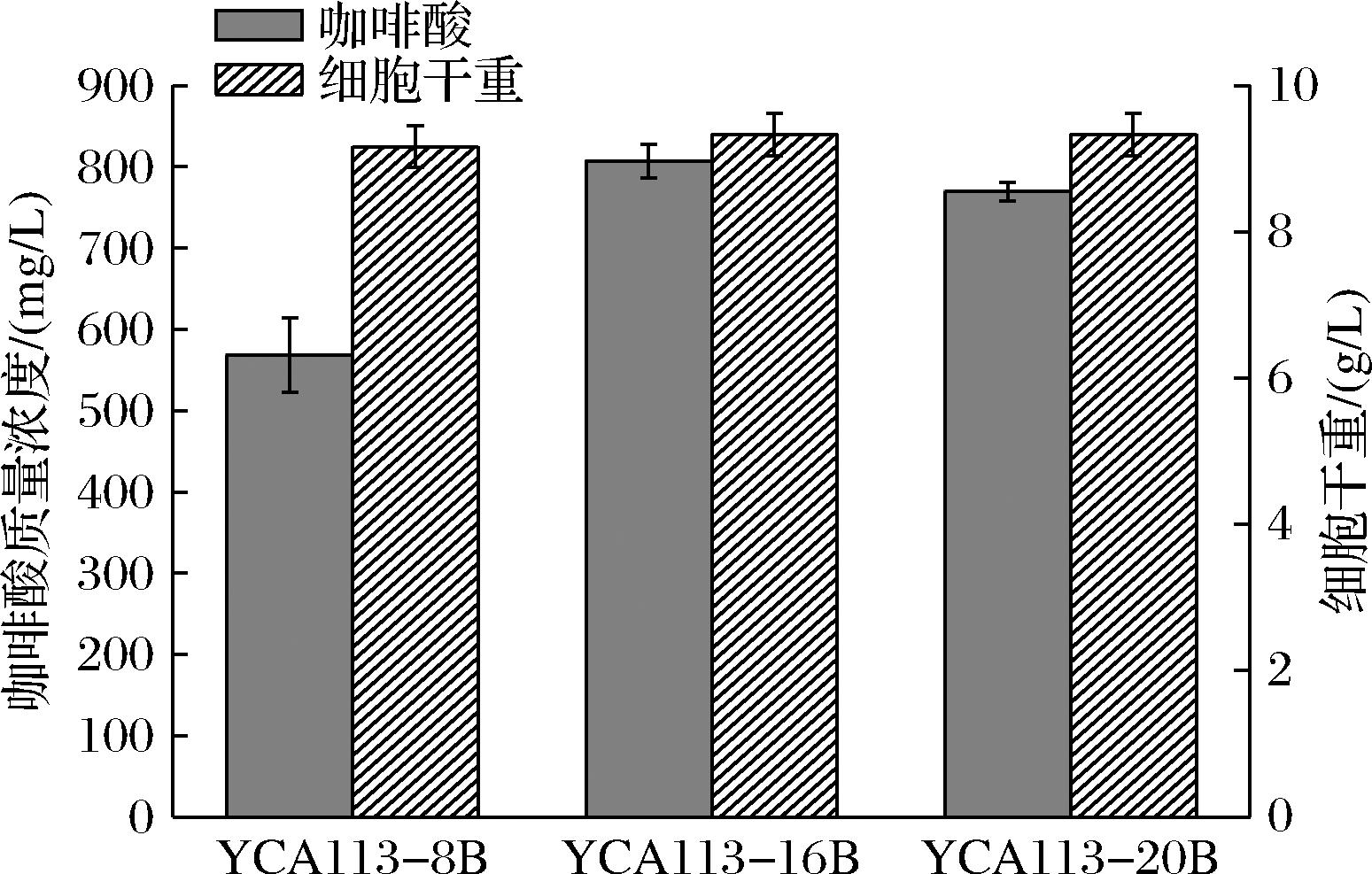
图2 敲除PHA2和TRP2对咖啡酸的影响
Fig.2 The effect of knocking out PHA2 and TRP2on caffeic acid production.
2.2 敲除TRP2对咖啡酸合成的影响
在酿酒酵母中,由莽草酸途径生成的分支酸,一方面转化生成预苯酸用于L-苯丙氨酸和L-酪氨酸的合成,另一方面,会在TRP2催化下,生成邻氨基苯甲酸,将碳代谢流转向L-色氨酸合成途径。为了进一步提高咖啡酸的产量,本研究利用CRISPR/Cas9基因编辑系统进一步将YCA113-16B菌株基因组中的TRP2基因进行敲除,获得YCA113-20B菌株。经摇瓶培养后,敲除TRP2基因后,YCA113-20B菌株咖啡酸产量略微下降(图2)。之前报道的酵母合成酪醇的研究中,LIU等[20]发现敲除TRP2基因后,酪醇的产量反而下降了19.7%,其推测是由于敲除TRP2基因后,影响了酵母生长所导致的。而在本研究中,敲除TRP2后,无论对酵母生物量和咖啡酸产量的影响不是特别明显。推测原因如下:色氨酸分支途径相对较复杂,有TRP1-TRP5五个酶通过六步反应将分支酸转化到色氨酸。其中在第一步反应中,除了TRP2能催化分支酸转化成邻氨基苯甲酸外,TRP3作为一个双功能的酶,含有谷氨酰胺酰胺转移酶和吲哚-3-甘油磷酸合酶两个独立的催化结构域,可与TRP2协同,催化分支酸向邻氨基苯甲酸的转化[21],因此仅敲除TRP2无法完全阻断色氨酸支路。同样地,在GUO等[22]的研究中,只敲除TRP3,酵母菌株生物量和酪醇的产量也无明显变化。因此,在后续的研究中,可以同时敲除TRP2和TRP3,考察其对咖啡酸合成的影响。此外,上述的研究也暗示TRP2或者TRP3可以作为酵母外源基因整合的位点。
2.3 重组菌株补料分批发酵
为了进一步提高咖啡酸的产量,计划选用摇瓶中咖啡酸产量相对较高的菌株进行补料分批发酵实验。然而,YCA113-16B是HIS3和MET15营养缺陷型菌株,且YCA113-16B菌株中,携带有URA3营养缺陷基因的TYPE3-M- gPHA2质粒容易在传代中丢失。为了在补料分批发酵中,避免组氨酸、甲硫氨酸和尿嘧啶缺乏影响细胞生长,首先,在去除了TYPE3-M-gPHA2质粒的YCA113-16B(-)菌株中,整合了URA3营养标记基因,获得了URA3能够稳定遗传的YCA113-23B菌株,在此基础上,回补了HIS3营养标记基因,获得了YCA113-23BH菌株,进一步回补了MET15营养标记基因,获得了YCA113-23BHM菌株。为了考察这些营养缺陷型基因回补后,对菌株咖啡酸和生物量的影响,先进行了摇瓶发酵实验。结果显示,在摇瓶发酵中,进行上述营养缺陷基因回补,对生物量和产量没有造成明显的影响(图3)。因此,最终选用了YCA113-23BHM菌株在5 L的发酵罐中进行了补料分批发酵,测试了其生产性能。结果如图4所示,在发酵8 h左右,葡萄糖耗尽,此时发酵液中乙醇含量达到了13.9 g/L,从10 h开始以1 g/(L·h)的速率补料,12.5 h时,乙醇浓度下降到了8.5 g/L,此时,将葡萄糖的补料速度提高到了2 g/(L·h),菌株进入了对数生长期,发酵24.5 h后,乙醇的含量只有0.2 g/L。因此将葡萄糖的补料速度提高到了4 g/(L·h)。但之后菌株的生长变慢。因此发酵36 h后将补糖速度调为2 g/(L·h)。发酵12.5 h后,咖啡酸快速积累,直到发酵84 h,咖啡酸的积累速度变缓,葡萄糖补料速度调成了1 g/(L·h)。经过103 h发酵,咖啡酸产量达到了9.3 g/L,是目前文献中报道的微生物合成咖啡酸的最高产量。但在补料分批发酵中,菌株的生物量较低,OD600最高约为137。因此需进一步优化发酵培养基以及补料工艺,以期提高生物量,从而提高咖啡酸产量。
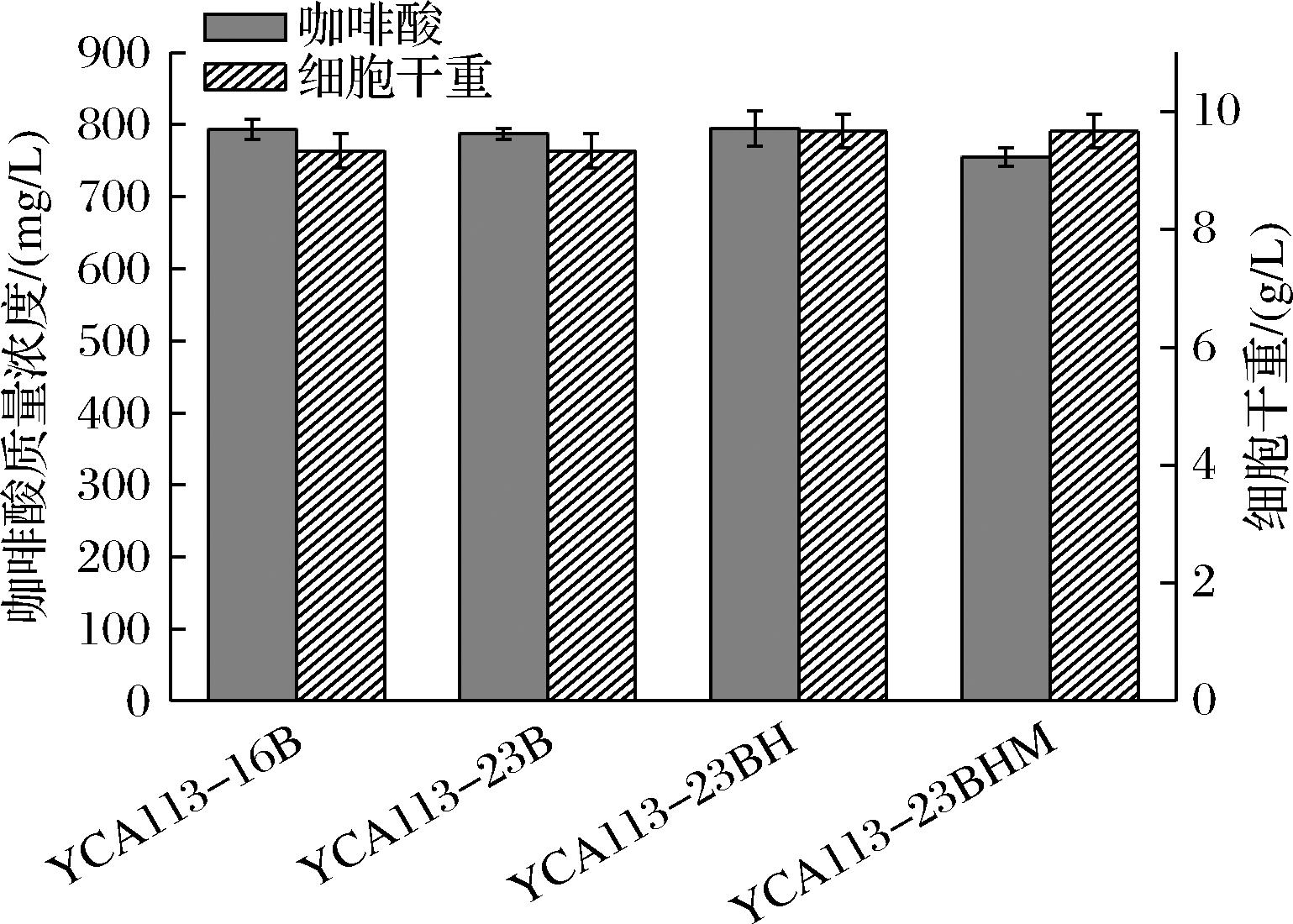
图3 回补URA3、HIS3和MET15营养缺陷基因对咖啡酸产量和生物量影响
Fig.3 Effects of URA3, HIS3, and MET15 auxotrophic marker genes complementation on caffeic acid production and biomass
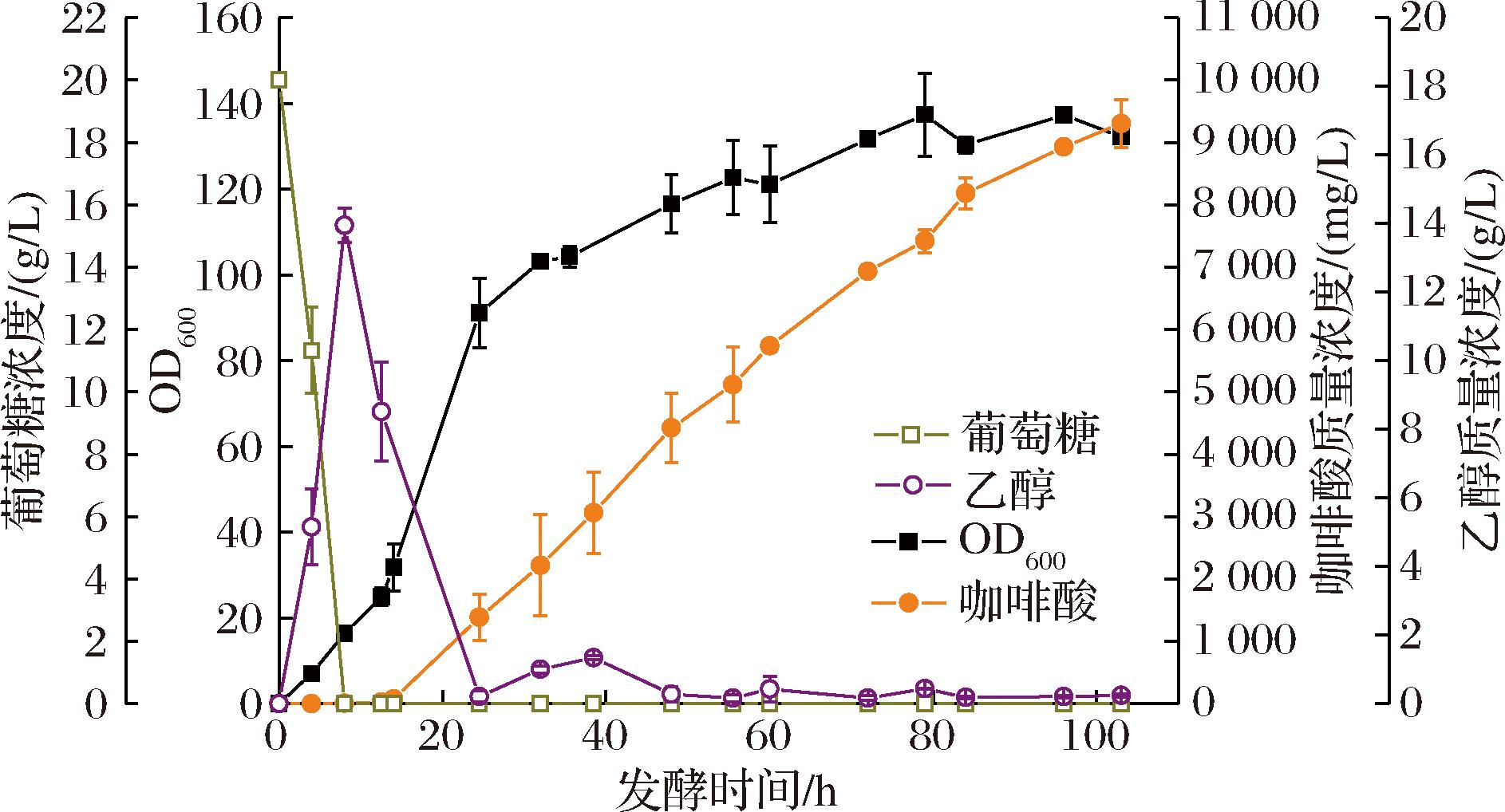
图4 YCA113-23BHM菌株分批补料发酵合成咖啡酸
Fig.4 Fed-batch fermentation of YCA113-23BHM to improve the production of caffeic acid
3 结论与讨论
咖啡酸作为一种重要的活性酚酸类化合物,具有抗氧化、抗癌、抗菌等生物学活性,在食品、医药、化妆品等领域具有广泛的应用。近年来,随着合成生物技术的发展,以大肠杆菌和酿酒酵母为底盘细胞,通过异源代谢途径的表达与优化,已成功实现了咖啡酸的生物合成,但产量仍普遍较低,距工业化应用仍有较大的距离。在前期的研究中,本课题组以酿酒酵母中L-酪氨酸为前体,构建了一株产咖啡酸菌株YCA113-8B,其摇瓶发酵产量为568.5 mg/L。为了进一步提高其产量,本研究进一步考察了敲除苯丙氨酸支路途径基因PHA2、以及色氨酸支路途径基因TRP2对咖啡酸合成效率的影响。结果表明敲除PHA2基因后咖啡酸的产量提高了42%。但进一步敲除TRP2基因后,咖啡酸产量变化不大。进一步通过回补URA3、HIS3、MET15营养标记基因,并在5 L生物反应器中进行补料分批发酵,使得酿酒酵母最终积累了9.3 g/L咖啡酸,是目前文献中报道的微生物产咖啡酸最高产量。本研究为咖啡酸的高效生物合成提供了方法借鉴,为咖啡酸衍生物的合成提供了优良的底盘细胞。在后续的研究中,为进一步提高咖啡酸产量,可以对莽草酸途径中的两个重要前体磷酸烯醇式丙酮酸以及赤藓糖4-磷酸的供应进行改造,以提高咖啡酸前体L-酪氨酸的代谢流。对香豆酸在4-羟基苯乙酸-3-单加氧酶(HpaB)和黄素还原酶(HpaC)催化下合成咖啡酸。但在表达HpaB和HpaC后,发酵液明显呈现棕色。很多研究文献表明L-酪氨酸也会在HpaB和HpaC的催化下直接生成L-多巴,L-多巴经氧化生成黑色素[17],从而造成了部分碳代谢流的损失。为此,可以对HpaB和HpaC的表达时间进行调控,在对香豆酸积累后,再开启HpaB和HpaC的表达,或者对HpaB和HpaC的催化底物专一性进行改造。此外,在补料分批发酵中,需进一步对基础培养基、补料策略进行优化,提高菌株的生物量,从而提高咖啡酸产量。
[1] CHEN J H, HO C T.Antioxidant activities of caffeic acid and its related hydroxycinnamic acid compounds[J].Journal of Agricultural and Food Chemistry, 1997, 45(7):2374-2378.
[2] CHAO P C, HSU C C, YIN M C.Anti-inflammatory and anti-coagulatory activities of caffeic acid and ellagic acid in cardiac tissue of diabetic mice[J].Nutrition &Metabolism, 2009, 6:33.
[3] ESP NDOLA K M M, FERREIRA R G, NARVAEZ L E M, et al.Chemical and pharmacological aspects of caffeic acid and its activity in hepatocarcinoma[J].Frontiers in Oncology, 2019, 9:541.
NDOLA K M M, FERREIRA R G, NARVAEZ L E M, et al.Chemical and pharmacological aspects of caffeic acid and its activity in hepatocarcinoma[J].Frontiers in Oncology, 2019, 9:541.
[4] LI S Z, LIANG C N, LIU G X, et al.De novo biosynthesis of chlorogenic acid using an artificial microbial community[J].Journal of Agricultural and Food Chemistry, 2021, 69(9):2816-2825.
[5] ZHOU P P, YUE C L, ZHANG Y C, et al.Alleviation of the byproducts formation enables highly efficient biosynthesis of rosmarinic acid in Saccharomyces cerevisiae[J].Journal of Agricultural and Food Chemistry, 2022, 70(16):5077-5087.
[6] XU Y P, GENG L J, ZHANG Y W, et al.De novo biosynthesis of salvianolic acid B in Saccharomyces cerevisiae engineered with the rosmarinic acid biosynthetic pathway[J].Journal of Agricultural and Food Chemistry, 2022, 70(7):2290-2302.
[7] WANG J, MAHAJANI M, JACKSON S L, et al.Engineering a bacterial platform for total biosynthesis of caffeic acid derived phenethyl esters and amides[J].Metabolic Engineering, 2017, 44:89-99.
[8] MAGNANI C, ISAAC V L B, CORREA M A, et al.Caffeic acid:A review of its potential use in medications and cosmetics[J].Analytical Methods, 2014, 6(10):3203-3210.
[9] KIM Y H, KWON T, YANG H J, et al.Gene engineering, purification, crystallization and preliminary X-ray diffraction of cytochrome P450 p-coumarate-3-hydroxylase (C3H), the Arabidopsis membrane protein[J].Protein Expression and Purification, 2011, 79(1):149-155.[10] BERNER M, KRUG D, BIHLMAIER C, et al.Genes and enzymes involved in caffeic acid biosynthesis in the actinomycete Saccharothrix espanaensis[J].Journal of Bacteriology, 2006, 188(7):2666-2673.
[11] CHOI O, WU C Z, KANG S Y, et al.Biosynthesis of plant-specific phenylpropanoids by construction of an artificial biosynthetic pathway in Escherichia coli[J].Journal of Industrial Microbiology &Biotechnology, 2011, 38(10):1657-1665.
[12] LIN Y H, YAN Y J.Biosynthesis of caffeic acid in Escherichia coli using its endogenous hydroxylase complex[J].Microbial Cell Factories, 2012, 11:42.
[13] HUANG Q, LIN Y H, YAN Y J.Caffeic acid production enhancement by engineering a phenylalanine over-producing Escherichia coli strain[J].Biotechnology and Bioengineering, 2013, 110(12):3188-3196.
[14] WANG L, LI N, YU S Q, et al.Enhancing caffeic acid production in Escherichia coli by engineering the biosynthesis pathway and transporter[J].Bioresource Technology, 2023, 368:128320.
[15] LIU L Q, LIU H, ZHANG W, et al.Engineering the biosynthesis of caffeic acid in Saccharomyces cerevisiae with heterologous enzyme combinations[J].Engineering, 2019, 5(2):287-295.
[16] CHEN R B, GAO J Q, YU W, et al.Engineering cofactor supply and recycling to drive phenolic acid biosynthesis in yeast[J].Nature Chemical Biology, 2022, 18(5):520-529.
[17] ZHOU P P, YUE C L, SHEN B, et al.Metabolic engineering of Saccharomyces cerevisiae for enhanced production of caffeic acid[J].Applied Microbiology and Biotechnology, 2021, 105(14-15):5809-5819.
[18] ZHOU P P, FANG X, XU N N, et al.Development of a highly efficient copper-inducible GAL regulation system (CuIGR) in Saccharomyces cerevisiae[J].ACS Synthetic Biology, 2021, 10(12):3435-3444.
[19] LYU X M, WANG F, ZHOU P P, et al.Dual regulation of cytoplasmic and mitochondrial acetyl-CoA utilization for improved isoprene production in Saccharomyces cerevisiae[J].Nature Communications, 2016, 7:12851.
[20] LIU H Y, TIAN Y J, ZHOU Y, et al.Multi-modular engineering of Saccharomyces cerevisiae for high-titre production of tyrosol and salidroside[J].Microbial Biotechnology, 2021, 14(6):2605-2616.
[21] CAO M F, GAO M R, SU STEGUI M, et al.Building microbial factories for the production of aromatic amino acid pathway derivatives:From commodity chemicals to plant-sourced natural products[J].Metabolic Engineering, 2020, 58:94-132.
STEGUI M, et al.Building microbial factories for the production of aromatic amino acid pathway derivatives:From commodity chemicals to plant-sourced natural products[J].Metabolic Engineering, 2020, 58:94-132.
[22] GUO W, HUANG Q L, FENG Y H, et al.Rewiring central carbon metabolism for tyrosol and salidroside production in Saccharomyces cerevisiae[J].Biotechnology and Bioengineering, 2020, 117(8):2410-2419.