鸭肉富含蛋白质、维生素、铁、硒等元素,但脂肪和胆固醇含量低,是一种营养丰富的食物来源[1]。酱卤鸭肉是鸭肉经预煮后,用香辛料和调味料加水煮制而成。酱卤鸭肉制品极大程度上保留了鸭肉的营养、风味,且种类繁多、鲜嫩适中,深受广大消费者的喜爱。然而,酱卤鸭肉制品水分含量高,加工过程中其中心温度较低,容易在运输、销售过程中受到微生物污染,这不仅会导致经济损失,也可能会威胁消费者的健康,同时限制了产品的销售周期和半径[2]。散装酱卤鸭肉制品从生产企业运输到门店,然后贮藏在冷藏柜中,再卖给消费者,该过程大约历时3~6 h。由于敞开放置,酱卤鸭肉制品在冷藏柜中贮藏阶段是微生物定植和繁殖的最佳时期,所以冷藏柜中及周围环境的卫生状况显著影响着散装酱卤鸭肉制品的货架期。消费者在购买酱卤鸭肉制品后未立即食用,后续的贮藏也会影响酱卤鸭肉制品的品质。因此研究酱卤鸭肉制品贮藏过程中的微生物多样性及其变化规律,对于指导酱卤鸭肉制品的贮藏以及后续采取的防腐策略具有重要指导意义。
免培养法和纯培养法是研究微生物多样性的2种主要方法。基于高通量测序的免培养法具有数据量大、精确性高、速度快等优点[3],但不能获得纯培养物,无法验证基于大数据分析获得的推论。纯培养法指从一个细胞或一群相同的细胞经过培养繁殖得到纯培养物的方法。然而,现阶段大量微生物无法利用纯培养手段在实验室条件下进行培养,使纯培养法无法真实反映样品的微生物多样性[4]。目前,已经有一些关于酱卤肉制品微生物多样性的研究报道。谢萍等[5]采用高通量测序技术分析了散装酱卤鸭肉在25 ℃贮藏过程中微生物群落多样性,发现魏斯氏菌属(Weissella spp.)、葡萄球菌属(Staphylococcus spp.)是贮藏后期的优势细菌。李德红[6]利用平板培养法分离气调包装酱鸭食管中的腐败微生物,获得了特基拉芽孢杆菌(Bacillus tequilensis)、香坊肠杆菌(Enterobacter xiangfangensis)和肉芽肿克雷伯氏菌(Klebsiella granulomatis)等6株可能优势腐败细菌。王志琦[7]综合了上述2种方法,首先利用高通量测序技术解析了阚疃板鸡在贮藏期间的微生物群落特征,推测热杀索丝菌(Brochothrix thermasphacta)是阚疃板鸡在25 ℃贮藏下的主要腐败菌之一,并且通过纯培养手段分离获得了1株B. thermasphacta。因此,以免培养获得的数据为指导,设计合适的纯培养方法,是一种很有潜力的研究微生物多样性和获得腐败微生物菌种资源的方法。
本研究以不同区域酱卤鸭脖和鸭翅为研究对象,采用高通量测序技术对其贮藏期间的微生物群落变化进行全面解析,并利用纯培养和分子生物学技术对酱卤鸭肉中可能致腐的微生物进行分离与鉴定,以期阐明空间(贮藏位置)和时间(贮藏时间)对食品中微生物多样性的影响以及获得可能的关键致腐微生物,为了解酱卤肉食品的腐败机制以及后续抑制细菌生长,有效控制酱卤肉制品的腐败提供理论基础。
1 材料与方法
1.1 材料、试剂和培养基
散装酱卤鸭脖和鸭翅(500 g/样)购自江西省南昌市4个不同的酱卤肉制品专卖门店。
细菌DNA提取试剂盒,北京天根生化科技有限公司;AxyPrepDNA凝胶回收试剂盒,美国AXYGEN公司;Phanta Max Super-Fidelity DNA Polymerase,南京诺唯赞生物科技有限公司;琼脂糖,西班牙biowest公司。
MRS肉汤培养基、琼脂粉,北京索莱宝科技有限公司。
1.2 仪器与设备
SW-CJ系列净化工作台,上海新苗医疗器械制造有限公司;隔水式培养箱,上海一恒科学仪器有限公司;DYY-6C电泳仪,北京六一生物科技有限公司;NanoDrop2000超微量分光光度计,美国Thermo Fisher Scientific公司;GeneAmp® 9700型聚合酶链式反应仪,美国应用生物系统公司。
1.3 样品采集与保存
国内某知名食品公司生产的酱卤鸭脖和鸭翅于上午10点左右运抵至各专卖店,散装贮藏在4 ℃冷藏柜中。当天下午14点左右,购买酱卤鸭脖和鸭翅,用灭菌锡箔纸包好,1 h内运回实验室,打开锡箔纸,贮藏于江西师范大学国家淡水鱼加工技术研发专业中心产品间(图1中Locus_S),调节房间温度为20 ℃。购买地点和贮藏地点示意图见图1。分别贮藏2、4、6 d后进行取样,依次编号为:样品采集点A的鸭脖样品(Locus_A中的AN_2,AN_4,AN_6);样品采集点A的鸭翅样品(Locus_A中的AW_2,AW_4,AW_6);样品采集点B的鸭脖样品(Locus_B中的BN_2,BN_4,BN_6);样品采集点B的鸭翅样品(Locus_B中的BW_2,BW_4,BW_6);样品采集点C的鸭脖样品(Locus_C中的CN_2,CN_4,CN_6);样品采集点C的鸭翅样品(Locus_C中的CW_2,CW_4,CW_6);样品采集点D的鸭脖样品(Locus_D中的DN_2,DN_4,DN_6);样品采集点D的鸭翅样品(Locus_D中的DW_2,DW_4,DW_6)。
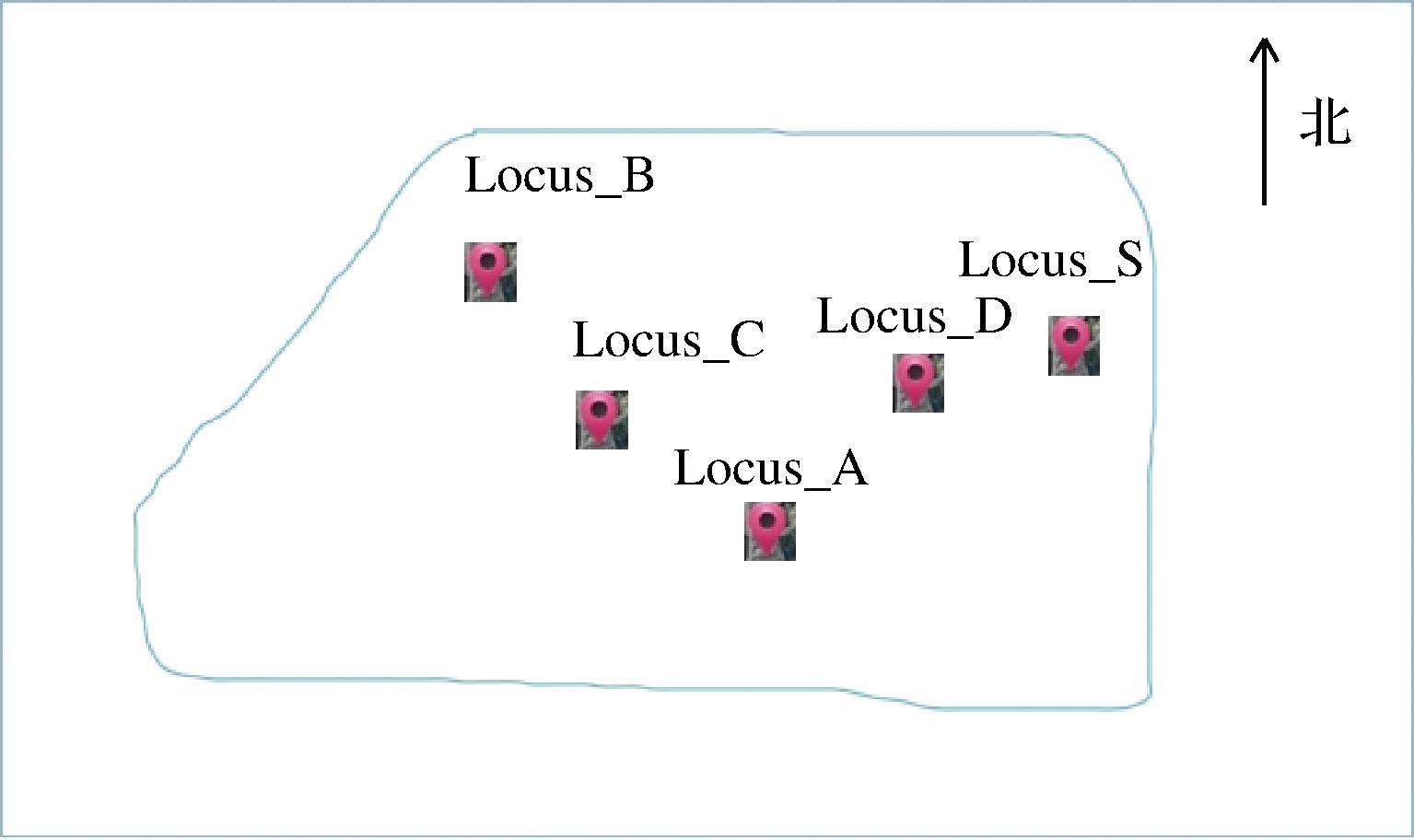
图1 样品采集点和贮藏点相对位置示意图
Fig.1 Schematic diagram of samples collection and storage locations
注:Locus_A~Locus_D为样品采集点,Locus_S为样品贮藏点。
1.4 细菌基因组DNA提取和PCR扩增
在超净工作台中,用无菌镊子从酱卤鸭脖的上中下3个不同位置分别取1 g鸭肉,置于1.5 mL灭菌离心管中,振荡混匀。按相同的方法收集酱卤鸭翅样品。根据天根生化细菌DNA试剂盒(DP302)说明书进行微生物群落总基因组DNA抽提。提取的基因组DNA使用1%(质量分数)的琼脂糖凝胶电泳检测质量,并使用NanoDrop 2000测定DNA浓度。以提取的DNA为模板,采用通用引物338F(5′-GTACTCCTACGGGAGGCAGCA-3′)和引物806R(5′-GTGGACTACHVGGGTWTCTAAT-3′)PCR扩增细菌菌群的16S rDNA V3~V4区基因序列。PCR扩增体系为:10×PCR缓冲液5 μL,dNTP(10 mmol/L)0.5 μL,DNA 10 ng,上下游引物(50 μmol/L)各0.5 μL,高保真Taq酶(5 U/μL)0.5 μL,加超纯水到50 μL。PCR扩增参数:95 ℃预变性5 min;95 ℃变性45 s,55 ℃退火50 s,72 ℃延伸45 s,30个循环;72 ℃再延伸10 min。PCR产物用AxyPrepDNA凝胶回收试剂盒纯化回收。
1.5 高通量测序与数据分析
纯化回收且质量合格的PCR产物送至上海美吉生物医药科技有限公司进行Illumina MiSeq高通量测序。使用Fastp软件对测序原始数据进行质控和过滤,使用Flash软件对Illumina测序得到的PE reads进行拼接,最终得到有效序列[8]。通过Uparse软件将序列按照相似性≥97%阈值进行操作分类单元(operational taxonomic unit,OTU)聚类[9],并进行嵌合体检查。采用主坐标分析法(principal co-ordinates analysis,PCoA)分析样品间细菌群落的β-多样性;利用Mothur软件进行样品内细菌群落α-多样性分析。采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析[10],基于138版本Silva(http://www.arb-silva.de)和13.5版本Greengenes(http://greengenes.secondgenome.com)数据库对细菌群落进行物种注释,并分别在属和种水平统计各样本的群落物种组成。
1.6 微生物分离、纯化与鉴定
为了分离出共有的微生物,以贮藏了4 d的所有酱卤鸭脖和鸭翅为分菌材料,在超净工作台中,用无菌镊子分别取0.5 g样品,置于1.5 mL灭菌离心管中。加500 μL灭菌生理盐水,漩涡振荡后,上清液用生理盐水稀释至10-4、10-5、10-6。取100 μL稀释液涂布于MRS固体平板,置于37 ℃培养箱中培养48 h。随机选取10个单菌落,在MRS液体培养基中静置培养12 h,-80 ℃保存甘油菌。用接种环蘸取少许保存的甘油菌,在MRS固体培养基中划线分离,37 ℃培养48 h。选取2株长势最好菌株,接种于MRS液体培养基,37 ℃静置培养48 h后,用普通光学显微镜观察细胞形态。将MRS液体培养基的菌液以8 000 r/min离心10 min后弃去培养基,根据DNA提取试剂盒说明书进行细菌总基因组DNA提取。使用细菌通用引物27F (5′-AACTGAGTTTGATCCTGGCTC-3′)和1492R (5′-TACGGTTACCTTGTTACGAC-TT-3′)扩增16S rDNA基因,凝胶电泳分析其PCR产物。PCR扩增条件为:95 ℃ 5 min,94 ℃ 30 s,在55 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃ 10 min。PCR产物经1%琼脂糖凝胶电泳检测合格后,送至上海生工生物工程股份有限公司进行双向测序。对测序结果进行序列拼接校准后与NCBI上GenBank数据库中的已知序列进行同源性比对分析。参考曾金秀等[11]的方法,下载同源性较高的16S rDNA的基因序列作为参比对象,使用MEGA 7.0软件中的邻接法构建系统发育树。最后,结合菌落和细胞形态以及分子生物学分析结果,确定菌株信息。
2 结果与分析
2.1 测序数据分析
高通量测序获得酱卤鸭脖和鸭翅样品不同贮藏时间的原始序列,经拼接、过滤后,测序数据统计见表1。24个样品共有989 839条有效序列,不同贮藏时间样品有效序列数均超过30 000,平均长度为428 bp。稀释曲线是以样本中随机抽取的序列数为横坐标,以对应的OTU数量为纵坐标,常用来反映测序数据是否科学[12]。酱卤鸭脖和鸭翅样品测序数据的稀释曲线表明,当测序量超过10 000 bp时,稀释曲线上升缓慢,达到30 000 bp时,曲线已经趋于平缓(图2),表明本次测序的深度已足够,说明取样合理,测序深度已基本覆盖样品中所有微生物,可以反映样品细菌多样性。
表1 测序数据统计
Table 1 Statistics of the sequencing data
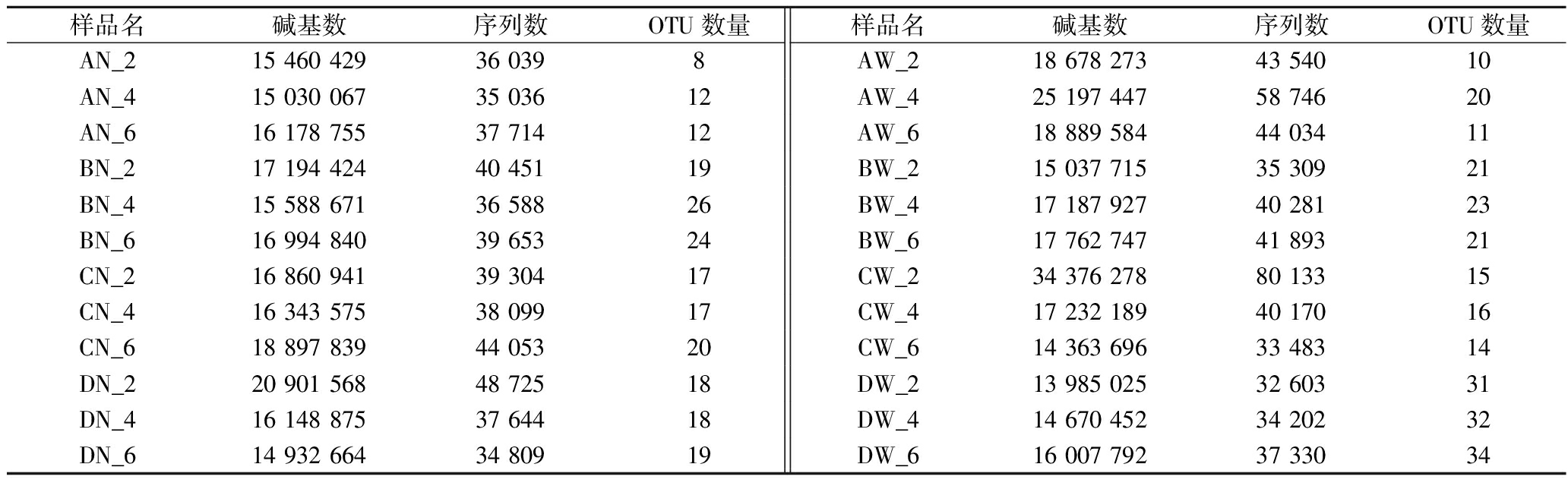
样品名碱基数序列数OTU数量样品名碱基数序列数OTU数量AN_215 460 42936 0398AW_218 678 27343 54010AN_415 030 06735 03612AW_425 197 44758 74620AN_616 178 75537 71412AW_618 889 58444 03411BN_217 194 42440 45119BW_215 037 71535 30921BN_415 588 67136 58826BW_417 187 92740 28123BN_616 994 84039 65324BW_617 762 74741 89321CN_216 860 94139 30417CW_234 376 27880 13315CN_416 343 57538 09917CW_417 232 18940 17016CN_618 897 83944 05320CW_614 363 69633 48314DN_220 901 56848 72518DW_213 985 02532 60331DN_416 148 87537 64418DW_414 670 45234 20232DN_614 932 66434 80919DW_616 007 79237 33034
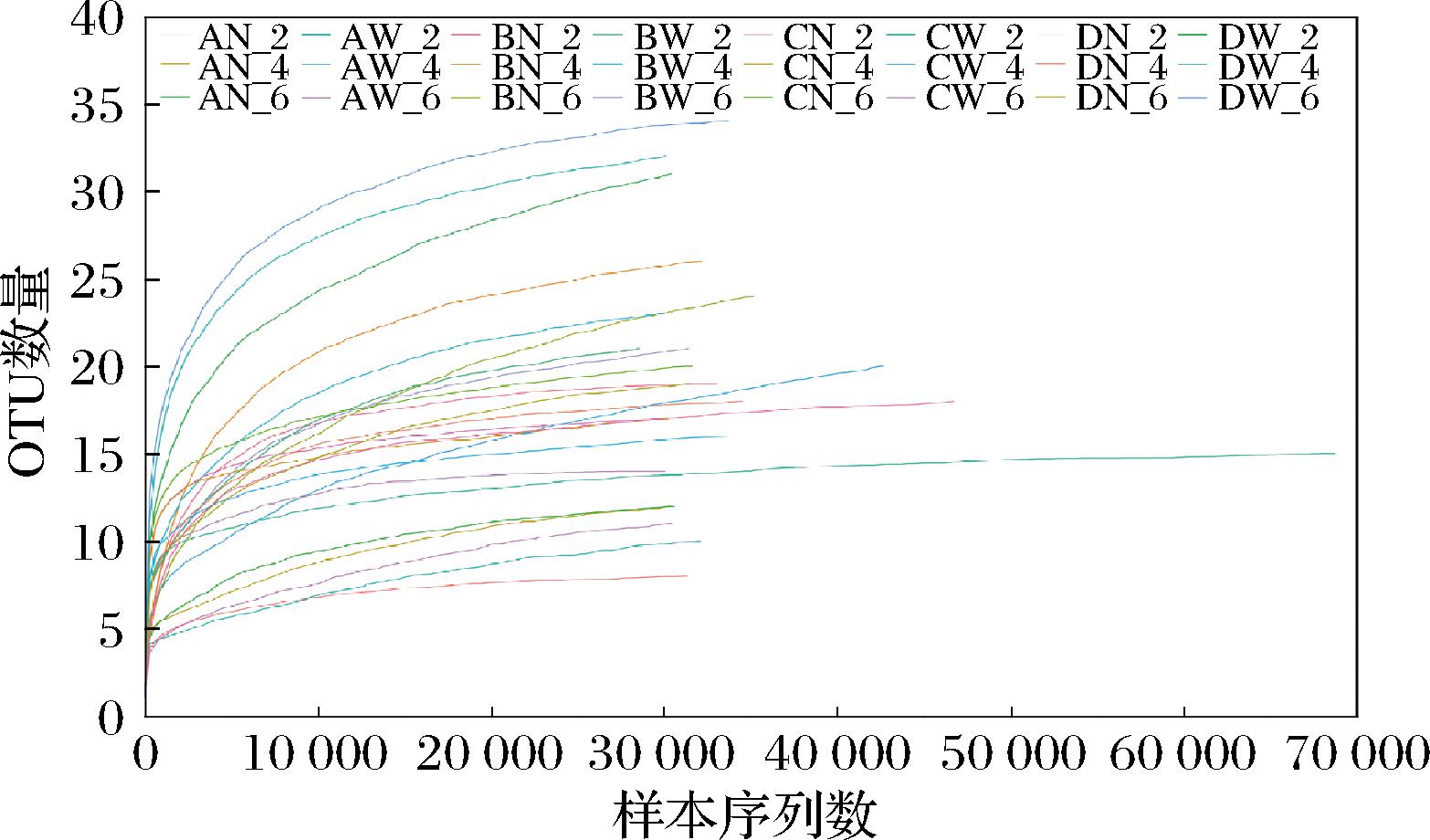
图2 酱卤鸭脖和鸭翅样品稀释曲线
Fig.2 Rarefaction curves of sauced duck neck and wing samples
2.2 β-多样性分析
β-多样性分析通过对不同微生物群落间的物种多样性进行组间比较分析,探索不同分组样本间群落组成的相似性或差异性[13];样品在PCoA图中距离越近,说明样品间的细菌群落组成越相似[14]。如图3所示,第1主成分和第2主成分的累计方差贡献率为74.18%,说明两个主成分可以叠加解释74.18%。整体来看,所有样品按地理位置聚集,其中Locus_B和Locus_C的样品分别聚类在不同的区域;尽管Locus_A和Locus_D的样品聚类在一个大的区域,但Locus_D样品聚集得更紧密,能和Locus_A样品分开,这些结果说明来源于不同的地理位置对样本的聚类具有决定性的影响。Locus_A和Locus_D样品聚集在一起的原因可做如下推测:在采集点贮藏的4 h中,Locus_A和Locus_D样品中的微生物多样性和物种丰度均较低,未形成稳定的群落结构,易受外界环境影响,当样品转移至Locus_S,新环境中的微生物逐渐占据优势,造成Locus_A和Locus_D样品中群落结构具有相似性。另外,对Locus_A样品来说,来源于酱卤鸭脖的样本(n=3)和酱卤鸭翅的样本(n=3)各有小范围的聚集,这说明酱卤肉制品的种类是影响聚类的第2个因素,这个现象在Locus_B、Locus_C和Locus_D中也可以体现。此外,对于同一采样点同一种酱卤肉制品的不同贮藏时间样品在图中有一定的差距,该现象以Locus_A和Locus_C来源的样品最明显,说明贮藏时间也是影响聚类的一种因素。综上所述,地理位置是影响酱卤鸭脖和鸭翅中腐败微生物聚类的主要因素,酱卤肉制品的种类和贮藏时间也是影响聚类的因素。黄郑朝[15]通过对不同区域传统发酵香肠细菌多样性研究发现,不同地理位置发酵香肠细菌多样性和群落结构之间具有明显的差异性,这种结果的原因可能是地理位置上的差异。这和我们的研究结果相似,说明地理位置会显著影响微生物的群落结构。
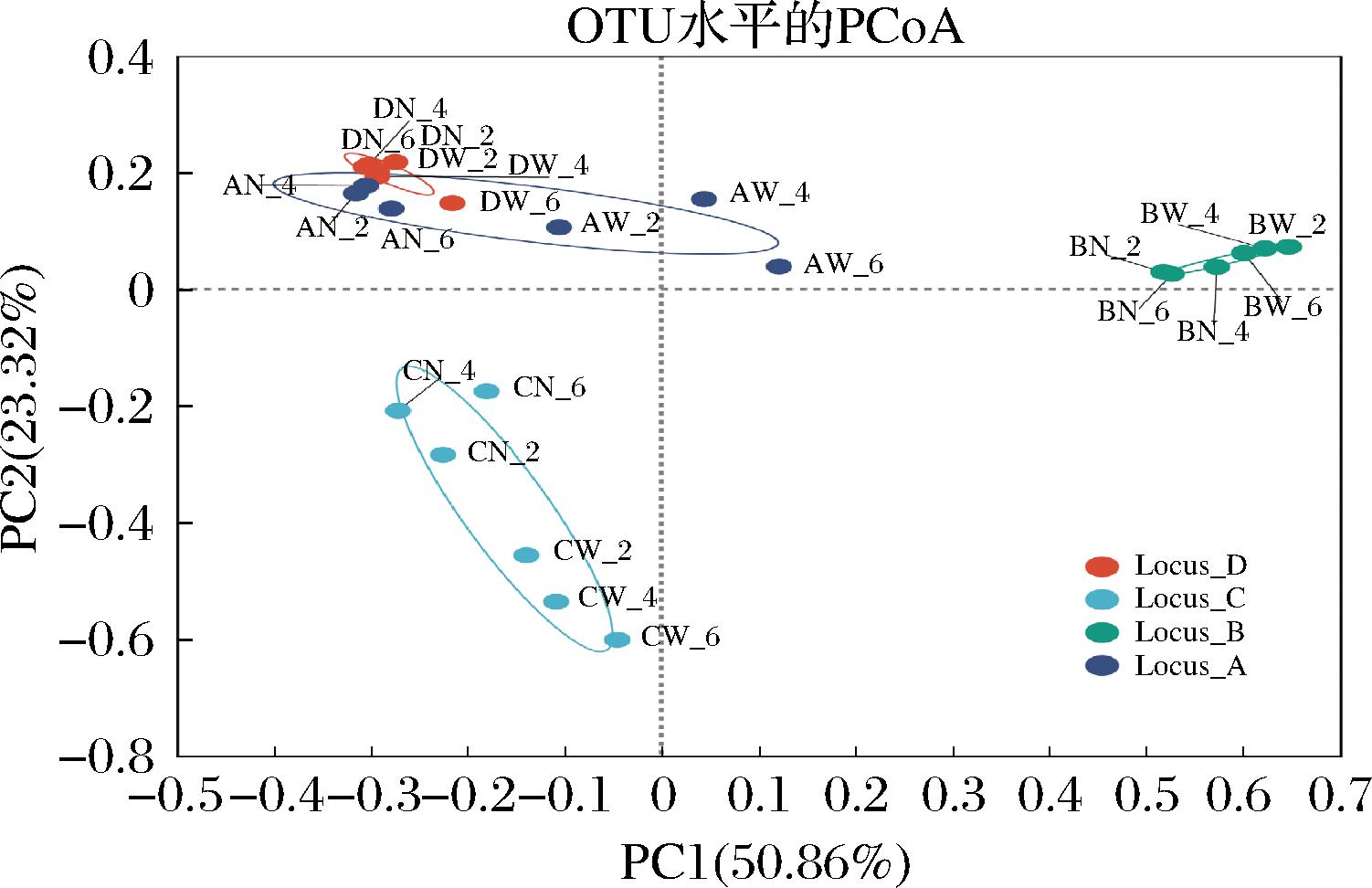
图3 酱卤鸭脖和鸭翅样本间PcoA结果
Fig.3 PCoA results in sauced duck neck and duck wing samples
2.3 α-多样性分析
α-多样性分析主要用于反映物种丰富度、均匀度以及多样性[16]。基于97%相似性水平下的OTU信息,利用Chao指数、Ace指数、Shannon指数及Invsimpson指数对样品的微生物丰富度和多样性进行评估。Chao指数、Ace指数越大,表明群落的物种丰富度越高;Shannon指数、Invsimpson指数越大,表明群落中物种多样性越高。从地理位置来看,Chao指数和Ace指数从大到小依次为Locus_D、Locus_B、Locus_C和Locus_A(图4-a、图4-b);Shannon指数和Invsimpson指数中Locus_B、Locus_C最大,Locus_D最小(图4-c、图4-d)。从酱卤肉制种类类来看,Locus_D中的鸭脖样品和鸭翅样品丰富度差异最明显(图4-a、图4-b);Locus_C中鸭脖样品多样性高于鸭翅样品,而其他3个点样品的情况刚好相反(图4-c、图4-d)。从取样时间上看,第2天的丰富度最低,第4天或第6天丰富度最高(图4-a、图4-b);大部分样品第6天的多样性最高,少数样品如BN第2天多样性最高(图4-c、图4-d)。这些结果再次说明地理位置、酱卤肉制种类和贮藏时间等因素同时影响着酱卤鸭脖和鸭翅的微生物多样性。
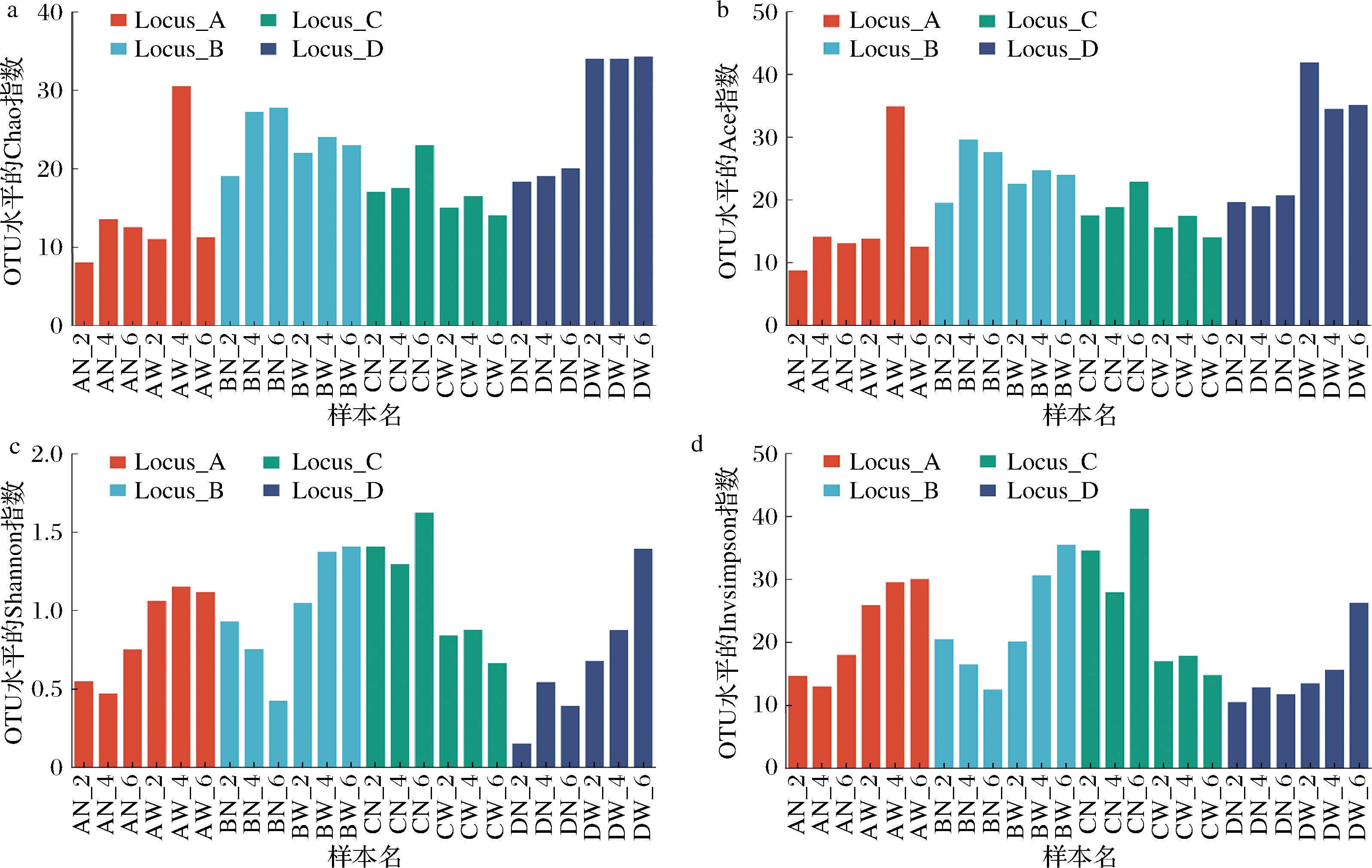
a-Chao指数;b-Ace指数;c-Shannon指数;d-Invsimpson指数
图4 酱卤鸭脖和鸭翅样品的α-多样性指数
Fig.4 α- Diversity indexes of sauced duck neck and duck wing samples
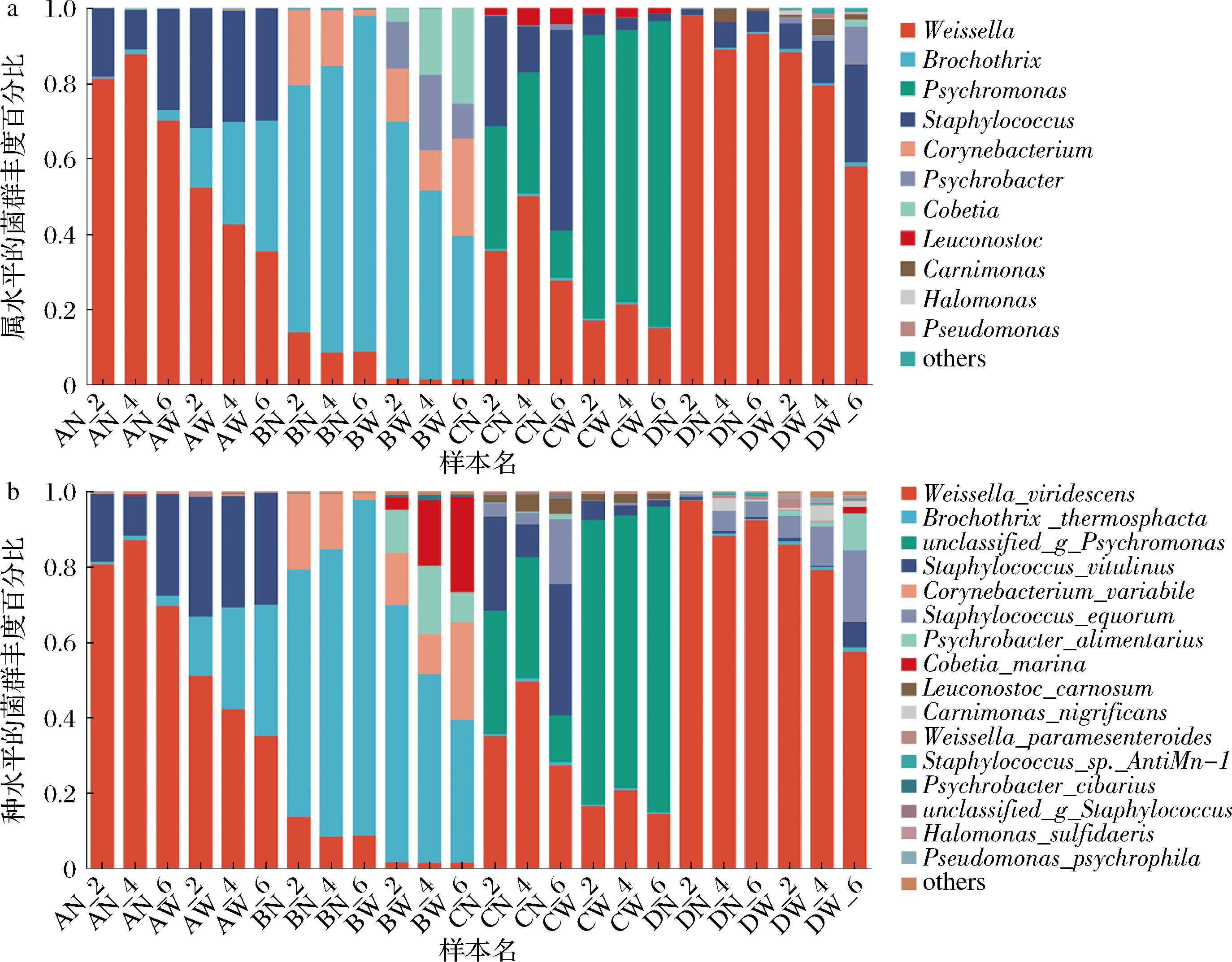
a-属水平;b-种水平
图5 酱卤鸭脖和鸭翅细菌属水平和种水平相对丰度
Fig.5 Relative abundance of genus levels and species levels in sauced duck neck and duck wing samples
2.4 细菌菌群组成分析
为进一步研究酱卤鸭脖和鸭翅贮藏过程中微生物多样性和动态演替过程,在属水平和种水平进行细菌群落的结构和差异分析。酱卤鸭脖和鸭翅在贮藏2 d后未发生明显变化,颜色鲜亮;贮藏4 d后样品有轻微的腐败,颜色变暗;样品贮藏6 d后样品有明显的腐败,颜色进一步变暗。如图5所示,Locus_A样品中,丰度排名第一的是Weissella spp.,其次为葡萄球菌属Staphylococcus spp.和索丝菌属(Brochothrix spp.),其总群落占有率分别为35.36%~81.29%,10.53%~29.84%和0.55%~34.73%,对应的优势菌种分别为绿色魏斯氏菌(Weissella viridescens)、小牛葡萄球菌(Staphylococcus vitulinus)和B.thermosphacta。与Locus_A类似,Locus_D样品中,魏斯氏菌属占绝对优势(57.96%~97.98%),但其优势物种的多样性比Locus_A样品高,除W. viridescens,S. vitulinus和B. thermosphacta外,马胃葡萄球菌(Staphylococcus equorum)、消化嗜冷杆菌(Psychrobacter alimentarius)、Carnimonas_nigrificans也被检测到。如前所述,Locus_A和Locus_D样品聚集在一起(图3),这可能归因于Locus_A和Locus_D相似的细菌菌群结构,且这些优势微生物可能主要来源于贮藏点S。李海燕等[17]通过研究不同生境甘草微生物群落特征发现,聚类在一起的甘草样品在属水平上主要的微生物相同,即主要微生物群落导致不同生境的甘草样品聚类在一起。这和我们的研究结果一致,因此我们推断造成Locus_A和Locus_D聚类在一起的原因是相似的细菌群落结构。对于Locus_B和Locus_C的酱卤鸭脖和鸭翅样品来说,贮藏不同时期具有更多的微生物多样性,其中Locus_B样品丰度最高的属是Brochothrix spp.,对应的物种是B. thermosphacta;Locus_C样品丰度最高的属是Weissella spp.、Psychrobacter spp.和Staphylococcus spp.,对应的物种是W. viridescens、P. alimentarius和S. vitulinus;另外,Corynebacterium variabile、unclassified_g_Psychromona、Cobetia marina、Leuconostoc carnosum等也是丰度较高的微生物,且C.variabile和unclassified_g_Psychromona分别主要存在于Locus_B和Locus_C样品中。从贮藏时间来看,主要优势微生物的丰度变化没有固定规律。例如,随贮藏时间增加,W. viridescens在AN样品中先增加后减少,而在AW样品中一直减少;B. thermosphacta在AW样品中一直增加,而在BW样品中一直减少。总体看来,酱卤鸭脖和鸭翅在贮藏过程中菌群变化较为复杂,主要的影响因素为样品贮藏的地理位置,这与王瑾瑜[18]报道的不同地区的酸面团微生物群落组成存在明显差异的结果类似。因此,需要对贮藏环境等严格把关,最大程度避免微生物污染。
2.5 共有和特有物种分析
为了探究不同贮藏期间酱卤鸭脖和鸭翅中的核心微生物,对所有样品的共有物种和特有物种进行分析。如图6所示,Locus_A、Locus_B、Locus_C和Locus_D有14个共有物种,分别有1、6、0、14个特有物种。共有物种可能是导致酱卤鸭脖和鸭翅腐败的关键物种,包括W. viridescens、S. vitulinus、B. thermosphacta、W.paramesenteroides、S.equorum、C.marina、P.alimentarius、P.cibarius、C.variabile、Pseudomonas psychrophila、unclassified_g_Psychrobacter、Rhodococcus erythropolis、unclassified_g_Psychromonas、unclassified_g_Weissella。丁珊珊等[19]研究发现W. viridescens能使真空包装低温火腿发生快速腐败;ILLIKOUD等[20]研究证明B. thermosphacta是导致肉类变质腐败的主要细菌之一;滕安国等[21]发现S. vitulinus存在于香肠的不同储藏时期。以上研究结果表明W. viridescens、S. vitulinus和B. thermosphacta是导致肉制品腐败的关键微生物。本次实验在室温(20 ℃)下贮藏,腐败后产品出现表面发黏、颜色变暗的现象,结合我们的发现和前人的研究,因此我们推测这些微生物也是酱卤鸭脖和鸭翅腐败的关键微生物。
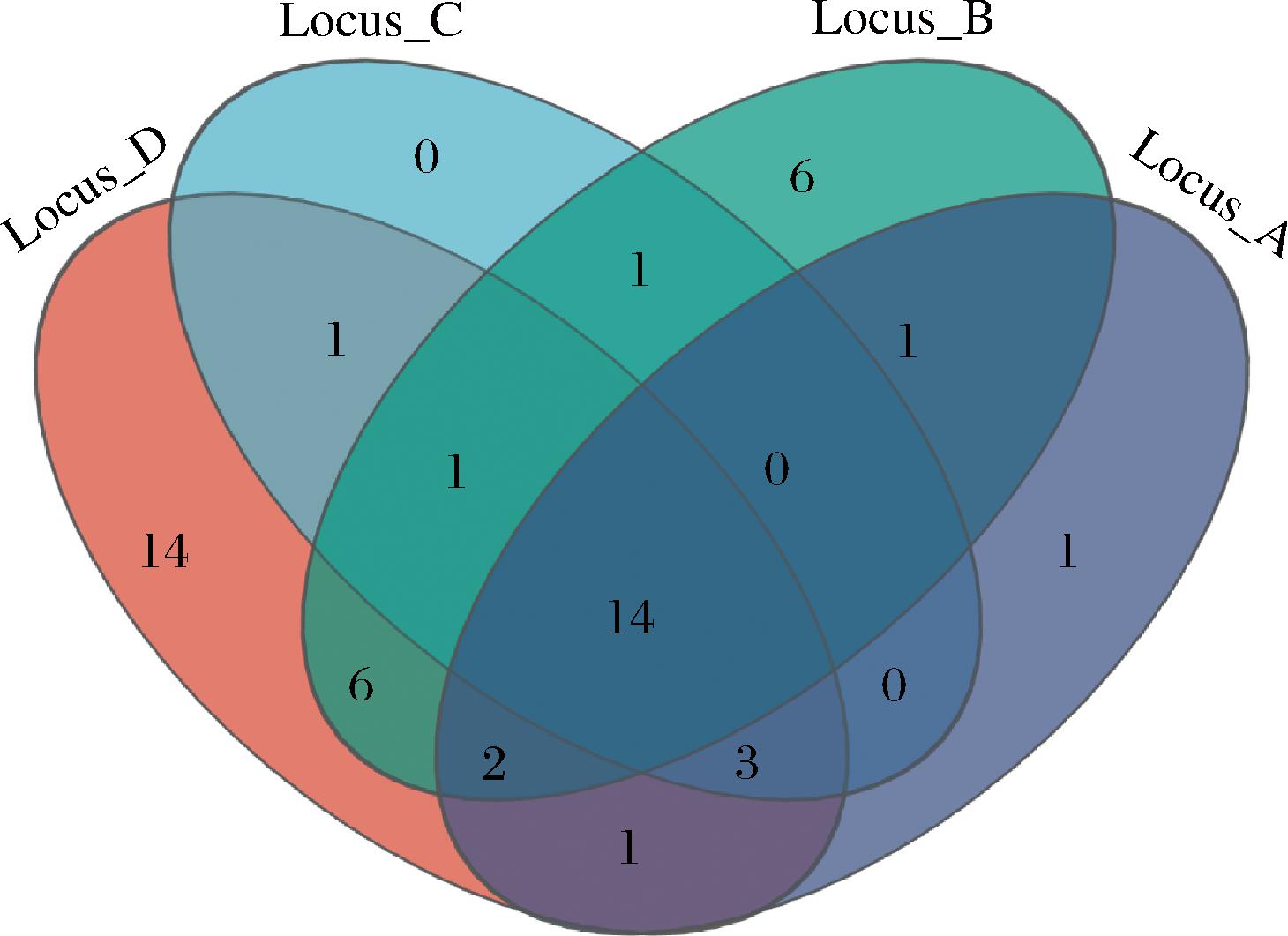
图6 酱卤鸭脖和鸭翅样本OTU数目Venn图
Fig.6 Venn plot of the number of OTU of sauced duck neck and wing samples
2.6 可能致腐微生物分离与鉴定
如前所述,W. viridescens、S. vitulinus和B. thermosphacta等可能是肉制品的关键致腐微生物。为了后续证实该推测以及阐明这些微生物的致腐机制,我们进行了微生物分离与鉴定研究。如图7-a所示,MRS固体平板上有圆形,淡黄色,表面光滑,边缘整齐,无褶皱的直径大约在1 mm左右的菌落。挑选2个长势良好的菌株,命名为6-M和6-N。革兰氏染色结合显微观察表明,6-M和6-N为革兰氏阳性菌,细胞呈不规则的短杆状,两端呈圆形或稍细,成对或短链排列(图7-b)。分子生物学鉴定分析显示,6-M和6-N的16S RNA序列同源性为100%,其与NCBI数据库中的W. viridescens均具有>99%的同源性,说明6-M和6-N是同一个菌株,统一命名为6-M。系统发育树分析表明,6-M与W.viridescens聚在一起,形成一个亚分支(图7-c)。综合6-M的菌落和细胞形态、分子生物学分析以及上述微生物多样性分析揭示的结果,证明6-M为W. viridescens,可用于后续致腐实验。小牛葡萄球菌和热杀索丝等微生物未分离成功,可能是MRS培养基不合适,后续可改变培养基配方进行分离。
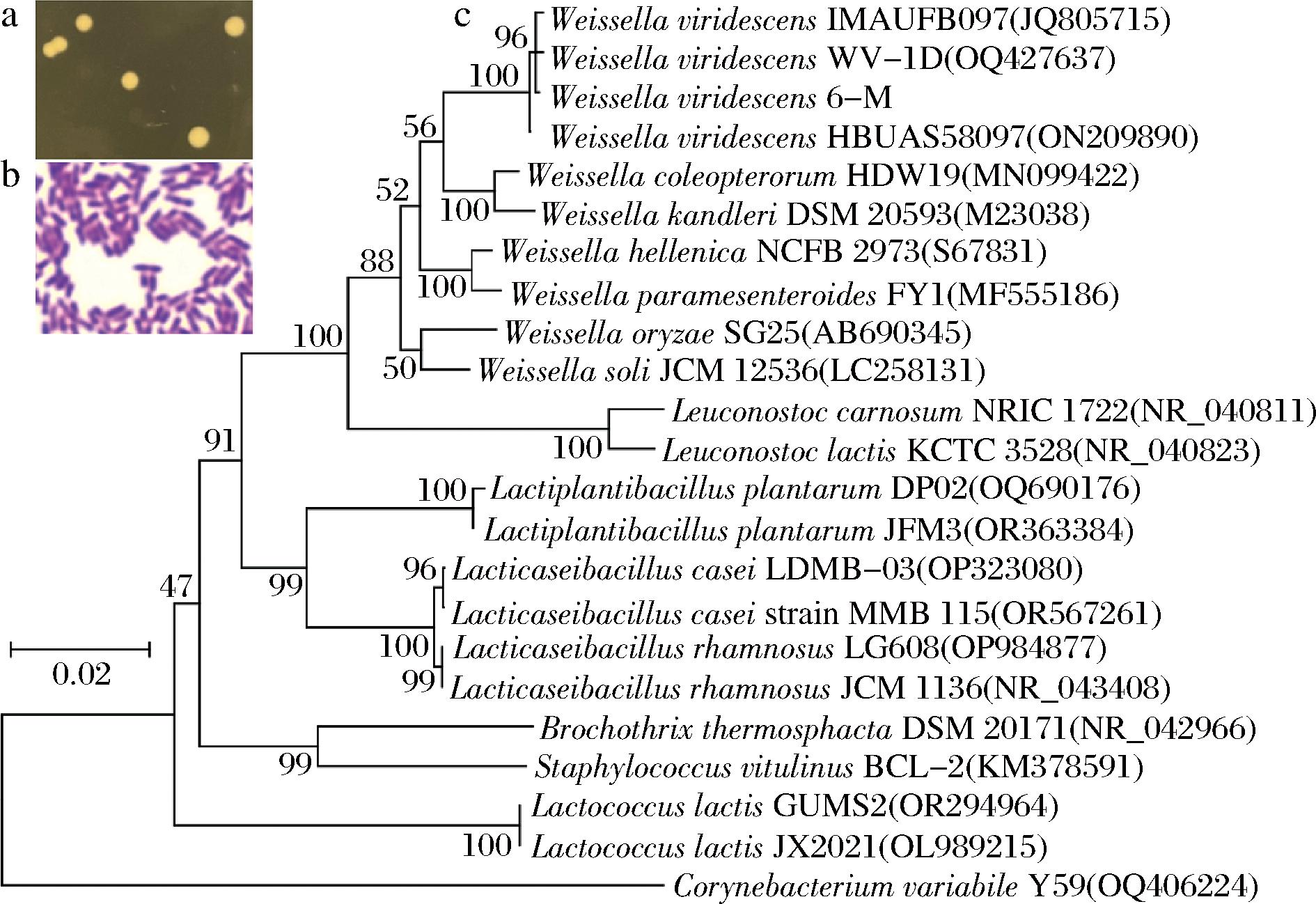
a-菌落形态;b-细胞形态;c-系统发育树
图7 菌落形态、细胞形态和系统发育树
Fig.7 The colonial morphology, cell morphology, and phylogenetic tree
3 结论
本研究利用高通量测序技术研究了散装酱卤鸭脖和鸭翅在贮藏过程中的微生物多样性变化规律,结果发现地理位置、酱卤肉制品种类和贮藏时间共同影响微生物的群落结构,其中地理位置是第一影响因素;W. viridescens、S. vitulinus和B. thermosphacta可能是酱卤鸭脖和鸭翅的关键致腐微生物。然后,通过纯培养和分子生物学手段从腐败酱卤鸭脖和鸭翅中分离并鉴定到1株W. viridescens 6-M,进一步证明了W. viridescens是能被分离,可被用于后续酱卤鸭脖和鸭翅等肉制品腐败研究的关键菌株。本研究为阐明酱卤鸭脖和鸭翅等肉制品的腐败机制提供了理论依据以及关键菌株资源。同时从空间(地理位置)和时间(贮藏时间)两个维度研究食品中的微生物多样性以及基于免培养指导微生物分离纯化的研究思路可扩展至食品与微生物交叉学科领域。因此,本研究具有一定学术理论价值。
[1] CAI Z D, RUAN Y F, HE J, et al.Effects of microbial fermentation on the flavor of cured duck legs[J].Poultry Science, 2020, 99(9):4642-4652.
[2] 谢萍, 徐明生, 尹忠平, 等.散装酱卤鸭肉制品中特定腐败菌的确定[J].食品安全质量检测学报, 2016, 7(7):2895-2903.XIE P, XU M S, YIN Z P, et al Determination of special spoilage organisms in bulk sauced duck products[J].Journal of Food Safety &Quality, 2016, 7(7):2895-2903.
[3] 陈丽, 王敬敬, 贺明君, 等.基于高通量测序分析玉米浸泡液细菌菌群结构及多样性[J].食品研究与开发, 2023, 44(17):178-181;217.CHEN L, WANG J J, HE M J, et al.Bacterial community structure and diversity of corn soak solution based on high-throughput sequencing[J].Food Research and Development, 2023, 44(17):178-181;217.
[4] 彭玺, 冯凯, 厉舒祯, 等.宏基因组学技术与微生物群落多样性分析方法[J].科技导报, 2022, 40(3):99-111.PENG X, FENG K, LI S Z, et al.Analytical methods for metagenomic technology and microbial community diversity[J].Science &Technology Review, 2022, 40(3):99-111.
[5] 谢萍, 徐明生, 尹忠平, 等.MiSeq测序研究散装酱卤鸭肉贮藏期间微生物群落多样性[J].现代食品科技, 2015, 31(11):120-126;106.XIE P, XU M S, YIN Z P, et al.MiSeq sequencing to study the diversity of microflora in bulk-marinated duck meat during storage[J].Modern Food Science and Technology, 2015, 31(11):120-126;106.
[6] 李德红. 气调包装酱鸭食管优势腐败菌分离鉴定及抑菌研究[D].哈尔滨:哈尔滨商业大学, 2020.LI D H.Isolation, identification and bacteriostasis of dominant spoilage bacteria in modified atmosphere packaging sauced duck’s esophagus[D].Harbin:Harbin University of Commerce, 2020.
[7] 王志琦. 阚疃板鸡菌相分析及保鲜技术研究[D].合肥:合肥工业大学, 2022.WANG Z Q.Study on microflora analysis and fresh-keeping technology of chicken[D].Hefei:Hefei University of Technology, 2022.
[8] 黄志远, 毛传亮, 万众, 等.储藏温度对石竹笋内生细菌多样性的影响[J].竹子学报, 2023, 42(2):48-55.HUANG Z Y, MAO C L, WAN Z, et al.Effect of storage temperature on the diversity of endophytic bacteria in Phyllostachys nuda shoots[J].Journal of Bamboo Research, 2023, 42(2):48-55.
[9] EDGAR R C, HAAS B J, CLEMENTE J C, et al.Uchime improves sensitivity and speed of chimera detection[J].Bioinformatics, 2011, 27(16):2194-2200.[10] ANUKAM K.Effects of ampicillin on the gut microbiome of an adult male as determined by 16S rRNA V4 metagenomics sequencing and greengenes bioinformatics suite[J].Journal of Advances in Microbiology, 2018, 7(4):1-18.
[11] 曾金秀, 尹红梅, 张权, 等.霉鱼源清酒乳杆菌的分离筛选及其在霉鱼发酵中的应用[J].食品与发酵工业, 2024, 50(6):56-64.ZENG J X, YIN H M, ZHANG Q, et al.Isolation and identification of Lactobacillus sakei of traditional fermented bighead carp and its application in inoculated fermentation[J].Food and Fermentation Industries, 2024, 50(6):56-64.
[12] 杨婧, 李娜娜, 刘子恒, 等.高粱单宁对清香型大曲白酒发酵过程中真菌群落结构的影响[J].食品科技, 2022, 47(7):1-9.YANG J, LI N N, LIU Z H, et al.Effect of sorghum tannin on fungi community structure during the brewing of Fen-flavor Daqu liquor[J].Food Science and Technology, 2022, 47(7):1-9.
[13] 肖岚, 何莲, 安潘宇, 等.萝卜丝入坛发酵对安岳坛子肉发酵过程中微生物演替变化的影响[J].食品工业科技, 2020, 41(20):72-78.XIAO L, HE L, AN P Y, et al.Effects of fermentation of shredded radish in jar on bacterial diversity during the fermentation process of Anyue ceramic-pot sealed meat[J].Science and Technology of Food Industry, 2020, 41(20):72-78.
[14] YAO Y, ZHANG J M, DING X L.Partial beta-amylolysis retards starch retrogradation in rice products[J].Journal of Agricultural and Food Chemistry, 2003, 51(14):4066-4071.
[15] 黄郑朝. 中国不同区域传统发酵香肠细菌多样性及发酵用菌株筛选研究[D].郑州:河南农业大学, 2020.HUANG Z C.Study on bacterial diversity of traditional fermented sausages in different regions of China and screening of fermentation strains[D].Zhengzhou:Henan Agricultural University, 2020.
[16] 庞春霞, 李艺, 虞任莹, 等.基于Illumina Miseq技术比较不同地区传统发酵大豆制品细菌多样性[J].食品工业科技, 2022, 43(8):133-140.PANG C X, LI Y, YU R Y, et al.Comparison of bacterial diversity of traditional fermented soybean products from different regions based on Illumina Miseq technology [J].Science and Technology of Food Industry, 2022, 43(8):133-140.
[17] 李海燕, 白雯静, 尹盼盼, 等.基于16S rRNA基因的不同基原、不同产地甘草微生物群落特征分析[J].食品安全质量检测学报, 2023, 9(19):225-234.LI H Y, BAI W J, YIN P P, et al.Microbial community characteristics analysis of licorice from different provenances and different habitats based on 16S rRNA gene[J].Journal of Food Safety and Quality, 2023, 9(19):225-234.
[18] 王瑾瑜. 基于高通量测序和机器学习方法解析酸面团微生物多样性[D].太原:山西大学, 2023.WANG J Y.Analysis of microbial diversity of sour dough based on high-throughput sequencing and machine learning method[D].Taiyuan:Shanxi University, 2023.
[19] 丁珊珊, 蔡淑珍, 韩衍青, 等.绿色魏斯氏菌对真空包装低温火腿的致腐效应研究[J].南京农业大学学报, 2020, 43(1):164-171.DING S S, CAI S Z, HAN Y Q, et al.Study on the spoilage effect of Weissella viridescens on vacuum-packed low-temperature ham[J].Journal of Nanjing Agricultural University, 2020, 43(1):164-171.
[20] ILLIKOUD N, ROSSERO A, CHAUVET R,et al.Genotypic and phenotypic characterization of the food spoilage bacterium Brochothrix thermosphacta[J].Food Microbiology, 2019, 81:22-31.
[21] 滕安国, 张芹, 蒋怡, 等.不同储藏时期香肠菌群结构的变化及优势腐败菌研究[J].现代食品科技, 2019, 35(2):40-46.TENG A G, ZHANG Q, JIANG Y, et al.Studyies on the microbial profiles and dominant spoilage bacteria in sausage during different storage periods [J].Modern Food Science and Technology, 2019, 35(2):40-46.