中国白酒以粮谷为主要原料,以大曲、小曲、麸曲、酶制剂及酵母等为糖化发酵剂,经蒸煮、糖化、发酵、蒸馏、陈酿、勾调而成的蒸馏酒[1]。长期以来,泥窖被用作为发酵浓香型白酒的反应器,窖泥在长期发酵过程中形成了复杂的生态系统,构建了特殊的微生态结构[2]。酶是具有生物催化能力的高分子蛋白质活性物质,是由窖泥微生物代谢过程中产生,在窖泥氮、磷等循环中起关键作用,同时还参与有机物的分解转化[3]。窖泥中脲酶与蛋白酶是参与氮循环的关键酶,在有机氮转化为无机氮的过程中发挥极其重要的作用[4],其中脲酶能够将有机氮水解成氨[5],蛋白酶将蛋白质与肽类物质分解为氨基酸,提供微生物能够利用的无机氮,因此蛋白酶和脲酶的活性直接影响着窖泥中氮转化的强度和供氮能力[6]。过氧化氢酶、脱氢酶能够促进窖泥中木质素的降解、蔗糖的水解、纤维素的分解、腐殖质的形成,促进窖泥微生物的物质循环,磷酸酶参与有机磷酸盐的水解[7]。窖泥微生物对酒质的影响是通过酶促反应实现的,已有研究表明酶活性的高低可以反映土壤微生物活性和功能的变化,也可以作为评价窖泥质量的重要指标[8]。因此研究不同位置窖泥酶活力及其细菌群落结构的相关性对于探究窖泥中氮素转化具有重要意义。
酶活力可以反映窖泥中氮代谢的强度,其活性大小不仅受窖泥品质、微生物活性的影响,还与窖泥的空间位置密切相关。吴树坤等[9]分析不同品质浓香型白酒窖泥与微生物群落的相关性,结果表明不同品质窖泥的酶活与微生物群落结构呈一定的规律性。微生物群落与酶活力是影响窖泥物质循环的重要因素,WANG等[10]发现氮是调控微生物群落与酶活力的重要因素,氮素转化也是窖泥中重要的代谢过程。张淼等[11]针对黑土区农田土壤的研究结果表明,除了理化因子,地理距离对土壤氮循环中关键过程微生物分布产生重要影响。目前关于窖泥的研究多是微生物结构解析,而研究不同位置窖泥细菌群落与酶活力的相关性、参与氮循环关键酶的差异性报道较少。
本研究以四川浓香型酒厂发酵正常窖池不同位置的窖泥作为样品,通过高通量测序技术获得窖泥微生物信息并测得窖泥理化数据及酶活力,使用聚类分析、冗余分析、相关性分析研究窖泥理化性质、细菌群落、氮循环中关键酶的差异性,试图揭示不同位置窖泥微生物对浓香型白酒发酵的影响,为进一步提高白酒风味品质提供方向。
1 材料与方法
1.1 实验材料
1.1.1 样品采集
样品取自四川某浓香型白酒窖池的窖泥,取样点如图1所示,从距离窖沿30 cm处的四面窖壁中心点取样混合,作为窖壁上层窖泥(wall top mud,WT);从距离窖底120 cm处的四面窖壁中心点取样混合,作为窖壁中层窖泥(wall middle,WM);窖底采用五点取样法,以窖底平面,在窖底确定对角线的中点作为中心抽样点,再在对角线上选择4个与中心样点距离相等的点作为样点,作为窖底窖泥(bottom mud,BM)。每个样品取3口窖池,分别命名为WT1~WT3,WM1~WM3,BM1~BM3,将每个窖泥样品取出混匀后分成2份装入无菌袋并冷藏,快速送回实验室,贮存在4 ℃冰箱和-80 ℃冰箱,分别用于检测理化指标和窖泥DNA的提取。
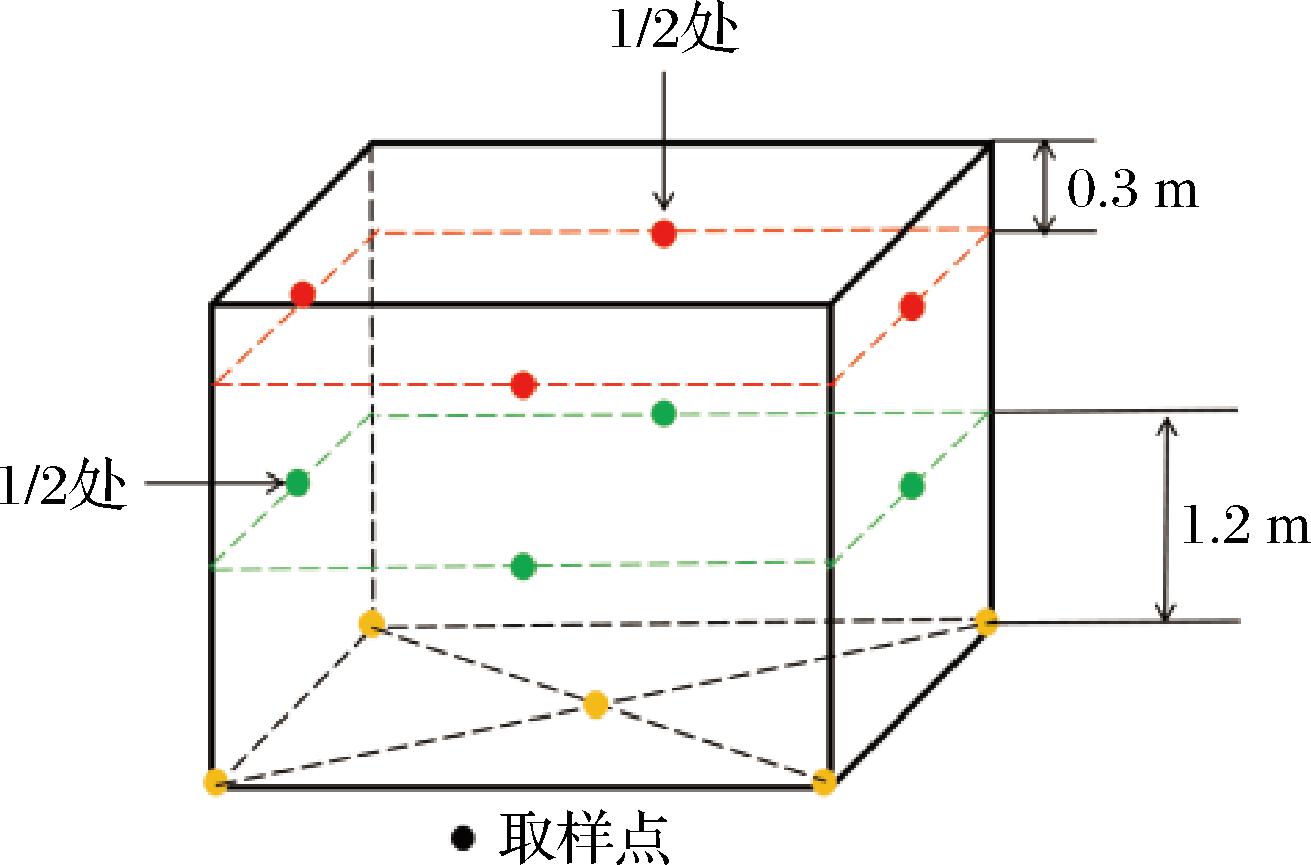
图1 窖泥的取样位置
Fig.1 Collection sample location of pit mud
1.1.2 试剂与仪器
E.Z.N.A.®soil试剂盒,上海晶诺生物科技有限公司;碘化汞、碘化钾、氢氧化钠、酒石酸钾钠、氯化铵、氟化铵、盐酸、钼酸铵、磷酸二氢钾、氯化亚锡、柠檬酸、苯酚钠、次氯酸钠、福林试剂、碳酸钠、三氯乙酸、磷酸苯二钠、硫酸、高锰酸钾、氯化三苯基四氮唑、连二亚硫酸钠,以上均为分析纯,成都市科隆化学品有限公司;UV-2400紫外分光光度计,上海尤尼柯仪器有限公司;PCR仪、电泳仪,美国Bio-Rad公司;高速冷冻离心机,德国Hettich公司;NanoDrop2000 DNA含量测定仪,美国Illumina公司。
1.2 实验方法
1.2.1 窖泥理化与酶活检测方法
pH值、酸性磷酸酶活力(acid phosphatase,ACP)、脲酶活力(urease,N_U)、过氧化氢酶(catalase,CAT)、蛋白酶(protease,D_U)、脱氢酶(dehydrogenase,T_U)用新鲜的窖泥测定,铵态氮含量(ammonium nitrogen content,A_N)、有效磷含量(available phosphorus content,P)用风干的窖泥测定,纳氏试剂比色法测定铵态氮含量;氟化铵-盐酸比色法测定有效磷含量[12];靛酚比色法测定脲酶活性;茚三酮比色法测定蛋白酶活性;苯磷酸二钠比色法测定酸性磷酸酶活性;容量法测定过氧化氢酶活性;氯化三苯基四氮唑法测定脱氢酶活力,5种酶活的具体检测步骤参考吴树坤等[9]。
1.2.2 窖泥总DNA的提取
根据E.Z.N.A.®soil试剂盒说明书进行总DNA提取,DNA浓度和纯度利用NanoDrop2000进行检测,同时利用质量分数为1%的琼脂糖凝胶电泳检测DNA提取质量。
1.2.3 PCR扩增
用338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)引物对V3~V4可变区进行PCR扩增,扩增程序为:95 ℃预变性3 min,27个循环(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s),最后72 ℃延伸10 min。扩增体系为20 μL,4 μL 5×FastPfu缓冲液,2 μL 2.5 mmol/L dNTPs,0.8 μL引物(5 μmol/L),0.4 μL FastPfu聚合酶;10 ng DNA模板。全部样本按照正式实验条件进行,每个样本3个重复,将同一样本的PCR产物混合后用质量分数为2%的琼脂糖凝胶电泳检测。参照电泳初步定量结果,将PCR产物送到上海美吉生物技术有限公司进行测序,用于后续分析。
1.2.4 实时PCR 法检测样本中 16S 基因的绝对含量
使用16S rDNA通用引物341F(5′-CCTACGGGAGGCAGCAG-3′)和518R(5′-TTACCGCGGCTGCTGGC-3′)扩增窖泥宏基因组DNA,得到16S rDNA混合扩增产物。具体步骤参照张会敏等[13]。
1.3 数据处理
原始测序序列使用Trimmomatic软件质控,使用FLASH软件进行拼接,使用的UPARSE软件,根据97%的相似度对序列进行OTU聚类,并在聚类的过程中去除单序列和嵌合体。利用RDP classifier对每条序列进行物种分类注释,比对Silva数据库(SSU123)。利用R语言和Origin2022软件画图。理化数据采用平均数+标准差![]() 表示,窖泥理化的差异性显著性分析通过软件SPSS实现。
表示,窖泥理化的差异性显著性分析通过软件SPSS实现。
2 结果与分析
2.1 窖泥理化性质与酶活
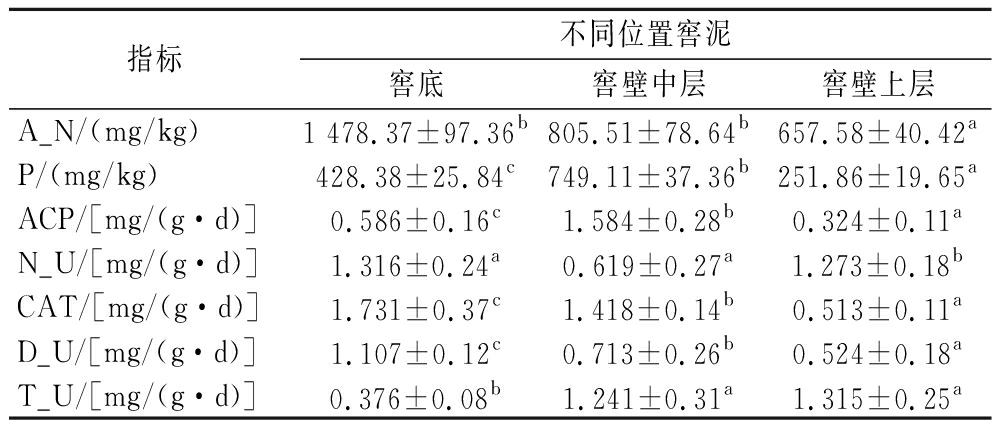
由表1可知,铵态氮、过氧化氢酶活性和蛋白酶活力随着窖窖泥位置的升高,含量减小;窖壁中层窖泥的有效磷含量和酸性磷酸酶活力都显著高于窖底、窖壁上层(P<0.05);窖壁中层窖泥的脲酶活力显著低于窖底、窖壁上层(P<0.05);脱氢酶活力随着窖泥位置的升高,活性增强。
表1 窖泥理化与酶活性质
Table 1 Physicochemical and enzymatic active substances of pit mud
指标不同位置窖泥窖底窖壁中层窖壁上层A_N/(mg/kg)1 478.37±97.36b805.51±78.64b657.58±40.42aP/(mg/kg)428.38±25.84c749.11±37.36b251.86±19.65aACP/[mg/(g·d)]0.586±0.16c1.584±0.28b0.324±0.11aN_U/[mg/(g·d)]1.316±0.24a0.619±0.27a1.273±0.18bCAT/[mg/(g·d)]1.731±0.37c1.418±0.14b0.513±0.11aD_U/[mg/(g·d)]1.107±0.12c0.713±0.26b0.524±0.18aT_U/[mg/(g·d)]0.376±0.08b1.241±0.31a1.315±0.25a
注:不同位置窖泥理化性质与酶活力的差异用小写字母表示,不同字母表示差异显著(P<0.05)。
2.2 不同位置窖泥细菌群落相似性分析
基于Beta多样性距离矩阵进行层次聚类分析,UPGMA算法构建树状结构,呈现窖池不同位置窖泥细菌群落分布的差异程度。由图2可知,所有窖泥样品分为两大簇,所有窖底、窖壁中层窖泥聚为一簇,窖壁上层窖泥单独聚为一簇,不同位置的窖泥理化性质具有差异,窖泥中细菌对环境的适应性不同,从而在窖池发酵过程中,窖泥细菌群落存在独特的空间分布特征。由图3可知,提取到的主成分1和主成分2的特征值是0.847和0.489,且累计方差占78.76%,说明成分划分合理。按照主成分1和主成分2的划分,不同位置的窖泥各自距离较集中,结合聚类和PCoA分析,二者结果基本一致,不同位置的窖泥根据细菌群落能进行较好的聚类,说明其细菌群落相似度较高。图4为通过绝对定量检测出的窖泥样本中16S rDNA的丰度,BM的16S rDNA的拷贝数显著高于WM、WT,但都在相同数量级上。

图2 不同位置窖泥细菌群落聚类树
Fig.2 Bacterial community clustering trees at different locations
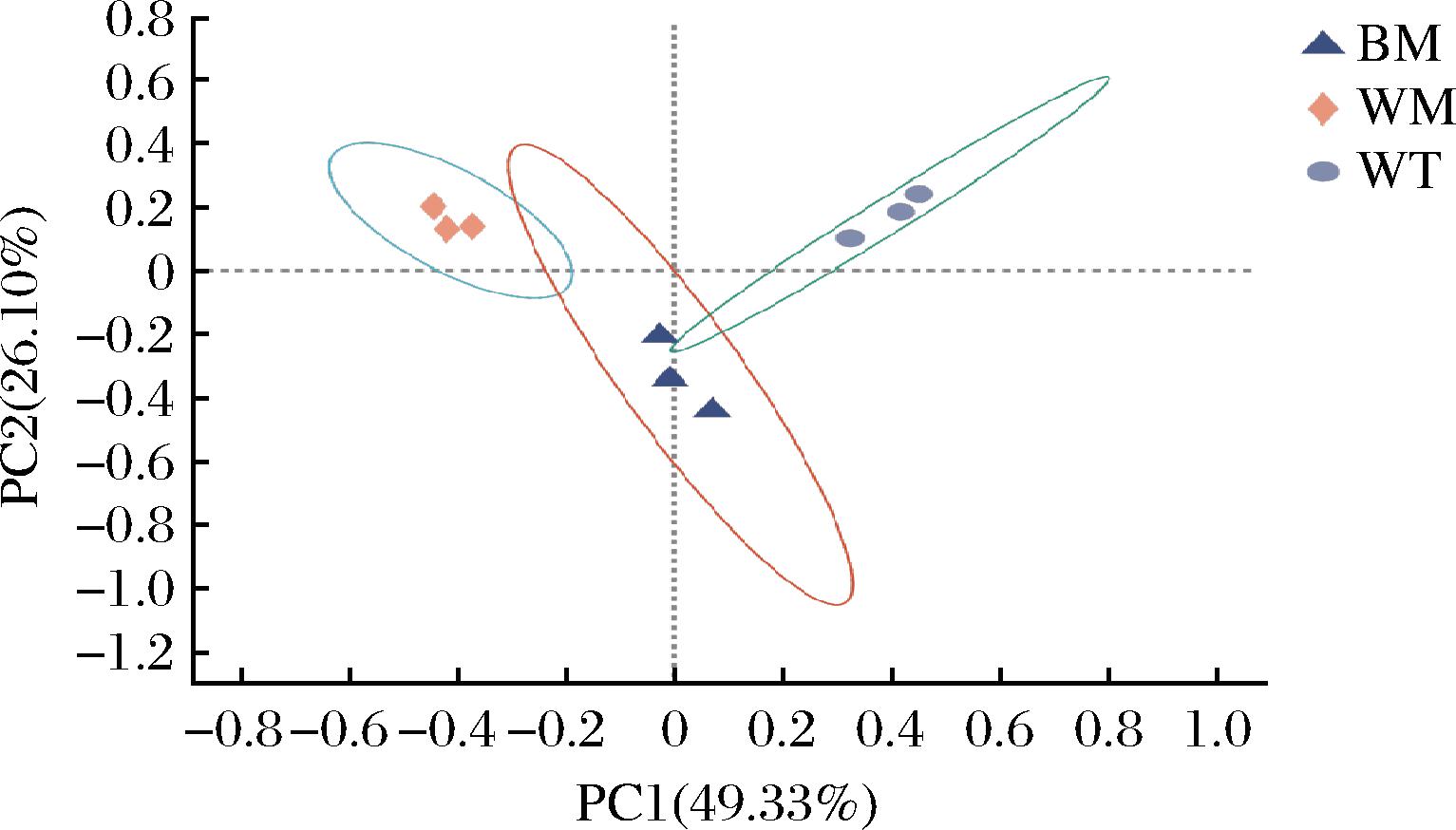
图3 细菌群落多样性PCoA图
Fig.3 Bacterial community diversity PCoA plot
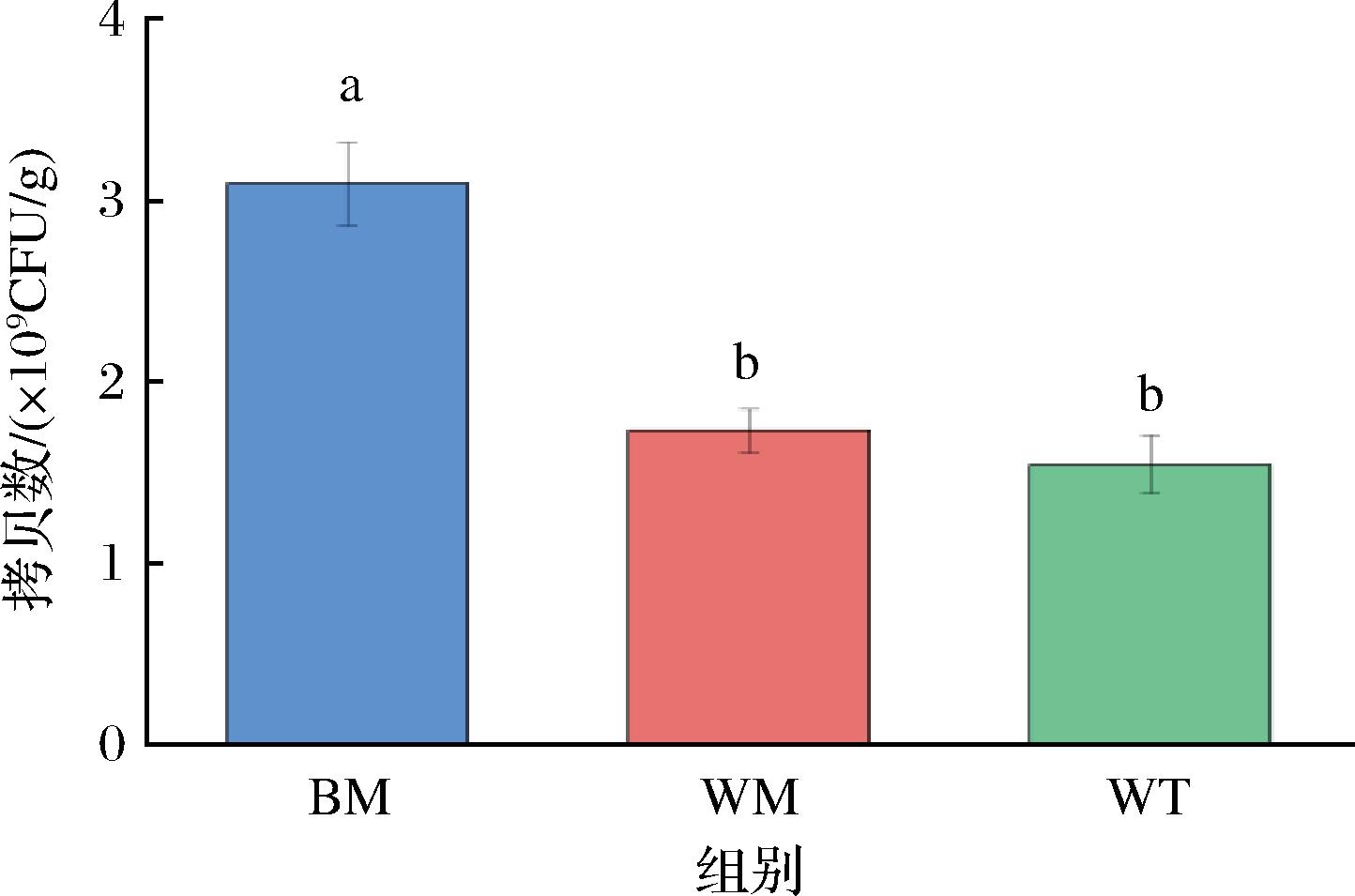
图4 三组窖泥样本中16S rDNA拷贝数
Fig.4 Numbers of 16S rDNA copies in three pit muds
2.3 窖泥细菌群落的门水平和属水平组成分析
将窖泥序列进行OTU物种注释,得到排名前5的优势菌门和排名前20的优势菌属,others表示排名靠后的菌属。如图5-a所示,厚壁菌门(Firmicutes)细菌是窖泥中的绝对优势菌门,窖泥中产酸产香的关键细菌群落隶属于Firmicutes门,不同位置窖泥Firmicutes门细菌的平均相对丰度都在76.4%以上;拟杆菌门(Bacteroidetes)、互养菌门(Synergistetes)细菌是窖底窖泥、窖壁上层窖泥的优势菌门,其平均相对丰度都在1%以上,Bacteroidetes能将窖泥中的Fe3+转化成Fe2+使窖泥颜色由红棕黄褐等转向灰白青,表现出老熟特征[14];窖壁中层窖泥特有的优势菌门是放线菌门(Actinobacteria);窖壁上层窖泥特有的优势菌门是绿弯菌门(Chlorflexi)。
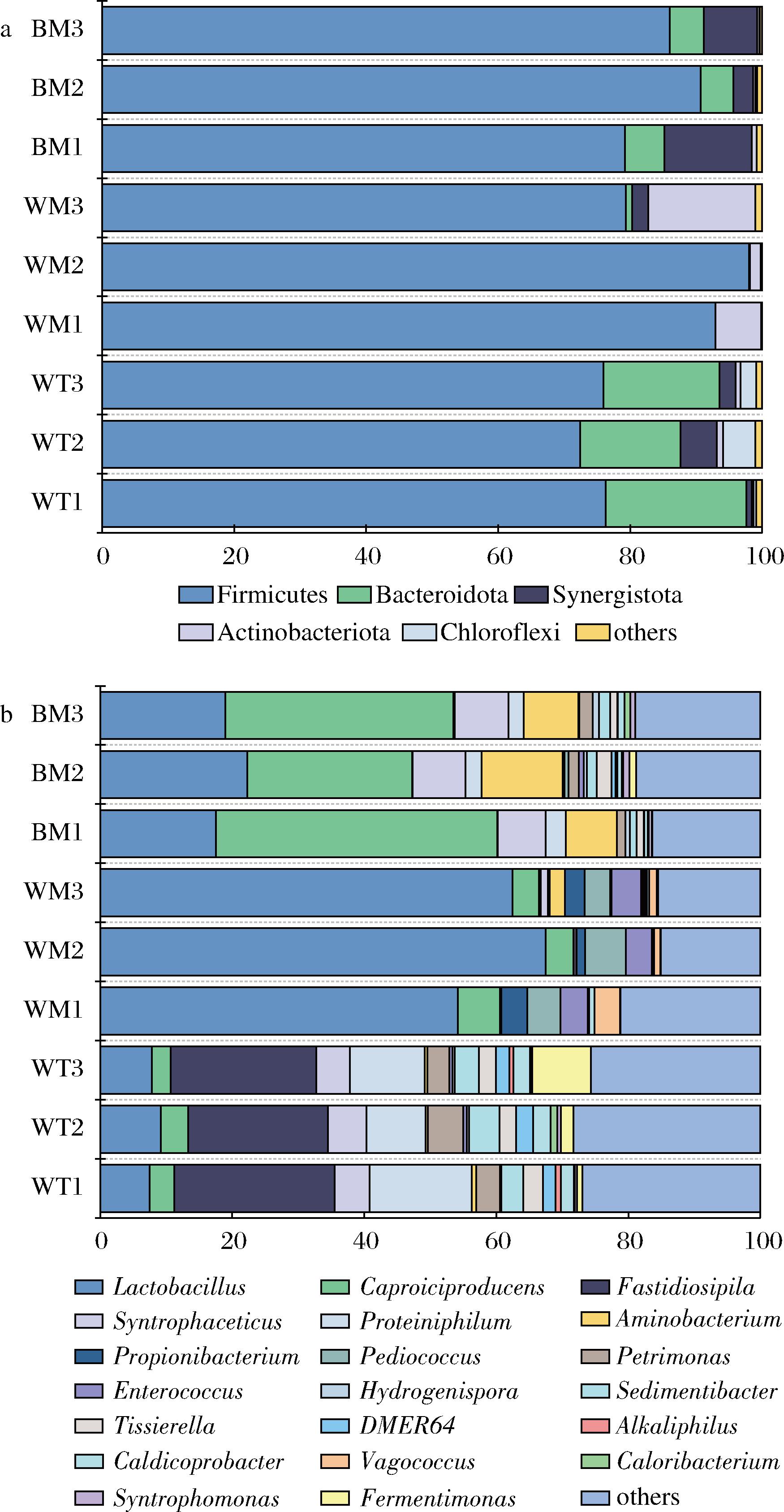
a-门水平;b-属水平
图5 细菌群落Bar图
Fig.5 Bacterial community Bar map
将相对丰度≥1%的属定义为优势菌属,如图5-b所示,不同位置窖泥属水平上的优势菌属存在差异,窖底窖泥的优势菌属有产己酸菌属(Caproiciproduces)、乳杆菌属(Lactobacillus)、乙酸互营菌属(Syntrophaceticus)、产乙酸嗜蛋白菌(Proteiniphilum)、氨杆菌属(Aminobacterium)、片球菌属(Petrimonas)、沉积菌属(Sedimentibacter)、泰氏菌属(Tissierella)等;窖壁中层窖泥的优势菌属有Lactobacillus、Caproiciproduces、丙酸杆菌属(Propionibacterium)、片球菌属(Pediococcus)、肠球菌属(Enterococcus)、漫游球菌属(Vagococcus)等;窖壁上层窖泥的优势菌属有Fastidiosipila、Lactobacillus、DMER64、Caldiocprobacter等。Lactobacillus是窖壁中层窖泥的第一优势菌属,从窖泥的空间位置分布来看,Lactobacillus的相对丰度变化较明显,在窖壁上层窖泥中的平均相对丰度最低(3.7%);Caproiciproduces是窖底窖泥的第一优势菌属,研究表明己酸菌和梭菌等菌群产生的中长链脂肪酸(如己酸)能抑制Lactobacillus的生长代谢[15],窖底窖泥Caproiciproducens相对丰度较高,其产生的中长链脂肪酸对Lactobacillus抑制作用增强,导致其优势地位下降,与前人研究结果相似[13];Fastidiosipila是窖壁上层窖泥的第一优势菌,该菌属在老窖泥中丰度较高,与郭明遗[16]研究结果一致。总之,窖泥中细菌群落结构的分布与窖池的空间位置密切相关。
2.4 不同位置窖泥酶活力与细菌属水平组成热图
提取丰度>1%的属,进一步利用热图展示不同位置窖泥酶活力和细菌群落群的空间分布特征。由图6可知,细菌群落与窖泥的空间位置有一定的关联,但部分指标存在显著差异,这与胡晓龙等[15]研究结果一致。不同位置窖泥的理化因子和酶活性分布特征如图7所示,相比窖底窖泥,窖壁中层和窖壁上层窖泥的铵态氮含量分别降低了45.51%、55.52%,过氧化氢酶活力分别降低了18.08%,70.36%,蛋白酶活力分别降低了35.59%,52.66%,蛋白酶将蛋白质水解成游离氨基酸,游离氨基酸与糖发生反应后合成谷氨酸,对白酒质量有很大影响;而脱氢酶活力分增加了2.3倍、2.5倍;酸性磷酸酶可以水解有机磷,提供微生物所需的微量元素,该酶在窖壁中层活性最高的原因可能是窖壁中层的有效磷含量最高。过氧化氢酶、蛋白酶的活性与Caproiciproduces、Aminobacterium的相对丰度变化规律相似,脱氢酶活性与Caldicoprobacter、Sedimentibacter等菌属的变化规律相似,是否具有显著的相关性还需进一步分析。
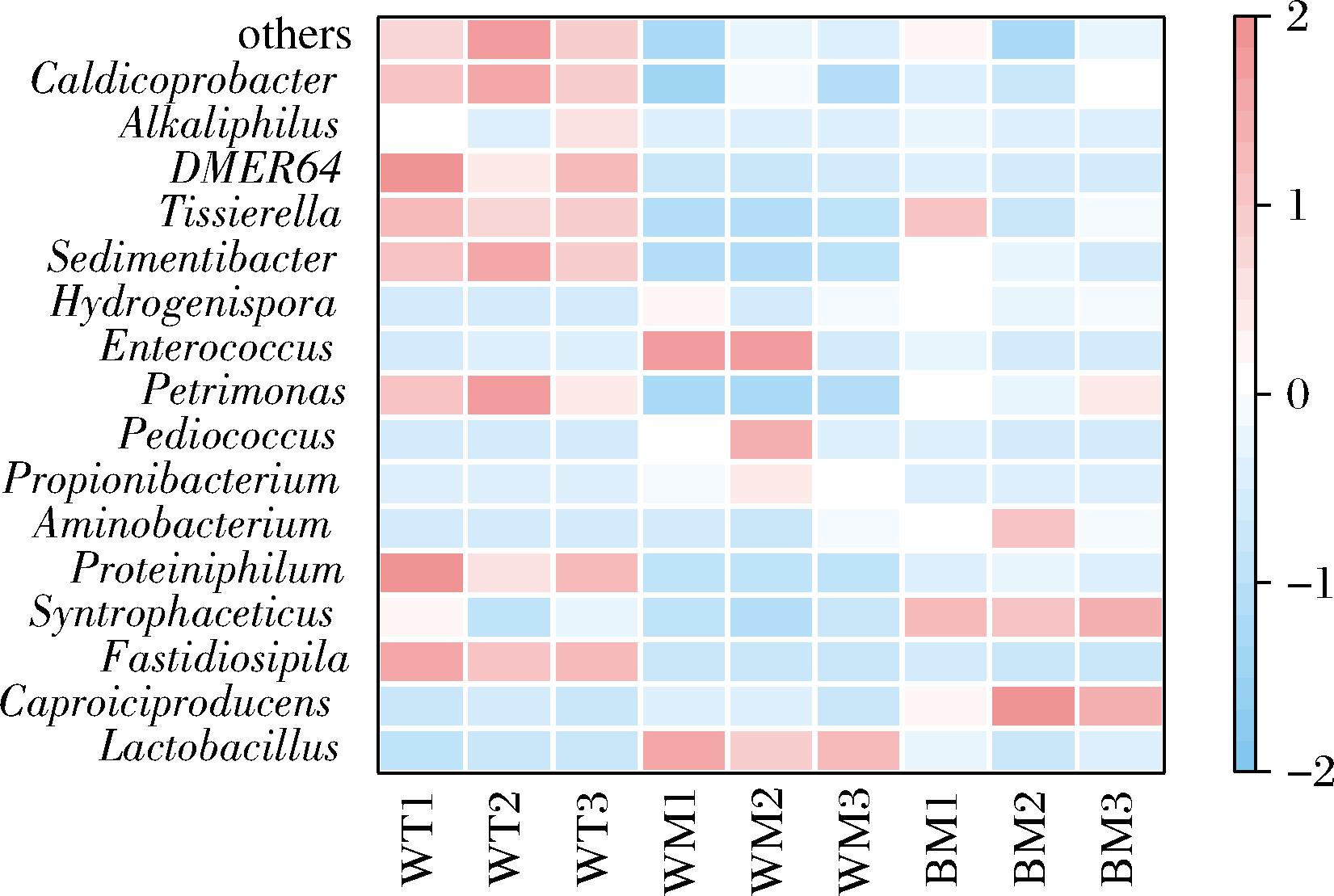
图6 不同位置窖泥属水平细菌群落组成热图
Fig.6 Heatmap of bacterial communities at the level of the genus pit mud at different locations
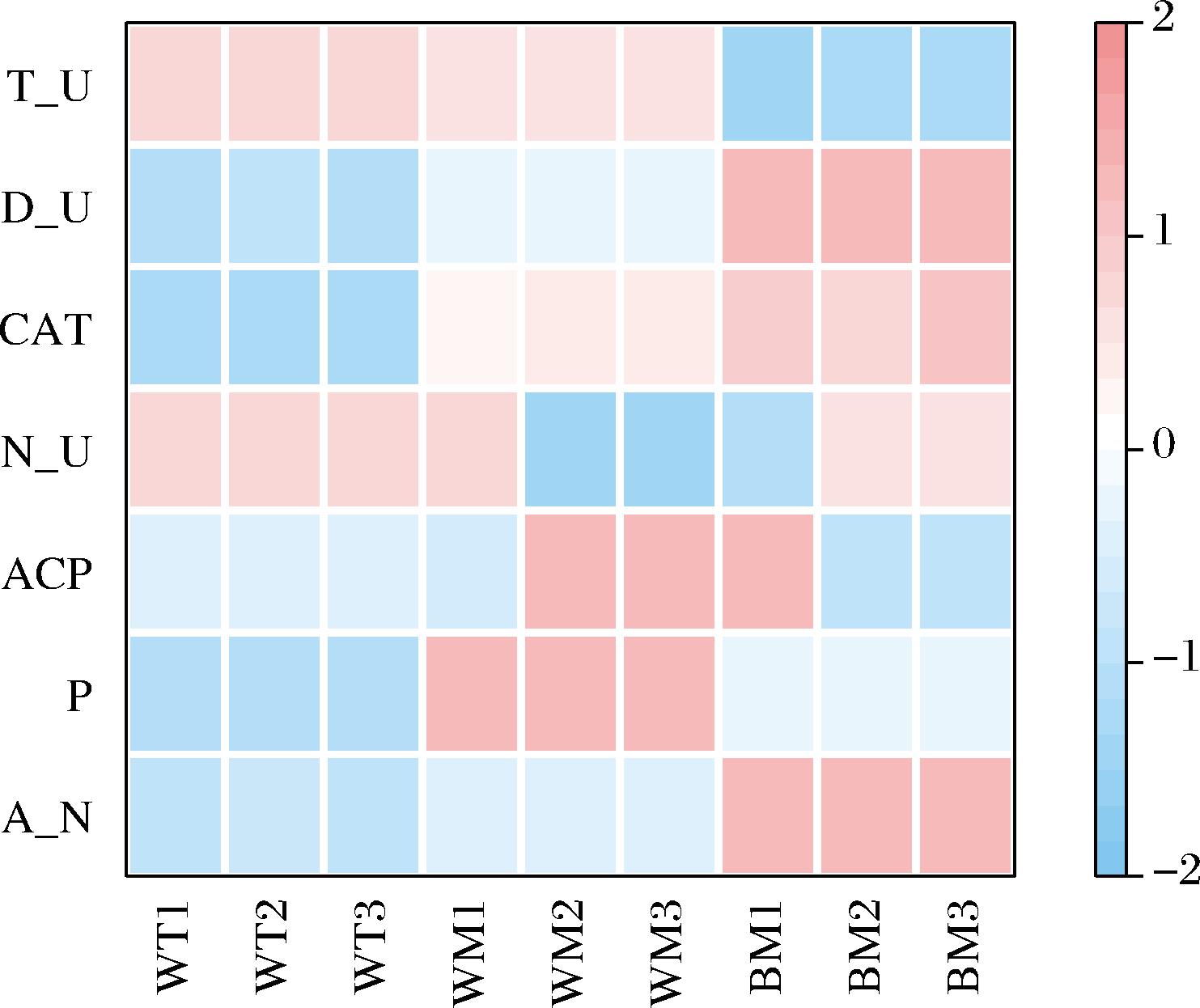
图7 不同位置窖泥理化和酶活力热图
Fig.7 Heatmap of physicochemical and enzyme communities at the level of activity of pit mud at different locations mud at different locations
2.5 不同位置窖泥理化酶活力与细菌群落相关性分析
通过对窖泥和理化指标进行相关性分析,图8结果表明,窖底窖泥的环境对铵态氮、蛋白酶活力的影响较大。由图9可以看出,铵态氮与Aminobacterium呈显著的正相关,与Enterococcus呈显著的负相关;脲酶活力与Syntrophaceticus、Petrimonas、Tissierella呈显著的正相关,与Propionibacterium、Enterococcus呈显著的负相关;有效磷和酸性磷酸酶活力对窖壁中层窖泥的影响力最大,其次是过氧化氢酶,酸性磷酸酶活性与Lactobacillus、Propionibacterium、Pediococcus、Enterococcus、Vagocossus呈显著的正相关;蛋白酶活力与Caproiciproduces、Aminobacterium呈显著的正相关。
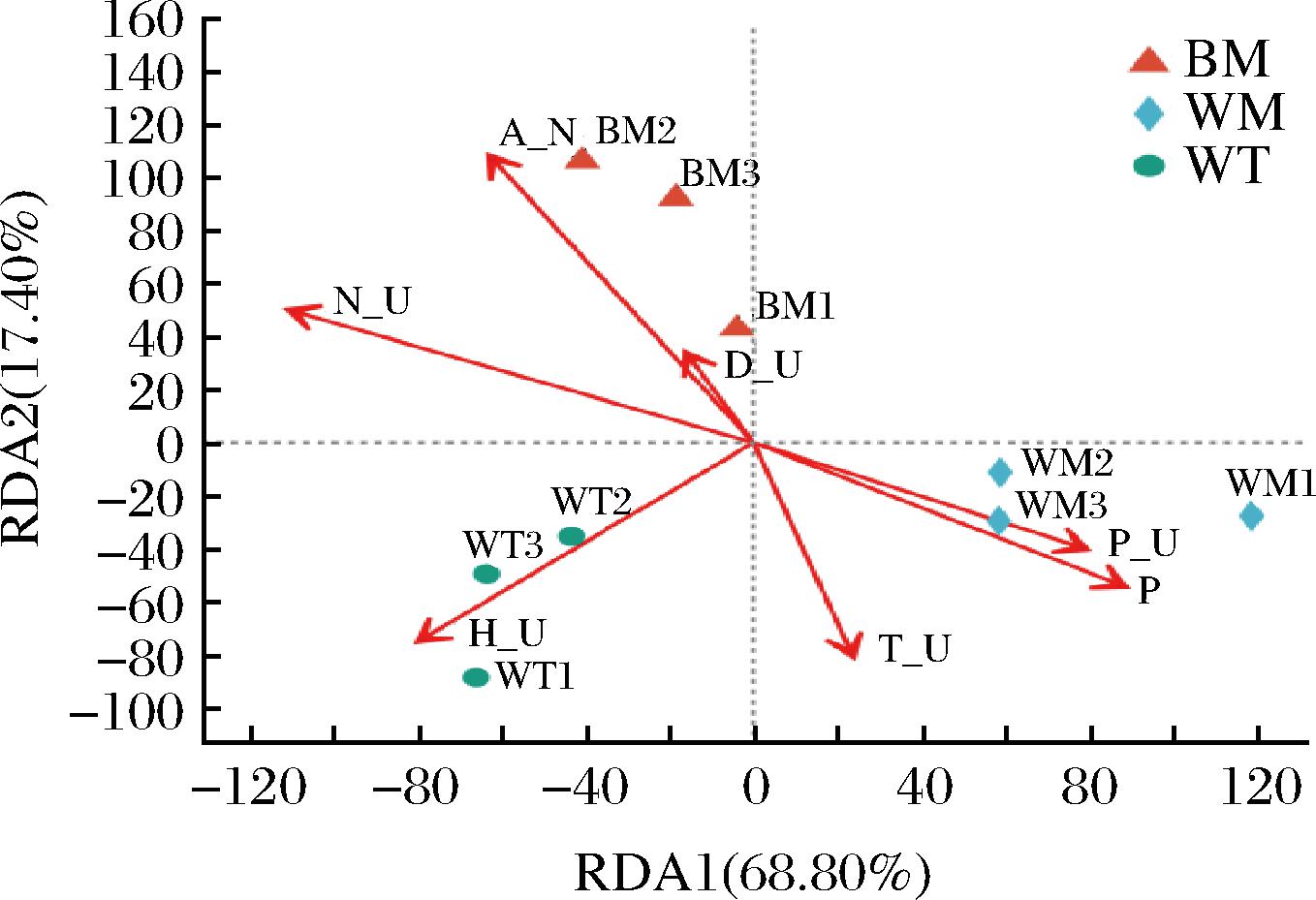
图8 酶活性与细菌群落结构相关性RDA图(属水平)
Fig.8 RDA plot of enzyme activity and bacterial community structure correlation (genus level)
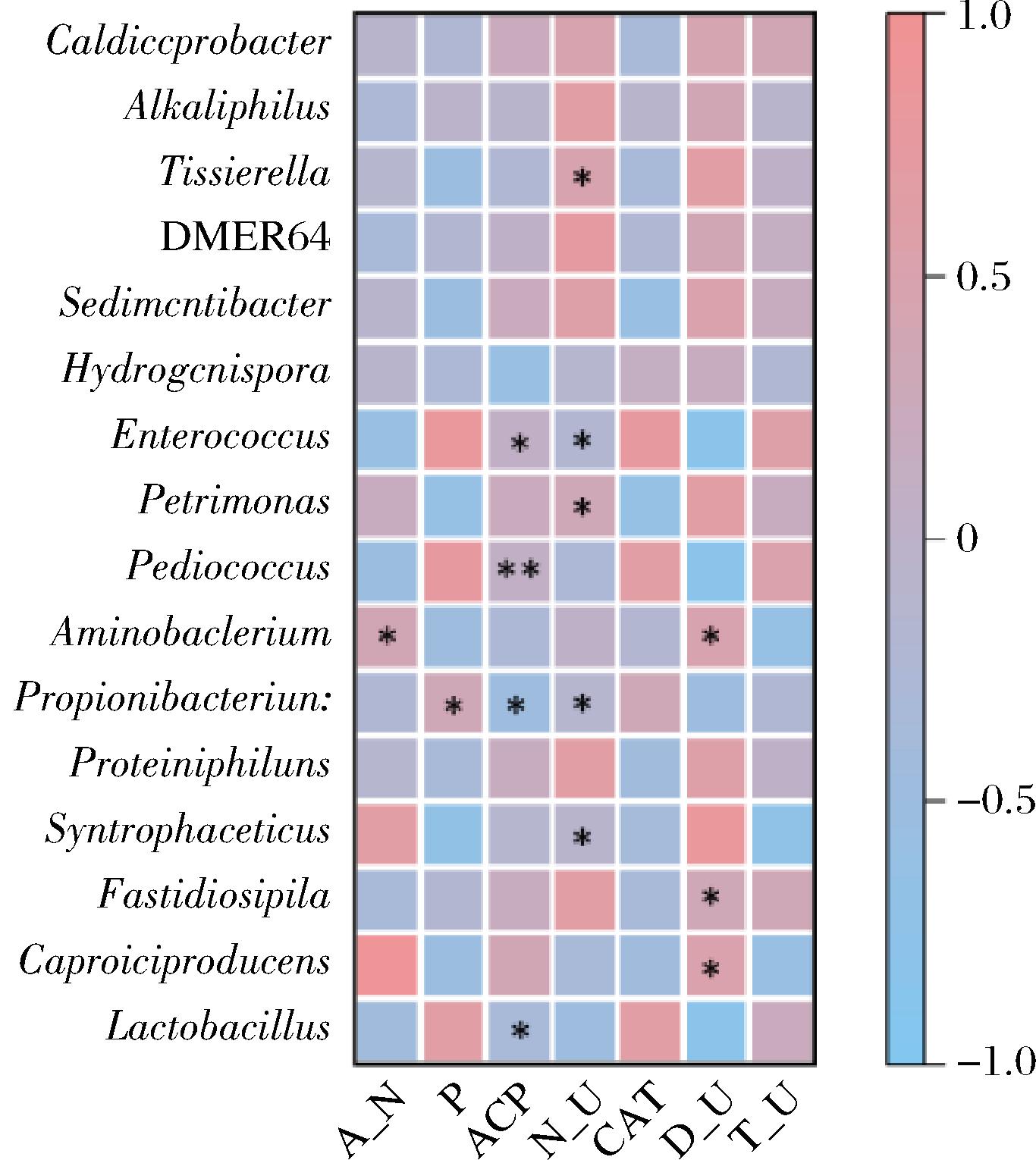
图9 酶活力与菌群相关性热图
Fig.9 Heat map of enzyme activity and microbiota correlation
2.6 不同位置窖泥参与氮代谢的功能酶差异分析
土壤微生物是氮代谢过程中的主要驱动者,在窖泥这个特殊微生态环境中,通过微生物的一系列协同作用也可以完成氮代谢中的一些循环过程,通过PICRUSt2标记基因序列预测窖泥相关酶功能和丰度信息,对Ammonium(氨)的合成途径进行了构建,如图10所示,窖泥中脲酶和蛋白酶是参与有机氮向无机氮形式转换的关键酶,分别在氮代谢和碳酰胺循环中发挥极其重要的作用,其中蛋白酶促使蛋白质、肽类水解成氨基酸,脲酶促进窖泥中碳酰胺水解成氨,水解后的氨基酸在谷氨酸合成酶(glutamate synthase,GOGAT)的作用下生成谷氨酸,再通过谷氨酸脱氢酶(glutamate dehydrogenase,GDH)的催化作用形成氨,氨能通过亚硝酸盐还原酶(nitrite reductase,nirS)的作用生成。

图10 窖泥不同氮循环功能酶的分布特征
Fig.10 Distribution characteristics of different nitrogen cycling functional enzymes
通过理化指标检测,发现不同位置窖泥的酶活存在显著的差异,随着窖池位置的深入,蛋白酶活力逐渐变高,这与常文化等[17]研究土壤蛋白酶变化趋势相反,可能是窖泥在驯化过程中,不断的对土壤微生物进行筛选,最后形成了窖泥独特的细菌群落结构,导致了蛋白酶活力与普通土壤的差异。脲酶在窖底和窖壁上层窖泥中活性最高,可能是窖泥中存在较高的腐殖质含量,利于微生物生长,其活跃代谢使得酶活力较高[18];结合图11与表1可知,窖泥中不同酶活力的差异与预测结果基本相符合,功能酶的预测结果表明,窖泥中的细菌群落具有完成氮代谢和氨循环的潜力,并且不同位置窖泥参与代谢的关键酶存在显著的差异。
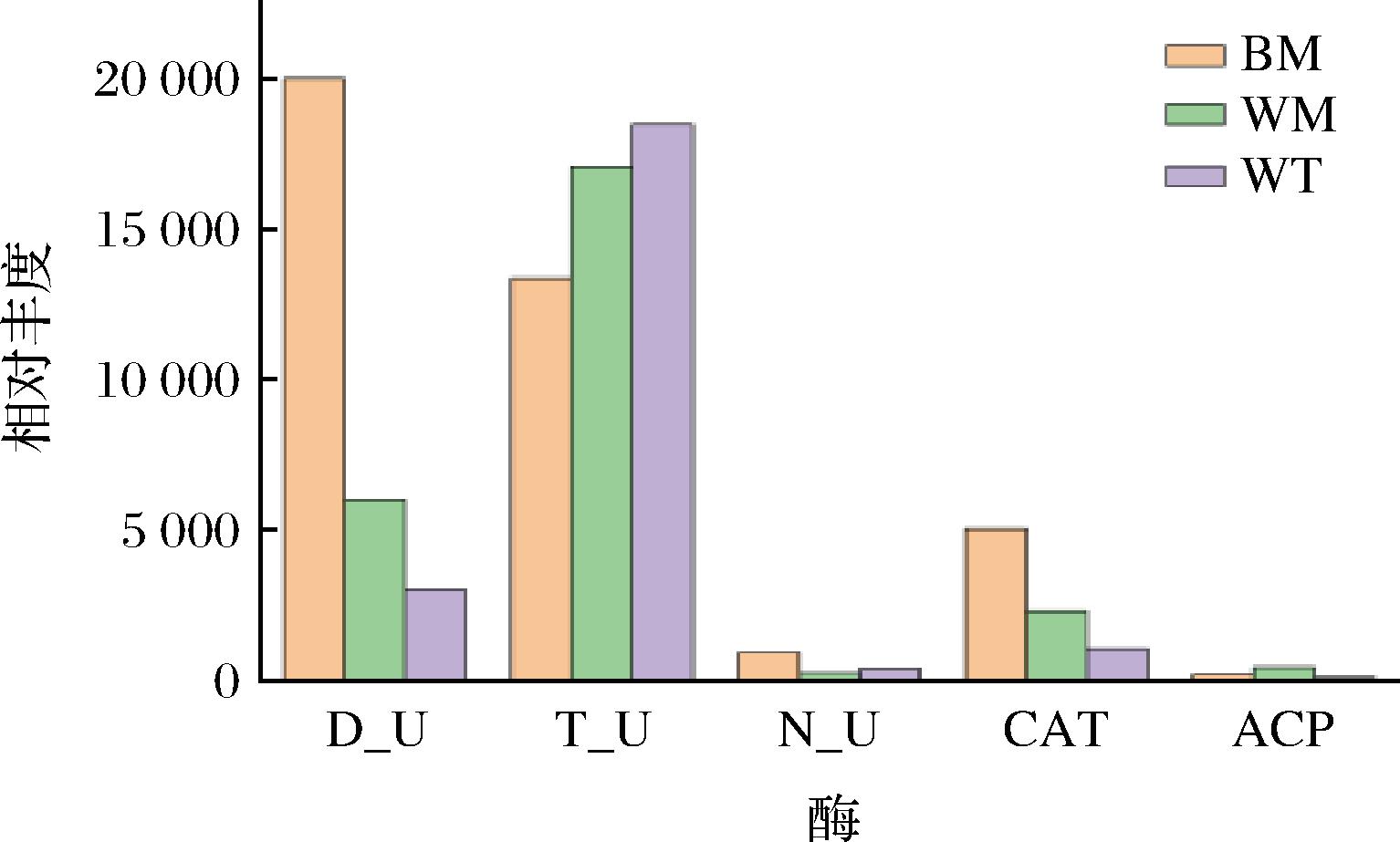
图11 酶活力差异与PICRUSt功能预测结果的比较
Fig.11 Comparison of differences in enzyme activity with PICRUSt function predictions
3 讨论
本研究分析了理化因子、酶活力及细菌群落的空间分布规律,铵态氮的含量随着窖泥的位置自下而上呈减小趋势,铵态氮是微生物自身繁殖、合成各种蛋白质以及酶类所必需的物质,并涉及氨基酸代谢、硝化和反硝化,此外,氮源作为微生物生长发育的主要营养元素,与细菌群落密切相关[19]。适量的铵态氮对维持窖泥生境,提高酒质有着重要作用,与之呈正相关的Aminobacterium的相对丰度随着窖泥的位置自上而下呈减小趋势,可推测铵态氮的含量影响微生物属的相对丰度,可能铵态氮通过硝化反应影响窖泥的pH值,间接影响窖泥的细菌群落结构,也可能是铵态氮作为氮源直接影响微生物的生长繁殖。窖壁中层窖泥酸性磷酸酶活力、脲酶活力最高,窖泥中的酸性磷酸酶用于分解有机磷释放出有效磷,促进窖泥细菌的代谢[20];有研究表明品级越高的窖泥,脲酶活性越低[21],脲酶能够特异性水解碳酰胺释放铵态氮,碳酰胺是存在于窖泥中可以转化为铵态氮的氮源,脲酶是转化过程中必不可少的水解酶,可能是维持2种氮源的平衡,利于窖泥这种特殊环境下细菌群落的代谢,碳酰胺和铵态氮两者之间转换机制还未见报道,后续研究脲酶对这2种氮源的转换可能会为浓香型白酒发酵条件的控制提供新的理论基础。窖池中不同位置窖泥的理化性质和细菌群具有较强的相关性,铵态氮与Aminobacterium呈较强的正相关,该菌属不仅可以降解氨基酸增加铵态氮的含量,还可以降解丝氨酸、甘氨酸并且以苏氨酸为底物,产生可以被氢气还原成乙醇的醋酸盐[22-23],浓香型白酒发酵过程中会产生乙醇,乙醇作为浓香型白酒关键风味物质己酸的有效底物,因此Aminobacterium可能有助于浓香型白酒的风味形成,并且在Aminobacterium参与氨基酸发酵过程中与产氢产甲烷菌协同作用,以减少氢胁迫[24],不同的理化性质以及长期演化的微生物群落,使得Aminobacterium成为窖底窖泥的优势菌属。
蛋白酶与Caproiciproducens呈显著的正相关,该菌属具有产生参与酸代谢酶类的特性,能够将乙醇作为底物合成己酸,能为白酒酿造代谢丰富的风味物质。酸性磷酸酶活性与Lactobacillus、Pediococcus、Enterococcus、Proteiniphilum呈显著的正相关,其中乳酸菌种群较多,研究表明,乳酸菌代谢过程中积累的乳酸可以作为梭菌合成丁酸和己酸等风味物质的底物[25],同时乳酸也是窖泥中菌群形成完整代谢功能的主要压力[26],通过抑制磷酸酶的活性是否可以达到降解乳酸的效果,将是后期的研究重点,脲酶活力与Syntrophaceticus、Proteiniphilum、Petrimonas、Tissierella、呈显著的正相关,Syntrophaceticus与甲烷菌共存时能够表现出乙酸氧化能力,将乙酸盐转化为氢气和二氧化碳,随后产甲烷菌利用氢气将二氧化碳还原成甲烷[27],利于甲烷菌的生长代谢,甲烷菌是窖泥老熟的标志,适量的Syntrophaceticus存在与窖泥中,可能加快窖泥的老熟,形成完整的窖泥菌群代谢链,这也可能是脲酶活性与窖泥质量相关的原因。Petrimonas能够进行无机盐的还原、多环芳烃的降解[28],Tissierella的降解产物中含有乙酸[29],与特定菌体在协同作用下可以降解长链饱和脂肪酸,生成乙酸[30],窖泥中功能菌代谢产生的功能酶是浓香型白酒独特风味形成的关键,窖池中不同位置窖泥的细菌群落结构不同,酶活性也具有差异,能够在白酒发酵过程中及时调控,通过改变环境条件促进有益功能菌的代谢可能有助与浓香型白酒的提升。
4 结论
不同位置窖泥的酶活力和理化性质差异显著,其中窖底窖泥蛋白酶、脲酶的活性最高,谷氨酸脱氢酶的丰度较高,而nirS酶丰度最低,表明窖底窖泥的氨来源主要是通过碳酰胺循环和氨基酸的分解。不同位置的窖泥细菌群落相对丰度存在显著的差异,此外窖泥理化性质与其细菌群落的RDA相关性分析发现,铵态氮含量、有效磷含量、过氧化氢酶含量、酸性磷酸酶对窖泥细菌群落的解释度较高,其影响着窖泥的细菌群结构特征,空间位置也对窖泥物质循环的关键酶产生了重要影响。
[1] HE M W, JIN Y, ZHOU R Q, et al.Dynamic succession of microbial community in Nongxiangxing daqu and microbial roles involved in flavor formation[J].Food Research International, 2022, 159:111559.
[2] CHEN L, LI Y Z, JIN L, et al.Analyzing bacterial community in pit mud of Yibin Baijiu in China using high throughput sequencing[J].Peer J, 2020, 8:e9122.
[3] 刘洢杋. 模拟氮磷沉降对北方针叶林土壤性质、微生物群落结构及氮循环过程的影响[D].桂林:广西师范大学, 2022.
LIU Y F.Effects of Simulated nitrogen and phosphorus deposition on soil properties, microbial community structure and nitrogen cycling in a boreal coniferous forest[D].Guilin:Guangxi Normal University, 2022.
[4] THANGARAJ B, RAJASEKAR D P, VIJAYARAGHAVAN R, et al.Cytomorphological and nitrogen metabolic enzyme analysis of psychrophilic and mesophilic Nostoc sp.:A comparative outlook[J].3 Biotech, 2017, 7(2):1-10.
[5] SU Y Z, LI Y L, CUI J Y, et al.Influences of continuous grazing and livestock exclusion on soil properties in a degraded sandy grassland, Inner Mongolia, northern China[J].CATENA, 2005, 59(3):267-278.
[6] WALLENSTEIN M D, MCMAHON S K, SCHIMEL P.Seasonal variation in enzyme activities and temperature sensitivities in Arctic tundra soils[J].Global Change Biology, 2009, 15(7):1631-1639.
[7] TAN X, CHANG S X, KABZEMS R, et al.Soil compaction and forest floor removal reduced microbial biomass and enzyme activities in a boreal aspen forest soil[J].Biology and Fertility of Soils, 2008, 44(3):471-479.
[8] 韩光, 张宿义, 卢中明.窖泥微生物、窖泥酶活性与窖泥养分相关性研究[J].酿酒科技, 2011(3):48-51;54.
HAN G, ZHANG S Y, LU Z M.Research on the correlations among soil microbes, soil enzyme activity, and soil nutrient in pit mud[J].Liquor-Making Science &Technology, 2011(3):48-51;54.
[9] 吴树坤, 刘梅, 邓杰, 等.不同品质浓香型窖泥的酶活与微生物群落的相关性[J].食品与发酵工业, 2018, 44(1):19-24.
WU S K, LIU M, DENG J, et al.Relation of microbial composition and enzyme activity index with different grades of Luzhou flavored clay[J].Food and Fermentation Industries, 2018, 44(1):19-24.
[10] WANG L X, PANG X Y, LI N, et al.Effects of vegetation type, fine and coarse roots on soil microbial communities and enzyme activities in eastern Tibetan Plateau[J].CATENA, 2020, 194:104694.
[11] 张淼, 刘俊杰, 刘株秀, 等.黑土区农田土壤氮循环关键过程微生物基因丰度的分布特征[J].土壤学报, 2022, 59(5):1258-1269.
ZHANG M, LIU J J, LIU Z X, et al.Distribution characteristics of microbial gene abundance in key processes of soil nitrogen cycling in black soil zone[J].Acta Pedologica Sinica, 2022, 59(5):1258-1269.
[12] LIU X Z, MENG W, LIANG G H, et al.Available phosphorus in forest soil increases with soil nitrogen but not total phosphorus:evidence from subtropical forests and a pot experiment[J].PloS one, 2014, 9(2):e88070.
[13] 张会敏, 王艳丽, 孟雅静, 等.浓香型白酒发酵新老窖泥理化因子和原核微生物群落结构差异分析[J].食品科学, 2020, 41(6):207-214.
ZHANG H M, WANG Y L, MENG Y J, et al.Differences in physicochemical properties and prokaryotic microbial communities between young and old pit mud from Chinese strong-flavor Baijiu brewing[J].Food Science, 2020, 41(6):207-214.
[14] 任海伟, 孙一帆, 王希, 等.不同窖龄及位置浓香型白酒窖泥中细菌群落结构的差异性分析[J].食品与发酵工业, 2023,49(9):103-111.
REN H W, SUN Y F, WANG X, et al.Difference analysis of bacterial community structure in pit mud with different ages and position[J].Food and Fermentation Industries, 2023,49(9):103-111.
[15] 胡晓龙, 余苗, 曹振华, 等.基于高通量测序的窖泥原核微生物群落多样性在退化窖池中的空间异质性[J].食品科学, 2021, 42(10):86-93.
HU X L, YU M, CAO Z H, et al.Spatial heterogeneity of prokaryotic microbial community diversity in pit mud from degraded cellar for the production of strong-flavor baijiu revealed by high throughput sequencing[J].Food Science, 2021, 42(10):86-93.
[16] 郭明遗. 基于宏基因组高通量测序窖泥微生态及其基因多样性研究[D].重庆:重庆大学, 2020.
GUO M Y.Metagenomic approach studying the taxonomic diversity and functional genes of the pit-mud of Chinese liquor[D].Chongqing:Chongqing University, 2020.
[17] 常文华, 马维伟, 李广, 等.尕海湿地不同退化梯度土壤脲酶与蛋白酶活性时空分布特征[J].土壤, 2022, 54(3):524-531.
CHANG W H, MA W W, LI G, et al.Temporal and spatial distribution characteristics of soil urease and protease activities in different degraded gradients of Gahai wetland[J].Soils, 2022, 54(3):524-531.
[18] 徐静静, 刘隋赟昊, 朱新萍, 等.巴音布鲁克天鹅湖高寒湿地不同水分梯度土壤微生物及酶活性的差异[J].新疆农业大学学报, 2017, 40(5):337-344.
XU J J, LIU S B H, ZHU X P, et al.Soil microorganism and enzymatic activity differences at various water gradients in swan lake alpine wetland of Bayanbulak grassland[J].Journal of Xinjiang Agricultural University, 2017, 40(5):337-344.
[19] GONG Y, MA N, TANG H.Analysis of microbial community diversity and physicochemical factors in pit mud of different ages based on high-throughput sequencing[J].Canadian Journal of Microbiology, 2022, 68(11):674-686.
[20] 李学思, 李绍亮, 曹振华, 等.窖池自然老熟过程中窖泥理化指标及产酒质量变化规律的研究[J].酿酒, 2019, 46(3):31-35.
LI X S, LI S L, CAO Z H, et al.Study on the change of physicochemical indexes and quality of wine produced in pit mud during natural aging[J].Liquor Making, 2019, 46(3):31-35.
[21] 宣灵, 汪英, 陈瑞, 等.宣酒功能窖泥应用于新窖池后品质变化[J].酿酒, 2015, 42(1):69-72.
XUAN L, WANG Y, CHEN R, et al.The change of quality of the functional pit mud of Xuan liquor was applied to new pool[J].Liquor Making, 2015, 42(1):69-72.
[22] HAMDI O, BEN HANIA W, POSTEC A, et al.Aminobacterium thunnarium sp.nov., a mesophilic, amino acid-degrading bacterium isolated from an anaerobic sludge digester, pertaining to the phylum Synergistetes[J].International Journal of Systematic and Evolutionary Microbiology, 2015, 65(Part 2):609-614.
[23] BAENA S, FARDEAU M L, LABAT M, et al.Aminobacterium mobile sp.nov., a new anaerobic amino-acid-degrading bacterium[J].International Journal of Systematic and Evolutionary Microbiology, 2000, 50(1):259-264.
[24] ZHENG Y, HU X L, JIA Z J, et al.Co-occurrence patterns among prokaryotes across an age gradient in pit mud of Chinese strong-flavor liquor[J].Canadian Journal of Microbiology, 2020, 66(9):495-504.
[25] FU J X, CHEN L, YANG S Z, et al.Metagenome and analysis of metabolic potential of the microbial community in pit mud used for Chinese strong-flavor liquor production[J].Food Research International, 2021, 143:110294.
[26] 孟雅静, 王艳丽, 丁峰, 等.浓香型白酒新、老窖池分层池底窖泥菌群总氢代谢与乳酸含量之间的关系[J].食品科学, 2021, 42(18):171-177.
MENG Y J, WANG Y L, DING F, et al.Relationship between microbial total hydrogen metabolism and lactic acid content in layered bottom pit muds from old and young pits used for the production of strong-flavor Baijiu[J].Food Science, 2021, 42(18):171-177.
[27] MANZOOR S, BONGCAM-RUDLOFF E, SCHNÜRER A, et al.Genome-guided analysis and whole transcriptome profiling of the mesophilic syntrophic acetate oxidising bacterium Syntrophaceticus schinkii[J].PLoS One, 2016, 11(11):e0166520.
[28] QIAN Y F, XU M Y, DENG T C, et al.Synergistic interactions of Desulfovibrio and Petrimonas for sulfate-reduction coupling polycyclic aromatic hydrocarbon degradation[J].Journal of Hazardous Materials, 2021, 407:124385.
[29] HARMS C, SCHLEICHER A, COLLINS M D, et al.Tissierella creatinophila sp.nov., a gram-positive, anaerobic, non-spore-forming, creatinine-fermenting organism[J].International Journal of Systematic Bacteriology, 1998, 48(Part 3):983-993.
[30] WU C G, DONG X Z, LIU X L.Syntrophomonas wolfei subsp.methylbutyratica subsp.nov., and assignment of Syntrophomonas wolfei subsp.saponavida to Syntrophomonas saponavida sp.nov.comb.nov[J].Systematic and Applied Microbiology, 2007, 30(5):376-380.