作为中国传统的发酵调味品,酱油以大豆、小麦和食盐为主要原料,经人工添加含有米曲霉等微生物的曲种发酵而成[1]。虽然酱油中含有大量的食盐,但由于制作的开放性,产品中亦可能存在部分微生物,货架期内若贮藏温度过高,则产品可能会出现胀气和变酸等质量问题[2]。以货架期内出现胀壶现象的酱油为研究对象,上官宗渺[3]发现其微生物类群主要由破布子乳杆菌(Lactobacillus pobuzihii)、丁酸梭菌(Clostridium butyricum)、埃切毕赤酵母(Pichia etchellsii)、巨大芽孢杆菌(Bacillus megaterium)和枯草芽孢杆菌(Bacillus subtilis)构成。杨卓等[4]通过基因测序和产气实验证明了工厂中胀罐酱油的厌氧产气菌为L.pobuzihii。蒋雪薇等[5]在成品变质酱油中解析出地衣芽胞杆菌(Bacillus licheniformis)、嗜盐芽胞杆菌(Bacillus halodurans)和B.megaterium等3株耐盐性较好的产气菌。张佳惠等[6]研究亦表明变质酱油中可能存在金黄色葡萄球菌(Staphylococcus aureus)等食源性致病菌,并且随着贮藏时间的延长,酱油中菌落总数会逐渐增加,食用后对身体健康存在极大的危害。由此可见,系统解析胀气酱油中微生物的类群,对提升货架期内酱油的稳定性和食用安全性均具有积极的意义。
目前大量研究人员对微生物的产气途径进行了分析,发现通过异型乳酸发酵部分微生物经戊糖磷酸途径可产生乳酸、乙酸和二氧化碳[7],另外微生物的乳糖发酵产酸产气也是一个重要途径[8]。具体来讲,部分酵母菌等真菌通过呼吸作用可产生二氧化碳[9],梭菌等嗜温及嗜热细菌可利用碳水化合物发酵产氢气[10];产甲烷菌在厌氧环境中可利用挥发性脂肪酸产生甲烷[11]。由此可见,通过发酵产气的微生物是多样的,但可引起生抽酱油胀气的具体微生物类群还有待进一步研究。利用宏基因组技术,SULAIMAN等[12]对酱油发酵过程中微生物类群和功能的变化进行了解析,结果发现发酵前期魏斯氏菌(Weissella)为优势菌群而发酵后期则转变为念珠菌(Candida),该研究亦同时揭示了不同发酵时期蛋白质和碳水化合物异养发酵的特征,这也直接证实了宏基因组技术在酱油微生物类群和功能解析中可行性。
湖北省某调味品公司生产的生抽酱油在留样贮藏期间出现了胀气现象,本研究采集了2个批次的胀袋生抽酱油样品,首先采用宏基因组测序技术对其微生物类群进行了解析,继而采用纯培养技术对其微生物菌株进行了分离鉴定,最后对分离出的微生物菌株进行了产气验证实验。通过本研究的开展以期为生抽酱油胀气问题的解决提供数据支持和理论指导。
1 材料与方法
1.1 材料与试剂
于2022年7月中旬和8月初,从湖北某酱油生产厂家采集在贮藏期间中出现涨袋现象的生抽酱油样品2份,分别编号为Q1和Q2,同时采集正常酱油样品2份,所有样品的生产时间均为2022年5月。
QIAGEN DNeasy mericon Food Kit DNA基因组提取试剂盒,德国QIAGEN公司;Illumina Novaseq 6000高通量测序平台配套试剂,美国Illumina公司;PDA、LB和MRS培养基,青岛海博生物技术有限公司;十六烷基三甲基溴化铵、酚、氯仿、异戊醇、氯化钠、乙酸钠、乙醇,国药集团化学试剂有限公司;PCR buffer、dNTP mix、r Taq酶、Solution I、pMD18-T载体和Loading buffer,宝生物工程(大连)有限公司;PCR产物清洁试剂盒,艾思进生物技术(杭州)公司;引物27F/1495R、NS1/NL4和M13F(-47)/M13R(-48),武汉天一辉远生物科技有限公司。
1.2 仪器与设备
Veriti FAST梯度PCR仪,美国ABI公司;164-5050基础电泳仪,美国Bio-Rad公司;UVPCDS8000凝胶成像分析系统,美国Protein Simple公司;Illumina novaseq 6000高通量测序平台,美国Illumina公司;R920型机架式服务器,美国DELL公司;DG250型厌氧工作站,英国Don Whitley公司。
1.3 实验方法
1.3.1 宏基因组测序及生物信息学分析
使用QIAGEN DNeasy mericon Food Kit DNA基因组提取试剂盒提取生抽酱油样品的宏基因组DNA,并将通过琼脂糖凝胶电泳检验合格的DNA寄往北京诺禾致源科技股份有限公司进行双端各150 bp的宏基因组测序,测序平台为Illumina HiSeq XTen。首先使用fastp v0.23.2软件(https://github.com/OpenGene/fastp)对下机序列进行质控,剔除长度<50 bp、平均质量值<20 bp和含有N碱基的序列[13];继而通过与人类基因组比对,去除实验中引入的人类基因组序列后得到可用于后续分析的有效序列;最后将有效序列与kraken2(v2.1.1)[14]和Bracken(v2.5)数据库[15]比对以解析出胀气生抽样品中菌群群落的结构信息。
1.3.2 基于纯培养技术胀袋生抽酱油中微生物的分离
使用倍比稀释法将酱油样品稀释至10-1梯度,各取1 mL原液和1 mL稀释液分别涂布于PDA、LB和MRS平板中,PDA平板在25 ℃有氧条件下正置培养5 d,LB平板在30 ℃有氧条件下倒置培养2 d,MRS平板在37 ℃无氧条件(85% N2,10% H2和5% CO2)下倒置培养2 d。挑取形态、大小和颜色各不相同的菌株,依照三区划线法连续划线3次后分别转接于PDA、LB和MRS的液体试管中,培养1 d后收集菌体进行过氧化氢酶实验和革兰氏染色,最后通过观察镜检结果初步判断分离株的种类[16]。
1.3.3 胀袋生抽酱油中可培养微生物的鉴定及登录号的申请
参照GUO等[17]的方法提取疑似细菌的DNA,并进行PCR扩增、连接、转化和鉴定,参照OOI等[18]的方法对疑似酵母菌的菌株进行鉴定,将鉴定的阳性克隆子寄往上海桑尼生物科技有限公司进行测序,拼接返回序列后于美国国家生物技术信息中心NCBI比对,最后将所有分离株序列上传至GeneBank数据库,获得细菌登录号分别为OP002178-OP002238,酵母菌登录号分别为OP018542-OP018543。
1.3.4 产气验证实验
a)将分离出的微生物从冻存管中各取200 μL逐一活化在装有5 mL液体培养基(PDA/LB/MRS)试管(10 mm×150 mm)中,分别于25、30、37 ℃下连续传3代后离心,去掉上层清液并在菌泥中添加1 mL浓度为0.85%的生理盐水后重悬待用。
b)取30 mL正常生抽酱油装入一次性透明自立吸嘴包装袋(30 mL),密封后于100 ℃沸水中保持15 min,随后在无菌条件下排尽空气待用。
c)在15 mm×150 mm的试管中装入10 mL液体培养基(PDA/LB/MRS)和排尽气泡的杜氏小管,于121 ℃灭菌15 min,放凉后待用。
验证实验一:参照张小丽等[19]的方法,并稍作修改,取200 μL A中重悬后的菌液在无菌条件下逐一接种于B方式处理后的酱油袋,同时制作不接菌的空白对照,分别在25、30、37 ℃条件下培养10 d,观察是否出现胀袋现象。
验证实验二:参照张小丽等[19]的方法,并稍作修改,取200 μL A中重悬后的菌液在无菌条件下逐一接种于C方式处理后的PDA、LB和MRS液体试管,同时制作不接菌的空白对照,分别于25、30、37 ℃培养10 d并观察结果。
1.3.5 微生物群落结构解析
使用MEGA5.0与R软件的“tidyverse”、“treeio”和“ggtree”包构建系统发育树;使用Origin 2018软件绘制相对含量的柱状图。
2 结果与分析
2.1 基于宏基因组测序技术解析胀袋生抽酱油的微生物类群
本研究首先对2个胀气和2个正常生抽酱油样品微生物基因组进行了提取,但2个正常样品因提取的基因组质量较差无法进行上机建库,究其原因可能与其微生物含量较少有关,因而本研究仅对2个胀袋生抽酱油样品的微生物类群进行了解析。在过滤掉人类基因组和低质量序列后,Q1和Q2样品共产出44 503 218条和47 657 592条序列,测序数据量分别为6.68 G和7.15 G,经比对共鉴定出526个属和1 199个种。平均相对含量>1.0%属和种的柱状图,如图1所示。

A-属;B-种
图1 基于宏基因组学技术胀袋生抽酱油中微生物群落在属和种水平上的构成分析
Fig.1 Analysis of microbial community composition at the genus and species levels in aerogenesis soy sauce based on metagenomics technology
由图1-A可知,胀袋生抽酱油样品中平均相对含量>1.0%的属主要由四链球菌属(Tetragenococcus)、联合乳杆菌属(Ligilactobacillus)、植物乳杆菌属(Lactiplantibacillus)、肠球菌属(Enterococcus)、乳杆菌属(Lactobacillus)、伴生乳杆菌属(Companilactobacillus)、腐败乳杆菌属(Loigolactobacillus)、促生乳杆菌属(Levilactobacillus)、链球菌属(Streptococcus)、魏斯氏菌属(Weissella)和黏液乳杆菌属(Limosilactobacillus)组成,平均相对含量分别为28.29%、27.40%、9.93%、4.81%、4.06%、2.95%、2.73%、1.76%、1.46%、1.41%和1.31%。由图1-B可知,平均相对含量>1.0%的种主要由嗜盐四联球菌(Tetragenococcus halophilus)、酸鱼联合乳杆菌(Ligilactobacillus acidipiscis)、植物乳植物杆菌(Lactiplantibacillus plantarum)、耐酸乳杆菌(Lactobacillus acetotolerans)、棒状腐败乳杆菌(Loigolactobacillus coryniformis)、短促生乳杆菌(Levilactobacillus brevis)和屎肠球菌(Enterococcus faecium)组成,平均相对含量分别为28.01%、25.50%、9.45%、2.49%、1.83%、1.53%和1.33%。值得一提的是,Q1和Q2样品微生物组成和含量存在较大差异,其中T.halophilus在Q1样品中最丰富,占比高达53.37%,而L.acidipiscis在Q2样品中最丰富,占比高达41.99%。ZHANG等[20]的研究表明,T.halophilus与酱油中天冬氨酸和谷氨酸等鲜味化合物的产生呈显著正相关,麻颖垚等[21]通过变性梯度凝胶电泳技术对胀袋酱油中的污染细菌进行了鉴定,亦发现其中存在L.acidipiscis。综上所述,纳入本研究的胀袋生抽酱油样品中可能主要以乳酸菌为主,但具体是由于哪类微生物产气从而引起了涨袋现象尚需进一步研究。并且,宏基因组测序技术是对样本中存在的所有DNA进行测序,其包含了活细胞和死细胞,这对于解析生抽酱油中产气微生物类群具有一定的干扰性,因此本研究进一步采用纯培养技术对样品中微生物菌株进行了分离鉴定。
2.2 基于纯培养技术解析胀袋生抽酱油的微生物类群
因只有2个胀袋生抽酱油样品成功完成了上机测序,本研究进一步使用纯培养技术对其微生物菌株进行了分离鉴定,从2个胀袋生抽酱油样品中共分离鉴定出49株细菌与2株酵母菌,其中细菌分离株的系统发育树如图2所示。
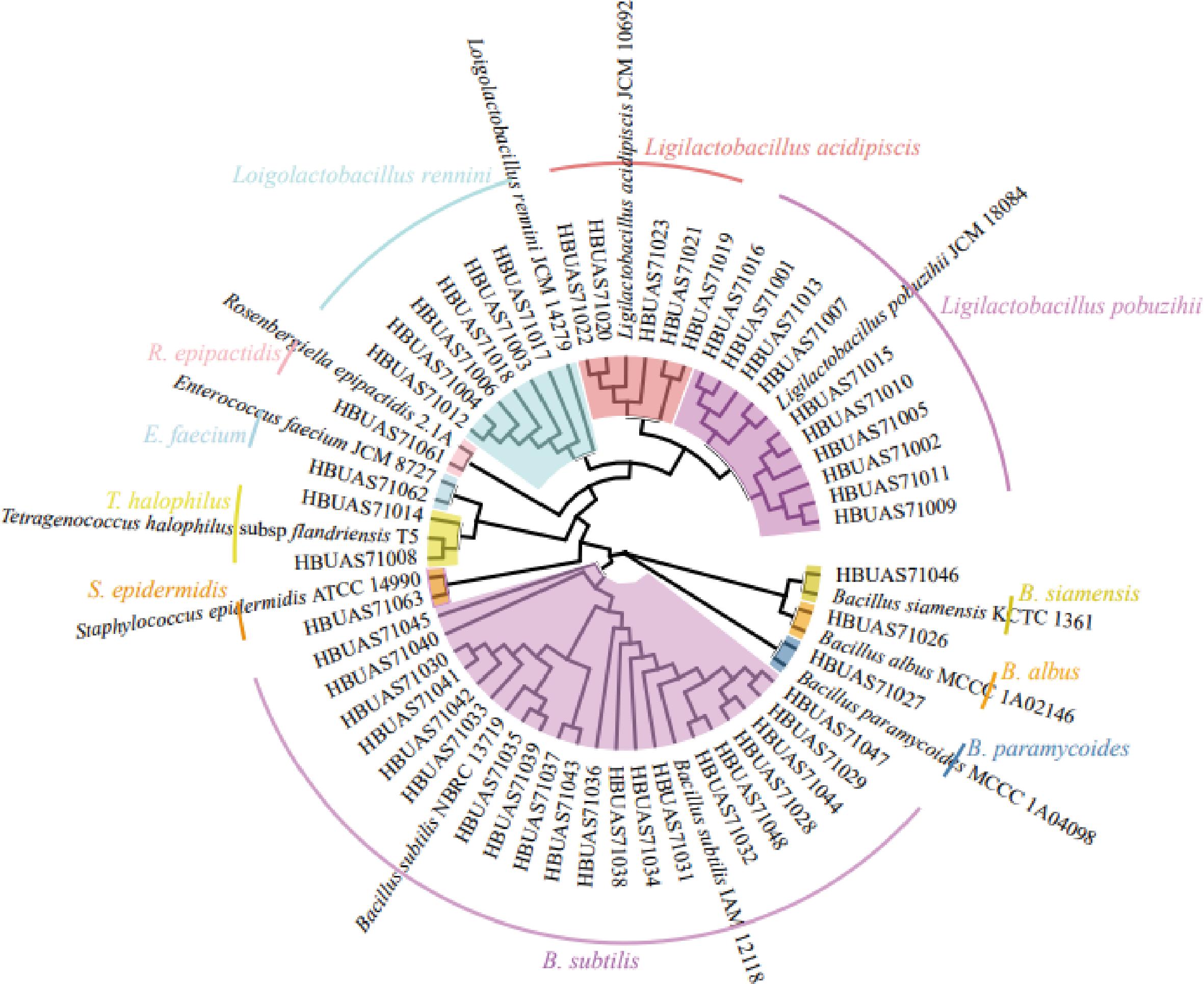
图2 基于16 S rRNA基因序列构建的胀袋生抽酱油中细菌的系统发育树
Fig.2 Phylogenetic tree of bacteria in aerogenesis soy sauce constructed based on 16 S rRNA gene sequences
由图2可知,49株细菌隶属于6个属下的11个种,分别为Ligilactobacillus下的破布子联合乳杆菌(L.pobuzihii)和L.acidipiscis,Loigolactobacillus下的蛋白原酶腐败乳杆菌(L.rennini),Bacillus下的枯草芽孢杆菌(B subtilis)、暹罗芽孢杆菌(B.siamensis)、白色芽孢杆菌(B.albus)和拟蕈状芽孢杆菌(B.paramycoides),Tetragenococcus下的T.halophilus,Rosenbergiella下的罗森伯氏菌(R.epipactidis),Enterococcus下的E.faecium,以及葡萄球菌(Staphylococcus)下的表皮葡萄球菌(S.epidermidis)。由图3亦可知,分别有23个和21个分离株隶属于芽孢杆菌和乳酸杆菌,分别占总分离株的45.10%和42.86%,因而基于纯培养技术,本研究发现胀气生抽中的细菌多由芽孢杆菌和乳酸杆菌组成。李娜[22]运用传统的纯培养技术发现,胀罐酱油中主要的污染菌为B.subtilis、油酸芽孢杆菌(B.oleronius)、弯曲芽孢杆菌(B.flexus)、L.pobuzihii、L.acidipiscis和豌豆乳杆菌(L.piscium),与本研究相比其未分离出隶属于Tetragenococcus、Rosenbergiella、Enterococcus和Staphylococcus的菌株,这可能是与样品采集地和分离方式等存在差异有关。值得一提的是,E.faecium具有较强的环境耐受性,是人类和动物的肠道共生菌[23],S.epidermidis主要存在于皮肤的微环境及粘膜中,属于条件致病菌的一种[24],它们在胀袋生抽酱油中被分离鉴定出,可能是源于酿造车间工人的不规范操作或样品灭菌不彻底。

图3 基于28 S rRNA基因序列构建的胀袋生抽酱油中酵母菌的系统发育树
Fig.3 Phylogenetic tree of yeasts in expanded aerogenesis soy sauce constructed based on 28 S rRNA gene sequences
从胀袋生抽酱油中分离的2株酵母菌系统发育树,如图3所示。
由图3可知,本研究仅从Q2样品中分离出2株酵母菌,经鉴定均为扣囊复膜酵母(Saccharomycopsis fibuligera),隶属于该种的微生物菌株能够从可溶性淀粉中积累海藻糖,并产生淀粉酶、酸性蛋白酶和β-葡萄糖苷酶,在各种发酵食品中,被广泛用作微生物发酵剂[25]。YAN等[26]的研究表明,S.fibuligera在酱油种曲发酵的初始阶段生长,其产生的各种酶有助于葡萄糖的积累,与酱油风味的形成密切相关。
本研究进一步对2份样品中可培养微生物构成情况进行了分析,结果如图4所示。
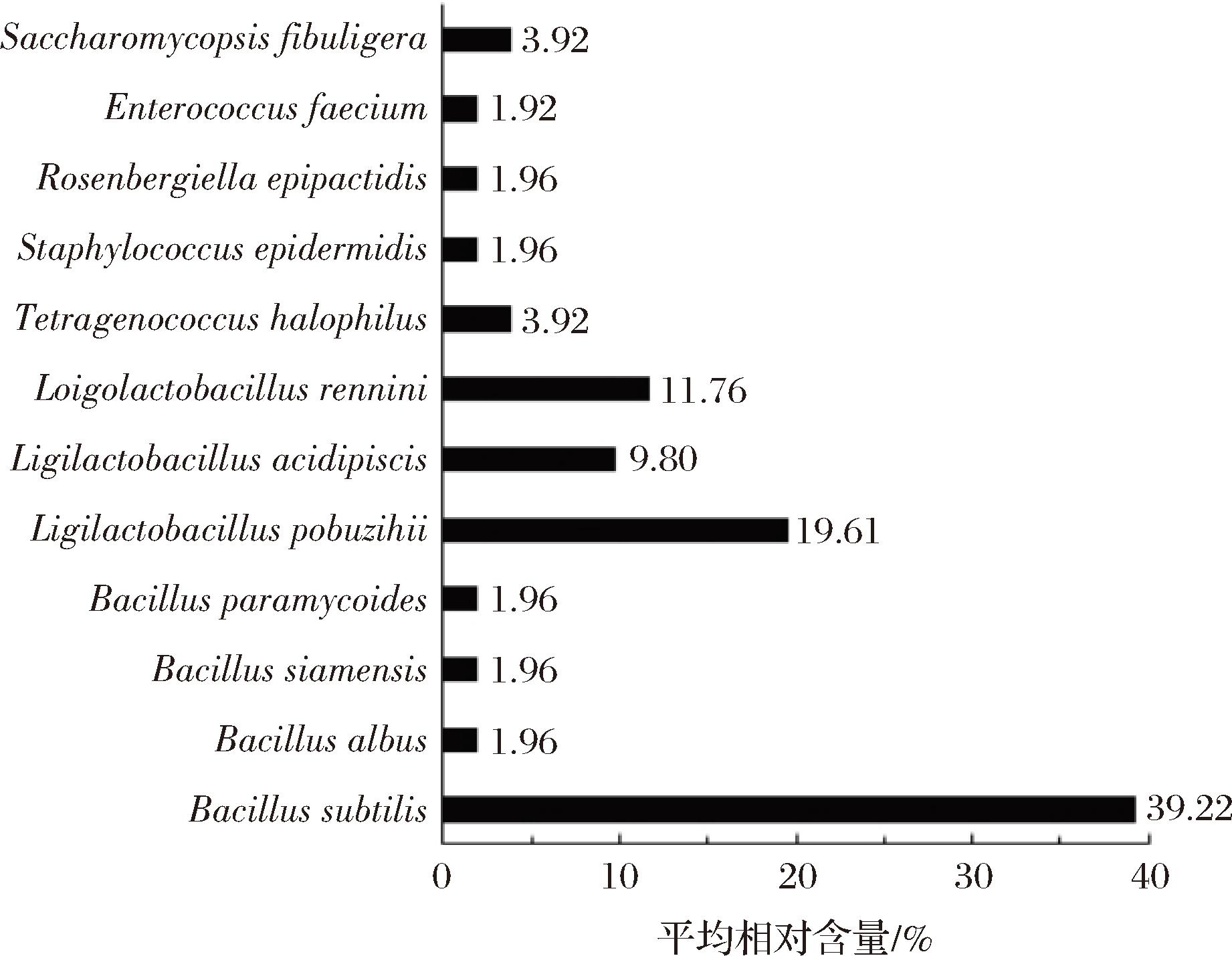
图4 基于纯培养技术胀袋生抽酱油中微生物群落构成分析
Fig.4 Analysis of microbial community composition in expanded aerogenesis soy sauce based on pure culture technique
由图4可知,2份样品中分离最多的菌株为B.subtilis,占分离株总数的39.22%,其次是L.pobuzihii、L.rennini和L.acidipiscis,分别占分离株总数的19.61%、11.76%和9.80%。秦南南等[27]从新疆某品牌的胀袋酱油中分离了2株产气菌,经鉴定分别为B.subtilis和巨大芽孢杆菌(B.megaterium),与本研究结论存在一定的相似性。值得一提的是,采用宏基因组和纯培养技术对胀气酱油微生物类群进行解析时,2种技术获得的结果存在一定的差异性。究其原因可能在于,酱油中部分微生物菌株处于休眠状态,受培养基成分和培养条件等因素的影响,在实验室条件下无法实现其纯培养。此外,宏基因组技术是基于微生物的全基因组DNA进行测序,其无法区分微生物菌株存活状态,因而可能将部分已经死亡的微生物菌株也进行了测序,导致了结果的不准确。
2.3 产气验证
为明确引起生抽酱油胀袋的微生物类群,本研究进一步对51株分离株进行了产气验证实验。通过观察第一个验证试验发现,51个分离株在酱油袋中均有少量淡黄色沉淀产生,但均未出现胀袋现象;通过观察第二个验证实验发现,在所有接菌培养的试管底部均有菌体沉淀产生但杜氏小管内无气泡。由此可见,本研究分离出的微生物菌株均不能引起酱油的胀袋。参照《伯杰细菌鉴定手册》[28]发现B.subtilis在含有葡萄糖的复杂培养基中生长较为缓慢,但在有氧条件下生长旺盛且能产生二氧化碳,手册中亦表明Staphylococcus epidermidis能分解精氨酸产氨、分解半胱氨酸可能产生少量硫化氢。由此可见,本研究的产气实验中隶属于B.subtilis和Staphylococcus epidermidis的分离株均未出现产气现象可能与其培养环境较为不适宜有关,但具体的原因还有待进一步的研究。吴浪涛研究表明,B.subtilis及其亚种(B.subtilis subsp natto)和L.pobuzihii含量较多时对酱油的胀气现象有很大影响[29]。然而,本研究分别接种B.subtilis和Ligilactobacillus pobuzihii后并未出现酱油胀袋。究其原因可能在于:a)本研究并未分离出造成酱油胀袋的微生物菌株,在后续研究中可采用培养组学策略,通过更换培养基、改变培养温度或转换培养条件等方法尽可能多的分离出微生物菌株,对酱油涨袋这一产业问题的解决可能具有积极意义;b)酱油胀袋可能是多菌株协同作用的结果,而本研究接种的菌株均为单一菌株,因而在后续研究中还需进一步考虑多菌株的协同作用;c)除了微生物产气的生物原因外,后续研究中亦需进一步考虑热胀冷缩的物理原因或酸碱反应的化学原因[30]。
3 结论
本研究采用宏基因组测序和纯培养技术对胀袋生抽酱油微生物类群进行了解析,宏基因组测序结果表明胀气酱油中主要微生物为T.halophilus和Ligilactobacillus acidipiscis,两者累计平均相对含量超过了50%。纯培养结果表明,胀气酱油中细菌被鉴定为6个属下的11个种,具有较高的多样性,且以B.subtilis、L.pobuzihii、L.rennini和L.acidipiscis为主,隶属于4个种的分离株占总分离株的80%以上,而酵母菌多样性较低仅以S.fibuligera为主。经验证实验发现,本研究分离的菌株均无法导致生抽酱油涨袋。由此可见,胀袋生抽酱油具有较高的微生物多样性,在后续研究中引入培养组学策略以获得更多的分离株,亦或考虑多菌株的协同作用,对酱油涨袋这一产业问题的解决可能具有积极的意义。
[1] ZHANG X L, LIU Z P, KANG B, et al.Effect of Lactobacillus plantarum or Enterococcus faecalis as co-inoculants with Aspergillus oryzae in koji making on the physicochemical properties of soy sauce[J].Journal of Food Science, 2022, 87(2):714-727.
[2] ZHAO G Z, LIU C, LI S, et al.Exploring the flavor formation mechanism under osmotic conditions during soy sauce fermentation in Aspergillus oryzae by proteomic analysis[J].Food &Function, 2020, 11(1):640-648.
[3] 上官宗渺. 酱油胀壶微生物的分离鉴定和预防措施[J].食品与发酵科技, 2020, 56(6):83-86;107.
SHANGGUAN Z M.Isolation of microorganism of pot-bulged soy sauce and the preventive measures[J].Food and Fermentation Sciences &Technology, 2020, 56(6):83-86;107.
[4] 杨卓, 杨欢岚, 姚泓, 等.基因测序结合传统微生物培养用于胀罐酱油中产气菌的检测[J].食品与发酵工业, 2017, 43(11):197-201.
YANG Z, YANG H L, YAO H, et al.Gene sequencing and traditional microbial culture for the detection of aerogen in swelled packing soy sauce[J].Food and Fermentation Industries, 2017, 43(11):197-201.
[5] 蒋雪薇, 周尚庭, 叶菁, 等.成品变质酱油中微生物的分离鉴定及变质原因分析[J].食品与机械, 2016, 32(2):46-50.
JIANG X W, ZHOU S T, YE J, et al.Identification of contamination microbes separated from finished deterioration sauce and analysis of deterioration reason[J].Food &Machinery, 2016, 32(2):46-50.
[6] 张佳惠, 王芳, 郭树静, 等.山东省酱油微生物污染检测及鉴定[J].食品工业, 2022, 43(6):196-198.
ZHANG J H, WANG F, GUO S J, et al.Detection and identification of microbiological contamination in soy sauce in Shandong Province[J].The Food Industry, 2022, 43(6):196-198.
[7] LESKA A, NOWAK A, MOTYL I.Isolation and some basic characteristics of lactic acid bacteria from honeybee (Apis mellifera L.) environment—A preliminary study[J].Agriculture, 2022, 12(10):1562.
[8] TABLA R, G MEZ A, SIMANCAS A, et al.Enterobacteriaceae species during manufacturing and ripening of semi-hard and soft raw ewe’s milk cheese:Gas production capacity[J].Small Ruminant Research, 2016, 145:123-129.
MEZ A, SIMANCAS A, et al.Enterobacteriaceae species during manufacturing and ripening of semi-hard and soft raw ewe’s milk cheese:Gas production capacity[J].Small Ruminant Research, 2016, 145:123-129.
[9] PAVL K M, FLEISCHER P, FLEISCHER P, et al.Evaluation of the carbon dioxide production by fungi under different growing conditions[J].Current Microbiology, 2020, 77(9):2374-2384.
K M, FLEISCHER P, FLEISCHER P, et al.Evaluation of the carbon dioxide production by fungi under different growing conditions[J].Current Microbiology, 2020, 77(9):2374-2384.
[10] WANG J L, YIN Y N.Clostridium species for fermentative hydrogen production:An overview[J].International Journal of Hydrogen Energy, 2021, 46(70):34599-34625.
[11] ZHEN X S, ZHANG X Y, LI S B, et al.Effect of micro-oxygen pretreatment on gas production characteristics of anaerobic digestion of kitchen waste[J].Journal of Material Cycles and Waste Management, 2020, 22:1852-1858.
[12] SULAIMAN J, GAN H M, YIN W F, et al.Microbial succession and the functional potential during the fermentation of Chinese soy sauce brine[J].Frontiers in Microbiology, 2014, 5:556.
[13] CHEN S F, ZHOU Y Q, CHEN Y R, et al.Fastp:An ultra-fast all-in-one FASTQ preprocessor[J].Bioinformatics, 2018, 34(17):884-890.
[14] WOOD D E, LU J, LANGMEAD B.Improved metagenomic analysis with Kraken 2[J].Genome Biology, 2019, 20(1):1-13.
[15] LU J, BREITWIESER F P, THIELEN P, et al.Bracken:Estimating species abundance in metagenomics data[J].PeerJ Computer Science, 2017, 3:e104.
[16] ZHANG Z D, HOU Q C, WANG Y R, et al.Evaluation of bacterial diversity and evolutionary dynamics of gut Bifidobacterium longum isolates obtained from older individuals in Hubei Province, China[J].Microbiology Spectrum, 2022, 10(1):e01 442-21.
[17] GUO Z, WANG Y R, XIANG F S, et al.Bacterial diversity in pickled cowpea (Vigna unguiculata [linn.] walp) as determined by Illumina MiSeq sequencing and culture-dependent methods[J].Current Microbiology, 2021, 78(4):1286-1297.
[18] OOI T S, TING A S Y, SIOW L F.Influence of selected native yeast starter cultures on the antioxidant activities, fermentation index and total soluble solids of Malaysia cocoa beans:A simulation study[J].LWT, 2020, 122:108977.
[19] 张小丽. 胀袋酱油中产气微生物的分离、鉴定、与特性研究[D].杭州:浙江工商大学, 2015.
ZHANG X L.Isolation, identification and characteristics of aerogen from explosion bags of soy sauce[D].Hangzhou:Zhejiang Gongshang University, 2015.
[20] ZHANG L J, ZHANG L, XU Y.Effects of Tetragenococcus halophilus and Candida versatilis on the production of aroma-active and umami-taste compounds during soy sauce fermentation[J].Journal of the Science of Food and Agriculture, 2020, 100(6):2782-2790.
[21] 麻颖垚, 郭天文, 岳跃成, 等.胀袋酱油中污染细菌的鉴定分析[J].中国调味品, 2020, 45(1):46-50.
MA Y Y, GUO T W, YUE Y C, et al.Identification and analysis of contaminated bacteria in bag-bulged soy sauce[J].China Condiment, 2020, 45(1):46-50.
[22] 李娜. 胀罐酱油污染微生物的分离鉴定与性质研究[D].广州:华南理工大学, 2015.
LI N.Separation, identification, and characterization of contaminant microorganism in swollen canned soy sauces[D].Guangzhou:South China University of Technology, 2015.
[23] ZHOU X W, WILLEMS R J L, FRIEDRICH A W, et al.Enterococcus faecium:from microbiological insights to practical recommendations for infection control and diagnostics[J].Antimicrobial Resistance &Infection Control, 2020, 9(1):130.
[24] MÉRIC G, MAGEIROS L, PENSAR J, et al.Disease-associated genotypes of the commensal skin bacterium Staphylococcus epidermidis[J].Nature Communications, 2018, 9(1):5034.
[25] LEE S M, JUNG J H, SEO J A, et al.Bioformation of volatile and nonvolatile metabolites by Saccharomycopsis fibuligera KJJ81 cultivated under different conditions-Carbon sources and cultivation times[J].Molecules, 2018, 23(11):2762.
[26] YAN Y Z, QIAN Y L, JI F D, et al.Microbial composition during Chinese soy sauce koji-making based on culture dependent and independent methods[J].Food Microbiology, 2013, 34(1):189-195.
[27] 秦南南, 夏俊芳, 沈玉敏, 等.胀袋酱油中产气微生物筛选鉴定及特性研究[J].中国调味品, 2018, 43(4):9-14.
QIN N N, XIA J F, SHEN Y M, et al.Screening and identification of gas-producing microorganisms in bulge-bag soy sauce and research on their characteristics[J].China Condiment, 2018, 43(4):9-14.
[28] BUKANNKNRE, JIBENSINE.伯杰氏系统细菌学手册[M].北京:科学出版社, 1984.
BUKANNKNRE, JIBENSINE.Bergey′s Manual of Determinative Bacteriology[M].Beijing:Science Press, 1984.
[29] 吴浪涛, 房峻, 吴新, 等.酱油变质微生物因素及乳酸菌在防腐中的应用[J].中国酿造, 2021, 40(7):22-27.
WU L T, FANG J, WU X, et al.Microbiological factors of spoilage in soy sauce and application of lactic acid bacteria in preventing spoilage[J].China Brewing, 2021, 40(7):22-27.
[30] 张洁. 酱油中产气代表性微生物的筛选和检测方法的建立[J].现代食品, 2020, 6(18):217-219.
ZHANG J.Screening and detection of representative microorganisms producing gas in soy sauce[J].Modern Food, 2020, 6(18):217-219.