中药肉桂为樟科的植物肉桂(Cinnamomum cassia Presl)干燥树皮,以“牡桂”之名始载于《神农本草经》,列为上品。肉桂自古药食同源,其性辛、甘、大热。归肾、脾、心、肝经,具补火助阳、引火归元、散寒止痛、温通经脉等作用[1]。现代药理学研究证明肉桂具有镇痛、抗菌、抗肿瘤的作用,对消化系统、心血管系统和免疫系统疾病也有良好的疗效[2-7]。2018年,中华人民共和国国家卫生健康委员会将肉桂列入了《按照传统既是食品又是中药材的物质目录》的征求意见稿中。2021年,广西壮族自治区中医药管理局将肉桂确定为“桂十味”道地药材品种。广西作为肉桂药材的道地产区,其主产区主要在广西防城港市、东兴市、玉林市和贵港市等。肉桂作为药食两用中药材,其相关产品涉及医药、食品、日用品、农药等多个领域,是国际上重要的药用植物品种[8]。肉桂用量大、栽种时间长、价格高、货源紧缺,因此在广西市场上常出现以肉桂同科植物阴香(Cinnamomum burmanni)冒充肉桂出售,其在外观、性状、颜色及气味等方面特征都与肉桂相似,容易造成混用的现象[9-10]。目前鉴别肉桂和阴香主要是通过性状、组织结构、形态特征、理化性质等传统鉴别方法[11-14]。这些方法易受样品形式、组织来源、贮存条件和人员操作等因素的影响,存在人为主观因素强、实验稳定性差等弊端,尤其是当肉桂和阴香经研磨成粉后,传统的鉴别方式很难将二者区别。因此需要建立一种高效、便捷、精确的区分肉桂和阴香的方法。
近年来,随着分子生物学技术在中药学领域的渗透与发展,基于DNA分子生物技术的分子鉴定方法称为中药4大鉴定方法(基原鉴定、性状鉴定、显微鉴定、理化鉴定)的重要补充,得到了不断的发展与应用。作为实用性强的分子生物技术,不仅具有鉴定方法不受个体形态特征限制、不受个体发育阶段影响,从基因水平上能客观区分物种的优势,还具有操作方法相对简便等优势。本研究通过提取不同产地的肉桂和阴香的基因组DNA,使用psbA-trnH引物扩增后测序,通过查找两者的psbA-trnH序列差异,设计并筛选出高特异性的鉴别引物,建立了可区别两者的聚合酶链式反应(polymerase chain reaction,PCR)体系,为药食同源物种肉桂的质量评价提供了新的DNA分子标记方法。
1 材料与方法
1.1 材料与试剂
肉桂药材的道地产区在广西,故本实验的样品均采集于广西区内,主要来自企业种植基地、普通农户自家田地散种以及野外采集。样品经广西食品药品检验所黄清泉主管药师鉴定,硅胶干燥,阴干保存,存放于广西食品药品检验所标本室,样品具体信息见表1。
表1 肉桂及其阴香的样品采集信息表
Table 1 Sample collection information table of Cinnamomum cassia and Cinnamomum burmanni

物种名称拉丁女学名样品编号采集地肉桂Cinnamomum cassiaR1来宾市金秀县金秀镇老山界肉桂Cinnamomum cassiaR2来宾市金秀县罗香乡肉桂Cinnamomum cassiaR3来宾市金秀县六巷乡肉桂Cinnamomum cassiaR4玉林市六万大山林场肉桂Cinnamomum cassiaR5玉林市大容山林场肉桂Cinnamomum cassiaR6玉林市北流市山围镇丰垌村肉桂Cinnamomum cassiaR7防城港市那梭镇肉桂Cinnamomum cassiaR8防城港市防城区肉桂Cinnamomum cassiaR9东兴市马路镇肉桂Cinnamomum cassiaR10东兴市马路镇肉桂Cinnamomum cassiaR11贵港市平南市马练镇马练村肉桂Cinnamomum cassiaR12靖西市五岭森林公园阴香Cinnamomum burman-niY1玉林市市区景观树阴香Cinnamomum burman-niY2玉林市大容山林场阴香Cinnamomum burman-niY3玉林市大容山林场阴香Cinnamomum burman-niY4防城港市滑石镇阴香Cinnamomum burman-niY5防城港市防城区阴香Cinnamomum burman-niY6来宾市金秀县金秀镇老山界阴香Cinnamomum burman-niY7来宾市金秀县桐木镇
琼脂糖,BIOWEST公司(批号:111885);GelRed,BIOTIUM公司(批号:41003);PrimeSTAR Max DNA Polymerase、DNA Marker,TAKARA公司(批号:AL52852A、AI5230SA);2X F8 LongFast PCR MasterMix,艾德莱公司(批号:331523AX);KOD OneTM PCR Master Mix,Toyobo公司(批号:041500);Platinu Ⅱ Hot-Start Green PCR Master Mix,ThermoFisher公司 (批号:01147162);Q5酶,NEW ENGLEND公司(批号:0161512);新型植物组织基因组DNA提取试剂盒,天根公司(批号:W0229);引物由华大基因合成,其他试剂均为国产分析纯。
1.2 仪器与设备
S1000梯度PCR扩增仪,美国Bio-Rad公司;TA96SG梯度PCR扩增仪,德国耶拿公司;Mini-sub cell GT琼脂糖凝胶电泳仪,美国Bio-Rad公司;Nano Value PLUS微量紫外分光光度计,Cytiva公司;GelDoc XR+全自动凝胶成像系统,美国BIO-RAD公司;ME203型电子天平,Mettler Toledo公司;MIKRO220R高速冷冻离心机,Hettich公司;XW-80A漩涡振荡仪,Kylin-Bell Lab Instruments公司。
1.3 实验方法
1.3.1 DNA提取
取适量样品用75%(体积分数)乙醇擦拭表面,晾干,用球磨仪研磨成粉末。取约20 mg,采用天根新型植物基因组DNA提取试剂盒提取样品的DNA,然后使用微量紫外分光光度计测定DNA的浓度和纯度,合格的DNA样品于-20 ℃保存备用。
1.3.2 PCR扩增及产物测序
利用植物药DNA条形码通用引物psbA-trnH序列对提取的DNA进行扩增并测序。PCR扩增反应体系总体积为20 μL,其中2X F8 LongFast PCR MasterMix 10.0 μL,psbA-trnH上下游引物(10 μmol/L)各0.5 μL,DNA模板1.0 μL,灭菌蒸馏水8.0 μL。PCR扩增程序:95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸30 s,运行35个循环;72 ℃延伸10 min。将扩增所得产物送至测序公司进行双向测序。
1.3.3 psbA-trnH序列分析
将测得的序列去除测序低质量数据,使用BioEdit软件对正向及反向序列进行拼接。使用MEGA 6.0软件对肉桂及其阴香的拼接序列进行对比分析,寻找可供特异性引物设计的单核苷酸多态性(single nucleotide polymorphism,SNP)位点。通过图1的序列比对发现,肉桂和阴香基本上是不连续的单碱基差异,在第34个碱基至第35个碱基处,肉桂为AA,阴香为GT;在第37个碱基至第38个碱基处,肉桂为AG,阴香为CT;在第40个碱基至第41个碱基处,肉桂为AC,阴香为TT。
图1 肉桂及阴香的psbA-trnH序列比对
Fig.1 psbA-trnH sequences alignment of Cinnamomum cassia and Cinnamomum burmanni
1.3.4 特异性引物设计
根据肉桂及阴香在叶绿体psbA-trnH基因序列中发现的特异性位点,使用引物设计软件Primer Premier 5分别设计肉桂及阴香的特异性引物。引物序列信息见表2。
表2 特异性引物信息表
Table 2 Specific primers information table
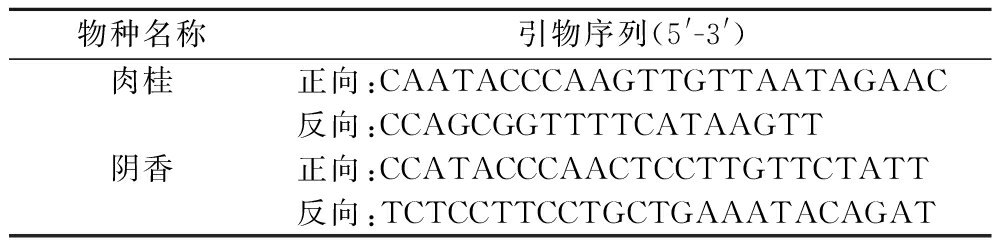
物种名称引物序列(5'-3')肉桂正向:CAATACCCAAGTTGTTAATAGAAC反向:CCAGCGGTTTTCATAAGTT阴香正向:CCATACCCAACTCCTTGTTCTATT反向:TCTCCTTCCTGCTGAAATACAGAT
2 结果与分析
2.1 PCR反应条件的筛选与优化
2.1.1 退火温度考察
使用艾德莱2X F8 LongFast PCR MasterMix酶,按PCR扩增反应体系,退火温度分别考察了50、52、54、56、58、60 ℃。经试验,如图2所示,当退火温度由50 ℃升高至60 ℃后,肉桂和阴香经肉桂引物扩增后,只有在54 ℃时肉桂在100~200 bp出现单一扩增条带,而阴香没有扩增条带,其他温度下扩增阴香均有干扰;因此选择54 ℃作为肉桂引物扩增时的最佳退火温度。另外,2个品种经阴香引物扩增后,在56 ℃时阴香在200~300 bp有明显的单一扩增条带, 而肉桂没有扩增条带,当温度进一步升高,条带亮度减弱,因此阴香引物扩增时的最优退火温度为56 ℃。
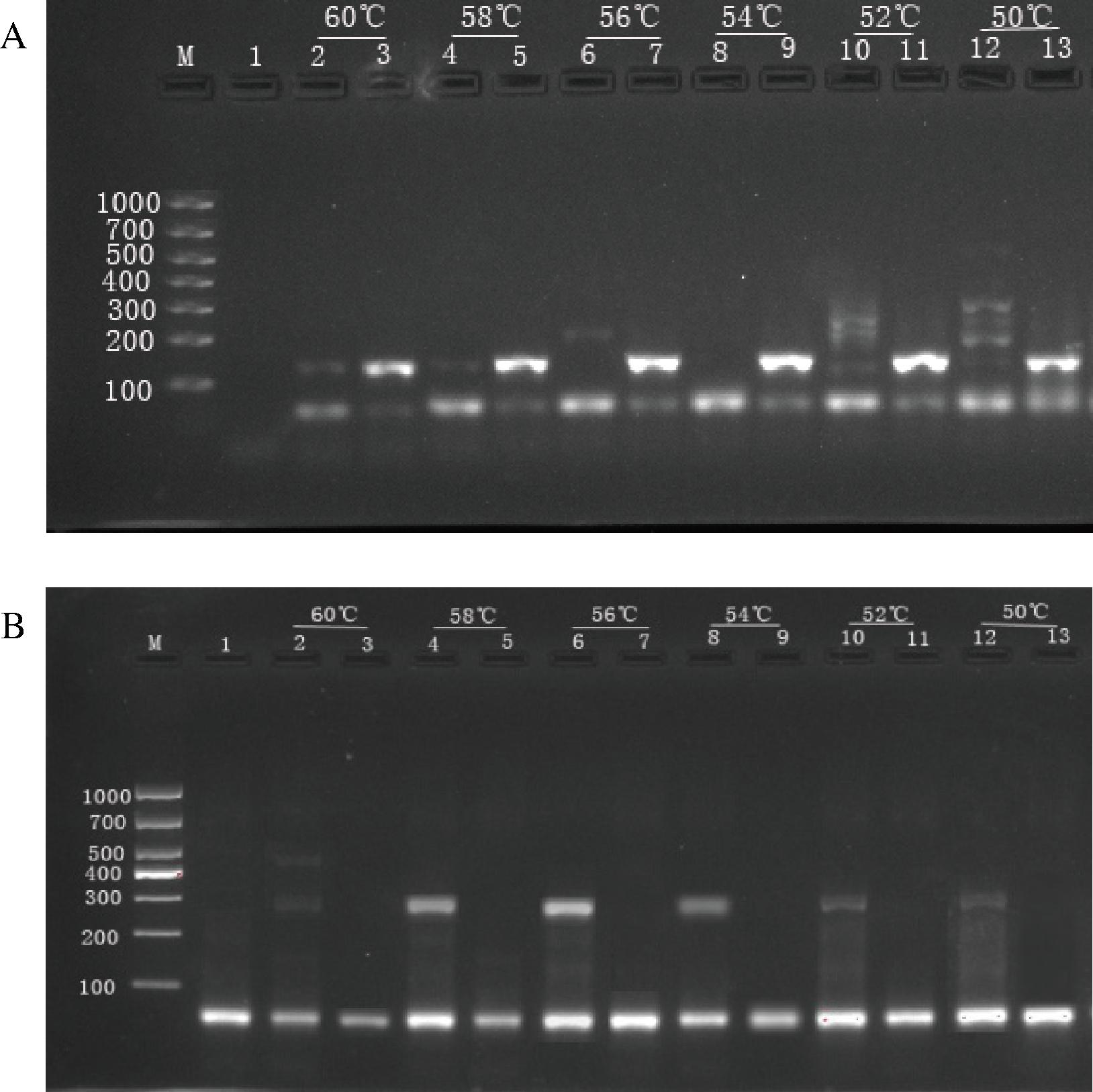
M-D1000 DNA Marker;1-空白对照;2、4、6、8、10、12-阴香药材;3、5、7、9、11、13-肉桂药材
A-肉桂引物退火温度考察;B-阴香引物退火温度考察
图2 不同退火温度条件下凝胶电泳检测结果
Fig.2 Gel electrophoresis test results at different annealing temperatures
2.1.2 反应循环数考察
如图3和图4所示,肉桂引物在35个循环的时候得到明显亮度的单一条带,阴香引物在40个循环出现明显亮度的单一条带,最终确定肉桂引物的循环数为35,阴香引物的循环数为40。
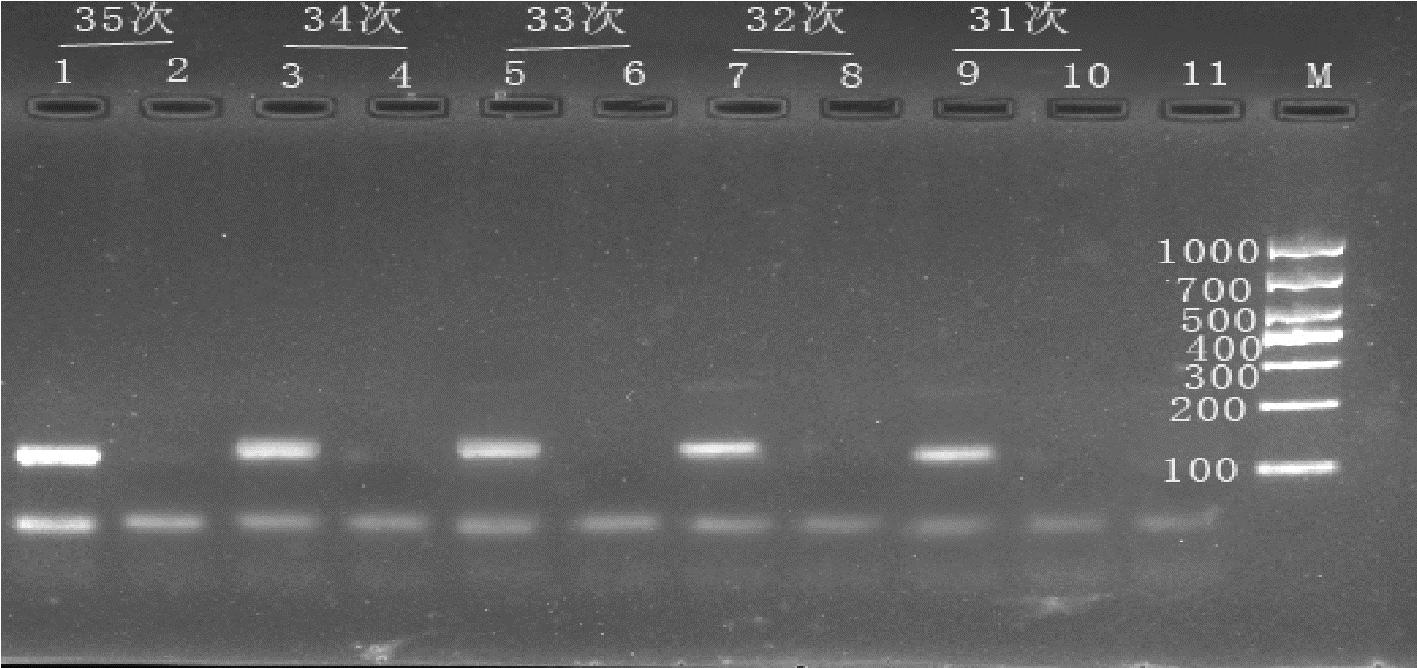
M-D1000 DNA Marker;11-空白对照;2、4、6、8、10-阴香药材;1、3、5、7、9-肉桂药材
图3 肉桂引物不同循环次数条件下凝胶电泳检测结果
Fig.3 Gel electrophoresis test results at different cycle numbers of Cinnamomum cassia primer
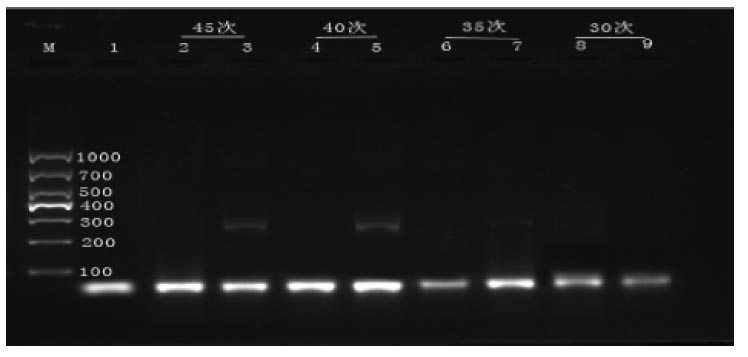
M-D1000 DNA Marker;1-空白对照;2、4、6、8-肉桂药材;3、5、7、9-阴香药材
图4 阴香引物不同循环次数条件下凝胶电泳检测结果
Fig.4 Gel electrophoresis test results at different cycle numbers of Cinnamomum burmanni primer
2.1.3 DNA模板量的考察
本实验考察了1、2、5、10、20、40 ng的DNA模板量,结果显示(图5、图6),肉桂引物在加入模板量为5 ng时有隐约可见的条带,加入量为20 ng时条带亮度明显;阴香引物在模板加入量为20 ng时才出现条带,40 ng时条带亮度明显。
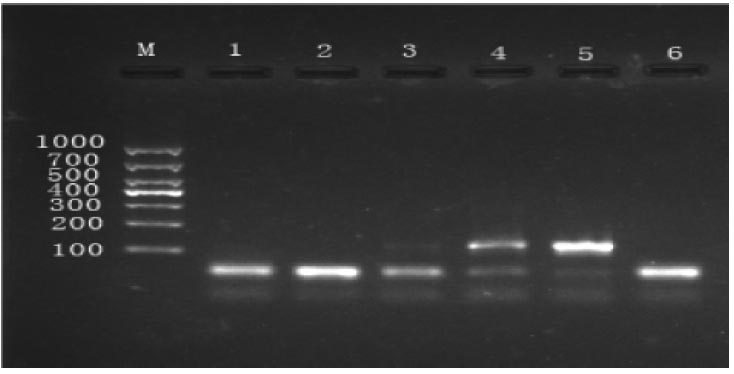
M-D1000 DNA Marker;1~5-1、2、5、10、20 ng;6-空白对照
图5 肉桂引物不同模板量下凝胶电泳检测结果
Fig.5 Gel electrophoresis test results at different template amounts of Cinnamomum cassia primer
M-D1000 DNA Marker;1~6-40、20、10、5、2、1 ng;7-空白对照
图6 阴香引物不同模板量下凝胶电泳检测结果
Fig.6 Gel electrophoresis test results at different template amounts of Cinnamomum burmanni primer
M-D1000 DNA Marker;1-空白对照;2-阴香药材粉末;3~7-肉桂和阴香混合粉末(阴香质量分数依次为50%、20%、10%、5%、1%)
图7 肉桂PCR掺伪检出限
Fig.7 The detection limit of adulteration in Cinnamomum cassia by PCR
2.1.4 掺伪检出限的考察
制备阴香质量分数分别为1%、5%、10%、20%、50%的肉桂和阴香混合粉末样品,提取基因组DNA后进行PCR实验,结果显示(图7),阴香质量分数为10%以上时出现阴香特异性条带,说明混合样品中阴香检出限为10%。
2.2 耐受性考察
2.2.1 引物酶的种类考察
本实验采用2X F8 LongFast PCR MasterMix(艾德莱)、PrimeSTAR Max DNA Polymerase(TAKARA)、KOD OneTM PCR Master Mix(Toyobo)等不同品牌的酶对鉴别反应结果影响的检测。结果表明(图8、图9),3种聚合酶都可扩增出肉桂和阴香的特异性条带,可鉴别出肉桂和阴香。阴香引物用KOD OneTM PCR Master Mix(Toyobo)牌子的酶扩增出的条带特别亮,可考虑用该引物酶进行扩增。
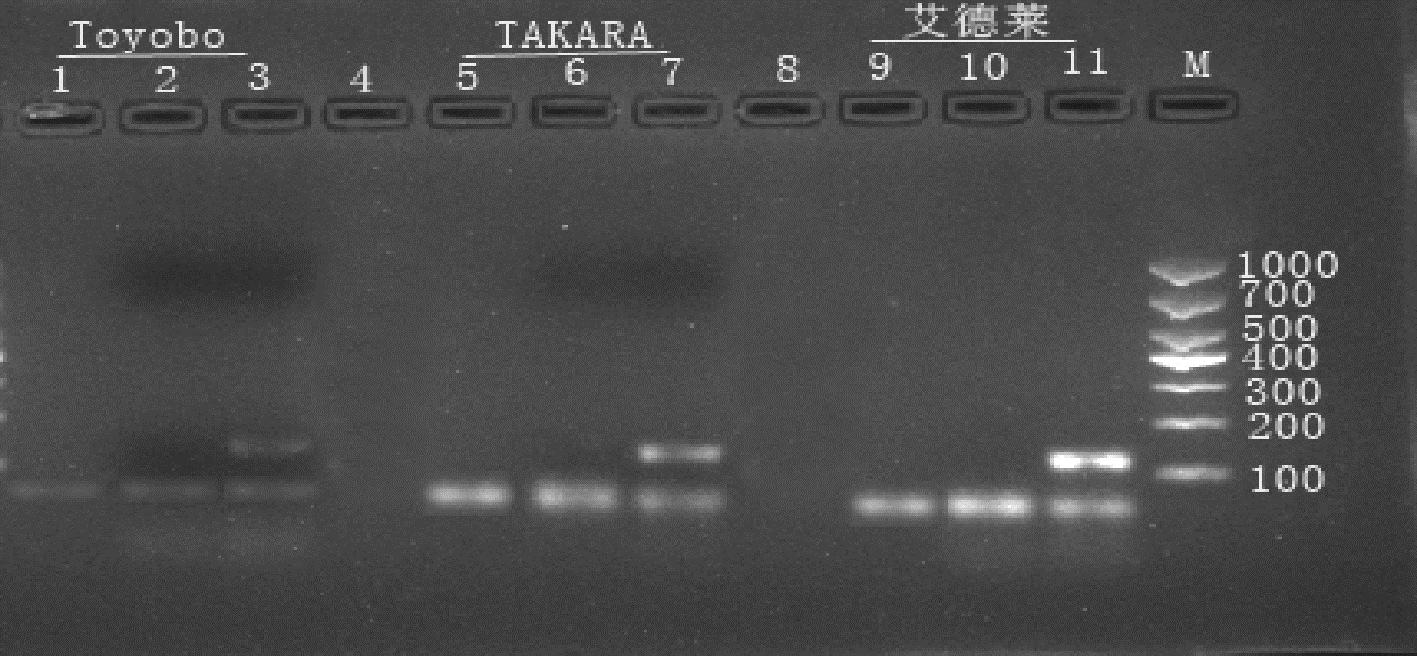
M-D1000 DNA Marker;1、5、9-空白对照;2、6、10-阴香药材;3、7、11-肉桂药材;4、8-空泳道
图8 肉桂引物不同引物酶条件下凝胶电泳检测结果
Fig.8 Gel electrophoresis test results at different primase of Cinnamomum cassia primer

M-D1000 DNA Marker;1、5、9-空白对照;2、6、
10-肉桂药材;3、7、11-阴香药材;4、8-空泳道
图9 阴香引物不同引物酶条件下凝胶电泳检测结果
Fig.9 Gel electrophoresis test results at different primase of Cinnamomum burmanni primer

M-D1000 DNA Marker;1、3、5、7-空白对照;2、4-肉桂药材;6、8-阴香药材
A-肉桂引物不同PCR仪考察;B-阴香引物不同PCR仪考察
图10 不同PCR仪条件下凝胶电泳检测结果
Fig.10 Gel electrophoresis test results at different PCR machines
2.2.2 PCR仪器品牌考察
本实验采用美国ABI veriti梯度PCR扩增仪和德国耶拿TA96SG梯度PCR扩增仪对建立的PCR体系的耐受性进行考察,结果均能得到很好的扩增效果(图10),说明所建立的PCR鉴别体系耐受性良好。
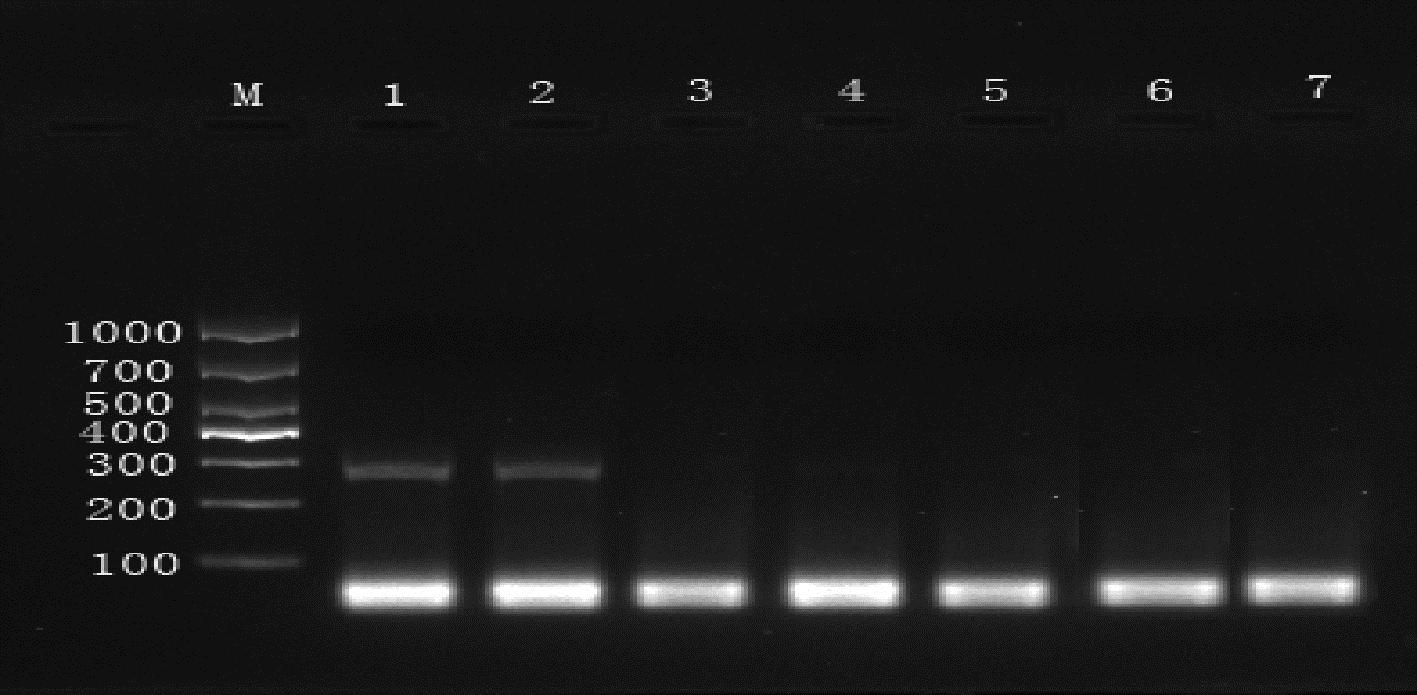
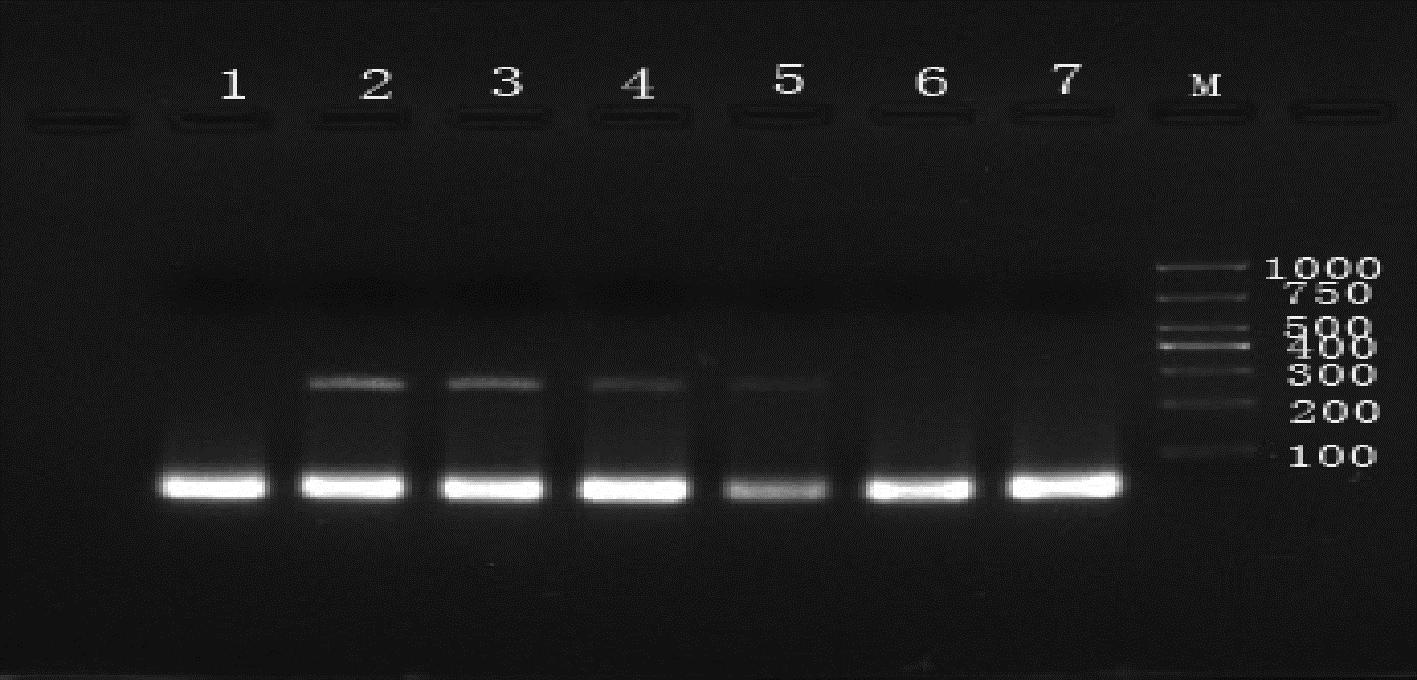
2.2.3 适用性及专属性考察
利用优化好的实验条件,对所有采集到的肉桂及阴香进行验证,结果显示(图11、图12),肉桂经肉桂引物扩增后在100~200 bp处出现特异性条带,阴香经阴香引物扩增后在200~300 bp处出现特异性条带,表明本方法具有良好的特异性,能达到对2个品种进行鉴别。
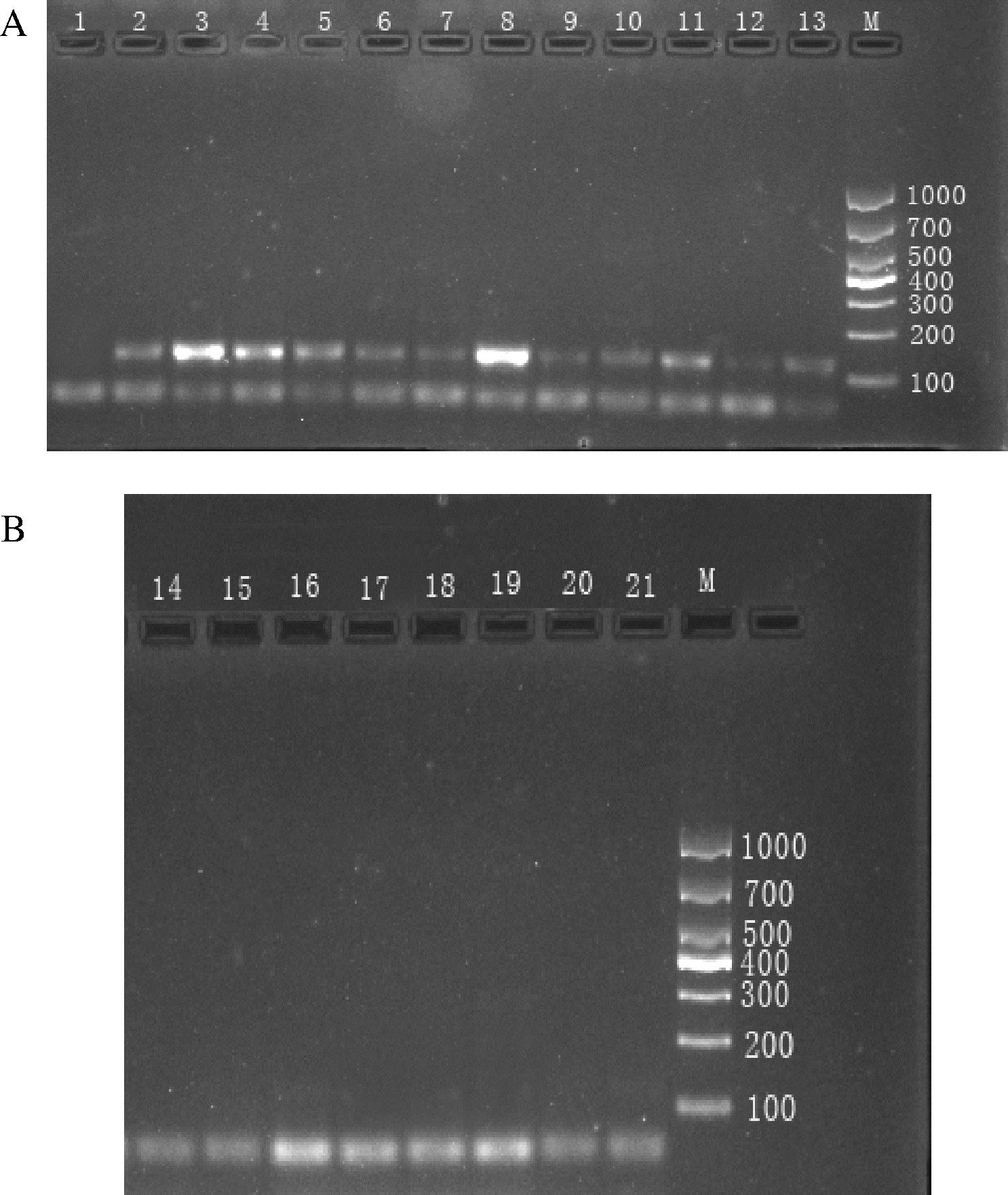
M-D1000 DNA Marker;1、14-空白对照;2~13-肉桂药材;15~21-阴香药材
A-肉桂药材PCR扩增产物凝胶电泳检测结果;B-阴香药材PCR扩增产物凝胶电泳检测结果
图11 肉桂及阴香药材凝胶电泳检测结果(肉桂引物)
Fig.11 Gel electrophoresis test results of Cinnamomum cassia and Cinnamomum burmanni (Cinnamomum cassia primer)
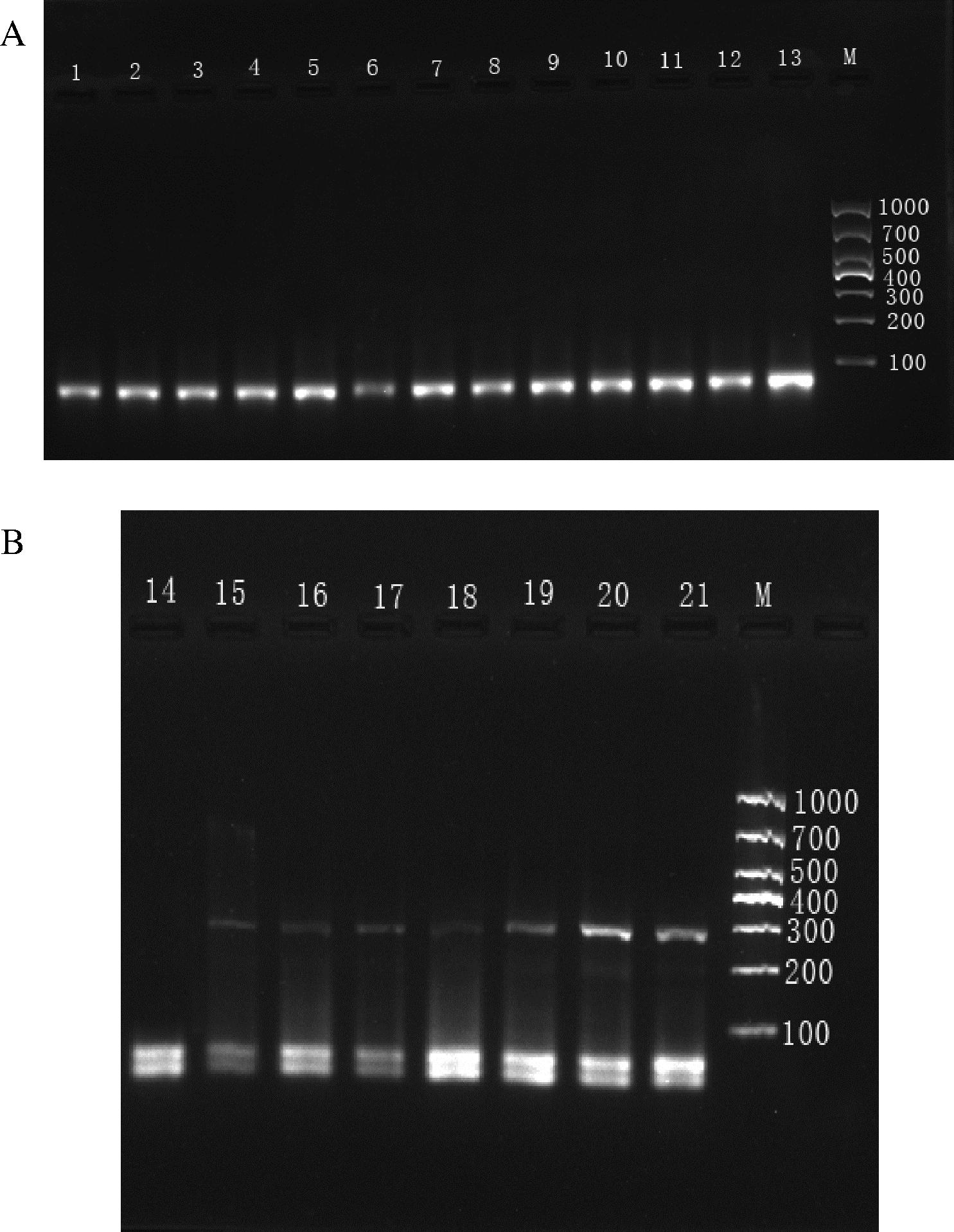
M-D1000 DNA Marker;1、14-空白对照;2~13-肉桂药材;15~21-阴香药材
A-肉桂药材PCR扩增产物凝胶电泳检测结果;B-阴香药材PCR扩增产物凝胶电泳检测结果
图12 肉桂及阴香药材凝胶电泳检测结果(阴香引物)
Fig.12 Gel electrophoresis test results of Cinnamomum cassia and Cinnamomum burmanni (Cinnamomum burmanni primer)
3 结论与讨论
近年来有文献报道采用分子生物学技术对肉桂及其近似物种进行研究。杨培等[15]通过叶绿体psbA-trnH条形码序列对肉桂及其近似品种的物种内及物种间的遗传关系进行了研究,成功构建了鉴别肉桂及其近似品种的系统发育树。但利用DNA条形码鉴别的弊端在于每次都需要对分析样品进行测序分析,且相近类群单位点的DNA条形码缺乏足够变化,所以不能准确鉴别所有种。DOH等[16]比较了来自越南、中国、日本的肉桂、锡兰肉桂和香樟等的DNA内部转录间隔区的核苷酸序列,可用于肉桂皮、桂枝、烟仔桂植物基原的鉴定,但未能对肉桂和阴香的植物基原进行鉴定。
本实验通过采用植物组织基因组DNA提取试剂盒提取,提取的DNA浓度高,纯度佳,保证了DNA模板的质量。引物设计是整个研究的关键点,直接关系到目标物种是否能够成功检出。本实验选取肉桂和阴香的psbA-trnH基因序列作为靶基因,利用MEGA 6.0软件对其基因序列进行序列同源性比较分析,寻找特异性SNP位点,利用Primer Premier 5分别设计肉桂及阴香的特异性引物,并通过考察退火温度、反应循环数、引物酶等获得各自最优的PCR反应体系。结果显示,肉桂经肉桂引物扩增后在100~200 bp处出现特异性条带,阴香无条带;阴香经阴香引物扩增后在200~300 bp处出现特异性条带,肉桂无条带。实验结果不仅可在普通的琼脂糖凝胶上分辨,还避免了因香辛料加工过程中DNA降解导致的扩增效率降低,提高了鉴定结果的准确性。
与其他分子标记方法相比,SNP方法因其简便快捷、灵敏度高的优点已在中药材的真伪鉴别中有应用[17-23]。本研究利用了基于SNP位点建立的PCR体系实现了肉桂和阴香的鉴别,在检测样品时无需再对样品进行测序,可以在较短时间内完成大批量样品的快速鉴别,具有稳定性好、准确度高、特异性强等特点,为保障肉桂的药材质量,肉桂初加工及深加工产品的植物来源提供了有力的技术支持。
[1] 国家药典委员会. 中华人民共和国药典-一部[M]. 北京: 中国医药科技出版社, 2020: 142-143.
Chinese Pharmacopoeia Commission. Pharmacopoeia of the People′s Republic of China (Part 1) [M]. Beijing: China Medical Science Press, 2020: 142-143.
[2] 李艳, 苗明三. 肉桂的化学、药理及应用特点[J]. 中医学报, 2015, 30(9):1335-1337.
LI Y, MIAO M S. Discussion on the chemical, pharmacology and application characteristics of cinnamon[J]. China Journal of Chinese Medicine, 2015, 30(9):1335-1337.
[3] 吴修富. 肉桂提取物的主要化学成分及药理活性研究进展[J]. 中国药房, 2015, 26(24):3454-3456.
WU X F. Research progress on main chemical constituents and pharmacological activities of Cinnamomum cassia extract[J]. China Pharmacy, 2015, 26(24):3454-3456.
[4] 陈菊莲. 中药肉桂与食用肉桂的对比及鉴别分析[J]. 世界最新医学信息文摘, 2018, 18(54):96.
CHEN J L. Comparison and identification analysis of Chinese medicine cinnamon and edible cinnamon[J]. World Latest Medicine Information, 2018, 18(54):96.
[5] 伍彩红, 舒眉, 李倩, 等. 广东、广西产肉桂资源调查研究[J]. 现代中药研究与实践, 2017, 31(5):14-17, 21.
WU C H, SHU M, LI Q, et al. Investigation on Cinnamomum cassia presl resource in Guangdong and Guangxi[J]. Research and Practice on Chinese Medicines, 2017, 31(5):14-17; 21.
[6] ZHANG C L, FAN L H, FAN S M, et al. Cinnamomum cassia presl: A review of its traditional uses, phytochemistry, pharmacology and toxicology[J]. Molecules, 2019, 24(19):3473.
[7] TAN H Y, NABIL W N N, XU H X. Clinical research on cinnamomi cortex: A scoping review[J]. Chinese Medicine and Culture, 2021, 4(4):243-250.
[8] 焦少珍. 世界桂皮生产、消费和贸易格局分析[J]. 世界农业, 2016(8):124-129.
JIAO S Z. Analysis on the production, consumption and trade pattern of cinnamon in the world[J]. World Agriculture, 2016(8):124-129.
[9] 肖培根. 新编中药志-第三卷[M]. 北京: 化学工业出版社, 2002: 575.
XIAO P G. Modern Chinese Materia Medica (Volume 3)[M]. Beijing: Chemical Industry Press, 2002: 575.
[10] 中国科学院植物研究所. 中国高等植物图鉴-第二册, II[M]. 北京: 科学出版社, 1972: 454.
Institute of Botany, Chinese Academy of Sciences. Higher Plants of China (Volume 2)[M]. Beijing: Science Press, 1972: 454.
[11] 安维杰. 肉桂及其常见伪品的性状鉴别[J]. 内蒙古中医药, 2011, 30(3):79-80.
AN W J. Character identification of Cinnamomum cassia and its common counterfeits[J]. Nei Mongol Journal of Traditional Chinese Medicine, 2011, 30(3):79-80.
[12] 庞运同, 董元玉. 肉桂及其伪品的鉴别[J]. 中国医院药学杂志, 2004, 24(12): 788-789.
PANG Y T, DONG Y Y. Identification of Cinnamomum cassia and its adulterants[J]. Chinese Journal of Hospital Pharmacy, 2004, 24(12): 788-789.
[13] 邱燕祥, 严娅娟, 张丹雁, 等. 肉桂及其常见混淆品的鉴别研究[J]. 安徽农业科学, 2015, 43(17):299-301; 316.
QIU Y X, YAN Y J, ZHANG D Y, et al. Study on identification of Cinnamomum cassia presl and its adulterants[J]. Journal of Anhui Agricultural Sciences, 2015, 43(17):299-301; 316.
[14] 徐洋洋, 王添敏, 初正云, 等. HPLC-DAD测定5种商品规格肉桂及两种伪品中桂皮醛的含量[J]. 中国实验方剂学杂志, 2014, 20(1):90-93.
XU Y Y, WANG T M, CHU Z Y, et al. Determination of cinimaldehyde in 5 different standards of Cinnamomum cassia and two kinds of adulterants by HPLC-DAD[J]. Chinese Journal of Experimental Traditional Medical Formulae, 2014, 20(1):90-93.
[15] 杨培, 周红, 马双姣, 等. 叶绿体psbA-trnH序列鉴定药食同源肉桂类药材[J]. 中国药学杂志, 2015, 50(17):1496-1499.
YANG P, ZHOU H, MA S J, et al. Authentication of raw material for edible and medicinal cinnamon based on plastid intergenic region psbA-trnH[J]. Chinese Pharmaceutical Journal, 2015, 50(17):1496-1499.
[16] DOH E J, KIM J H, OH S E, et al. Identification and monitoring of Korean medicines derived from Cinnamomum spp. by using ITS and DNA marker[J]. Genes &Genomics, 2017, 39(1):101-109.
[17] 李慧, 钱润, 田娜, 等. 红天麻、乌天麻及其杂交天麻的PCR鉴别[J]. 中国中药杂志, 2020, 45(15):3666-3671.
LI H, QIAN R, TIAN N, et al. Identification of Gastrodia elata and its hybrid by polymerase chain reaction[J]. China Journal of Chinese Materia Medica, 2020, 45(15):3666-3671.
[18] 衣洁菡, 李燕青, 孙珂, 等. 山豆根及其混伪品的DNA分子标记鉴定方法[J]. 中药材, 2022, 45(5):1076-1080.
YI J H, LI Y Q, SUN K, et al. Identification method of DNA molecular markers for Sophora tonkinensis roots and its adulterants[J]. Journal of Chinese Medicinal Materials, 2022, 45(5):1076-1080.
[19] 江雨欣, 王锐莹, 张旭, 等. 八角茴香及其混伪品的DNA分子标记鉴别[J]. 食品科技, 2022, 47(8):307-311.
JIANG Y X, WANG R Y, ZHANG X, et al. Authentication of anisi stellati fructus and its adulterants using DNA molecular markers[J]. Food Science and Technology, 2022, 47(8):307-311.
[20] 赵琳娜, 刘娜, 王学硕, 等. 基于叶绿体rpoC1序列在山药分子鉴定中的应用[J].食品安全质量检测学报, 2021, 12(14):5644-5650.
ZHAO L N, LIU N, WANG X S, et al. Application of chloroplast rpoC1 sequence in molecular identification of Chinese yam[J]. Journal of Food Safety &Quality, 2021, 12(14):5644-5650.
[21] 汪香君, 刘墨祎, 李迎, 等. 人参、三七和西洋参多重PCR方法建立及应用[J]. 食品与发酵工业, 2022, 48(24):288-293.
WANG X J, LIU M Y, LI Y, et al. Establishment and application of multiplex PCR method for identification of Panax ginseng, Panax notoginseng, and Panax quinquefolius[J]. Food and Fermentation Industries, 2022, 48(24):288-293.
[22] THONGKHAO K, TUNGPHATTHONG C, SUKRONG S. A PCR-lateral flow immunochromatographic assay (PCR-LFA) for detecting Aristolochia species, the plants responsible for aristolochic acid nephropathy[J]. Scientific Reports, 2022, 12:12188.
[23] KIM W J, YANG S, CHOI G, et al. Establishment of conventional PCR and real-time PCR assays for accurate, rapid and quantitative authentication of four mistletoe species[J]. Phytochemistry, 2020, 176:112400.