肌苷(inosine),又称次黄苷、次黄嘌呤核苷等,是复合鲜味剂“I+G”的重要原料,广泛应用于食品生产领域,另外,肌苷作为抗病毒药物的前体,以及具有镇静、扩张血管等作用在医药领域也发挥着重要作用[1-2],因此肌苷是一种具有较高经济和社会价值的生物制品。目前微生物发酵法是生产肌苷的主要方法,但是目前已有肌苷生产菌株的生产效率仍旧较低,且存在营养缺陷、副产物积累和携带质粒等弊端,影响发酵法生产肌苷的工业化应用。
肌苷的生物合成是从5-磷酸核糖焦磷酸(phosphoribosyl pyrophosphate,PRPP)和谷氨酰胺(glutamine,Gln)开始,首先经过十步酶促反应生成肌苷酸(inosinic acid,IMP)[3],IMP在5′-核苷酸酶的催化下生成肌苷(图1)。IMP作为肌苷合成的直接前体物,也是嘌呤生物合成的中心代谢产物,其生物合成受到严格的调控,另外,腺苷和鸟苷合成支路与肌苷合成竞争碳流,这是限制肌苷大量合成的关键因素。因此,为了构建肌苷生产菌,常规的育种策略包括增强IMP的从头合成途径,强化前体物供应,解除IMP合成的反馈调节机制,阻断降解途径和竞争途径等。ASAHARA等[4]通过敲除枯草芽孢杆菌KMBS436中阻遏蛋白编码基因purR和嘌呤操纵子核糖开关,优化嘌呤操纵子的启动子序列,解除了嘌呤合成所受的反馈调节机制,并加强了嘌呤的从头合成途径;并进一步通过敲除腺苷酸琥珀酸合成酶的基因(purA)和IMP脱氢酶基因(guaB),阻断肌苷合成的竞争途径,最终使肌苷产量从1.8 g/L 提升至6.0 g/L。SHIMAOKA 等[5]在大肠杆菌突变菌 I-9 m中过表达了purFK326Q和prsD128A突变基因,解除了限速酶受到的反馈抑制并加强了前体物PRPP的合成,使肌苷产量达到了7.5 g/L。LI等[6]通过敲除枯草芽胞杆菌BS017中的purA基因来阻断腺苷合成支路,肌苷产量从0.15 g/L提升至7.6 g/L。PEIFER等[7]通过阻断谷氨酸棒杆菌(ATCC13032)中的腺苷和鸟苷合成支路,使IMP产量增加了45倍。WANG等[8]通过对枯草芽孢杆菌定向进化,降低腺苷酸琥珀酸合成酶的活性,使肌苷产量提高了约4.7倍。以往的研究发现,调整鸟苷和腺苷的合成通量对IMP合成起着非常关键的作用,但是直接阻断鸟苷和腺苷途径会造成缺陷型,导致菌体生长和肌苷生产受限,虽然补充一定量的鸟嘌呤和腺嘌呤会使生长恢复,但是必须严格控制用量,这增加了工艺控制的难度和生产成本。因此如何调控鸟苷和腺苷的合成通量,协调菌体生长和肌苷生产是肌苷生产菌株构建的难点。
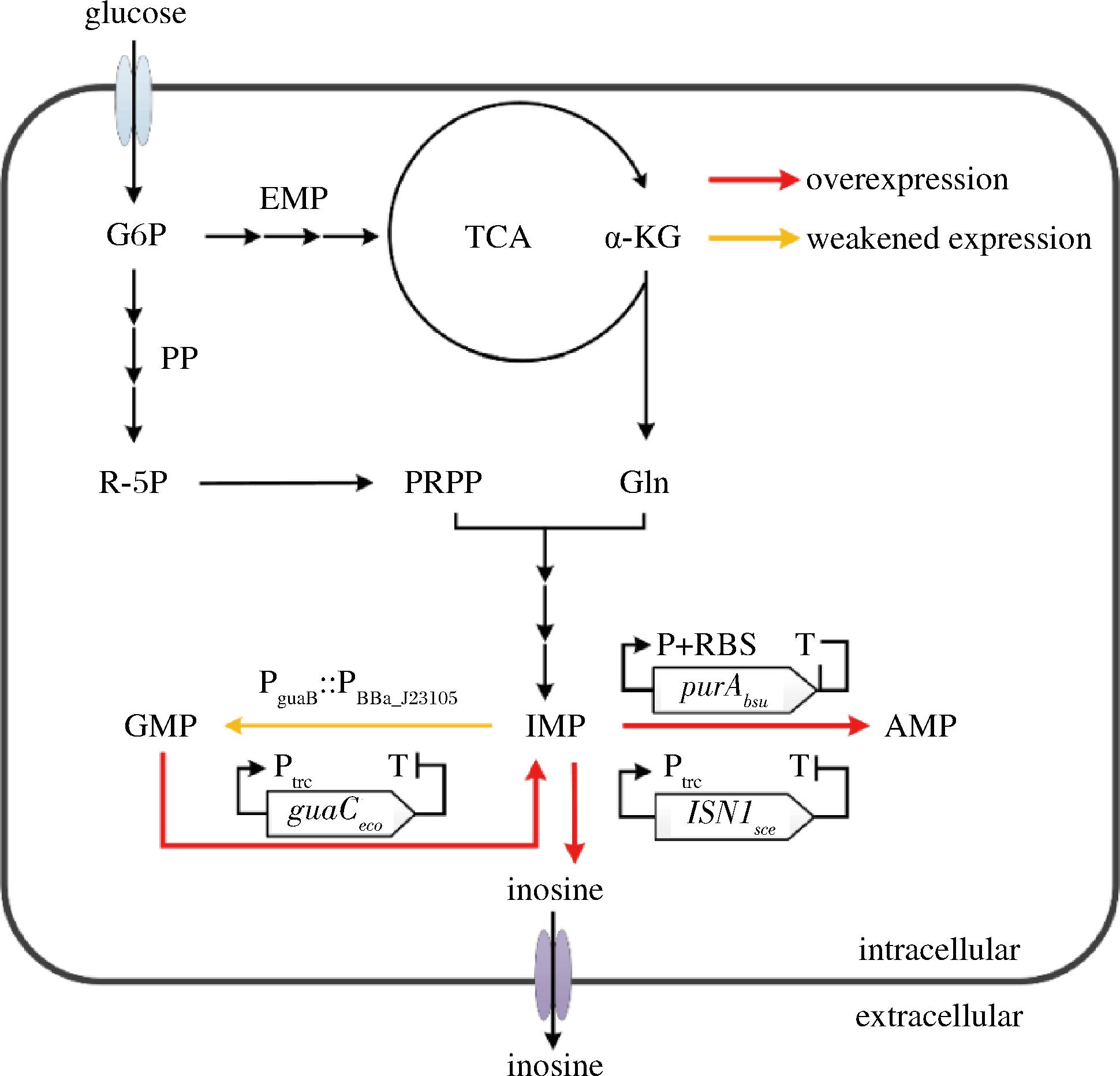
图1 肌苷合成途径及本研究的改造策略
Fig.1 Inosine synthetic pathway and the transformation strategies of this study
注:AMP-腺苷酸;GMP-鸟苷酸;IMP-肌苷酸;PRPP-5-磷酸核糖
焦磷酸;Gln-谷氨酰胺;R-5P-5-磷酸核糖;G6P-6-磷酸-葡萄糖;α-KG-α-酮戊二酸;EMP-糖酵解途径;PP-磷酸戊糖途径;TCA-三羧酸循环
本课题在前期的研究工作中,通过一系列代谢工程策略的组合运用,构建了一株肌苷生产菌株[9]。该菌株虽不是缺陷型,但是由于腺苷合成途径太弱,菌株肌苷生产性能仍受到了一定的限制,为了获得更高的肌苷生产效率,需要添加一定量的腺嘌呤。本研究在5 L发酵罐上对腺嘌呤的添加量进行了优化,发现在一定范围内随着腺嘌呤添加量的提升,肌苷的产量也显著提升,但是相应副产物的积累也显著增加。为此,本研究又继续对肌苷酸节点代谢进行了优化,包括进一步强化腺苷代谢途径,减弱鸟苷合成支路,以及通过5′-核苷酸酶的筛选和强化以进一步增强肌苷的合成(图1),降低鸟苷酸等副产物的积累,并避免了腺嘌呤的添加,为肌苷的发酵法工业化生产奠定基础。
1 材料与方法
1.1 菌株和质粒
菌株和质粒见表1。
表1 菌株和质粒
Table 1 Strains and plasmids

菌株/质粒基因型来源MG1655野生型本实验室INO4-5ΔdeoD,ΔyaiE,ΔyghE::Ptrc-pur1-2-3-4-5-6baz,ΔpurR, ΔrihB,ΔrihA,ΔrihC,Δrph::Ptrc-purFbazK316Q, ΔpurA::PpurA-purAb-suP242 N,ΔyjiT::Ptrc-pbuEbsu本实验室INO5-1INO4-5,gsk::PBBa_J23114-purAbsu本研究 INO5-2INO4-5,gsk::PBBa_J23105-purAbsu本研究 INO5-3INO4-5,gsk::PBBa_J23110-purAbsu本研究 INO5-4INO4-5,gsk::PBBa_J23106-purAbsu本研究 INO5-5INO4-5,gsk::PBBa_J23101-purAbsu本研究 INO5-2aINO4-5,gsk::PBBa_J23105-RBS10-purAbsu本研究 INO5-2bINO4-5,gsk::PBBa_J23105-RBS10-purAbsu本研究 INO5-2cINO4-5,gsk::PBBa_J23105-RBS10-purAbsu本研究 INO6-1aINO5-2b,PguaB::PBBa_J23114本研究 INO6-1bINO5-2b,PguaB::PBBa_J2310本研究 INO6-1cINO5-2b,PguaB::PBBa_J23110本研究 INO6-1dINO5-2b,PguaB::PBBa_J23106本研究 INO6-1eINO5-2b,PguaB::PBBa_J23101本研究 INO6-2INO6-1b,ybeM::Ptrc-guaCeco本研究 INO7-1INO6-2,ycgH::Ptrc-yrfG本研究 INO7-2INO6-2,ycgH::Ptrc-ushA本研究 INO7-3INO6-2,ycgH::Ptrc-umpH本研究 INO7-4INO6-2,ycgH::Ptrc-umpG本研究 INO7-5INO6-2,ycgH::Ptrc-yfkNbsu本研究 INO7-6INO6-2,ycgH::Ptrc-ISN1sce本研究 pREDCas9Sper, Cas9 and λ-Red recombinase expression vec-to[10] pGRBAmpr, gRNA expression vector[10]
1.2 培养基
斜面培养基(g/L):酵母粉5,蛋白胨10,氯化钠5,牛肉膏10,葡萄糖5,琼脂20。
种子培养基(g/L):酵母粉5,蛋白胨3,KH2PO4·3H2O 1.3,MgSO4·7H2O 0.5,FeSO4·7H2O 10 mg/L,MnSO4·H2O 10 mg/L。维生素B1、维生素B3、维生素B5、维生素B12和维生素H各2 mg/L。
发酵培养基(g/L):酵母粉4,蛋白胨5,KH2PO4·3H2O 2,MgSO4·7H2O 3,柠檬酸钠·2H2O 2,FeSO4·7H2O 10 mg/L,MnSO4·H2O 10 mg/L。维生素B1、维生素B3、维生素B5、维生素B12和维生素H各2 mg/L。
1.3 菌株构建及发酵方法
1.3.1 基因编辑方法
本研究采用CRISPR/Cas9技术[10-11]对菌株基因组进行改造,该方法中包括pGRB和pREDCas9两个质粒。将含有pGRB同源序列和特定sgRNA序列的双链DNA与线性化pGRB在同源重组酶作用下形成gRNA表达质粒;重组片段则通过重叠PCR制备。首先将pREDCas9质粒导入大肠杆菌感受态细胞中,再将pGRB质粒和重组片段导入到含有pREDCas9质粒的感受态细胞,然后筛选阳性重组子,最后依次消除pGRB和pREDCas9质粒,完成菌株构建。
1.3.2 摇瓶发酵
将菌株接种至斜面培养基中,37 ℃培养12 h后,将菌体接种至发酵培养基中,37 ℃、200 r/min培养28 h,发酵培养基中添加苯酚红作为指示剂,根据培养基颜色变化手动补加氨水以维持培养基pH值为7.0左右。
1.3.3 分批补料发酵
菌株在装有种子培养基的5 L发酵罐中进行菌体活化,发酵过程pH为7.0左右,温度37 ℃,溶氧保持在15%~25%,当OD600达到10~16时,按15%的接种量接入5 L发酵罐中进行发酵培养,发酵过程维持pH值为7.0左右,温度35 ℃,溶氧控制在20%~30%。发酵过程中流加质量浓度为800 g/L的葡萄糖溶液,维持罐中葡萄糖质量浓度在0.1~5 g/L。
1.4 基因转录水平测定
使用试剂盒Easep® Super RNA kit(Promega,上海)提取总RNA,然后使用试剂盒PrimeScriptTM RT Master Mix (TaKaRa, 大连)进行反转录,相关操作参照说明书。通过RT-PCR测定基因相对转录量,采用嵌合荧光法(TB Green® Premix Ex TaqTM Ⅱ,Takara),16S rRNA作为内标,RT-PCR结果用2-ΔΔCt法进行分析。
1.5 检测方法
菌体生物量以发酵液在600 nm处的吸光度(OD600)表征。
肌苷及其他嘌呤类副产物浓度采用HPLC方法检测:所用仪器为Thermo U3000;色谱柱为phenomenex Gemini 5u C18 110A(150 mm×4.6 mm);流动相为2%乙腈和0.05%三氟乙酸的混合水溶液;柱温30 ℃;流速 1 mL/min;检测波长260 nm。
1.6 数据统计分析
发酵数据代表3组平行发酵数据的平均值和标准差。利用t检验双尾分布对两组发酵参数进行单向方差分析。“*”表示P<0.05,表示结果差异显著;“**”表示P<0.01,表示结果差异极其显著。
2 结果与分析
2.1 腺嘌呤添加对INO4-5发酵的影响
INO4-5是实验室前期构建的一株肌苷生产菌,分别对嘌呤从头合成途径、肌苷的降解途径和肌苷转运系统进行了改造。特别是弱化了腺苷酸琥珀酸合成酶(PurA)的活性,在不造成缺陷型的前提下实现了肌苷的积累,但是由于PurA的活性过低,生长还是受到了明显抑制,添加一定量的腺嘌呤可恢复菌体生长并促进肌苷的生产。本研究在5 L发酵罐上对腺嘌呤的添加量进行了优化。结果如图2所示,在不添加腺嘌呤时,菌体生长和肌苷生产性能都比较低,而随着腺嘌呤添加量的增加,菌体的生长和肌苷的产量在也逐渐增加。当腺嘌呤添加量为3 g/L时,肌苷的最大产量可以达到30.6 g/L,OD600值可达85.8,继续增加腺嘌呤的添加量虽然使得最大OD600值提高至92.4,但对肌苷生产影响不大。结果表明INO4-5中PurA的活性过低,虽然实现了不用额外添加腺嘌呤也能积累一定量的肌苷,但是由于菌体生长受到明显抑制,需要补充定量的腺嘌呤才能获得较高的肌苷产量。另外对不同腺嘌呤的添加量下,INO4-5发酵液中的副产物进行了检测(图2-C),发现有腺苷酸,鸟嘌呤和鸟苷酸等副产物的积累,并且这些副产物的浓度也会随着腺嘌呤添加量的增加而增加。腺嘌呤的添加会增加发酵工艺的复杂程度,提高发酵成本;鸟苷酸等副产物的积累会抑制嘌呤操纵子的表达[8],不利于肌苷的合成,降低碳源的利用率,同时增加了下游的提取难度。为解决这两个问题则需进一步优化IMP节点的代谢流。
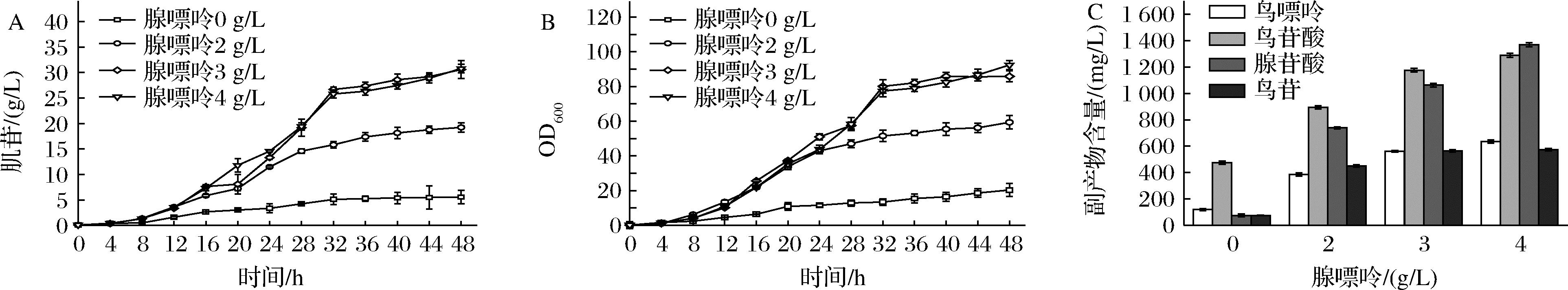
A-肌苷产量曲线;B-菌体生长曲线;C-副产物浓度
图2 腺嘌呤添加量对肌苷发酵的影响
Fig.2 Effect of adenine addition on inosine fermentation
2.2 优化purA基因的转录表达对肌苷发酵的影响
肌苷合成的直接前体是肌苷酸,但是肌苷酸除了在5′-核苷酸酶的作用生成肌苷外,还会在腺苷酸琥珀酸合成酶(purA基因编码)的作用下生成腺苷酸琥珀酸,从而流向腺苷的合成,也会在IMP脱氢酶(guaB编码)的作用下生成黄苷酸(xanthylic acid,XMP),进而流向鸟苷合成途径。因此肌苷的过量生产需要适当弱化腺苷和鸟苷合成支路碳流来实现[12],使代谢通量转向肌苷生产。INO4-5中腺苷合成支路的PurA活性太弱,导致菌体内的ATP供应不足,抑制了菌体的生长并限制了肌苷生产,因此在发酵过程中仍需要添加腺嘌呤才能获得较好的发酵结果,这无疑增加了肌苷发酵生产的成本。因此为了避免腺嘌呤的添加,同时满足菌体生长和肌苷生产的需要,需要对PurA的活性进行适当的强化。为此,本研究引入了来源于枯草芽孢杆菌的purAbsu基因,并分别使用了5个表达强度依次增加的启动子(BBa_J23114、BBa_J23105、BBa_J23110、BBa_J23106和BBa_J23101,启动子来源于https://parts.igem.org/Promoters/Catalog/Anderson)来启动purAbsu基因的表达,构建了菌株INO5-1~5。将INO4-5和INO5-1~5六株菌进行摇瓶发酵,结果如图3-A所示。结果显示,强化purAbsu基因后,菌体的生长得以恢复,并且随着启动子强度的增强,菌体的生长也越来越好。但是purAbsu的表达过强,会导致代谢流过多流向腺苷支路,从而减少肌苷的生产,其中,INO5-2的肌苷产量最高,达到6.4 g/L,最大OD600值可恢复至37。另外,基因的表达也跟核糖体结合位点(ribosomebinding site,RBS)有关,于是本研究对INO5-2中purAbsu基因的RBS进行了替换,继续微调purAbsu基因的表达强度。选用了RBS03、RBS09和RBS10[13] 3个强度依次增强的RBS,分别构建了菌株INO5-2a~c。将INO5-2与INO5-2a~c同时进行摇瓶发酵实验,结果如图3-B所示,RBS的替换对菌体生长的影响不显著,而INO5-2b的肌苷产量达到了6.9 g/L,相比对照菌INO5-2提高了7.5%。
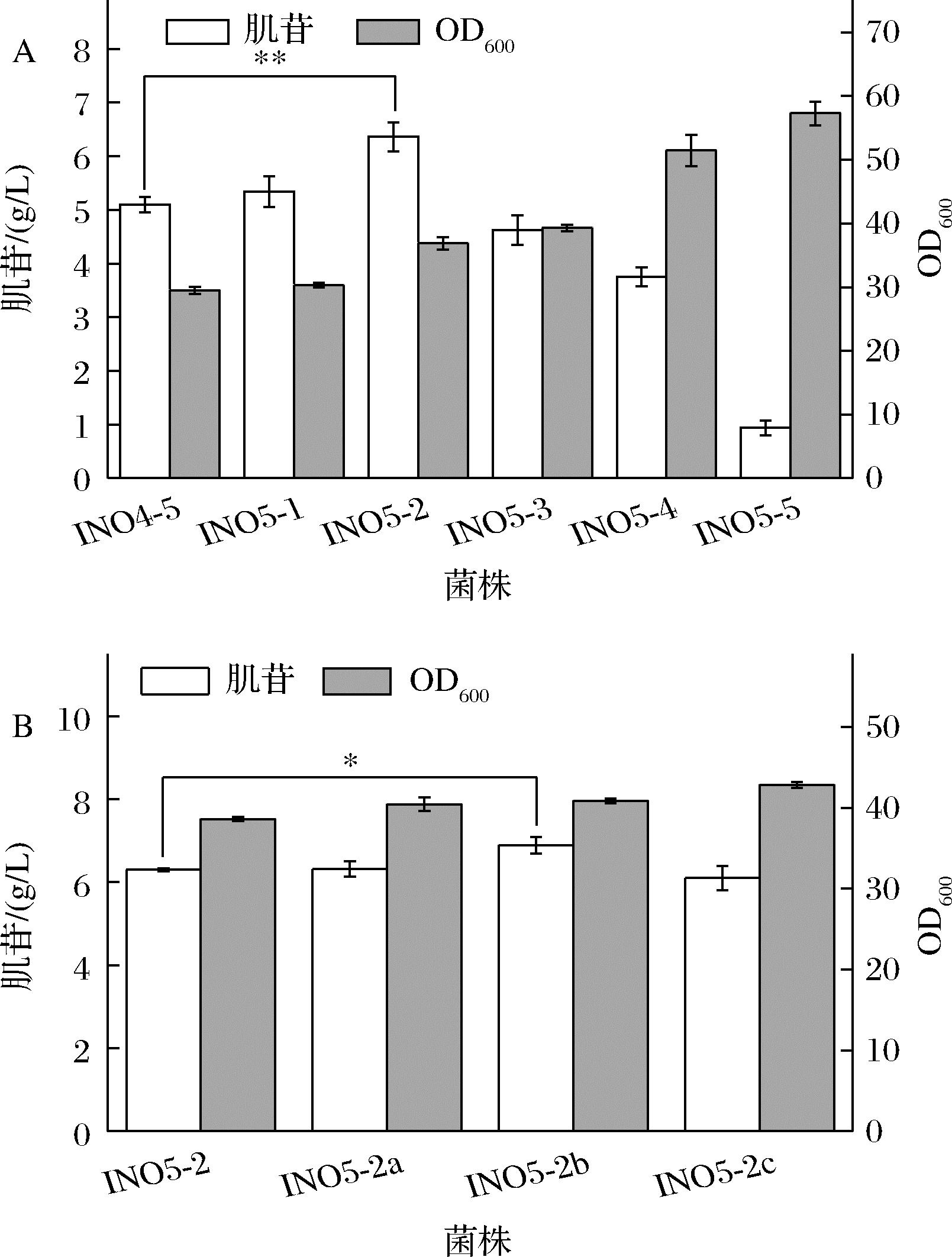
A-不同强度启动子启动purAbsu对肌苷发酵的影响;B-替换purAbsu的RBS对肌苷发酵的影响
图3 改造腺苷合成支路对肌苷发酵的影响
Fig.3 Effect of modifying adenosine synthesis bypass on inosine fermentation
注:*表示显著(P<0.05),**表示差异极显著(P<0.01)(下同)。
将本研究中摇瓶肌苷产量最高的INO5-2b在5 L发酵罐中进行分批补料发酵,结果如图4-A所示,不用额外添加腺嘌呤,INO5-2b发酵48 h可以生产肌苷29.5 g/L,与INO4-5的发酵产量(添加3 g/L腺嘌呤)相当。结果说明通过合理调整purAbsu的表达强度可以很好地平衡菌体生长与肌苷生产的平衡,在不造成缺陷型的前提下实现肌苷的高效生产。但经检测发现,发酵结束时,发酵液中仍旧积累了鸟苷酸、鸟苷和鸟嘌呤等副产物(图4-B),未检测到腺苷酸,INO4-5在添加腺嘌呤的发酵过程中会积累腺苷酸可能是腺嘌呤通过补救合成途径生成的。因此,为了降低甚至阻断这些副产物的合成,本研究接着对INO5-2b的鸟苷合成支路进行了改造。
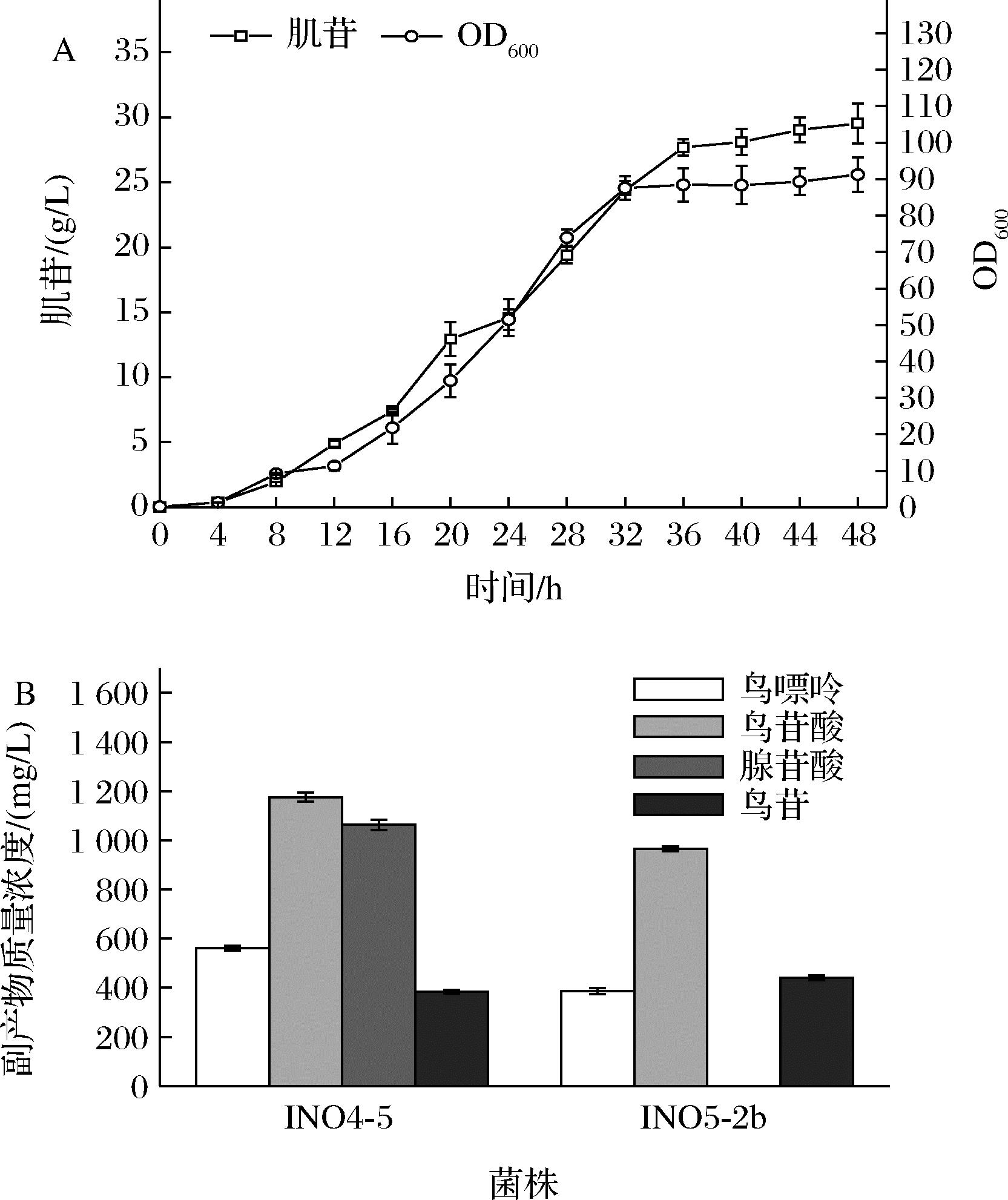
A-发酵生产曲线;B-副产物浓度
图4 菌株INO5-2b的5 L发酵罐发酵结果
Fig.4 Fermentation results of strain INO5-2b in a 5 L fermentor
2.3 鸟苷合成途径改造对肌苷发酵的影响
INO5-2b在发酵过程中,鸟苷途径的一些副产物如鸟苷酸、鸟嘌呤和鸟苷均有不同程度积累。有报道指出,上述副产物在胞内积累会损害细胞活力,使菌体快速老化[14],还会抑制PRPP合成酶(Prs)的活性[7]以及嘌呤操纵子基因的转录表达,不利于肌苷的生产。为了验证这些副产物积累对肌苷生产的影响,本研究将INO5-2b分别在添加不同浓度鸟苷酸、鸟苷或鸟嘌呤的条件下进行发酵实验,分别选择了0、0.2、0.4和0.8 g/L 4个梯度(图5)。结果显示,随着鸟苷酸和鸟嘌呤的浓度增加,其对肌苷产量和菌体生长的抑制作用也愈发显著,而在实验范围内,鸟苷的添加未对肌苷产量和菌体生长产生显著影响。
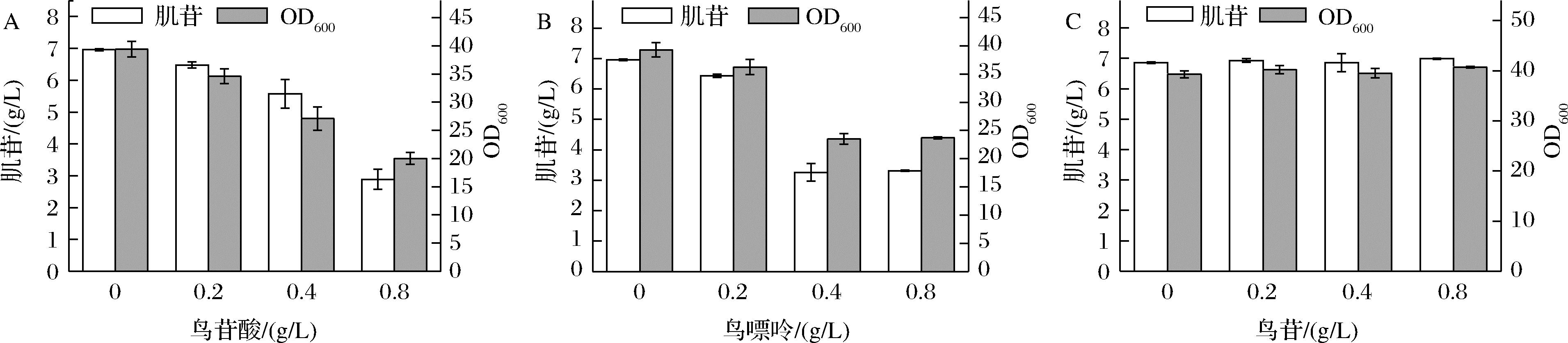
A-鸟苷酸对肌苷发酵的影响;B-鸟嘌呤对肌苷发酵的影响;C-鸟苷对肌苷发酵的影响
图5 鸟苷支路副产物对肌苷发酵的影响
Fig.5 Effects of guanosine bypass by-products on inosine fermentation
为了减少鸟苷支路副产物的积累,本研究首先将guaB基因自身的启动子进行替换,以期降低guaB基因的表达,减弱鸟苷合成支路。仍旧选用了强度依次增强的启动子BBa-J23114、BBa-J23105、BBa-J23110、BBa-J23106和BBa-J23101,分别替换了INO5-2b的guaB自身启动子,构建了菌株INO6-1a~e, 将INO5-2b和INO6-1a~e六株菌进行摇瓶发酵验证,结果显示(图6-A),与对照菌株INO5-2b相比,INO6-1a的OD600由39.5降低到了23.7,降低了40%,肌苷产量为5.9 g/L;INO6-1b的OD600相比对照菌株基本无变化,而肌苷产量由6.9 g/L提升至7.5 g/L,提高了8.7%;INO6-1c和INO6-1d同出发菌株相比,菌体生长和肌苷产量都未发生明显变化;INO6-e相比对照菌株,菌体生长无明显差异,但是肌苷产量下降至5.5 g/L,降低了20%。对发酵不同阶段guaB基因的转录量进行检测,结果显示(图6-B)发酵6 h和12 h时,相比于对照菌株,INO6-1a和INO6-1b的guaB基因的转录量分别降低了40.1%和15.2%,INO6-1c和INO6-1 d的guaB基因的转录量与对照菌相当,而INO6-1e的guaB基因转录量增加了65%。从副产物的检测情况来看(表2),发酵结束时,INO6-1a的发酵基本检测不到鸟苷酸和鸟嘌呤,INO6-1b中鸟苷酸、鸟嘌呤和鸟苷的质量浓度分别为0.051、0.126和0.167 g/L,较对照菌分别降低了76.4%、52.1%和18.1%;INO6-1c和INO6-1 d中3种副产物的浓度和对照菌株相当;而INO6-1e的鸟苷酸、鸟苷和鸟嘌呤的浓度分别较出发菌株提高了11.1%、4.6%和19.6%。结果说明通过启动子替换,降低guaB基因的转录量,从而减弱鸟苷合成途径,不仅有助于肌苷产量的提升,还有效降低了鸟苷酸等副产物的积累,但是guaB基因的转录强度太弱,则会抑制菌体的生长。综合比较来看,用启动子BBa_J23105替换guaB基因原有启动子的效果最佳,在保证菌体生长的前体下,有效提升了肌苷产量并减低了副产物的积累。
表2 不同工程菌株摇瓶发酵副产物积累情况
Table 2 Byproducts accumulation of different engineered strains in shake flasks

菌株鸟苷酸/(g/L)鸟嘌呤/(g/L)鸟苷/(g/L)INO5-2b0.216±0.028 0.263±0.013 0.204±0.047 INO6-1a<0.001**<0.001**0.144±0.054**INO6-1b0.051±0.005**0.126±0.005**0.167±0.012**INO6-1c0.184±0.007*0.182±0.016**0.184±0.014*INO6-1d0.221±0.0060.265±0.0120.163±0.020*INO6-1e0.240±0.0200.275±0.0080.244±0.034INO6-20.010±0.002**0.130±0.020**0.014±0.003**
注:*表示结果差异显著(P<0.05),**表示结果差异极显著(P<0.01)。
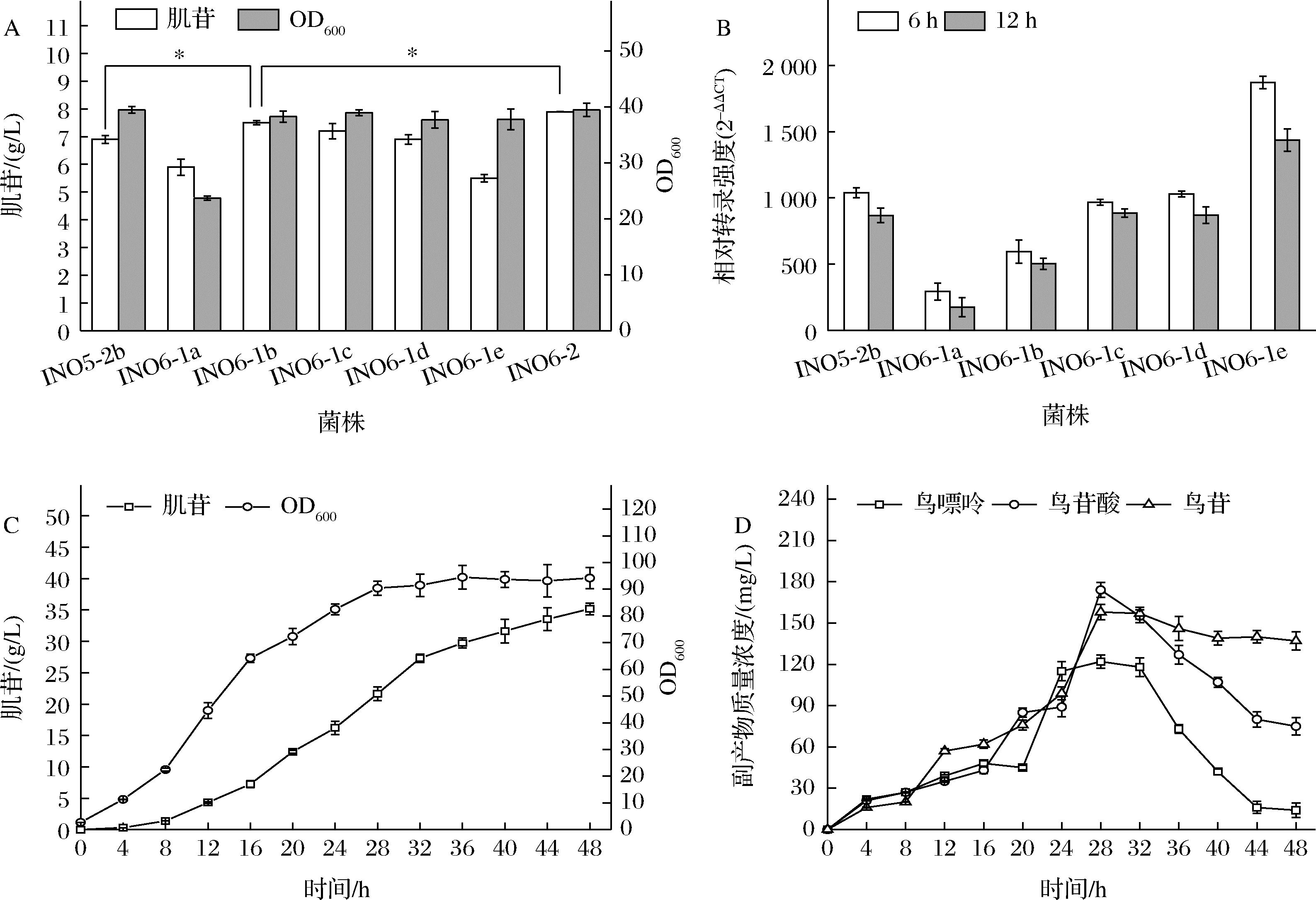
A-摇瓶发酵结果;B-guaB基因转录水平;C-INO6-2的5 L发酵罐发酵结果;D-INO6-2发酵过程中副产物浓度
图6 鸟苷支路改造对肌苷发酵的影响
Fig.6 Effect of modifying guanosine synthesis bypass on inosine fermentation
另外,GMP还原酶(guaC基因编码)可催化鸟苷酸生成肌苷酸,理论上有助于降低鸟苷合成支路副产物并促进肌苷积累。于是本研究在INO6-1b中对guaC基因进行了过表达,构建了菌株INO6-2。摇瓶结果显示(图6-A),相比INO6-1b,INO6-2的肌苷产量由7.5 g/L提升至7.9 g/L,提高了5.3%,副产物检测结果显示,发酵液中几乎检测不到鸟苷酸和鸟苷,只有少量的鸟嘌呤存在(表2)。将INO6-2在5 L发酵罐进行发酵实验(图6-C),结果显示INO6-2经5 L发酵罐测试,OD600达到了94.2,肌苷的产量达到了35.2 g/L,比INO5-2b提升了19.3%,发酵过程中鸟苷支路副产物均处于较低的水平(图6-D)。
2.4 强化5′-核苷酸酶对肌苷发酵的影响
5′-核苷酸酶作为菌体内参与合成肌苷的最后一个酶,水解5′-核苷酸生成核苷和无机磷酸盐。但是大多数的5′-核苷酸酶的底物特异性差,可作用于各种嘌呤或嘧啶核苷酸等。因此筛选活性更高、特异性更强的5′-核苷酸酶将有利于肌苷的生产。大肠杆菌中有多种5′-核苷酸酶[15-16],分别由yrfG、ushA、umpH和umpG等基因编码;目前常用于发酵生产肌苷的菌株枯草芽孢杆菌5′-核苷酸酶基因为yfkN[17];在酿酒酵母中发现了催化肌苷酸生成肌苷的特异性5′-核苷酸酶基因ISN1[18]。本研究将上述具有催化肌苷酸生成肌苷的核苷酸酶基因yrfG、ushA、umpH、umpG、yfkN和ISN1,分别整合在了INO6-2基因组的ycgH假基因位点上,并由Ptrc启动子启动表达,构建了菌株INO7-1~6。摇瓶发酵结果如图7-A所示。
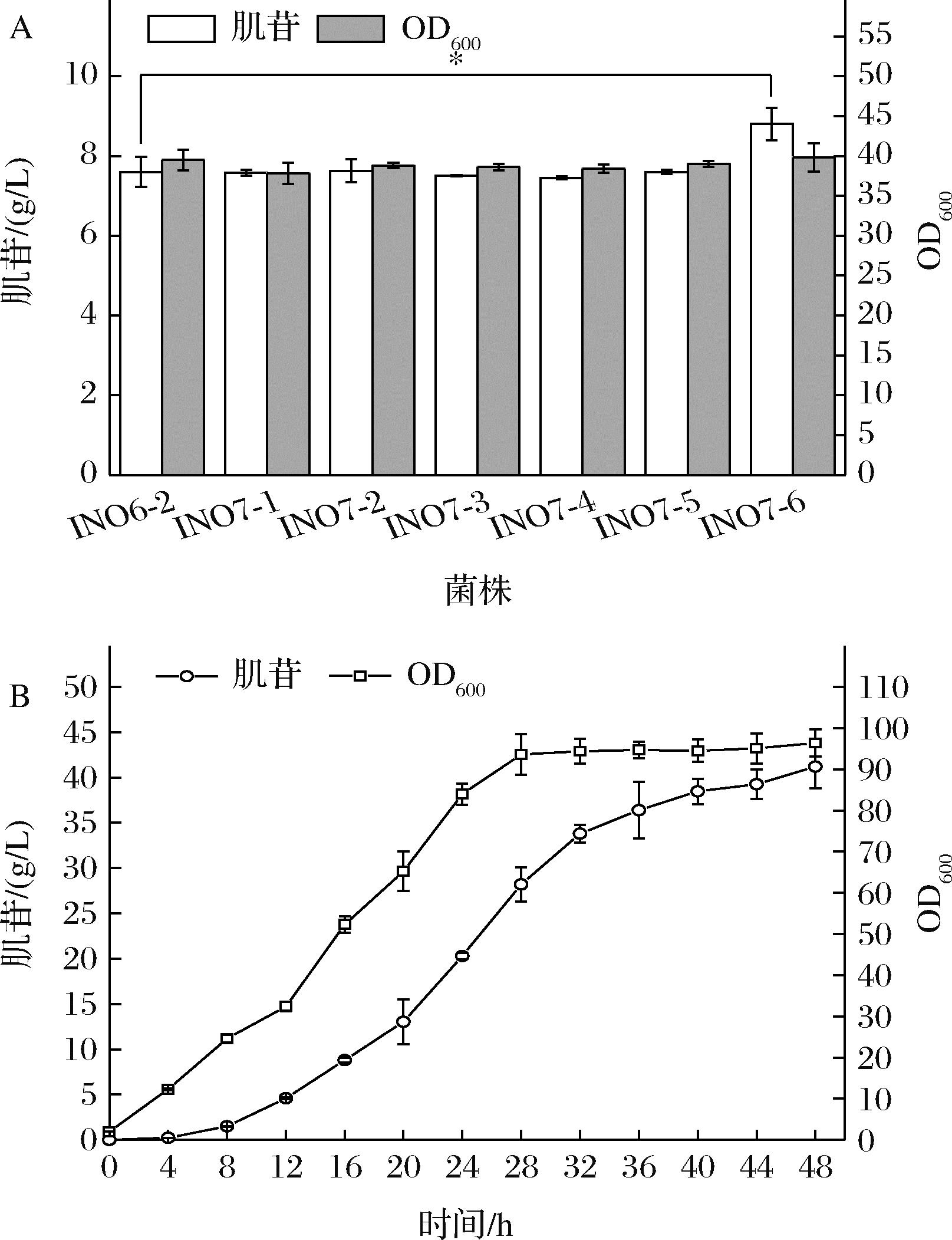
A-INO6-2和INO7-1~6的摇瓶发酵结果;B-工程菌INO7-6的5 L发酵罐发酵结果
图7 强化5′-核苷酸酶对肌苷发酵的影响
Fig.7 Effect of enhancing 5′- nucleotidase on inosine fermentation
结果显示,与对照菌INO6-2相比,INO7-1~5这五株菌的生长和肌苷生产基本无差别,说明来源于大肠杆菌和枯草芽孢杆菌的5′-核苷酸酶基因的过表达对菌体生长和肌苷生产量没有影响。而INO7-6的菌体生长相比对照菌变化不大,但是肌苷产量由7.6 g/L提升至8.8 g/L,相比对照菌株提升了15.8%。说明来源于酿酒酵母的ISN1基因的表达有效促进了肌苷的生产。为测试菌株 INO7-6的放大生产性能,在5 L发酵罐中进行了分批补料发酵,发酵结果如图7-B所示,菌株在发酵初期生长缓慢,28 h OD600逐渐达到稳定,并进入稳定期。0~12 h,肌苷合成速率较慢;12~32 h,肌苷保持了较高的合成速率,32 h之后肌苷合成速率逐渐减慢。48 h的肌苷的最终产量为41.2 g/L,葡萄糖产率为0.17 g/g葡萄糖,生产强度为0.86 g/(L·h)。
3 结论与讨论
为了解决肌苷生产菌INO4-5在发酵过程中副产物高、需外源添加腺嘌呤等问题,并进一步提高其肌苷的发酵效率,本研究对其肌苷酸节点代谢流进行了优化。首先为了强化腺苷合成途径,表达了来源于枯草芽孢杆菌的腺苷酸琥珀酸合成酶编码基因purAbsu,并使用不同强度的启动子和核糖体结合位点调整其转录表达的强度,所得最优菌株在不添加腺嘌呤的前提下,5 L罐上的肌苷发酵产量达到了29.5 g/L;鸟嘌呤和鸟苷酸等副产物的积累会抑制菌体的生长和肌苷生产,为了降低这些副产物的积累,本研究对鸟苷合成支路进行了改造,通过启动子的替换,弱化了IMP脱氢酶编码基因guaB的转录表达,并过表达guaC基因,促使鸟苷酸回流至肌苷酸,使鸟苷酸等副产物的含量显著降低,并有效提高了肌苷产量。最后对5′-核苷酸酶进行了筛选和强化,发现引入来自酿酒酵母的特异性5′-核苷酸酶基因(ISN1),可有效促进肌苷生产。最终所得菌株INO7-6,摇瓶发酵28 h肌苷产量为8.8 g/L;在5 L发酵罐上进行分批补料发酵48 h,肌苷产量达到41.2 g/L,生产强度为0.86 g/(L·h),葡萄糖的产率为0.17 g/g葡萄糖。菌株INO7-6不含质粒,且在发酵过程不用额外添加腺嘌呤或鸟嘌呤等营养物,可简化发酵工艺并降低生产成本,具有良好的工业化应用前景。为进一步提高肌苷发酵生产效率,下一步的研究可以针对中心代谢进行改造,比如重新分配糖酵解与磷酸戊糖途径的碳流,增强前体物PRPP的供应水平,促进葡萄糖向肌苷的转化,提高转化率,以满足肌苷工业化生产的需求。
[1] HASK G, SITKOVSKY M V, SZAB
G, SITKOVSKY M V, SZAB C.Immunomodulatory and neuroprotective effects of inosine[J].Trends in Pharmacological Sciences, 2004, 25(3):152-157.
C.Immunomodulatory and neuroprotective effects of inosine[J].Trends in Pharmacological Sciences, 2004, 25(3):152-157.
[2] LEDESMA-AMARO R, JIMÉNEZ A, SANTOS M A, et al.Biotechnological production of feed nucleotides by microbial strain improvement[J].Process Biochemistry, 2013, 48(9):1263-1270.
[3] ZHANG Y, MORAR M, EALICK S E.Structural biology of the purine biosynthetic pathway[J].Cellular and Molecular Life Sciences, 2008, 65(23):3699-3724.
[4] ASAHARA T, MORI Y, ZAKATAEVA N P, et al.Accumulation of gene-targeted Bacillus subtilis mutations that enhance fermentative inosine production[J].Applied Microbiology and Biotechnology.2010, 87(6):2195-2207.
[5] SHIMAOKA M, TAKENAKA Y, KURAHASHI O, et al.Effect of amplification of desensitized purF and prs on inosine accumulation in Escherichia coli[J].Journal of Bioscience and Bioengineering, 2007, 103(3):255-261.
[6] LI H J, ZHANG G Q, DENG A H, et al.De novo engineering and metabolic flux analysis of inosine biosynthesis in Bacillus subtilis[J].Biotechnology Letters, 2011, 33(8):1575-1580.
[7] PEIFER S, BARDUHN T, ZIMMET S, et al.Metabolic engineering of the purine biosynthetic pathway in Corynebacterium glutamicum results in increased intracellular pool sizes of IMP and hypoxanthine[J].Microbial Cell Factories, 2012,11:138.
[8] WANG X Y, WANG G L, LI X L, et al.Directed evolution of adenylosuccinate synthetase from Bacillus subtilis and its application in metabolic engineering[J].Journal of Biotechnology, 2016, 231:115-121.
[9] 朱彦凯, 刘铁重, 伍法清, 等.代谢工程改造大肠杆菌生产肌苷[J].食品与发酵工业, 2022, 48 (24):1-9.
ZHU Y K, LIU T Z, WU F Q, et al.Metabolic engineering of Escherichia coli for inosine production[J].Food and Fermentation Industries, 2022, 48 (24):1-9.
[10] LI Y F, LIN Z Q, HUANG C, et al.Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing[J].Metabolic Engineering, 2015, 31:13-21.
[11] WU M Y, SUNG L Y, LI H, et al.Combining CRISPR and CRISPRi systems for metabolic engineering of E.coli and 1,4-BDO biosynthesis[J].ACS Synthetic Biology, 2017, 6(12):2350-2361.
[12] MATSUI H, KAWASAKI H, SHIMAOKA M, et al.Investigation of various genotype characteristics for inosine accumulation in Escherichia coli W3110[J].Bioscience Biotechnology and Biochemistry, 2001, 65(3):570-578.
[13] ZHANG Q, YAO R, CHEN X L, et al.Enhancing fructosylated chondroitin production in Escherichia coli K4 by balancing the UDP-precursors[J].Metabolic Engineering, 2018, 47:314-322.
[14] LEDESMA-AMARO R, BUEY R M, REVUELTA J L.Increased production of inosine and guanosine by means of metabolic engineering of the purine pathway in Ashbya gossypii[J].Microbial Cell Factories, 2015, 14(1):1-8.
[15] KUZNETSOVA E, PROUDFOOT M, GONZALEZ C F, et al.Genome-wide analysis of substrate specificities of the Escherichia coli haloacid dehalogenase-like phosphatase family[J].Journal of Biological Chemistry, 2006, 281(47):36149-36161.
[16] PROUDFOOT M, KUZNETSOVA E, BROWN G, et al.General enzymatic screens identify three new nucleotidases in Escherichia coli.Biochemical characterization of SurE, YfbR, and YjjG[J].Journal of Biological Chemistry, 2004, 279(52):54687-54694.
[17] KUNST F, OGASAWARA N, MOSZER I, et al.The complete genome sequence of the gram-positive bacterium Bacillus subtilis[J].Nature, 1997, 390(6657):249-256.
[18] ITOH R, SAINT-MARC C, CHAIGNEPAIN S, et al.The yeast ISN1 (YOR155c) gene encodes a new type of IMP-specific 5′-nucleotidase[J].BMC Biochemistry, 2003, 4:4.