脂肪酶(EC3.1.1.3)又称三酰基甘油水解酶,属于羧酸酯水解酶家族中的一类[1],可表现出水解、酰胺化、酯化、酯交换、醇解等活性[2]。脂肪酶作为一种多用途生物催化剂,在食品、制药、纺织品、化妆品行业和生物柴油等方面有着广泛的应用。研究发现,脂肪酶可从动物、植物、天然或重组微生物中获得[3],在以微生物来源的脂肪酶中,真菌被广泛认为是脂肪酶的首要来源,如诺维信商业化脂肪酶B(SP435)和Lipozyme RMIM分别来源于南极假丝酵母和米黑根毛霉[4]。此外,研究报道根霉、曲霉等丝状真菌中含有多个编码脂肪酶的基因[5-6],如黑曲霉CBS513.88基因组中含有70多个编码脂肪酶的基因。XING等[7]在大肠杆菌中异源表达了黑曲霉脂肪酶EXANL1,该重组脂肪酶在碱性条件下稳定,酶活保留率在90%以上。
酯类物质是白酒中重要的风味物质,如乙酸乙酯、己酸乙酯、乳酸乙酯、丁酸乙酯等短链脂肪酸乙酯对白酒风味起着关键的作用[8-9]。白酒中酯类物质的形成除酵母细胞代谢合成外,酶催化的酯化反应也是一个重要途径[10]。脂肪酶可催化脂肪酸和乙醇生成脂肪酸乙酯,反应通常在有机相中进行,目前已报道的在水相体系下可催化合成脂肪酸乙酯的脂肪酶还非常有限[12-13],且以中、碱性脂肪酶居多。例如,蒋翠翠等[14]筛选获得一种来源于表皮葡萄球菌和一种来源于解脂耶氏酵母的脂肪酶,可有效提高酸肉中脂肪酸乙酯等风味物质的生成,2种脂肪酶的最适反应pH值均为8.0;本课题组在前期研究中对黑曲霉中的脂肪酶Lip A和Lip B分别进行了克隆表达,发现Lip B在水相条件下可以催化合成乳酸乙酯,2种脂肪酶的最适反应pH也均为碱性条件[15]。但是,由于传统酿造过程大部分在酸性环境下进行,目前应用于该领域的高效酸性脂肪酶的研究报道还较少,为使脂肪酶在改善传统发酵产品风味品质方面更好地发挥作用,在酿造领域体现其更大的应用价值,仍需挖掘更多的酸性脂肪酶基因并对其催化特性进行深入研究。
为了更深入地研究黑曲霉来源的脂肪酶的催化特性及其催化合成脂肪酸乙酯的能力,本研究将黑曲霉脂肪酶Tgl进行异源表达,研究该酶的酶学性质及催化酯类物质的合成能力,为脂肪酶在食品行业中广泛应用和工业化酶制剂种类的丰富提供理论基础。
1 材料与方法
1.1 材料与试剂
大肠杆菌(Escherichia coli) DH5α、E.coli Rosetta (DE3)、质粒pET-28a(+),均由本实验室保存。
2×Taq Master Mix、2×Phanta Max Master Mix,Vazyme公司;Quick CutTM Dpn I,TaKaRa公司;对硝基苯酚酯(C2/C4/C6/C8/C12/C16),Aladdin公司;异丙基硫代-β-D-半乳糖苷(isopropyl-beta-D-thiogalactopyranoside,IPTG)、Bradford蛋白定量试剂盒,Solarbio公司;Ni柱和PD-10脱盐柱,GE公司;30 kDa超滤管,Millipore公司;乙酸、丁酸、戊酸、己酸、庚酸、辛酸、乳酸等均为分析纯,天津市津东天正精细化学试剂厂。
1.2 仪器与设备
HE-120多功能水平电泳槽,上海天能科技公司;全自动凝胶成像仪,美国SYNGENE;PCT-200型PCR基因扩增仪,Eppendorf公司;7890A气相色谱仪 美国安捷伦科技有限公司。
1.3 实验方法
1.3.1 目的基因的获取和重组质粒的构建
本研究通过NCBI在线网站(https://www.ncbi.nlm.nih.gov/)获得黑曲霉(Aspergillus niger CBS513.88)的基因组序列。通过对黑曲霉基因组中的开放阅读框进行对比,获得脂肪酶Tgl编码基因(Gene ID:4988961)。将去掉内含子和信号肽的目的基因片段送至金唯智有限公司进行合成,并进行大肠杆菌密码子优化。载体pET-28a(+)经EcoR I线性化后与目的片段在连接酶的作用下,37 ℃反应30 min,构建重组载体pET-28a(+)-Tgl。
1.3.2 脂肪酶Tgl表达载体的构建
将重组产物化转至E.coli DH5α感受态中后,涂布在含有卡那霉素抗性的LB固体培养基上,筛选验证正确的转化子,将正确的转化子在LB液体培养基中培养并提取重组质粒转化至E.coli Rosetta (DE3)感受态中,然后涂布在含有卡那霉素和氯霉素的LB固体培养基中,筛选出验证正确的转化子即为表达菌株。
1.3.3 脂肪酶Tgl的诱导表达和纯化
将重组菌株转接至5 mL含有卡那霉素和氯霉素抗性的LB培养基的试管中,在37 ℃,220 r/min的摇床中过夜培养,然后按1%接种量转接到含100 mL的LB液体培养基至菌液OD600=0.6~0.8,加入IPTG(终浓度为1 mmol/L)25 ℃培养20 h,离心收集菌体。加入PBS溶液洗涤菌体后重悬菌体,然后超声破碎菌体,离心收集上层清液。将上清液加入到镍柱中,经不同浓度咪唑溶液洗脱,收集洗脱液进行SDS-PAGE分析。
1.3.4 脂肪酶Tgl生物信息学分析
1.3.4.1 脂肪酶Tgl多序列对比
通过Swiss-Model在线网站将已表征过晶体结构的高同源性脂肪酶氨基酸序列在ESPript3.0(https://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)在线网站中与脂肪酶Tgl的氨基酸序列进行对比。
1.3.4.2 脂肪酶Tgl三维结构模拟
采用AlphaFold2(https://alphafold2.biodesign.ac.cn/)对脂肪酶Tgl的三维结构进行模拟。
1.3.5 蛋白含量的测定
将1.3.3节中获得的蛋白洗脱液使用30 kDa超滤管去除咪唑分子后,采用Bradford蛋白浓度测定试剂盒测定纯化后重组脂肪酶Tgl的浓度[16]。
1.3.6 脂肪酶Tgl活力的测定
脂肪酶活力的测定采用对硝基苯酚法[17]。反应方法如下:向400 μL的Tris-HCL缓冲液(50 mmol/L,pH 8.0)中加入50 μL pNPA异丙醇溶液(10 mmol/L)混匀后置于恒温水浴锅中保温1 min,后加入50 μL酶液,继续在水浴锅中反应5 min后,立即拿出加入500 μL无水乙醇终止反应,在波长为410 nm下测定吸光值。在该条件下,将1 min内催化生成1 μmol对硝基苯酚所需要的酶量定义为1 U。酶活力计算如公式(1):
酶活力![]()
(1)
式中:A为410 nm处测量得到的吸光值;1 000为mmol向μmol单位转换;t为反应时间,min;V为反应总体积,L;n为稀释倍数;Q为所需酶量,mL。
1.3.7 脂肪酶Tgl酶学性质分析
1.3.7.1 最适反应温度和温度稳定性测定
反应底物使用对硝基苯酚乙酸酯,分别向pH 8.0的Tris-HCL缓冲液加入50 μL酶液,设置3个平行,分别将其放入温度为20~70 ℃的水浴锅中进行反应,将其中最高酶活力设为100%,计算其他条件下的相对酶活。将等体积的酶液分别置于不同温度的水浴中保温,每隔2 h取样测定酶活,将其中最高酶活力设为100%,计算其他条件下的相对酶活力。
1.3.7.2 最适反应pH和pH稳定性测定
分别配制20 mmol/L不同pH值(pH 4.0~11.0)的缓冲液作为反应体系,50 ℃进行反应,结束后将反应体系pH调整为8.0测定酶活力,将其中最高酶活力设为100%,计算其他条件下的相对酶活力。将等体积的酶液分别置于不同pH值(pH 4.0~11.0)的缓冲液中,每隔2 h后取样测定酶活力,将其中最高酶活力设为100%,计算其他条件下的相对酶活力。
1.3.7.3 脂肪酶Tgl底物特异性分析
将不同链长的对硝基苯酚酯用异丙醇溶解,按照各自的分子质量配制成浓度为10 mmol/L的底物溶液。在Tris-HCl缓冲液(pH 8.0,50 mmol/L)中加入底物,并在50 ℃水浴锅中测定脂肪酶Tgl的活力,将酶活力最高的设置为100%,计算其他条件下的相对酶活力。
1.3.7.4 不同有机溶剂对脂肪酶Tgl活力的影响
将酶液与有机试剂(甲醇、无水乙醇、异丙醇、正己烷、十二烷、DMSO、乙酸乙酯、乙腈)按照9∶1 (体积比)混合均匀,置于4 ℃放置1 h后,对照组为用缓冲液代替有机试剂,分别测定酶活。将对照组的酶活力设置为100%,同时测定不同浓度(20%~100%)下乙醇对酶活力的影响。
1.3.7.5 Km和Vmax的测定
分别向pH 8.0的缓冲液中加入不同浓度(0.1、0.3、0.5、0.7、0.9、1.0、2.0 mmol/L)的不同底物(pNP-C2~16),50 ℃反应5 min后测定吸光度值,根据Lineweaver-Burk双倒数作图法计算得到脂肪酶Tgl的Km和Vmax值。
1.3.7.6 脂肪酶Tgl催化合成脂肪酸乙酯
分别配制20%和60%的乙醇水溶液作为溶剂,乙酸、丁酸、戊酸、己酸、庚酸、乳酸、辛酸的浓度均为3.43 mol/L,向反应体系中加入等体积的7种酸和5 mg的冻干酶粉,反应体系为2 mL,以加入相同质量的高温灭活脂肪酶Tgl作空白对照离心,在摇床内40 ℃、200 r/min,反应时间12 h,结束后使用正己烷萃取,取萃取液留作气相色谱分析。
脂肪酸乙酯的气相色谱检测方法:色谱柱为HP-INNOWAX(30 m×0.32 mm×0.5 μm),检测器为FID。进样器温度为260 ℃,进样量为1.0 μL,检测器温度为300 ℃。柱温在65 ℃保持5 min,以5 ℃/min上升到150 ℃,保持2 min,共24 min。载气为氮气,色谱柱流速为0.8 mL/min。
2 结果与分析
2.1 脂肪酶Tgl表达菌株的构建及表达纯化
将载体pET-28a(+)通过EcoR Ι限制性酶切位点线性化后,脂肪酶基因无缝克隆至载体pET-28a(+)(Kanamycin)构建重组质粒。筛选阳性克隆进行菌落PCR验证,结果如图1-a所示,得到大小为819 bp的目的基因条带,含his标签,与Tgl编码基因大小一致。重组菌株经过诱导、破碎、纯化后,SDS-PAGE电泳验证结果如图1-b所示。脂肪酶Tgl蛋白分子的理论分子质量为30 kDa,由图中可见,在30 kDa附近出现特异性条带,实际的蛋白分子质量比理论值偏高的原因是在C端加入His标签以及凝胶电泳存在误差引起的。测定纯化后脂肪酶Tgl的比酶活力达1 500.43 U/g,其比酶活力远远高于植物小麦芽来源的脂肪酶8.52 U/g[18],与米曲霉来源脂肪酶的比酶活1 424.73 U/g[19]相近。
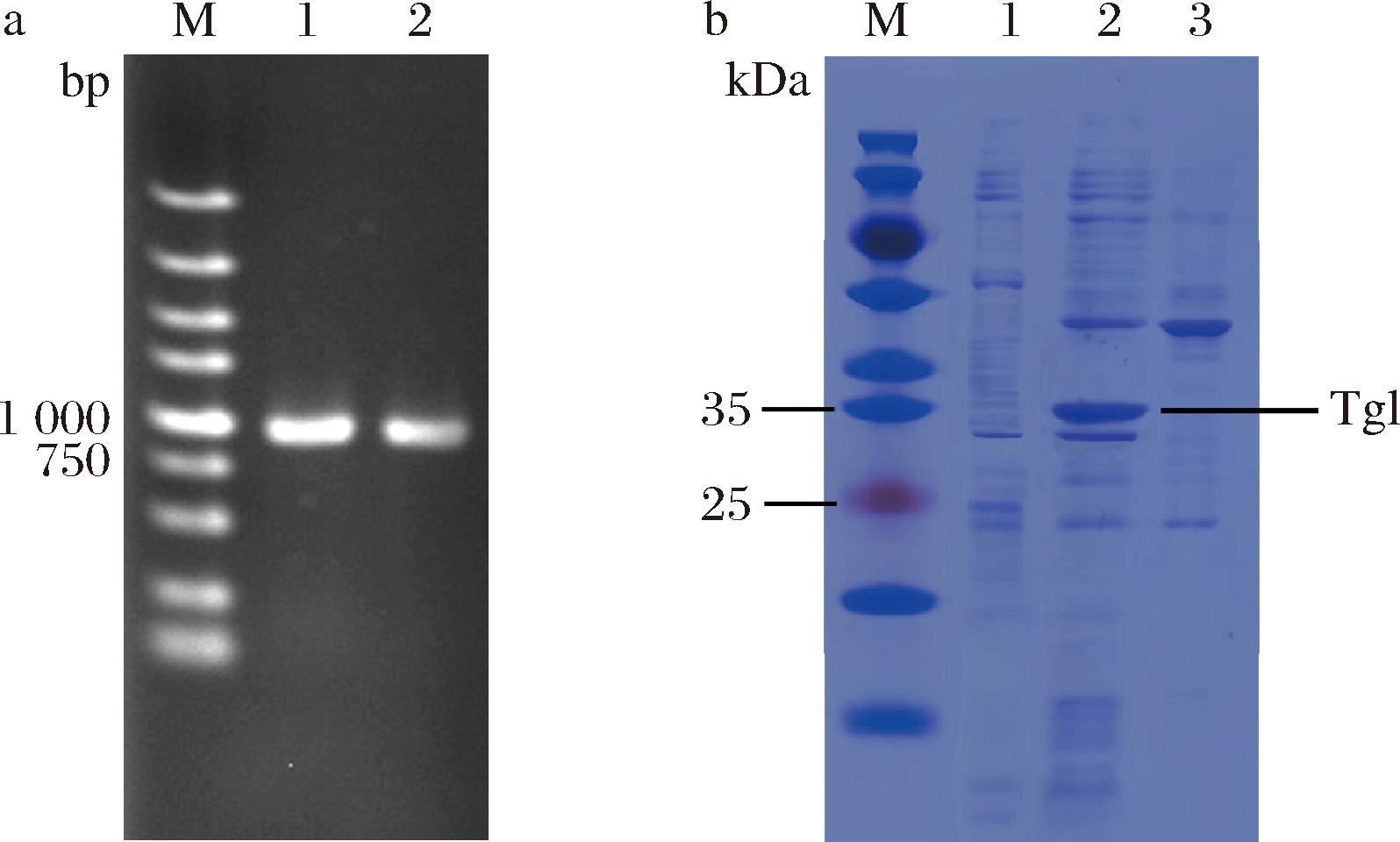
a-菌落PCR(M-Marker;1、2-目的条带819 bp);b-SDS-PAGE分析(M-蛋白Marker;1-空白对照;2-20 mmol/L咪唑洗脱液;3-500 mmol/L咪唑洗脱液)
图1 菌落PCR验证及脂肪酶Tgl的SDS-PAGE分析
Fig.1 Colony PCR verification and lipase Tgl SDS-PAGE analysis
2.2 脂肪酶Tgl的生物信息学分析
2.2.1 脂肪酶Tgl多序列对比
通过对Swiss-Model数据库中已经表征过晶体结构的脂肪酶序列进行分析,以Tgl氨基酸序列为目标,按照同源性从高到低依次显示与来源于A.niger的脂肪酶Tgl高同源性的氨基酸序列,分别为扩展青霉(Penicillium Expansum,3 g7n)、嗜热丝孢菌(Thermomyces,1 du4)、绵毛嗜热丝孢菌(Thermomyces lanuginosa,4 gi1)、绵毛嗜热丝孢菌(Thermomyces lanuginosa,6xok),通过比对分析发现(图2),Tgl结构中的A75-D83小段序列为脂肪酶Tgl的“盖子”结构,β-sheet 1-8分别为T40-E47、D51-S59、K63-M69、T132-H139、I161-L166、T186-N192、T213-S217、V225-C227,Ser139、Asp195、His248为脂肪酶的催化活性中心,其中Ser139是催化亲核氨基酸,Ser139、Asp195、His248三个催化中心用蓝色星号标出。
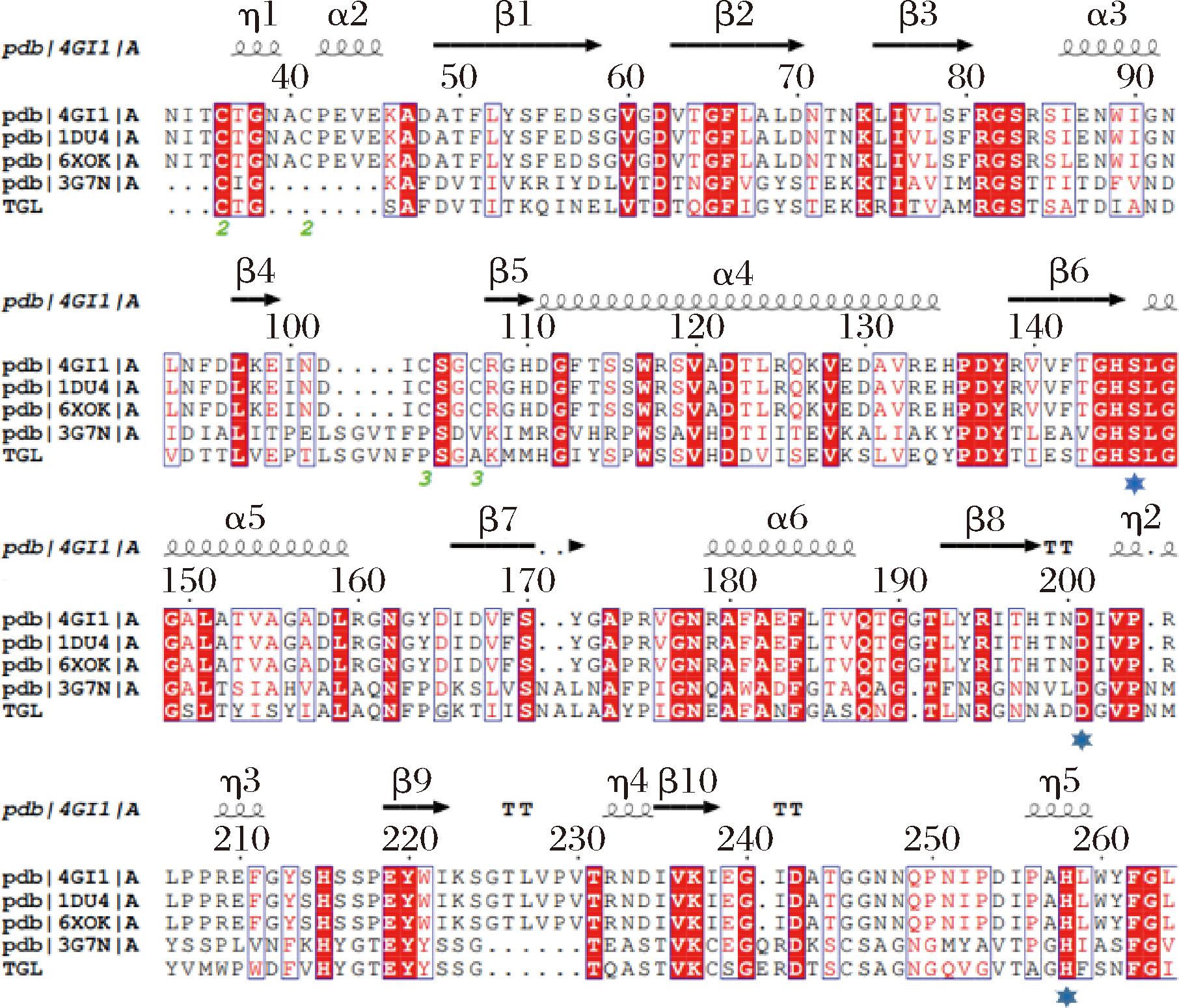
图2 脂肪酶Tgl氨基酸序列比对
Fig.2 Amino acid sequence alignment of lipase Tgl
注:红色区域为完全保守区,蓝色星星标记的为催化三联体。
2.2.2 脂肪酶Tgl三维结构模拟
本研究在Swiss-Model数据库中,获得已经表征过蛋白晶体结构的高同源性序列(PDB:3 g7n),以3 g7n为模板,利用AlphaFold软件预测脂肪酶Tgl的三维结构,结果如图3所示。图3-c为预测脂肪酶Tgl与3g7n三维结构的对比,其RMSD值为0.404,其中AlphaFold预测的结果与3g7n蛋白结构的差别主要体现在C端和N端的loop区,而核心区域的预测结果较3g7n蛋白结构差别不大,AlphaFold预测的Tgl的三维结构具有较高的可信度(图3-d)。

a-脂肪酶Tgl的AlphaFold软件预测结果(脂肪酶Tgl中浅蓝色为α-螺旋结构,紫色为β-sheet结构,浅粉色为loop结构);b-脂肪酶PDB:3g7n结构(脂肪酶Tgl中红色为α-螺旋结构,黄色为β-sheet结构,绿色为loop结构);c-AlphaFold软件预测结果和3g7n对比(浅蓝色为3g7n,红色为AlphaFold2软件预测结果);d-脂肪酶Tgl的AlphaFold软件预测可信度图
图3 脂肪酶Tgl蛋白结构预测
Fig.3 Structure prediction of lipase Tgl protein
2.3 脂肪酶Tgl酶学性质的表征
2.3.1 温度与pH对脂肪酶Tgl酶活力的影响
以对硝基苯酚乙酸酯为底物,温度对脂肪酶Tgl活力的影响如图4-a所示,当反应温度为50 ℃时,相对酶活力最高,为100%;在40~70 ℃,相对酶活力维持在75%以上;而在低于40 ℃时相对酶活力随温度的降低迅速下降。这表明脂肪酶Tgl对低温更加敏感,在低温环境下,脂肪酶的活力受到抑制。同样黑曲霉来源的脂肪酶Lip A、Lip B和酸性脂肪酶ANL的最适温度分别为40、50、45 ℃[20],说明黑曲霉来源的脂肪酶最适温度在40~50 ℃。脂肪酶Tgl在40~70 ℃下的稳定性如图4-b所示,在50 ℃下孵育0~12 h后残余酶活力数据表现最好,同时脂肪酶Tgl在40 ℃和50 ℃条件下孵育6 h,酶的相对酶活力仍能够维持在80%以上,然后随着孵育时间延长,在40 ℃条件下的脂肪酶Tgl的活力显著下降;而在60 ℃和70 ℃下孵育6 h的脂肪酶Tgl的相对酶活力均降至50%以下,当孵育时间延长至10~12 h时,该酶活力已经基本丧失。由此可见,黑曲霉来源的脂肪酶Tgl的耐热性能较好。
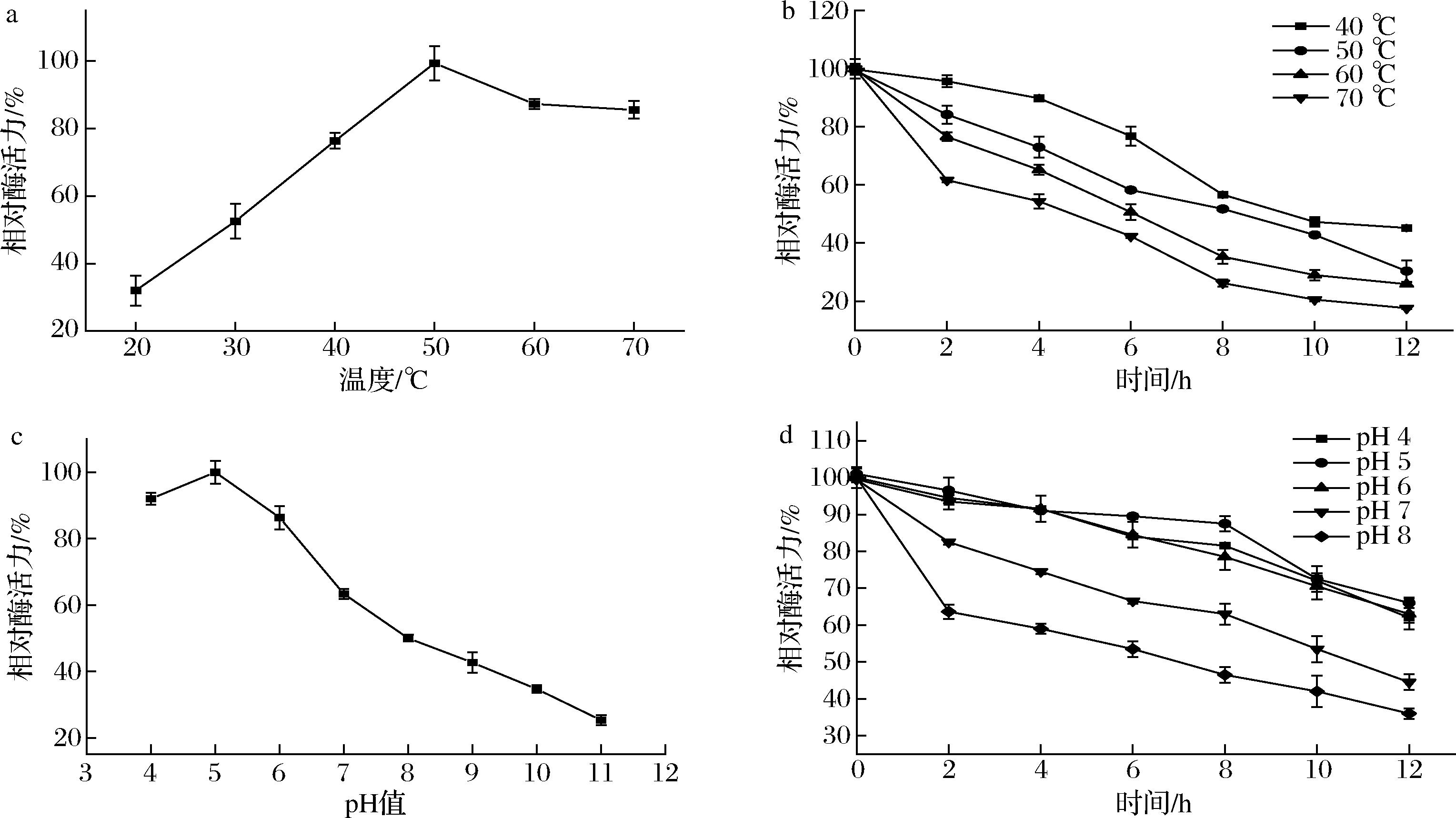
a-反应温度;b-温度稳定性;c-反应pH;d-pH稳定性
图4 温度和pH对脂肪酶Tgl活力的影响
Fig.4 Effect of temperature and pH on lipase Tgl activity
由图4-c可知,脂肪酶Tgl的最适反应pH值为5.0,在pH 4.0~6.0的弱酸性反应环境下具有较高的酶活性,其相对酶活力均保持在85%以上,但在中性和碱性条件下酶活力迅速下降,仅为其在弱酸环境下酶活力的一半。文献中已报道的其他来源的脂肪酶的最适反应pH值多为碱性。例如,来源于米根霉WPG和解淀粉芽孢杆菌G7的脂肪酶最适反应pH均为8.0[21-22],来源于木糖葡萄球菌来源的脂肪酶最适反应pH为 8.2[23],来源于黑曲霉的脂肪酶Lip A的最适反应pH也是8.0,由此可见,即使相同来源的脂肪酶其酶学性质也有可能存在显著的差异。进一步在pH 4.0~8.0的环境下对脂肪酶Tgl的稳定性进行研究,结果如图4-d所示。脂肪酶Tgl在pH 4.0~6.0的弱酸环境下的酶活力相差不大,均能够在孵育12 h后仍保留65%以上的相对酶活力,具有较好的稳定性,其中在pH 5.0时脂肪酶Tgl的稳定性表现最好,孵育8 h后,其相对酶活力仍在90%以上,随着孵育时间的延长,其相对酶活力逐渐下降至70%;在中性和弱碱性环境下,脂肪酶Tgl的相对酶活力随着孵育时间的延长迅速下降,在孵育12 h后,其相对酶活力不足50%。对比文献报道,来源于黑曲霉的酸性脂肪酶ANL在pH 2.5~5.0条件下稳定性最高[20],同样来源于黑曲霉AN0512的脂肪酶在pH 3.0~7.0条件下稳定性最高[24],它在pH 3.0下几乎保持了95%的活性。相比较之下,脂肪酶Tgl的稳定性在弱酸环境下表现更好,更适合用于酿酒工业中。
2.3.2 脂肪酶Tgl的底物特异性研究
酶的底物特异性是探究酶学性质的一个重要指标,脂肪酶Tgl的底物特异性分析结果如图5所示,脂肪酶Tgl的最适底物为pNP-C2,其相对酶活力随着底物碳链长度的延长而不断降低,对pNP-C6、pNP-C8、pNP-C12底物水解活性分别是pNP-C2酶活力的74.95%、86.12%和52.74%。在测定的所有底物中,脂肪酶Tgl对pNP-C12和pNP-C16底物的水解活性最低,仅有对pNP-C2水解活力的50%左右,这表明Tgl对长链底物的水解能力有限,受限于蛋白质三维构象,短链底物进入酶的催化口袋较长链底物容易,使得该酶对短链底物的催化效率较长链底物高,表现出对长链底物较低的水解活性。与本研究中脂肪酶Tgl相比,Aspergillus carneus来源的脂肪酶作用于pNP-C12时表现出较好的活性,比对pNP-C16时活性高5倍[25],由此可见,针对不同应用领域,可选择不同底物偏好性的脂肪酶。
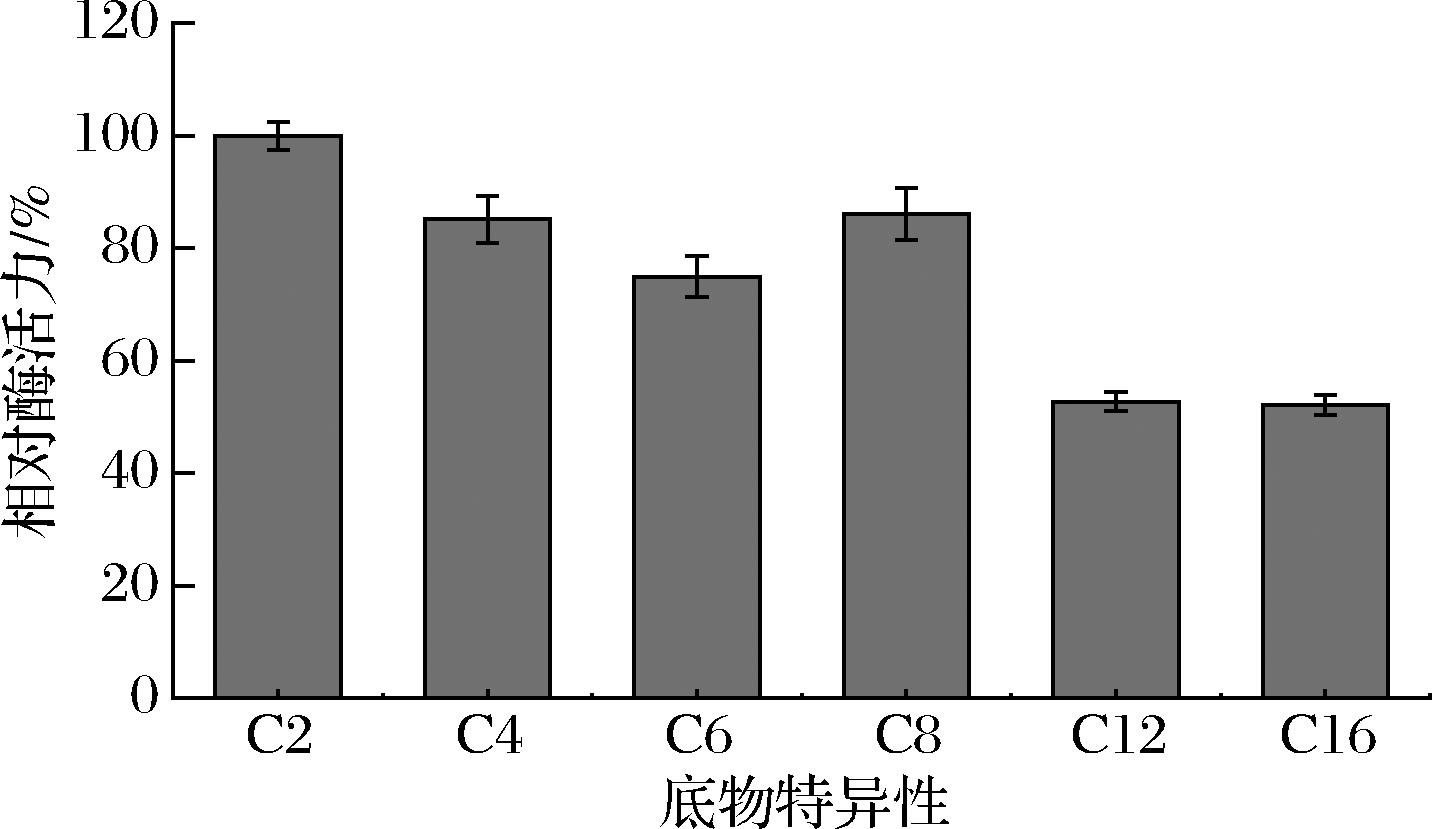
图5 脂肪酶Tgl的底物特异性
Fig.5 Substrate specificity of lipase Tgl
2.3.3 有机溶剂对脂肪酶Tgl活力的影响
使用对硝基苯酚乙酸酯进行酶活力测定,对照组用PBS缓冲液替代有机溶剂,其相对酶活力设置为100%,测定结果如图6-a所示。所有的实验组有机试剂对脂肪酶Tgl均表现出不同程度的抑制作用,这是因为大多数有机溶剂会削弱蛋白质的疏水键,使脂肪酶分子内斥力增加,造成蛋白质的三级结构延展,最终导致脂肪酶丧失催化活性[22]。由图6可知,乙醇和异丙醇对脂肪酶Tgl的抑制作用最为明显,乙醇浓度越高,对脂肪酶Tgl的抑制作用越大,如图6-b所示,当乙醇浓度超过60%时,脂肪酶Tgl的相对酶活力不足50%。正己烷和DMSO对脂肪酶Tgl的抑制作用最小。
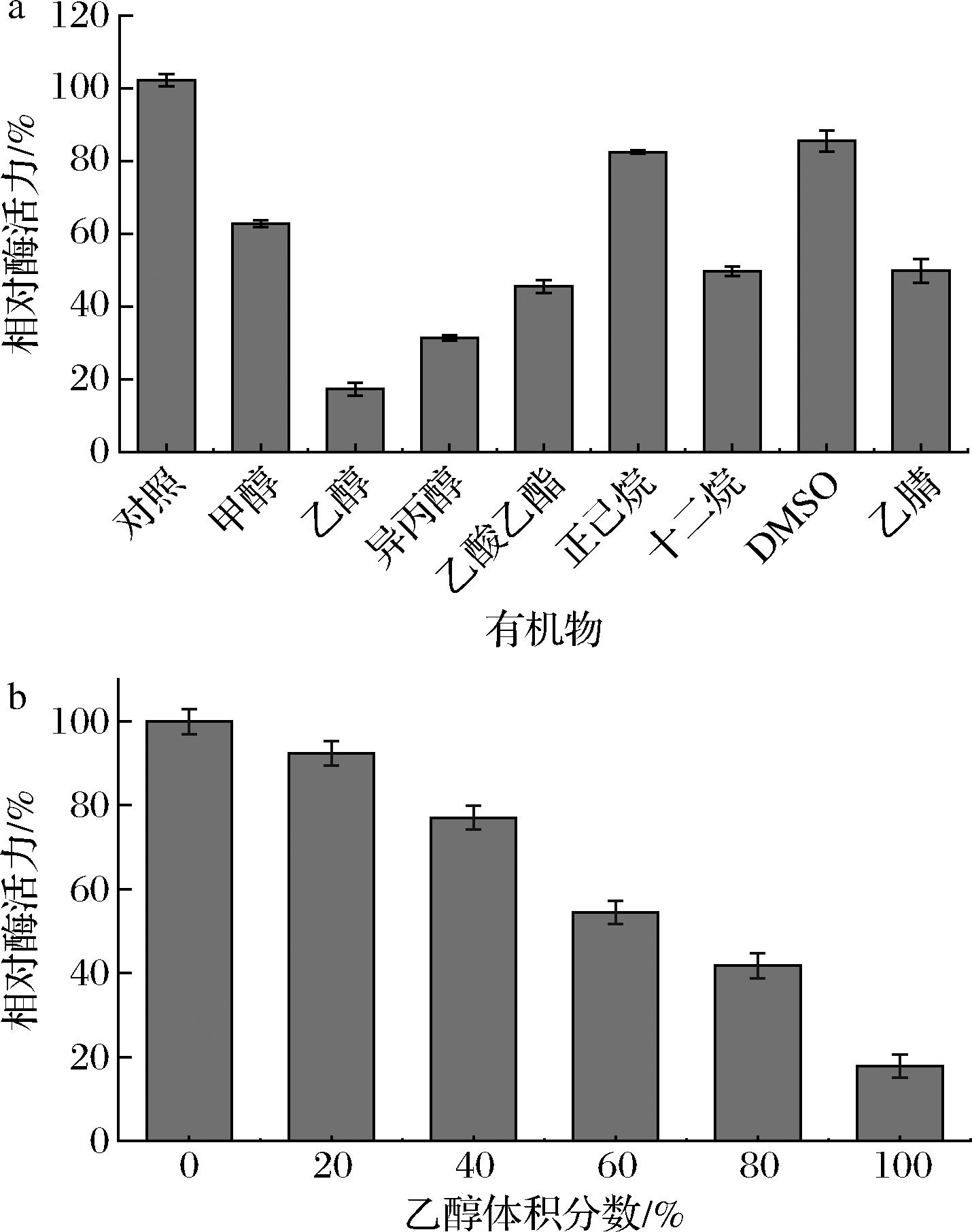
a-不同有机溶剂;b-不同乙醇浓度
图6 有机溶剂对脂肪酶Tgl活力的影响
Fig.6 Effect of organic solvent on lipase Tgl activity
2.3.4 脂肪酶Tgl的催化动力学研究
以对硝基苯酚乙酸酯为底物,分别测定不同底物浓度下的酶活力值,建立动力学曲线回归方程,并确定其拟合良好。由回归方程可计算得到脂肪酶Tgl在不同底物下的Km、Kcat和Kcat/Km,计算结果如表1所示。米氏常数Km值大小表示酶与底物的亲和力的强度,Km越低,与底物的亲和力越高。与文献报道相比,同样以对硝基苯酚乙酸酯为底物时,脂肪酶Tgl的Km(0.03 mmol/L)要远远小于同样来源的脂肪酶Lip A的Km(0.71 mmol/L)和Rhizopus oryzae来源的脂肪酶Km(0.13 mmol/L)[27],其催化效率同样也远大于脂肪酶Lip A的Kcat/Km(95/ms)。
表1 脂肪酶Tgl的催化动力学参数
Table 1 Catalytic kinetic parameters of lipase Tgl
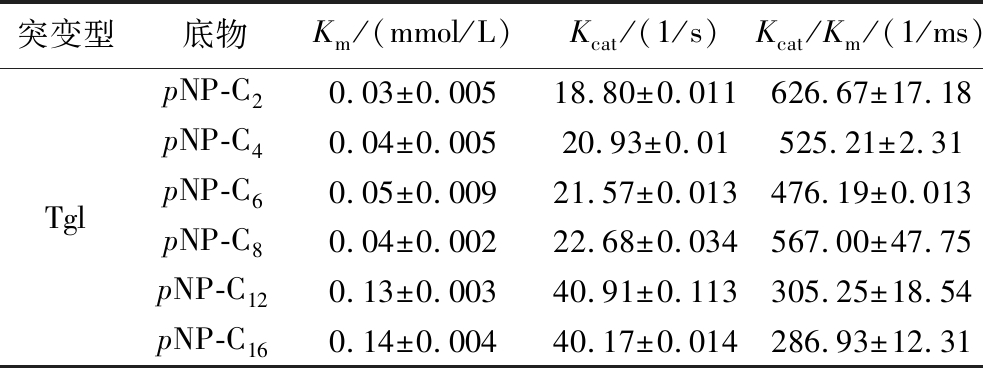
突变型底物Km/(mmol/L)Kcat/(1/s)Kcat/Km/(1/ms)pNP-C20.03±0.00518.80±0.011626.67±17.18pNP-C40.04±0.00520.93±0.01525.21±2.31TglpNP-C60.05±0.00921.57±0.013476.19±0.013pNP-C80.04±0.00222.68±0.034567.00±47.75pNP-C120.13±0.00340.91±0.113305.25±18.54pNP-C160.14±0.00440.17±0.014286.93±12.31
2.3.5 脂肪酶Tgl水相中合成脂肪酸乙酯的能力
脂肪酶Tgl对7种不同链长的酸与乙醇反应生成脂肪酸乙酯的酯化反应的催化能力,结果如图7所示。在20%乙醇的水相反应体系中(图7-a),与添加灭活的酶粉的对照组相比,脂肪酶Tgl对乙酸乙酯合成的催化效率最高,但是对己酸乙酯、庚酸乙酯、辛酸乙酯合成的催化效率很差,几近于空白对照。在60%的乙醇水反应体系中脂肪酶Tgl催化酯合成的结果如图7-b所示。与20%乙醇的水相反应体系相比,己酸乙酯、庚酸乙酯、辛酸乙酯的产量发生了极大的变化,其中,己酸乙酯的产量最大,为171.08 mg/L,其次是庚酸乙酯,其产量为115.615 mg/L。实验结果表明,庚酸乙酯的产量提升最为明显,证明脂肪酶Tgl的酯化能力方面对C7链长的底物特异性最高;且脂肪酶Tgl在60%的乙醇水反应体系中酶活力受抑制的情况下,其酯化效率仍远远高于未受明显抑制的20%乙醇体系,酯化能力更加稳定。目前,关于脂肪酶的酯化反应多是在有机相中进行,例如,固定化脂肪酶lipase TL IM在有机介质中被用于合成柚皮苷脂肪酸酯[27],本研究中的脂肪酶Tgl可在弱酸性的水相反应体系中进行酯化反应生成脂肪酸乙酯更适合酿酒工业的应用。

a-20%乙醇∶酸反应体系;b-60%乙醇∶酸反应体系
图7 脂肪酶Tgl在不同乙醇浓度体系下合成脂肪酸乙酯的能力
Fig.7 The ability of lipase Tgl in the synthesis of ethyl fatty acid esters under different ethanol concentrations
3 结论
本文成功克隆表达了来源于黑曲霉的脂肪酶Tgl。通过多序列比对和结构模拟,确定A75-D83为脂肪酶Tgl的“盖子”结构,及其3个催化中心Ser139、Asp195和His248。经酶学性质分析表明,脂肪酶Tgl的最适反应温度和pH分别为50 ℃和5.0,且在40~50 ℃和pH 4.0~6.0条件下具有较好的稳定性。该酶对不同碳链长度的底物均表现出催化活性,当作用于pNP-C2时,催化活性最高。脂肪酶Tgl对有机试剂均表现出不同程度的耐受性,在40%的乙醇体系下,Tgl的残余酶活仍接近80%。此外,脂肪酶Tgl对乙酸和庚酸的底物特异性较高,20%的乙醇体系中,Tgl催化合成乙酸乙酯的产量最高,而在60%乙醇体系中催化合成己酸乙酯的产量最高。该研究为黑曲霉酸性脂肪酶应用于发酵产品的品质提升提供了理论基础,对其在食品行业中广泛应用和工业化酶制剂种类的丰富具有重要意义。
[1] LEVISSON M, VAN DER OOST J, KENGEN S W M.Carboxylic ester hydrolases from hyperthermophiles[J].Extremophiles, 2009, 13(4):567-581.
[2] CHANDRA P, ENESPA, SINGH R, et al.Microbial lipases and their industrial applications:A comprehensive review[J].Microbial Cell Factories, 2020, 19(1):169.
[3] STERGIOU P Y, FOUKIS A, FILIPPOU M, et al.Advances in lipase-catalyzed esterification reactions[J].Biotechnology Advances, 2013, 31(8):1846-1859.
[4] MAHADIK N D, PUNTAMBEKAR U S, BASTAWDE K B, et al.Production of acidic lipase by Aspergillus niger in solid state fermentation[J].Process Biochemistry, 2002, 38(5):715-721.
[5] FILHO D G, SILVA A G, GUIDINI C Z.Lipases:Sources, immobilization methods, and industrial applications[J].Applied Microbiology and Biotechnology, 2019, 103(18):7399-7423.
[6] SANDANA MALA J G, KAMINI N R, PUVANAKRISHNAN R.Strain improvement of Aspergillus niger for enhanced lipase production[J].The Journal of General and Applied Microbiology, 2001, 47(4):181-186.
[7] XING S Q, ZHU R N, LI C Q, et al.Gene cloning, expression, purification and characterization of a sn-1,3 extracellular lipase from Aspergillus niger GZUF36[J].Journal of Food Science and Technology, 2020, 57(7):2669-2680.
[8] CONG S Z, TIAN K M, ZHANG X, et al.Synthesis of flavor esters by a novel lipase from Aspergillus niger in a soybean-solvent system[J].3 Biotech, 2019, 9(6):244.
[9] XU Y Q, ZHAO J R, LIU X, et al.Flavor mystery of Chinese traditional fermented Baijiu:The great contribution of ester compounds[J].Food Chemistry, 2022, 369(1):130920.[10] XU Y Q, WANG X C, LIU X, et al.Discovery and development of a novel short-chain fatty acid ester synthetic biocatalyst under aqueous phase from Monascus purpureus isolated from Baijiu[J].Food Chemistry, 2021, 338:128025.
[11] XU Y Q, HUANG H Q, LU H Y, et al.Characterization of an Aspergillus niger for efficient fatty acid ethyl ester synthesis in aqueous phase and the molecular mechanism[J].Frontiers in Microbiology, 2022, 12:820380.
[12] ZHAO J R, XU Y Q, LU H Y, et al.Molecular mechanism of LIP05 derived from Monascus purpureus YJX-8 for synthesizing fatty acid ethyl esters under aqueous phase[J].Frontiers in Microbiology, 2023, 13:1107104.
[13] KOBAYASHI T.Lipase-catalyzed syntheses of sugar esters in non-aqueous media[J].Biotechnology Letters, 2011, 33(10):1911-1919.
[14] 蒋翠翠, 尚昊, 张素芳, 等.产脂肪酶菌株的筛选、酶学特性及其接种对酸肉风味物质的影响[J].食品科学, 2023, 44(10):106-113.JIANG C C, SHANG H, ZHANG S F, et al.Screening and enzymatic properties of lipase-producing strains and effects of their inoculation on flavor compounds of fermented sour meat[J].Food Science, 2023, 44(10):106-113.
[15] 王雅琪, 凡林林, 李文瑶, 等.黑曲霉脂肪酶Lip A的异源表达与酶学性质分析[J].食品与发酵工业, 2023, 49(5):25-31.WANG Y Q, FAN L L, LI W Y, et al.Heterologous expression and enzymatic characterization of Aspergillus Niger lipase Lip A[J].Food and Fermentation Industries, 2023, 49(5):25-31.
[16] TRUBITSINA L I, TISHCHENKO S V, GABDULKHAKOV A G, et al.Structural and functional characterization of two-domain laccase from Streptomyces viridochromogenes[J].Biochimie, 2015, 112:151-159.
[17] YAO J, GUI L, YIN S C.A novel esterase from a soil metagenomic library displaying a broad substrate range[J].AMB Express, 2021, 11(1):38.
[18] 王聪, 杜金华, 张开利, 等.小麦芽脂肪酶酶学性质以及制麦过程的酶活变化[J].食品与发酵工业, 2013, 39(5):105-110.WANG C, DU J H, ZHANG K L, et al.Characterization of wheat malt lipase in malting[J].Food and Fermentation Industries, 2013, 39(5):105-110.
[19] 田家英, 张江华, 崔励, 等.固定化脂肪酶制备条件优化及催化应用[J].大连工业大学学报, 2022, 41(6):408-413.TIAN J Y, ZHANG J H, CUI L, et al.Optimization of conditions for preparing immobilized lipase and its catalytic performance[J].Journal of Dalian Polytechnic University, 2022, 41(6):408-413.
[20] ZHU S Y, XU Y, YU X W.Improved homologous expression of the acidic lipase from Aspergillus niger[J].Journal of Microbiology and Biotechnology, 2020, 30(2):196-205.
[21] SALAH R B, MOSBAH H, FENDRI A, et al.Biochemical and molecular characterization of a lipase produced by Rhizopus oryzae[J].FEMS Microbiology Letters, 2006, 260(2):241-248.
[22] KHAN M T, KAUSHIK A C, RANA Q U A, et al.Characterization and synthetic biology of lipase from Bacillus amyloliquefaciens strain[J].Archives of Microbiology, 2020, 202(6):1497-1506.
[23] MOSBAH H, SAYARI A, MEJDOUB H, et al.Biochemical and molecular characterization of Staphylococcus xylosus lipase[J].Biochimicaet Biophysica Acta, 2005, 1723(1-3):282-291.
[24] LIU G, HU S Q, LI L, et al.Purification and characterization of a lipase with high thermostability and polar organic solvent-tolerance from Aspergillus niger AN0512[J].Lipids, 2015, 50(11):1155-1163.
[25] SAXENA R K, DAVIDSON W S, SHEORAN A, et al.Purification and characterization of an alkaline thermostable lipase from Aspergillus carneus[J].Process Biochemistry, 2003, 39(2):239-247.
[26] NAMBOODIRI V M, CHATTOPADHYAYA R.Purification and biochemical characterization of a novel thermostable lipase from Aspergillus niger[J].Lipids, 2000, 35(5):495-502.
[27] 张帆, 程路峰, 曹红, 等.基于酶反应动力学理论优化脂肪酶活力测定体系[J].中国油脂, 2023,48(5):146-152.ZHANG F, CHENG L F, CAO H, et al.Optimization of lipase activity assay system based on enzyme reaction kinetics theory[J].China Oils and Fats, 2023, 48(5):146-152.
[28] 杜理华, 徐亮亮, 成柄灼.一种脂肪酶催化柚皮苷酯化反应的研究[J].浙江工业大学学报, 2017, 45(1):51-55.DU L H, XU L L, CHENG B Z.A study on lipase catalyzed esterification of naringin[J].Journal of Zhejiang University of Technology, 2017, 45(1):51-55.