高血压是一种常见的慢性疾病,是心脑血管疾病最重要的危险因素。世界卫生组织调查报告显示,从1990年到2021年,全球高血压患者人数增加了一倍,到达12.8亿[1],血管紧张素转化酶(angiotensin converting enzyme, ACE)抑制剂是治疗高血压的有效药物[2],但临床所用的ACE抑制剂大多为化学合成的药剂,会产生许多不良反应,如咳嗽、味觉功能紊乱及皮疹等[3-4]。自FERRIRA等[5]首次从南美洲蝮蛇的毒液中发现ACE抑制肽以来,国内外学者已经从各种动植物和海洋生物原料中制备并分离出多种ACE抑制多肽。这些天然ACE抑制肽安全性高,无毒副作用,对高血压患者可以起到降压作用,而对血压正常者无降压作用,还具有免疫调节、减肥和易消化吸收的功能,有着化学合成药物不可比拟的优越性,成为公认的具有巨大发展前景的食品功能因子[6-9]。虽然目前很多学者从食源性蛋白中分离和制备了大量的ACE肽,但是目前真正研发成产品,并应用于生产和临床的天然ACE抑制肽并不多。主要是由于ACE抑制肽的作用机制尚不清楚,严重制约其在食品工业和的临床中的应用[10]。
目前定量构效关系(quantitative structure-activity relationship, QSAR)已成为制药行业开发的一个必不可少的工具[11-12]。QSAR是探究共同作用机制的一系列化学物质的生物活性变化与其结构、物理和化学性质变化的关系[13]。由于多肽物质结构与功能活性相关性质,通过实验或计算手段比使用体外或体内方法能更有效地确定其生物活性,因此统计验证的QSAR模型能够预测同一系列中新物质的生物活性,代替耗时和费力的化学合成和生物评价过程。因此,QSAR可以节省大量的时间、金钱和人力资源。
本研究通过收集食源性ACE抑制二肽的序列建立肽库,并采用Z-scales、VHSE和SVHEHS三种不同的氨基酸描述符对其进行结构表征,随后采用自交协方差法(auto cross covariances, ACC)处理数据,以单个氨基酸侧链的疏水值、立体性质、电性参数和氢键贡献参数为自变量,以ACE抑制二肽的lg(IC50)为因变量,通过偏最小二乘回归法(partial least squares regression, PLS)建立ACE抑制二肽数学模型,并采用“留一法”进行交叉验证,阐明二肽结构与ACE抑制活性的构效关系,并对氨基酸序列二肽的ACE抑制进行预测和验证,以期找到ACE抑制肽准确的定量构效关系,为进一步阐明食源性ACE抑制肽的作用机理提供参考,为其深度开发利用奠定基础。
1 材料与方法
1.1 食源性ACE抑制二肽肽库构建
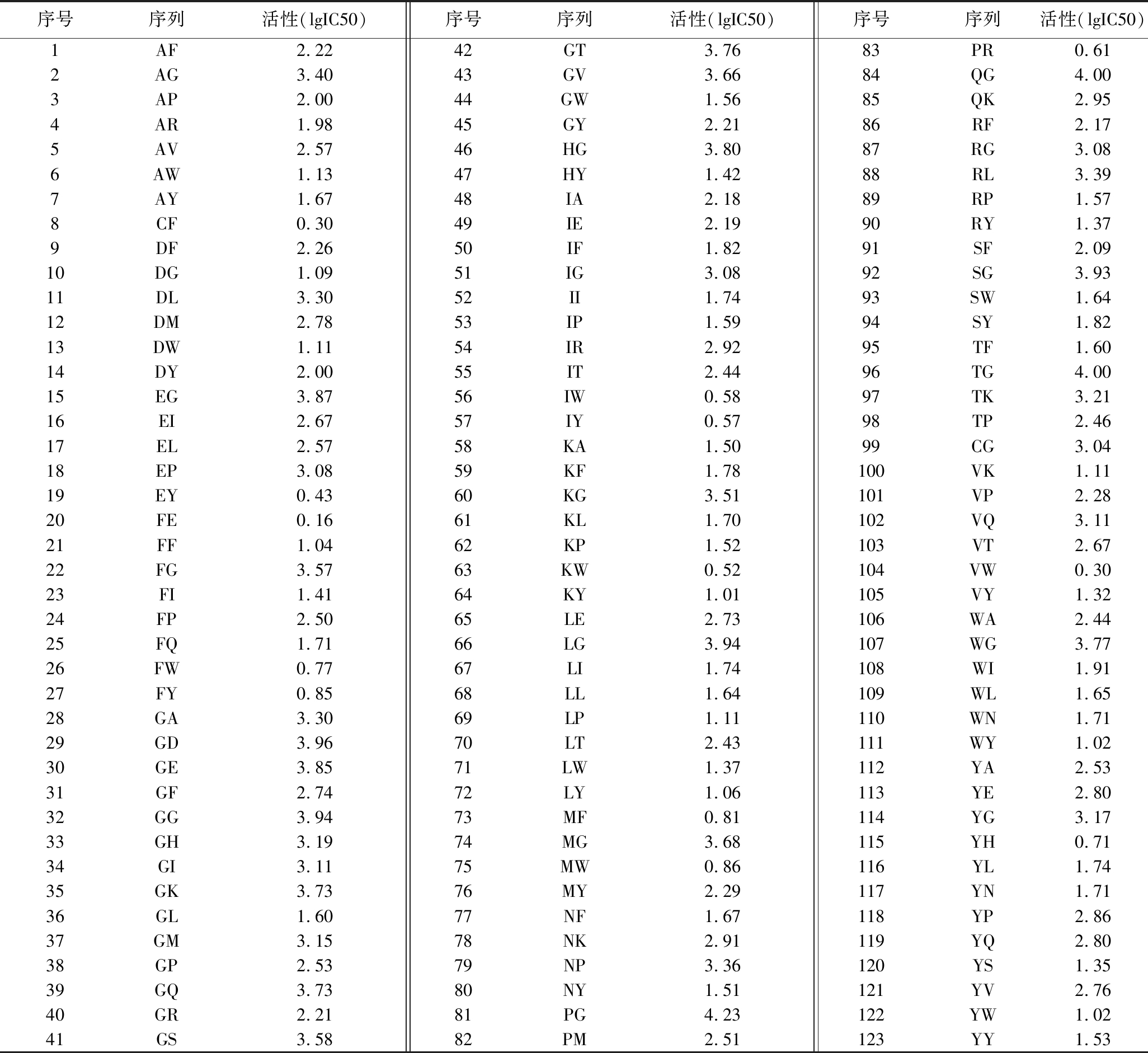
通过收集国内外文献中报道的食源性ACE抑制二肽组建肽库(https://doi.org/10.13995/j.cnki.11-1802/ts.036602),对于不同来源中肽序列相同的,但报道的IC50值却存有差异的ACE抑制肽,选较优的IC50值,为了便于分析和建模,本研究采用lg(IC50)对数据作统一化处理[14]。
1.2 基于Z-scales、VHSE和VHEHS三种描述符的二肽结构描述
1.2.1 20种天然氨基酸Z-scales描述符
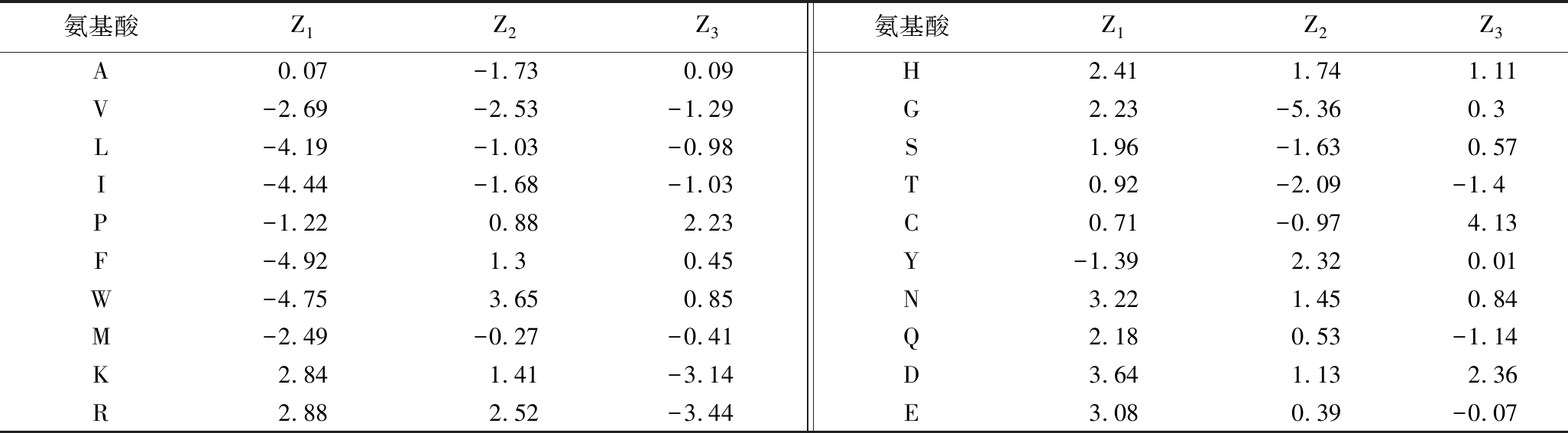
根据HELLBERG等[15]所提出的Z-scales描述符,从天然氨基酸的29种物理和化学参数中,采用主成分分析方法提取与ACE抑制肽相关性较高的参数作为自变量,分别为氨基酸残基的疏水性Z1、立体性Z2和电性Z3(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)。
1.2.2 20种天然氨基酸VHSE氨基酸描述符
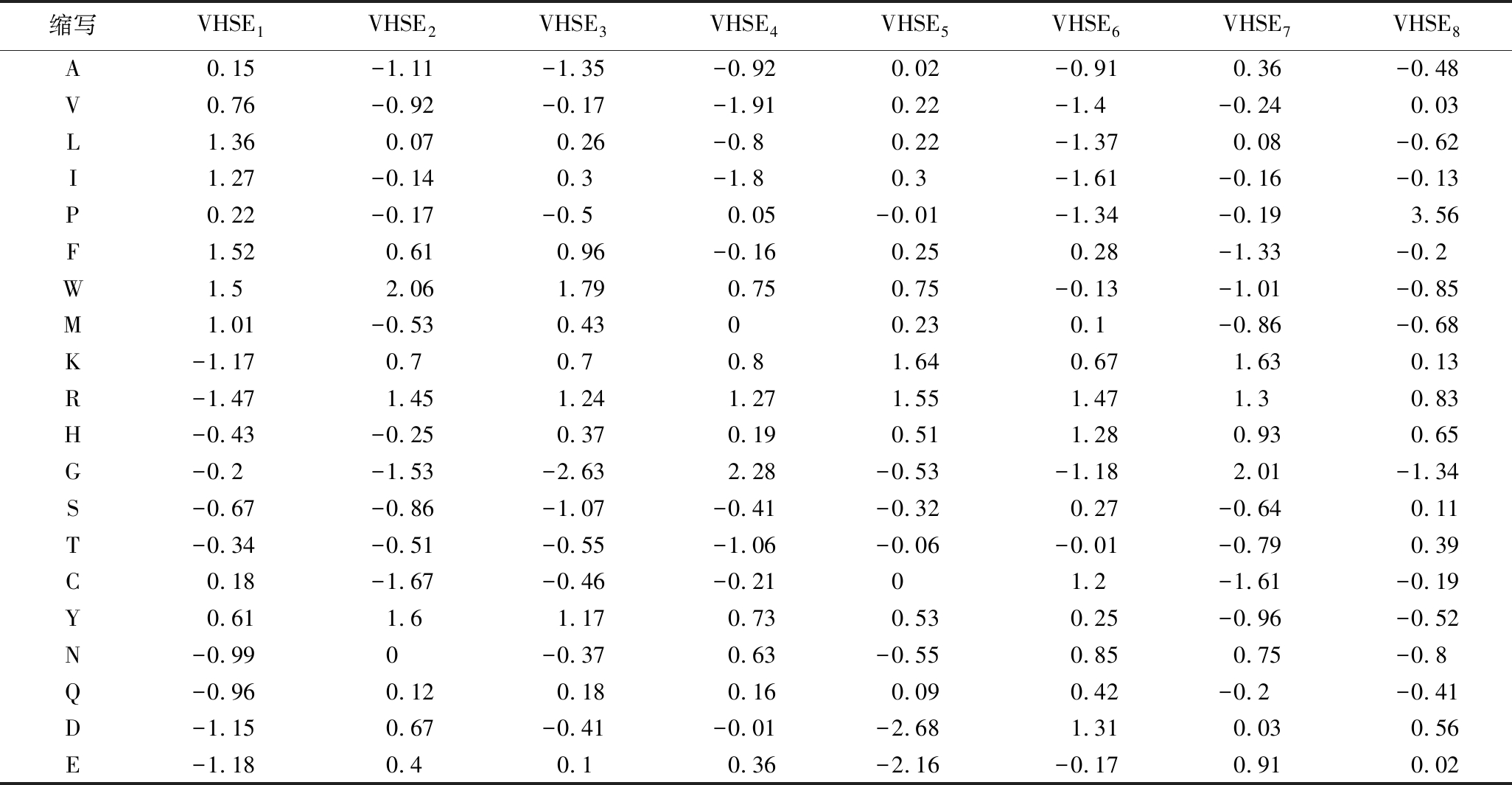
VHSE氨基酸描述符是将20种氨基酸的性质依照疏水性、立体性和电性特征分成3种类型的数据,然后分别对这3种参数进行主成分分析[16],并将该矢量称之为VHSE,其中VHSE1、VHSE2表示氨基酸的疏水性特征值,VHSE3、VHSE4表示氨基酸的立体特征值,VHSE5~VHSE8则表示氨基酸的电性特征值[17](https://doi.org/10.13995/j.cnki.11-1802/ts.036602)。
1.2.3 20种天然氨基酸VHEHS描述符
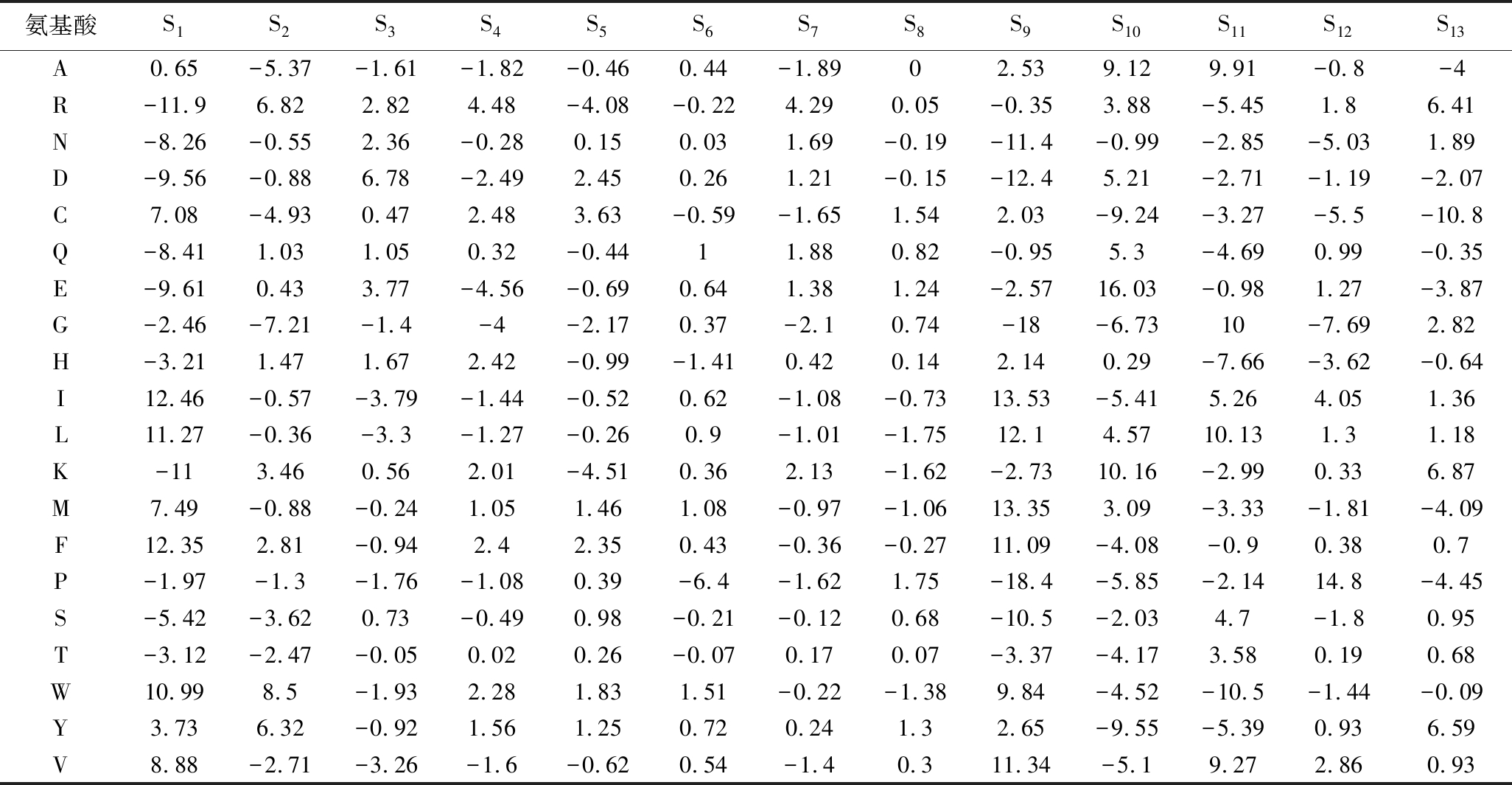
VHEHS描述符是从AA index据库中收集了关于20种天然氨基酸的457个理化性质参数变量,这些参数分为疏水、电性、氢键贡献及立体特征四类,对这四类参数变量进行主成分分析,称该分矢量为SVHEHS[18]。其中S1~S2代表氨基酸疏水性特征,S3~S6代表氨基酸电性特征,S7~S8代表氨基酸氢键贡献参数特征,S9~S13代表氨基酸的立体特征(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)。
1.3 123个食源性ACE抑制二肽活性及Z-scales、VHSE、SVHEHS描述值
应用附表2~附表4(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)中的Z-scales、VHSE、SVHEH三种描述符对这123个ACE抑制二肽进行结构表征,并将每个二肽中氨基酸残基的参数作为自变量,以ACE抑制二肽的lg(IC50)作为模型的因变量,应用PLS对多重自变量和因变量进行多元回归分析,建立QSAR模型。数据统计分析采用MATLAB R2018a软件在PC上完成。
1.4 PLS建立食源性ACE抑制二肽定量构效模型
对收集到的123个样本,采用PLS建立数学模型,分析模型方程的标准回归系数,剔除对因变量影响不显著的自变量,重新建立数学模型,得到优化的较佳模型。该方法主要原理是:首先要将列举的变量数据矩阵进行双线性分解[19],即如公式(1)所示:
X=TPn+F
(1)
T为矩阵X的线性拟合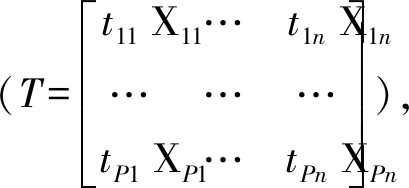 t为自变量的潜变量,F为常数,如公式(2)所示:
t为自变量的潜变量,F为常数,如公式(2)所示:
Y=UQn+E
(2)
U为矩阵Y的线性拟合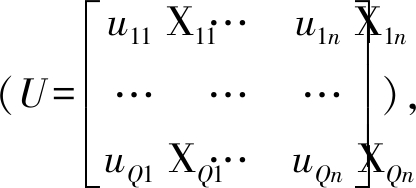 u为因变量的潜变量,E为常数。
u为因变量的潜变量,E为常数。
在此基础上,PLS要求从矩阵X的分解中得到的潜变量t与从矩阵Y的分解中得到的潜变量u有最大的重叠或关联性,如公式(3)所示:
u=vt+e
(3)
式中:e为残差矢量,系数v则由PLS决定。根据上述要求潜因变量T和U应最大限度的代表数据矩阵X和数据矩阵Y中的信息。
PLS算法:
以ACE抑制二肽的lg(IC50)为因变量Y,以ACE抑制二肽的理化性质为自变量X,并建立矩阵:[Y1X1X2X3 …],并对矩阵中的数据进行筛选,剔除掉活性差的数据;对矩阵数据进行标准化处理,然后对矩阵数据计算回归系数;剔除矩阵数据中回归系数小于0.05的数据,创建修改后的特征矩阵;特征矩阵X中添加一列1,以考虑线性回归模型中的截距,重新建立回归系数;将修改后的特征矩阵X与回归系数相乘,得到预测Y值;循环的每次迭代都会从特征矩阵X(以xi表示)和目标向量Y(以yi表示)中剔除一个数据点,以创建验证集,其余数据用于训练;通过计算模型的Q2和R2对模型的预测能力和拟合能力进行检验。
1.5 模型的检验与预测
模型建立后,用“留一法”检验模型的稳定性和预测能力,即每次从样本中抽出一种肽,用其余的二肽建模并预测该抽出的二肽的lg(IC50)值,从而得到了各个二肽的lg(IC50)预测值,应用所得数据计算出交叉验证相关系数Q2,如公式(4)所示:
Q2=1-∑(yexp-ypre)2/∑(yexp-yave)2
(4)
式中:yexp表示实际测的活性值;ypre表示交叉验证的活性预测值;yave表示所有肽的平均活性值。
1.6 多肽ACE抑制能力测定
用50 mmol/L硼酸钠缓冲液制得5 mmol/L马脲酰组氨酰亮氨酸(hippuryl-His-Leu-OH,HHL)溶液,将ACE抑制二肽稀释成不同浓度溶液,将20 μL ACE抑制二肽溶液与120 μL HHL溶液混合,然后加入10 μL ACE溶液(0.1 U/mL),于37 ℃下反应60 min。加入50 μL 1 mmol/L盐酸(终止反应,加入1 mL乙酸乙酯充分振荡萃取,10 000 r/min离心5 min,收集酯层并干燥。冷却后加入去离子水溶解残余物,于228 nm处测量吸光度。以硼酸盐缓冲液作为空白对照。在反应前,150 μL盐酸(1 mmol/L)灭活ACE作为阳性对照[],如公式(5)所示:
ACE抑制率![]()
(5)
式中:Aa为样品吸光度;Ab空白对照吸光度;Ac阳性对照吸光度。
将样品溶液稀释至不同浓度,测定ACE抑制率,以ACE抑制率为纵坐标,样品浓度为横坐标,利用MATLAB R2018a进行线性拟合计算IC50。
1.7 分子对接模拟
首先从PDB数据库(https://www.rcsb.org/)中下载ACE蛋白文件(2X8J),使用AutoDockTools 1.5.7对ACE蛋白进行去水、去配体和加全氢,并设置为受体,使用ChemDraw 21绘制3种ACE抑制二肽分子,并使用AutoDockTools 1.5.7对小分子进行加全氢、检测扭转键,并设置为最低能量构型,然后设置为配体。在此基础上通过AutoDockTools 1.5.7逐一对接。对接完成后,使用PyMOL进行结果可视化分析。
1.8 统计与分析
数据统计分析采用MATLAB R2018a软件在PC上完成。试验数据Origin Pro 2021软件进行作图。
2 结果与分析
2.1 基于的Z-scales描述符QSAR模型建立
2.1.1 PLS模型初步建立
以所组建的肽库中的123个ACE抑制二肽利用PLS研究建立ACE抑制二肽的结构与ACE抑制活性关系的QSAR模型方程,该方程的回归方程标准回归系数值见附表5(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)。
2.1.2 模型优化与构效关系分析
由附表5(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)可知,自变量X6的标准回归系数绝对值小于0.05,对模型的影响不显著,可以剔除该自变量进一步优化模型,重新建立数学模型。得出标准化回归方程:
Y=0.295 1X1-0.220 7X2+0.172 4X3+0.338 3X4-0.603 5X5;
将标准化变量表示为原变量回归方程:
Y=2.437 1+0.095 3X1-0.082 1X2+0.132 5X3+0.114 7X4-0.213 0X5。
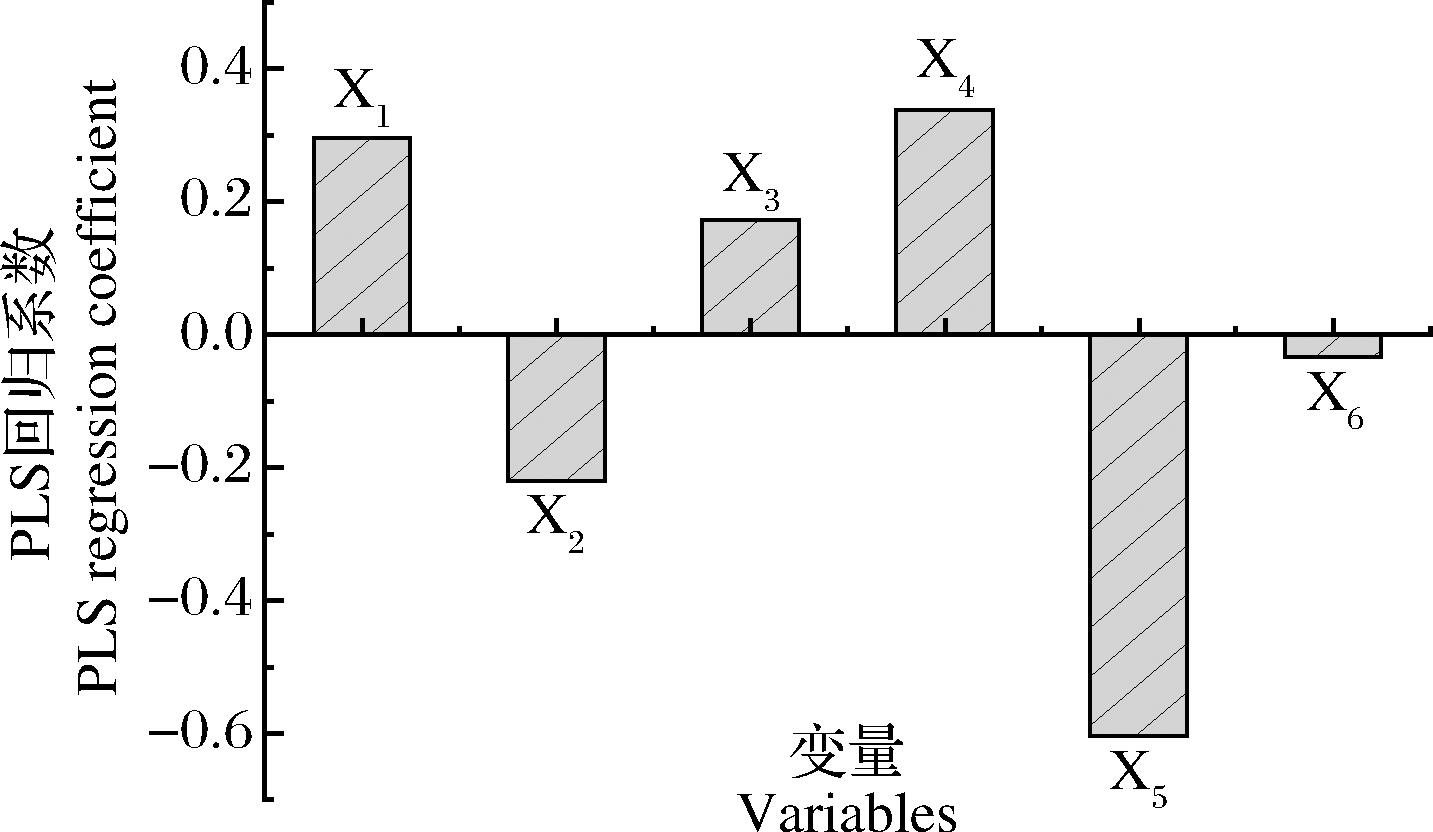
样本数为123,R2为0.847 7(R2表示模型的拟合能力,R2>0.600),说明模型能够解释的样本占总样本的比例越大,模型的相关性越好,拟合效果越好。由回归模型的相关系数可知(附图1,https://doi.org/10.13995/j.cnki.11-1802/ts.036602),其中X1、X4与组成的单个氨基酸的疏水特性相关,说明疏水特性越强该肽ACE抑制活性越低,X2、X5与组成的单个氨基酸的立体特性相关,说明立体特性越强该肽ACE抑制活性越高。
2.1.3 QSAR模型的检验和预测
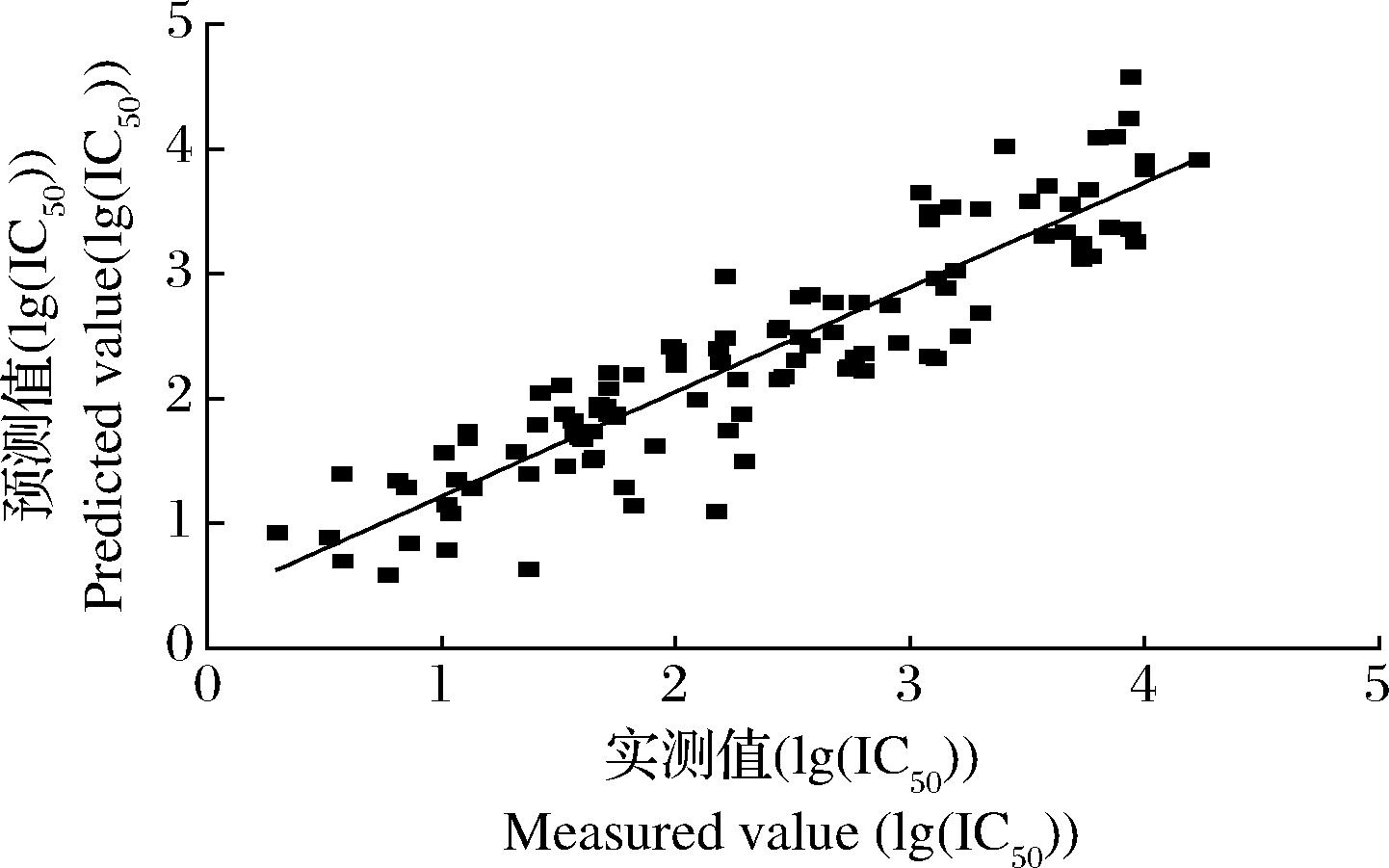
为了考察所建模型的稳定性和预测能力,用“留一法”对数据作交叉检验,计算抽出的123个二肽样本的ACE抑制活性的预测值,求出交叉验证相关系数Q2=0.828 4(Q2>0.500说明模型稳定性好,预测能力也就越强)。将二肽ACE抑制活性的预测值和实验值作拟合线性图(附图2,https://doi.org/10.13995/j.cnki.11-1802/ts.036602),所建模型得到的预测值和文献实验值具有很好的线性关系(R2=0.910 2),表明所建模型具有良好的预测能力。
2.2 基于的VHSE描述子QSAR模型建立
2.2.1 PLS模型初步建立
以所组建的肽库中的123个ACE抑制二肽利用PLS研究建立ACE抑制二肽的结构与ACE抑制活性关系的QSAR模型方程,该方程的回归方程标准回归系数值见附表6(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)。
2.2.2 模型优化与构效关系分析
由附表6(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)可知,自变量X8、X13的标准回归系数绝对值小于0.05,对模型影响不显著,可以剔除该自变量进一步优化模型,重新建立数学模型:
Y=-0.299 5X1-0.070 8X2-0.136 3X3+0.240 3X4-0.107 8X5-0.142 6X6-0.224 6X7-0.413 2X9-0.917 5X10+0.440 3X11+0.229 6X12-0.218 6X14-0.115 3X15-0.152 3X16;
将标准化变量表示为原变量回归方程:
Y=2.449 4-0.278 7X1-0.054 2X2-0.115 5X3+0.189 4X4X4-0.119 3X5-0.140 4X6-0.193 2X7-0.478 9X9-0.788 2X10+0.302 0X11+0.202 8X12-0.288 8X14-0.127 0X15-1 173X16。
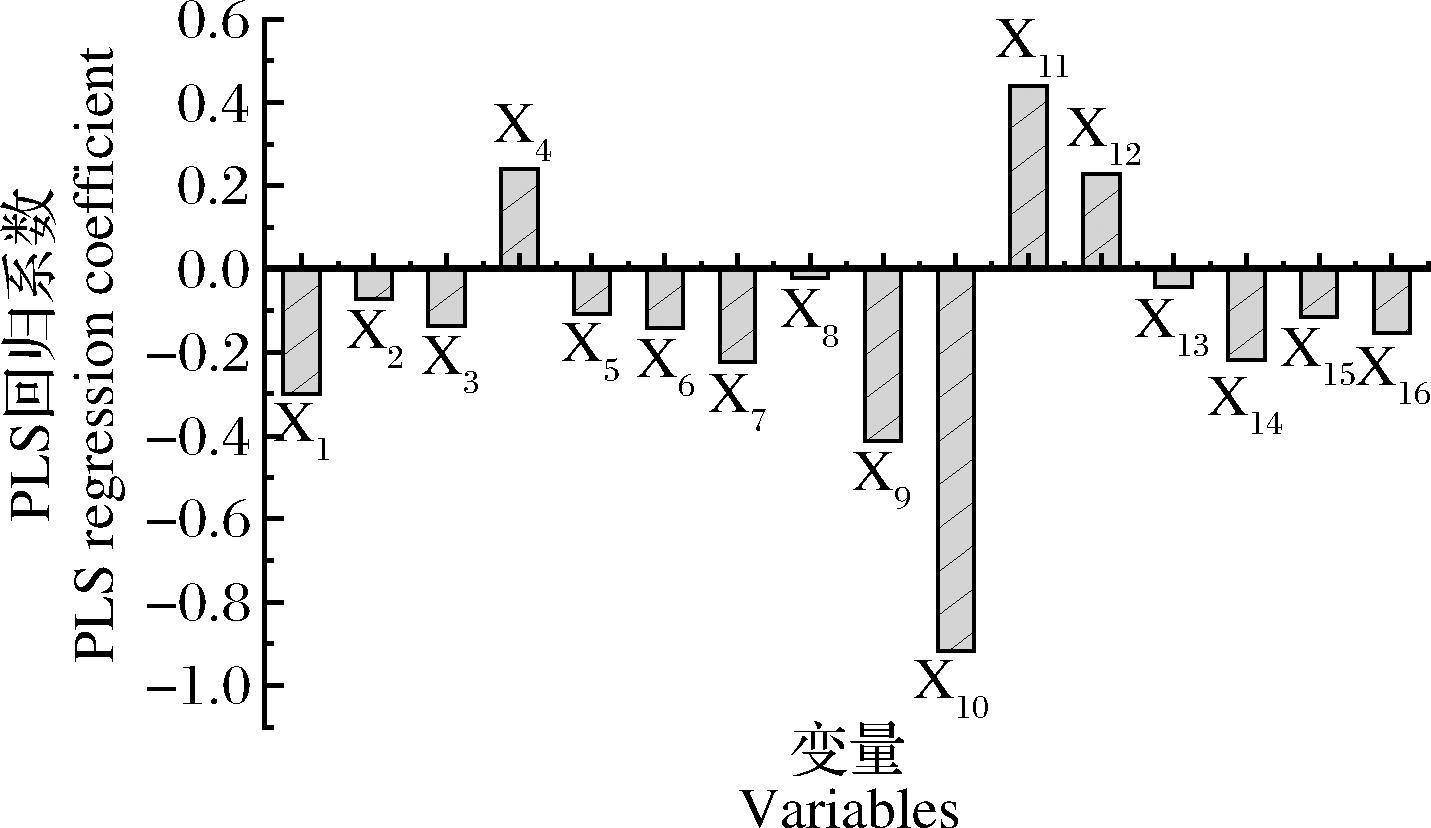
样本数为123,R2为0.856 8(R2>0.600 0),说明模型的相关性较好,拟合效果较好。由回归模型的相关系数可知(附图3,https://doi.org/10.13995/j.cnki.11-1802/ts.036602),X1、X2、X3、X5、X6、X7、X8、X9、X10、X13、X14、X15、X16与二肽的活性呈正相关,X4、X11、X12与二肽的活性呈负相关,ACE抑制二肽从N端开始第一位氨基酸的疏水特性(X1)立体特性(X4)以及第二位氨基酸的疏水特性(X9、X10)、立体特性(X11、X12)对ACE抑制二肽的活性较大,N端第二位氨基酸的疏水特性影响较为显著,其疏水特性对活性起正向作用。
2.2.3 QSAR模型的检验和预测
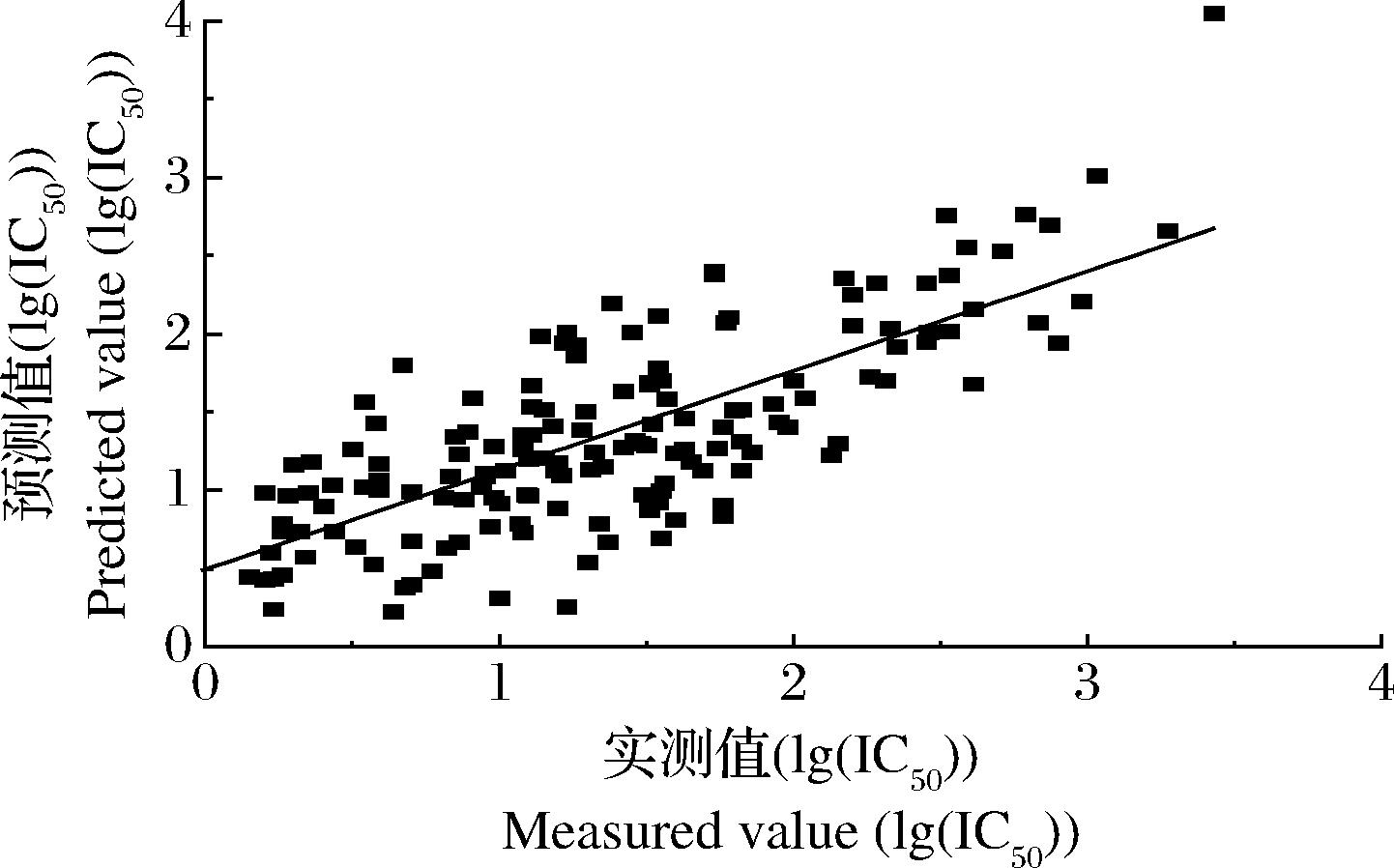
为了考察所建模型的稳定性和预测能力,用“留一法”对数据作交叉检验,计算抽出的123个二肽样本的ACE抑制活性的预测值,求出交叉验证相关系数Q2=0.805 2。将二肽ACE抑制活性的预测值和实验值作拟合线性图(附图4,https://doi.org/10.13995/j.cnki.11-1802/ts.036602),所建模型得到的预测值和文献实验值具有很好的线性关系(R2=0.897 9)。表明所建模型具有良好的预测能力。
2.3 基于的SVHEHS描述子QSAR模型建立
2.3.1 PLS模型初步建立
以所组建的肽库中的123个ACE抑制二肽利用PLS研究建立ACE抑制二肽的结构与ACE抑制活性关系的QSAR模型方程,该方程的回归方程标准回归系数值见附表7(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)。
2.3.2 模型优化与构效关系分析
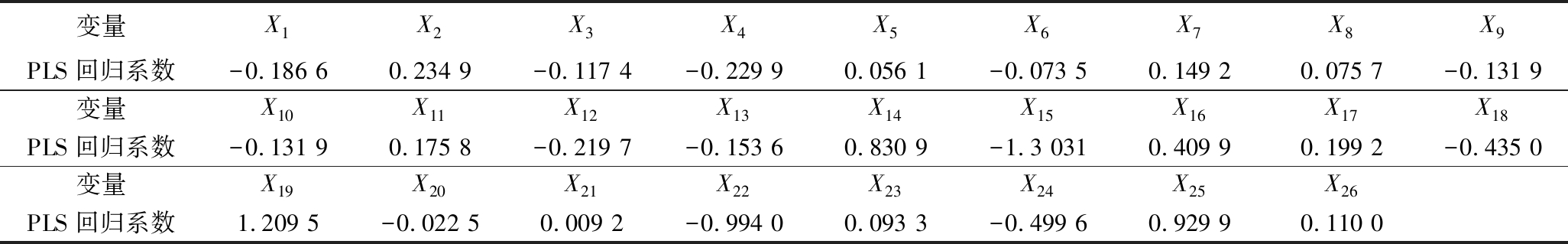
由附表7可知,自变量X20、X21的标准回归系数绝对值小于0.05,对模型影响不显著,可以剔除该自变量进一步优化模型,重新建立数学模型:
Y=-0.186 6X1+0.234 9X2-0.117 4X3-0.229 9X4+0.056 1X5-0.073 5X6+0.149 2X7+0.075 7X8-0.131 9X9-0.131 9X10+0.175 8X11-0.219 7X12-0.153 6X13+0.830 9X14-1.303 1X15+0.409 9X16+0.199 2X17-0.435 0X18+1.209 5X19-0.994 0X22+0.093 3X23-0.499 6X24+0.929 9X25+0.110 0X26;
将标准化变量表示为原变量回归方程:
Y=2.351 9-0.022 2X1+0.048 5X2-0.047 7X3-0.093 1X4+0.029 2X5-0.071 0X6+0.093 8X7+0.076 2X8-0.011 8X9-0.018 1X10+0.025 4X11-0.053 4X12-0.045 6X13+0.105 5X14-0.261 8X15+0.216 0X16+0.075 2X17-0.237 7X18+0.527 9X19-0.082 6X22+0.013 6X23-0.071 5X24+0.153 0X25+0.031 6X26。
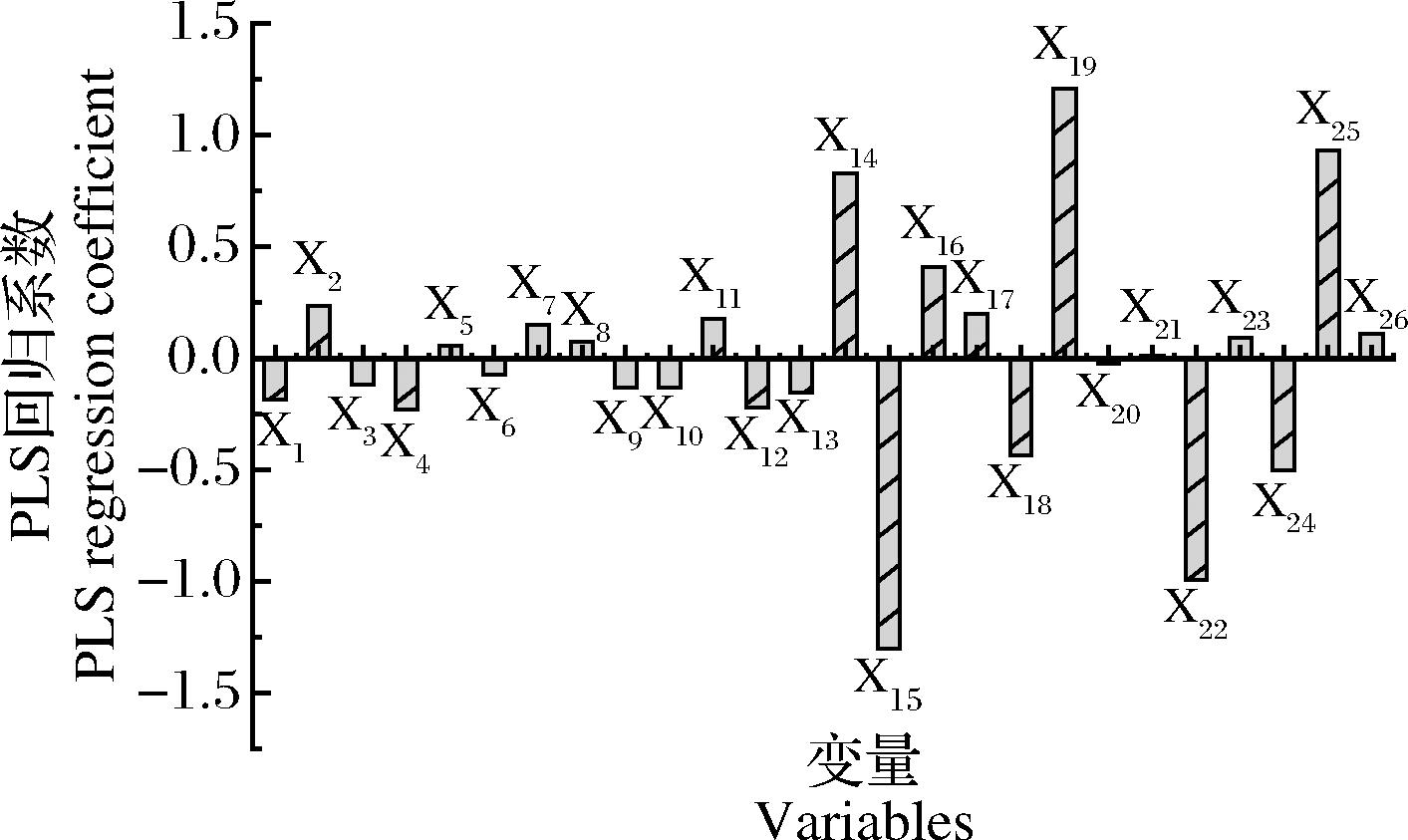
样本数为123,R2为0.858 1(R2>0.600 0),说明模型的相关性好,拟合效果好;所得模型的自变量标准回归系数见附图5(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)。由回归模型的相关系数可知,X1、X3、X4、X6、X9、X10、X12、X13、X15、X18、X20、X22、X24与二肽的活性呈正相关,X2、X5、X7、X8、X11、X14、X16、X17、X19、X21、X23、X25、X26与二肽的活性呈负相关,ACE抑制二肽从N端开始第二位氨基酸的其疏水特征(X14、X15)、电性特征(X19)、立体特征(X22、X24、X25)对ACE抑制肽活性影响较大,尤其第二位氨基酸疏水特征影响较为显著,其疏水特性对活性起正向作用。
2.3.3 QSAR模型的检验和预测
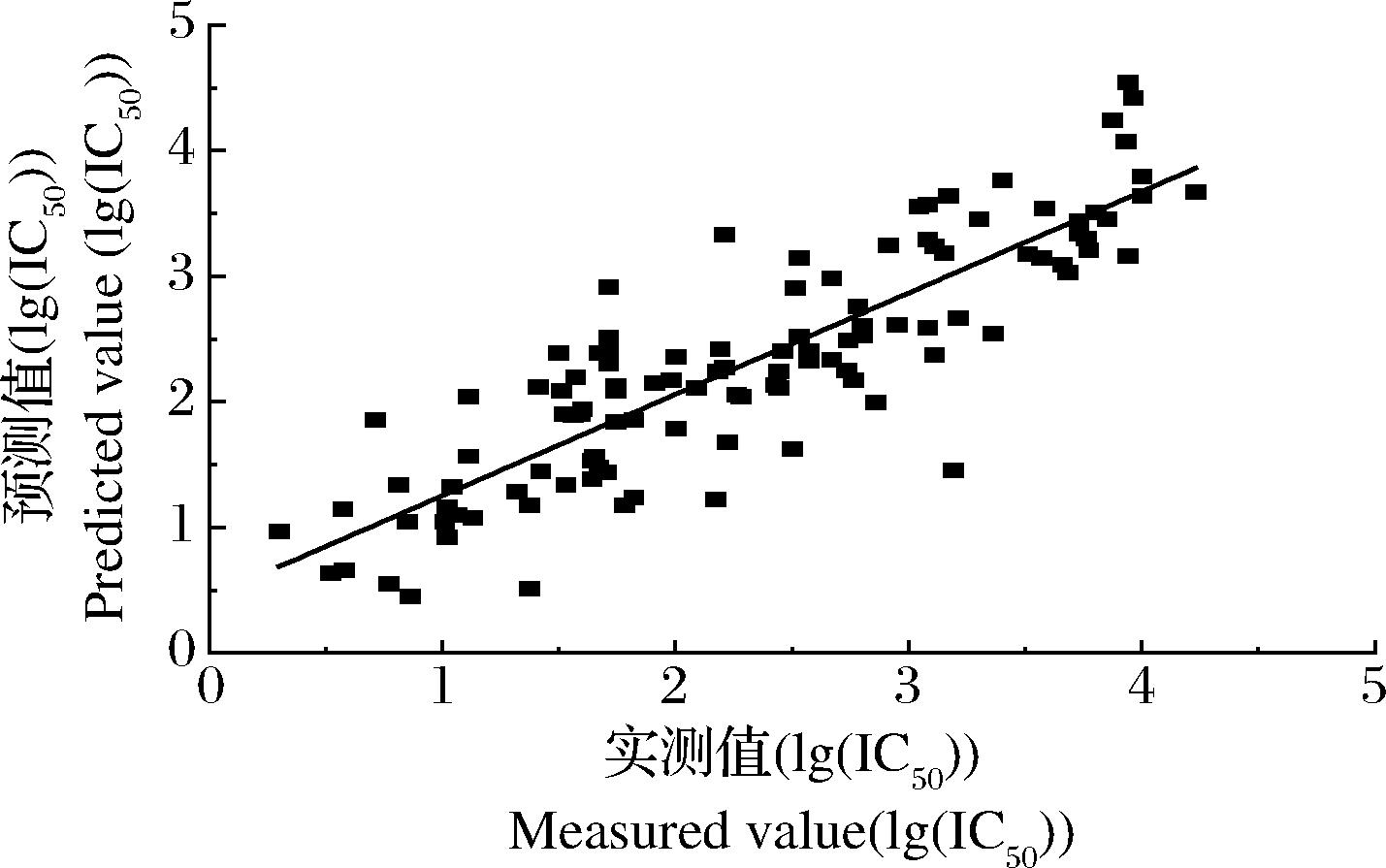
为了考察所建模型的稳定性和预测能力,用“留一法”对数据作交叉检验,计算抽出的123个二肽样本的ACE抑制活性的预测值,求出交叉验证相关系数Q2=0.7453。将二肽ACE抑制活性的预测值和实验值作拟合线性图(附图6,https://doi.org/10.13995/j.cnki.11-1802/ts.036602),所建模型得到的预测值和文献实验值具有很好的线性关系(R2=0.865 8),表明所建模型预测能力良好。
2.4 不同氨基酸描述符对ACE抑制二肽定量构效建模效果比较
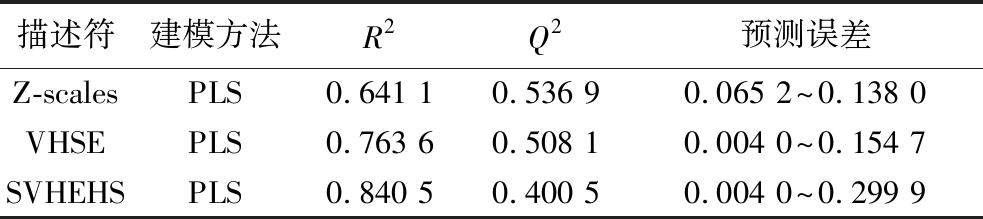
采用3种氨基酸描述符对123种ACE抑制二肽的性质进行表征,通过PLS法进行建模,不同氨基酸描述符的参数如附表8(https://doi.org/10.13995/j.cnki.11-1802/ts.036602)所示,3种建模方法,Z-scales描述符R2为0.847 7,Q2为0.828 4,对已知活性的ACE抑制二肽的预测误差为0.008 5~0.182 1;VHSE描述符R2为0.856 8,Q2为0.805 2,对已知活性的ACE抑制二肽的预测误差为0.030 4~0.210 0。SVHEHS描述符R2为0.858 1,Q2为0.745 3,对已知活性的ACE抑制二肽的预测误差为0.019 6~0.096 7。3种氨基酸描述符中,SVHEHS描述符的拟合能力最强,但是其预测能力较弱,Q2为0.845 3,而Z-scales描述符预测能力相较另外2种氨基酸描述符较强,但3种建模方法的拟合能力和预测能力没有明显差异性,且3种建模方法均较为合适地对ACE抑制二肽进行定量构效分析。
2.5 ACE抑制二肽预测与合成
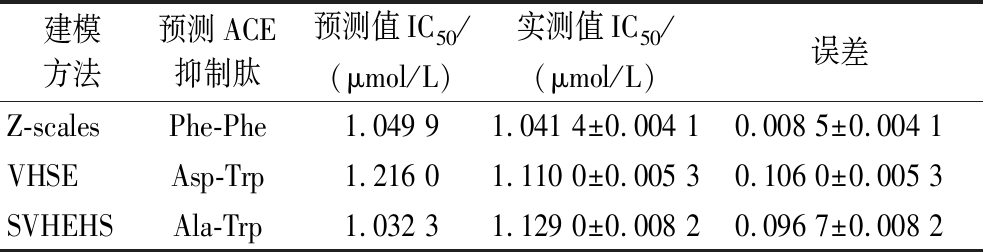
通过3种描述符建立定量构效模型,分别对ACE抑制二肽进行预测,分别预测出活性较优的ACE抑制二肽,并通过化学合成,测定其体外活性(附表9,https://doi.org/10.13995/j.cnki.11-1802/ts.036602),其中基于Z-scales描述符建立的定量构效模型预测出活性较优的ACE抑制二肽为Phe-Phe,预测活性IC50为1.049 9 μmol/L,通过化学合成,测得体外活性测定IC50为(1.041 4±0.004 1) μmol/L;基于VHSE描述符建立的定量构效模型预测出活性较优的ACE抑制二肽Asp-Trp,预测活性IC50为1.216 0 μmol/L,通过化学合成,测得体外活性IC50为(1.110 0±0.005 3) μmol/L;基于SVHEHS描述符建立的定量构效模型预测出较优的ACE抑制二肽为Ala-Trp,预测活性IC50为1.03 23 μmol/L,通过化学合成,测得体外活性IC50为(1.129 0±0.008 2) μmol/L。
2.6 分子对接
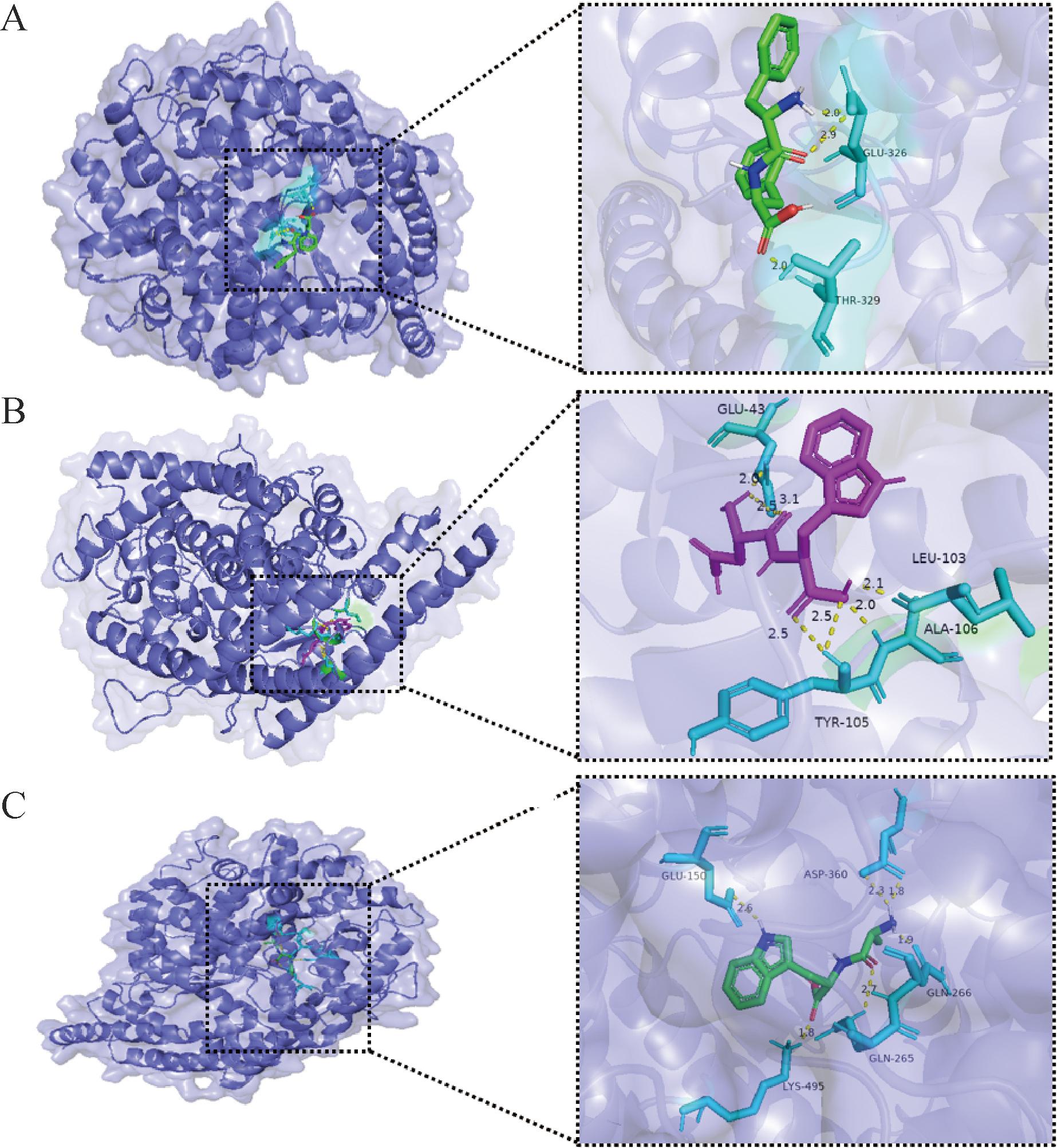
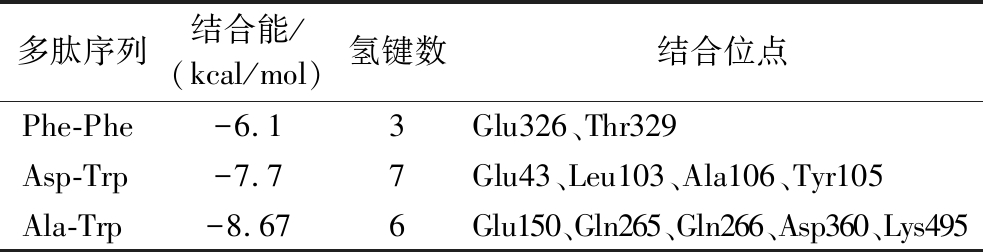
通过3种ACE抑制肽与ACE蛋白(2X8 J)进行分子对接发现,ACE抑制二肽Phe-Phe、Asp-Trp、Ala-Trp均可与ACE蛋白进行结合(附图7,附表10,https://doi.org/10.13995/j.cnki.11-1802/ts.036602)。从Phe-Phe与ACE的相互作用图可以看出,Phe-Phe与ACE活性口袋中的Glu326、Thr329氨基酸残基相互作用,共形成3个氢键(附图7-A,https://doi.org/10.13995/j.cnki.11-1802/ts.036602);从Asp-Trp与ACE的相互作用图可以看出,Asp-Trp与ACE活性口袋中的Glu43、Leu103、Ala106、Tyr105氨基酸残基相互作用,共形成7个氢键(附图7-B,https://doi.org/10.13995/j.cnki.11-1802/ts.036602);从Ala-Trp与ACE的相互作用图可以看出,Ala-Trp与ACE活性口袋中Glu150、Gln265、Gln266、Asp360、Lys495氨基酸残基相互作于,共形成6个氢键(附图7-C,https://doi.org/10.13995/j.cnki.11-1802/ts.036602)。其中Phe-Phe与ACE的结合能为-6.1 kcal/mol,Asp-Trp与ACE的结合能为-7.7 kcal/mol,Ala-Trp与ACE的结合能为-8.76 kcal/mol。
3 讨论
定量构效关系模型是一种寻找结构与活性之间关系的数学表达方法,其基本假设是化合物的生物学活性与其结构密切相关,如电子属性,疏水性和空间属性,这导致数学模型中有多个变量数据[21]。QSAR研究方法最早由MEKENYAN等[22]确立,用于构建分子的电效应和立体效应与活性相联系的定量模型。随着计算机辅助手段的发展,QSAR从二维发展到六维。建模的分析方法有多元线性回归(multivariable linear regression, MLR)、PLS、支持向量随机(support vector machine, SVM)、主成分分析(principal component analysis, PCA)、逐步线性回归(stepwise regression, SLR)、人工神经网络(artificial neural network, ANN)和遗传算法-偏最小二乘(genetic algorithm-PLS, GA-PLS)等[23-24]。目前,定量构效关系在研究化学和生物相互作用等许多方面具有很重要的作用。例如多肽QSAR建模已成功地用于研究抗菌剂ACE抑制剂和苦味肽。
多肽的QSAR研究中,首先对一系列多肽类似物进行适当的数学描述是至关重要的,现在普遍采用氨基酸残基的物理化学性质来定量描述多肽的化学结构。本文利用了Z-scales、VHSE、SVHEHS三种描述符分别对自组建的ACE抑制肽库进行了描述,并采用PLS进行了QSAR研究,通过3种不同描述符定量构效模型发现:Z-scales描述符对ACE抑制二肽氨基酸残基的理化特性描述较为模糊,该描述符对氨基酸残基每种特性仅用一种参数进行描述,建立的模型拟合能力较弱(R2=0.847 7),但是由于其参数较为简单,因此该模型的噪音较弱,预测能力较强(Q2=0.828 4);VHSE描述符利用主成分分析对氨基酸的疏水特性参数、立体特性参数和电性特征参数进行选择,选择对其理化特性影响较大的参数,对氨基酸理化特性描述较为精确,建立的模型拟合能力较强(R2=0.856 8),但是预测能力较弱(Q2=0.805 2);VHEHS描述符可对氨基酸进行更为详细的描述,建立的模型拟合能力最强(R2=0.858 1),但是预测能力最弱(Q2=0.745 3)。因此氨基酸描述符对氨基酸理化特性描述的越为准确,越能描述出多肽结构与功能的关系[25]。梅虎等[17]通过VHSE描述符对苦味二肽定量构效模型建模,R2和Q2分别为0.881和0.843,对血管缓激肽促进剂二肽进行定量构效建模,R2和Q2分别为0.959 0和0.690 0,并且发现氨基酸的疏水、立体特性与其生物活性密切相关。仝建波等[26]通过对20种天然氨基酸的721个3D结构信息描述符经过主成分分析得到氨基酸描述符SVTD,并通过对72个ACE抑制二肽进行PLS建立定量构效关系模型,其相关系数R2=0.998 0,Q2=0.804 0。
由3种描述符定量构效模型可知,ACE抑制肽N端第二位氨基酸的疏水特征越强,其ACE抑制能力越强,例如带有色氨酸、酪氨酸、苯丙氨酸等带有苯环的芳香族氨基酸ACE抑制肽活性较强;其次该位置氨基酸的立体特征越强,ACE抑制能力越强,例如带有色氨酸、酪氨酸、精氨酸等氨基酸的ACE抑制肽活性较强。WU等[27]表示在C端含有疏水性或芳香性氨基酸,如色氨酸、脯氨酸和苯丙氨酸,以及疏水性氨基酸残基如甘氨酸、亮氨酸、异亮氨酸和缬氨酸等的ACE抑制肽会增强其抑制ACE的能力。CHEUNG等[28]对ACE抑制三肽的构效关系分析表明,C端为芳香族氨基酸且N端为支链氨基酸的三肽具有较高的活性。因此ACE抑制肽的疏水性、立体特征对ACE抑制肽活性有积极的影响。
受体蛋白与小分子配体的结合强度取决于对接过程中结合能的变化,从热力学的角度来看,结合能越低,分子之间结合的越稳定紧密。另外氢键、疏水作用力、离子共价结合能力、范德华力等都是影响配体与受体结合能力的重要作用力,其中氢键能显著提高多肽和ACE结合的稳定性,因此探究多肽与ACE之间的作用力,能间接判断该多肽的抑制能力。本研究通过分子对接模拟发现,Phe-Phe与ACE活性口袋中的Glu326、Thr329氨基酸残基相互作用形成3个氢键,结合能为-6.1 kcal/mol;Asp-Trp与ACE活性口袋中的Glu43、Leu103、Ala106、Tyr105氨基酸残基相互作用形成7个氢键,结合能为-7.7 kcal/mol;Ala-Trp与ACE活性口袋中Glu150、Gln265、Gln266、Asp360、Lys495氨基酸残基相互作用形成6个氢键,结合能为-8.76 kcal/mol,3种ACE抑制二肽均能与ACE进行结合,而且多肽C端为色氨酸时,该多肽与ACE的结合能更强。管骁等[29]通过分子对接发现ACE抑制活性与氨基酸的疏水性有关,C端是ACE抑制肽与ACE活性位点结合的关键,当C端含有疏水性氨基酸残基时更利于发挥ACE抑制活性。邵燕秋等[30]研究发现鳗鱼骨胶原蛋白ACE抑制肽酶解前、疏水性氨基酸(脯氨酸、丙氨酸、缬氨酸、甲硫氨酸、异亮氨酸、亮氨酸、苯丙氨酸)含量分别为29.91%,酶解后疏水性氨基酸含量增加到32.31%,酶解后ACE抑制肽抑制率得到了显著提升,这表明疏水性氨基酸是发挥ACE抑制活性的重要氨基酸。
4 结论
本文利用Z-scales、VHSE、SVHEHS三种描述符分别对123个二肽样本进行结构表征,采用PLS方法建立了3种ACE抑制二肽的QSAR模型。结果表明,3种氨基酸描述符均能对ACE抑制肽的结构与功能之间的关系进行表述,氨基酸残基的疏水性与立体特性对ACE抑制肽的活性有积极的影响。通过分子对接模拟发现,ACE抑制肽通过与ACE以氢键结合的方式发挥其生物学功能。表明通过偏最小二乘法对Z-scales、VHSE、SVHEHS三种描述符对ACE抑制二肽构建QSAR模型是可行的。研究结果对ACE抑制肽的检测、预测和筛选具有积极的意义。
[1] PAGIDIPATI N J, PETERSON E D.Can system solutions be scaled to control high blood pressure and lipids?[J].JAMA Cardiology, 2023, 8(1):21-22.
[2] HANIF K, BID H K, KONWAR R. Reinventing the ACE inhibitors: Some old and new implications of ACE inhibition[J]. Hypertension Research: Official Journal of the Japanese Society of Hypertension, 2010, 33(1):11-21.
[3] WANG Z L, ZHANG S S, JIN H W, et al.Angiotensin-I-converting enzyme inhibitory peptides:Chemical feature based pharmacophore generation[J].European Journal of Medicinal Chemistry, 2011, 46(8):3428-3433.
[4] 刘桂剑, 程宽, 朱文青, 等.高血压的药物治疗进展[J].中国临床药理学与治疗学, 2022, 27(4):446-449.LIU G J, CHENG K, ZHU W Q, et al.Progress of hypertension pharmacological treatment[J].Chinese Journal of Clinical Pharmacology and Therapeutics, 2022, 27(4):446-449.
[5] FERREIRA S H, BARTELY D C, GREENE L J.Isolation of bradykinin-potentiating peptides from Bothrops jararaca venom[J].Biochemistry, 1970, 9(13):2583-2593.
[6] ZHAO W Z, XUE S Y, YU Z P, et al.Novel ACE inhibitors derived from soybean proteins using in silico and in vitro studies[J].Journal of Food Biochemistry, 2019, 43(9):e12975.
[7] SUN X P, WANG M, XU C J, et al.Positive effect of a pea-clam two-peptide composite on hypertension and organ protection in spontaneously hypertensive rats[J].Nutrients, 2022, 14(19):4069.
[8] CHEN J W, WANG Y M, ZHONG Q X, et al.Purification and characterization of a novel angiotensin-I converting enzyme (ACE) inhibitory peptide derived from enzymatic hydrolysate of grass carp protein[J].Peptides, 2012, 33(1):52-58.
[9] GU R Z, LI C Y, LIU W Y, et al.Angiotensin I-converting enzyme inhibitory activity of low-molecular -weight peptides from Atlantic salmon (Salmo salar L.) skin[J].Food Research International, 2011, 44(5):1536-1540.[10] LI R J, ZHOU X L, SUN L P, et al.Identification, in silico screening, and molecular docking of novel ACE inhibitory peptides isolated from the edible symbiot Boletus griseus-Hypomyces chrysospermus[J].LWT, 2022, 16(9):114008.
[11] 孙强, 王瑞丹, 黄纪念, 等.亚临界水制备芝麻ACE抑制肽的分离纯化、构效、分子对接[J/OL].食品科学, 2023.https://kns.cnki.net/kcms/detail/11.2206.TS.20220819.0949.028.html.SUN Q, WANG R D, HUANG J N, et al.Separation, purification, structure-activity and molecular docking of sesame ACE inhibitory peptides prepared by subcritical water[J/OL].Food Science, 2023.https://kns.cnki.net/kcms/detail/11.2206.TS.20220819.0949.028.html.
[12] 唐鹏杰, 韩秋煜, 包斌, 等.海洋生物活性肽的制备、分离纯化及构效关系研究进展[J].食品工业科技, 2022, 43(24):488-495. TANG P J, HAN Q Y, BAO B, et al.Research progress on preparation, separation, purification and structure-function relationship of marine bioactive peptides[J].Science and Technology of Food Industry, 2022, 43(24):488-495.
[13] DAS M, HALDER A, CHATTERJEE R, et al.In vitro structure-activity relationship study of a novel octapeptide angiotensin-I converting enzyme (ACE) inhibitor from the freshwater mussel Lamellidens marginalis[J].International Journal of Peptide Research and Therapeutics, 2023, 29(2):18.
[14] 吴婷. 一组新的氨基酸描述子在多肽定量构效关系中的应用[D].成都:电子科技大学, 2021.WU T.Application of a group of new amino acid descriptors in peptide quantitative structure-activity relationship[D].Chengdu:University of Electronic Science and Technology of China, 2021.
[15] HELLBERG S, SJÖESTRÖEM M, SKAGERBERG B, et al.Peptide quantitative structure-activity relationships, a multivariate approach[J].Journal of Medicinal Chemistry, 1987, 30(7):1126-1135.
[16] 张艳萍, 成忠, 俞远志.基于3种建模法的食源性ACE抑制二肽QSAR研究[J].浙江科技学院学报, 2015, 27(3):174-182. ZHANG Y P, CHENG Z, YU Y Z.QSAR study of angiotensin I-converting enzyme inhibitory dipeptides based on three kinds of modeling methods[J].Journal of Zhejiang University of Science and Technology, 2015, 27(3):174-182.
[17] 梅虎, 周原, 孙立力, 等.氨基酸结构描述子矢量VHSE及其在肽QSAR中的应用[J].化学通报, 2005(7):534-540.MEI H, ZHOU Y, SUN L L, et al.A new set of descriptors of amino acids and its application in peptide QSAR[J].Chemistry, 2005(7):534-540.
[18] 刘静, 彭剑秋, 管骁.基于多元线性回归的血管紧张素转化酶抑制肽定量构效关系建模研究[J].分析科学学报, 2012, 28(1):16-22.LIU J, PENG J Q, GUAN X.Modeling the QSAR of ACE inhibitory peptides based on MLR[J].Journal of Analytical Science, 2012, 28(1):16-22.
[19] 翟明明, 高畅.基于偏最小二乘法的四氢异喹啉类化合物抗AchE活性定量构效关系研究[J].沈阳药科大学学报, 2021, 38(10):1093-1099.ZHAI M M, GAO C.Quantitative structure-activity relationship study on anti-AchE activity of tetrahydroisoquinoline compounds based on partial least squares[J].Journal of Shenyang Pharmaceutical University, 2021, 38(10):1093-1099.
[20] 王静, 马劲, 朱柏佳, 等.酶解法制备核桃谷蛋白-1 ACE抑制肽的工艺研究[J/OL].中国油脂, 2023.https://kns.cnki.net/kcms/detail//61.1099.TS.20230222.1356.013.html.WANG J, MA J, ZHU B J, et al.Preparation of ACE inhibitory peptide of walnut glutenin-1 by enzymatic hydrolysis[J/OL].China Oils and Fats, 2023.https://kns.cnki.net/kcms/detail//61.1099.TS.20230222.1356.013.html.
[21] ZHANG J, YANG L B, TIAN Z Q, et al.Large-scale screening of antifungal peptides based on quantitative structure-activity relationship[J].ACS Medicinal Chemistry Letters, 2021, 13(1):99-104.
[22] MEKENYAN O.Dynamic QSAR techniques:Applications in drug design and toxicology[J].Current Pharmaceutical Design, 2002, 8(17):1605-1621.
[23] JALALI-HERAVI M, KYANI A.Use of computer-assisted methods for the modeling of the retention time of a variety of volatile organic compounds:A PCA-MLR-ANN approach[J].Journal of Chemical Information and Computer Sciences, 2004, 44(4):1328-1335.
[24] 杨琦, 杨许花, 杨雪妍, 等.血管紧张素转换酶抑制肽定量构效关系研究进展[J].中国酿造, 2021, 40(6):23-27.YANG Q, YANG X H, YANG X Y, et al.Research progress on quantitative structure-activity relationship of angiotensin converting enzyme inhibitory peptide[J].China Brewing, 2021, 40(6):23-27.
[25] 苗青青. 系统分析和比较氨基酸描述子在生物活性肽的定量构效关系研究中的应用[D].成都:电子科技大学, 2020.MIAO Q Q. Application of systematic analysis and comparison of amino acid descriptors in quantitative structure-activity relationship research of bioactive peptides[D].Chengdu: University of Electronic Science and Technology of China, 2020.
[26] 仝建波, 刘淑玲, 来水利, 等.一种新三维氨基酸描述子SVTD及在肽QSAR的应用[J].分析科学学报, 2008,24(5):522-526.TONG J B, LIU S L, LAI S L, et al.A New descriptor of amino acids-SVTD and its applications in peptide QSAR[J].Journal of Analytical Science, 2008,24(5):522-526.
[27] WU Q Y, JIA J Q, YAN H, et al.A novel angiotensin-I converting enzyme (ACE) inhibitory peptide from gastrointestinal protease hydrolysate of silkworm pupa (Bombyx mori) protein:Biochemical characterization and molecular docking study[J].Peptides, 2015, 68:17-24.
[28] CHEUNG H S, WANG F L, ONDETTI M A, et al.Binding of peptide substrates and inhibitors of angiotensin-converting enzyme.Importance of the COOH-terminal dipeptide sequence[J].The Journal of Biological Chemistry, 1980, 255(2):401-407.
[29] 管骁, 刘静, 苏淅娜.ACE抑制三肽与ACE相互作用的分子机制[J].分析测试学报, 2014, 33(10):1116-1122.GUAN X, LIU J, SU X N.Study on flexible molecular docking of ACE inhibitory tripeptides and ACE[J].Journal of Instrumental Analysis, 2014, 33(10):1116-1122.
[30] 邵燕秋, 黄卉, 李来好, 等.鳗鱼骨胶原蛋白ACE抑制肽的制备及性质研究[J].南方水产科学, 2022, 18(6):137-145.SHAO Y Q, HUANG H, LI L H, et al.Preparation and properties of collagen ACE inhibitory peptides from bone of eel (Anguilla japonica)[J].South China Fisheries Science, 2022, 18(6):137-145.