自1923年首次从猪肉饼得到分离的德尔卑沙门氏菌作开始,1923—1944年间共发生了11起德尔卑沙门氏菌食物中毒事件;1968年在澳大利亚也发生一起德尔卑沙门氏菌导致的婴儿腹泻问题[1-3]。近年来,德尔卑沙门氏菌引起的沙门菌病不断频发,所感染的菌株通常具有较强毒力,严重甚至导致人的死亡,多起事件引起人们对德尔卑沙门氏菌的高度重视,然而在目前的形势下,抗生素的滥用导致沙门氏菌耐药性的不断提高[4]。沙门氏菌进入食物链的途径是复杂多样的,可能发生在食品的一个或多个环节中,食品行业因受到沙门氏菌污染而造成的经济损失正在不断扩大。
近年来,对德尔卑沙门氏菌的研究取得了一定的进展,但仍存在一些不足之处。首先,当前的研究现状中已发现了德尔卑沙门氏菌的多种亚型和血清型,并建立了相关的监测和预防措施[5]。此外,还揭示了德尔卑沙门氏菌的致病机制以及其与食物安全之间的关系。然而,在研究的深入程度和范围上,还存在一些不足之处,目前的研究主要集中在菌株鉴定、药物敏感性和遗传变异等方面。然而,对其产生毒力的分子机制、抗生素的耐药性以及免疫应答等方面的了解还相对有限[6]。此外,由于沙门氏菌在不同地区和环境中的发生和传播具有高度多样性,如何建立更准确和可靠的流行病学监测和预测模型仍是尚未解决的问题。因此,针对以上研究不足,对该细菌进行全基因组测序,建立德尔卑沙门氏菌从分子水平到生态系统层面的研究进行全面深入的分析,将有助于更好地了解并有效应对此病原体的威胁。
围绕猪源沙门氏菌的生物学和分子特性,从120份猪肉样本中进行细菌分离通过纯化出1株野生型沙门氏菌,通过16S rRNA 基因鉴定和血清型鉴定该沙门氏菌血清型,并进行药敏试验、毒力基因检测和全基因组测序分析。同时针对该菌株进行环境胁迫实验与生物被膜产量测定,结晶紫实验旨在检测沙门氏菌的生物被膜,高度复杂化和组织化的生物膜功能形式依赖于基质中各分子间的相互协作,它塑造生物被膜细菌之间的空间,提供稳定性,营造细菌的生存环境,生物被膜更有利于营养的获取[7]。该研究的目的是进一步探索德尔卑沙门氏菌的生理学特性与机制,为今后深入开展德尔卑沙门氏菌生存机制提供了重要的参考数据,研究结果不仅为沙门氏菌耐药机制的研究提供了理论依据,也为合理使用抗生素治疗沙门氏菌病提供了依据。
1 材料与方法
1.1 材料与仪器
1株野生型猪源沙门氏菌,由安徽凤阳县菜市场猪肉中分离筛选得到,经过16S rRNA基因序列测定为德尔卑沙门氏菌;德尔卑沙门氏菌CMCC50719,中国医学微生物菌种保藏中心;结晶紫染色液,北京百灵威科技有限公司;Ezup柱式细菌基因组DNA抽提试剂盒、药敏片,上海生工生物有限公司;Real Time PCR 试剂盒(SYBR® Green Premix Pro Taq HS qPCR Kit II),TaKaRa公司;胰酪大豆胨琼脂培养基(tryptone soya agar,TSA),胰酪大豆胨液体培养基(tryptone soya broth,TSB),广东环凯微生物科技有限公司。
752型紫外分光光度计,上海光谱仪器有限公司;TC-EA PLUS基因扩增仪,杭州博日科技有限公司;TGL-16微量核酸蛋白测定仪、DHG-9030 A高速冷冻台式离心机、BSC-1100ⅡA2-X生物安全柜,湖南湘仪实验仪器开发有限公司;LDZX-50 KBS立式高压蒸汽灭菌锅、ZHP-100F恒温培养振荡器,上海三发科学仪器有限公司;DHG-9030 A电热恒温鼓风干燥箱、BDF-86V50-80超低温冰箱,济南鑫贝西生物技术有限公司;ME403E电子天平,梅特勒-托利多仪器(上海)有限公司;Light Cycler® 96实时荧光定量PCR仪,Roche公司;Qubit Flex荧光定量仪,赛默飞世尔科技(中国)有限公司;E220evolution超声打断仪, 美国Covaris公司。
1.2 实验方案
1.2.1 沙门氏菌的分离鉴定及16S rRNA鉴定
将收集回来的猪肉样品放置于超净工作台中,无菌环境下将样品切成5~6 g块状放入新鲜的TSB培养基中,37 ℃培养18 h。参考NADI等[8]的沙门氏菌菌种鉴定方法,采用水煮法提取基因组,通过实行分离纯化,保存甘油之中放置在-80 ℃冰箱备用以及生化鉴定。多次纯化后的样本在TSB培养基中进行增菌,后接种于沙门氏菌显色培养基平板中,利用革兰氏染色法对品红色泽的单菌落进行检验,并利用沙门氏菌生化检测试剂盒进行生化鉴定,对于样本提取基因组DNA进行PCR法鉴定。
对经过生化鉴定后的样本菌株进行16S rRNA(引物序列见表1)同源性比较分析[9]。经过过夜的TSB培养,利用细菌基因组DNA 提取试剂盒提取基因组DNA,于-20 ℃中保存备用。扩增后的 DNA 经检验送至上海美吉生物医药科技有限公司进行测序。将 16S rRNA 基因测序结果通过BLAST在GenBank中进行同源比对分析。
表1 引物序列
Table 1 Primer sequence

基因引物序列bpTm/℃araCF:CCTGGCACCTCGCGCTATACR:CCACACACGGATCGTGGCG82660dnaNF:ATGAAATTTACCGTTGAACGTGAR:AATTTCTCATTCGAGAGGATTGC83358hemPF:GAAGCGTTAGTGAGCCGTCTGCGR:ATCAGCGACCTTAATATCTTGCCA66660hisDF:GAAACGTTCCATTCCGCGCAGACR:CTGAACGGTCATCCGTTTCTG89460purEF:ATGTCTTCCCGCAATAATCCR:TCATAGCGTCCCCCGCGGATC51058sucAF:AGCACCGAAGAGAAACGCTGR:GGTTGTTGATAACGATACGTAC64360thrAF:GTCACGGTGATCGATCCGGTR:CACGATATTGATATTAGCCCG8526016S rRNAF:CTCTTGCCATCAGATGTGCCR:TTCTTCATACACGCGGCATG20160stnF:GGTCGGTCCCACTTTCTTTTGR:GGTCAGTCAGGATGCCCAA18560invAF:AAAGAAGGGTCGTCGTTAGGAR:GTAGACGCTCCGCAAGTT13060sseLF:TAATCTGGAAGGGGCATGTAGR:GACCTGGGGCATAGAGT11760hliAF:CGATGCTCTTTATCTTCCGACAR:CGTTCTGGTCATCCTGCTC15360mgtCF:GCAGCGTATAAATCAACTCCCR:CGAACCTAACCCTTGTAAACAG15160fliAF:CCTGTTGCGGAGTATCGTCAR:CGTTAACACCAGTTGCTCGC13760pipAF:CAGAGTGTGTGGTGGAGTACCR:GAAGTGTTCCTGAGCCACGG24860
1.2.2 血清型与多位点序列分型(multilocus sequence typing,MLST)鉴定
参照国标GB 4789.4—2016《食品安全国家标准 食品微生物学检验 沙门氏菌检验》的方法诊断血清,分别确定菌株的O抗原和H抗原类型,以此确定沙门氏菌的血清型[10]。提取样本细菌的DNA,利用生工Ezup柱式细菌基因组DNA抽提试剂盒进行提取。对7个管家基因arcC、dnaN、hemP、hisD、purE、sucA、thrA进行PCR扩增。PCR扩增体系为25 μL:12.5 μL 2×PCR Mix、1 μL基因上下游引物(10 μmol/L)、1 μL DNA模板、9.5 μL ddH2O;PCR反应条件为:① 95 ℃预变性5 min、② 95 ℃ 变性60 s、③ 60 ℃退火30 s、④ 72 ℃ 延伸45 s、②~④ 步重复35次、⑤ 72 ℃ 延伸10 min。引物序列见表3,将扩增产物送至上海生工生物有限公司进行测序,测序结果采用DNA Star软件根据Pub MLST的相关要求进行修正,与MLST数据库中序列进行比对分析,获取7个看家基因位点的等位基因数值和MLST。
1.2.3 药敏实验、毒力基因验证以及生物被膜结晶紫染色实验
1.2.3.1 药敏性实验
采用抑菌圈法[11],各取1 mL经TSB活化培养后的样本置于灭菌后的离心管中,120 r/min离心3 min,弃上清液。沉淀用1 mL生理盐水重悬,振荡混匀后梯度稀释,取适宜稀释度细胞悬液涂布TSA平板。在平板中的三角位置等距放置3片抗生素药敏片,37 ℃培养18 h后,使用游标卡尺精确测量抑敏圈直径,参照CHEN等[12]的标准制定表2,来判定菌株敏感性,结果分为敏感、中度敏感及耐药。
表2 药物敏感实验判定标准
Table 2 Criteria for determination of drug sensitivity tests

类别名称(英文名)含量/μg判定标准(抑菌圈直径)/mmRIS青霉素类苯唑西林(OXA)20≤1314-16≥17氨苄西林(AMP)20≤1314~16≥17糖肽类多黏菌素B(PB)20≤88~11 ≥12头孢类头孢噻吩(CEP)20≤14 15~17 ≥18氨基糖苷类卡那霉素(KAN)20≤1314~17≥18四环素类米诺环素(MNO)20≤1415~18≥19喹诺酮类环丙沙星(CIP)20≤1515~21≥21磺胺类复方新诺明(SXT)20≤1011~15≥16
注:试验重复3次,取平均值;R-耐药;I-中介;S-敏感(下同)。
1.2.3.2 毒力基因验证
提取样本细菌的DNA,本实验选取了7种毒力基因,引物序列见表1。利用qPCR反应体系,25 μL反应体系包括:DNA模板 2 μL,AceQ® qPCR SYBR® Green Master Mix (High ROX Premixed) 12.5 μL,基因特异性上下游引物(10 μmol/L) 各1 μL,RNase-free ddH2O 8.5 μL。Ct值<35,结果判定为阳性(+),未出现Ct值或Ct值>35判断为阴性(-)。
1.2.3.3 生物被膜定量分析
将样本接种于含有9 mL TSB的灭菌试管中,37 ℃分别培养24、48、72、96、120 h后,弃除菌液,灭菌蒸馏水洗涤3次后用0.1%结晶紫溶液染色20 min,再取相应量体积分数95%乙醇脱色10 min,测定570 nm处的吸光度。根据生物性重复做3次平行实验,同时设置空白培养液作为对照,生物被膜合成性以德尔卑沙门氏菌标准菌株作为对照。
1.2.4 环境胁迫抗性实验
将沙门氏菌经TSB培养基37 ℃培养18 h,取1 mL培养液于灭菌后的离心管中,120 r/min离心3 min,弃上清液。参考刘佳玫等[13]的方法按以下方法进行沙门氏菌抗胁迫能力实验。
温度处理:用1 mL生理盐水重悬菌体,高温在40 ℃、50 ℃、60 ℃温度中处理30 min;低温则4 ℃冰箱冷藏处理。
酸胁迫:将无菌生理盐水利用柠檬酸将pH值调整为1.5、2.0和2.5,使用移液枪各取1 mL加入离心管中重悬菌体(处理30 min)。
碱胁迫:将无菌生理盐水利用NaOH将pH值调整为11、12和13,使用移液枪各取1 mL加入离心管中重悬菌体(处理15 min)。
NaClO溶液处理:将菌体分别重悬于无菌水与NaClO制成的25、50、100 mg/L NaClO溶液中处理30 min,用过量的Na2S2O3终止反应。
乙醇处理:将菌体分别重悬于无菌水与无水乙醇配制成的体积分数65%、70%乙醇溶液中处理5 min,以德尔卑沙门氏菌标准菌株(CMCC50719)作为对照。以上菌体在胁迫环境下处理一定时间后,120 r/min离心3 min,弃上清液。沉淀用1 mL生理盐水重悬,振荡混匀后梯度稀释,取适宜稀释度细胞悬液涂布TSA平板计数。根据胁迫前后的菌体数,按公式(1)计算存活率。
存活率![]()
(1)
1.2.5 全基因组测序及生物信息学分析
通过TSA平板划线,将样本于TSB培养基中以37 ℃ 180 r/min培养16 h,菌液于2 mL无菌离心管中并加入体积分数30%甘油中进行保存,送至深圳华大基因生物公司进行测序。采用BGI自主平台对样品进行测序,利用Qubit荧光定量仪检测DNA样品的浓度,1%的琼脂糖凝胶电泳检测DNA样品的完整性,使用超声打断仪超声波打断DNA样品,通过调整打断参数,获得符合长度要求的短DNA片段[14]。对基因组DNA浓度、纯度以及完整性进行相关检验,扩增产物用试剂Agencourt AMPure XP-Medium进行片段筛选,结果达到要求后再进行建库,库检合格后,进行基因组组分、功能注释、比较基因组学等分析[15]。将基因与已知的数据Cluster of Orthologous Groups of Proteins(COG)、Kyoto Encyclopedia of Genes and Genomes(KEGG)、Gene Ontology (GO)、Virulence Factors of Pathogenic Bacteria (VFDB)、Antibiotic Resistance Genes Database (CARD)等比对现有蛋白做功能预测和注释[16-20]。
2 结果与分析
2.1 沙门氏菌鉴定结果
利用沙门氏菌显色培养基将菌株过夜培养后出现显色,初步判定为沙门氏菌。提取分离菌株的DNA作为模板,16S rRNA为引物,利用PCR进行扩增。将PCR产物送至生工进行测序,将测序结果在BLAST中进行比对,与沙门氏菌的同源性在99%以上,初步鉴定分离出的菌株为沙门氏菌。通过数据库分析,菌株抗原式为1.4.12;f,g,;-。通过血清型鉴定和MLST分子分型,得出该分离株为德尔卑沙门氏菌,ST型为ST1498,并将其命名为G-1B。
2.2 生物学结果
2.2.1 药敏性实验结果
针对分离菌株G-1B对其进行8种抗生素的药敏性实验,以标准德尔卑沙门氏菌CMCC50719作为对照,观察与标准菌株的不同进行判断。表3结果表明,G-1B菌株对于喹诺酮类的CIP、氨基糖苷类的KAN和磺胺类的SXT药物较为敏感。而对于糖肽类PB、四环素类MNO则是中度敏感。达到耐药水准的则是青霉素类的OXA、AMP和头孢菌素类CEP。而作为对照的标准菌株的耐药性,CIP、KAN、SXT是达到了S, PB、CEP、MNO则是R。与对照的标准菌株实验结果来看,G-1B已被证明产生了异质性并且具有多重耐药性。
表3 药敏性分析
Table 3 Drug susceptibility test
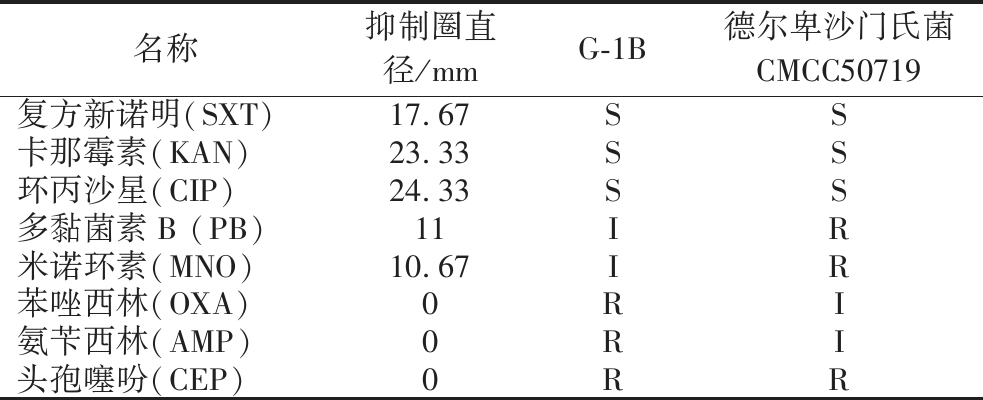
名称抑制圈直径/mmG-1B德尔卑沙门氏菌CMCC50719复方新诺明(SXT)17.67SS卡那霉素(KAN)23.33SS环丙沙星(CIP)24.33SS多黏菌素B (PB)11IR米诺环素(MNO)10.67IR苯唑西林(OXA)0RI氨苄西林(AMP)0RI头孢噻吩(CEP)0RR
2.2.2 毒力基因验证结果
本实验选取的7种毒力基因验证:SseL、hilA、mgtC、FilA、Stn、invA以及pipA经qPCR的结果见表4。以16S rRNA作为内参基因,所有目的基因均收集到荧光信号,表明G-1B内这7种毒力基因均呈表达,存在强致病风险需要进一步进行研究。
表4 qPCR毒力基因验证
Table 4 Toxicity gene verification by qPCR

基因Ct值2-ΔΔCtSsel10.54±0.1621.83hilA12.52±0.122-0.15mgtC12.23±0.0420.14FilA15.08±0.212-2.71Stn10.65±0.2221.72invA13.11±0.082-0.74pipA11.38±0.1720.99
2.2.3 生物被膜定量分析
24 h前G-1B和标准菌株的结膜能力基本相同,但是从第24 h开始后,标准菌株的产量开始高于G-1B,到达72 h时,2种菌株的结膜能力趋于稳定状态,而G-1B明显低于标准菌株,此时吸光度要低于标准菌株接近1.5倍(图1)。生物被膜被认为是细菌适应恶劣环境而形成的一种保护机制,而在本次实验中G-1B的结膜能力低于标准菌株,该现象可能会对沙门氏菌的生存和繁衍造成负面影响,可能由多种因素引起,包括环境压力、抗生素的长期使用和基因变异等。因此需要对G-1B进行后续实验分析,进一步研究其生物被膜能力下降的原因和机制。
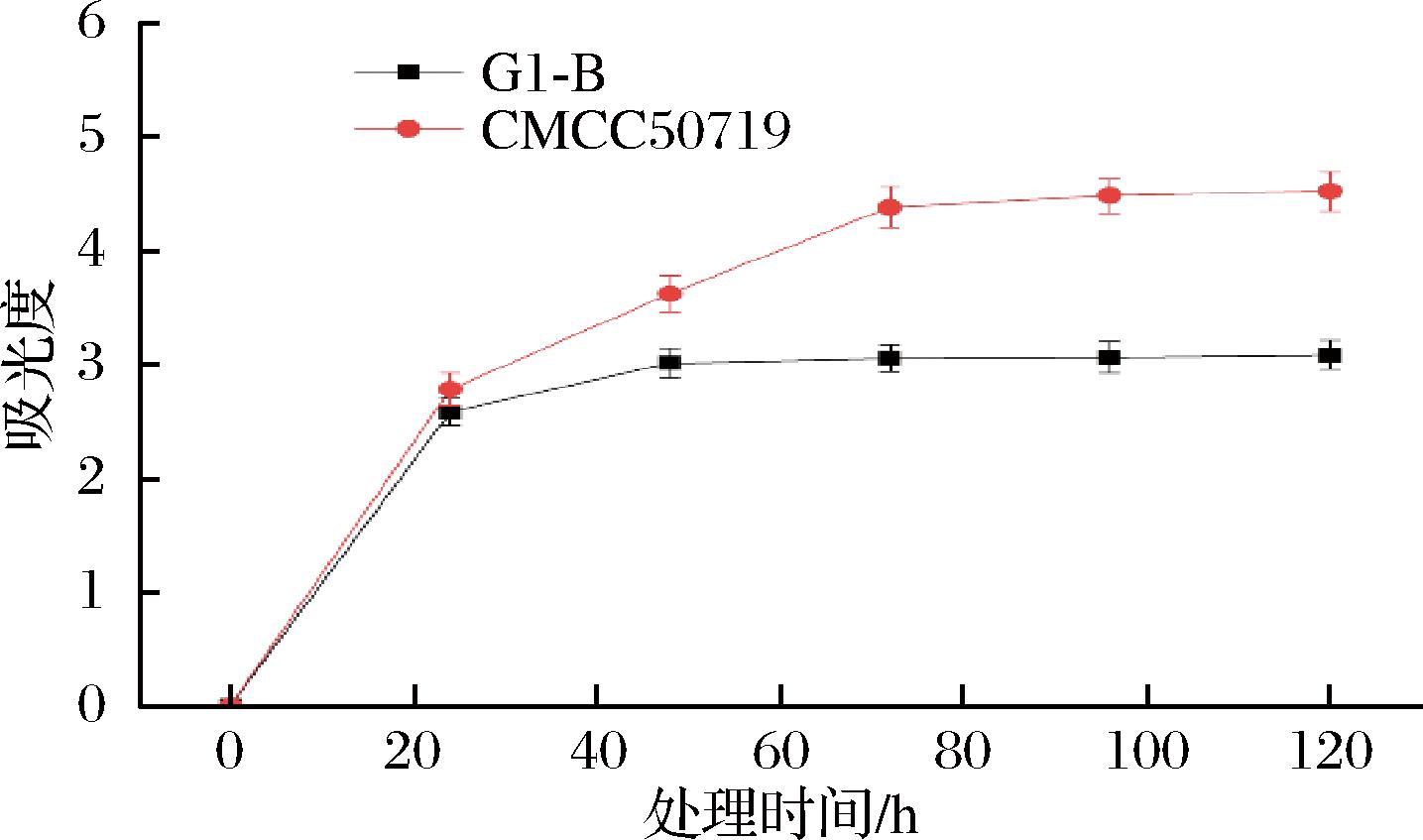
图1 结晶紫染色结果
Fig.1 Crystalline violet staining results
2.2.4 环境胁迫实验结果
为了研究在自然环境下的生存状态与标准型的德尔卑沙门氏菌的生存能力的对比,对凤阳县本地市场发现的这株野生型德尔卑沙门氏菌G-1B进行环境胁迫的实验。
高温加热处理是对肉类制品灭菌最常见的手段,从高温胁迫中发现了G-1B对于高温耐受的能力要高于标准菌株,尤其是当温度从40 ℃提升至60 ℃时,2株菌的存活率都降低了30%~40%,而G-1B对于这种高温耐受性较为明显(图2-a)。低温保鲜是延长及储存食品常用的一种保藏技术,4 ℃低温存活率的实验中,将G-1B和标准菌株进行19 d的低温试验。如图2-b所示,G-1B并没有高于标准菌株的耐低温能力,随着时间的增加,G-1B的生存率逐渐下降。温度对于G-1B的影响在高温的甚为明显,低温的环境下的存活率不如标准菌株,由此可见对于长期处在低温环境下,可以降低沙门氏菌的活性。酸胁迫时,酸性环境可以明显抑制沙门氏菌,pH=2.0时,G-1B的存活率已经到了10%以下,pH=1.5时,已经完全失活(图2-c)。相比较而言,对比标准菌株,G-1B的耐酸性有了提升,而这可能与区域以及存活环境有关。但是到了碱性环境胁迫实验中(图2-d),G-1B的生存能力明显低于标准菌株,pH值到达13时,标准菌株依然有21.86%的存活率,以上结果表明,碱性环境是凤阳县当地肉制品加工厂更有效的有效杀菌措施,能够对肉类加工器械等起到更为明显的灭菌效果。在消毒剂的胁迫实验中(图2-e),当NaClO质量浓度达到50 mg/L时,标准菌株对于此种消毒剂就没有了任何生存迹象,当提升至100 mg/L时,G-1B有25.95%的存活率,这表明凤阳地区对NaClO消毒剂已经产生了一定的抗逆性。使用乙醇溶液时,则完全相反,体积分数为70%时,G-1B明显低于标准菌株的存活率。
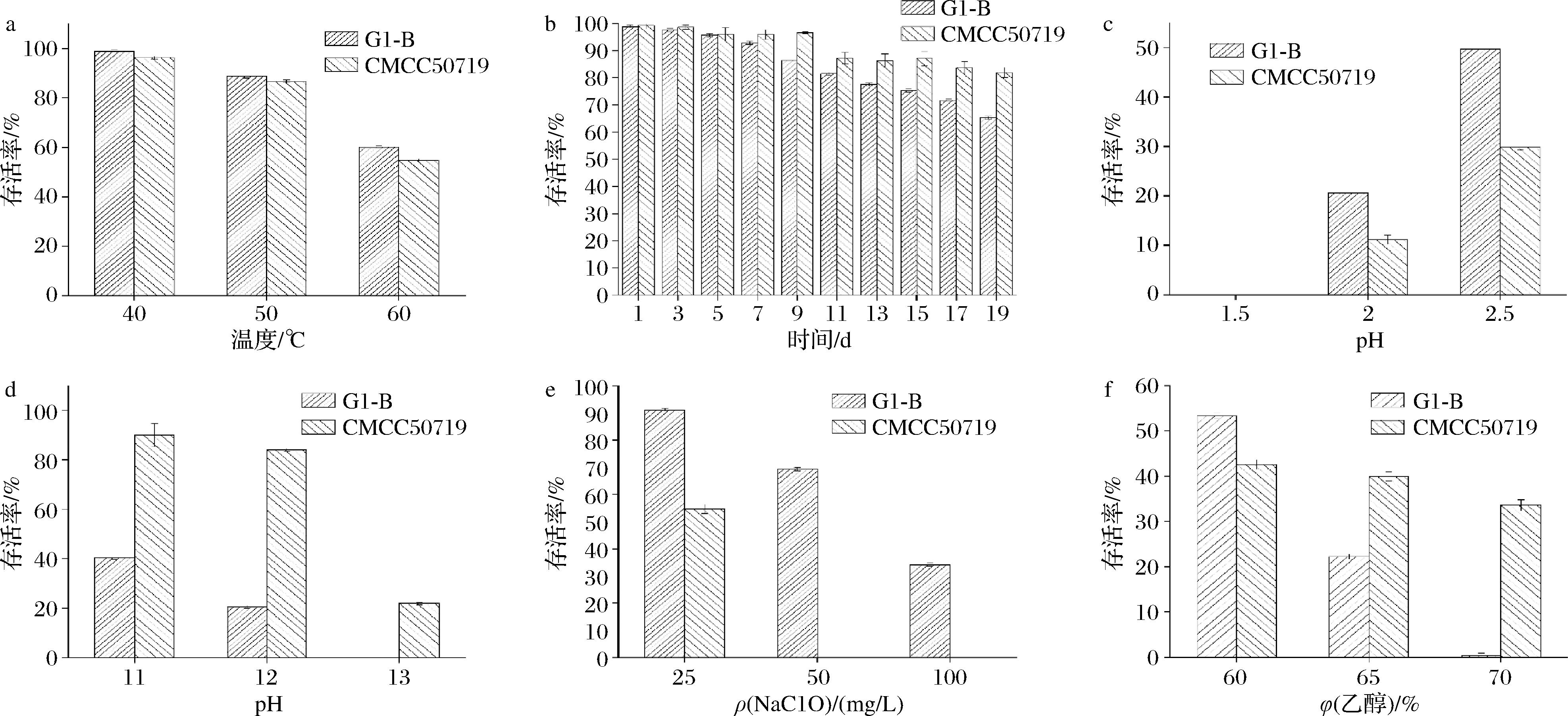
a-高温胁迫;b-低温4 ℃胁迫;c-柠檬酸胁迫;d-NaOH胁迫;e-消毒剂NaClO胁迫;f-乙醇胁迫存活率
图2 环境胁迫实验结果
Fig.2 Results of environmental stress experiment
2.3 全基因组测序
2.3.1 基本信息
对G-1B进行全基因组测序,最终得到基因组大小为4 805 494,预测出4 660个基因,总长度为4 180 794 bp,非编码RNA共预测出了84个tRNA,22个rRNA和65个新sRNA,GC含量为53.26%。基因平均长度为897.17。G-1B的全基因组图谱组分如图3。
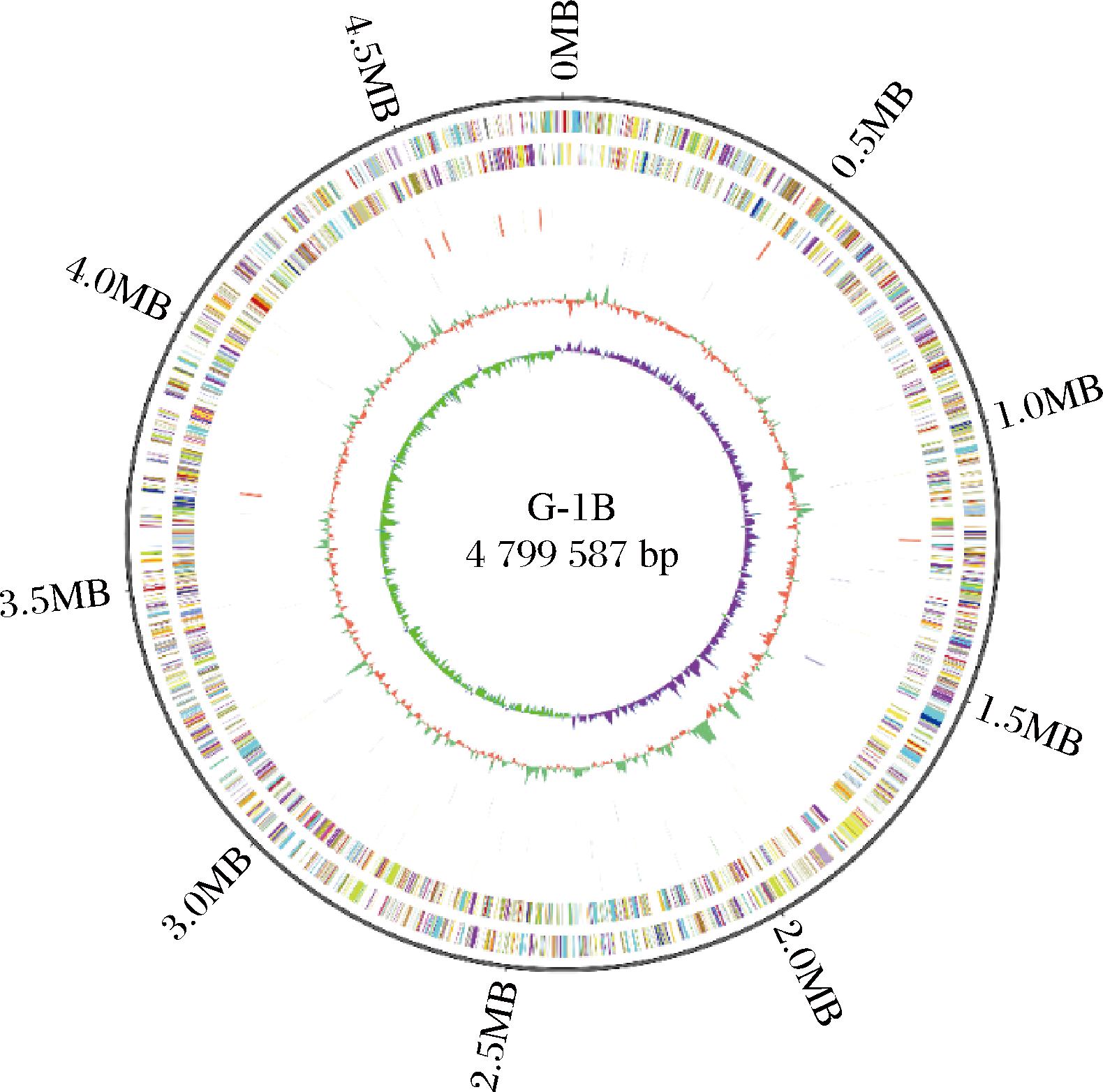
图3 G-1B基因组图谱
Fig.3 G-1B genome map of strains
G-1B基因组圈图的最外面一圈表示基因组长度信息;第二圈和第三圈分别表示正链、负链上的编码序列(coding sequence,CDS),不同颜色的 CDS 表示不同的 COG 功能类别;第四圈为 rRNA 和 tRNA;第五圈为 GC 含量,向外的绿色部分表示该区域 GC 含量高于全基组平均 GC 含量,向内的橘色部分表示该区域 GC 含量低于全基因组平均 GC 含量,峰值越高表示与平均 GC 含量差值越大;最内一圈为GC skew值。
2.3.2 基因注释
用BLAST进行基因和功能注释。对预测到的基因分别在KEGG、COG、NR、GO等11个数据库进行功能注释,结果如表5所示。
表5 基因集注释结果统计
Table 5 Gene set annotation results
菌株VFBDARDBCAZYIPRSwiss-ProtCOGCARDGOKEGGNRT3SS总计G-1B559371314 1093 5483 689423 0483 2334 6446304 647
对G-1B基因组中具有生物学功能的蛋白编码基因进行 COG 注释,结果发现共有3 689个蛋白编码基因被注释到。如图4所示,基因功能注释结果共分为细胞4大类,23个小类。其中Carbohydrate transport and metabolism(碳水化合物转运及代谢)的注释结果最为丰富,共413个;其次为Amino acid transport and metabolism genes(氨基酸转运及代谢基因),共379个基因,Transcription(转录355个基因)、
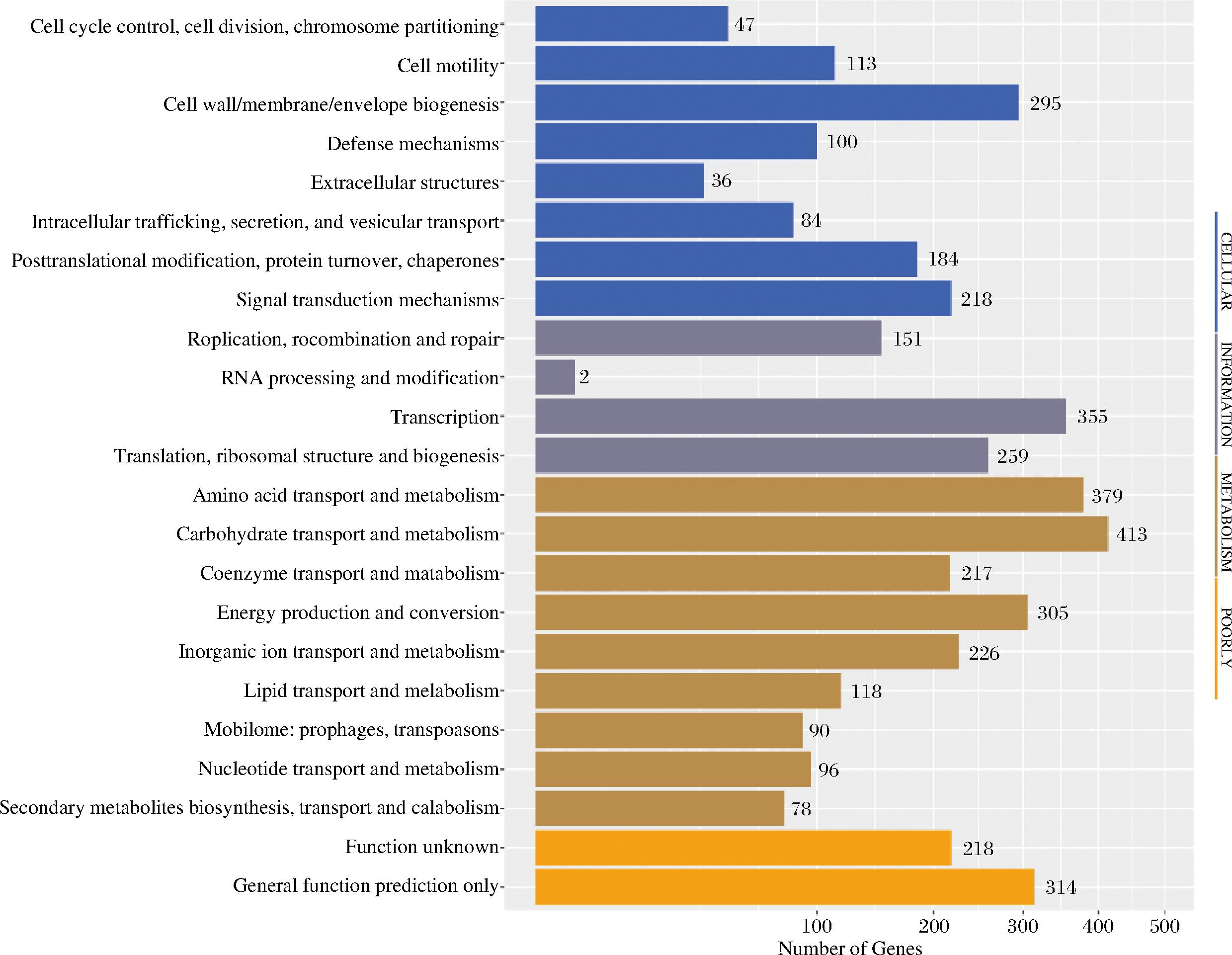
图4 COG 功能注释分布图
Fig.4 COG function annotation distribution plot
Energy production and conversion(能源生产和转换305个基因)、Cell wall/membrane/envelope biogenesis(细胞壁/膜/包膜的生物发生295个基因)的基因也得到较多的注释,此外还有218个功能未知的基因,有待今后进一步研究。
2.3.3 毒力基因及耐药基因
经VFDB数据库的分析发现了的毒力基因如表6所示,这些基因所编码的产物主要参与功能有黏附、铁摄取、细菌分泌系统、细胞膜形成等。本次测序在VFDB数据中共出现了80个毒力岛1、2相关的编码基因,见表7。
表6 毒力基因分布
Table 6 Distribution of toxic genes
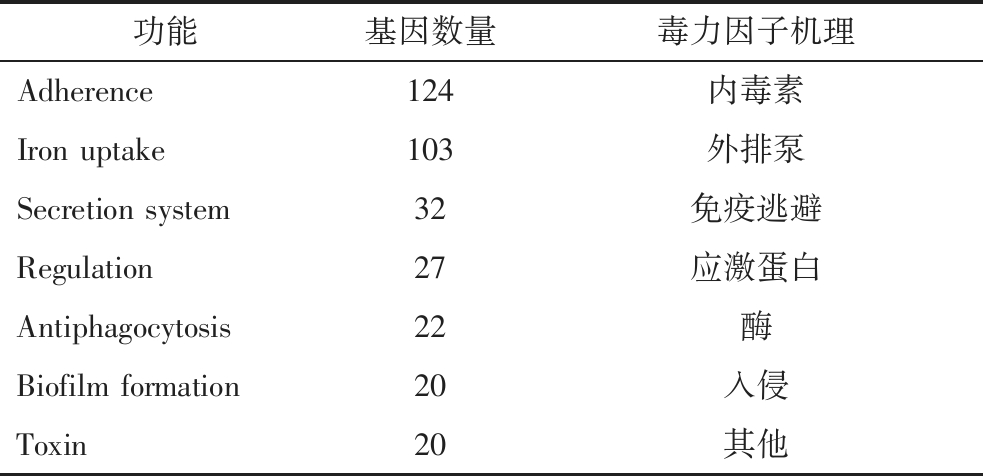
功能基因数量毒力因子机理Adherence124内毒素Iron uptake103外排泵Secretion system32免疫逃避Regulation27应激蛋白Antiphagocytosis22酶Biofilm formation20入侵Toxin20其他
表7 毒力岛1、2相关的编码基因
Table 7 Toxicity island 1,2 related coding genes

毒力岛基因功能SPI-1InvH细胞黏附/入侵蛋白InvF入侵蛋白InvG假定蛋白InvE细胞侵袭蛋白InvAⅢ分泌系统主要输出蛋白InvBⅢ分泌系统蛋白InvCⅢ分泌系统ATP酶InvI分泌蛋白(与毒力有关)InvJ抗原递呈蛋白spaOⅢ分泌系统C环蛋白spaPⅢ型分泌系统次要出口蛋白spaQⅢ型分泌系统次要出口蛋白spaRⅢ型分泌系统次要出口蛋白spaSⅢ分泌系统出口装置开关蛋白sicASipC 和 SipB 的伴侣蛋白sipB致病性岛1效应蛋白sipC/sspCⅢ型分泌系统亲水转运体,孔蛋白sipD细胞侵袭蛋白IacP酰基载体蛋白sicP分泌系统伴侣蛋白sptP酪氨酸磷酸酶和 GTP 酶激活蛋白iagB入侵蛋白前体hilA入侵蛋白转录激活因子hilD入侵蛋白调节蛋白prgH/EprHⅢ分泌装置蛋白
续表7

毒力岛基因功能prgIⅢ分泌系统针丝蛋白prgJⅢ型分泌系统内杆蛋白prgKⅢ分泌系统 MS 内环蛋白orgA/MxiKⅢ分泌装置蛋白orgB氧调节侵袭蛋白orgC假定蛋白hilC假定AraC家族转录调节器sprB转录调节因子slrPⅢ型分泌系统效应子TTSS-1sopDSop效应蛋白(转位效应因子)hopAN1Ⅲ型效应器sipA细胞侵袭蛋白avrA分泌效应蛋白ipaH假定蛋白sopA分泌效应蛋白sopE2入侵相关蛋白sopB/sigD肌醇磷酸磷酸酶sopE鸟嘌呤核苷酸交换因子SPI-2SsrB双组分调节因子SsrA分泌系统调节器SsaC外膜蛋白SsaDⅢ型分泌蛋白SsaE分泌系统蛋白SseB分泌系统效应子SscAⅢ型分泌低钙反应伴侣SseC易位机制组件SseD易位机制组件SseE分泌效应蛋白SseG分泌系统装置SsaG分泌系统装置SsaH致病岛蛋白SsaIⅢ型分泌系统SsaJ假定蛋白SsaK致病岛蛋白SsaL分泌系统蛋白SsaM致病岛蛋白SsaV分泌系统装置SsaNⅢ型分泌系统SsaOⅢ型分泌系统SsaP分泌系统装置蛋白SsaQⅢ型分泌蛋白SsaRⅢ型分泌系统蛋白SsaSⅢ型分泌系统蛋白SsaTⅢ型分泌蛋白SsaUⅢ型分泌蛋白TTSS-2PipB2假定蛋白SseL去泛素化酶SifA转位效应蛋白SseF易位效应蛋白SifB易位效应蛋白SseT分泌效应蛋白SspH2富含亮氨酸的重复蛋白PipB易位效应蛋白SopD2Ⅲ型分泌蛋白
经过CARD数据库的分析,G-1B的基因中存在多种类型的耐药基因,具体情况见表8。这些基因可能会导致它在不同的药物中发挥相应的抗性作用。
表8 耐药基因情况
Table 8 Drug resistance genes
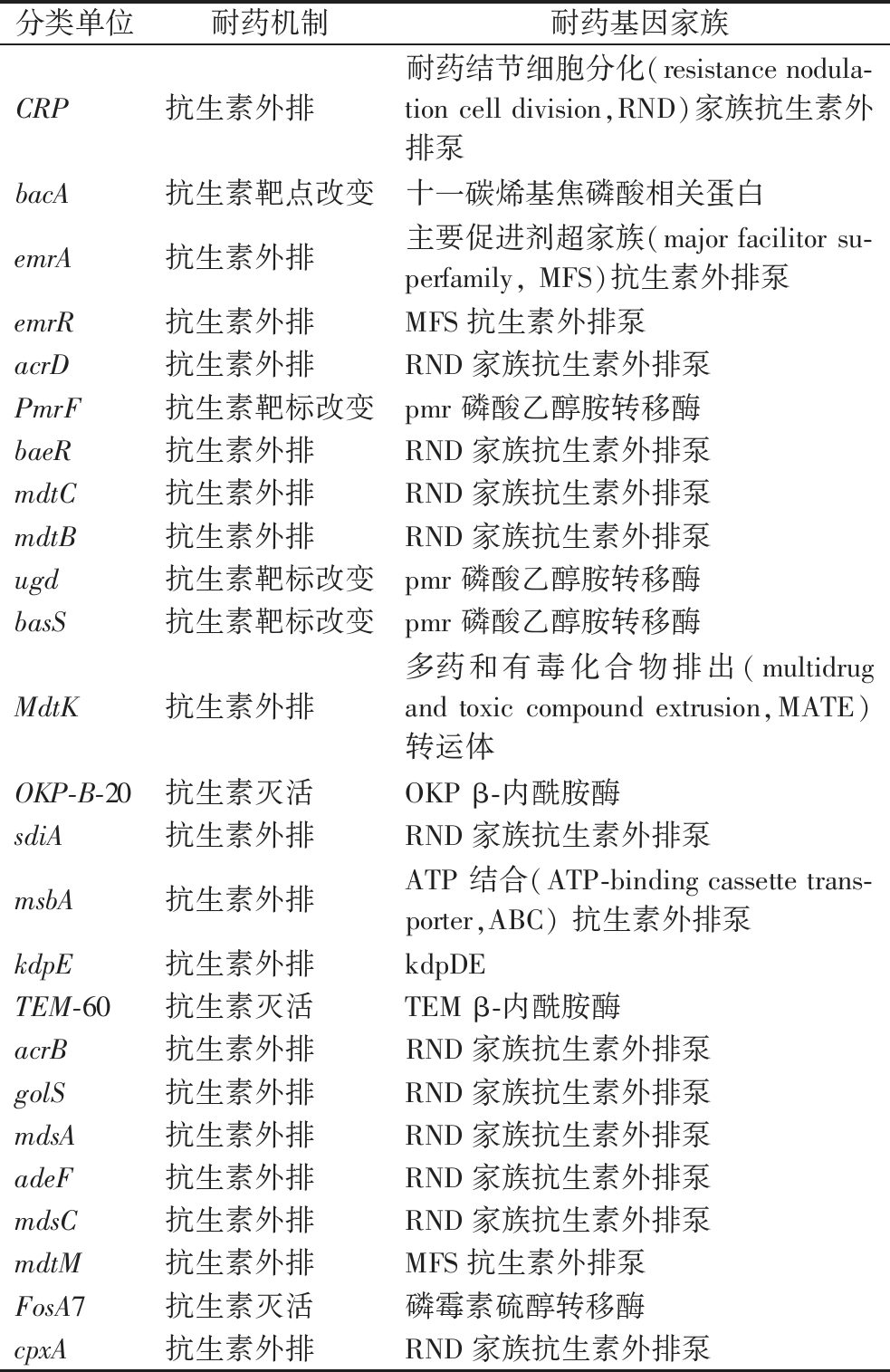
分类单位耐药机制耐药基因家族CRP抗生素外排耐药结节细胞分化(resistance nodula-tion cell division,RND)家族抗生素外排泵bacA抗生素靶点改变十一碳烯基焦磷酸相关蛋白emrA抗生素外排主要促进剂超家族(major facilitor su-perfamily, MFS)抗生素外排泵emrR抗生素外排MFS抗生素外排泵acrD抗生素外排RND家族抗生素外排泵PmrF抗生素靶标改变pmr 磷酸乙醇胺转移酶baeR抗生素外排RND家族抗生素外排泵mdtC抗生素外排RND家族抗生素外排泵mdtB抗生素外排RND家族抗生素外排泵ugd抗生素靶标改变pmr 磷酸乙醇胺转移酶basS抗生素靶标改变pmr 磷酸乙醇胺转移酶MdtK抗生素外排多药和有毒化合物排出(multidrug and toxic compound extrusion,MATE)转运体OKP-B-20抗生素灭活OKP β-内酰胺酶sdiA抗生素外排RND家族抗生素外排泵msbA抗生素外排ATP 结合(ATP-binding cassette trans-porter,ABC) 抗生素外排泵kdpE抗生素外排kdpDETEM-60抗生素灭活TEM β-内酰胺酶acrB抗生素外排RND家族抗生素外排泵golS抗生素外排RND家族抗生素外排泵mdsA抗生素外排RND家族抗生素外排泵adeF抗生素外排RND家族抗生素外排泵mdsC抗生素外排RND家族抗生素外排泵mdtM抗生素外排MFS抗生素外排泵FosA7抗生素灭活磷霉素硫醇转移酶cpxA抗生素外排RND家族抗生素外排泵
3 结论与讨论
通过环境胁迫研究发现,G-1B相较于标准菌株,对于强酸、强碱、乙醇有着较为明显的敏感性,可以作为有效的措施进行杀菌,高温、低温、NaClO的杀菌效果并不充分,存在着灭杀不彻底的风险,尤其在高浓度NaClO、NaOH的极端胁迫下,仍具有20%以上的存活率,推测出本株野生型德尔卑沙门氏菌G-1B产生了地区、机制、耐药等方面的异质性。
G-1B在本次全基因组测序中共发现了559个毒力基因以及37个耐药性基因。在这些基因当中,毒力基因分布在32种不同的蛋白质分泌系统中,TypeVI型的基因数量居首,达到了21个,而VI型蛋白质分泌系统则具备更强大的功效,它不仅能够将溶菌效应物输送到靶细胞,还具备抗菌作用,从而损害宿主细胞的结构[21],例如mgtC为沙门氏菌的存活提供了强有力的支持,这也使得它们的致病能力变得极为显著。AcrAB-TolC的外排系统可以检测到许多不同类型的抗生素,包括青霉素、四环素、磺胺类、氨基糖苷类和消毒剂,它们可以表达为多种不同的耐药性,这些耐药性可以由CRP、bacA、emrA、acrD、PmrF、baeR、mdtC、ugd和basS基因所表达。由此证明G-1B属于多重耐药菌株,于β-内酰胺类药物、青霉素类药物、氨基糖苷类药物acrD、aadA四环素类药物、喹诺酮类药物以及磺胺类药物具有耐药性,而且可能会导致它们对磷霉素、β-内酰胺类、氨基糖苷类和喹诺酮类药物的敏感。而通过本次药敏性实验分析,由于CIP与SXT两种抗生素的机理为抑制核酸合成,KAN是抑制蛋白质合成,猜测可能由于地区与种属的问题而导致了耐药异质性[22]。G-1B具有了与标准菌株较大差异的结果,猜测与地区差异以及屠宰环境、运输过程等有关。这与本次实验中的药敏性实验结果大致相同。有研究发现,其82株沙门氏菌中对头孢类药物敏感[23],而在本次实验中G-1B对于头孢类产生了耐药作用;2017年我国将多黏菌素禁止作为饲料添加剂,与贺恒旭等[24]研究禽源沙门氏菌耐药研究数据相比较而言,本研究中对多黏菌素耐药仍然达到了中等耐药水平,需要有关部门采取相关措施,加强抗生素管理;姚玉芳等[25]研究发现黄山市的43株沙门氏菌多数对AMP和CEP达到了耐药性,与本次药敏性实验结果近乎一致,这表明在安徽凤阳地区的猪源德尔卑沙门氏菌可能出现了多重耐药性且分布广泛的现象,应进行针对性防控。
本研究在安徽凤阳地区分离得到一株猪源德尔卑沙门氏菌G-1B,对其进行全基因组测序分析,该菌株为多重耐药菌且携带大量毒力基因,基因组中存在较多毒力岛和插入序列,在全基因组测序中,为了判断G-1B的同源性,与邦戈沙门氏菌(Salmonella bongori)和肠炎沙门氏菌(Salmonella enterica)做了同源对比,G-1B与二者的同源性达到了90%以上。本次发现的野生型德尔卑沙门氏菌产生了多重耐药的异质性且携带大量毒力基因,需要深入研究。本研究为测猪源沙门氏菌流行,毒力、耐药性发展及后续深入研究提供了参考。
[1] PECKHAM C F, SAVAGE W G.An outbreak of pork pie poisoning at Derby[J].The Journal of Hygiene, 1923, 22(1):69-76.
[2] EFS A, EUROPEAN CENTRE FOR DISEASE PREVENTION AND CONTROL.The European union summary report on trends and sources of zoonoses, zoonotic agents and food-borne outbreaks in 2011[J].EFSA Journal, 2013, 11(4):3129.
[3] LI Q, YIN J, LI Z, et al.Serotype distribution, antimicrobial susceptibility, antimicrobial resistance genes and virulence genes of Salmonella isolated from a pig slaughterhouse in Yangzhou, China[J].AMB Express, 2019, 9(1):210.
[4] 白明焕, 万姿廷, 罗睿心, 等.2020—2021年四川省自贡市食品中沙门氏菌的污染及耐药性研究[J].河南预防医学杂志, 2022, 33(10):799-803.
BAI M H, WAN Z T, LUO R X, et al.Contamination status and antimicrobial resistance of Salmonella in foods in Zigong city, Sichuan Province from 2020 to 2021[J].Henan Journal of Preventive Medicine, 2022, 33(10):799-803.
[5] STAES I, PASSARIS I, CAMBRÉ A, et al.Population heterogeneity tactics as driving force in Salmonella virulence and survival[J].Food Research International, 2019, 125:108560.
[6] KENNEY L J.The role of acid stress in Salmonella pathogenesis[J].Current Opinion in Microbiology, 2019, 47:45-51.
[7] 黄建江, 朱小兰.苯胺结晶紫染色法在细菌染色中的应用[J].诊断病理学杂志, 2021, 28(5):409; 416.
HUANG J J, ZHU X L.Application of aniline crystal violet staining in bacterial dyeing[J].Chinese Journal of Diagnostic Pathology, 2021, 28(5):409; 416.
[8] NADI Z R, SALEHI T Z, TAMAI I A, et al.Evaluation of antibiotic resistance and prevalence of common Salmonella enterica serovars isolated from foodborne outbreaks[J].Microchemical Journal, 2020, 155:104660.
[9] 杨佰启, 温贵兰, 邱燕, 等.屠宰场猪肉中猪霍乱沙门氏杆菌的分离鉴定及16S rRNA的序列分析[J].黑龙江畜牧兽医, 2019(6):73-77;171.
YANG B Q, WEN G L, QIU Y, et al.Isolation, identification and 16S rRNA sequence analysis of Salmonella choleraesuis from pork in slaughterhouses[J].Heilongjiang Animal Science and Veterinary Medicine, 2019(6):73-77;171.
[10] 李娜. 沙门氏菌的检测与预防研究进展[J].食品安全导刊, 2022(11):178-180.
LI N.Research progress on detection and prevention of Salmonella[J].China Food Safety Magazine, 2022(11):178-180.
[11] 陈德纯, 周晏阳, 周泷, 等.一株牦牛源产细菌素植物乳杆菌SWUN5815全基因组测序及序列分析[J].中国农业大学学报, 2021, 26(5):61-71.
CHEN D C, ZHOU Y Y, ZHOU L, et al.Whole-genome sequencing and sequence analysis of a Lactobacillus plantarum SWUN5815 producing bacteriocin from yak[J].Journal of China Agricultural University, 2021, 26(5):61-71.
[12] CHEN Z Q, BAI J, WANG S J, et al.Prevalence, antimicrobial resistance, virulence genes and genetic diversity of Salmonella isolated from retail duck meat in southern China[J].Microorganisms, 2020, 8(3):444.
[13] 刘佳玫, 栗军杰, 陆兆新, 等.酸适应海德尔堡沙门氏菌对环境胁迫耐受性分析[J].食品科学, 2016, 37(21):209-213.
LIU J M, LI J J, LU Z X, et al.Tolerance of acid-adapted Salmonella heidelberg to various environmental stresses[J].Food Science, 2016, 37(21):209-213.
[14] FAINO L, SEIDL M F, DATEMA E, et al.Single-molecule real-time sequencing combined with optical mapping yields completely finished fungal genome[J].mBio, 2015, 6(4):e00936-e00915.
[15] EID J, FEHR A, GRAY J, et al.Real-time DNA sequencing from single polymerase molecules[J].Science, 2009, 323(5910):133-138.
[16] GALPERIN M Y, MAKAROVA K S, WOLF Y I, et al.Expanded microbial genome coverage and improved protein family annotation in the COG database[J].Nucleic Acids Research, 2015, 43(DI):D261-D269.
[17] KANEHISA M, GOTO S, HATTORI M, et al.From genomics to chemical genomics:New developments in KEGG[J].Nucleic Acids Research, 2006, 34(Database issue):D354-D357.
[18] ASHBURNER M, BALL C A, BLAKE J A, et al.Gene ontology:Tool for the unification of biology.The Gene Ontology Consortium[J].Nature Genetics, 2000, 25(1):25-29.
[19] CHEN L H, ZHENG D D, LIU B, et al.VFDB 2016:Hierarchical and refined dataset for big data analysis:10 years on[J].Nucleic Acids Research, 2016, 44(D1):D694-D697.
[20] JIA B F, RAPHENYA A R, ALCOCK B, et al.CARD 2017:Expansion and model-centric curation of the comprehensive antibiotic resistance database[J].Nucleic Acids Research, 2017, 45(D1):D566-D573.
[21] 罗逊, 丁碧荷, 王印, 等.一株兔源A型多杀性巴氏杆菌的分离鉴定和全基因组测序及分析[J].微生物学通报, 2024, 51(2):582-598.
LUO X, DING B H, WANG Y, et al.Isolation, identification, and whole genome sequencing of a rabbit-derived type A Pasteurella multocida strain[J].Microbiology China, 2024, 51(2):582-598.
[22] 支威, 马海燕, 仇永凤, 等.猪源沙门氏菌耐药性及耐药基因的分析[J].生物技术通报, 2018, 34(3):170-176.
ZHI W, MA H Y, QIU Y F, et al.Analysis of antimicrobial resistance and resistance genes of Salmonella from swine[J].Biotechnology Bulletin, 2018, 34(3):170-176.
[23] 何名扬, 朱必婷, 王鸣秋, 等.武汉地区食源性沙门氏菌耐药特征分析[J].食品安全质量检测学报, 2021, 12(1):78-85.
HE M Y, ZHU B T, WANG M Q, et al.Analysis of drug resistance characteristics of food-borne Salmonella in Wuhan Area[J].Journal of Food Safety &Quality, 2021, 12(1):78-85.
[24] 贺恒旭, 刘璐函, 刘俊峰, 等.生猪屠宰环节沙门氏菌携带状况、耐药性及其耐药基因分析[J].食品安全质量检测学报, 2021, 12(8):3034-3040.
HE H X, LIU L H, LIU J F, et al.Prevalence, antimicrobial resistance phenotypes and antimicrobial resistance genes of Salmonella isolates from pig slaughtering[J].Journal of Food Safety &Quality, 2021, 12(8):3034-3040.
[25] 姚玉芳, 张竹慧, 杨小双.黄山市43株沙门氏菌耐药分析及PFGE分子分型[J].安徽预防医学杂志, 2022, 28(3):202-205;211.
YAO Y F, ZHANG Z H, YANG X S.Drug resistance analysis and PFGE molecular typing of 43 Salmonella strains in Huangshan City[J].Anhui Journal of Preventive Medicine, 2022, 28(3):202-205;211.