单核细胞增生李斯特氏菌(Listeria monocytogenes),简称单增李斯特菌。相比其他食源性致病菌,人类因食用被单增李斯特菌污染的食物而引起的李斯特菌病是相对罕见的,但所引起的相关疾病却是致命的。侵袭性李斯特菌病往往会引起脑膜炎、败血症等,病死率约为20%~30%。据相关报道,单增李斯特菌所引起的李斯特菌病往往发生在易感人群(孕妇、新生儿以及65岁以上的人群)和极易感人群(免疫功能低下的人群,如艾滋病患者、癌症患者和器官移植患者)中。其中,妊娠期感染会对胎儿造成严重影响,可能会引起流产、死产或胎儿出生缺陷[1]。这归因于单增李斯特菌丰富的毒力因子、独特的细胞内生活方式以及跨越多种宿主屏障的能力,如肠道屏障、血脑屏障和胎盘屏障。由于这些原因,它也被用作研究致病性细菌如何介导宿主-病原体相互作用的模型细菌。单增李斯特菌感染周期主要包括3个阶段:宿主细胞的侵袭和黏附、宿主细胞内增殖和宿主细胞间扩散,每个阶段的实现都取决于特定毒力因子的协调作用,而这些协调作用又受到调控因子的严格控制[2]。
在早期的研究中,科研人员已对单增李斯特菌毒力因子进行了广泛研究,如肌动蛋白组装诱导蛋白(actin assembly-inducing protein,ActA)、内化素A(internalin A,InlA)、内化素B(internalin B,InlB)、李斯特菌溶血素O(listeriolysin O,LLO)等。近些年,随着分子生物信息学技术、组学技术的快速发展,单增李斯特菌在毒力因子及其调控机制得到了更进一步的表征和鉴定,了解这些新发现对于深入理解单增李斯特菌和宿主之间的相互关系具有重要意义。基于此,本文回顾整理了单增李斯特菌在感染期间关键毒力因子、调控因子和最新的生物学进展,着重讨论了调控因子在宿主外环境和内环境的相互作用,为进一步预防和控制单增李斯特菌感染提供理论参考。
1 主要毒力因子
单增李斯特菌的黏附、侵袭、繁殖、扩散和侵入人体各种组织和器官的能力取决于众多的毒力因子,如表1所示。目前研究表明,单增李斯特菌最重要的毒力因子受inlA-inlB基因座、毒力岛1(Listeria pathogenicity island 1,LIPI-1)、毒力岛3(Listeria pathogenicity island 3,LIPI-3)和毒力岛4(Listeria pathogenicity island 4,LIPI-4)编码。值得注意的是,inlA-inlB基因座和LIPI-1的表达都受到正调节因子A(positive regulatory factor A,PrfA)的调控。
表1 单增李斯特菌主要毒力因子及主要功能
Table 1 Main virulence factors and functions involved in L.monocytogenes

毒力因子调控基因主要功能参考文献InlA、InlBinlA、inlBInlA与细胞受体E-cadherin相互作用而内化。InlB与细胞受体Met相互作用而内化。[3]ActAactA介导肌动蛋白聚合驱动的细菌运动,促进病原体在宿主细胞间的扩散和宿主组织内传播。[5]LLOhly在宿主细胞表面制造膜穿孔,促进单增李斯特菌内化为吞噬小体。在吞噬小体内诱导孔的形成,促进细菌逃离吞噬小体,并协助病原体在细胞内复制。[10,13]PlcA、PlcBplcA、plcB介导病原体从吞噬小体中逃逸。介导病原体逃离宿主的自噬防御。[14,16]
1.1 内化素
内化素是李斯特菌致病性相关基因编码的表面蛋白。由inlA-inlB操纵子编码的InlA和InlB是单增李斯特菌中最早被鉴定的内化素,介导病原体对宿主非吞噬细胞的黏附和内化,其特征是含有富含亮氨酸重复序列(leucine-rich repeats,LRRs)的n端结构域,与它们各自的宿主细胞受体E-钙黏蛋白(E-cadherin)和肝细胞生长因子受体Met的胞外结构域结合,导致宿主肌动蛋白细胞骨架的局部聚合,从而引起细菌内化(图1)。在InlB依赖性内化过程中,细胞表面受体Met和下游磷脂酰肌醇3-激酶(phosphatidylinositol 3-kinase,PI3K)/蛋白激酶B(protein kinase B,PKB)信号通路会被激活,导致宿主细胞去乙酰化酶sirtuin 2(deacetylase sirtuin 2,SIRT2)易位到细胞核,并使赖氨酸18(histone H3 on lysine 18,H3K18)上的组蛋白H3(histone H3)去乙酰化(图1)。而在近年来相关研究中发现,InlB介导对宿主细胞的内化作用不仅涉及肌动蛋白聚合,还涉及“极化胞吐作用”的过程。在这个过程中,丝氨酸/苏氨酸蛋白激酶(mammalian target of rapamycin,mTOR)、蛋白激酶C-α(protein kinase C-α,PKC-α)和Ras相关蛋白Ral-A(ras-related protein Ral-A,RalA)通过控制细丝蛋白A(filamin A,FlnA)在进入过程中促进胞吐,但InlA介导的内化是否涉及胞吐作用还尚不明确[3]。InlA和E-cadherin的相互作用在单增李斯特菌穿越肠道屏障和胎盘屏障中起到重要作用,但介导的内化作用局限于有限数量的上皮细胞,而Inlb介导的内化作用针对多种细胞,如肝细胞、上皮细胞和内皮细胞等。长期以来,无法通过小鼠模型评估InlA在单增李斯特菌毒力中的作用,此外豚鼠和兔的Met也无法被InlB识别或刺激,表现出单增李斯特菌的物种特异性[4]。
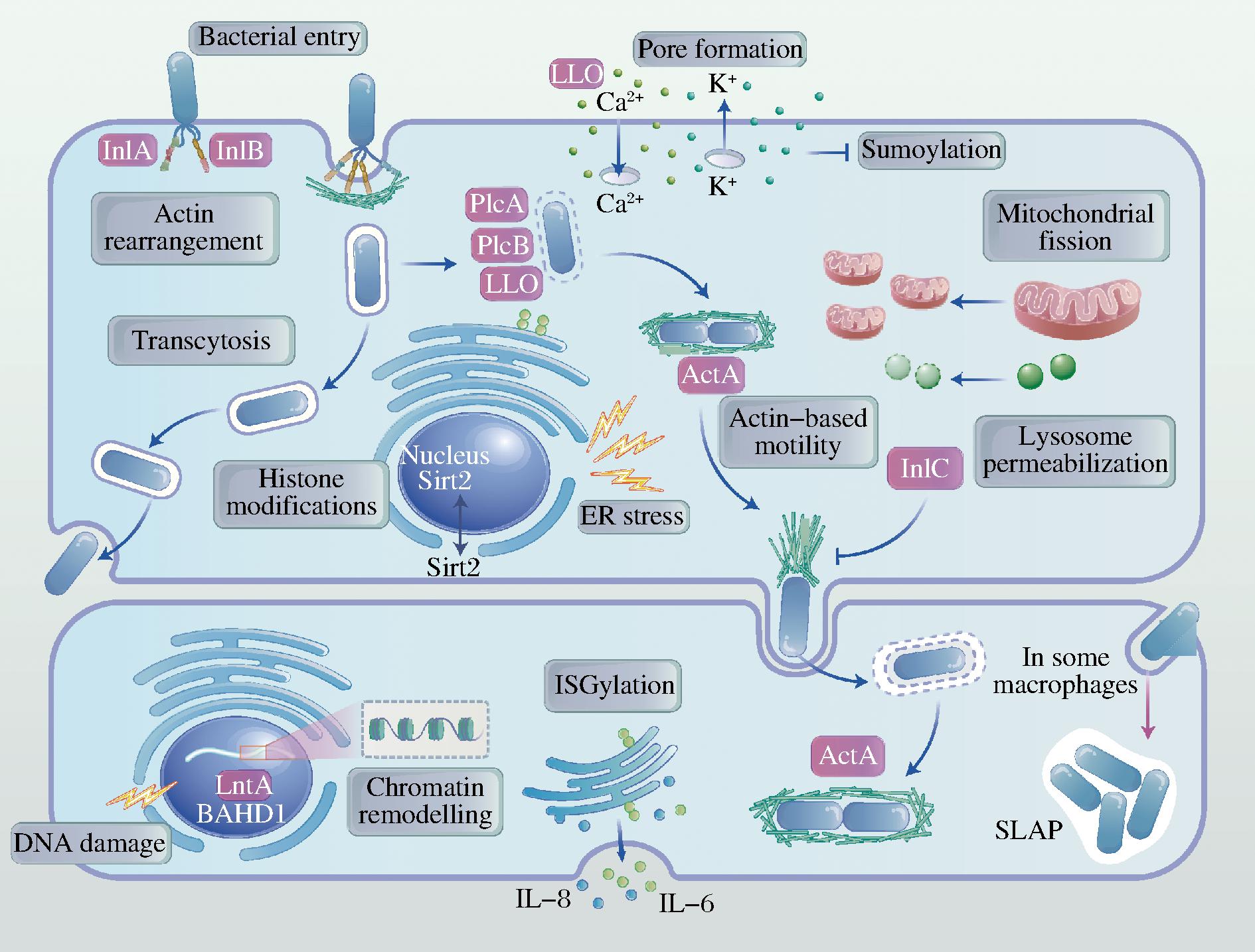
图1 单增李斯特菌的细胞感染周期(根据参考文献[2]改绘)
Fig.1 L.monocytogenes cellular infection cycle (redrawn from reference[2])
1.2 LIPI-1
LIPI-1在所有致病的单增李斯特菌菌株中是保守的,包含参与宿主侵袭和细胞增殖的关键基因prfA、actA、hly、plcA和plcB,这些基因的表达均受主要毒力调控因子PrfA的调控。
1.2.1 ActA
由actA基因编码的ActA由639个氨基酸组成,通过其C端区附着在细菌细胞壁上。相关研究表明,ActA本身介导了肌动蛋白聚合驱动的细菌运动,促进了单增李斯特菌在细胞间的扩散和宿主组织内传播(图1)。在这个过程中,ActA和InlC通过协同作用干扰细胞骨架和调节细胞内皮质张力,促进宿主细胞的染色体易位,干扰宿主细胞的有丝分裂,推迟细胞周期,从而促进单增李斯特菌在宿主细胞内的定殖和传播[5]。相关研究认为,ActA介导细菌通过细胞质进入邻近细胞的同时,靠近吞噬小体膜的肌动蛋白可能通过LLO介导的孔隙进入,其聚合可使孔隙变宽。这导致吞噬小体的破坏,促进细菌从吞噬小体逃逸[6]。在上皮细胞中,单增李斯特菌利用ActA的仿生特性,将Ena/VASP和Arp2/3复合物组成的宿主蛋白伪装自己,从而逃避自噬的识别[7]。此外,ActA对单增李斯特菌向胎盘侵入至关重要,并在向胎儿的垂直传播中发挥了关键作用[8]。在低营养、抗性(低温、pH、抗生素、消毒剂等)条件下,单增李斯特菌会形成生物被膜以抵抗外部不利条件,相关研究发现,ActA还介导细菌间的相互作用和生物被膜的形成,促进了单增李斯特菌在肠道内的聚集和定殖,提高了在环境中传播的可能性[9]。
1.2.2 溶血素
由hly基因编码的LLO已被证明能促进宿主内部的感染,主要是细胞外环境、吞噬小体和细胞质。在细胞外环境,LLO通过在宿主细胞中制造膜穿孔,促进单增李斯特菌内化为吞噬小体(图1)。LLO通过诱导离子通量的组合,允许Ca2+内流,促进了单增李斯特菌对人喉表皮样癌细胞(Hep-2)的侵袭[10]。Ca2+内流会诱导宿主细胞内多种信号通路激活,包括细胞外调节蛋白激酶1(extracellular regulated protein kinases 1,ERK-1)、细胞外调节蛋白激酶2(extracellular regulated protein kinases 2,ERK-2)、p38丝裂原活化蛋白激酶(p38 mitogen-activated protein kinase,p38 MAPK)和应激活化蛋白激酶(stress-activated protein kinase,SAPK)通路的激活、磷脂酰肌醇(phosphatidylinositol,PI)代谢、核转录因子(nuclear factor kappa-B,NF-κB)核易位、促炎细胞因子白细胞介素6(interleukin 6,IL-6)、白细胞介素8(interleukin 8,IL-8)、粒细胞巨噬细胞集落刺激因子(granulocyte macrophage colony-stimulating factor,GM-CSF)和白细胞介素1α(interleukin 1α,IL-1α)的分泌等[11]。在SAMBA-LOUAKA等[12]的一项研究中发现,LLO介导DNA损伤感应子减数分裂重组同源物11(meiotic recombination 11,Mre11)的降解,改变感染时引起的DNA损伤反应(DNA damage response,DDR),从而促进了单增李斯特菌对宿主细胞的感染。此外,相关研究发现单增李斯特菌感染期间还可诱导多种细胞凋亡,如淋巴细胞、树突状细胞等。在吞噬小体内,LLO诱导孔的形成,以促进细菌逃离宿主吞噬小体,并协助病原体在细胞内复制。在此过程中产生的膜损伤可使膜磷脂在磷脂酶作用下水解,从而导致质膜完全破裂[13]。
1.2.3 磷脂酶
单增李斯特菌可以分泌2种不同的磷脂酶C,分别是由plcA基因编码的磷脂酰肌醇特异性磷脂酶C(phosphatidylinositol-specific phospholipase C,PlcA)和plcB基因编码的非特异性磷脂酰胆碱磷脂酶C(non-specific phosphotidylcholine phospholipase C,PlcB)。相关研究证明PlcA在介导单增李斯特菌从小鼠原代巨噬细胞吞噬小体中逃逸发挥重要作用(图1)[14]。在进入宿主细胞后,单增李斯特菌从形成的初级单膜液泡中逃逸,使细菌能够进入宿主细胞的细胞质。细菌在细胞质内复制,利用肌动蛋白为基础的运动扩散到邻近的细胞。在这个过程中,LLO已被证明是绝对必须的,而相关研究发现,即使LLO表达缺失,PlcB在侵袭上皮细胞时协助空泡逃逸,介导单增李斯特菌在宿主细胞间的传播。PlcB最初以酶原形式表达,然后在酸性环境下被金属蛋白酶激活而发挥作用。此外,PlcB也被认为是单增李斯特菌引起脑膜炎的一个重要毒力因子[15]。在上文中,LLO可以介导单增李斯特菌触发相应机制以逃离宿主的自噬防御,自噬是宿主细胞内功能失调的细胞器降解和循环的过程,自噬也针对侵袭的病原体,在先天免疫中发挥重要作用。而在一项研究发现,PlcA和PlcB降低了自噬通量和磷脂酰肌醇3-磷酸(phosphatidylinositol 3-phosphate,PI3P)水平,导致自噬体前结构停滞,阻止了胞质细菌的有效靶向。这揭示了PlcA和PlcB在抑制自噬通量中的关键作用,有利于细菌逃离宿主的自噬防御[16]。
1.3 LIPI-3
单增李斯特菌至少由4个进化谱系(Ⅰ、Ⅱ、Ⅲ和Ⅳ)组成,具有不同但重叠的生态位。大多数分离出的单增李斯特菌分离物属于Ⅰ和Ⅱ谱系,如在人类临床病例常见的血清型,包括血清型1/2a(Ⅱ谱系)和血清型1/2b和4b(Ⅰ谱系)。Ⅱ谱系菌株在食品中很常见,广泛存在于自然和农场环境中,而且也经常从动物李斯特菌病病例和零散的人类临床病例中分离出来。然而,大多数人类李斯特菌病的暴发都与Ⅰ谱系有关。此外,一些研究表明,在许多国家,与第Ⅱ谱系菌株相比,第Ⅰ谱系菌株在人类分离物中的比例更高。而Ⅲ谱系和Ⅳ谱系菌株是罕见的,主要分离自动物源。目前,单增李斯特菌LIPI-3似乎只在单增李斯特菌的Ⅰ谱系分离株中发现。LIPI-3由8个基因组成,与李斯特菌溶血素S(listeriolysin S,LLS)的产生和高毒力相关[17]。
LLS是一种翻译后修饰的溶血素。在小鼠模型中,LLS对机体的致病性起作用,此外在多形核嗜中性粒细胞(polymorphonuclear neutrophil,PMN)中对单增李斯特菌的存活也起到重要作用。然而在最近的研究中发现,LLS不会引起特异性免疫反应,对真核细胞没有细胞毒性,也不会影响单增李斯特菌的细胞感染。但LLS可改变宿主肠道菌群,促进单增李斯特菌在肠道的定殖和进一步扩散感染。这在一定程度上解释了谱系Ⅰ在李斯特菌病暴发中所占比例过高的原因[18]。MEZA-TORRES等[19]采用亚细胞分级和免疫金标记技术研究LLS对细胞毒性作用、杀菌机制和LLS表达机制时发现,LLS是第一个显示出接触依赖性抑制机制的噻唑/恶唑修饰小菌素(thiazole/oxazole modified microcin,TOMM),在体内感染期间诱导肠道共生细菌的膜透化和去极化,并专门针对原核细胞。
1.4 LIPI-4
MAURY等[20]整合了整合流行病学数据和比较基因组学,发现了与单增李斯特菌致病性有关的新基因簇LIPI-4。该基因簇编码一个纤维二糖家族磷酸转移酶系统(phosphotransferase system,PTS),这种PTS已被确定为与母婴和中枢神经系统感染有关的超毒性因子,具有重要的临床意义。LIPI-4也与单增李斯特菌高毒力密切相关,但其原因有待确定。近些年,含有LIPI-4高毒力的单增李斯特菌菌株逐渐在即食食品、牛奶等食品和加工环境被分离出来,这可能对食品安全和公共卫生构成潜在威胁[21-23]。
2 单增李斯特菌毒力调控因子和关键转录调控因子
当宿主细胞被单增李斯特菌黏附、侵袭后,PrfA和管家基因σB对单增李斯特菌在宿主细胞内的生长和传播起着重要作用。
2.1 PrfA
PrfA直接调控一组10~15个蛋白质编码基因以及至少一种非编码RNA(non-coding RNA,ncRNA),所有功能已知的PrfA依赖基因似乎都与单增李斯特菌毒力相关。由PrfA调控的关键功能包括细胞内生存和细胞间传播,参与这些功能的PrfA依赖性基因在一个也编码PrfA的毒力基因岛(毒力岛1)中编码。该毒力岛中依赖PrfA的毒力基因包括actA、hly、plcA、plcB等[24]。PrfA特定基因的转录水平依赖于3个不同的因素:(1)PrfA蛋白浓度;(2)PrfA活性;(3)靶基因启动子的结构。单增李斯特菌必须在时间和空间上严格控制PrfA调控基因的表达,以确保在感染过程中成功黏附、侵袭。PrfA的无节制激活会给细胞带来负面影响[25]。除了PrfA调控基因的激活外,PrfA调控基因组编码的蛋白质翻译后调控也增强了毒力基因的分级表达,如LLO在细胞质中的表达对于单增李斯特菌逃离吞噬小体是必不可少的,然而细胞质中高水平的LLO活性可以过早地杀死宿主细胞,从而抑制了单增李斯特菌在细胞间的传播[13]。
2.1.1 prfA的转录调控
由于PrfA在许多毒力基因的转录激活中发挥着重要作用,因此必须对PrfA的水平和活性进行精确调控。
prfA的转录是由3个不同的启动子控制。除了直接来自prfA上游的两个启动子(称为prfAP1和prfAP2)外,prfA还从一个远端启动子prfAP3转录,产生一个双区plcA-prfA转录子。prfAP1是一个简单的依赖σA的启动子,没有任何明显的附加调节元件,而prfAP2是一个复杂的启动子,包括重叠的σA和σB的识别序列和一个假定的PrfA蛋白结合区。prfAP3是plcA上游的唯一启动子,它代表了一个σA依赖的启动子,其上游有一个典型的PrfA蛋白结合区[26]。目前有明确的研究结果表明,prfA转录的调节包括一个由prfAP3介导的正调节反馈回路,其介导的调节反馈回路在宿主细胞感染时促进PrfA快速上调中发挥了重要作用,在PrfA激活后,prfAP3的正调节导致了更高水平的prfA转录[25]。除了prfAP3启动子介导的正调节反馈回路外,也有研究表明prfAP2和相关的调控元件介导了负反馈调节[27]。
2.1.2 prfA的转录后调控
除了prfA的转录调控外,单增李斯特菌还通过转录后调控对PrfA的表达产生影响。PrfA的转录后调控主要包括以下3个方面:(1)通过RNA的稳定性和翻译起始密码子的可及性进行调控;(2)通过与PrfA的辅助因子的结合来调控其活性;(3)通过降解和隔离来调节蛋白质水平。这些不同的调控方式协调发挥作用,以增加单增李斯特菌在宿主外环境的适应性,并确保宿主内的成功感染。此外,σB直接或间接地影响PrfA的水平和活性,因为许多参与PrfA转录后调控机制的基因都是从σB依赖的启动子进行转录[28]。OLLINGER等[29]结合DNA微阵列技术、逆转录定量PCR和表型实验发现当PrfA有活性时,σB负责降低PrfA调控蛋白的表达。σB依赖性调节PrfA蛋白的表达,降低了PrfA活性菌株在人肝癌细胞(Hep-G2)中的毒性,突出了PrfA和σB之间调节功能的重要性。这些调控机制协同作用,提高了单增李斯特菌在不同环境中的适应性和感染能力。
2.2 σB
自1998年首次发现单增李斯特菌σB因子以来,已经有300多个基因被证明受σB因子的转录调控(约占基因组的10%)[30]。虽然σB因子可以识别的启动子序列为GTTTNW-N13-16-GGGWADW,但由于其调控成员参与细胞的各种功能,它也间接影响大量缺乏该启动子序列的基因表达。利用多组学策略以及生理和遗传学方法,目前已经确定了许多直接或间接受σB因子控制的基因和功能,包括一般应激反应、多种运输系统、转录后调节、毒力、代谢、耐药性以及一些功能未知的基因[31]。
相关研究表明,σB因子在宿主细胞内活性较低,在一定程度上表明单增李斯特菌在细胞间的传播较少依赖于σB因子的调控表达[32]。GULDIMANN等[33]的一项研究中发现,在不同的环境条件下,σB因子只在部分单增李斯特菌中表达,而PrfA在整个群体中均有表达。由此可见,单增李斯特菌σB因子的表达是随机的,而PrfA的表达是分级的。由于σB因子表达的高生理成本,仅在亚群中激活σB可能对单增李斯特菌有利,而毒力基因在宿主所有细胞中表达可增加细胞感染成功率。
2.3 PrfA和σB之间的相互作用
为了在宿主外环境和内环境中生长,单增李斯特菌必须依赖其应激反应和毒力的主要调控因子σB和PrfA的表达。σB和PrfA之间复杂的相互作用提供了不同微环境中基因表达的模式。两者之间的相互作用主要包括在宿主外的相互作用、在宿主消化道中的相互作用和在宿主细胞中的相互作用。
2.3.1 PrfA和σB在宿主外的相互作用
在宿主体外,单增李斯特菌必须在σB因子和PrfA表达之间达到适当的平衡,以保证适应不同的环境条件。单增李斯特菌的一个重要的宿主外生存策略来自于它附着在物体表面并形成生物被膜的能力。生物被膜内的微生物细胞组织成复杂的结构,在这些结构中,它们嵌入自产的细胞外聚合物质(extracellular polymeric substances,EPS)基质中,这些物质负责生物被膜表面的黏附和凝聚。EPS还赋予生物被膜一些特征,如结构复杂性、更高的去除和破坏抵抗力以及更高的抗微生物剂抵抗力。此外,在生物被膜发育的最后阶段,微生物能够从生物被膜上分离并分散到外部环境中(浮游形式)[34]。PrfA和σB在特定生长条件下对生物被膜的形成起协同作用。在高盐(质量分数6% NaCl)和低温(15 ℃)条件下,单增李斯特菌σB活性被诱导,形成稳定的生物被膜。此外,相关研究也表明σB因子在单增李斯特菌表面附着和进一步生物被膜的形成起到重要作用[35]。目前,σB对生物被膜形成或表面附着的确切分子机制仍然是未知的。同样,PrfA似乎也在生物被膜形成中发挥作用,缺乏PrfA的突变体菌株在生物被膜的形成上有一定缺陷[25]。
2.3.2 PrfA和σB在宿主消化道中的相互作用
在侵袭宿主细胞之前,单增李斯特菌必须在宿主胃肠道内物理、化学消化和防御系统的条件下存活,主要包括胃部酸应激、高渗透、胆汁应激、营养竞争和肠道菌群产生的抗菌化合物。单增李斯特菌也必须启动早期毒力基因的表达以保证对宿主细胞的成功侵袭[36]。在胃肠道中,单增李斯特菌依赖σB的表达来应对不利环境,这也可能提高与其他胃肠道微生物群竞争的能力。与宿主外环境的生长和存活相比,在胃肠道中,单增李斯特菌需要调控prfA的表达,以确保对寄主细胞侵袭。这种转变可能是通过解除prfA mRNA的转录后抑制和增加prfAP2启动子σB依赖的prfA转录表达来实现的,相关研究也表明σB和PrfA之间的相互作用对于促进单增李斯特菌在胃肠道的生存至关重要[37]。
由于消化道的环境条件变化迅速且剧烈,单增李斯特菌必须通过感知信号来协调应激反应和毒力基因的表达,以快速有效地应对不断变化的条件。相关研究对比单增李斯特菌在小鼠肠道和脑心浸液培养基中的基因表达水平,在小鼠肠道中发现了广泛的转录重塑,共有437个基因上调,769个基因下调,许多与应激反应和低氧生存有关。其中有232个基因受到σB的控制,如编码侵袭蛋白InlA和InlB以及表面蛋白(如InlH、Lmo0610和Lmo2085)的相关基因,这些蛋白可能在黏附和穿越肠道屏障中发挥重要作用。在一项研究中,hly突变体菌株在肠道内的存活能力严重受损,这表明单增李斯特菌在肠道内的定殖生长与LLO密切相关[38]。此外在一项关于ActA对单增李斯特菌聚集、肠道定殖的研究中,C端缺失的ActA蛋白突变体菌株能够在细胞间感染传播,但在生物被膜形成和聚集方面有一定影响,感染4天后会被迅速从肠腔中清除,在小鼠肠道定殖中存在明显缺陷[39]。由于inlA、inlB、hly和actA的表达受到PrfA的调控,因此在一定程度上,PrfA也是单增李斯特菌在宿主肠道定殖以及对肠道上皮细胞的初始侵袭所必需的。但值得注意的是,σB介导的侵袭过程是由于对inlA转录的直接影响,而不是对prfA转录的影响。
宿主肠道内存活与细胞内侵袭之间的过渡是一个复杂的过程,不仅涉及PrfA调控基因的表达,还涉及单增李斯特菌蛋白组和转录组的重塑,以确保存活并逃避宿主免疫系统。甚至在对宿主细胞侵袭之前,单增李斯特菌就必须在通过胃肠道的过程中预先适应这种转变。通过胃肠道屏障是随后感染的一个先决条件,感知特定信号为必要条件。单增李斯特菌在不同胁迫条件下的传代生长为随后的感染侵袭过程提供了准备。相关研究发现,酸适应后单增李斯特菌表现出对热胁迫、渗透胁迫、结晶紫和乙醇的耐受性增加,将耐酸能力增强的突变体通过腹腔注射方式接种至小鼠模型体内,发现突变体菌株对小鼠的致死率比野生型菌株要高。这表明在酸适应后,单增李斯特菌突变体的自然耐酸性和毒力可能会增强[40]。此外,胆汁暴露似乎也是重塑基因转录为宿主侵袭提供准备的信号[41]。
2.3.3 PrfA和σB在在宿主细胞内的相互作用
单增李斯特菌内化后面临着不同的应激条件,包括低营养、pH和氧化应激等。适应性基因表达允许病原体在宿主细胞内微环境中遭遇宿主防御时持续存活和传播。CHATTERJEE等[42]通过全基因组微阵列和突变分析方法研究了单增李斯特菌在宿主细胞的基因表达情况,大约17%的总基因组表达以适应细胞内生长,包括参与糖酵解和柠檬酸循环的基因下调,参与替代碳源利用的基因上调,以及寡肽、脂肪酸、支链氨基酸的运输和代谢。在这过程中。σB因子本身的表达在整个感染期间没有受到显著影响,但由σB调控的相关基因上调。σB突变体在细胞内生长与野生型菌株无显著区别,这表明细胞内细菌的基因表达模式与体外稳定期细菌生长的基因表达模式不同。在一项关于prfA、σB突变的单增李斯特菌菌株在健康人类血液的生存研究中发现,毒力基因在血液中高度过表达,这些基因的表达在单增李斯特菌逃离吞噬小体并在宿主细胞和组织内传播起到重要作用。与在培养基中生长时的基因表达相比,在血液中上调了545个基因,下调了716个基因,除了上调与毒力和生长有关的基因(prfA)外,还抑制鞭毛编码等基因的表达,作为逃避宿主识别和宿主固有免疫系统的适应性机制[43]。细胞侵袭过程中差异表达的基因数量较多,包括编码毒力相关功能、细菌代谢和改变哺乳动物细胞生理功能的基因,这表明单增李斯特菌需要广泛的调控机制来确保对宿主细胞成功感染。
σB因子的表达对单增李斯特菌在消化道内存活中发挥了主要作用,而PrfA则是宿主细胞内的主要调节因子。单增李斯特菌在宿主细胞内上调PrfA,通过与辅助因子谷胱甘肽(glutathione,GSH)结合而激活蛋白,以确保PrfA依赖性的最佳激活水平。单增李斯特菌能够感知其环境,诱导毒力基因的分级表达。具体来说,hly和plcA主要在吞噬小体中表达,而actA和inlC在细胞质中被诱导表达。hly缺失菌株无法从宿主细胞吞噬小体中逃脱,在受感染的组织培养细胞中未检测到actA和plcB的表达,说明单增李斯特菌能够感受宿主细胞内的不同区域并调节相应的毒力基因。此外,支链氨基酸和L-谷氨酰胺也被确定为触发诱导毒力基因表达的可能信号[25]。
在宿主细胞内生长的单增李斯特菌,PrfA可在GSH和prfAP3启动子的自动调节下充分激活,可最大限度诱导prfA的转录。PrfA对GSH的亲和力相对较低,其解离常数(Kd)约为4~5 mmol/L,这使得PrfA可以逐渐被激活,而不会直接性地全部激活。这种逐渐激活可能是单增李斯特菌能够表达不同毒力基因的可能原因,且在一定程度上受到GSH含量水平的影响[44]。
2.4 其他毒力调控因子
除了PrfA和σB因子之外,其他调控因子如VirR、Hfq、MogR等也对单增李斯特菌毒力调控有一定的影响(表2)。
表2 单增李斯特菌主要毒力调控因子及主要功能
Table 2 Main virulence regulators and functions involved in L.monocytogenes

毒力调控因子调控基因主要功能参考文献PrfAprfA参与对众多毒力因子的调控,如actA、hly、pl-cA、plcB等。[24]σB-参与对一般应激反应、多种运输系统、转录后调节、毒力、代谢、耐药性等基因的调控。[31]VirRvirR通过表面成分修饰的全面调节来控制毒力表达。[45]Hfqhfq参与对渗透压、乙醇等应激反应的基因的调控。对致病性也有一定贡献。[48]MogRmogR在宿主细胞内抑制鞭毛蛋白基因的表达。[50]
VirR调控因子是通过特征标记突变技术识别出的涉及单增李斯特菌毒力的新基因。在一项研究中,virR缺失严重降低了单增李斯特菌对模型小鼠的致病能力。此外,转录组学鉴定了12个由VirR调控的基因,包括dlt-操纵子。已有研究表明该操纵子对单增李斯特菌的毒力调控起重要作用[45]。另一个VirR调控的基因与mprF同源,mprF基因编码一种蛋白,该蛋白用L-赖氨酸修饰膜磷脂酰甘油,形成左旋赖氨酸磷脂酰甘油酯蛋白,该蛋白可抵抗人源或细菌源的阳离子抗菌肽。因此,VirR似乎是通过对表面成分修饰的全面调节来控制毒力,这些修饰可能影响与宿主细胞的相互作用,包括先天免疫系统的组成部分[46]。
在革兰氏阴性菌中,RNA结合蛋白Hfq已成为多种生理过程中的重要调控因子,包括抗逆性和毒力。对现有基因组序列的分析表明,在革兰氏阴性菌和革兰氏阳性菌中都存在Hfq同源物[47]。相关研究结果发现Hfq参与了单增李斯特菌对渗透压和乙醇胁迫的能力,并有助于单增李斯特菌在氨基酸限制条件下的长期生存。然而对于酸和氧化应激,Hfq不是必需的。此外,hfq的应力诱导转录依赖于σB因子。感染研究表明,在单增李斯特菌对小鼠发挥致病性过程中Hfq起重要作用,但对培养细胞(巨噬细胞、肠上皮细胞)的感染无作用[48]。
鞭毛是一种复杂的表面结构,是许多细菌运动的主要手段,也是许多细菌病原体介导黏附、侵袭和毒力因子分泌的必要机制。然而,细菌鞭毛在感染时会激活宿主免疫应答,因此在定殖后,细菌鞭毛的产生通常受到抑制。事实上,在生理温度下,许多兼性细胞内病原体的鞭毛运动基因表达被抑制,包括单增李斯特菌[49]。在一项关于MogR转录抑制因子调控单增李斯特菌鞭毛运动基因的非层级表达和毒力的研究中,MogR在37 ℃的细胞外生长和细胞内感染期间严格抑制鞭毛蛋白(flagellin,FlaA)的表达。在小鼠的感染模型中,在没有MogR的情况下单增李斯特菌会过度产生鞭毛蛋白。鞭毛蛋白产生的失调导致细菌不再能够有效地进入宿主细胞,致病性降低[50]。
单增李斯特菌在复杂环境下需要高度敏感和稳定的调控机制来微调保护机制和毒力因子的表达,这些因素会共同影响其生存和致病性。深入理解这些调控机制可能有助于识别该病原体潜在弱点,进而为开发新一代抗菌药物提供科学依据。
3 总结与展望
综上所述,当单增李斯特菌进入宿主体内,便可通过复杂的调控机制侵袭不同的真核细胞,逃避宿主的免疫系统。此外,该病原体穿过肠道、血脑和胎盘屏障,进而对人体造成严重伤害(败血症、脑膜炎、流产和死胎等)。目前尽管在了解单增李斯特菌毒力机制方面取得了重大进展,但在分子水平上对其发病过程的认识仍然非常片面,如单增李斯特菌如何跨越宿主的生理屏障并与其靶细胞相互作用而致病的机制,以及单增李斯特菌肠道易位后如何对抗宿主的防御机制并长期存活等,这些问题都还有待深入研究。未来的展望如下:
(1)结合转录组学、蛋白质组学和代谢组学等组学技术来进一步揭示单增李斯特菌和宿主细胞的相互作用机制。
(2)利用体内成像技术、基于序列标签的微生物种群分析(sequence tag-based analysis of microbial populations,STAMP)等新兴技术结合动物模型探究单增李斯特菌在宿主体内的种群动态变化,量化不同感染部位之间病原体的种群相关性,对于进一步了解单增李斯特菌在宿主体内的传播途径具有重要意义。
(3)构建可用于研究单增李斯特菌毒性的3D新型细胞模型,如肠道、大脑、胎盘等细胞模型,并基于这些重要器官的体外模型对单增李斯特菌的靶向机制、感染后宿主的免疫反应及细菌的毒力机制开展研究,为单增李斯特菌的精准防控提供科学参考。
[1] CHENG Y, DONG Q L, LIU Y T, et al.Systematic review of Listeria monocytogenes from food and clinical samples in Chinese mainland from 2010 to 2019[J].Food Quality and Safety, 2022, 6:fyac021.
[2] RADOSHEVICH L, COSSART P.Listeria monocytogenes:Towards a complete picture of its physiology and pathogenesis[J].Nature Reviews.Microbiology, 2018, 16(1):32-46.
[3] BHALLA M, VAN NGO H, GYANWALI G C, et al.The host scaffolding protein filamin A and the exocyst complex control exocytosis during InlB-mediated entry of Listeria monocytogenes[J].Infection and Immunity, 2018, 87(1):e00689-e00618.
[4] DISSON O, MOURA A, LECUIT M.Making sense of the biodiversity and virulence of Listeria monocytogenes[J].Trends in Microbiology, 2021, 29(9):811-822.
[5] COSTA A C, PINHEIRO J, REIS S A, et al.Listeria monocytogenes interferes with host cell mitosis through its virulence factors InlC and ActA[J].Toxins, 2020, 12(6):411.
[6] NGUYEN B N, PORTNOY D A.An inducible cre-lox system to analyze the role of LLO in Listeria monocytogenes pathogenesis[J].Toxins, 2020, 12(1):38.
[7] COLIN A, BLANCHOIN L.Cytoskeleton regulation:Distinct steps in Arp2/3 complex activation[J].Current Biology:CB, 2022, 32(5):R220-R222.
[8] 吴梦洁, 董庆利, 鲁新新, 等.单增李斯特菌感染胎盘相关毒力因子的研究进展[J].生物工程学报, 2022, 38(6):2139-2152.
WU M J, DONG Q L, LU X X, et al.Placenta-specific virulence factors involved in Listeria monocytogenes infection[J].Chinese Journal of Biotechnology, 2022, 38(6):2139-2152.
[9] JANEŽ N,  KRLJ B, STERNI
KRLJ B, STERNI A M, et al.The role of the Listeria monocytogenes surfactome in biofilm formation[J].Microbial Biotechnology, 2021, 14(4):1269-1281.
A M, et al.The role of the Listeria monocytogenes surfactome in biofilm formation[J].Microbial Biotechnology, 2021, 14(4):1269-1281.
[10] VADIA S, SEVEAU S.Fluxes of Ca2+ and K+ are required for the listeriolysin O-dependent internalization pathway of Listeria monocytogenes[J].Infection and Immunity, 2014, 82(3):1084-1091.
[11] PHELPS C C, VADIA S, ARNETT E, et al.Relative roles of listeriolysin O, InlA, and InlB in Listeria monocytogenes uptake by host cells[J].Infection and Immunity, 2018, 86(10):e00555-e00518.
[12] SAMBA-LOUAKA A, PEREIRA J M, NAHORI M A, et al.Listeria monocytogenes dampens the DNA damage response[J].PLoS Pathogens, 2014, 10(10):e1004470.
[13] MCDOUGAL C E, SAUER J D.Listeria monocytogenes:The impact of cell death on infection and immunity[J].Pathogens, 2018, 7(1):8.
[14] SHAHID A D, LU Y, IQBAL M A, et al.Listeria monocytogenes crosses blood brain barrier through Rho GTPases induced migration of macrophages and inflammatory interleukin expression[J].Microbial Pathogenesis, 2021, 159:105143.
[15] MONTURIOL-GROSS L, VILLALTA-ROMERO F, FLORES-D AZ M, et al.Bacterial phospholipases C with dual activity:Phosphatidylcholinesterase and sphingomyelinase[J].FEBS Open Bio, 2021, 11(12):3262-3275.
AZ M, et al.Bacterial phospholipases C with dual activity:Phosphatidylcholinesterase and sphingomyelinase[J].FEBS Open Bio, 2021, 11(12):3262-3275.
[16] QUEREDA J J, MOR N-GARC
N-GARC A A, PALACIOS-GORBA C, et al.Pathogenicity and virulence of Listeria monocytogenes:A trip from environmental to medical microbiology[J].Virulence, 2021, 12(1):2509-2545.
A A, PALACIOS-GORBA C, et al.Pathogenicity and virulence of Listeria monocytogenes:A trip from environmental to medical microbiology[J].Virulence, 2021, 12(1):2509-2545.
[17] LEE S M, PARSONS C, CHEN Y, et al.Contrasting genetic diversity of Listeria pathogenicity islands 3 and 4 harbored by nonpathogenic Listeria spp[J].Applied and Environmental Microbiology, 2023, 89(2):e0209722.
[18] QUEREDA J J, MEZA-TORRES J, COSSART P, et al.Listeriolysin S:A bacteriocin from epidemic Listeria monocytogenes strains that targets the gut microbiota[J].Gut Microbes, 2017, 8(4):384-391.
[19] MEZA-TORRES J, LELEK M, QUEREDA J J, et al.Listeriolysin S:A bacteriocin from Listeria monocytogenes that induces membrane permeabilization in a contact-dependent manner[J].Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(40):e2108155118.
[20] MAURY M M, TSAI Y H, CHARLIER C, et al.Uncovering Listeria monocytogenes hypervirulence by harnessing its biodiversity[J].Nature Genetics, 2016, 48(3):308-313.
[21] LI W W, GUO Y C, CUI Q P, et al.Whole-genome sequencing-based characterization of clinical Listeria monocytogenes isolates in China, 2013-2019[J].Foodborne Pathogens and Disease, 2023, 20(4):158-168.
[22] ZHANG Y Y, DONG S L, CHEN H H, et al.Prevalence, genotypic characteristics and antibiotic resistance of Listeria monocytogenes from retail foods in bulk in Zhejiang Province, China[J].Frontiers in Microbiology, 2019, 10:1710.
[23] ZHANG H Z, CHEN W J, WANG J, et al.10-year molecular surveillance of Listeria monocytogenes using whole-genome sequencing in Shanghai, China, 2009-2019[J].Frontiers in Microbiology, 2020, 11:551020.
[24] WIKTORCZYK-KAPISCHKE N, SKOWRON K, WA ECKA-ZACHARSKA E.Genomic and pathogenicity islands of Listeria monocytogenes-overview of selected aspects[J].Frontiers in Molecular Biosciences, 2023, 10:1161486.
ECKA-ZACHARSKA E.Genomic and pathogenicity islands of Listeria monocytogenes-overview of selected aspects[J].Frontiers in Molecular Biosciences, 2023, 10:1161486.
[25] GABALLA A, GUARIGLIA-OROPEZA V, WIEDMANN M, et al.Cross talk between SigB and PrfA in Listeria monocytogenes facilitates transitions between extra- and intracellular environments[J].Microbiology and Molecular Biology Reviews:MMBR, 2019, 83(4):e00034-e00019.
[26] HENDERSON L O, GABALLA A, ORSI R H, et al.Transcriptional profiling of the L.monocytogenes PrfA regulon identifies six novel putative PrfA-regulated genes[J].FEMS Microbiology Letters, 2020, 367(22):fnaa193.
[27] TIENSUU T, GUERREIRO D N, OLIVEIRA A H, et al.Flick of a switch:Regulatory mechanisms allowing Listeria monocytogenes to transition from a saprophyte to a killer[J].Microbiology, 2019, 165(8):819-833.
[28] DOREY A, MARINHO C, PIVETEAU P, et al.Role and regulation of the stress activated sigma factor sigma B (σB) in the saprophytic and host-associated life stages of Listeria monocytogenes[J].Advances in Applied Microbiology, 2019, 106:1-48.
[29] OLLINGER J, BOWEN B, WIEDMANN M, et al.Listeria monocytogenes sigmaB modulates PrfA-mediated virulence factor expression[J].Infection and Immunity, 2009, 77(5):2113-2124.
[30] LIU Y C, ORSI R H, BOOR K J, et al.Home alone:Elimination of all but one alternative sigma factor in Listeria monocytogenes allows prediction of new roles for σB[J].Frontiers in Microbiology, 2017, 8:1910.
[31] ORSI R H, CHATURONGAKUL S, OLIVER H F, et al.Alternative σ factors regulate overlapping as well as distinct stress response and metabolic functions in Listeria monocytogenes under stationary phase stress condition[J].Pathogens, 2021, 10(4):411.
[32] KANNAN S, BALAKRISHNAN J, GOVINDASAMY A.Listeria monocytogens - Amended understanding of its pathogenesis with a complete picture of its membrane vesicles, quorum sensing, biofilm and invasion[J].Microbial Pathogenesis, 2020, 149:104575.
[33] GULDIMANN C, GUARIGLIA-OROPEZA V, HARRAND S, et al.Stochastic and differential activation of σB and PrfA in Listeria monocytogenes at the single cell level under different environmental stress conditions[J].Frontiers in Microbiology, 2017, 8:348.
[34] WANG Y, WU Y Z, NIU H M, et al.Different cellular fatty acid pattern and gene expression of planktonic and biofilm state Listeria monocytogenes under nutritional stress[J].Food Research International, 2023, 167:112698.
[35] HSU C Y, CAIRNS L, HOBLEY L, et al.Genomic differences between Listeria monocytogenes EGDe isolates reveal crucial roles for SigB and wall rhamnosylation in biofilm formation[J].Journal of Bacteriology, 2020, 202(7):e00692-e00619.
[36] 朱雅慧, 王佳莹, 朱洪日, 等.单增李斯特菌在模拟人体胃环境中的耐受性[J].中国食品学报, 2018, 18(12):39-45.
ZHU Y H, WANG J Y, ZHU H R, et al.Tolerance of Listeria monocytogens to the simulating human stomach[J].Journal of Chinese Institute of Food Science and Technology, 2018, 18(12):39-45.
[37] DAVIS M L, RICKE S C, DONALDSON J R.Establishment of Listeria monocytogenes in the gastrointestinal tract[J].Microorganisms, 2019, 7(3):75.
[38] 李巧巧, 王欣雨, 潘星羽, 等.溶血素基因缺失对单增李斯特菌在小鼠肠道内定殖的影响[J].畜牧与兽医, 2022, 54(9):104-108.
LI Q Q, WANG X Y, PAN X Y, et al.Effect of the hemolysin gene deletion on Listeria monocytogenes colonization in the intestinal tracts of mice[J].Animal Husbandry &Veterinary Medicine, 2022, 54(9):104-108.
[39] TRAVIER L, GUADAGNINI S, GOUIN E, et al.ActA promotes Listeria monocytogenes aggregation, intestinal colonization and carriage[J].PLoS Pathogens, 2013, 9(1):e1003131.
[40] SIBANDA T, BUYS E M.Listeria monocytogenes pathogenesis:The role of stress adaptation[J].Microorganisms, 2022, 10(8):1522.
[41] GUARIGLIA-OROPEZA V, ORSI R H, GULDIMANN C, et al.The Listeria monocytogenes bile stimulon under acidic conditions is characterized by strain-specific patterns and the upregulation of motility, cell wall modification functions, and the PrfA regulon[J].Frontiers in Microbiology, 2018, 9:120.
[42] CHATTERJEE S S, HOSSAIN H, OTTEN S, et al.Intracellular gene expression profile of Listeria monocytogenes[J].Infection and Immunity, 2006, 74(2):1323-1338.
[43] TOLEDO-ARANA A, DUSSURGET O, NIKITAS G, et al.The Listeria transcriptional landscape from saprophytism to virulence[J].Nature, 2009, 459(7249):950-956.
[44] PORTMAN J L, DUBENSKY S B, PETERSON B N, et al.Activation of the Listeria monocytogenes virulence program by a reducing environment[J].mBio, 2017, 8(5):e01595-e01517.
[45] ALLEN K J, WA ECKA-ZACHARSKA E, CHEN J C, et al.Listeria monocytogenes:An examination of food chain factors potentially contributing to antimicrobial resistance[J].Food Microbiology, 2016, 54:178-189.
ECKA-ZACHARSKA E, CHEN J C, et al.Listeria monocytogenes:An examination of food chain factors potentially contributing to antimicrobial resistance[J].Food Microbiology, 2016, 54:178-189.
[46] RAKIC MARTINEZ M, WIEDMANN M, FERGUSON M, et al.Assessment of Listeria monocytogenes virulence in the Galleria mellonella insect larvae model[J].PLoS One, 2017, 12(9):e0184557.
[47] YAO H, KANG M Q, WANG Y T, et al.An essential role for hfq involved in biofilm formation and virulence in serotype 4b Listeria monocytogenes[J].Microbiological Research, 2018, 215:148-154.
[48] CHRISTIANSEN J K, LARSEN M H, INGMER H, et al.The RNA-binding protein Hfq of Listeria monocytogenes:Role in stress tolerance and virulence[J].Journal of Bacteriology, 2004, 186(11):3355-3362.
[49] SUBRAMANIAN S, KEARNS D B.Functional regulators of bacterial flagella[J].Annual Review of Microbiology, 2019, 73:225-246.
[50] CHO S Y, NA H W, OH H B, et al.Structural basis of flagellar motility regulation by the MogR repressor and the GmaR antirepressor in Listeria monocytogenes[J].Nucleic Acids Research, 2022, 50(19):11315-11330.