白酒具有悠久的酿造历史,其独特的生产工艺和风味特征使其成为世界蒸馏酒的典型代表,2022年白酒产业实现销售收入6 626.5亿元,在我国具备庞大的消费群体和文化基础[1]。微生物作为白酒酿造的核心资源,在白酒酿造过程中产生多种生物酶和风味物质及其前体物质,决定了白酒风味的丰富性和独特性。白酒酿造微生物组中菌种的种类、丰度、种群间的互作以及代谢的多样性对白酒品质具有重要影响,是白酒香型和风格呈现多样化的主要原因[2-3]。因此,明确功能微生物的分类学地位、来源,维持或提高菌种的优良性能,调控优化酿造微生态环境,是提高白酒发酵品质和稳定性的重要保障。
白酒酿造微生态具备复杂性和独特性,由于培养基质选择不合理、微生物处于存活但难以培养(viable but non-culturable,VBNC)状态、多种微生物间的互作依赖性、部分微生物的低丰度和缓慢生长等原因,导致用传统的培养技术对白酒酿造生境中微生物的可培养性较低,绝大多数酿酒微生物还未能得到分离培养,白酒微生物组具有“黑箱”属性[4],严重阻碍了对白酒微生物组多样性、菌种功能及代谢机制、菌种间互作调控路径的全面认识,极大限制了白酒酿造过程的定向调控和技术创新[5-6]。目前,大曲、酒醅、窖泥等酿造环境中关键核心菌种的功能和微生物间协同互作的方式研究还不透彻,白酒酿造中发挥重要作用的关键特征微生物菌种并未进行全面系统性的梳理分析,白酒酿造微生物菌种还存在资源分散、来源不明、分类地位不准确、功能特性不清晰等问题,亟需对白酒特殊酿造生境中的特征性微生物资源进行深度挖掘和科学鉴定,为破解白酒酿造微生物“黑箱”提供基础材料。本文系统归纳了白酒酿造过程中分离培养的新菌种资源及其功能评价进展,围绕白酒酿造特殊生境,对其中新菌种和未/难培养资源的分离培养策略进行了系统分析,为白酒酿造微生物“暗物质”的挖掘和利用提供技术支撑。
1 白酒中新菌种资源挖掘
白酒是世界六大蒸馏酒之一,其工艺复杂,涉及原料处理、酒曲发酵制备、窖池发酵、蒸馏取酒、老熟陈酿和勾兑调味等过程。与其他蒸馏酒发酵工艺不同,白酒酿造过程涉及多菌种混合发酵,微生物的来源主要包括酿造环境、原料、水、酒曲、窖池,经过长年累月的积累形成了独特的微生物群落,蕴含着丰富的微生物资源[7]。微生物菌种在酿造过程中发挥产生物酶、风味物质、健康因子等多种功能,是白酒独特风味特征形成的核心驱动因素。
1.1 分离自白酒中的新菌种
微生物新种资源的挖掘与后续功能开发,可有效促进白酒酿造微生物功能及代谢机理的深入研究,加强白酒优势酿造微生物资源的有效保护与高效利用,为白酒酿造微生物组的微生态调控提供理论支撑。随着分子生物学技术的不断发展,近21世纪以来通过非培养技术对白酒酿造各个过程中微生物群落结构已经有了较为全面的解析,获得了较多的微生物信息[8]。然而对于一些低丰度或未知物种仍无法了解,需要通过分离培养的方法,对白酒酿造过程中未/难培养微生物及新菌种进行进一步的发掘与研究。
依据原核生物系统学国际委员会(International Committee on Systematics of Prokaryotes,ICSP)的规定,结合原核生物国际命名法规(International Code of Nomenclature of Prokaryotes,ICNP)要求,对照目前分类单元的描述、更新及命名发布在原核生物名称名录(List of Prokaryotic names with Standing in Nomenclature,LPSN)和国际微生物学系统与进化杂志(International Journal of Systematic and Evolutionary Microbiology,IJSEM)中的微生物新种,总结了白酒酿造相关工艺环节或环境中分离得到科学命名的新种资源25种,其中20种新种分离自浓香型白酒。浓香型白酒中的新种主要来自于窖池发酵过程中的窖泥和黄水,由于窖池发酵特殊的厌氧或微氧条件,分离得到的新种以厌氧或兼性厌氧细菌为主。此外,在五粮液酿造车间古窖池的空气中分离到一株酵母新种Moniliella aeria sp.nov.,这也是白酒相关新种中唯一的酵母新种。除浓香型白酒相关新种外,其余的4种新种分别分离自酱香型白酒或芝麻香型白酒的高温大曲中。高温大曲在发酵过程中温度可到达60~70 ℃[9],根据这一特性,在分离时均选择与发酵过程相近的高温(≥45 ℃)进行培育,成功分离得到了具有耐高温特性的新菌种。
从时间上来看,在2005—2006年间,中科院微生物所首次由河北省浓香型白酒厂的窖泥中分离得到4种来源于白酒的新菌种。此后,在2007—2013年间,白酒新菌种资源的挖掘处于空白阶段。2014年开始,每年持续有白酒相关新菌种的发表,在2021年到达爆发阶段,仅一年就发表了新种6种。近年来,白酒企业与高校及科研院所合作密切,不断强化白酒酿造过程中关键微生物资源的挖掘与保护,新菌种资源挖掘和科学命名的主体已由科研院所转移到相关白酒企业,这也与白酒酿造的科技创新水平的提升密切相关。
白酒相关新菌种中,有5种为梭菌属(Clostridium)下新种,梭菌属是窖泥中的优势菌属,同时也是浓香型白酒微生态系统中风味化合物形成的主要微生物之一,梭菌属可将淀粉、蛋白质和嘌呤等物质转化成有机酸(乙酸、丁酸和己酸)、醇类等风味物质[10-11]。互营单胞菌属(Syntrophomonas)也是窖泥中的典型的优势菌属,可与产甲烷菌互作促进脂肪酸降解[12]。乳酸菌在白酒生产过程中分布广泛,在窖泥、大曲和酒醅中均占据优势,如乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)和迟缓乳杆菌属(Lentilactobacillus)[13-14],其代谢产物中乳酸和乙酸都是白酒重要的风味物质。芽孢杆菌属(Bacillus)作为白酒自然发酵过程中的优势菌属,不仅对白酒的风味有着巨大贡献,其次级代谢产物与其他微生物相互作用,维持窖泥生态环境的稳定[12-13]。己小杆菌属(Caproicibacterium)在酒厂中分布广泛,平均丰度为11.4%,是窖泥厌氧发酵过程中优势己酸生产菌[15]。高温大曲中,具有耐高温特性的菌种往往占据优势,如高温放线菌属(Thermoactinomyces)[16-17]。白酒新菌种资源中,除了上述优势菌属外,还存在着一些低丰度的关键菌种,如Novisyntrophococcus fermenticellae[18]可与其他菌种互作产短链脂肪酸;Thermophilibacter immobilis和Blautia liquoris代谢产乙酸;Proteiniphilum propionicum专性利用蛋白质和多肽,主要代谢产物为丙酸与乙酸,在偏中性pH值下生长更为良好,是优质窖泥的特征微生物[19]。具体见表1。
表1 分离自白酒的微生物新种
Table 1 New microbial species isolated from Baijiu
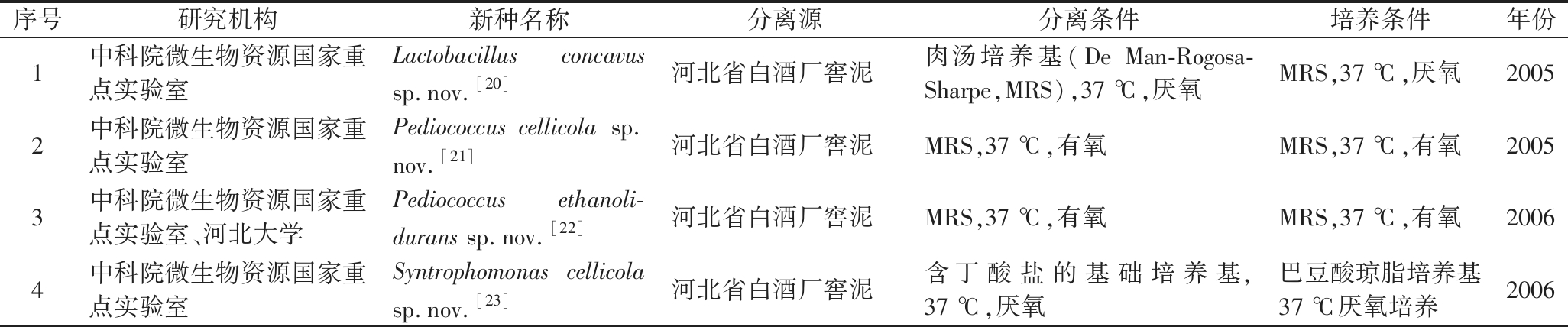
序号研究机构新种名称分离源分离条件培养条件年份1中科院微生物资源国家重点实验室Lactobacillus concavus sp.nov.[20]河北省白酒厂窖泥肉汤培养基(De Man-Rogosa-Sharpe,MRS),37 ℃,厌氧MRS,37 ℃,厌氧20052中科院微生物资源国家重点实验室Pediococcus cellicola sp.nov.[21]河北省白酒厂窖泥MRS,37 ℃,有氧MRS,37 ℃,有氧20053中科院微生物资源国家重点实验室、河北大学Pediococcus ethanoli-durans sp.nov.[22]河北省白酒厂窖泥MRS,37 ℃,有氧MRS,37 ℃,有氧20064中科院微生物资源国家重点实验室Syntrophomonas cellicola sp.nov.[23]河北省白酒厂窖泥含丁酸盐的基础培养基,37 ℃,厌氧巴豆酸琼脂培养基 37 ℃厌氧培养2006
续表1

序号研究机构新种名称分离源分离条件培养条件年份5四川大学、农业部沼气研究所Clostridium swellfunia-num sp.nov.[24]四川泸州水井坊浓香型白酒窖泥乳酸培养基(lactate-medi-um,LM),37 ℃,厌氧孵育,滚管分离LM/葡萄糖培养基(glucose-medium,GM)37 ℃,厌氧培养20146中国食品发酵工业研究院有限公司、山东扳倒井股份有限公司、北京林业大学Thermoactinomyces daqus sp.nov.[25]山东省扳倒井芝麻香型白酒高温大曲Reasoner’s 2A培养基(R2A),55 ℃,有氧酵母麦芽/胰酪大豆胨琼脂培养基(ISP Medium No.2/Trypti-case Soy Broth, ISP 2/TSB),55 ℃,有氧20157山东大学、绵阳师范大学Clostridium luticellarii sp.nov.[26]四川省浓香型白酒窖泥强化梭菌培养基(rein-forced Clostridium medium,RCM),37 ℃,厌氧RCM,37 ℃,厌氧20158中国农业科学院、重庆工商大学Paenibacillus vini sp.nov.[27]四川省泸州老窖窖泥大豆酪蛋白琼脂培养基(tryptone soy agar,TSA), 30 ℃,有氧胰酪大豆胨琼脂培养基( trypticase soy broth,TSB),37 ℃,有氧20159中科院环境与应用微生物重点实验室、四川大学Clostridium liquoris sp.nov.[28]四川省绵竹市浓香型白酒窖泥改良 Chalmers培养基 (modified Chalmers medi-um,MCM),30 ℃,厌氧改良MCM,37 ℃,厌氧201610中国食品发酵工业研究院有限公司、山东扳倒井股份有限公司Scopulibacillus daqui sp.nov.[29]山东省扳倒井芝麻香型白酒高温大曲R2A,50 ℃,厌氧R2A,50 ℃,厌氧201611大连理工大学、大连民族大学Franconibacter daqui sp.nov.[30]贵州省仁怀市茅台大曲葡萄糖牛肉膏蛋白胨培养基(glucose, beef extract and peptone,GBP),45 ℃,有氧GBP,37 ℃,厌氧/有氧201712江南大学、国家固态酿造工程技术研究中心Clostridium fermenticellae sp.nov.[31]浓香型白酒窖泥RCM,37 ℃,厌氧RCM,37 ℃,厌氧201913贵州大学Thermoflavimicrobium daqui sp.nov.[32]贵州省仁怀市茅台风味大曲改良Gao2培养基48 ℃富集,ISP 2分离,有氧ISP 2,45 ℃,有氧201914中国食品发酵工业研究院有限公司、五粮液股份有限公司Bacillus aquiflavi sp.nov.[33]宜宾浓香型白酒黄水TSA,37 ℃,厌氧TSA,37 ℃,厌氧202015北京工商大学Pseudoxanthomonas beig-ongshangi sp.nov.[34]四川省白酒窖泥R2A,30 ℃营养琼脂,30 ℃202116江南大学、国家固态酿造工程技术研究中心Novisyntrophococcus fer-menticellae gen.nov., sp.nov.[35]浓香型白酒窖泥RCM,37 ℃,厌氧RCM,37 ℃,厌氧202117泸州老窖股份有限公司Thermophilibacter immo-bilis sp.nov.[36]泸州老窖窖泥R2A,35 ℃,分离PYG-mod,37 ℃,厌氧202118农业农村部农村可再生能源开发与应用重点实验室、沼气研究所Aminipila luticellarii sp.nov.[37]浓香型白酒窖泥RCM,37 ℃,厌氧RCM,37 ℃,厌氧202119江南大学、国家固态酿造工程技术研究中心Caproicibacterium amylo-lyticum gen.nov., sp.nov.[38]浓香型白酒窖泥GAM,37 ℃,厌氧GAM,37 ℃,厌氧202120泸州老窖股份有限公司、江南大学、国家固态酿造工程技术研究中心Blautia liquoris sp.nov.[39]浓香型白酒窖泥GAM,37 ℃,厌氧GAM,37 ℃,厌氧202121中科院微生物资源国家重点实验室、国家固态酿造工程技术研究中心Lentilactobacillus laojiao-nis sp.nov.[40]四川省泸州老窖窖泥改良MRS,37 ℃,厌氧改良MRS,37 ℃,微需氧202222五粮液股份有限公司Moniliella aeria sp.nov.[41]四川省宜宾市五粮液酿造车间空气PDA,28 ℃PDA,28 ℃202223江南大学、中国轻工业浓香型白酒固态发酵重点实验室Caproicibacterium lactati-fermentans sp.nov.[42]浓香型白酒窖泥改良梭菌生长培养基(mod-ified Clostridia growth medi-um, MCGM),37 ℃,厌氧mCGM,37 ℃,厌氧202224江南大学、中国轻工业浓香型白酒固态发酵重点实验室Proteiniphilum propi-onicum sp.nov.[19]五粮液窖泥改良Chalmers培养基(modified Chalmers,MC),37 ℃,厌氧改良蛋白胨-酵母提取物培养基(modified peptone-yeast extract, mPY),37 ℃,厌氧202225五粮液股份有限公司Clostridium aroma-tivorans sp.nov.[43]五粮液窖泥54b,35 ℃,厌氧RCM,37 ℃,厌氧2023
1.2 白酒新菌种功能挖掘
新菌种资源的挖掘不仅可为解析白酒酿造过程中微生物组成、群落演替规律及菌群互作提供科学支撑,新菌种资源的功能挖掘也为白酒酿造品质提升提供新思路。菌株大曲高温放线菌CICC 10681(Thermoactinomyces daqus H-18),是我国白酒大曲中分离的第一株新菌种,基因和表型分析表明该菌株具备良好的安全性;具备发达的菌丝体,可有效改善大曲质构性状;耐高温,适应大曲高温发酵环境;具备产高温蛋白酶能力,能够提升大曲蛋白酶活力等理化指标,采用该菌株对大曲中微生物组进行了微生态调控,有效提升了白酒大曲品质,为白酒酿造品质提升提供了技术支撑[17, 44]。
从老窖窖泥中分离的新菌种老窖互营球菌JN500902(Novisyntrophococcus fermenticellae)和老窖梭菌JN500901(Clostridium fermenticellae)共培养可显著缩短生长周期,并提高发酵液中浓香型白酒典型风味化合物前体物质丁酸和己酸的含量[18]。进而通过基因组和转录组联合分析,由表型到分子,系统的阐明了这两株菌之间通过代谢物相互作用促进短链脂肪酸产生的互作机制,拓宽了对窖泥微生物间复杂的相互作用的了解[45]。
黄水芽孢杆菌3H-10(Bacillus aquiflavi)是由浓香型白酒黄水中分离得到的新种,也是目前唯一一株分离自黄水的新种。通过对菌株的全基因组序列比较分析和体外功能评价发现,该菌株具有优良的产蛋白酶能力同时具有良好的安全性,在白酒发酵中具有潜在应用价值[46-47],并进一步对该菌株的乙醇耐受机制进行了解析[48],为后续该菌株在白酒实际生产中的应用奠定了理论基础。此外,从五粮液的古窖泥中分离到了新种解乳酸己小杆菌JNU-WLY1368(Caproicibacterium lactatifermentans),该菌种的发现解决了浓香型白酒窖泥中主导功能菌认知不准确的问题,并通过比较基因组学和生理生化特性研究,揭示了该菌种对多碳源、高酸度的白酒酿造环境的生态适应性机制,为提升窖泥微生态质量奠定了重要理论和实践基础[49]。
目前,在25种白酒新菌种资源中,对其功能性、安全性等的系统评价还处于起步阶段,新菌种资源作为功能微生物菌种在白酒酿造中发挥的重要作用还未充分挖掘,新菌种资源在原生态位存在但未培养状态对白酒酿造微生物组的影响还有待深入研究。因此,系统开展新菌种资源的功能开发,建立菌种选育、精准鉴定、功能评价、安全性评价、工艺开发和产业化应用的全生命周期研发技术体系已成为白酒新菌种资源开发的发展趋势。
2 白酒中新菌种资源挖掘策略
由于白酒酿造生境的复杂性,大曲、酒醅、窖泥等酿造环境中微生物间共同协作的方式研究还不透彻[50],培养基质选择不合理等因素[51-52],导致用传统的培养技术对不同生境微生物的可培养性都很低[53],例如土壤可培养的微生物占比约为0.3%,淡水约为0.25%,海水不到0.1%,严重阻碍了对微生物群落多样性的全面认识。
2.1 传统分离培养方法改进
传统平板分离培养技术仍是最普遍且适用的微生物资源挖掘方法,也是白酒中新菌种最主要的分离手段。根据目标菌种的生物学特性,改进培养方法和分离条件,可以有效提升微生物分离培养的成功率和多样性。
白酒酿造是一个复杂的混菌发酵过程,存在许多低丰度或处于活但非可培养(viable but non-culturable,VBNC)状态的菌株。许多研究表明,通过富集培养可显著提高菌株存活率,增加可培养微生物的多样性。例如,白酒相关新菌种Clostridium swellfunianum[42]、Clostridium fermenticellae[31]和Thermophilibacter immobilis[36]在被分离前均经过了1~2周的富集培养。陈园等[54]通过多次富集培养,由窖泥中分离得到1株产氨细菌。郭莹[55]将窖泥接种于培养基中,连续3次富集7 d,成功分离出具有产己酸能力的菌株,并应用于窖泥养护液的制备。
依据微生物生长环境、代谢特性和分离目标的不同,设计特异性培养基、添加生长所必需的特定营养成分或抑制其他菌种生长的抑菌剂,可以有效提高目标微生物的分离效率。例如,ZHAO等[40]在基础MRS培养基中添加10%的酒醅浸出液和2.5%制霉菌素,在更好的还原微生物原本生长条件的同时抑制了真菌的生长,成功从窖泥中分离出了乳酸菌新种Lentilactobacillus laojiaonis。卢萌萌等[56]选择了改良CGM、NBM和MCI三种培养基分别对浓香型白酒窖泥进行富集,结果表明不含葡萄糖并降低氮源的寡营养培养基更有利于窖泥中低丰度微生物的生长,适用于窖泥中未培养微生物的进一步分离。浩楠等[57]在MRS培养基中加入放线菌酮,用于葡萄酒中乳酸菌的分离,成功得到一株具有本地特色的优势乳酸菌。
生长坏境中独特的营养条件决定了微生物的种类和结构。然而,通过传统分离培养技术无法模拟微生物在自然生长环境中原位生长所需的全部营养物质,或培养基营养过剩导致部分微生物生长受限。面对以上挑战,近年来,许多研究者通过膜扩散技术以实现微生物的原位培养,选择合适孔径的通透膜可以使小分子营养物质透过并阻止细胞的通过,同时避免了由于分离微生物代谢产物的积累导致后续微生物生长受限[58]。基于膜扩散的分离芯片法(isolation chip,Ichip)是常用的原位培养方法,该装置可实现高通量操作,已广泛应用于土壤、海洋微生物的分离筛选,近年来在白酒领域也展开了研究。邹云曼等[59]通过Ichip原位培养法和传统培养法对不同贮藏时期的酱香型白酒大曲中微生物进行分离,从5个储存期的酱香型高温大曲样品中共分离纯化出679株细菌,其中原位培养法471株,传统培养法208株。结果表明,与传统培养法相比,原位培养能培养出更多种类的细菌,分离效率是传统培养法的两倍。同时,原位培养也能筛选出高通量测序得出的、难培养的功能微生物。
白酒酿造过程中微生物的自然群落结构及菌株间相互作用关系复杂,许多酿造过程的关键微生物在自然群落中丰度较低,生长速度缓慢,且常规培养条件与自然生长环境差异较大,这些限制因素导致了使用传统分离培养条件无法对酿造过程中许多具有良好代谢及发酵功能的菌株进行分离。针对以上问题,根据目标菌种生长特性的不同,不断优化分离培养条件,通过长时间富集培养、增加基础培养基中特定营养成分、添加抑制特定微生物类群生长的抑菌剂、原位培养或使用寡营养培养基可有效增加白酒酿造过程中未/难培养微生物的分离效率。
2.2 培养组学
培养组学这一概念是在2012年由LAGIER等[60-61]提出,是通过多种培养条件大量分离微生物,并通过基质辅助激光解吸电离飞行时间质谱和16S rRNA测序方法对分离株进行鉴定。旨在从同一样品中尽可能多而全的分离出可培养的微生物。培养组学最早用于肠道中微生物的分离,人们通过测序技术已经对肠道中微生物的群落结构有了较为详尽的了解,然而其中80%以上的细菌尚未被分离培养。LAGIER及其团队[60]共使用212种培养条件,由3份粪便样品中分离鉴定了32 500个单菌落,共340个物种,其中包含了31个潜在新种。培养组学已经在肠道微生物研究方面有了较为广泛的应用,同时也扩展到了包括白酒在内的其他领域。孙乐平[62]根据酒糟和窖泥的扩增子测序结果,通过培养组学的方法,针对其中的细菌和真菌,设计了69种不同培养条件,使用商业化培养基、原始环境模拟培养基和通过KOMODO(known media database)培养基推荐系统检索获得的预测培养基进行培养,共分离鉴定到457株细菌菌株和53株真菌菌株,其中110株细菌菌株被划定为潜在新种。
培养组学的方法可以根据样品中微生物组成的不同,设置满足不同微生物生长需求的培养条件,较为全面的分离样品中的微生物,但同时也存在着通量低、耗时长和工作量大的缺点。随着人工智能和深度学习的不断成熟和普及,HUANG等[63]开发了一个基于机器学习引导机器人自动化实现菌株分离和基因分型的培养组学平台(culturomics by automated microbiome imaging and isolation,CAMII),该平台由4个关键要素组成:1)采集形态学的成像系统及人工智能引导下的菌落选择算法;2)一个自动的挑取菌落的机器人用于高通量分离及排列菌落;3)具有成本效益的流水线快速生成挑选的分离物的基因组数据;4)分离物的实物库和可以搜索到菌株菌落形态、表型和基因型的数据库。HUANG等[63]通过CAMII平台建立了20个健康人群的肠道菌群数据库,总共对102 071个菌落进行图像分析,并挑取了26 997个菌落通过16S rRNA基因测序进行分类鉴定。这一技术证明了菌落形态的定量分析可以进行分类学预测,加强了目标属的分离,并揭示了微生物之间潜在的相互作用,有望改变下一代微生物培养组学。目前,培养组学在白酒微生物领域得到了成功应用。但同时需要注意的是,白酒酿造不同阶段微生物复杂多变,不同样品间微生物组成差异大,需结合高通量测序技术,有针对性的对样品中特定菌属及其相对丰度设计相应的培养基,优化培养条件,并结合人工智能选取代表性菌落,在减小工作量的同时分离得到更多可培养的微生物。
2.3 单细胞分选技术
通过微流控技术和细胞分选技术,在微观尺度下对复杂微生物组中菌种单细胞进行控制和操作,结合荧光原位杂交[64]、荧光激活细胞分选[65]、拉曼光谱细胞检测分选技术[66],可实现对微生物单细胞的靶向分离,获得特异性目标微生物菌种单细胞,结合培养基设计和培养条件优化,所得单细胞可在微液滴等培养基中生长繁殖,减少了目标菌种在原位微生物群落中与其他菌种的竞争和拮抗作用,更利于低丰度、生长缓慢的微生物的培养。微流控和细胞分选技术,可高通量分选大批量的菌种单细胞,显著提高菌种培养效率,减少微生物新菌种资源挖掘的实验周期。单细胞分选技术在肠道微生物、土壤微生物、海洋微生物等领域新菌种挖掘、新功能选育等方向已有多种应用,如ZHOU等[67]利用微流控平板划线技术,从白蚁肠道中分离了8种潜在新菌种。CHEN等[68]采用微流控技术结合趋化性筛选,从土壤中分离出了能降解咪唑啉酮的苍白杆菌属(Ochrobactrum)类群。HU等[69]利用微流控技术结合长时间培养策略,从海洋样本中分离了15种潜在新菌种资源。QIAO等[70]通过基于微流体技术的荧光激活液滴分选技术,实现了白酒大曲样品中微生物的单细胞分选,从中成功分离筛选出9株具有脂肪酶活性的细菌。
微流控技术和细胞分选技术可用于高通量培养多种样品中的微生物且可显著提高分离效率,同时可以克服传统分离方法存在的种间竞争等问题,但由于设备的专业性和复杂性,在应用时也存在一定局限。白酒酿造过程中微生物生长的窖泥、大曲和酒醅等基质成分复杂,这些环境中的非生物成分对分子标记和细胞分选时的流动性存在一定的干扰。此外,白酒酿造过程中以厌氧微生物为主,单细胞分选设备难以在常规厌氧工作站进行安装和操作,需要更大的厌氧环境。目前,单细胞分选技术已用于白酒发酵环境中功能微生物的筛选,但尚未在白酒新菌种资源挖掘中成熟应用,还需进一步探索。
3 总结与展望
随着高通量测序技术的快速发展,微生物组中大量未培养微生物的基因组信息被挖掘,白酒酿造过程的微生物群落组成及演替规律日益清晰,但围绕新菌种和未培养菌种的分离培养方法仍未出现质变,可分离培养的菌种占比不足1%。微生物菌种作为白酒酿造的核心关键资源,通过培养基优化、培养条件改善、培养组学和单细胞分选等技术应用,可有效丰富可培养微生物菌种类群,为开发新型功能菌种资源提供技术支撑,系统挖掘白酒酿造特殊生境中的新菌种资源,并开展新菌种和未/难培养菌种的功能挖掘及产业应用,可有效支撑对白酒酿造过程微生物菌种发挥功能作用的机制解析,有助于揭示微生物在发酵过程中与代谢物质、微生物群落间的互作关系,为白酒酿造的微生态调控、微生物组重构研究提供坚实物质基础。
[1] 宋书玉. 中国酒业迈进“十四五”[J].中国酒, 2021(4):56-59.
SONG S Y.China’s wine industry enters “14th Five-Year Plan”[J].China Wine, 2021(4):56-59.
[2] 王媚, 傅小红, 冯学愚, 等.浓香型白酒发酵微生物多样性研究进展与技术创新[J].酿酒科技, 2014(8):85-88.
WANG M, FU X H, FENG X Y, et al.Research progress in microbial diversity of Nongxiang Baijiu(liquor)and its technical innovation[J].Liquor-Making Science &Technology, 2014(8):85-88.
[3] ZOU W, ZHAO C Q, LUO H B.Diversity and function of microbial community in Chinese strong-flavor Baijiu ecosystem:A review[J].Frontiers in Microbiology, 2018, 9:671.
[4] COLLINS G, KAVANAGH S, MCHUGH S, et al.Accessing the black box of microbial diversity and ecophysiology:Recent advances through polyphasic experiments[J].Journal of Environmental Science and Health.Part A, Toxic/Hazardous Substances &Environmental Engineering, 2006, 41(5):897-922.
[5] 李斌斌, 吴丹妮, 聂国兴, 等.未/难培养微生物可培养策略研究:机遇与挑战[J].微生物学通报, 2023, 50(2):832-844.
LI B B, WU D N, NIE G X, et al.Isolation and culture techniques of uncultured microorganisms:Challenges and opportunities[J].Microbiology China, 2023, 50(2):832-844.
[6] 王镓璇, 郝淑月, 任清.中国传统发酵食品发酵系统中的未培养微生物研究进展[J].食品与发酵工业, 2023, 49(11):306-314.
WANG J X, HAO S Y, REN Q.Advances in the uncultured microorganisms in the fermentation system of traditional Chinese fermented food[J].Food and Fermentation Industries, 2023, 49(11):306-314.
[7] QIAN M C, HUGHES P, CADWALLADER K.Overview of Distilled Spirits[M].ACS Publications, 2019, 1321:125-144.
[8] 胡梓晴, 刘晓艳, 白卫东, 等.高通量测序技术在白酒微生物多样性中的研究进展[J].中国酿造, 2023, 42(5):15-21.
HU Z Q, LIU X Y, BAI W D, et al.Research progress of high-throughput sequencing technology in microbial diversity of Baijiu production[J].China Brewing, 2023, 42(5):15-21.
[9] 王柏文, 吴群, 徐岩, 等.中国白酒酒曲微生物组研究进展及趋势[J].微生物学通报, 2021, 48(5):1737-1746.
WANG B W, WU Q, XU Y, et al.Recent advances and perspectives in study of microbiome in Chinese Jiuqu starter[J].Microbiology China, 2021, 48(5):1737-1746.
[10] 钱玮, 陆震鸣, 柴丽娟, 等.泸型酒酒醅与窖泥中梭菌群落结构、演替和功能差异[J].生物工程学报, 2020, 36(6):1190-1197.
QIAN W, LU Z M, CHAI L J, et al.Differences of the structure, succession and function of Clostridial communities between Jiupei and pit mud during Luzhou-flavour Baijiu fermentation[J].Chinese Journal of Biotechnology, 2020, 36(6):1190-1197.
[11] 何培新, 李芳莉, 郑燕, 等.浓香型白酒窖泥梭菌的分离及其挥发性代谢产物分析[J].中国酿造, 2017, 36(4):45-49.
HE P X, LI F L, ZHENG Y, et al.Isolation and volatile metabolites of Clostridium spp.in pit mud of strong-flavor Baijiu[J].China Brewing, 2017, 36(4):45-49.
[12] 张应刚, 许涛, 郑蕾, 等.窖泥群落结构及功能微生物研究进展[J].微生物学通报, 2021, 48(11):4327-4343.
ZHANG Y G, XU T, ZHENG L, et al.Research progress on community structure and functional microorganisms of pit mud[J].Microbiology China, 2021, 48(11):4327-4343.
[13] 吴树坤, 谢军, 卫春会, 等.四川不同地区浓香型大曲微生物群落结构比较[J].食品科学, 2019, 40(14):144-152.
WU S K, XIE J, WEI C H, et al.Comparison of microbial community structure of starter cultures (Daqu) for Luzhou-flavor liquor in different regions of Sichuan[J].Food Science, 2019, 40(14):144-152.
[14] 赵东, 郑佳, 彭志云, 等.高通量测序技术解析五粮液窖泥原核微生物群落结构[J].食品与发酵工业, 2017, 43(9):1-8.
ZHAO D, ZHENG J, PENG Z Y, et al.Prokaryotic microbial community in Wuliangye pit mud using NextSeq high-throughput sequencing technology[J].Food and Fermentation Industries, 2017, 43(9):1-8.
[15] WANG H L, GU Y, ZHOU W C, et al.Adaptability of a caproate-producing bacterium contributes to its dominance in an anaerobic fermentation system[J].Applied and Environmental Microbiology, 2021, 87(20):e0120321.
[16] 张应刚, 邓宇.基于高通量测序的不同窖龄窖泥微生物结构与多样性分析[J].中国沼气, 2019, 37(3):3-8.
ZHANG Y G, DENG Y.Microbial structure and diversity of cellar mud at different winery cellar age based on high-throughput sequencing[J].China Biogas, 2019, 37(3):3-8.
[17] 刘红强, 于学健, 翟磊, 等.耐高温菌株大曲高温放线菌CICC 10681培养条件的响应面优化[J].食品与发酵工业, 2021, 47(14):123-129.
LIU H Q, YU X J, ZHAI L, et al.Response surface optimization of the culture conditions of thermotolerant Thermoactinomyces daqus CICC 10681[J].Food and Fermentation Industries, 2021, 47(14):123-129.
[18] 孙红, 柴丽娟, 方冠宇, 等.窖泥中梭菌和互营球菌交互作用对生长和短链脂肪酸代谢的影响[J].食品与发酵工业, 2022, 48(15):24-32.
SUN H, CHAI L J, FANG G Y, et al.Effect of co-culture of Clostridium strains and Novisyntrophococcus fermenticellae, isolated from pit mud, on growth and short-chain fatty acid metabolism[J].Food and Fermentation Industries, 2022, 48(15):24-32.
[19] LIU Q J, ZHENG H Z, WANG H L, et al.Proteiniphilum propionicum sp.nov., a novel member of the Phylum Bacteroidota isolated from pit clay used to produce Chinese liquor[J].International Journal of Systematic and Evolutionary Microbiology, 2022, 72(11):10.1099/ijsem.0.005612.
[20] TONG H C, DONG X Z.Lactobacillus concavus sp.nov., isolated from the walls of a distilled spirit fermenting cellar in China[J].International Journal of Systematic and Evolutionary Microbiology, 2005, 55(5):2199-2202.
[21] ZHANG B, TONG H C, DONG X Z.Pediococcus cellicola sp.nov., a novel lactic acid coccus isolated from a distilled-spirit-fermenting cellar[J].International Journal of Systematic and Evolutionary Microbiology, 2005, 55(5):2167-2170.
[22] LIU L M, ZHANG B, TONG H C, et al.Pediococcus ethanolidurans sp.nov., isolated from the walls of a distilled-spirit-fermenting cellar[J].International Journal of Systematic and Evolutionary Microbiology, 2006, 56(Pt 10):2405-2408.
[23] WU C G, LIU X L, DONG X Z.Syntrophomonas cellicola sp.nov., a spore-forming syntrophic bacterium isolated from a distilled-spirit-fermenting cellar, and assignment of Syntrophospora bryantii to Syntrophomonas bryantii comb.nov[J].International Journal of Systematic and Evolutionary Microbiology, 2006, 56(Pt 10):2331-2335.
[24] HUANG Y, WEI Z X, CONG L L, et al.Clostridium prolinivorans sp.nov., a thermophilic bacterium isolated from an anaerobic reactor degrading propionate[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(10):5190-5196.
[25] YAO S, LIU Y, ZHANG M J, et al.Thermoactinomyces daqus sp.nov., a thermophilic bacterium isolated from high-temperature Daqu[J].International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 1):206-210.
[26] WANG Q, WANG C D, LI C H, et al.Clostridium luticellarii sp.nov., isolated from a mud cellar used for producing strong aromatic liquors[J].International Journal of Systematic and Evolutionary Microbiology, 2015, 65(12):4730-4733.
[27] CHEN X R, SHAO C B, WANG Y W, et al.Paenibacillusvini sp.nov., isolated from alcohol fermentation pit mud in Sichuan Province, China[J].Antonie Van Leeuwenhoek, 2015, 107(6):1429-1436.
[28] YIN Q, TAO Y, ZHU X Y, et al.Clostridium liquoris sp.nov., isolated from a fermentation pit used for the production of Chinese strong-flavoured liquor[J].International Journal of Systematic and Evolutionary Microbiology, 2016, 66(2):749-754.
[29] YAO S, ZHAI L, XIN C H, et al.Scopulibacillus daqui sp.nov., a thermophilic bacterium isolated from high temperature Daqu[J].International Journal of Systematic and Evolutionary Microbiology, 2016, 66(11):4723-4728.
[30] GAO Z Q, SU C L, YANG X Y, et al.Franconibacter daqui sp.nov., a facultatively alkaliphilic species isolated from a Daqu sample[J].International Journal of Systematic and Evolutionary Microbiology, 2017, 67(12):4962-4966.
[31] XU P X, CHAI L J, QIU T, et al.Clostridium fermenticellae sp.nov., isolated from the mud in a fermentation cellar for the production of the Chinese liquor, Baijiu[J].International Journal of Systematic and Evolutionary Microbiology, 2019, 69(3):859-865.
[32] LI D N, HUANG W, QIU S Y.Thermoflavimicrobium daqui sp.nov., a thermophilic microbe isolated from Moutai-flavour Daqu[J].International Journal of Systematic and Evolutionary Microbiology, 2019, 69(9):2709-2716.
[33] XIE J Y, CHENG K, ZHAO D, et al.Bacillus aquiflavi sp.nov., isolated from yellow water of strongly flavored Chinese Baijiu[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(5):3406-3412.
[34] SUN Z B, DAI F, YAN Y, et al.Pseudoxanthomonas beigongshangi sp.nov., a novel bacteria with predicted nitrite and nitrate reduce ability isolated from pit mud of Baijiu[J].Antonie Van Leeuwenhoek, 2021, 114(8):1307-1314.
[35] CHAI L J, FANG G Y, XU P X, et al.Novisyntrophococcus fermenticellae gen.nov., sp.nov., isolated from an anaerobic fermentation cellar of Chinese strong-flavour Baijiu[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(9):10.1099/ijsem.0.004991.
[36] LU L F, YANG Y, ZHENG L, et al.Reclassification of Olsenella gallinarum as Thermophilibacter gallinarum comb.nov.and description of Thermophilibacter immobilis sp.nov., isolated from the mud in a fermentation cellar used for the production of Chinese Luzhou-flavour Baijiu[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(12):10.1099/ijsem.0.005192.
[37] WEI Z X, MA S C, CHEN R, et al.Aminipila luticellarii sp.nov., an anaerobic bacterium isolated from the pit mud of strong aromatic Chinese liquor, and emended description of the genus Aminipila[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(10):10.1099/ijsem.0.004710.
[38] GU Y, ZHU X J, LIN F, et al.Caproicibacterium amylolyticum gen.nov., sp.nov., a novel member of the family Oscillospiraceae isolated from pit clay used for making Chinese strong aroma-type liquor[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(4):10.1099/ijsem.0.004789.
[39] LU L F, YANG Y, CHAI L J, et al.Blautia liquoris sp.nov., isolated from the mud in a fermentation cellar used for the production of Chinese strong-flavour liquor[J].International Journal of Systematic and Evolutionary Microbiology, 2021, 71(10):10.1099/ijsem.0.005041.
[40] ZHAO Q W, YANG S P, BAO G H, et al.Lentilactobacillus laojiaonis sp.nov., isolated from the mud in a fermentation cellar for the production of Chinese liquor[J].International Journal of Systematic and Evolutionary Microbiology, 2022, 72(5):10.1099/ijsem.0.005349.
[41] LEI X J, ZHENG J, ZHAO D, et al.Moniliella aeria sp.nov., a novel yeast isolated from the air of a Wuliangye Baijiu-making workshop[J].International Journal of Systematic and Evolutionary Microbiology, 2022, 72(7):005464.https://doi.org/10.1099/ijsem.0.005464.
[42] WANG H L, GU Y, ZHAO D, et al.Caproicibacterium lactatifermentans sp.nov., isolated from pit clay used for the production of Chinese strong aroma-type liquor[J].International Journal of Systematic and Evolutionary Microbiology, 2022, 72(1):10.1099/ijsem.0.005206.
[43] LUO Q C, ZHENG J, ZHAO D, et al.Clostridium aromativorans sp.nov., isolated from pit mud used for producing Wuliangye Baijiu[J].Antonie Van Leeuwenhoek, 2023, 116(7):739-748.
[44] 冯慧军, 翟磊, 于学健, 等.大曲高温放线菌CICC 10681在不同温度下的脂肪酸代谢变化[J].食品与发酵工业, 2018, 44(11):49-54.
FENG H J, ZHAI L, YU X J, et al.Fatty acids metabolism changes of Thermoactinomyces daqus CICC 10681 at different temperatures[J].Food and Fermentation Industries, 2018, 44(11):49-54.
[45] SUN H, CHAI L J, FANG G Y, et al.Metabolite-based mutualistic interaction between two novel clostridial species from pit mud enhances butyrate and caproate production[J].Applied and Environmental Microbiology, 2022, 88(13):e0048422.
[46] 刘红强, 程坤, 于学健, 等.黄水芽胞杆菌Bacillus aquiflavi 3H-10全基因组序列分析及功能探究[J].食品与发酵工业, 2022, 48(15):41-46.
LIU H Q, CHENG K, YU X J, et al.Genome sequence analysis and mining of Bacillus aquiflavi 3H-10[J].Food and Fermentation Industries, 2022, 48(15):41-46.
[47] 程坤, 刘蕊, 孙思佳, 等.基于全基因组测序的黄水芽胞杆菌Bacillus aquiflavi 3H-10安全性评价[J].食品与发酵工业, 2022, 48(17):49-56.
CHENG K, LIU R, SUN S J, et al.Safety evaluation of Bacillus aquiflavi 3H-10 based on whole genome sequencing[J].Food and Fermentation Industries, 2022, 48(17):49-56.
[48] 程坤, 刘蕊, 郑佳, 等.黄水芽胞杆菌抗逆性潜力挖掘及乙醇耐受机制分析[J].食品与发酵工业, 2022, 48(11):24-29.
CHENG K, LIU R, ZHENG J, et al.Stress resistance potential mining of Bacillus aquiflavi 3H-10 and the mechanism of its ethanol tolerance[J].Food and Fermentation Industries, 2022, 48(11):24-29.
[49] 筱鹂. 江南大学与五粮液微生物新种科技成果获国际领先评价[J].酿酒科技, 2023(1):60.
XIAO L.Jiangnan University’s research achievements on wuliangye liquor-making microbes have reached international leading level[J].Liquor-Making Science &Technology, 2023(1):60.
[50] 周玥, 刘小锦, 朱晨光, 等.细菌中群体感应调节系统[J].微生物学报, 2004, 44(1):122-126.
ZHOU Y, LIU X J, ZHU C G, et al.Quorum sensing in bacteria[J].Acta Microbiologica Sinica, 2004, 44(1):122-126.
[51] 周楠, 姜成英, 刘双江.从环境中分离培养微生物:培养基营养水平至关重要[J].微生物学通报, 2016, 43(5):1075-1081.
ZHOU N, JIANG C Y, LIU S J.Cultivation of microorganisms from environments:Nutrient level of the culture medium is of great importance[J].Microbiology China, 2016, 43(5):1075-1081.
[52] 郭斌, 吴晓磊, 钱易.提高微生物可培养性的方法和措施[J].微生物学报, 2006, 46(3):508-511.
GUO B, WU X L, QIAN Y.Approaches for increasing the culturability of microorganisms[J].Acta Microbiologica Sinica, 2006, 46(3):508-511.
[53] KAEBERLEIN T, LEWIS K, EPSTEIN S S.Isolating “uncultivable” microorganisms in pure culture in a simulated natural environment[J].Science, 2002, 296(5570):1127-1129.
[54] 陈园, 王超凯, 李觅, 等.窖泥产氨菌的分离筛选与发酵培养基的优化[J].食品与发酵科技, 2023, 59(2):43-49.
CHEN Y, WANG C K, LI M, et al.Isolation and screening of ammonia producing bacteria from pit mud and optimization of fermentation medium[J].Food and Fermentation Science &Technology, 2023, 59(2):43-49.
[55] 郭莹. 窖泥主要功能菌分离及窖泥养护液制备研究[D].郑州:河南工业大学, 2021.
GUO Y.Study on Separation of main functional bacteria from pit mud and preparation of curing liquid of pit mud[D].Zhengzhou:Henan University of Technology, 2021.
[56] 卢萌萌, 任聪, 聂尧, 等.白酒酿造窖泥未培养微生物菌群的可培养化策略[J].食品与发酵工业, 2020, 46(19):9-16.
LU M M, REN C, NIE Y, et al.Cultivation strategy for unculturable microbiota in pit mud involved in strong-flavor Baijiu fermentation[J].Food and Fermentation Industries, 2020, 46(19):9-16.
[57] 浩楠, 马腾臻, 贠建民, 等.甘肃河西走廊产区野生乳酸菌筛选及酿酒特性研究[J].食品与发酵工业, 2019, 45(10):142-148.
HAO N, MA T Z, YUN J M, et al.Isolation and brewing characteristics of wild-type lactic acid bacteria from Gansu Hexi Corridor[J].Food and Fermentation Industries, 2019, 45(10):142-148.
[58] LEWIS W H, TAHON G, GEESINK P, et al.Innovations to culturing the uncultured microbial majority[J].Nature Reviews.Microbiology, 2021, 19(4):225-240.
[59] 邹云曼, 邱树毅, 郑佳, 等.基于原位培养和传统培养分析比较不同储存期酱香大曲的细菌群落[J].食品与发酵工业, 2023, 49(15):38-46.
ZOU Y M, QIU S Y, ZHENG J, et al.Comparative analysis of bacterial community of high temperature Daqu with different storage periods based on in situ cultivation and traditional cultivation[J].Food and Fermentation Industries, 2023, 49(15):38-46.
[60] LAGIER J C, ARMOUGOM F, MILLION M, et al.Microbial culturomics:Paradigm shift in the human gut microbiome study[J].Clinical Microbiology and Infection:The Official Publication of the European Society of Clinical Microbiology and Infectious Diseases, 2012, 18(12):1185-1193.
[61] LAGIER J C, HUGON P, KHELAIFIA S, et al.The rebirth of culture in microbiology through the example of culturomics to study human gut microbiota[J].Clinical Microbiology Reviews, 2015, 28(1):237-264.
[62] 孙乐平. 扩增子测序与培养组学联用分析白酒酒糟和窖泥中微生物的多样性[D].北京:北京工商大学, 2020.
SUN L P.Culturing the Baijiu microorganism from fermentation vinasse and pit mud with culturomics and amplicon-based metagenomic approaches[D].Beijing:Beijing Technology and Business University, 2020.
[63] HUANG Y M, SHETH R U, ZHAO S J, et al.High-throughput microbial culturomics using automation and machine learning[J].Nature Biotechnology, 2023, 41(10):1424-1433.
[64] WAGNER M, HAIDER S.New trends in fluorescence in situ hybridization for identification and functional analyses of microbes[J].Current Opinion in Biotechnology, 2012, 23(1):96-102.
[65] HAROON M F, SKENNERTON C T, STEEN J A, et al.In-solution fluorescence in situ hybridization and fluorescence-activated cell sorting for single cell and population genome recovery[J].Methods in Enzymology, 2013, 531:3-19.
[66] LAU A Y, LEE L P, CHAN J W.An integrated optofluidic platform for Raman-activated cell sorting[J].Lab on a Chip, 2008, 8(7):1116-1120.
[67] ZHOU N, SUN Y T, CHEN D W, et al.Harnessing microfluidic streak plate technique to investigate the gut microbiome of Reticulitermes chinensis[J].Microbiology Open, 2019, 8(3):e00654.
[68] CHEN D W, LIU S J, DU W B.Chemotactic screening of imidazolinone-degrading bacteria by microfluidic SlipChip[J].Journal of Hazardous Materials, 2019, 366:512-519.
[69] HU B Y, XU B X, YUN J L, et al.High-throughput single-cell cultivation reveals the underexplored rare biosphere in deep-sea sediments along the Southwest Indian Ridge[J].Lab on a Chip, 2020, 20(2):363-372.
[70] QIAO Y X, ZHAO X Y, ZHU J, et al.Fluorescence-activated droplet sorting of lipolytic microorganisms using a compact optical system[J].Lab on a Chip, 2017, 18(1):190-196.