甲醛是一种在常温下具有刺激性气味的无色气体,当处在高浓度甲醛环境时会增加急性中毒的风险,长时间接触低剂量的甲醛则可能导致慢性中毒甚至癌症[1]。海产品中的甲醛主要分为非法添加以延长保质期的外源性甲醛和海产品自身分解氧化三甲胺(trimetlylamine oxide,TMAO)后产生的内源性甲醛[2]。我国目前禁止以任何形式将甲醛加入食品中,因此海产品的甲醛含量主要与内源性甲醛有关[3]。内源性甲醛可通过非生物途径和生物途径(包括酶途径和微生物途径)产生[4]。非生物途径指TMAO在高温条件下分解产生甲醛[5],而生物途径的必要条件是酶[主要是氧化三甲胺脱甲基酶(trimethylamine N-oxide demethylase,TMAOase)]或微生物的作用[6]。对于低温贮藏的海产品而言,绝大多数微生物的生长受到抑制。由于TMAOase能被特定的辅因子(如半胱氨酸和Fe2+)激活,酶途径成为低温贮藏海产品内源性甲醛生成的最主要来源,因此通过抑制TMAOase的活性降低海产品内源性甲醛生成是延长其保鲜期的有效措施。
TMAOase是一种广泛存在于海产鱼类内脏组织或微生物(如嗜冷杆菌属、红杆菌属、玫瑰变色菌属、玫瑰杆菌属和雷辛格氏菌属等)中的酶,在冷冻条件下仍能保持活性[7]。植物多酚化合物作为一种天然活性成分,能与甲醛发生反应并抑制TMAOase的活性,近年来被广泛应用于海产品的保鲜中。G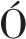 MEZ-ESTACA等[8]观察到红茶多酚提取物在冷冻储存期间可以抑制切碎鳕鱼肉中TMAOase的部分活性,并且不会影响鳕鱼的蛋白功能和凝胶化。LI等[9]发现儿茶素对于从鱿鱼肝脏中纯化得到的TMAOase的抑制作用为非竞争性抑制,且二者间的相互作用主要为疏水相互作用和氢键。然而上述研究主要基于抑制动力学及光谱学分析方法解释多酚与TMAOase之间的相互作用,并未涉及两者之间的关键结合位点和构效关系,无法清晰揭示多酚对TMAOase的抑制作用机制。
MEZ-ESTACA等[8]观察到红茶多酚提取物在冷冻储存期间可以抑制切碎鳕鱼肉中TMAOase的部分活性,并且不会影响鳕鱼的蛋白功能和凝胶化。LI等[9]发现儿茶素对于从鱿鱼肝脏中纯化得到的TMAOase的抑制作用为非竞争性抑制,且二者间的相互作用主要为疏水相互作用和氢键。然而上述研究主要基于抑制动力学及光谱学分析方法解释多酚与TMAOase之间的相互作用,并未涉及两者之间的关键结合位点和构效关系,无法清晰揭示多酚对TMAOase的抑制作用机制。
分子模拟技术近年来被广泛用于多酚与酶相互作用的研究[10-12],为解释两者间相互作用的活性位点及作用类型提供了重要参考。有研究表明不同聚合度的多酚对于酶的抑制效果不同,多酚的聚合度与其酶抑制能力呈正相关关系[13-14]。笔者前期通过搜寻UniProt数据库(https://www.uniprot.org/)发现目前鉴定的TMAOase基因编码序列主要为微生物来源,其中嗜冷杆菌属所表达的TMAOase在低温贮藏海产品内源性甲醛的生成中起重要作用,因此本研究拟采用多序列同源建模的方法构建嗜冷杆菌属来源TMAOase的三维结构,通过分子对接及分子动力学模拟等分子模拟技术探究不同聚合度多酚(原花青素单体儿茶素和表儿茶素、二聚体原花青素B2及三聚体原花青素C1)与TMAOase之间的非共价相互作用。分析其作用位点及主要作用力,从中筛选出与TMAOase相互作用最强的化合物,并进一步验证TMAO存在时,多酚对TMAOase的抑制机制,为后续通过实验阐述其抑制机理提供理论支撑。
1 材料与方法
1.1 受体蛋白的准备工作
从UniProt数据库中获取TMAOase的氨基酸序列信息(UniProtID:A0A6M4P6B0_9GAMM)后,提交至trRosetta服务器进行多模板同源建模[15]。通过SAVES网站(http://services.mbi.ucla.edu/SAVES/)中的PROCHECK和ERRAT程序对构建好的模型质量进行评估[16-17]。
1.2 配体小分子的准备工作
儿茶素(PubChem CID:73160)、表儿茶素(PubChem CID:72276)、原花青素B2(PubChem CID:122738)、原花青素C1(PubChem CID:169853)和TMAO(PubChem CID:1145)的3D结构从PubChem数据库(https://pubchem.ncbi.nlm.nih.gov/)下载,并使用具有MM2力场的Chem 3D软件进行配体的能量最小化。
1.3 分子对接
采用AutoDock Vina 1.2.0[18]软件进行盲分子对接,以探索TMAOase和不同配体小分子之间的相互作用。使用AutoDockTools1.5.7工具为受体蛋白和配体添加极性氢原子和Gastieger电荷。由于蛋白质的活性口袋未知,将搜索框大小包含整个蛋白质以获得二者结合的最佳构象。蛋白质搜索框的网格坐标X、Y和Z中心分别为28.823、16.038和-27.145,搜索框x、y和z方向的尺寸分别为60.639、75.389和70.473,其他优化参数设置为默认值。整个对接过程中,保持蛋白质结构刚性不变,采用半柔性对接,并释放配体小分子所有的扭转键。基于AutoDock Vina中预定义的评分函数,获得具有最低结合亲和力(kcal/mol)的对接姿势,通过PyMOL 2.4.0和Ligplot 2.2.5软件可视化并分析具有最低结合能的最佳对接姿势[19-20]。
1.4 分子动力学模拟
以分子对接得到的最佳对接构象为初始结构,使用GROMACS 2021.4[21]软件包中的Amber99SB-ildn力场和显式溶剂化条件进行模拟。将单独的TMAOase或者蛋白-配体复合物放置在充满TIP3P水分子溶剂的立方体模拟盒中,模拟盒边缘与蛋白质表面之间的最小距离设置为1 nm,并通过添加相反电荷的Na+或Cl-中和体系电荷。采用最陡下降法进行能量最小化,共50 000步,最大值设置为1 000.0 kJ/mol/nm,以消除不良接触和空间碰撞。通过0.1 ns的正则系综平衡和等温等压系综平衡使系统温度达到300 K、压力达到0.1 MPa,并在此条件下进行100 ns的分子动力学模拟。模拟结束后分析轨迹并计算均方根偏差(root mean square deviation,RMSD)、均方根波动(root mean square fluctuation,RMSF)、氢键、旋转半径(radius of gyration,Rg)、溶剂可及表面积(solvent accessible surface area,SASA);使用DSSP[22]程序分析蛋白质的二级结构含量变化;提取最后10 ns的稳定轨迹并使用g_mmpbsa[23]软件通过分子力学-泊松玻尔兹曼表面积(molecular mechanics-Poisson Bolzmann surface area,MM-PBSA)方法计算蛋白与配体之间的结合自由能。
1.5 数据统计分析
实验结果以平均值±标准差表示,分子模拟结果采用Origin 2019b软件绘图,采用SPSS 26软件中的Duncan检验进行显著性分析,P<0.05表示差异显著。
2 结果与分析
2.1 TMAOase三维模型合理性的评估
trRosetta服务器通过HHsearch法检索PDB70数据库中与TMAOase序列一致性程度后得到较高的5个模板(PDB ID分别为4PAB_A,4P9S_A,3GSI_A,1PJ6_A和I9 J5_A)后,进行多模板同源建模得到最终的三维模型(图1-a),TM-score得分为0.927,表明该模型的可信度较高。
a-TMAOase的同源模型;b-模型Ramachandran图;c-模型ERRAT图
图1 TMAOase的同源模型及其评估
Fig.1 Homologous model of TMAOase and its evaluation
作为一种蛋白质结构验证的程序,PROCHECK通过绘制Ramachandran plot判断蛋白结构的合理性。Ramachandran plot通常分为3个区域,黄色区域为最佳允许区,浅黄色是区域为最大允许区,而其他区域(白色区)为三维构象中可能存在的不合理氨基酸残基[24]。如图1-b所示,TMAOase中87.8%和12.0%的氨基酸处于最佳允许区和最大允许区中,只有0.3%的氨基酸位于不合理的区域,因此蛋白质的结构符合立体化学能规则,本研究所构建的结构模型合理。
对于ERRAT分析来说,整体质量因子的值越高,模型的准确性就越高[17]。同源建模得到的TMAOase三维模型的ERRAT值为89.726%(图1-c),说明结构预测产生的模型的准确性良好。而10~20号残基附近的氨基酸残基为环区,其具有较高的柔韧性,导致误差值高于两条误差线。此外,其他氨基酸残基的误差值均低于误差线,表明该模型的精度较高,进一步证实了预测模型的高合理性。基于上述结果,采用trRosetta服务器构建的TMAOase三维结构可作为后续分子对接和分子动力学模拟研究的受体蛋白。
2.2 不同聚合度多酚与TMAOase之间的分子对接研究
分子对接能有效预测受体蛋白和配体之间非共价结合的结合位点和作用方式[25]。图2为TMAOase与多酚的最佳对接构象及二维相互作用,4种多酚化合物均能进入TMAOase的活性口袋被TMAOase的氨基酸残基所包围,形成稳定的复合物。根据图2对TMAOase与4种多酚抑制剂的亲和力和相互作用残基进行汇总得到电子版增强出版附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.036354),从而揭示不同多酚化合物与受体蛋白的关键结合位点。
表1 四种抑制剂对TMAOase二级结构百分含量的影响
Table 1 Effects of four inhibitors on the secondary structure of TMAOase
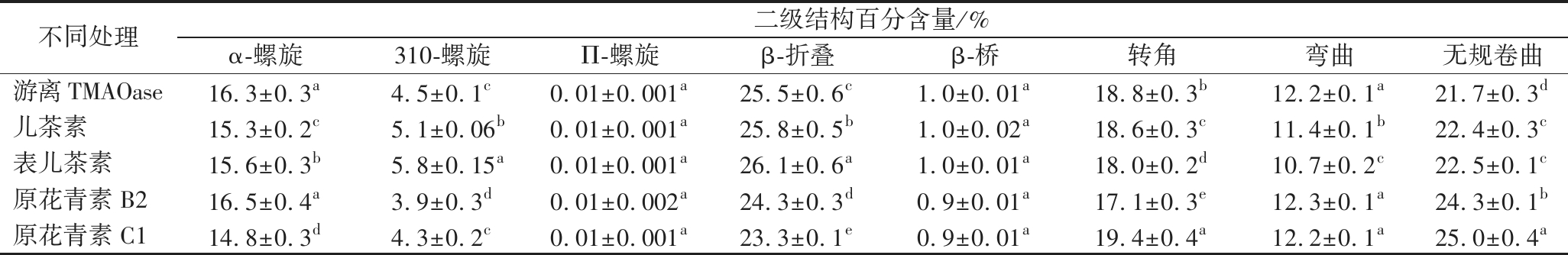
不同处理二级结构百分含量/%α-螺旋310-螺旋Π-螺旋β-折叠β-桥转角弯曲无规卷曲游离TMAOase16.3±0.3a4.5±0.1c0.01±0.001a25.5±0.6c1.0±0.01a18.8±0.3b12.2±0.1a21.7±0.3d儿茶素15.3±0.2c5.1±0.06b0.01±0.001a25.8±0.5b1.0±0.02a18.6±0.3c11.4±0.1b22.4±0.3c表儿茶素15.6±0.3b5.8±0.15a0.01±0.001a26.1±0.6a1.0±0.01a18.0±0.2d10.7±0.2c22.5±0.1c原花青素B216.5±0.4a3.9±0.3d0.01±0.002a24.3±0.3d0.9±0.01a17.1±0.3e12.3±0.1a24.3±0.1b原花青素C114.8±0.3d4.3±0.2c0.01±0.001a23.3±0.1e0.9±0.01a19.4±0.4a12.2±0.1a25.0±0.4a
注:具有相同字母标定的数据不存在显著性(P>0.05)(下同)。
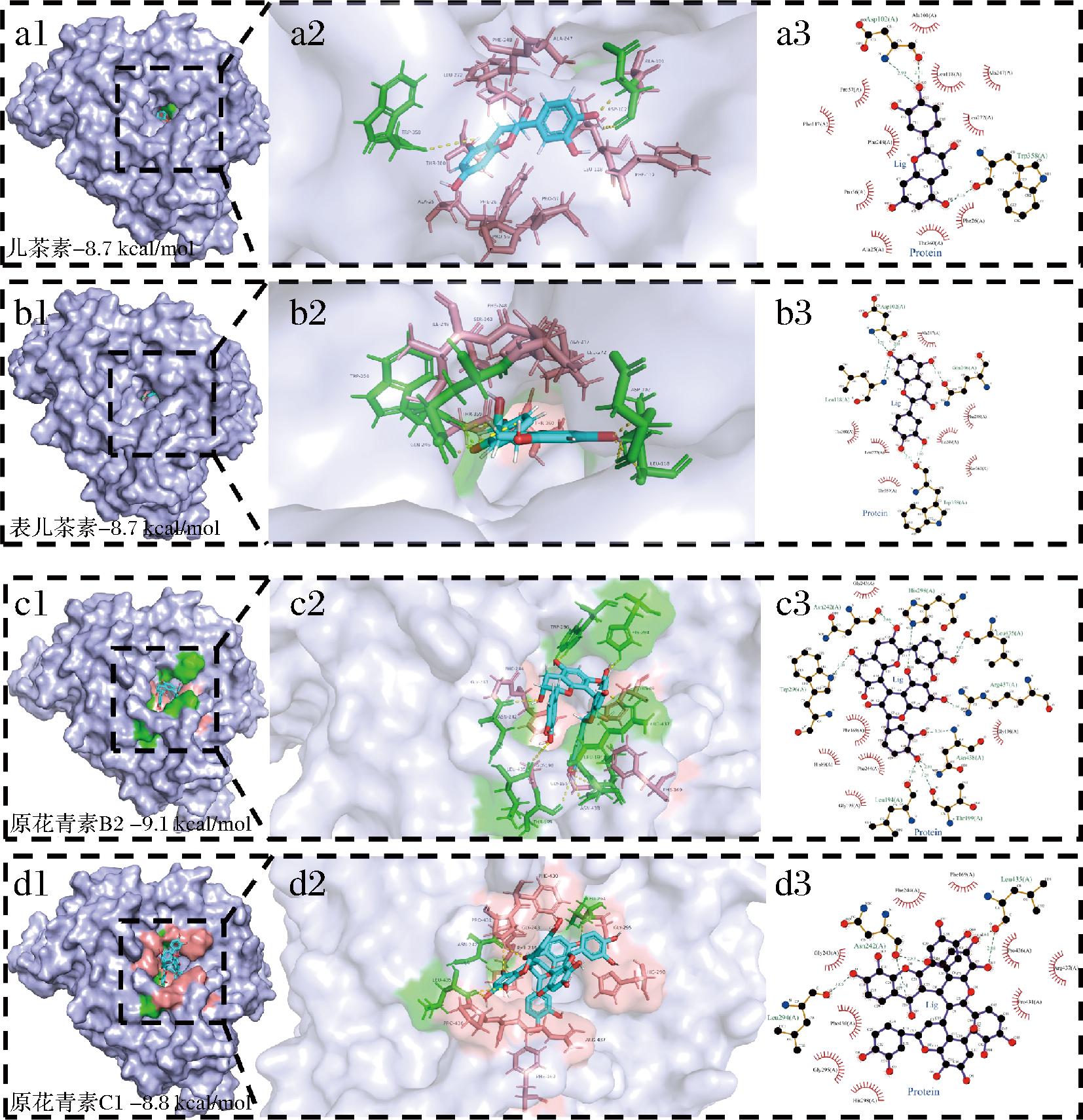
a-TMAOase-儿茶素体系;b-TMAOase-表儿茶素体系;c-TMAOase-原花青素B2体系;d-TMAOase-原花青素C1体系
图2 TMAOase与4种抑制剂配体的对接姿势和相互作用
Fig.2 Docking posture and interaction of TMAOase with four inhibitor ligands
注:数字1-3分别代表TMAOase与抑制剂配体之间的结合位点、结合构象和相互作用图。
使用Ligplot软件可视化分析TMAOase与4种抑制剂配体之间的相互作用(主要为疏水相互作用和氢键),如图2-a3、图2-b3所示,儿茶素与TMAOase内Ala-25、Phe-26、Pro-56、Pro-57、Ala-101、Phe-117、Leu-118、Ala-247、Phe-248、Leu-272和The-360发生强烈的疏水相互作用,并与Asp-102形成2条氢键,与Trp-358形成1条氢键。而表儿茶素则在TMAOase的活性口袋内与Ala-247、Phe-248、Leu-249、Leu-272、Thr-359、The-360和Ser-363形成疏水相互作用;与Asp-102形成3条氢键,与Trp-358形成2条氢键,与Gln-246形成1条氢键。此外,从图2-a1、图2-b1和电子版增强出版附表1可以看出,儿茶素与表儿茶素的结合能相同,均为-8.7 kcal/mol,且二者在TMAOase空腔内的结合位点相似,均与Ala-247、Phe-248、Leu-272和The-360产生疏水相互作用,同时和Asp-102和Trp-358形成氢键。但因结构差异,形成的氢键数目及参与疏水作用的氨基酸残基略有不同。对比儿茶素与表儿茶素的结构,推测产生上述现象的原因可能是二者相互为同分异构体,这与刘红艳等[26]分析漆酶与甲酚同分异构体(邻甲酚、间甲酚、对甲酚)之间相互作用得到的结果相似。
与儿茶素和表儿茶素几乎全部埋藏在TMAOase的内部(图2-a1、图2-b1)相比,原花青素B2和原花青素C1并没有完全进入蛋白内部(图2-c1、图2-d1),但因其聚合度更高,可为TMAOase提供更多的氢键结合位点和疏水相互作用,表现为TMAOase与2种高聚合度多酚的结合强度更高(图2-c3、图2-d3)。其中原花青素B2被TMAOase的7个疏水性氨基酸(His-89、Phe-169、Gly-195、Gly-198、Gly-243、Phe-244)包围并发生疏水相互作用;能和Thr-199和Asn-438均形成2条氢键,与Asn-242、Thr-296、His-298、Leu-435和Arg-437形成1条氢键。而原花青素C1与Phe-169、Leu-194、Gly-243、Gly-295、His-298、Phe-430、Pro-431、Pro-436和Arg-437产成疏水相互作用;并和Asn-242、Leu-435均形成2条氢键,和Leu-294形成1条氢键。对比二者与TMAOase之间的结合能发现,聚合度适中的原花青素B2(-9.1 kcal/mol)与酶的结合能强于高聚合度的原花青素C1(-8.8 kcal/mol),且明显高于单体儿茶素或表儿茶素(-8.7 kcal/mol)。进一步分析高聚合度多酚与酶的结合位点(电子版增强出版附表1)发现,原花青素B2和C1均被疏水性的Phe-169、Gly-243包围并形成疏水相互作用,同时均与Asn-242和Leu-435形成氢键。因此,上述现象表明原花青素B2和C1的结合位点相似,但原花青素B2对于TMAOase的结合能力优于C1,推测可能是因为高聚合度的原花青素C1体积相对较大,无法全部进入TMAOase的结合口袋。基于分子对接的结果,4种多酚抑制剂均可通过疏水相互作用和氢键作用与TMAOase结合,引起竞争性/非竞争性抑制,阻碍其催化TMAO。而配体的结合能力受到聚合度的影响,二聚体原花青素B2对TMAOase的结合能力最强。
2.3 不同聚合度多酚与TMAOase之间的分子动力学模拟研究
分子动力学模拟不仅可以预测蛋白结构、蛋白折叠以及蛋白动力学和热力学的性质,还能考察配体与蛋白的结合/解离过程,了解受体蛋白与配体分子之间的结构变化,为蛋白和配体之间相互作用的研究提供丰富的动态信息[27]。因此,本研究以分子对接得到的最佳构象作为起始位置,对单独的TMAOase和蛋白-多酚复合物进行100 ns分子动力学模拟,以探讨TMAOase与多酚之间的相互作用及稳定性程度,同时研究多酚结合前后对TMAOase结构的影响。
2.3.1 RMSD
RMSD通常用于分析蛋白质等生物分子在模拟体系下的结构稳定性和运动变化,RMSD值的波动越小,其整体结构越稳定[28]。当RMSD值的波动小于0.1 nm时,可认为模拟系统达到平衡状态[29]。如图3-a所示,游离蛋白体系和蛋白-多酚抑制剂体系中蛋白的RMSD在70 ns左右均基本达到平衡并围绕平均值上下波动,表明所有模拟体系在模拟过程中达到平衡,结构相对稳定。消除蛋白质的平动转动轨迹后,计算配体的原子位置相对于蛋白的RMSD以检查配体结合蛋白质的紧密程度(图3-b)。结果表明,所有抑制剂配体的RMSD值在前20 ns内发生显著波动,70 ns后收敛平衡并与TMAOase的结合较为紧密,这被载入运动轨迹所证实。因此,分子动力学模拟结果可靠度高,能用于进行后续的处理分析。

a-4种抑制剂对TMAOase中RMSD的影响;b-4种抑制剂配体相对TMAOase位置的距离;c-4种抑制剂对TMAOase时间平均位置的RMSF;d-4种抑制剂对TMAOase中Rg的影响
图3 游离TMAOase和TMAOase-抑制剂配体的稳定性分析
Fig.3 Stability analysis of native TMAOase and TMAOase-inhibitor ligand
以无抑制剂结合的TMAOase体系为参考,对比4种不同聚合度多酚对酶空间构象变化的影响(图3-a)。蛋白质的RMSD值通常与配体的抑制能力成负相关[30]。较高聚合度的原花青素B2和C1降低了TMAOase的RMSD值,在一定程度上稳定了TMAOase的结构,使其紧密度增加,抑制酶与催化底物的结合。而低聚合度的儿茶素和表儿茶素对于TMAOase的RMSD在模拟后期(95~100 ns)与游离状态时蛋白的RMSD相似,与蛋白的结合对于蛋白构象变化的影响并不明显。
2.3.2 RMSF
RMSF可用于监测特定氨基酸残基的波动程度和整个蛋白质分子的柔韧性变化[31]。如图3-c所示,由于多酚的结合,TMAOase中37、65、298、361和388号氨基酸残基(红框)附近的RMSF显著降低,这与ZHAN等[32]发现辣椒素的结合降低了β-乳球蛋白的RMSF从而限制波动的现象一致。可能归因于多酚与上述位点残基之间的相互排斥和疏水相互作用,以及与蛋白受体的亲水残基形成氢键,最终导致局部残基的结构发生变化[33]。而这些残基可能是多酚与TMAOase结合的关键作用位点,且位于分子对接所显示的结合口袋附近,证实了分子对接结果的可靠性。此外,与天然状态下的TMAOase相比,抑制剂体系在氨基酸编号为1、26、180、340和440(黑框)附近的RMSF均发生不同程度的升高,通过观察序列后发现这些氨基酸残基大部分是环的一部分,其余为同源建模的模板链头部及尾部,推测抑制剂可能会引起环区不同幅度的运动[34]。
2.3.3 Rg
Rg代表蛋白质结构的整体紧密性,Rg值越小,则蛋白质结构的紧密度越好,系统越稳定[35]。由图3-d可看出,与游离的TMAOase相比,儿茶素和表儿茶素略微增加了TMAOase的Rg值,表明当聚合度低的抑制剂小分子结合到蛋白内部时,蛋白质紧密性降低,其结构转变为松散状态,这可能是因为将聚合度低的小分子插入TMAOase的内部时将导致蛋白质向外扩增,使其体积膨胀,结构松散[29]。而分子对接的结合位点(图2a-1和图2a-b1)和动力学模拟的轨迹中也证实了这一结果。
三聚体原花青素C1与TMAOase的Rg值在模拟后期(90~100 ns)几乎重叠,高聚合度配体的结合区域几乎没有影响到TMAOase的结构。但二聚体在95 ns后略微降低了TMAOase的Rg值,使其平均值从2.27 nm降低至2.24 nm,表明二聚体原花青素B2的结合使得蛋白表面更紧固,将蛋白质压得更紧,这与ZHANG等[36]发现姜黄素的存在使肌球蛋白紧密度增加的现象相似。
2.3.4 氢键
分子对接的结果显示氢键在维持蛋白与4种不同多酚之间的非共价相互作用发挥了重要作用。为进一步了解4种蛋白-多酚体系之间的稳定性差异,采用gromacs计算蛋白-配体之间潜在的氢键数量。如电子版增强出版附图1(https://doi.org/10.13995/j.cnki.11-1802/ts.036354)所示,氢键对稳定蛋白质结合口袋内抑制剂配体的结合做出了一定贡献,单体和二聚体与TMAOase之间相互作用的平均氢键数量分别为3.2和3.4,三聚体的平均氢键数量为4.5,表明分子对接所形成的氢键并不稳定,在模拟条件下可能会发生断裂。而我们推测三聚体与TMAOase形成更多氢键可能是因为聚合度的增加使其与TMAOase之间发生相互作用的酚羟基数量增加,从而形成了更多的氢键。
此外,分子内氢键在蛋白质折叠中起着核心作用,可以维持蛋白质表面原子和溶剂分子之间的相互作用[34]。如如电子版增强出版附图1-b所示,游离TMAOase中氢键的总体平均值为322.63,当抑制剂配体结合到TMAOase后,儿茶素和表儿茶素使TMAOase内部的氢键数量分别降低至320.23和320.54,原花青素B2使TMAOase的氢键降低至316.90左右,而原花青素C1并未显著改变氢键数量,上述现象表明多酚可通过破坏TMAOase的氢键来降低其活性。
2.3.5 SASA
SASA可评估氨基酸残基在于溶剂中的暴露程度,疏水区埋在结构紧凑的分子内部,随着SASA值的增加而暴露出来[37]。如电子增强出版附图2(https://doi.org/10.13995/j.cnki.11-1802/ts.036354)所示,在多酚的存在条件下,TMAOase的总SASA值在模拟前70 ns内不断下降,可能是由于随着疏水作用的增强和溶剂水分子的大量排出,多酚和TMAOase之间的结合逐渐稳定[34]。每个体系的SASA在70 ns后围绕各自的平均值上下波动保持相对恒定,表明在模拟后期,游离蛋白和蛋白-多酚复合物的轨迹达到平衡,结构相对稳定,这与Rg和RMSD的结果相似,验证了动力学模拟结果的可靠性。
如电子版增强出版附图2(https://doi.org/10.13995/j.cnki.11-1802/ts.036354)进一步发现抑制剂均不同程度增加了蛋白质疏水表面积和亲水表面积,从而略微增加了TMAOase的总SASA。这与NI等[38]关于牡荆素抑制α-葡萄糖苷酶活性的结果一致,而SHI等[39]证明三萜皂苷(人参皂苷和甘草酸)的结合对牛α-乳清蛋白的疏水和亲水性SASA也具有类似的作用。
2.3.6 二级结构
二级结构是评价蛋白质稳定性的重要指标,以游离的TMAOase为对比,计算不同多酚结合对TMAOase二级结构含量的影响。如电子版增强出版附图3为100 ns模拟过程中游离蛋白和蛋白配体复合物的二级结构含量变化,可以观察到主要二级结构(α-螺旋、β-折叠、无规卷曲和转角)的含量几乎恒定,表明在模拟过程中蛋白质体系的结构稳定。
进一步分析模拟过程中TMAOase二级结构的平均含量(表1)发现,由于小分子的结合,除β-桥和Π-螺旋外,其余二级结构均发生微小变化,α-螺旋、310-螺旋、弯曲和转角的含量变化均在1%上下浮动,而无规卷曲的含量增加和β-折叠的含量下降较为明显,表明二者可能发生相互转化。而二级结构含量的微小变化表明多酚诱导的构象变化仅限于结合位点附近,并不涉及蛋白质折叠的明显变化[36]。相比其他多酚化合物,原花青素B2对TMAOase二级结构含量变化的影响较为显著。
2.3.7 结合能
为评估不同聚合度多酚与TMAOase的结合能力及结合中的主要作用能,采用MM-PBSA方法计算抑制剂与TMAOase的结合能。如表2所示,4种蛋白-多酚复合物总结合自由能均为负值,表明4种多酚均可自发地与蛋白质形成稳定的复合物[40],这与分子对接结果一致(图2)。
表2 四种抑制剂与TMAOase的结合能量 单位:kJ/mol
Table 2 Binding energy of four inhibitors and TMAOase

配体结合总能量范德华能静电作用能极性溶剂化能非极性溶剂化能儿茶素-83.928±11.623a-111.220±16.985a-111.228±16.985b115.725±11.960c-15.722±11.960a表儿茶素-83.950±9.626a-146.738±9.913b-61.390±7.426a140.040±10.015b-15.862±0.625a原花青素B2-106.120±5.062b-206.480±4.811c-112.415±4.706b235.785±6.599a-23.010±6.599a原花青素C1-82.879±6.289a-190.081±6.181c-113.618±14.163b244.638±8.569a-23.818±1.409a
从结合能来看,4种多酚与TMAOase之间的结合能从强到弱依次为原花青素B2[(-106.120±5.062) kJ/mol]、表儿茶素[(-83.950±9.626) kJ/mol]、儿茶素[(-83.928±11.623) kJ/mol]、和原花青素C1[(-82.879±16.289) kJ/mol,表明原花青素B2的结合能力最强。将结合自由能进一步分解为范德华能、静电作用能、极性溶剂化能和非极性溶剂化能(通常将SASA用作非极性溶剂化能),如表2所示,多酚与酶之间的范德华能、静电作用能和非极性溶剂化能均为负值,意味着这些能量有利于结合过程的自发进行,其中范德华力的贡献最大,静电作用次之,具有较低能量的SASA表明疏水作用并不明显。而极性溶剂化能阻碍了复合物的形成,这可能是因为溶剂对静电相互作用具有强烈的屏蔽作用[39]。根据此部分结果,范德华力和静电作用力是促进多酚和TMAOase结合的主要作用力,其中原花青素B2的结合作用最强。
2.4 验证模拟
如电子版增强出版附图4(https://doi.org/10.13995/j.cnki.11-1802/ts.036354)所示,TMAO(催化底物)可进入TMAOase的空腔形成稳定复合物(-3.0 kcal/mol),且TMAO最有利的结合构象被7个疏水性氨基酸残基(Gly-236、Thr-240、Ile-249、His-250、Ser-381、Gly-382和Ile-384)包围,同时Arg-383与其结构中的氮原子形成1条氢键。进一步以分子对接得到的TMAO与原花青素B2的结合位置为起始构象(图4绿色),进行100 ns的分子动力学模拟,验证当有/无TMAO存在时,原花青素B2对TMAOase的抑制效果。
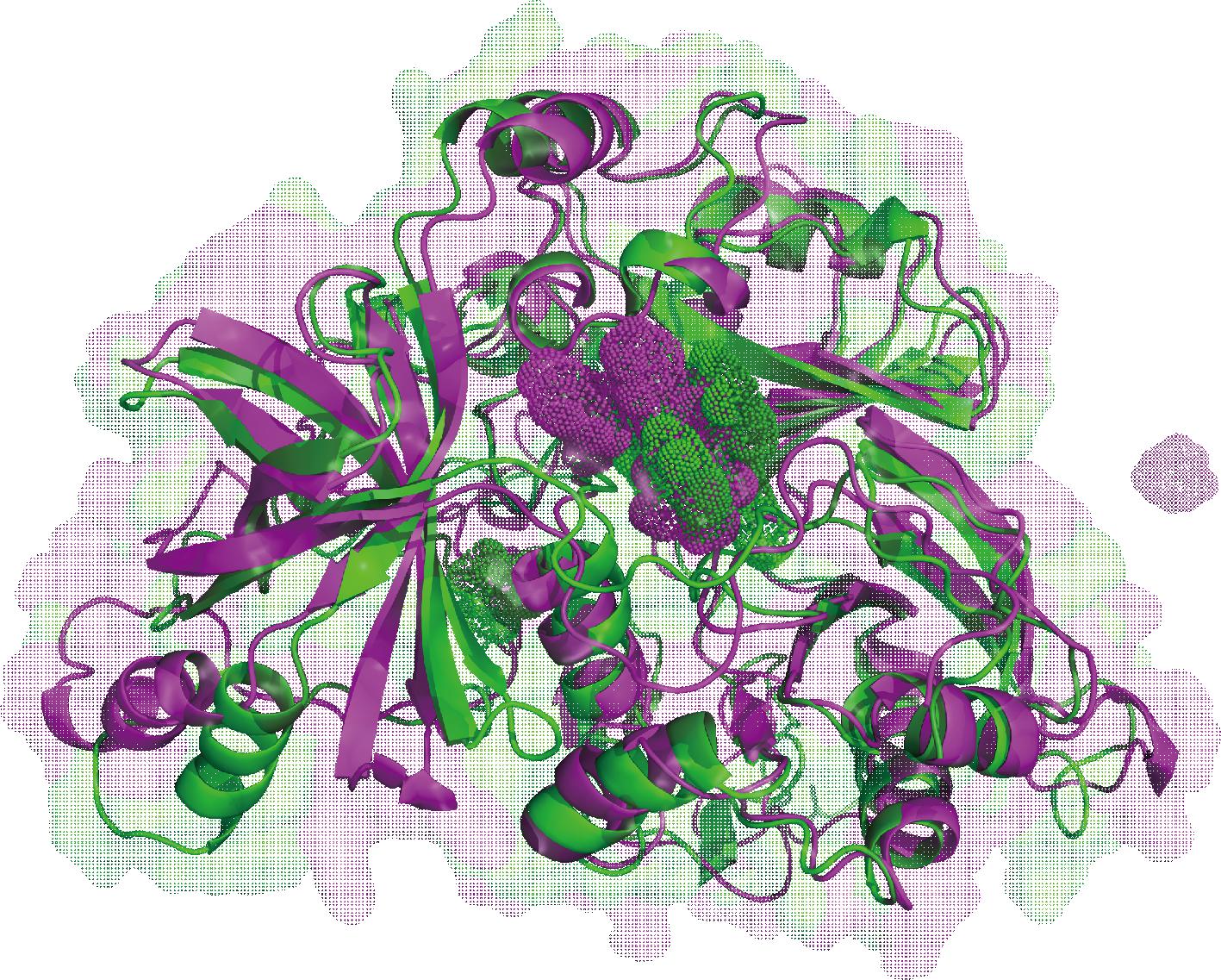
图4 模拟结束后TMAOase-TMAO-原花青素B2的结构变化(点状态为配体)
Fig.4 Structural changes of TMAOase-TMAO-Procyanidin B2 after simulation (dot state is ligand)
图4为100 ns模拟前后整个体系的构象变化,原花青素B2在100 ns后依旧与TMAOase结合稳定,而催化底物从蛋白内部脱离,依据二者的结合位置初步推测原花青素B2对TMAOase的抑制作用为非竞争性抑制(图4紫色),仅蛋白活性位点的结构发生变化,后续结合RMSD等指标进一步分析其抑制机制。
2.4.1 RMSD
图5-a显示了在100 ns模拟过程中,酶、底物和抑制剂同时存在时TMAOase的RMSD值波动。模拟时间约50 ns后,体系的RMSD值达到平衡,围绕平均值0.35 nm上下波动,表明体系变得平衡且稳定,分子动力学模拟的结果合理。当原花青素B2存在时,TMAOase的RMSD值低于游离状态和催化状态,可能因为TMAOase的催化活性位点受到抑制剂的影响发生构象变化,使蛋白质自由度降低,稳定了蛋白质结构。

a-抑制剂对TMAOase-TMAO中蛋白的RMSD影响;b-两种配体相对于TMAOase位置的距离;c-两种配体对TMAOase中Rg的影响;d-抑制剂对TMAOase-TMAO中蛋白分子内部氢键数量的影响
图5 抑制剂对TMAOase-TMAO体系的影响
Fig.5 Effect of Inhibitors on TMAOase-TMAO System
进一步计算抑制剂和底物的原子位置相对于蛋白的RMSD值并结合Visual Molecular Dynamics(VMD)软件可视化轨迹以检查不同配体结合蛋白质的紧密程度(图5-b)。结果表明,原花青素B2存在时催化底物与酶的结合极其不稳定,模拟时间约32 ns后开始脱离蛋白活性中心,但仍在蛋白内部,随着模拟时间的延长,50 ns后彻底脱离蛋白在模拟盒内移动,而多酚在TMAOase内部的结合非常稳定,RMSD值在0.45 nm上下小范围波动。上述现象表明原花青素B2的抑制作用为非竞争性抑制,当结合抑制剂后,TMAOase的活性催化位点构象发生改变,阻止催化底物和蛋白的相互识别,最终抑制TMAOase的催化活性。
2.4.2 Rg
在前50 ns的模拟内,抑制剂使酶的Rg值降低(图5-c)。可能是因为其活性位点受到抑制剂的非竞争抑制作用被压缩使结构更加致密,导致催化底物TMAO无法结合TMAOase的活性位点,进而从蛋白内部脱离。当催化底物脱离蛋白后,蛋白的Rg略有波动,逐渐变得稳定,与RMSD分析的结果一致。
2.4.3 氢键分析
如图5-d所示,前50 ns内3种体系(游离TMAOase、TMAOase-TMAO和TMAOase-TMAO-原花青素B2)中TMAOase的内部氢键平均数量为315、321和314。表明催化底物可通过增加TMAOase内部的氢键数量稳定蛋白结构从而进行催化反应,而原花青素B2可以破坏催化底物所增加的氢键数量甚至略微降低TMAO的内部氢键数量来抑制其催化活性。
3 结论
本研究通过分子对接及分子动力学等分子模拟技术探究了4种不同聚合度的多酚对TMAOase的抑制作用机制。PROCHECK和ERRAT的评估表明同源建模得到的蛋白三维结构合理,可用于分子对接和分子动力学模拟。分子对接结果显示4种抑制剂均进入TMAOase的疏水空腔内且稳定结合,其中氢键和疏水相互作用力是维持复合物稳定性的主要结合力。与三聚体原花青素C1和互为同分异构体的单体表儿茶素或儿茶素相比,二聚体原花青素B2对于TMAOase的结合能力最强,其主要与酶的疏水性氨基酸His-89、Phe-169、Gly-195、Gly-198、Gly-243和Phe-244产生疏水相互作用以及和Thr-199、Asn-438形成氢键。
分子动力学模拟表明,4种多酚的结合使得蛋白质的结构和动力学参数发生了微小的变化。其中,表儿茶素和儿茶素对于TMAOase结构的影响相似,均使得蛋白质体积膨胀,结构松散,而原花青素B2和C1的结合可使蛋白质的构象更加致密,表面更紧固。4种多酚均不同程度增加了TMAOase的表面疏水性同时降低其分子内氢键数量,但多酚诱导的蛋白质构象变化仅限于其结合位点附近,并不涉及二级结构的显著变化。原花青素B2对TMAOase的结合能力强于儿茶素、表儿茶素及原花青素C1,同时范德华力也参与了多酚与TMAOase之间的结合且为主要作用力。最终的验证模拟表明原花青素B2的抑制作用为非竞争性抑制,不仅可通过降低TMAOase的体积使催化口袋变得致密,导致催化底物TMAO无法在疏水空腔稳定存在,还能通过降低蛋白分子内部氢键的数量来抑制其催化活性。综上所述,原花青素B2是一种极具前景的TMAOase抑制剂,未来有望被开发为降低海产品内源性甲醛生成的新型生物保鲜剂。
[1] VER SSIMO M I S, GAMELAS J A F, FERNANDES A J S, et al.A new formaldehyde optical sensor:Detecting milk adulteration[J].Food Chemistry, 2020, 318:126461.
SSIMO M I S, GAMELAS J A F, FERNANDES A J S, et al.A new formaldehyde optical sensor:Detecting milk adulteration[J].Food Chemistry, 2020, 318:126461.
[2] JINADASA B K K K, ELLIOTT C, JAYASINGHE G D T M.A review of the presence of formaldehyde in fish and seafood[J].Food Control, 2022, 136:108882.
[3] 毛伟峰, 赵天琪, 隋海霞, 等.我国8省份市售海产品及其制品中甲醛含量的调查分析[J].中国食品卫生杂志, 2015, 27(1):61-64.
MAO W F, ZHAO T Q, SUI H X, et al.Investigation of formaldehyde content of seafood and its products from 8 provinces in China[J].Chinese Journal of Food Hygiene, 2015, 27(1):61-64.
[4] 励建荣, 朱军莉.食品中内源性甲醛的研究进展[J].中国食品学报, 2011, 11(9):247-257.
LI J R, ZHU J L.Research advance in endogenous formaldehyde in foods[J].Journal of Chinese Institute of Food Science and Technology, 2011, 11(9):247-257.
[5] 李颖畅, 李双燕, 杜凤霞, 等.二氢杨梅素对煎烤鱿鱼片贮藏过程中氧化三甲胺降解及品质的影响[J].食品科学, 2021, 42(7):247-252.
LI Y C, LI S Y, DU F X, et al.Effect of dihydromyricetin on trimethylamine oxide degradation and quality attributes in grilled squid fillets during storage[J].Food Science, 2021, 42(7):247-252.
[6] SUMMERS G, WIBISONO R D, HEDDERLEY D I, et al.Trimethylamine oxide content and spoilage potential of New Zealand commercial fish species[J].New Zealand Journal of Marine and Freshwater Research, 2017, 51(3):393-405.
[7] 刘雪飞, 马吾霞, 李颖畅, 等.芳樟醇抑制嗜冷杆菌引起的秘鲁鱿鱼甲醛升高[J].中国食品学报, 2021, 21(12):214-221.
LIU X F, MA W X, LI Y C, et al.Inhibitory effects of linalool on the increase in formaldehyde of Dosidicus gigas caused by Psychrobacter sp[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(12):214-221.
[8] G MEZ-ESTACA J, G
MEZ-ESTACA J, G MEZ-GUILLEN M C, MAR
MEZ-GUILLEN M C, MAR N-PE
N-PE ALVER D, et al.Functional aptitude of Hake minces with added TMAO-demethylase inhibitors during frozen storage[J].Food Chemistry, 2020, 309:125683.
ALVER D, et al.Functional aptitude of Hake minces with added TMAO-demethylase inhibitors during frozen storage[J].Food Chemistry, 2020, 309:125683.
[9] LI Y C, SONG S Z, LI Y Y, et al.Novel insights into the inhibitory mechanism of (+)-catechin against trimethylamine-N-oxide demethylase[J].Food Chemistry, 2022, 373:131559.
[10] WU M, LIU M, WANG F Y, et al.The inhibition mechanism of polyphenols from Phyllanthus emblica Linn.fruit on acetylcholinesterase:A interaction, kinetic, spectroscopic, and molecular simulation study[J].Food Research International, 2022, 158:111497.
[11] DAI T T, LI T, HE X H, et al.Analysis of inhibitory interaction between epigallocatechin gallate and alpha-glucosidase:A spectroscopy and molecular simulation study[J].Spectrochimica Acta.Part A, Molecular and Biomolecular Spectroscopy, 2020, 230:118023.
[12] MARADESHA T, PATIL S M, AL-MUTAIRI K A, et al.Inhibitory effect of polyphenols from the whole green jackfruit flour against α-glucosidase, α-amylase, aldose reductase and glycation at multiple stages and their interaction:Inhibition kinetics and molecular simulations[J].Molecules, 2022, 27(6):1888.
[13] ZHONG H R, XUE Y, LU X Y, et al.The effects of different degrees of procyanidin polymerization on the nutrient absorption and digestive enzyme activity in mice[J].Molecules, 2018, 23(11):2916.
[14] BUCHHOLZ T, MELZIG M F.Polyphenolic compounds as pancreatic lipase inhibitors[J].Planta Medica, 2015, 81(10):771-783.
[15] DU Z Y, SU H, WANG W K, et al.The trRosetta server for fast and accurate protein structure prediction[J].Nature Protocols, 2021, 16(12):5634-5651.
[16] LASKOWSKI R A, MACARTHUR M W, MOSS D S, et al.PROCHECK:A program to check the stereochemical quality of protein structures[J].Journal of Applied Crystallography, 1993, 26(2):283-291.
[17] COLOVOS C, YEATES T O.Verification of protein structures:Patterns of nonbonded atomic interactions[J].Protein Science:a Publication of the Protein Society, 1993, 2(9):1511-1519.
[18] EBERHARDT J, SANTOS-MARTINS D, TILLACK A F, et al.AutoDock vina 1.2.0:New docking methods, expanded force field, and python bindings[J].Journal of Chemical Information and Modeling, 2021, 61(8):3891-3898.
[19] SEELIGER D, DE GROOT B L.Ligand docking and binding site analysis with PyMOL and Autodock/Vina[J].Journal of Computer-Aided Molecular Design, 2010, 24(5):417-422.
[20] LASKOWSKI R A, SWINDELLS M B.LigPlot+:Multiple ligand-protein interaction diagrams for drug discovery[J].Journal of Chemical Information and Modeling, 2011, 51(10):2778-2786.
[21] ABRAHAM M J, MURTOLA T, SCHULZ R, et al.GROMACS:High performance molecular simulations through multi-level parallelism from laptops to supercomputers[J].SoftwareX, 2015, 1-2:19-25.
[22] TOUW W G, BAAKMAN C, BLACK J, et al.A series of PDB-related databanks for everyday needs[J].Nucleic Acids Research, 2015, 43(Database issue):D364-D368.
[23] KUMARI R, KUMAR R, CONSORTIUM O S D D, et al.G_mmpbsa:A GROMACS tool for high-throughput MM-PBSA calculations[J].Journal of Chemical Information and Modeling, 2014, 54(7):1951-1962.
[24] FRIESNER R A, BANKS J L, MURPHY R B, et al.Glide:A new approach for rapid, accurate docking and scoring.1.Method and assessment of docking accuracy[J].Journal of Medicinal Chemistry, 2004, 47(7):1739-1749.
[25] 杨婷婷, 杨婧, 袁子又, 等.抗菌肽BCp12保鲜膜在乳扇贮藏保鲜中的应用[J].食品科学, 2023, 44(1):189-198.
YANG T T, YANG J, YUAN Z Y, et al.Application of edible films containing antimicrobial peptide BCp12 in preservation of milk fan[J].Food Science, 2023, 44(1):189-198.
[26] 刘红艳, 唐琳, 谢世伟, 等.基于分子模拟和光谱法分析漆酶与甲酚的相互作用[J].分析测试学报, 2020, 39(3):323-329.
LIU H Y, TANG L, XIE S W, et al.Study on interaction between laccase and cresols based on molecular docking and spectral analysis[J].Journal of Instrumental Analysis, 2020, 39(3):323-329.
[27] HU X, ZENG Z, ZHANG J, et al.Molecular dynamics simulation of the interaction of food proteins with small molecules[J].Food Chemistry, 2023, 405(Pt A):134824.
[28] NIE H N, DONG H, CHEN Y L, et al.Effects of spray drying and freeze drying on the structure and emulsifying properties of yam soluble protein:A study by experiment and molecular dynamics simulation[J].Food Chemistry, 2023, 409:135238.
[29] LU Y C, ZHAO R, WANG C, et al.Deciphering the non-covalent binding patterns of three whey proteins with rosmarinic acid by multi-spectroscopic, molecular docking and molecular dynamics simulation approaches[J].Food Hydrocolloids, 2022, 132:107895.
[30] CARDOSO W B, MENDANHA S A.Molecular dynamics simulation of docking structures of SARS-CoV-2 main protease and HIV protease inhibitors[J].Journal of Molecular Structure, 2021, 1225:129143.
[31] HABIBIAN-DEHKORDI S, FARHADIAN S, GHASEMI M, et al.Insight into the binding behavior, structure, and thermal stability properties of β-lactoglobulin/Amoxicillin complex in a neutral environment[J].Food Hydrocolloids, 2022, 133:107830.
[32] ZHAN F C, DING S, XIE W Y, et al.Towards understanding the interaction of β-lactoglobulin with capsaicin:Multi-spectroscopic, thermodynamic, molecular docking and molecular dynamics simulation approaches[J].Food Hydrocolloids, 2020, 105:105767.
[33] CEN C N, CHEN J, WANG W Q, et al.Exploring the interaction mechanism of dietary protein ovalbumin and folic acid:A combination research of molecular simulation technology and multispectroscopy[J].Food Chemistry, 2022, 385:132536.
[34] ZHOU H L, BIE S K, LI J, et al.Comparison on inhibitory effect and mechanism of inhibitors on sPPO and mPPO purified from ‘Lijiang snow’ peach by combining multispectroscopic analysis, molecular docking and molecular dynamics simulation[J].Food Chemistry, 2023, 400:134048.
[35] WANG B L, KOU S B, LIN Z Y, et al.Investigation on the binding behavior between BSA and lenvatinib with the help of various spectroscopic and in silico methods[J].Journal of Molecular Structure, 2020, 1204:127521.
[36] ZHANG L, WANG P, YANG Z Y, et al.Molecular dynamics simulation exploration of the interaction between curcumin and myosin combined with the results of spectroscopy techniques[J].Food Hydrocolloids, 2020, 101:105455.
[37] ZHANG X G, LU Y C, ZHAO R, et al.Study on simultaneous binding of resveratrol and curcumin to β-lactoglobulin:Multi-spectroscopic, molecular docking and molecular dynamics simulation approaches[J].Food Hydrocolloids, 2022, 124:107331.
[38] NI M T, HU X, GONG D M, et al.Inhibitory mechanism of vitexin on α-glucosidase and its synergy with acarbose[J].Food Hydrocolloids, 2020, 105:105824.
[39] SHI R J, CHEN W, PAN F, et al.Characterization of the binding behavior, structure and foaming properties of bovine α-lactalbumin combined with saponin by the multi-spectroscopic and silico approaches[J].Food Hydrocolloids, 2022, 124:107259.
[40] WANG H T, ZHU J M, ZHANG H W, et al.Understanding interactions among aldehyde compounds and porcine myofibrillar proteins by spectroscopy and molecular dynamics simulations[J].Journal of Molecular Liquids, 2022, 349:118190.