枯草芽孢杆菌(Bacillus subtilis)是一种革兰氏阳性非致病菌,被美国食品和药品管理局认定为GRAS(Generally Recognized As Safe)菌株,它可以直接向培养基中分泌蛋白质,而且具有成熟的遗传操作工具,这使得枯草芽孢杆菌在工业、医疗和食品等领域具有广泛的应用。然而,B.subtilis表达系统中各元件之间的适配性及对不同蛋白分泌的影响尚不清晰[1]。
根据启动子转录开关的特性,启动子分为诱导型启动子、自诱导型启动子和组成型启动子[2]。常用的诱导型启动子有T7(由β-D-硫代半乳吡喃糖苷诱导),PmtlA(由甘露醇诱导),Pxyl(木糖诱导),PSacB(蔗糖诱导)等[3-6]。近年来,自诱导型启动子受到越来越多的关注,并越来越多地应用于工业生产,例如Pcry3A和PmanP[7-8]。此外,组成型启动子P43、Plaps和Pshuttle09等由于无需额外添加诱导剂在工业界广受欢迎[9-10]。
P43来源于B.subtilis,是甘油脱氨酶的启动子,是目前B.subtilis中较强的组成型启动子之一。SONG等[11]发现,与PHpaⅡ相比,P43可使支链淀粉酶的活力提高1倍左右,达8.7 U/mL。P43被用于生产异甲基对硫磷水解酶,开发基于食品级表达质粒的枯草芽孢杆菌表达系统[12-13]。
核糖体结合位点(ribosomebinding site,RBS)又称为Shine Dalgarno(SD)序列,是位于起始密码子AUG上游一段富含嘌呤的DAN序列。有研究表明,SD序列与起始密码子两者之间的间隔长度对转录和翻译的效率起着重要作用[14]。研究表明,利用“RBS-Calculator”软件可以预测获得许多的RBS,该方法合成的RBS可有效调控蛋白表达[15-16]。MAO等[17]发现,通过对RBS的优化,在B.subtilis中异源表达二乙酰基化二糖脱乙酰酶的胞外酶活力相较优化前提升了30%,达4 807.6 U/mL。
迄今为止,已发现的枯草芽孢杆菌蛋白质分泌途径包括一般蛋白质分泌途径(Sec途径)、双精氨酸分泌途径(Tat途径)和ABC转运子途径。大约90%的胞外蛋白质是通过Sec和Tat途径分泌的。信号肽是这些蛋白质经由这些途径运输所必需的元件。为了提高胞外蛋白的产量,研究人员越来越重视研究发现更多更好用的信号肽。例如,DEGERING等[18]构建了一个信号肽库,其中包括来自枯草芽孢杆菌的173个预测信号肽和来自地衣芽孢杆菌的220个预测信号肽。ZHANG等[19]研究比较了6种信号肽对碱性聚半乳糖醛酸裂合酶(polygalacturonate lyase, PGL)分泌的影响,确定最适合信号肽bpr后,通过分批补料发酵使碱性PGL活力达到632.6 U/mL。ZHU等[20]以青霉素G酰基转移酶为指示基因,分析了24条Tat途径中的信号肽,对筛选出的信号肽SymzC做进一步研究,最大酶活力达到0.26 U/mL。SNYDER等[21]发现信号肽SphoD引导的绿色荧光蛋白可以主动输出到细胞外环境中,而无需经典的Tat途径分泌。ZHANG等[12],在P43启动子的控制下,SnprB作为信号肽,在分子伴侣PrsA的促进下,脂氧合酶的产量在上清液中达到76 U/mL。
大多数研究人员致力于在单因子水平上优化表达元件,忽略了元件之间的相互作用。因此,本文构建了12个质粒,包含不同启动子(P43、Pshuttle09)、核糖体结合位点(RBS1、RBS5)和信号肽(SnprB、SphoD、SymzC),以研究各个元件之间的适配性,并筛选高效分泌表达盒(图1)。此外,在优化表达元件的基础上,本文以壳聚糖酶Csn21c作为目标蛋白,进一步从5种不同培养基中筛选了最适发酵培养基,大幅提高了壳聚糖酶Csn21c的分泌水平,为该壳聚糖酶Csn21c的生产应用奠定基础。
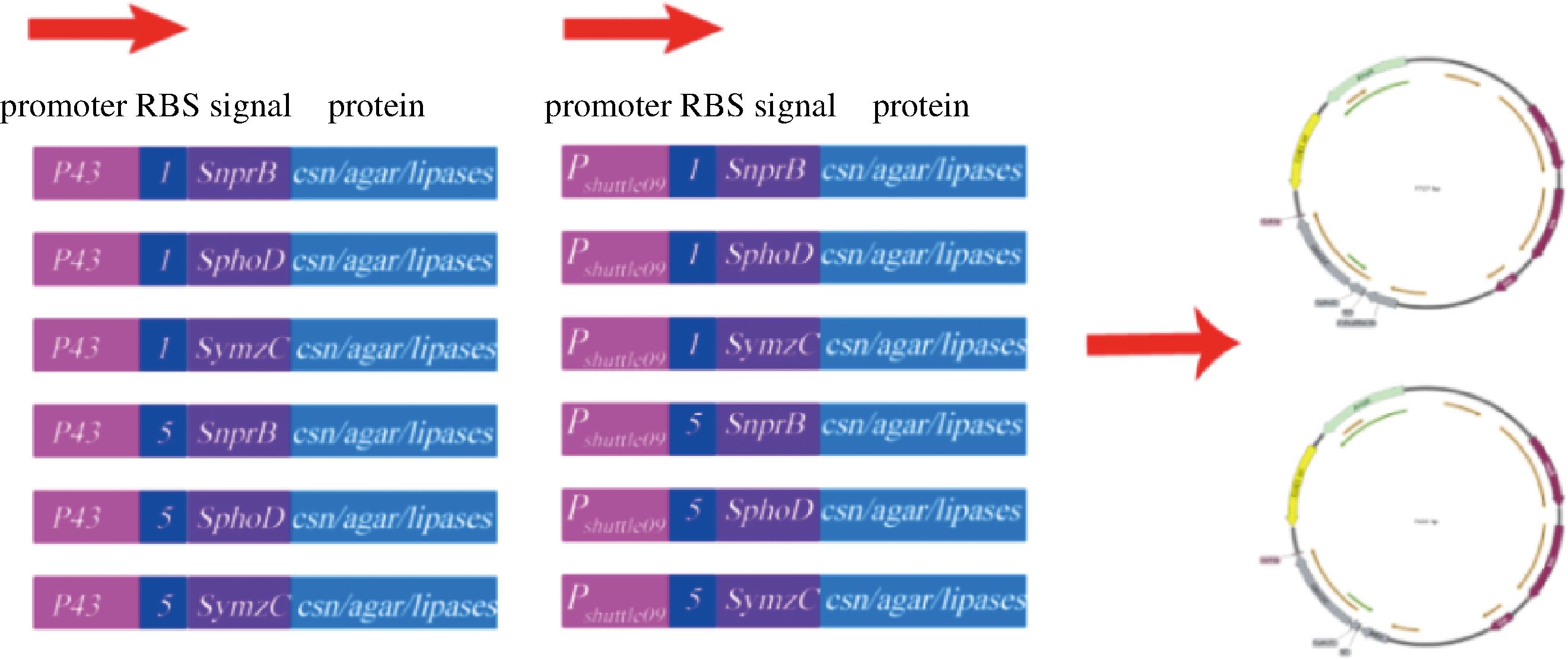
图1 包含不同启动子(P43、Pshuttle09)、核糖体结合位点(RBS1、RBS5)和信号肽(SnprB, SphoD, SymzC)的12个表达盒组合
Fig.1 Twelve cassette combinations containing different promoters (P43, Pshuttle09), RBSs (RBS1, RBS5) and signal peptides (SnprB, SphoD, SymzC)
1 材料与方法
1.1 实验材料
1.1.1 菌株与质粒
本研究使用的所有菌株和质粒列于表1和表2。Escherichia coli DH5α用于载体构建和扩增,青岛擎科天成生物技术有限公司;B.subtilis WB800用于目的基因的表达、表达质粒pP43 NMK,中国南京农业大学阎新教授赠送。
表1 本研究所用菌株
Table 1 Strains used in this study

菌株特征来源菌株特征来源E.coli DH5αrecA1 endA1 gyrA96 thi hsdR17(r-K m+K) relA1 supE44 φ80ΔlacZΔM15 Δ(lacZYA-argF)U169青岛擎科B.Subtilis WB800(168) ΔnprE, Δ aprA, Δepr, Δ brbpr, Δmpr, ΔnprB, Δvpr, ΔwprA[1]WBA1B.subtilis WB800 转入 pP431NAK本研究构建WBA2B.subtilis WB800 转入 pP435NAK本研究构建WBA3B.subtilis WB800 转入 pP431PAK本研究构建WBA4B.subtilis WB800 转入 pP435PAK本研究构建WBA5B.subtilis WB800 转入 pP431YAK本研究构建WBA6B.subtilis WB800 转入 pP435YAK本研究构建WBA7B.subtilis WB800 转入 pP091NAK本研究构建WBA8B.subtilis WB800 转入 pP091PAK本研究构建WBA9B.subtilis WB800 转入 pP095PAK本研究构建WBA10B.subtilis WB800 转入 pP095PAK本研究构建WBA11B.subtilis WB800 转入 pP091YAK本研究构建WBA12B.subtilis WB800 转入 pP095YAK本研究构建WBC1B.subtilis WB800 转入 pP431NCK本研究构建WBC2B.subtilis WB800 转入 pP435NCK本研究构建WBC3B.subtilis WB800 转入 pP431PCK本研究构建WBC4B.subtilis WB800 转入 pP435PCK本研究构建WBC5B.subtilis WB800 转入 pP431YCK本研究构建WBC6B.subtilis WB800 转入 pP435YCK本研究构建WBC7B.subtilis WB800 转入 pP091NCK本研究构建WBC8B.subtilis WB800 转入 pP091PCK本研究构建WBC9B.subtilis WB800 转入 pP095PCK本研究构建WBC10B.subtilis WB800 转入 pP095PCK本研究构建WBC11B.subtilis WB800 转入 pP091YCK本研究构建WBC12B.subtilis WB800 转入 pP095YCK本研究构建WBL1B.subtilis WB800 转入 pP431NLK本研究构建WBL2B.subtilis WB800 转入 pP435NLK本研究构建WBL3B.subtilis WB800 转入 pP431PLK本研究构建WBL4B.subtilis WB800 转入 pP435PLK本研究构建WBL5B.subtilis WB800 转入 pP431YLK本研究构建WBL6B.subtilis WB800 转入 pP435YLK本研究构建WBL7B.subtilis WB800 转入 pP091NLK本研究构建WBL8B.subtilis WB800 转入 pP091PLK本研究构建WBL9B.subtilis WB800 转入 pP095PLK本研究构建WBL10B.subtilis WB800 转入 pP095PLK本研究构建WBL11B.subtilis WB800 转入 pP091YLK本研究构建WBL12B.subtilis WB800 转入 pP095YLK本研究构建
表2 本研究所用质粒
Table 2 Plasmids used in this study

质粒特征来源质粒特征来源pP43NMKP43, ApR, KnR[19]pP431NAKpP43NMK导入, P43, RBS1, nprB, agwh50c基因本研究构建pP435NAKpP43NMK导入, P43, RBS5, nprB, agwh50c基因本研究构建pP431PAKpP43NMK导入, P43, RBS1, phoD, agwh50c基因本研究构建pP435PAKpP43NMK导入, P43, RBS5, phoD, agwh50c基因本研究构建pP431YAKpP43NMK导入, P43, RBS1, ymzC, agwh50c基因本研究构建pP435YAKpP43NMK导入, P43, RBS5, ymzC, agwh50c基因本研究构建pP091NAKpP43NMK导入, Pshuttle09, RBS1, nprB, agwh50c基因本研究构建pP095NAKpP43NMK导入, Pshuttle09, RBS5, nprB, agwh50c基因本研究构建pP091PAKpP43NMK导入, Pshuttle09, RBS1, phoD, agwh50c基因本研究构建pP095PAKpP43NMK导入, Pshuttle09, RBS5, phoD, agwh50c基因本研究构建pP091YAKpP43NMK导入, Pshuttle09, RBS1, ymzC, agwh50c基因本研究构建pP095YAKpP43NMK导入, Pshuttle09, RBS5, ymzC, agwh50c基因本研究构建pP431NCKpP43NMK导入, P43, RBS1, nprB, csn21c基因本研究构建pP435NCKpP43NMK导入, P43, RBS5, nprB, csn21c基因本研究构建pP431PCKpP43NMK导入, P43, RBS1, phoD, csn21c基因本研究构建pP435PCKpP43NMK导入, P43, RBS5, phoD, csn21c基因本研究构建pP431YCKpP43NMK导入, P43, RBS1, ymzC, csn21c基因本研究构建pP435YCKpP43NMK导入, P43, RBS5, ymzC, csn21c基因本研究构建pP091NCKpP43NMK导入, Pshuttle09, RBS1, nprB, csn21c基因本研究构建pP095NCKpP43NMK导入, Pshuttle09, RBS5, nprB, csn21c基因本研究构建pP091PCKpP43NMK导入, Pshuttle09, RBS1, phoD, csn21c基因本研究构建pP095PCKpP43NMK导入, Pshuttle09, RBS5, phoD, csn21c基因本研究构建pP091YCKpP43NMK导入, Pshuttle09, RBS1, ymzC, csn21c基因本研究构建pP095YCKpP43NMK导入, Pshuttle09, RBS5, ymzC, csn21c基因本研究构建pP431NLKpP43NMK导入, P43, RBS1, nprB,lip15基因本研究构建pP435NLKpP43NMK导入, P43, RBS5, nprB,lip15基因本研究构建pP431PLKpP43NMK导入, P43, RBS1, phoD,lip15基因本研究构建pP435PLKpP43NMK导入, P43, RBS5, phoD,lip15基因本研究构建pP431YLKpP43NMK导入, P43, RBS1, ymzC,lip15基因本研究构建pP435YLKpP43NMK导入, P43, RBS5, ymzC,lip15基因本研究构建pP091NLKpP43NMK导入, Pshuttle09, RBS1, nprB,lip15基因本研究构建pP095NLKpP43NMK导入, Pshuttle09, RBS5, nprB,lip15基因本研究构建pP091PLKpP43NMK导入, Pshuttle09, RBS1, phoD,lip15基因本研究构建pP095PLKpP43NMK导入, Pshuttle09, RBS5, phoD,lip15基因本研究构建pP091YLKpP43NMK导入, Pshuttle09, RBS1, ymzC,lip15基因本研究构建pP095YLKpP43NMK导入, Pshuttle09, RBS5, ymzC,lip15基因本研究构建
1.1.2 主要试剂及培养基
P505-d1聚合酶、Taq聚合酶混合体系2×Taq Master Mix、无缝拼接试剂盒,南京诺唯赞生物科技有限公司;胶回收试剂盒,美国Omega Bio-Tek公司;快速质粒小提试剂盒,天根生化科技(北京)有限公司;PCR所用引物合成及测序由青岛生工生物科技有限公司提供;其他试剂及药品未作特殊说明均为国产分析纯。
LB(Luria-Bertani)液体培养基(g/L):蛋白胨10,NaCl 10,酵母提取物5,115 ℃灭菌30 min后备用(LB固体培养基,添加15~20 g/L的琼脂粉)。
氨苄青霉素溶液:称取0.1 g的氨苄青霉素粉末放置于1.5 mL的离心管中,用1.0 mL的超纯水溶解,在超净台中过孔径0.22 μm的水相滤膜,放于-20 ℃冰箱备用,且添加量为0.1%(体积分数),最终使用质量浓度为100 μg/mL。
100.0 mg/mL卡那霉素溶液:称取1.0 g卡那霉素粉末放置于1.5 mL的离心管中,用1.0 mL的超纯水溶解,在超净台中过孔径0.22 μm的水相滤膜,放于-20℃冰箱备用,且添加量为0.05%(体积分数),最终使用质量浓度为50 μg/mL。
GMI溶液(100 mL):1×最低盐溶液95.6 mL,200 g/L葡萄糖2.5 mL,50 g/L水解酪蛋白0.4 mL,100 g/L酵母粉1 mL,10 mg/mL氨基酸溶液0.5 mL。
GMII溶液(100 mL):1×最低盐溶液96.98 mL,200 g/L葡萄糖2.5 mL,50 g/L水解酪蛋白0.08 mL,100 g/L酵母粉0.04 mL,1 mol/L MgCl2 0.25 mL,1 mol/L CaCl2 0.05 mL,10 mg/mL氨基酸溶液0.1 mL。
TB(terrific broth)培养基:12 g/L蛋白胨,24 g/L酵母粉,4 mL/L甘油,17 mmol/L KH2PO4,72 mmol/L K2HPO4。
SOC(the super optimal broth with catabolite repression)培养基:20 g/L蛋白胨,5 g/L酵母粉,0.5 g/L NaCl,2.5 mmol/L KCl,10 mmol/L MgCl2,20 mmol/L葡萄糖。
SR(super-rich)培养基(g/L):蛋白胨 25,酵母粉 20, K2HPO4 0.3,葡萄糖 30。
ZTY培养基(g/L):酵母粉 25,葡萄糖 31.4, KH2PO4 1.29, NaCl 0.5, MgSO4·7H2O 0.5(注:ZTY培养基为本实验室人员通过单因素优化得到的可使枯草芽孢杆菌富集培养的丰富培养基)。
1.2 实验方法
1.2.1 琼胶酶AgWH50C表达载体的构建
目的基因agwh50c是以Agarivorans gilvus WH0801基因组为模板,用引物Sn-50C-F和50C-R扩增获得agwh50c片段,表达载体的骨架是以质粒pP43NMK为模板,通过引物Bone-F、Bone-R扩增获得,通过将目的基因与载体进行无缝拼接,获得质粒pP431NAK。在此载体上构建其他琼胶酶AgWH50C表达载体。
对于质粒pP435NAK的构建,以pP431NAK质粒为模板,通过引物P43-5-B-F和P43-5-B-R扩增获得同源臂含有RBS5片段的线性化质粒,再应用无缝拼接试剂盒进行连接,即获得质粒pP435NAK,即pP431NAK质粒表达盒P43-RBS1-SnprB中RBS1替换为RBS5片段,即为质粒pP435NAK。
对于质粒pP091NAK的构建,通过引物P09-F和P09-R扩增获得Pshuttle09片段,以pP431NAK质粒为模板,通过引物P09-RBS1-B-F、Bone2-R扩增载体,再应用无缝拼接试剂盒进行连接,即获得质粒pP091NAK。
对于质粒pP431PAK的构建,通过引物P43-RBS1-D-F和D-R扩增获得SphoD片段,以pP431NAK质粒为模板,通过引物Bone-P43-F和Bone-P43-RBS1-R扩增载体,再应用无缝拼接试剂盒进行连接,即获得质粒pP091PAK。对于其他质粒的构建方法同pP435NAK质粒、pP091NAK质粒、pP431PAK质粒的构建方法。
1.2.2 壳聚糖酶Csn21c表达载体的构建
以链霉菌Streptomyces albolongus ATCC 27414基因组为模板,通过引物Sn-csn21c-F、csn21c-R扩增获得目的片段csn21c。
pP431NCK、pP435NCK、pP091NCK和pP095NCK质粒的构建:以链霉菌S.albolongus ATCC 27414基因组为模板,通过引物Sn-csn21c-F、csn21c-R扩增目的片段csn21c。分别以质粒pP431NAK、pP435NAK、pP091NAK和pP095NAK为模板,通过引物Bone-F,Bone-N-R扩增载体骨架。之后的构建方法同pP431NAK。
pP431PCK、pP435PCK、pP091PCK和pP095PCK质粒的构建:通过引物Sp-csn21c-F、csn21c-R扩增目的片段csn21c。分别以质粒pP431PAK、pP435PAK、pP091PAK和pP095PAK为模板,通过引物Bone-F、Bone-P-R扩增载体骨架。之后的构建方法同上。
pP431YCK、pP435YCK、pP091YCK和pP095YCK质粒的构建:通过引物Sy-csn21c-F、csn21c-R扩增目的片段csn21c。分别以质粒pP431YAK、pP435YAK、pP091YAK和pP095YAK为模板,通过引物Bone-F、Bone-Y-R扩增载体骨架。之后的构建方法同上。
1.2.3 脂肪酶Lip15表达载体的构建
脂肪酶Lip15来源于链霉菌Streptomyces bacillaris ATCC 15855T,其相关质粒的构建方法同壳聚糖酶Csn21c。
1.2.4 目的基因的转化与表达
将所得重组质粒,转化至E.coli DH5α感受态细胞中。E.coli DH5α在含100 μg/mL氨苄青霉素的5 mL LB培养基中,37 ℃、220 r/min培养过夜。
1.2.5 B.subtilis WB800感受态的制备及转化
采用化学法制备感受态,具体操作如下:
取甘油管保存的B.subtilis WB800在无抗的LB固体培养基上划线,于37 ℃培养过夜;用接种环接一单菌落于5 mL GMI溶液中,30 ℃,125 r/min振荡培养过夜;次日取2 mL上一步所得培养液转接到18 mL GMI中,37 ℃、250 r/min培养5 h;从上一步骤的培养液取10 mL转接到90 mL GMII中,37 ℃,125 r/min培养90 min后,于4 ℃、5 000×g离心10 min收集菌体;用10 mL GMII将菌体重新悬浮,加入20 mL已灭菌的30%甘油,使其终体积分数为10%,将其混匀后,分装到1.5 mL的离心管中(每管0.5 mL),置于-80 ℃保存;转化时取出离心管,将其置于45 ℃水浴锅中溶化,然后加入适量DNA(约0.5 μg),吹打混匀后,于37 ℃,200 r/min孵育30 min后涂布相应抗性(Kana)平板上,37 ℃恒温培养过夜。
含重组质粒的枯草芽孢杆菌在含50 μg/mL卡那霉素的液体LB培养基中,30 ℃、220 r/min培养,每间隔一段时间测其OD600和相应酶活力。优化培养基为TB培养基,SOC培养基,SR培养基和本实验室配制的ZTY培养基。通过测量600 nm光密度(OD600)监测细胞生长情况。本研究所用引物如表3所示。
表3 本研究所用引物
Table 3 Primers used in this study

引物序列 5’→3’Bone-FTGATGAAAGCTTGGCGTAATC Bone-RTGCAGCTGAGGCATGTGTTACP43-5-B-FAGAAAGGAGGTGATCATGCGCAACTTGP43-5-B-RCGCATGTGTACATTCCTCTCTTACCTATAATGGTACCGCTATCAP09-FGAGATTTTTTTGAGCAACTGATCGTCACAATGCGCCATCAAACP09-RGTGTACATTCCTCTCTTACGATAACACATTCATGGATCCCP43-5-C-FAGAAAGGAGGTGATCTTGTTTGAAAGTGAAGCAGAP43-5-C-RGCTGGCTCCATACTAGTCCGCCCGCACGCCCCGAACAGTAAAAAGACBone-P43-5-RGATCACCTCCTTTCTCTATAATGGTACCGCTATCABone-P43-FCGGACTAGTATGGAGCCAGCAAATCAGCCCP43-5-D-FAGAAAGGAGGTGATCATGGCATACGACAGTCP43-5-D-RGCTCCATACTAGTCCGTACTTCAAAGGCBone-09-5-RGATCACCTCCTTTCTGATAACACATTCATGGATCCCBone-09-5-FGGACTAGTATGGAGCCAGCAAATCAGC09-5-C-FAGAAAGGAGGTGATCTTGTTTGAAAGTGAAGCAGA09-5-C-FTGGCTCCATACTAGTCCGCCCGCACGCCCCGAACAGTAAAAAG09-5-D-FAGAAAGGAGGTGATCATGGCATACGACAGTCGTTTTG09-5-D-RACTAGTCCGTACTTCAAAGGCCCCAACCGACTGP43-RBS1-RCATTCCTCTCTTACCTATAATGGTACCGCTATCACP43-RBS1-B-FGTAAGAGAGGAATGTACACATGCGCAACTTGACCP43-RBS1-C-FGTAAGAGAGGAATGTACACTTGTTTGAAAGTGAAGCAGP43-RBS1-D-FGTAAGAGAGGAATGTACACATGGCATACGP09-RBS1-RCCTCTCTTACGATAACACATTCATGGATCCCACP09-RBS1-B-FGTAAGAGAGGAATGTACACATGCGCAACTTGACCP09-RBS1-C-FGTAAGAGAGGAATGTACACTTGTTTGAAAGTGAAGCAGAP09-RBS1-D-FGTAAGAGAGGAATGTACACATGGCATACGACAGTCBone2-RAGTTGCTCAAAAAAATCTCGGTCD-RGCTCCATACTAGTCCGTACTTCAAAGGCC-RGCTGGCTCCATACTAGTCCGCCCGCACGCCCCGAACAGTAAAAAGACSn-50C-FTAACACATGCCTCAGCTGCAATGGAGCCAGCAAATCAGCCCSp-50C-FTCGGTTGGGGCCTTTGAAGTACGGACTAGTATGGAGCCAGCAAATCASy-50C-FTTTTACTGTTCGGGGCGTGCGGGATGGAGCCAGCAAATCAGCCC50C-RCAGTGGTGGTGGTGGTGGTGGCTCGAGCCCTAAACACGAAAGCGTTSn-csn21c-FTAACACATGCCTCAGCTGCAATGCCTGTCGTGCGCCGTCGTACSp-csn21c-FGCCTTTGAAGTACGGACTAGTATGCCTGTCGTGCGCCGTCGTACSy-csn21c-FGGGCGTGCGGGCGGACTAGTATGCCTGTCGTGCGCCGTCGcsn21c-RCAGTGGTGGTGGTGGTGGTGGCTGTTGATGCTGAAGCTGTCSn-lip15-FGTAACACATGCCTCAGCTGCAATGAAGATGTCCAGACTTGTSp-lip15-FGGTTGGGGCCTTTGAAGTACGGACTAGTATGAAGATGTCCAGACTTGTSy-lip15-FGTTCGGGGCGTGCGGGCGGACTAGTATGAAGATGTCCAGACTTGTlip15-RCAGTGGTGGTGGTGGTGGTGGGCAGCCGAGTTGAAGB-ep095p-FGTTTGATGGCGCATTGTGACGATCAGTTGCTCAAAAAAATCTCGB-ep095p-RTCGGTTGGGGCCTTTGAAGTACGGACTAGTATGCCTGTCGTGCep-09-FCTGATCGTCACAATGCGCCATCAAACCGTTep-P-RTACTTCAAAGGCCCCAACCGACTGGGCAATB-ep435y-FCGAAAACATACCACCTATCAAGCTGCTCAAAAAAATCTCGB-ep435y-RTTTTACTGTTCGGGGCGTGCGGGCGGACTAGTATGCCTGTCGTep-43-FGCAGCTTGATAGGTGGTATGTTTTCGCTTGep-Y-RCCCGCACGCCCCGAACAGTAAAAAGACAG
1.2.6 琼胶酶AgWH50C的酶活力测定方法
本研究采用3,5-二硝基水杨酸法(3,5-dinitrosalicylic acid, DNS)[22]测定琼胶酶的活性。反应条件为:150 μL发酵上清液与50 μL含0.3%低熔点琼脂糖的20 mmol/L磷酸钠缓冲液(pH 6.0)混合,37 ℃反应30 min,加入300 μL的DNS煮沸10 min终止反应,直至样品冷却后在540 nm处测定吸光度。琼胶酶AgWH50C酶活力单位为37 ℃下1 min内释放1 μmol还原糖的酶量。
1.2.7 壳聚糖酶Csn21c的酶活力测定方法
采用DNS法[23]测定壳聚糖酶Csn21c的活性。该酶的反应底物为溶于体积分数1%醋酸溶液的2%(体积分数,下同)的壳聚糖。反应体系为10 μL的发酵上清液加入到190 μL 2%的壳聚糖溶液中混匀,55 ℃反应15 min,加入300 μL的DNS煮沸10 min终止反应,直至样品冷却后在540 nm处测定吸光度。壳聚糖Csn21c酶活力单位定义为在55 ℃条件下1 min内产生1 μmol葡萄糖胺的酶量。
1.2.8 脂肪酶Lip15的酶活力测定方法
反应体系为先加入200 μL上清液与400 μL Tris-HCl缓冲液(pH 8.0, 100 mmol/L)混合,于37 ℃反应5 min,然后再加入20 μL对硝基丁酸苯酯(0.02 mol/L,溶于异丙醇和二甲亚砜缓冲液中),37 ℃反应5 min后[24],加入650 μL 质量分数1%的SDS终止反应,在405 nm处测定上清液的吸光度。一个单位的脂肪酶Lip15酶活力被定义为1 min内形成1 μmol对硝基苯酚所需的酶量。
1.2.9 SDS-PAGE分析
上清液样品在4 ℃、1 000 ×g离心10 min,用50 mmol/L Tris/HCl,pH 8.0重悬细胞。根据检测蛋白的大小,分别使用4%的浓缩胶和12.5%的分离胶。16 μL待测样品与4 μL的5×loading buffer混匀,沸水浴10 min后上样。SDS凝胶试剂盒购自上海EpiZyme生物技术有限公司。
2 结果与分析
2.1 不同元件组合表达盒的分泌酶活力比较
2.1.1 不同元件组合表达琼胶酶AgWH50C的胞外酶活力比较
本研究用到的AgWH50C是GH50家族的β-琼胶酶,具有外切酶特性,目前已在大肠杆菌BL21中成功表达[22]。该酶在化妆品和制药行业具有很大的应用前景。本研究候选表达元件包括2个启动子(P43, Pshuttle09),2个RBS(RBS1, RBS5)和3个信号肽(SnprB, SphoD, SymzC),自候选启动子,RBS及信号肽中随机选择一个组装成一个表达盒,故共有12种元件组合,因此共构建了12个琼胶酶AgWH50C表达质粒(图2)。通过电转化将12个质粒分别转化到B.subtilis WB800中。培养4 h后,每隔2 h测定一次细胞密度和胞外酶活力,确定各菌株酶活力的最高点。将所有重组菌株表达琼胶酶AgWH50C的胞外酶活力进行比较如图2-a所示,与原载体pP431NAK相比,其中重组质粒pP435YAK的表达效率最高,pP435YAK能使琼胶酶AgWH50C胞外酶活力提高184.25%,其次是pP095PAK、pP095NAK、pP091NAK,AgWH50C胞外酶活力分别提高了155.80%、58.41%、15.35%。其余的表达盒胞外AgWH50C活性均比原载体pP431NAK低。图2-b和图2-c比较了这4种质粒转化所得菌株的胞外酶活力和细胞密度,可以看出,当菌株生长进入稳定期,胞外琼胶酶AgWH50C的酶活力基本也达到稳定。大多数菌株酶活力在9 h时达到最高。

a-含有不同表达盒的B.subtilis WB800胞外分泌琼胶酶AgWH50C的酶活力;b-含有4个最优AgWH50C表达盒的B.subtilis WB800胞外分泌琼胶酶AgWH50C的酶活力随时间变化曲线;c-含有4个最优AgWH50C表达盒的B.subtilis WB800的生长曲线;d-含有不同表达盒的B.subtilis WB800胞外分泌壳聚糖酶Csn21c的酶活力;e-含有4个最优Csn21c表达盒的B.subtilis WB800胞外分泌壳聚糖酶Csn21c的酶活力随时间变化曲线;f-含有4个最优Csn21c表达盒的B.subtilis WB800的生长曲线;g-含有不同表达盒的B.subtilis WB800胞外分泌脂肪酶Lip15的酶活力;h-含有4个最优Lip15表达盒的B.subtilis WB800胞外分泌脂肪酶Lip15的酶活力随时间变化曲线;i-含有4个最优Lip15表达盒的B.subtilis WB800的生长曲线;j-强表达效果载体筛选
图2 12个表达盒对3种不同蛋白的表达效果
Fig.2 The expression effect of 12 expression boxes on three different proteins
2.1.2 不同元件组合表达壳聚糖酶Csn21c的胞外酶活力比较
Csn21c是本课题组前期研究中发掘的壳聚糖酶并在大肠杆菌BL21中成功表达[25],其酶解产物为D-氨基葡萄糖和壳二糖,壳聚糖酶Csn21c在水产品高价值利用方面具有很大潜力,这是选择其作为目标蛋白的原因。
对以不同元件组合表达壳聚糖酶Csn21c的枯草芽孢杆菌进行发酵,每隔12 h取样,测定各菌株的细胞密度及其胞外酶活力。如图2-d所示,对其单因素进行比较,可以发现启动子Pshuttle09优于P43,核糖体结合位点RBS5优于RBS1三种信号肽中SphoD要优于SnprB和SymzC。其中载体pP095PCK的壳聚糖酶Csn21c胞外酶活力最高,较原载体pP431NCK提高了164.62%。只有载体pP431YCK的酶活力低于原载体。载体pP095NCK、pP435PCK和pP435YCK使Csn21c酶活力增加约40%,酶活力和细胞密度随时间变化曲线如图2-e和图2-f所示。生长进入稳定期(>24 h)后,酶活力继续增加。
2.1.3 不同元件组合表达脂肪酶Lip15的胞外酶活力比较
脂肪酶Lip15为本课题组新发掘的脂肪酶。如图2-h所示,仅质粒 pP431PLK、pP091YLK、pP095YLK的胞外表达效率低于原始质粒pP431NLK,其余8个元件组合的胞外脂肪酶活性均略高于原始质粒pP431NLK。其中载体pP435YLK的脂肪酶Lip15胞外酶活力最高,比原始质粒pP431NLK胞外酶活力提高了约52.8%。pP435YLK略优于pP091PLK、pP435NLK和pP095PLK(图2-g),在稳定期(>12 h)酶活力达到最高(图2-h和图2-i)。
为筛选对3种酶均具有强分泌效果的表达盒,首先对每种蛋白的12个表达盒按胞外酶活力进行排序,挑选出各个蛋白胞外酶活力最高的4个载体(琼胶酶AgWH50C较优载体为pP091NAK、pP095NAK、pP095PAK和pP435YAK,壳聚糖酶Csn21c较优载体为pP095NCK、pP435PCK、pP095PCK和pP435YCK,脂肪酶Lip15较优载体为pP435NLK、pP091PLK、pP095PLK和pP435YLK)。图2-j显示,pP435Y*K和pP095P*K是对3种蛋白均具有强表达效果的载体,因此二者有望成为对多种蛋白均具有强分泌效果的通用载体(图2-j)。
2.2 pP095PCK载体表达壳聚糖酶Csn21c的SDS-PAGE分析
本文通过SDS-PAGE的方法比较载体pP431NCK和pP095PCK细胞外和细胞内壳聚糖酶Csn21c(33 kDa)的表达情况,进一步分析细胞外表达量提高的原因。如图3所示,可以发现pP095PCK表达的壳聚糖酶Csn21c在细胞外和细胞内的表达量均高于原载体pP431NCK。结果表明,与pP431NCK相比,载体pP095PCK可以提高转录和翻译水平,进一步提高外源表达量。
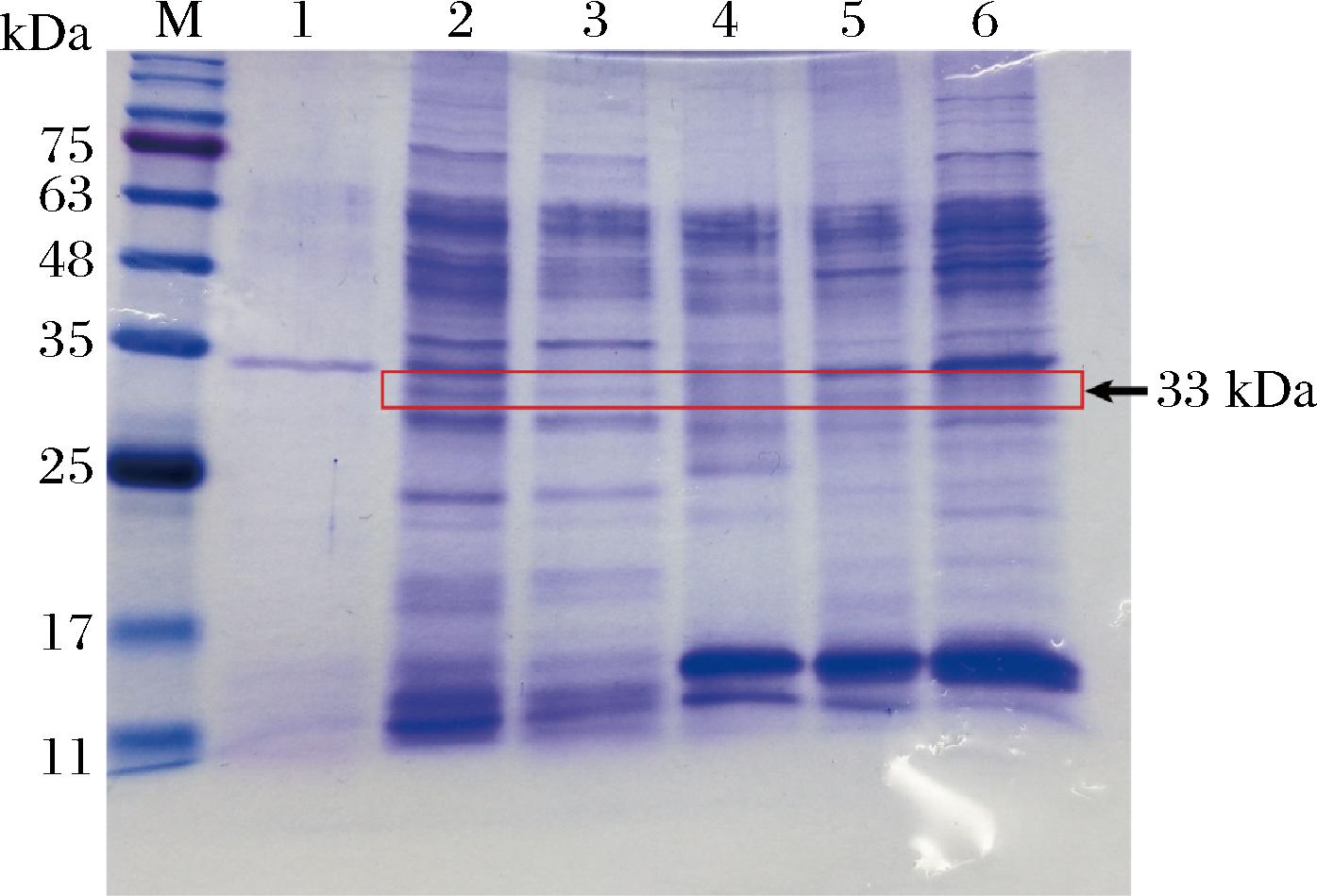
M-Marker;1~3分别为携带pP43NMK、pP095PCK、pP431NCK菌株发酵液的上清液;4~6分别为携带pP43NMK、pP095PCK、pP431NCK菌株胞内壳聚糖酶Csn21c
图3 载体pP431NCK和pP095PCK细胞内外壳聚糖酶Csn21c表达的SDS-PAGE分析
Fig.3 SDS-PAGE analysis of extracellular and intracellular chitonsanase Csn21c expression by pP431NCK and pP095PCK
2.3 发酵培养基优化
本文比较了5种培养基(LB培养基、TB培养基、SOC培养基、SR培养基和ZTY培养基)对pP095PCK质粒表达壳聚糖酶Csn21c的影响。细胞生长数据显示(图4-b),重组菌株在SR培养基中生长的速度比LB培养基高约3倍,然而壳聚糖酶Csn21c的表达与LB培养基中接近,这说明SR培养基中单位细胞的壳聚糖酶表达量仅为LB培养基的1/3。TB培养基不仅能使重组菌株更好地生长,而且能使壳聚糖酶Csn21c的酶活力提高128.31%,达到21.14 U/mL,是LB培养基的2.28倍(图4)。因此,确定TB培养基为壳聚糖酶Csn21c的最适发酵培养基。

a-五种培养基胞外酶活力比较;b-生长曲线;c-酶活力随时间的变化曲线
图4 不同培养基对pP095PCK质粒表达壳聚糖酶Csn21c的影响
Fig.4 Effect of five culture media on pP095PCK-mediated chitinsanase Csn21c expression
3 结论与讨论
在本研究中,以3种蛋白作为指示蛋白,比较了12种不同的表达元件组合。由图2可知,启动子Pshuttle09的转录效率高于P43,在翻译效率方面,RBS5优于RBS1。而载体pP091Y*K和pP095Y*K在这些蛋白中的表达能力较低,其原因可能是Pshuttle09和Symzc的适配性较低。在蛋白质方面,脂肪酶Lip15与载体的相容性较琼胶酶AgWH50C和壳聚糖酶Csn21c更好,在不同的组合方式下均能保持较高的相对活性。而这些载体与琼胶酶AgWH50C适配性较低,除载体pP095PAK和pP435YAK外,其余载体均表现较低的表达效率。进一步通过SDS-PAGE的方法确定较好用的载体pP095PCK表达的壳聚糖酶Csn21c在细胞外和细胞内的表达量均高于原载体pP431NCK。因此,载体pP095PCK可以提高转录和翻译水平,进一步提高外源表达量。本研究筛选获得了有潜力成为通用载体的高效蛋白分泌元件组合pP435Y*K和pP095P*K,为各种蛋白质在枯草芽孢杆菌表达系统中的高效分泌提供了新的载体。
[1] LI W F, ZHOU X X, LU P.Bottlenecks in the expression and secretion of heterologous proteins in Bacillus subtilis[J].Research in Microbiology, 2004, 155(8):605-610.
[2] SONG Y F, NIKOLOFF J M, ZHANG D W.Improving protein production on the level of regulation of both expression and secretion pathways in Bacillus subtilis[J].Journal of Microbiology and Biotechnology, 2015, 25(7):963-977.
[3] CHEN P T, SHAW J F, CHAO Y P, et al.Construction of chromosomally located T7 expression system for production of heterologous secreted proteins in Bacillus subtilis[J].Journal of Agricultural and Food Chemistry, 2010, 58(9):5392-5399.
[4] HERAVI K M, WENZEL M, ALTENBUCHNER J.Regulation of mtl operon promoter of Bacillus subtilis:Requirements of its use in expression vectors[J].Microbial Cell Factories, 2011, 10:83.
[5] BHAVSAR A P, ZHAO X, BROWN E D.Development and characterization of a xylose-dependent system for expression of cloned genes in Bacillus subtilis:Conditional complementation of a teichoic acid mutant[J].Applied and Environmental Microbiology, 2001, 67(1):403-410.
[6] LIU S L, DU K.Enhanced expression of an endoglucanase in Bacillus subtilis by using the sucrose-inducible sacB promoter and improved properties of the recombinant enzyme[J].Protein Expression and Purification, 2012, 83(2):164-168.
[7] LEE S J, PAN J G, PARK S H, et al.Development of a stationary phase-specific autoinducible expression system in Bacillus subtilis[J].Journal of Biotechnology, 2010, 149(1-2):16-20.
[8] WENZEL M, MÜLLER A, SIEMANN-HERZBERG M, et al.Self-inducible Bacillus subtilis expression system for reliable and inexpensive protein production by high-cell-density fermentation[J].Applied and Environmental Microbiology, 2011, 77(18):6419-6425.
[9] ZHANG X Z, CUI Z L, HONG Q, et al.High-level expression and secretion of methyl parathion hydrolase in Bacillus subtilis WB800[J].Applied and Environmental Microbiology, 2005, 71(7):4101-4103.
[10] YANG M M, ZHANG W W, JI S Y, et al.Generation of an artificial double promoter for protein expression in Bacillus subtilis through a promoter trap system[J].PLoS One, 2013, 8(2):e56321.
[11] SONG W, NIE Y, MU X Q, et al.Enhancement of extracellular expression of Bacillus naganoensis pullulanase from recombinant Bacillus subtilis:Effects of promoter and host[J].Protein Expression and Purification, 2016, 124:23-31.
[12] ZHANG X Z, CUI Z L, HONG Q, et al.High-level expression and secretion of methyl parathion hydrolase in Bacillus subtilis WB800[J].Applied and Environmental Microbiology, 2005, 71(7):4101-4103.
[13] XIA Y, CHEN W, ZHAO J X, et al.Construction of a new food-grade expression system for Bacillus subtilis based on theta replication plasmids and auxotrophic complementation[J].Applied Microbiology and Biotechnology, 2007, 76(3):643-650.
[14] VELLANOWETH R L, RABINOWITZ J C.The influence of ribosome-binding-site elements on translational efficiency in Bacillus subtilis and Escherichia coli in vivo[J].Molecular Microbiology, 1992, 6(9):1105-1114.
[15] SALIS H M, MIRSKY E A, VOIGT C A.Automated design of synthetic ribosome binding sites to control protein expression[J].Nature Biotechnology, 2009, 27(10):946-950.
[16] WANG J P, AI X L, MEI H, et al.High-throughput identification of promoters and screening of highly active promoter-5′-UTR DNA region with different characteristics from Bacillus thuringiensis[J].PLoS One, 2013, 8(5):e62960.
[17] MAO X Z, HUANG Z Y, SUN G Y, et al.High level production of diacetylchitobiose deacetylase by refactoring genetic elements and cellular metabolism[J].Bioresource Technology, 2021, 341:125836.
[18] DEGERING C, EGGERT T, PULS M, et al.Optimization of protease secretion in Bacillus subtilis and Bacillus licheniformis by screening of homologous and heterologous signal peptides[J].Applied and Environmental Microbiology, 2010, 76(19):6370-6376.
[19] ZHANG J J, KANG Z, LING Z M, et al.High-level extracellular production of alkaline polygalacturonate lyase in Bacillus subtilis with optimized regulatory elements[J].Bioresource Technology, 2013, 146:543-548.
[20] ZHU F M, JI S Y, ZHANG W W, et al.Development and application of a novel signal peptide probe vector with PGA as reporter in Bacillus subtilis WB700:Twenty-four tat pathway signal peptides from Bacillus subtilis were monitored[J].Molecular Biotechnology, 2008, 39(3):225-230.
[21] SNYDER A J, MUKHERJEE S, GLASS J K, et al.The canonical twin-arginine translocase components are not required for secretion of folded green fluorescent protein from the ancestral strain of Bacillus subtilis[J].Applied and Environmental Microbiology, 2014, 80(10):3219-3232.
[22] LIU N, MAO X Z, YANG M, et al.Gene cloning, expression and characterisation of a new β-agarase, AgWH50C, producing neoagarobiose from Agarivorans gilvus WH0801[J].World Journal of Microbiology &Biotechnology, 2014, 30(6):1691-1698.
[23] GAO L, SUN J N, SECUNDO F, et al.Cloning, characterization and substrate degradation mode of a novel chitinase from Streptomyces albolongus ATCC 27414[J].Food Chemistry, 2018, 261:329-336.
[24] RASOL R, RASHIDAH A R, NAZUHA R N, et al.Psychrotrophic lipase producers from Arctic soil and sediment samples[J].Polish Journal of Microbiology, 2014, 63(1):75-82.
[25] GUO N, SUN J N, WANG W, et al.Cloning, expression and characterization of a novel chitosanase from Streptomyces albolongus ATCC 27414[J].Food Chemistry, 2019, 286:696-702.