肿瘤的生长和转移与血管生成密不可分,肿瘤抑素是继血管生成抑素、内皮抑素、色素上皮衍生因子之后最新发现的来源于人基底膜IV型胶原的肿瘤新生血管抑制因子[1-2]。IV型胶原由α1~α6六条链以不同的组合方式组成不同的三聚体,这些α链包括N末端7S结构域、中间三螺旋结构域和C末端球状非胶原结构域1(NC1),其被认为在血管生成过程中对内皮细胞增殖和行为起着至关重要的作用[3-4]。Tumstatin是IV型胶原蛋白α3链的一段C末端球状非胶原结构域,分子质量为28 kDa,具有N末端抗血管生成和C末端抗肿瘤细胞活性[5]。肿瘤抑素能与肿瘤细胞、内皮细胞表面αVβ3整合素结合,诱导内皮细胞凋亡并通过FAK/Akt/NF途径减缓肿瘤细胞生长。研究发现Tumstatin能够使内皮细胞特异性凋亡、新生血管生成受到抑制从而抑制肿瘤生长、浸润和转移[6-7]。
与传统的放、化疗相比,肿瘤抑素治疗肿瘤具有抗肿瘤作用强等特点,对多种肿瘤细胞都具有活性[8],因此实现可溶性Tumstatin大量制备具有重要意义。目前,国内外研究者的研究主要集中在Tumstatin抗血管生成和抗肿瘤作用机制,肿瘤抑素的可溶性异源表达研究鲜有报道,表达宿主包括原核表达系统大肠杆菌和毕赤酵母及人胚肾细胞等真核细胞[9-13]。颜廷东[14]将人tumstatin基因与pET28a载体连接转入大肠杆菌BL21(DE3),得到占菌体总蛋白量的30%以上的包涵体Tumstatin,LUO等[13]将tumstatin基因与促溶标签MBP在大肠杆菌BL21融合表达,融合蛋白占菌体总蛋白20%以上,且实验结果表明重组蛋白具有抑制内皮细胞的增殖、促进内皮细胞凋亡和等能力;GU等[15]实现了Tumstatin在大肠杆菌中以不溶性形式蛋白表达,且目的蛋白占细菌总蛋白30%以上,并证明重新折叠的重组蛋白具有抑制肿瘤细胞活性。综上所述,大肠杆菌多以包涵体的形式异源表达Tumstatin,且目的蛋白含量仅占菌体总蛋白约20%~30%。
可溶性融合标签能帮助一些不溶性蛋白正确折叠,增强目的蛋白的溶解度。常见的融合标签包括硫氧还原蛋白(thioredoxin A,TrxA)标签、谷胱甘肽巯基转移酶(glutathione S-transferase,GST)标签、麦芽糖结合蛋白(maltose binding protein,MBP)标签、NusA蛋白(N-utilization substance A)标签以及小分子泛素样修饰蛋白(small ubiquitin-like modifier,SUMO)标签等多种标签[16-17]。但是,这些标签并不适用于每一种蛋白的表达,如何增加蛋白的溶解度仍是一个重要的课题。抗冻蛋白含有大量的苏氨酸和丝氨酸,能与水分子形成氢键,即使是疏水性的抗冻蛋白也能通过扭曲的主链形成氢键来结合大量的水分子,因此抗冻蛋白具有优异的溶解性和亲水性[18-19]。在此基础上,XIE等[20]开发了一种新型的人工蛋白XXA(小球藻来源的一种抗冻蛋白的反向蛋白),研究发现与常用的促溶性融合标签相比XXA蛋白能显著提高目的蛋白的可溶性表达。进一步通过对XXA预测的结构分析发现XXA的C端的亲水侧聚集了大量同种电荷,研究者认为位于XXA表面的大量相同电荷是提高蛋白可溶性表达的关键因素,XXA的强库仑斥力阻止了包涵体蛋白的聚集和形成。
本研究为实现Tumstatin蛋白的可溶性表达,构建了包括MBP、GST、TrxA、SUMO、XXA标签分别与tumstatin基因融合表达的不同重组菌,筛选出能可溶性表达Tumstatin蛋白的菌株,并在摇瓶水平对其进行诱导条件优化,为Tumstatin的可溶性表达以及应用奠定了基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株和质粒
本研究所用菌株和质粒如表1所示。
表1 本研究所用菌株和质粒
Table 1 Strains and plasmids used in this study

菌株和质粒特性和用途来源E.coli BL21(DE3)大肠杆菌表达宿主实验室保藏E.coli BL21(DE3)/pET28a-XXA-TumE.coli BL21(DE3)携带pET28a-XXA-Tum 质粒本研究构建E.coli BL21(DE3)/pET28a-MBP-TumE.coli BL21(DE3)携带 pET28a-MBP-Tum质粒本研究构建E.coli BL21(DE3)/pET28a-SUMO-TumE.coli BL21(DE3)携带 pET28a-SUMO-Tum质粒本研究构建E.coli BL21(DE3)/pET32a- TumE.coli BL21(DE3)携带pET32a-Tum质粒本研究构建E.coli BL21(DE3)/pGEX-6p-1- TumE.coli BL21(DE3)携带 pGEX-6p-1- Tum质粒本研究构建E.coli BL21(DE3)/pET28a- TumE.coli BL21(DE3)携带 pET28a- Tum 质粒本研究构建pET28a大肠杆菌表达载体实验室保藏pET32a大肠杆菌表达载体,含TrxA 标签实验室保藏pGEX-6p-1大肠杆菌表达载体,含 GST 标签实验室保藏pUC57-TumpUC57携带 Tum 基因天霖生物科技无锡有限公司pET28a-XXApET28a携带XXA基因天霖生物科技无锡有限公司pET28a-TumpET28a携带片段基因本研究构建pET28a-XXA-TumpET28a携带片段 His-XXA-Tum本研究构建pET28a-MBP-TumpET28a携带片段 MBP-Tum-His本研究构建pET28a-SUMO-TumpET28a携带片段 SUMO-Tum-His本研究构建pET32a-TumpET32a携带片段 Tum-His本研究构建pGEX-6p-1-TumpGEX-6p-1携带片段Tum-His本研究构建
1.1.2 引物
本研究所用引物如表2所示。
表2 本研究所用的引物
Table 2 Primers used in this study

引物名称引物序列(5′-3′)酶切位点28a-Tum-FCCCAAGCTTCCTGGCCTGAAAGGCHind Ⅲ28a-Tum-RCCGCTCGAGTGCGGCCGCTTAGTGACGTTTTTTCAT-GCAAACCTGGXho IGST-Tum-FCGCGGATCCCCTGGCCTGAAAGGCAAACGBamH IGST-Tum-RCCCAAGCTTGTGACGTTTTTTCATGCAAACCTGGHind ⅢMBP-Tum-FCGCGGATCCCCTGGCCTGAAAGGCAAACGBamH IMBP-Tum-RCCCAAGCTTGTGACGTTTTTTCATGCAAACHind ⅢXXA-FCGCGGATCCCCTAAATTACGCGACGCCGCBamH IXXA-RCCCAAGCTTCAGGGGCCCCTGGAACHind ⅢSUMO-FCGAGCTCATGTCAGATAGTGAAGTAAAC-CAAGAAGCCSac ISUMO-RCGCGGATCCCAATGGTCCTTGGAATAGCACTTCAAGBamH I32a-Tum-FttctggttctggccatatgCTGGAAGTTCTGTTCCAGGGGNde I32a-Tum-RcgagtgcggccgcaagcttGTGACGTTTTTTCATGCAAACCTGGHind Ⅲ
注:引物下划线为酶切位点,小写为同源臂。
1.1.3 试剂与培养基
限制性内切酶、T4DNA连接酶,大连Takara公司;GoldBand 3-color Regular Range Protein Marker,上海Yeasen 公司;2×Taq Master Mix、ClonExpress® Ⅱ One Step Cloning Kit、2×Phanta flash MasterMix、质粒DNA提取、片段纯化、凝胶回收试剂盒,南京诺唯赞生物科技股份有限公司;PrePack GSH Purose 4 Fast Flow预装柱,江苏千纯公司;硫酸卡那霉素、氨苄青霉素钠,上海麦克林生化科技有限公司,其他试剂均为国产分析纯。
LB培养基(g/L):NaCl 10,酵母粉5,蛋白胨10,115 ℃灭菌20 min。
TB培养基(g/L):酵母粉24,胰蛋白胨12,甘油5,KH2PO42.31,K2HPO4 12.54,115 ℃灭菌20 min。
1.2 仪器与设备
S100D型 PCR仪、Chemi Doc凝胶成像仪,美国Bio-Rad公司;DYY-6C核酸电泳仪,北京六一仪器厂;Pico17高速离心机,美国Thermo Fisher Scientific公司;HYL-C型组合式摇床,太仓市实验设备厂;Sonic VCX-750型超声波细胞破碎仪,南京新辰生物科技有限公司;SCG 蛋白纯化系统,苏州赛谱仪器有限公司;VS-1306超净工作台,苏州安泰空气技术有限公司;TU-100C恒温金属浴,上海一恒科技有限公司。
1.3 实验方法
1.3.1 人源肿瘤抑素融合表达质粒的构建
以质粒PUC57-Tum为模板,利用PCR扩增,通过酶切、酶连、热激转化、菌落PCR、测序等分子生物学手段最终得到6个重组菌株:E.coli BL21(DE3)/pET28a-Tum、E.coli BL21(DE3)/pET28a-XXA-Tum、E.coli BL21(DE3)/pET28a-MBP-Tum、E.coli BL21(DE3)/pET28a-SUMO-Tum、E.coli BL21(DE3)/pET32a- Tum、E.coli BL21(DE3)/pGEX-6p-1- Tum。
1.3.2 人源肿瘤抑素融合蛋白的诱导表达及Western-blot检测
从保菌管中分别沾取上述重组菌并划线于对应的带有卡那霉素、氨苄青霉素抗性的LB固体平板上37 ℃倒置培养24 h,挑取单菌落接种于15 mL LB液体培养基中,37 ℃,200 r/min培养12 h,以1∶50体积比接种于50 mL LB培养基中,37 ℃、200 r/min培养至OD600=1.5左右添加异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)至终浓度为0.5 mmol/L,16 ℃、200 r/min培养24 h,12 000 r/min离心5 min后弃上清液收集菌体,用pH 8.0 Tris-HCl缓冲液(0.5 mol/L NaCl,20 mmol/L咪唑,0.5 mol/L Tris)洗涤3次,Tris-HCl重悬菌体,按照4 ℃、工作1 s、停止2 s,总共15 min的条件破碎细胞,4 ℃、12 000 r/min离心8 min,取上清液、沉淀通过SDS-PAGE分析蛋白表达情况。将SDS-PAGE电泳后的蛋白在350 mA转移到PVDF膜上80 min后用5% 牛血清白蛋白(bovine serum albumin, BSA)的三羟甲基氨基甲烷缓冲液(Tris buffered saline with Tween 20, TBST。成分组成为:10 mmol/L Tris-HCl、150 mmol/L NaCl、0.1% Tween 20)封闭膜,然后与一抗在4 ℃下孵育过夜,然后用TBST冲洗膜并在室温下与二抗孵育1 h。最后用TBST洗涤并使用鲁米诺试剂观察免疫反应信号。
1.3.3 人源肿瘤抑素融合蛋白表达发酵工艺优化
1.3.3.1 诱导剂IPTG浓度
从甘油管中沾取重组菌菌液进行划线,挑取单菌落于15 mL LB中37 ℃、200 r/min培养12 h,按照2%的接种量接种于TB培养基,于37 ℃、200 r/min 条件下培养2 h(OD600=1.4~1.6),分别添加IPTG至终浓度为0.1、0.2、0.4、0.6、0.8、1.0 mmol/L,分3个平行,于16 ℃、200 r/min培养24 h,测定OD600值,取1 mL样品,12 000 r/min、5 min 收集菌体、破碎细胞,测定蛋白表达量。
1.3.3.2 诱导剂添加时间
从甘油管中沾取重组菌菌液进行划线,挑取单菌落于15 mL LB中37 ℃、200 r/min培养12 h,按照2%的接种量接种于TB培养基,于37 ℃、200 r/min条件下培养,在优化IPTG浓度基础上分别于1、2、4、6、8、12 h添加诱导剂,分3个平行,于16 ℃、200 r/min培养24 h,其他条件一致,测定OD600值,取1 mL样品,12 000 r/min、5 min收集菌体、破碎细胞,测定蛋白表达量。
1.3.3.3 诱导温度
从甘油管中沾取重组菌菌液进行划线,挑取单菌落于15 mL LB中37 ℃、200 r/min培养12 h,按照2%的接种量接种于TB培养基,在优化的诱导剂添加浓度、诱导剂添加时间基础上,分别于16、25、30、37 ℃诱导培养24 h,分3个平行,其他条件一致,测定OD600值,取1 mL样品,12 000 r/min、5 min收集菌体、破碎细胞,测定蛋白表达量。
1.3.3.4 诱导时间
从甘油管中沾取重组菌菌液进行划线,挑取单菌落于15 mL LB中37 ℃、200 r/min培养12 h,按照2%的接种量接种于TB培养基,在优化的诱导剂添加浓度、诱导剂添加时间基础上、诱导温度基础上,分别诱导培养8、16、24、36 h,分3个平行,其他条件一致,测定OD600值,取1 mL样品,12 000 r/min、5 min 收集菌体、破碎细胞,测定蛋白表达量。
1.3.4 人源肿瘤抑素蛋白的纯化
首先,用过膜的去离子水以15 mL/min的流速冲洗蛋白纯化系统2 min以及利用2 mL/min的流速冲洗PrePack Ni-NTA Perose 6FF预装柱20 min以除去乙醇;2 mL/min的流速用平衡液-A液(500 mmol/L NaCl、20 mmol/L咪唑、20 mmol/L Tris-HCl,pH 8.0)冲洗柱子20 min以平衡柱子;2 mL/min的流速对过膜的重组菌破碎液上清进行上样;2 mL/min的流速用A液冲洗柱子20 min;2 mL/min的流速分别用终浓度的为50、100、300、500 mmol/L咪唑的洗脱液-B液(500 mmol/L NaCl、相应浓度的咪唑、20 mmol/L Tris-HCl,pH 8.0)进行洗脱25 min,收集出峰样品并用SDS-PAGE进行分析。
1.3.5 蛋白表达量的测定
参考李正杰等[21]的方法,对破碎后的上清液样品利用考马斯亮蓝染色法测定总蛋白浓度,对上清液样品进行SDS-PAGE分析,采用Image Lab软件对SDS-PAGE电泳图进行蛋白条带灰度扫描,根据目的蛋白占所占总蛋白百分比计算出目的蛋白的量,该方法仅用于诱导条件优化蛋白表达量的确定。
2 结果与分析
2.1 人源肿瘤抑素表达载体的构建及验证
按照1.3.1节的方法成功获得6个重组菌:pET28a -Tum、pET28a-XXA-Tum、pET28a-MBP-Tum、pET32a- Tum以及pGEX-6p-1- Tum、pET28a-SUMO-Tum。将上述质粒转入BL21(DE3)中,获得了相应的单基因表达菌株和融合表达菌株,提取质粒进行双酶切验证。如图1-a和图1-b所示,用Hind Ⅲ、Xho I双酶切pET28a -Tum 和pET28a-XXA-Tum,分别得到5 277 bp(pET28a载体)和5 919 bp(XXA标签和pET28a载体)以及共同的754 bp(tumstatin基因);同样用Hind Ⅲ和BamH I分别双酶切载体pET32a-Tum、pET28a-MBP-Tum、pGEX-6p-1-Tum,分别得到大小为746 bp(tumstatin基因)和5 756 bp(图1-c)、749 bp(tumstatin基因)和6 455 bp(MBP标签和pET28a)(图1-d)、746 bp(tumstatin基因)和4 817 bp(pGEX-6-p-1载体);如图1-f,载体pET28a-SUMO-Tum用BamH I和Xho I双酶切得到781 bp(tumstatin基因)和5 538 bp(SUMO标签和pET28a载体)。上述重组质粒通过双酶切验证结果与理论一致,且测序结果完全正确,表明各重组菌构建成功。
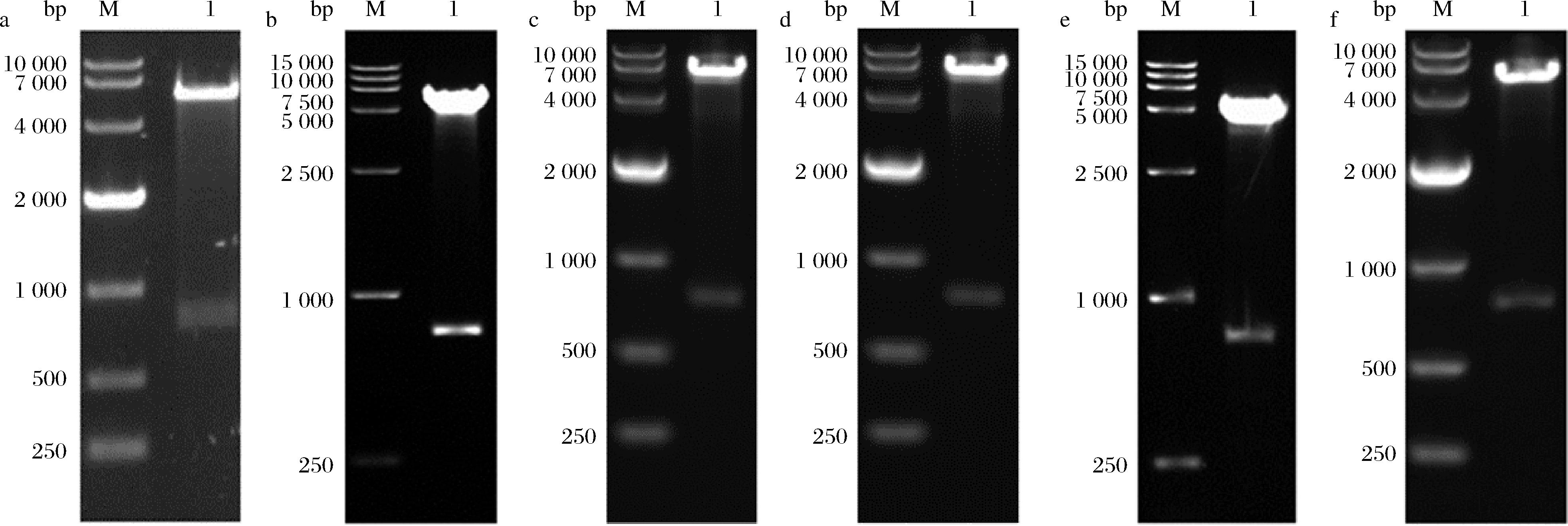
M-Maker,DL15000、DL10000(Takara);1-重组质粒酶切结果a、b-pET28a-Tum和pET28a-XXA-Tum Hind Ⅲ、Xho I双酶切结果;c、d、e-pET32a-Tum、pET28a-MBP-Tum、pGEX-6p-1-Tum Hind Ⅲ和BamH I双酶切结果;f-pET28a-SUMO-Tum BamH I和Xho I双酶切结果
图1 重组质粒酶切验证
Fig.1 Recombinant plasmid digestion to validate
2.2 人源肿瘤抑素融合蛋白的诱导表达
对构建好的6株重组菌进行诱导发酵24 h,通过SDS-PAGE分析细胞破碎后的上清液及沉淀表达情况,结果如图2-a~图2-f所示。Tumstatin蛋白相对分子质量为28 kDa,单基因表达菌株在细胞破碎沉淀中检测到相应条带,而对照中无相应条带;XXA标签大小为22.5 kDa,融合蛋白XXA/Tumstatin理论分子质量为50.9 kDa。如图2-a所示重组菌E.coli BL21/pET28a-XXA-Tum细胞破碎上清中检测到与理论分子质量一致的条带,通过扫描对条带进行灰度扫描,发现目的蛋白占总蛋白45%以上,空载菌株中未检测到相应条带;E.coli BL21/pGEX-6p-1-Tum、E.coli BL21/pET28a-MBP-Tum、E.coli BL21/pET28a-SUMO-Tum、E.coli BL21/pET32a-Tum表达的融合蛋白理论上分别55.3、72.7、41、42 kDa,重组菌E.coli BL21/pGEX-6p-1-Tum、E.coli BL21/pET28a-MBP-Tum的细胞破碎沉淀分别检测到与分子质量一致的条带,E.coli BL21/pET28a-SUMO-Tum、E.coli BL21/pET32a-Tum未检测到相应条带。结果表明,成功获得了一株能可溶性表达融合蛋白XXA/Tumstatin的重组菌E.coli BL21/pET28a-XXA-Tum,使用该重组菌进行后续实验。

M-Maker;1-空载破碎后沉淀;2-空载破碎后上清液;3-重组菌破碎后沉淀;4-重组菌破碎后上清液a-E.coli BL21(DE3)/pET28a-XXA-Tum;b-E.coli BL21(DE3)/pGEX-6p-1-Tum;c-E.coli BL21(DE3)/pET28a-SUMO-Tum;d-E.coli BL21(DE3)/pET28a-MBP-Tum;e-E.coli BL21(DE3)/pET32a-Tum;f-E.coli BL21(DE3)/pET28a-Tum
图2 各重组菌融合蛋白表达情况
Fig.2 Expression of fusion protein of each recombinant bacteria
2.3 重组菌表达人源肿瘤抑素融合蛋白的纯化及Western-Blot
如图3-a、图3-b所示,纯化结果和利用使用抗His标签抗体的Western-Blot分析显示,在50.9 kDa处出现目的条带,表明成功构建了能表达融合蛋白XXA-Tumstatin的重组菌BL21/pET28a-XXA-Tum。

a-蛋白纯化SDS-PAGE结果(M-Maker;1-未纯化样品;2~3-纯化样品);b-Western-Blot结果(M-Maker;1-细胞破碎上清液)
图3 蛋白纯化及Western-Blot验证
Fig.3 Protein purification and Western-Blot verification
2.4 人源肿瘤抑素融合蛋白表达诱导条件优化
2.4.1 重组菌BL21/pET28a-XXA-Tum生长曲线测定
将BL21/pET28a-XXA-Tum于TB培养基、37 ℃、200 r/min培养24 h,每隔2 h取样测定OD600值,绘制生长曲线。如图4所示,0~10 h为对数生长前期,10~18 h为对数生长后期,18~24 h为稳定期,根据该生长曲线对重组菌进行诱导条件的优化。
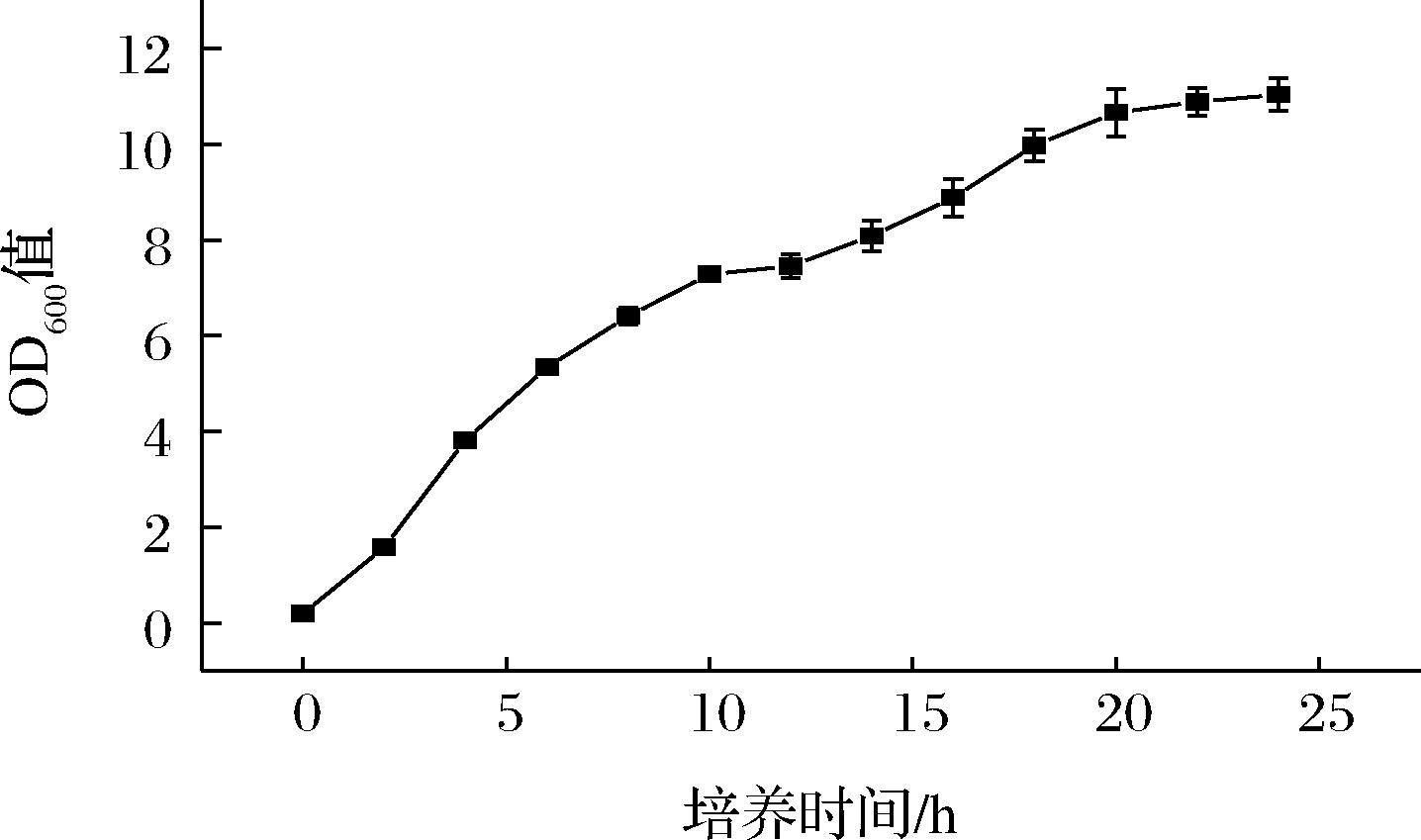
图4 重组菌生长曲线
Fig.4 Growth curve of recombinant bacteria
2.4.2 IPTG浓度
重组菌中所含的重组质粒pET28a-XXA-Tum为诱导型的Tac启动子,需要IPTG诱导目的蛋白表达。但IPTG诱导浓度太低不足以结合阻遏蛋白而诱导目的蛋白的转录而降低蛋白表达,太高则会影响重组菌的生长和蛋白表达,并且IPTG比较昂贵[22]。因此,本研究中首先针对IPTG的浓度开展研究。结果如图5-a所示,随着诱导剂IPTG浓度的升高,菌体生长受影响,目的蛋白的表达也受到影响。从结果来看,添加终浓度0.1 mmol/L的IPTG时OD600值最高为14.4,蛋白质量浓度达到1.01 mg/mL,并且考虑到过高浓度IPTG会抑制菌体生长进而影响蛋白表达,因此选择0.1 mmol/L的IPTG为最适浓度。

a-诱导剂浓度;b-诱导剂添加时间;b-诱导温度;d-诱导时间
图5 发酵工艺优化
Fig.5 The optimization of fermentation process
注:大写字母表示OD600值的显著性统计,小写字母表示蛋白浓度的显著性统计,不同字母代表组间具有显著性差异(P<0.05)。
2.4.3 诱导剂添加时间
在大肠杆菌中表达异源蛋白时,通常会影响细胞的正常生长和代谢,过早表达会给重组菌带来代谢负担,太晚诱导强度可能不够,影响细胞生长从而降低目的蛋白的产生[23-24]。结果如图5-b所示,诱导剂添加过早或者过晚都会影响菌株的生长和代谢,2 h添加诱导剂时重组菌生长和蛋白表达都为最优,OD600值和蛋白质量浓度分别达到16.0和0.92 mg/mL,因此选择2 h添加诱导剂。
2.4.4 诱导温度
温度是影响细胞生长和代谢的主要因素之一,低温能够促进蛋白正确折叠,高温不仅影响质粒稳定性而且促使蛋白快速合成而发生错误折叠[25]。结果如图5-c所示,随着温度的升高,重组菌的生长呈现降低的趋势,蛋白表达也逐渐降低。16 ℃诱导时,菌蛋白表达最高为1.06 mg/mL,其余3个温度诱导蛋白表达无明显差异,选择16 ℃为最优诱导温度。
2.4.5 诱导时间
诱导时间过短无法获得适量的目的蛋白,诱导时间过长会延长发酵周期、增加成本。结果如图5-d所示,随着诱导时间的延长,菌体量与蛋白表达量都逐渐增多,诱导时间达36 h时,菌体OD600值和蛋白质量浓度分别为19.8和1.09 mg/mL。诱导时间太长会导致培养基不足,细胞不能完成正常代谢,结合周期和成本问题,选择诱导时间为36 h。
3 结论与讨论
大肠杆菌因其遗传背景清晰和目的蛋白生产水平高仍然是研究最广泛重组蛋白表达宿主[26],但是由于没有真核细胞的翻译后修饰系统,大肠杆菌在表达真核蛋白基因时常常因为不正确折叠形成包涵体或者被靶向蛋白酶体降解,在N端添加融合标签一定程度上能解决这一问题[27]。融合标签虽然能提高某一类外源蛋白的溶解度,但无法证明某种融合标签能提高所有蛋白的溶解度。为实现人源Tumstatin在大肠杆菌中可溶性表达,本研究将不同促溶标签与tumstatin基因融合表达,发现XXA标签能促进Tumstatin蛋白高效可溶性表达,表达的重组蛋白占菌体总蛋白的45%以上。
TrxA、SUMO、MBP、GST等标签虽然是促进可溶性表达的最佳N端融合蛋白,但融合蛋白的溶解性取决于融合蛋白的大小、疏水性以及能否正确折叠等[28]。XXA蛋白标签因表面具有大量同种电荷而能阻止包涵体聚集和形成能力可能成为大肠杆菌表达异源蛋白的强有力促溶标签,然而融合标签提高目的蛋白的具体机制仍不明确,因此很难找到一个适合所有蛋白的万能标签,如何利用高通量技术及分子生物学技术筛选建立促溶标签工具箱仍是重要的研究课题。
研究进一步对重组菌E.coli BL21/pET28a-XXA-Tum进行摇瓶水平上的诱导条件优化,筛选出该重组菌最佳的诱导条件为:IPTG浓度0.1 mmol/L、2 h添加诱导剂、诱导温度16 ℃、诱导时间36 h。本研究对微生物生产可溶性人源功能性蛋白或多肽有一定借鉴作用,也为大量获取可溶性Tumstatin蛋白及研究Tumstatin蛋白在肿瘤治疗中的作用奠定基础,后续实验将纯化Tumstatin蛋白并进行细胞实验验证其功能活性。
[1] FOLKMAN J.Tumor angiogenesis:Therapeutic implications[J].The New England Journal of Medicine, 1971, 285(21):1182-1186.
[2] MAESHIMA Y, COLORADO P C, TORRE A, et al.Distinct antitumor properties of a type IV collagen domain derived from basement membrane[J].The Journal of Biological Chemistry, 2000, 275(28):21340-21348.
[3] LEBLEU V S, DAI J L, TSUTAKAWA S, et al.Identification of unique α4 chain structure and conserved antiangiogenic activity of α3 NC1 type IV collagen in zebrafish[J].Developmental Dynamicsn, 2023, 252(7):1046-1060.
[4] MAESHIMA Y, COLORADO P C, KALLURI R.Two RGD-independent alpha vbeta3 integrin binding sites on tumstatin regulate distinct anti-tumor properties[J].The Journal of Biological Chemistry, 2000, 275(31):23745-23750.
[5] HAMANO Y, YZEISBERG M, SUGIMOTO H, et al.Physiological levels of tumstatin, a fragment of collagen IV alpha3 chain, are generated by MMP-9 proteolysis and suppress angiogenesis via alpha vbeta3 integrin[J].Cancer Cell, 2003, 3(6):589-601.
[6] MAESHIMA Y, SUDHAKAR A, LIVELY J C, et al.Tumstatin, an endothelial cell-specific inhibitor of protein synthesis[J].Science, 2002, 295(5552):140-143.
[7] BOOSANI C S, MANNAM A P, COSGROVE D, et al.Regulation of COX-2 mediated signaling by alpha3 type IV noncollagenous domain in tumor angiogenesis[J].Blood, 2007, 110(4):1168-1177.
[8] 王淑静, 张艺, 刘佳, 等.肿瘤抑素抗肿瘤活性肽的研究进展[J].中国肿瘤, 2012, 21(1):56-59.
WANG S J, ZHANG Y, L J, et al.Research progress in anti-tumor peptide of tumstatin[J].China Cancer, 2012, 21(1):56-59
[9] DERRY C J, PICKERING M, BAKER C, et al.Identification of the Goodpasture antigen, alpha 3(IV) NC1, and four other NC1 domains of type IV collagen, by amino-terminal sequence analysis of human glomerular basement membrane separated by two-dimensional electrophoresis[J].Experimental Nephrology, 1994, 2(4):249-256.
[10] 顾取良, 张添元, 罗进贤, 等.人Tumstatin在毕赤酵母中的表达和活性分析[J].生物工程学报, 2006, (3):451-456.
GU Q L, ZHANG T Y, LUO J X, et al.Expression of human tumstatin in Pichia pastoris and its bioactivity[J].Chinese Journal of Biotechnology, 2006,22(3):451-456.
[11] ESIPOV R, BEYRAKHOVA K, LIKHVANTSEVA V, et al.Antiangiogenic and antivascular effects of a recombinant tumstatin-derived peptide in a corneal neovascularization model[J].Biochimie, 2012, 94(6):1368-1375.
[12] 姚丽娟. 重组融合蛋白Tumstatin-EGFP分泌型真核表达以及生物活性的研究[D];合肥:安徽医科大学, 2009.
YAO L J.Secreted eukaryotic expression and biological activity of recombinent fusion protein Tumstatin-EGFP[D];Hefei:Anhui Medical University, 2009.
[13] LUO Y Q, WANG L H, YI Q, et al.Expression of soluble, biologically active recombinant human tumstatin in Escherichia coli[J].Clinical and Experimental Medicine, 2008, 8(1):37-42.
[14] 颜廷东. 人肿瘤抑素的克隆、表达及其生物活性实验研究[D].北京:中国协和医科大学, 2007.
YAN T D.The study of cloning,expression and bioassay of human tumstatin[D].Beijing:Peking Union Medical College, 2007.
[15] GU Q L, ZHANG T Y, LUO J X, et al.Expression, purification, and bioactivity of human tumstatin from Escherichia coli[J].Protein Expression and Purification, 2006, 47(2):461-466.
[16] ESPOSITO D, CHATTERJEE D K.Enhancement of soluble protein expression through the use of fusion tags[J].Current Opinion in Biotechnology, 2006, 17(4):353-358.
[17] YARI K, AFZALI S, MOZAFARI H, et al.Molecular cloning, expression and purification of recombinant soluble mouse endostatin as an anti-angiogenic protein in Escherichia coli[J].Molecular Biology Reports, 2013, 40(2):1027-1033.
[18] LIOU Y-C, TOCILJ A, DAVIES P L, et al.Mimicry of ice structure by surface hydroxyls and water of a β-helix antifreeze protein[J].Nature, 2000, 406(6793):322-324.
[19] SUN T J, LIN F-H, CAMPBELL R L, et al.An antifreeze protein folds with an interior network of more than 400 semi-clathrate waters[J].Science, 2014, 343(6172):795-798.
[20] XIE X, WU P, HUANG X C, et al.Retro-protein XXA is a remarkable solubilizing fusion tag for inclusion bodies[J].Microbial Cell Factories, 2022, 21(1):51.
[21] 李正杰, 顾正华, 石贵阳, 等.Nanog蛋白在大肠杆菌中的可溶性表达及发酵优化[J].食品与发酵工业, 2020, 46(17):15-21.
LI Z J, GU Z H, SHI G Y, et al.Soluble expression of human Nanog protein in Escherichia coli and its fermentation optimization[J].Food and Fermentation Industries, 2020, 46(17):15-21.
[22] DUAN X G, CHEN J, WU J.Optimization of pullulanase production in Escherichia coli by regulation of process conditions and supplement with natural osmolytes[J].Bioresource Technology, 2013, 146:379-385.
[23] ZOU C, DUAN X, WU J.Enhanced extracellular production of recombinant Bacillus deramificans pullulanase in Escherichia coli through induction mode optimization and a glycine feeding strategy[J].Bioresource Technology, 2014, 172:174-179.
[24] YUAN S L, DUAN H Q, LIU C J, et al.The role of thioredoxin and disulfide isomerase in the expression of the snake venom thrombin-like enzyme calobin in Escherichia coli BL21 (DE3)[J].Protein Expression and Purification, 2004, 38(1):51-60.
[25] DUAN X G, HU S B, QI X H, et al.Optimal extracellular production of recombinant Bacillus circulans β-galactosidase in Escherichia coli BL21(DE3)[J].Process Biochemistry, 2017, 53:17-24.
[26] SU L Q, JIANG Q, YU L G, et al.Enhanced extracellular production of recombinant proteins in Escherichia coli by co-expression with Bacillus cereus phospholipase C[J].Microbial Cell Factories, 2017, 16(1):24.
[27] DYSON M R, SHADBOLT S P, VINCENT K J, et al.Production of soluble mammalian proteins in Escherichia coli:Identification of protein features that correlate with successful expression[J].BMC Biotechnology, 2004, 4(1):32.
[28] HAMMARSTRÖM M, HELLGREN N, VAN DEN BERG S, et al.Rapid screening for improved solubility of small human proteins produced as fusion proteins in Escherichia coli[J].Protein Science, 2002, 11(2):313-321.