嗜热链球菌(Streptococcus thermophilus)是发酵乳制品中应用最广泛的细菌之一,也是酸乳主要的发酵剂。多数嗜热链球菌在生长过程中能够产生胞外多糖(exopolysaccharides, EPS),对于发酵乳制品的生产而言,嗜热链球菌在发酵过程产生的EPS可以改善发酵乳制品的质构、黏度、持水性等感官和理化特性,可以减少其他改善产品质构的添加剂的使用,如乳清蛋白、淀粉、果胶等,最终,可以减少产品标签并降低生产成本。此外,近期研究表明,嗜热链球菌产生的EPS具有明确的生物活性和益生菌特性,在各种医药工业中作为抗氧化剂、抗癌、抗炎和免疫调节剂具有潜在的开发和利用潜力。因此,EPS成为了研究者最感兴趣的发酵产物之一。
EPS的产量、分子质量及结构是决定其理化性质的重要因素。一般而言,嗜热链球菌的EPS产量相对较低,在脱脂乳培养基中为50~400 mg/L[1],多为杂多糖(heteropolysaccharide, HePS),分子质量为10~2 000 kDa,通常由3~8个不同的单糖构成重复单元,其中D-半乳糖(D-galactose,D-Gal)、D-葡萄糖(D-glucose,D-Glu)、L-鼠李糖(L-rhamnose,L-Rha)及N-乙酰半乳糖胺(N-acetylgalactosamine,GalNac)是最为常见的单糖。嗜热链球菌EPS的合成途径非常复杂,其通过Wzx/Wzy依赖途径合成,又依赖位于细菌染色体的eps基因簇指导完成EPS重复单元的合成组装、聚合及输出等过程[2]。迄今为止,已有27个嗜热链球菌菌株eps基因簇完成测序,通过对其进行比较分析,发现存在至少20种不同的eps基因簇和67个糖基转移酶,不同的eps基因簇结构将直接导致EPS产量和化学结构的差异[3]。
一直以来,提升嗜热链球菌EPS产量,了解EPS的调控机制,使其能应用于工业生产是研究者关注的重点。菌株生长条件能够强烈影响乳酸菌中胞外多糖的产量、分子质量大小及单糖组成[4-5]。对于嗜热链球菌LY03,当使用乳糖和葡萄糖作为共同碳源时,其EPS产量显著提升[6]。碳源与氮源的比例也对EPS产生具有显著影响,较高的碳氮比以及足够的碳源和氮源浓度可提高EPS的产量[7-9]。此外,其他的影响因素包括培养的pH值、温度、氨基酸、磷酸盐、矿物质以及维生素[6,10-14]。尽管已有大量的研究对EPS的调控机制做出了解释,但仍存在不少疑问。几乎所有的研究都是在脱脂乳或M17培养基等复杂体系中进行优化培养,尽管这样的复杂培养基体系更贴近与实际生产,但却不利于研究单一具体的营养物质(如碳源、氨基酸、维生素和核酸碱基等)对EPS生物合成,进而难以准确确定调控EPS的调控靶点。因此,当研究乳酸菌定量和定性生产EPS以及EPS合成的调节时,使用限定化学成分培养基(chemical defined medium, CDM)尤为重要。在前期研究中通过优化CDM中成分和浓度以提升嗜热链球菌937在CDM中EPS的产量,结果发现,在以2%乳糖为碳源时,将组氨酸、异亮氨酸和谷氨酸浓度提升至15 mmol/L时,嗜热链球菌937 EPS的产量相较于在CDM中显著提升,但关于3种氨基酸是否对嗜热链球菌937 EPS的单糖组成及分子质量产生影响仍需进一步探索[15]。
本研究对嗜热链球菌937全基因组序列分析,得到嗜热链球菌937完整的EPS合成途径。通过对比嗜热链球菌937分别在组氨酸、异亮氨酸和谷氨酸初始浓度不同的CDM中的生长代谢、EPS产量和性质、eps基因簇上关键基因的转录水平的差异,探索氨基酸对嗜热链球菌EPS生物合成的影响,进一步了解其潜在的调控机制。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
S.thermophilus 937(CGMCC NO.25410)为已知的高产胞外多糖菌株,由江苏省乳品生物技术与安全控制重点实验室提供。
1.1.2 培养基
M17液体培养基,青岛海博生物技术有限公司;限定化学成分培养基(CDM),参考LETORT等[16]的描述略作改动,包含42种营养素、包括1种碳源、20种游离氨基酸、9种维生素以及多种无机盐成分,具体成分及浓度如表1所示。CDM培养基的pH值调整为6.7,并使用0.22 μm孔径的滤膜进行过滤除菌。在CDM基础上,将组氨酸、异亮氨酸和谷氨酸浓度由1 mmol/L提升至15 mmol/L,将所得培养基,即CDM+组氨酸(His)、异亮氨酸(Ile)和谷氨酸(Glu)命名为CDM+HIG。
表1 限定化学成分培养基成分
Table 1 Composition of the chemically defined medium
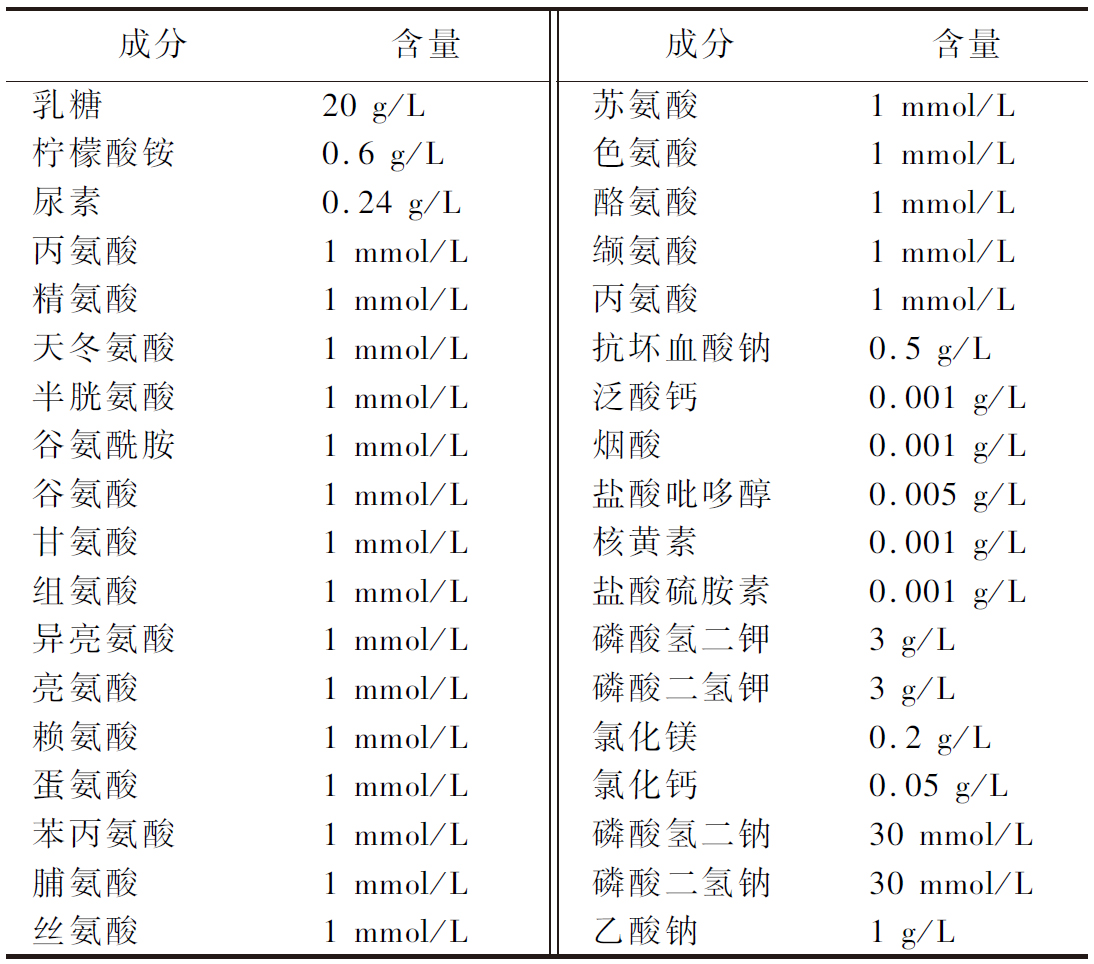
成分含量成分含量乳糖20 g/L苏氨酸1 mmol/L柠檬酸铵0.6 g/L色氨酸1 mmol/L尿素0.24 g/L酪氨酸1 mmol/L丙氨酸1 mmol/L缬氨酸1 mmol/L精氨酸1 mmol/L丙氨酸1 mmol/L天冬氨酸1 mmol/L抗坏血酸钠0.5 g/L半胱氨酸1 mmol/L泛酸钙0.001 g/L谷氨酰胺1 mmol/L烟酸0.001 g/L谷氨酸1 mmol/L盐酸吡哆醇0.005 g/L甘氨酸1 mmol/L核黄素0.001 g/L组氨酸1 mmol/L盐酸硫胺素0.001 g/L异亮氨酸1 mmol/L磷酸氢二钾3 g/L亮氨酸1 mmol/L磷酸二氢钾3 g/L赖氨酸1 mmol/L氯化镁0.2 g/L蛋氨酸1 mmol/L氯化钙0.05 g/L苯丙氨酸1 mmol/L磷酸氢二钠30 mmol/L脯氨酸1 mmol/L磷酸二氢钠30 mmol/L丝氨酸1 mmol/L乙酸钠1 g/L
1.1.3 试剂
三氯乙酸(trichloroacetic acid, TCA)、乙醇、浓硫酸、苯酚、三氟乙酸(trifluoroacetic acid, TFA)、氢氧化钠、醋酸钠,国药集团化学试剂有限公司;标准品甘露糖、鼠李糖、半乳糖醛酸、半乳糖、葡萄糖、葡萄糖醛酸、阿拉伯糖、木糖、岩藻糖、氨基葡萄糖、N-乙酰-D-氨基葡萄糖、D-果糖、D-核糖、氨基半乳糖、L-古洛糖醛酸、D-甘露糖醛酸,博睿糖生物公司;UNlQ-10柱式Trizol总RNA提取试剂盒,生工生物工程(上海)股份有限公司;HiScript II 1 st Strand cDNA Synthesis Kit (+gDNA wiper)试剂盒、AceQ qPCR SYBR Green Master Mix试剂盒,南京诺唯赞生物科技股份有限公司。
1.2 仪器与设备
Bioscreen CTM系统,Lab-systems公司;ABI Prism 7300 Detection System,Applied Biosystems公司;高效液相色谱仪,Shimadzu公司;离子色谱仪、酶标仪,Thermo Fisher公司;氮吹仪,力辰科技有限公司。
1.3 实验方法
1.3.1 菌株培养
嗜热链球菌937在M17液体培养基中42 ℃活化3代后备用,在接种前将嗜热链球菌937在M17液体培养基预培养至对数生长末期。为保证M17液体培养基中营养成分不干扰实验结果,菌株在接入生长培养基前,离心(10 000×g,5 min,25 ℃)收集菌体并用PBS缓冲液清洗2次,最终重悬于PBS中并调整OD600=1.00±0.05,重悬菌液按2%接种于生长培养基中,42 ℃培养,并使用Bioscreen CTM系统每隔15 min测定1次菌株在生长培养基中的OD600值。
1.3.2 嗜热链球菌937基因组分析
基因圈图、假定的蛋白质编码序列(coding sequence,CDS)鉴定、CDS功能注释在美吉生物云平台进行(www.majorbio.com)。eps基因簇通过BLASTN(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch)进行比对鉴定。
1.3.3 EPS分离及特性分析
EPS分离参照DE等[13]所描述的方法略作改动进行,培养完成后,将样品95 ℃加热15 min,使酶失活。样品冷却至室温后,涡旋混匀。取5 mL样品转入离心管中,4 ℃,12 000×g离心15 min,保留上清液。在上清液中加入80%(质量分数)TCA溶液至终浓度为20%,4 ℃静置8 h,离心(4 ℃,12 000×g,15 min),保留上清液,加入3倍体积无水乙醇,4 ℃静置过夜(>16 h)。离心(4 ℃,12 000×g,20 min),保留沉淀。将沉淀溶解于1 mL RO水中,装入透析袋(分子质量8 000~12 000 Da)。室温透析48 h,每4 h更换1次水。透析完成后,样品转入离心管中,离心(12 000×g,10 min)除去不溶物,保留上清液。使用苯酚硫酸法测定EPS浓度[17]。
EPS分子质量测定:精密称取样品和标准品,样品配制成5 mg/mL溶液,12 000×g离心10 min,上清液用0.22 μm的微孔滤膜过滤,然后将样品转置于1.8 mL进样小瓶中。使用高效液相色谱仪进行检测,色谱柱:BRT105-104-102串联凝胶柱(8 mm×300 mm);流动相:0.05 mol/L NaCl溶液;流速:0.6 mL/min;柱温:40 ℃;进样量:20 μL;检测器:示差检测器RI-10A。
EPS单糖组份检测:精密称量10 mg样品置于安瓿瓶中,加入3 mol/L TFA 10 mL,120 ℃水解3 h。准确吸取酸水解溶液转移至管中氮气吹干,加入10 mL RO水涡旋混匀,吸取100 μL加入900 μL去离子水,12 000×g离心5 min。取上清进IC分析。使用离子色谱仪进行检测,色谱柱:DionexCarbopacTMPA20 (3 mm×150 mm);流动相:A:H2O;B:15 mmol/L NaOH、100 mmol/L NaOAc;流速:0.3 mL/min;进样量:5 μL;柱温:30 ℃;检测器:电化学检测器。
1.3.4 RNA提取与q-PCR分析
收集1 mL在CDM和CDM+HIG中培养至3 h和5 h的细胞,8 000×g离心5 min,使用UNlQ-10柱式Trizol总RNA提取试剂盒提取细胞总RNA。利用样品在260 nm和280 nm吸光值检测样品RNA的浓度和纯度。使用诺唯赞HiScript Ⅱ 1 st Strand cDNA Synthesis Kit (+gDNA wiper)试剂盒进行逆转录。使用ABI Prism 7300检测系统进行q-PCR分析,所有引物均利用Oilgo 7.0软件根据全基因组数据进行设计(表2)。所有q-PCR反应均使用诺唯赞AceQ qPCR SYBR Green Master Mix试剂盒进行。
表2 q-PCR引物
Table 2 q-PCR primers

基因正向引物(5′-3′)反向引物(5′-3′)epsATAAAGACTAGAATTTGGCATCAGGTTCTAAATTTACG-GTGAepsBTGATTTCTCGAACACGGTCAAAGAAATTCATAGT-GGGCTTepsCGATTTCACGTACCAATACACCCTAAATTGCCAGAGT-CACCepsDCTAGCAAATGAAATCGCCAATTAAAGTGATTGCGATT-AGCTC16SCTAACTACGTGCCAGCAGCGGTTGAGCCACAGCCTT-TA
目的基因mRNA转录水平采用相对定量法进行计算,使用16S rDNA作为内参基因,CDM中样品作为对照组。
1.4 数据分析
每个实验至少进行3次生物平行,每次生物平行至少有3次技术平行。使用GraphPad Prism(适用于Windows的9.0.0版,GraphPad软件,美国加利福尼亚州圣地亚哥市)对数据进行分析,使用双尾计算的t检验或使用Dunnett或Tukey多重比较检验的单因素方差分析,如图例所示。
2 结果与分析
2.1 嗜热链球菌937基因组一般特征
嗜热链球菌937的基因组草案分布在60个支架上,总长度为1 861 615 bp,平均GC含量为39.11%,其中包括一条含有15个rRNA操纵子的线性染色体、57个tRNA基因和2 061个蛋白质编码基因(CDS)(图1)。在CDS中,KEGG和COG数据库分别注释到了1 156和1 675个功能基因组。根据KEGG和COG注释结果,在与碳水化合物运输和代谢相关的功能组中分别发现了106和90个基因,这意味着嗜热链球菌937可能在碳水化合物利用和EPS生物合成方面具有优势。CRISPR/Cas系统是一种针对噬菌体感染的原核免疫防御系统。在嗜热链球菌937基因组中发现了9个单独的CRISPR/Cas位点,表明菌株嗜热链球菌937可能对各种噬菌体感染具有良好的适应性免疫力。
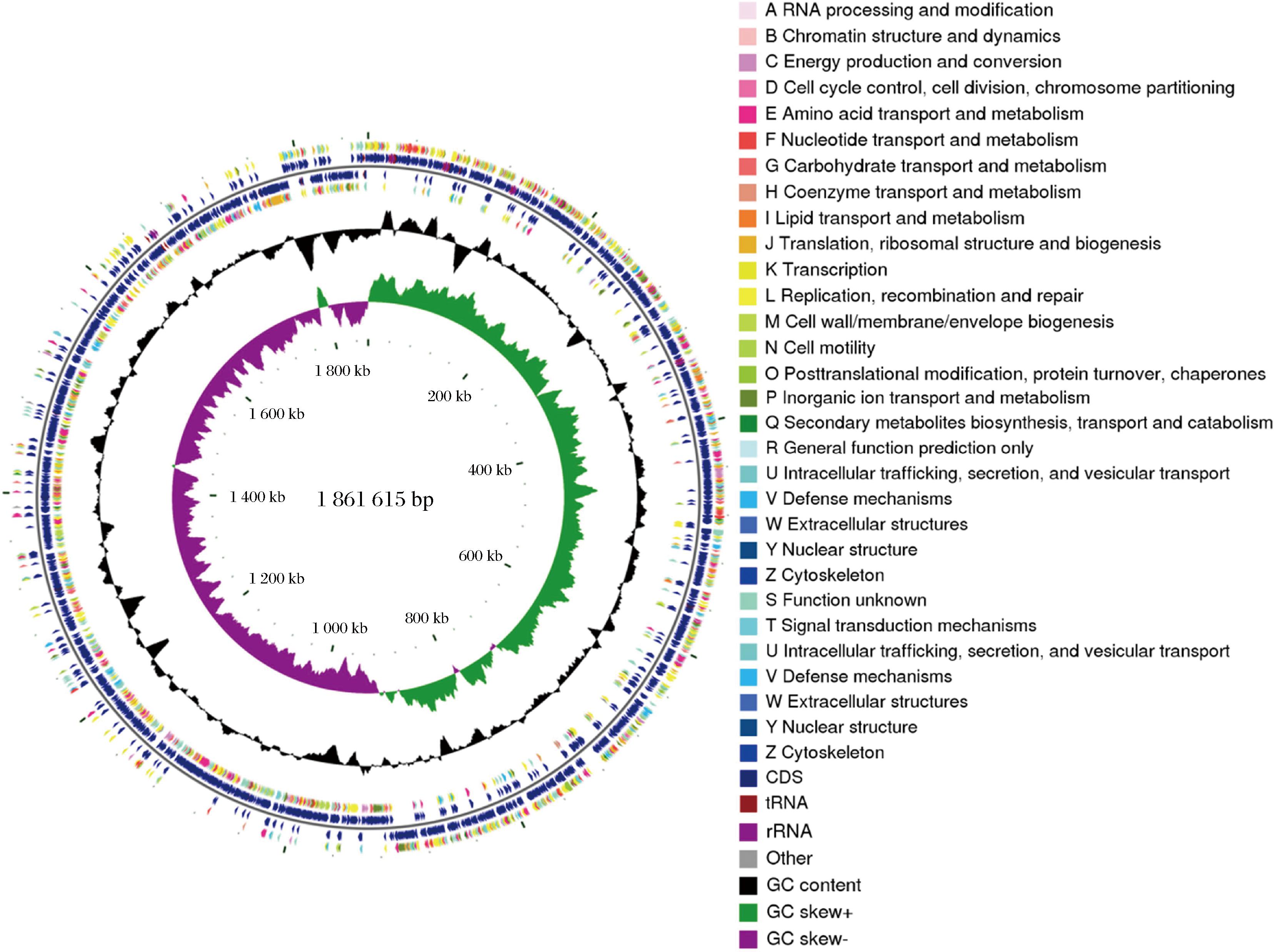
图1 嗜热链球菌937基因圈图
Fig.1 Circular genome map of Streptococcus thermophilus 937
2.2 基于全基因组数据分析嗜热链球菌937 EPS生物合成途径
2.2.1 嗜热链球菌937碳水化合物转运系统
嗜热链球菌主要通过Wzx/Wzy依赖途径合成f-EPS。完整的合成途径可分为4个步骤:a)碳水化合物的转运和代谢;b)糖前体(核苷酸糖)的生成;c)重复单元的组装;d)多糖的聚合与释放。对S.thermophilus937测得的全基因组序列与COG(clusters of orthologous groups of proteins)数据库进行比对,对注释到的f-EPS生物合成相关基因,按照f-EPS生物合成步骤进行分类总结,结果如表3~表5、图2、图3所示。
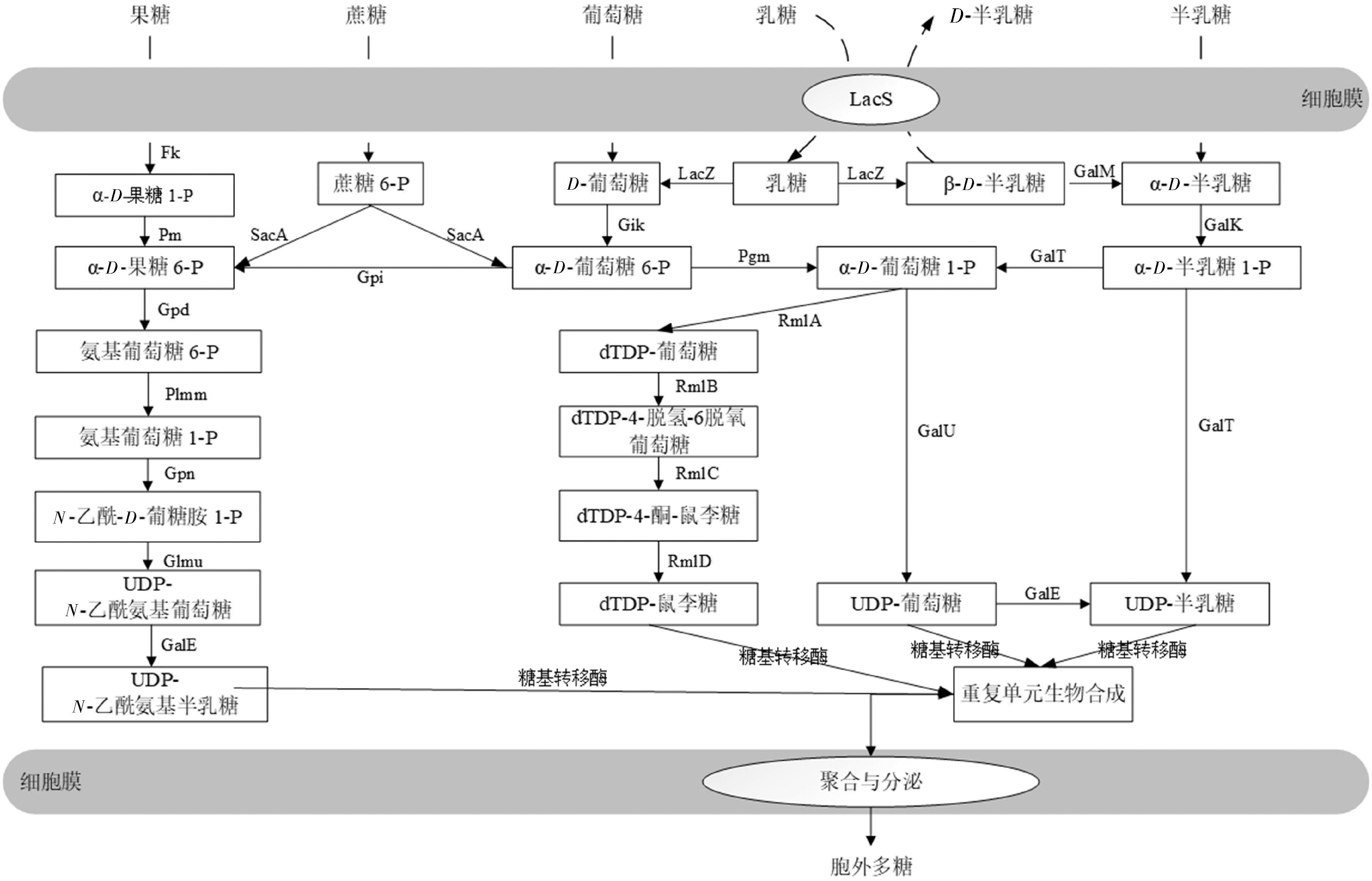
图2 嗜热链球菌937 EPS生物合成途径
Fig.2 EPS biosynthesis pathway of Streptococcus thermophilus 937
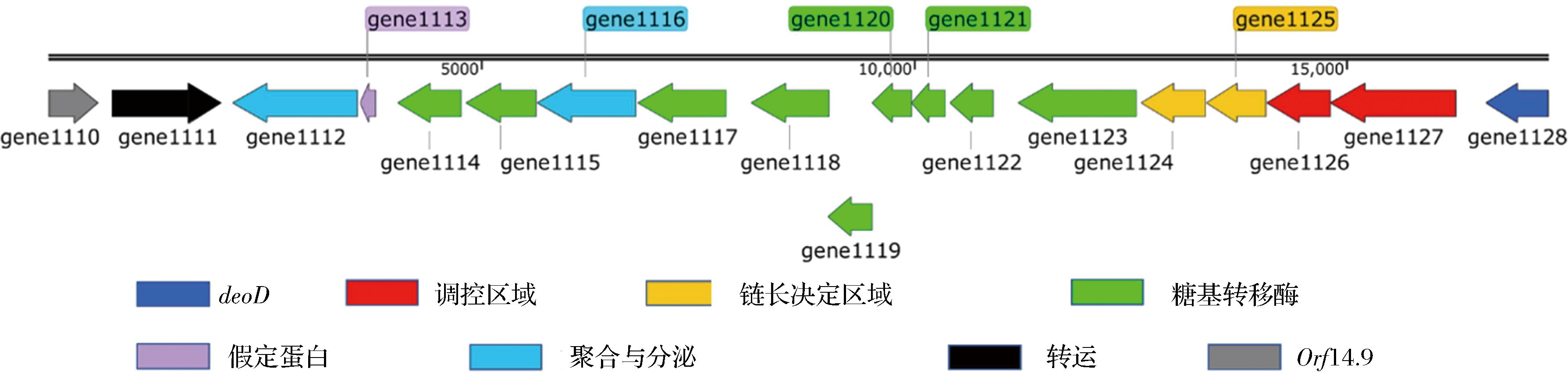
图3 嗜热链球菌937 eps基因簇
Fig.3 eps Gene cluster of Streptococcus thermophilus 937
表3 嗜热链球菌937糖转运系统相关基因
Table 3 Genes related to sugar transport system of Streptococcus thermophilus 937

位点名称基因描述gene0226-glucose PTS enzyme component IIABC, truncatedgene0227-PTS system glucose-specific EIICBAgene0365manZPTS system mannose-specific EIIDgene0366manYPTS system mannose-specific EIICgene0367manXPTS system mannose-specific EIIABgene0446fruBPTS system fructose-specific EIIABCgene0447fruBPTS system fructose-specific EIIABCgene0554-PTS transporter subunit EIICgene1434celBcellobiose PTS system EIICgene1475lacSlactose permeasegene1814scrKPTS system sucrose-specific EIIBCAgene1984-PTS system trehalose-specific EIIBCgene0810-sugar ABC transporter ATP-binding protein
由表3可知,嗜热链球菌937具有编码葡萄糖、果糖、蔗糖、海藻糖、甘露糖和纤维二糖特异性的PTS系统基因和乳糖渗透酶的基因,这表明在嗜热链球菌937除乳糖外,其余碳源可能均由PEP-PTS转运。
2.2.2 嗜热链球菌937核苷酸糖合成途径
由图2和表4可知,嗜热链球菌937具有合成EPS单元糖前体核苷酸糖的潜力,所有参与核苷酸糖合成的基因都在基因组中被鉴定。乳糖经乳糖渗透酶(LacS)进入细胞内,被β-半乳糖苷酶(LacZ)分解为葡萄糖和半乳糖。其中半乳糖可经Leloir途径形成葡萄糖1-磷酸参与核苷酸糖的形成,也可经半乳糖激酶(GalK)作用后形成UDP-半乳糖(UDP-Gal),UDP-半乳糖可在GalE的作用下,与UDP-葡萄糖相互转化。葡萄糖则经葡萄糖激酶(GK)作用,形成葡萄糖-6-磷酸,并通过α-磷酸葡萄糖变位酶(PGM)作用形成葡萄糖-1-磷酸,这是核苷酸糖合成的关键中间体。葡萄糖-1-磷酸可通过UDP-葡萄糖磷酸酶(GalU)形成UDP-葡萄糖;或通过dTDP-葡萄糖焦磷酸化酶(TGP)、dTDP-葡萄糖-4,6-脱水酶(RmlB)和dTDP-4-脱氢鼠李糖-3,5-差向异构酶(TRS)形成dTDP-鼠李糖。果糖通过PEP-PTS转运到细胞质中,并通过果糖激酶磷酸化为果糖-1-磷酸,然后通过磷酸变位酶转化为果糖-6-磷酸。果糖-6-磷酸在葡萄糖胺-6-磷酸脱氨酶、磷酸葡萄糖胺变位酶、葡萄糖胺-1-磷酸的催化下转化为UDP-N-乙酰半乳糖胺。蔗糖通过PEP-PTS转运至细胞质后,磷酸化为蔗糖-6-磷酸,通过蔗糖水解酶(SacA)水解为果糖-6-磷酸和葡萄糖-6-磷酸,通过上述酶的作用形成UDP-N-乙酰半乳糖胺和dTDP-鼠李糖。
表4 嗜热链球菌937核苷酸糖合成相关基因
Table 4 Genes related to nucleotide sugar synthesis of Streptococcus thermophilus 937

位点基因名称EC号产物gene1474lacZ3.2.1.23β-半乳糖苷酶 (β-galactosidase, LacZ)gene1475LacS2.7.1.207乳糖渗透酶(lactose permease, LacS)gene0780glk2.7.1.2葡萄糖激酶 (glucokinase, GlK)gene0841pgm5.4.2.2α-磷酸葡萄糖变位酶 (α-phosphate glucose, Pgm)gene1923galU2.7.7.9UDP-葡萄糖焦磷酸酶 (UDP-glucose pyrophosphatase, GalU)gene1184gene1438galE5.1.3.2UDP-半乳糖-4-差异构酶 (UDP-galactose-4-epimerase, GalE)gene1477gene1478galT2.7.7.12半乳糖1-P尿苷转移酶 (galactose 1-P uridyl transfer-ase, GalT)gene1476galM5.1.3.3半乳糖变位酶 (galactose mutarotase, GalM)gene1479galK2.7.1.6半乳糖激酶 (galactokinase, GalK)gene1283rfbA2.7.7.24dTDP-葡萄糖焦磷酸化酶 (dTDP-glucose-pyrophosphory-lase, RmlA)gene1281rfbB4.2.1.46dTDP-葡萄糖-4,6-脱水酶 (dTDP-glucose 4,6-de-hydratase, RmlB)gene1282rfbC5.1.3.13dTDP-4-脱氢血红蛋白酶3,5-差向异构酶 (dTDP-4-dehydrorhamnose-3,5-epimerase, RmlC)gene1554rfbD1.1.1.133dTDP-4-脱氢血红蛋白还原酶 (dTDP-4 keto-L-rham-nose reductase, RmlD)gene1222pfkA2.7.1.11果糖激酶 (6-phosphofructokinase, Fk)gene0230gpi5.3.1.9磷酸葡萄糖异构酶 (glucose-6-phosphate isomerase, Gpi)gene1815sacAEC.3.2.1.26蔗糖-6-磷酸水解酶 (sucrose 6-phosphate hydrolase, SacA)gene0928glmS2.6.1.16谷氨酰胺-果糖-6-磷酸转氨酶 (glutamine-fructose-6-phosphate transaminase, GFPT)gene1290plmM5.4.2.10磷酸葡萄糖胺变位酶 (phosphoglucosamine mutase, Plmm)gene0621glmU2.7.7.2N-乙酰氨基葡萄糖-1-磷酸尿苷转移酶 (N-acetylglu-cosamine-1-phosphate uridyltransferase, Gpn)gene0598nagB3.5.99.6氨基葡萄糖-6-磷酸脱氨酶 (glucosamine-6-phosphate deaminase, Gpd)
2.2.3 嗜热链球菌937eps基因簇
EPS的合成还需要位于嗜热链球菌染色体上的eps生物合成基因簇的指导来完成组装,聚合和输出。如图3和表5所示,嗜热链球菌937染色体上存在一个合成EPS的基因簇,该基因簇由18个基因组成。包含转录调控基因epsA;磷酸调节模块基因epsBCD,其中epsCD调控EPS链长;编码糖基转移酶基因,其中cpsE(gene1123和gene1121)编码半乳糖糖基转移酶,负责将UDP-半乳糖转移到异戊二烯醇磷酸酯(脂载体) 上,同时基因(gene1120~gene1117)编码糖基转移酶,转移相应的核苷酸糖指导EPS重复单元的形成。其中gene1122因截短成为假基因无法发挥转移酶功能。gene 1113为假想的蛋白质,尚不清楚其功能。gene1115和gene1111分别编码Wzy聚合酶和Wzx翻转酶,负责EPS的输出。在eps基因簇5′上游同样发现编码嘌呤核苷磷酸化酶的高度保守基因deoD,在5′上游发现编码与嗜热链球菌生长相关的基因orf14.9。
表5 嗜热链球菌937 eps基因簇
Table 5 eps gene cluster of Streptococcus thermophilus 937

基因位点长度/bp最优比对一致性产物gene1110576S. thermophilus STCH_06100orf14.9gene11111 257S. thermophilus STCH_06100可移动元素蛋白 (mobile element protein)gene11121 434S. thermophilus STCH_06100Wzx,多糖生物合成蛋白 [Wzx, Polysaccharide biosynthesis protein, CpsM(V)]gene1113186S. thermophilus STCH_06100假定蛋白质 (hypothetical protein)gene1114735S. thermophilus STCH_06100糖基转移酶 (glycosyltransferase)gene1115810S. thermophilus STCH_06100Wzy,多糖生物合成蛋白 [Wzy, Polysaccharide biosynthesis protein, CpsM(V)]gene11161 143S. thermophilus STCH_06100多糖生物合成蛋白[polysaccharide biosynthesis protein, cpsH(V)]gene11171 020S. thermophilus STCH_06100β-1,3-葡糖基转移酶 (β-1,3-glucosyltransferase)gene1118897S. thermophilus STCH_06100α-1,3-L-鼠李糖基转移酶 (α-1,3-L-rhamnosyltransferase)gene1119516S. thermophilus STCH_06100α-1,3-L-鼠李糖基转移酶(α-1,3-L-rhamnosyltransferase)gene1120450S. thermophilus STCH_06100N-乙酰葡糖胺转移酶 (N-acetylglucosamine transferase, CpsG)gene1121384S. thermophilus STCH_06100半乳糖基转移酶 (galactosyl transferase, CpsE)gene1122498S. thermophilus STCH_06100十一碳烯基磷酸半乳糖磷酸转移酶 (undecaprenyl-phosphate galactose phosphotrans-ferase)gene11231 368S. thermophilus STCH_06100半乳糖基转移酶 (galactosyl transferase)gene1124741S. thermophilus STCH_06100酪氨酸蛋白激酶 (tyrosine-protein kinase, EpsD)gene1125693S. thermophilus STCH_0699酪氨酸蛋白激酶跨膜调节剂 (tyrosine-protein kinase transmembrane modulator, EpsC)gene1126732S. thermophilus STCH_06100酪氨酸蛋白磷酸酶 (tyrosine-protein phosphatase, EpsB)gene11271 461S. thermophilus STCH_06100胞外多糖生物合成转录激活剂 (exopolysaccharide biosynthesis transcriptional activator, EpsA)gene1128711S. thermophilus IDCC2201100嘌呤核苷磷酸化酶 (purine-nucleoside phosphorylase,deoD)
结合核苷酸糖合成途径可以发现,嗜热链球菌937可以合成的核苷酸糖包括UDP-葡萄糖、UDP-半乳糖、UDP-鼠李糖及UDP-N-乙酰半乳糖胺,但eps基因簇中没有编码UDP-N-乙酰半乳糖胺转移酶的基因,仅有编码UDP-N-乙酰葡萄糖胺转移酶的基因,这可能使得嗜热链球菌937合成EPS的单糖组份不含有UDP-N-乙酰半乳糖胺,同样的结果在嗜热链球菌ASCC 1275中也可以发现[18]。
2.3 氨基酸对嗜热链球菌937生长及EPS产量的影响
测定嗜热链球菌937在CDM和CDM+HIG中培养10 h过程中OD600和EPS产量变化,结果如图4所示。嗜热链球菌937在CDM和CDM+HIG中的光学密度并无显著差异,仅在5 h时,CDM+HIG中OD600值略高于CDM。发酵3 h后,CDM中EPS产量开始低于CDM+HIG中;发酵5 h后CDM+HIG中EPS产量达到最高为58.77 μg/mL,而CDM中EPS产量仅为38.84 μg/mL。表明在CDM中提升3种氨基酸浓度后,嗜热链球菌937 EPS的产量得到了显著提升。
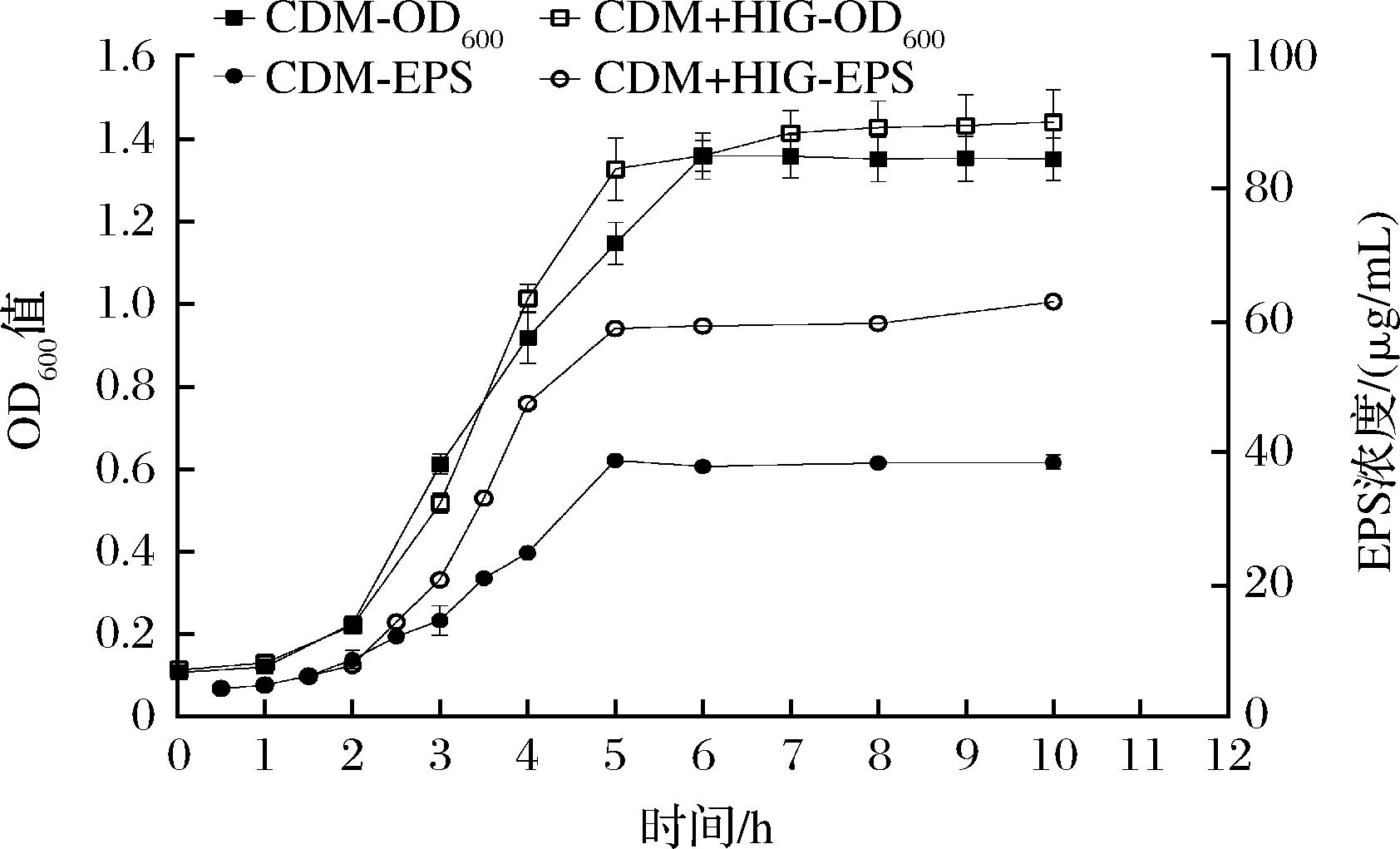
图4 氨基酸对嗜热链球菌937生长及EPS产量影响
Fig.4 Effects of amino acids on the growth and EPS production of Streptococcus thermophilus 937
注:CDM+HIG=CDM中提升组氨酸、异亮氨酸和谷氨酸浓度为15 mmol/L。
2.4 氨基酸对嗜热链球菌937 EPS性质影响
分别对CDM和CDM+HIG分离得到的EPS进行分子质量和单糖组份测定,结果如表6所示。在CDM+HIG中分离得到的EPS分子质量为1 086 844 Da,大于CDM中。2种培养基中EPS的单糖成分相同均为氨基半乳糖、鼠李糖、氨基葡萄糖、半乳糖和葡萄糖,但单糖组成的摩尔比存在差异。CDM+HIG中EPS的氨基半乳糖、鼠李糖和氨基葡萄糖摩尔比大于CDM,而半乳糖和葡萄糖摩尔比小于CDM。这表明提升3种氨基酸的浓度后,不仅影响了嗜热链球菌937的EPS产量,同时也影响了其分子质量和单糖组成。
表6 氨基酸对嗜热链球菌937 EPS分子质量及单糖组份影响
Table 6 Molecular mass and monomer composition of the f-EPS produced by Streptococcus thermophilus 937 in different chemically-defined medium

组别分子质量/kDa摩尔比氨基半乳糖鼠李糖氨基葡萄糖半乳糖葡萄糖CDM884.14±13.310.225±0.0060.174±0.0180.030±0.0030.265±0.0200.306±0.007CDM+HIG1 086.68±26.080.232±0.0020.212±0.0050.053±0.0010.214±0.0030.289±0.007
此外,嗜热链球菌937 EPS单糖组份中所含有的鼠李糖、半乳糖及葡萄糖与其全基因序列所得结论一致,但在单糖组份中还发现了氨基葡萄糖和氨基半乳糖,目前所报道的乳酸菌EPS中较少涉及这2种单糖组份,而在Citrolpacter sp.[19]、Staphylococcus aureus[20]、Staphylococcus saprophyticus[21]的EPS中可以发现这2种单糖的存在,推测可能与检测方法有关,乳酸菌EPS单糖组成的测定方法主要依靠高效液相或是气相质谱法,难以检测到氨基糖,而用离子色谱仪可以检测到,但这2种单糖组份的合成方式以及相应的糖基转移酶还需要进一步的研究来明确。
2.5 氨基酸对嗜热链球菌937 EPS合成关键基因影响
为了更进一步了解3种氨基酸是如何对嗜热链球菌937 EPS产量产生影响的,在3种培养基中分别提取了培养至3 h和5 h的嗜热链球菌937菌体的RNA,用以评估EPS生物合成相关基因的转录水平。通过荧光实时定量PCR对eps基因簇上相关基因:epsA、epsB、epsC、epsD的转录水平进行测定,结果如图5所示。由图5-A可知,发酵至3 h时,CDM+HIG中epsABCD、的转录水平显著上调(P<0.000 1;P<0.05)。由图5-B可知,发酵至5 h时,各基因转录水平无显著差异,这可能与细胞生长至稳定期有关。

A-培养3 h;B-培养5 h
图5 嗜热链球菌937在CDM和CDM+HIG中胞外多糖合成关键基因转录变化
Fig.5 The transcriptional change in the key genes of exopolysaccharide biosynthesis by Streptococcus thermophilus 937 in CDM and CDM+HIG respectively
注:样品通过双侧t检验分析;*代表P<0.05;****代表P<0.000 1。CDM+HIG=CDM中提升组氨酸、异亮氨酸和谷氨酸浓度为15 mmol/L。
还较少有文献报道氨基酸对epsABC转录水平具有调控作用。相关的研究结果发现,嗜热链球菌LY03在复杂培养基中培养时,碳氮源的比例会影响其EPS的产量和分子质量,且EPS分子质量随氮源浓度的上升而下降[6]。嗜热链球菌ASCC 1275在蔗糖为碳源的培养基中EPS产量最高,其eps1C1D2C2D基因高度表达,此外,还观察到与精氨酸代谢、丙氨酸/天冬氨酸和谷氨酸代谢相关的基因和蛋白质过度表达[22-23]。但在牛乳中添加乳清蛋白后,虽然EPS产量达到了最大值,但并未发现EPS合成相关基因转录水平上升,仅有精氨酸合成相关的基因转录和蛋白表达水平上升[24],这可能是由于培养基不同造成的。
3 结论
通过嗜热链球菌937的基因组测序结果发现了参与碳水化合物转运、核苷酸糖合成和EPS生物合成的基因。基因组分析表明,嗜热链球菌937存在转运葡萄糖、蔗糖、海藻糖、甘露糖及纤维二糖的特异性PTS转运系统和乳糖渗透酶;菌株具有合成EPS的糖前体的UDP-葡萄糖、UDP-半乳糖和dTDP-鼠李糖的潜力;同时菌株染色体上存在一个指导EPS合成的基因簇。与CDM相比,当提升组氨酸、异亮氨酸和谷氨酸浓度至15 mmol/L后,嗜热链球菌937的EPS产量和分子质量提升且单糖组成比例发生改变,这与eps基因簇相关基因的转录水平上调有关。本研究结果在基因水平上为嗜热链球菌EPS生物合成提供了更好的解释。
[1] SVENSSON M, WAAK E, SVENSSON U, et al.Metabolically improved exopolysaccharide production by Streptococcus thermophilus and its influence on the rheological properties of fermented milk[J].Applied and Environmental Microbiology, 2005, 71(10):6398-6400.
[2] BOELS I C, VAN KRANENBURG R, HUGENHOLTZ J, et al.Sugar catabolism and its impact on the biosynthesis and engineering of exopolysaccharide production in lactic acid bacteria[J].International Dairy Journal, 2001, 11(9):723-732.
[3] ZHOU Y, CUI Y H, QU X J.Exopolysaccharides of lactic acid bacteria:Structure, bioactivity and associations:A review[J].Carbohydrate Polymers, 2019, 207:317-332.
[4] CUI Y H, JIANG X, HAO M Y, et al.New advances in exopolysaccharides production of Streptococcus thermophilus[J].Archives of Microbiology, 2017, 199(6):799-809.
[5] ZEIDAN A A, POULSEN V K, JANZEN T, et al.Polysaccharide production by lactic acid bacteria:From genes to industrial applications[J].FEMS Microbiology Reviews, 2017, 41(Supp_1):S168-S200.
[6] DEGEEST B, DE V L.Indication that the nitrogen source influences both amount and size of exopolysaccharides produced by Streptococcus thermophilus LY03 and modelling of the bacterial growth and exopolysaccharide production in a complex medium[J].Applied and Environmental Microbiology, 1999, 65(7):2863-2870.
[7] ZISU B, SHAH N P.Effects of pH, temperature, supplementation with whey protein concentrate, and adjunct cultures on the production of exopolysaccharides by Streptococcus thermophilus 1275[J].Journal of Dairy Science, 2003, 86(11):3405-3415.
[8] LI D, LI J X, ZHAO F, et al.The influence of fermentation condition on production and molecular mass of EPS produced by Streptococcus thermophilus 05-34 in milk-based medium[J].Food Chemistry, 2016, 197(Part A):367-372.
[9] DE VUYST L, VANDERVEKEN, VEN V D, et al.Production by and isolation of exopolysaccharides from Streptococcus thermophilus grown in a milk medium and evidence for their growth-associated biosynthesis[J].Journal of Applied Microbiology, 1998, 84(6):1059-1068.
[10] MOZZI F, GIORI G, OLIVER G, et al.Exopolysaccharide production by Lactobacillus casei.I:Influence of salts[J].Milchwissenschaft-Milk Science International, 1995, 50:186-188.
[11] GROBBEN G J, CHIN-JOE I, KITZEN V A, et al.Enhancement of exopolysaccharide production by Lactobacillus delbrueckii subsp.bulgaricus NCFB 2772 with a simplified defined medium[J].Applied and Environmental Microbiology, 1998, 64(4):1333-1337.
[12] LOOIJESTEIJN P J, BOELS I C, KLEEREBEZEM M, et al.Regulation of exopolysaccharide production by Lactococcus lactis subsp.cremoris by the sugar source[J].Applied and Environmental Microbiology, 1999, 65(11):5003-5008.
[13] DE VUYST L, ZAMFIR M, MOZZI F, et al.Exopolysaccharide-producing Streptococcus thermophilus strains as functional starter cultures in the production of fermented milks[J].International Dairy Journal, 2003, 13(8):707-717.
[14] ASLIM B, ![]() Z N, BEYATLI Y, et al.Exopolysaccharide production by Lactobacillus delbruckii subsp. bulgaricus and Streptococcus thermophilus strains under different growth conditions[J].World Journal of Microbiology and Biotechnology, 2005, 21(5):673-677.
Z N, BEYATLI Y, et al.Exopolysaccharide production by Lactobacillus delbruckii subsp. bulgaricus and Streptococcus thermophilus strains under different growth conditions[J].World Journal of Microbiology and Biotechnology, 2005, 21(5):673-677.
[15] WA Y C, ZHANG C C, SUN G L, et al.Effect of amino acids on free exopolysaccharide biosynthesis by Streptococcus thermophilus 937 in chemically defined medium[J].Journal of Dairy Science, 2022, 105(8):6460-6468.
[16] LETORT C, JUILLARD V.Development of a minimal chemically-defined medium for the exponential growth of Streptococcus thermophilus[J].Journal of Applied Microbiology, 2001, 91(6):1023-1029.
[17] DUBOIS M, GILLES K A, HAMILTON J K, et al.Colorimetric method for determination of sugars and related substances[J].Analytical Chemistry, 1956, 28(3):350-356.
[18] WU Q L, TUN H M, LEUNG F C, et al.Genomic insights into high exopolysaccharide-producing dairy starter bacterium Streptococcus thermophilus ASCC 1275[J].Scientific Reports, 2014, 4:4974.
[19] HIA H C, JANG J H, IKE M, et al.Improved borate method for the rapid distinction of glucosamine and galactosamine in an exopolysaccharide produced by Citrolpacter sp.[J].Biotechnology Letters, 2004, 26(16):1307-1311.
[20] YUDINA O N, GENING M L, TSVETKOV Y E, et al.Synthesis of five nona-β-(1→6)-D-glucosamines with various patterns of N-acetylation corresponding to the fragments of exopolysaccharide of Staphylococcus aureus[J].Carbohydrate Research, 2011, 346(7):905-913.
[21] ZAMIL S S, AHMAD S, CHOI M H, et al.Production of poly-N-acetylglucosamine by Staphylococcus saprophyticus BMSZ711:Characterization and production optimization[J].Bioresource Technology, 2010, 101(18):7188-7191.
[22] PADMANABHAN A, TONG Y, WU Q L, et al.Transcriptomic insights Into the growth phase- and sugar-associated changes in the exopolysaccharide production of a high EPS-producing Streptococcus thermophilus ASCC 1275[J].Frontiers in Microbiology, 2018, 9:1919.
[23] PADMANABHAN A, TONG Y, WU Q L, et al.Proteomic analysis reveals potential factors associated with enhanced EPS production in Streptococcus thermophilus ASCC 1275[J].Scientific Reports, 2020, 10(1):807.
[24] WU Q L, CHU H, PADMANABHAN A, et al.Functional genomic analyses of exopolysaccharide-producing Streptococcus thermophilus ASCC 1275 in response to milk fermentation conditions[J].Frontiers in Microbiology, 2019, 10:1975.