大曲是一种典型的“多酶多菌”的微生态制品,中国名优白酒的生产大多采用大曲作为发酵剂。白酒发酵是大曲微生物和环境微生物共同作用的过程,特别是大曲进入固态酿酒体系后,还存在着“二次制曲”的过程[1];制曲过程中,大曲微生物生长繁殖代谢形成的香味成分,跟随曲粉进入糟醅体系,随蒸馏提取进入酒中,成为酒体中的呈香呈味成分[2]。在曲与酒以及风味特征的关系上,酒曲具有先导地位,“曲乃酒之骨”,就说明了酒曲对白酒质量的重要性[2]。所以了解大曲中微生物的群落结构对解释白酒的生产机理是很重要的。
从科赫发明平板分离法至今的一百多年中,人类利用传统培养法对微生物产生认知,但是这种认识仅占微生物种类的不到1%,对全面认识微生物而言是微小的。随着现代分子生物学技术的快速发展,免培养方法可以跨越微生物培养过程开展微生物研究,包括磷脂脂肪酸技术(phospholipid fatty acid,PLFA)[3]、聚合酶链式反应(polymerase chain reaction,PCR)-变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)技术[4]、PCR-单链构象多态性(Single strand conformation polymorphism,SSCP)技术[5]、基因克隆文库技术[6]、高通量测序(high-throughput sequencing,HTS)技术等。目前最常用的微生物免培养方法涉及高通量、大规模并行的基于扩增子的测序和基于公开可用的参考数据库进行后续的分类学归属[7-8]技术。HTS[9]可以同时对多个样品中的微生物进行测序分析,而且具有通量高、速度快、准确度高、实时检测、数据信息量大等特点,能在很短时间内获取大量的数据,是最常用的测序技术。
通过现代分子生物学技术,人们对白酒生产过程中主要功能微生物的分离鉴定和微生物群落结构的研究越来越深入,也有了越来越多的成果和应用。目前,研究大曲微生物的技术已经非常成熟和广泛,只是在方法学上存在一些差异。通过HTS挖掘大曲微生物的生物信息时提取的DNA质量至关重要,这是对DNA提取方法的一个巨大考验。量化DNA的提取效果,能对不同DNA提取方法起到重要的指导作用。闫亮珍等[10]对汾酒大曲中微生物DNA提取方法进行了优化,发现酶物理化学法提取获得的高质量DNA能全面反映汾酒酿造过程中的微生物群落结构多样性。葛媛媛等[11]从常温研磨、蛋白酶K和引入RNase的角度对传统CTAB法进行改良,并结合试剂盒提取法,提出了一种高效提取高温大曲微生物总DNA的方法。张春辉等[12]研究了5种基于不同裂解机理的提取方法对高温大曲中总基因组提取效果的影响,结果表明,基于SDS-酶法裂解原理的提取方法获得的结果最优,获得的总基因组片段大约为21 kbp,纯度为较好,能不经纯化直接用于16S rDNA的扩增。潘明等[13]对浓香型大曲中微生物总DNA的PCR扩增进行了优化,结果发现利用SDS-酶法能够最大量地提取曲药中总DNA。尽管现在有许多针对大曲DNA的提取方法,但是对于中温和中高温的浓香型大曲的提取方法还有待进一步优化,如何使大曲中微生物细胞裂解、充分释放DNA,有效去除杂质(植物、腐殖酸、蛋白质、酚类化合物等[14]),得到高纯度的DNA可能是解构浓香型大曲微生物群落的重要影响因素。
目前,大多数对于大曲DNA提取方法的探究都是基于提取方法本身,没有讨论样品前处理对DNA提取的微生物产量、微生物多样性以及微生物物种差异的影响,所以本文研究了原位和洗脱2种前处理对2种DNA提取方法的影响。对2种有代表性的大曲进行了分类取样,对大曲样品采取原位或洗脱的前处理,采用SDS-based提取法或试剂盒提取法对大曲微生物DNA进行提取,通过对DNA提取质量、微生物群落多样性指数及物种组成的差异展开评估,以确定适合于大曲微生物基因组DNA的提取方法,为在分子生物学水平上研究白酒大曲微生物资源打下一定基础。
1 材料与方法
1.1 材料与试剂
本研究所用的大曲样本都以小麦为原料,编号为TP的大曲样本经过28 d的安曲、培菌发酵、翻曲、转化发酵等过程,入库贮存3个月,采集于遂宁某浓香型酒厂;编号为LJ的大曲样本经过40 d的堆曲、二次翻曲、转曲等堆积培养过程,出房后曲库贮存3个月,采集于泸州某酱香型白酒厂。所有样品均是取混合粉粹的生产用曲。取样后,保存在-80 ℃,用于后续提取DNA。
1.2 仪器与设备
S1000型PCR仪,伯乐生命医学产品有限公司;JY600C型电泳仪,北京君意东方电泳设备有限公司;Tanon-1600型凝胶成像系统,上海天能科技有限公司;LYNX4000型高速冷冻离心机、ND-ONE-W型Nano drop One超微量紫外分光光度计,上海赛默飞世尔有限公司。
1.3 实验方法
带有Barcode标记的扩增引物由上海美吉生物医药科技有限公司提供;PCR扩增所用Taq酶(Taq Master Mix)、dNTPs等,近岸蛋白质科技有限公司(novoprotein);FastBeat土壤DNA提取试剂盒,北京艾德莱生物科技有限公司。
1.3.1 大曲DNA的提取前处理方法和提取方法
本文对大曲DNA的提取是将2种前处理(原位处理和洗脱处理)和2种提取方法(SDS-based提取法和试剂盒提取法)相结合共形成4种大曲DNA提取方法:原位SDS-based提取法、洗脱加SDS-based提取法、原位试剂盒法和洗脱加试剂盒法。
洗脱的前处理方法参考文献[15],称取5 g左右的大曲样本,放置于50 mL事先灭菌的离心管内,加入pH 7.0的磷酸缓冲液(PBS)20 mL,用振荡器振荡5~10 min,静置2~3 min,6 000 r/min离心10 min,转移上清液至新的50 mL离心管。在含有大曲的离心管中再次加入20 mL磷酸缓冲液洗涤菌体,离心获得上清液。重复上述步骤2次。合并上述上清液并在10 000 r/min下离心10 min,获得大曲中洗脱下来的微生物。
原位处理,即大曲样品未经过洗脱前处理,直接从大曲样品中裂解细胞,其DNA的SDS-based提取法参照文献[16]的方法。
试剂盒提取法:称取0.2~0.3 g左右大曲样本或将洗脱处理得到的样本置于FastBeat土壤DNA提取试剂盒配套的离心管内,后续的实验步骤参考试剂盒说明书流程实施。
1.3.2 DNA提取得率的计算
粗核酸沉淀经70%乙醇洗涤,并重悬于无菌去离子水中,得到100 μL的最终体积,该溶液用Nano Drop One超微量紫外分光光度计检测DNA的浓度,按照公式(1)计算DNA的得率:
(1)
式中:Y,DNA得率,ng/g;c,DNA溶液质量浓度,ng/μL;V,DNA溶液体积,μL;m,提取DNA时称取的大曲质量,g。
1.3.3 扩增子文库的构建、测序及荧光定量PCR
每个样本做3个重复的扩增子文库,分别针对细菌16S rRNA基因的V3-V4区和真菌ITS1区构建扩增子文库。引物对为515F(5′-GTGYCAGCMGCCGCGGTAA-3′)/806R(5′-GGACTACHVGGGTWTCTA-AT-3′)[17]和ITS1F(5′-CTHGRTCATTTAGAGGAASTAA-3′)/ITS2R(5′-GCTRCGYTCTTCATCGWTGC-3′)[18],分别用于构建细菌和真菌核糖体基因文库。PCR的扩增体系为:在50 μL反应体系中加入25 μL Taq Master Mix(2×)、含有不同Barcode标记的正反向引物各1.0 μL、模板DNA 1 μL、补双蒸水至50 μL;细菌16S rRNA基因的PCR扩增程序为:95 ℃预变性4 min(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s),循环27次,72 ℃终延伸10 min,10 ℃保温。真菌ITS1区基因的扩增体系为:94 ℃预变性4 min,(94 ℃变性30 s,53 ℃退火30 s,72 ℃延伸45 s)循环36次,72 ℃终延伸10 min,10 ℃保温。每个扩增子文库分别做3次平行的PCR技术重复,将同一文库的PCR产物合并,通过2%的琼脂糖凝胶(含EB)120 V电泳30 min,凝胶成像仪观察PCR扩增条带的有无,而后再进行切胶回收及定量检测,以提高PCR产物的稳定性。构建的扩增子文库通过上海美吉生物医药科技有限公司的Illumina Miseq PE300测序平台上机测序。
定量实时PCR(quantitative real-time PCR,qPCR)采用染料法(SYBR Green I),选择引物对338 F(5′-ACTCCTACGGGAGGCAGCAG-3′)/806 R(5′-GGACTACHVGGGTWTCTAAT-3′)[19]和ITS1F(5′-CTTGGTCATTTAGAGGAAGTAA-3′)/ITS2R(5′-GCTGCGTTCTTCATCGATGC-3′)[20]用于确定细菌16S rRNA基因和真菌ITS基因的拷贝数。每个样本做3次重复,相关标准品的制备和qPCR定量实验由上海美吉生物医药科技有限公司完成。
1.4 高通量测序数据的分析
测序数据的分析通过上海美吉生物医药科技有限公司的i-sanger云平台完成。高通量数据的质控以及处理步骤见参考文献[21]。OTU注释时去除了异常序列(如未知、古细菌、叶绿体和线粒体序列)[22],利用OriginPro 2023b(学习版)以及美吉生信云平台(http://www.majorbio.com/)绘制相关图形(多物种差异检验柱形图)。
原始数据NMDC40044159存储在国家微生物科学数据中心(NMDC),链接为https:/ /nmdc.cn/resource/genomics/sra/detail /NMDC40044159。
2 结果与分析
2.1 不同DNA提取方法的DNA得率、纯度的比较
本试验为了减少由操作引起的误差,每个DNA提取方法进行3次平行的提取操作,合并后的DNA通过Nano Drop One超微量紫外分光光度计和凝胶电泳检测DNA提取质量。大曲DNA样本信息及提取得率等信息见图1。
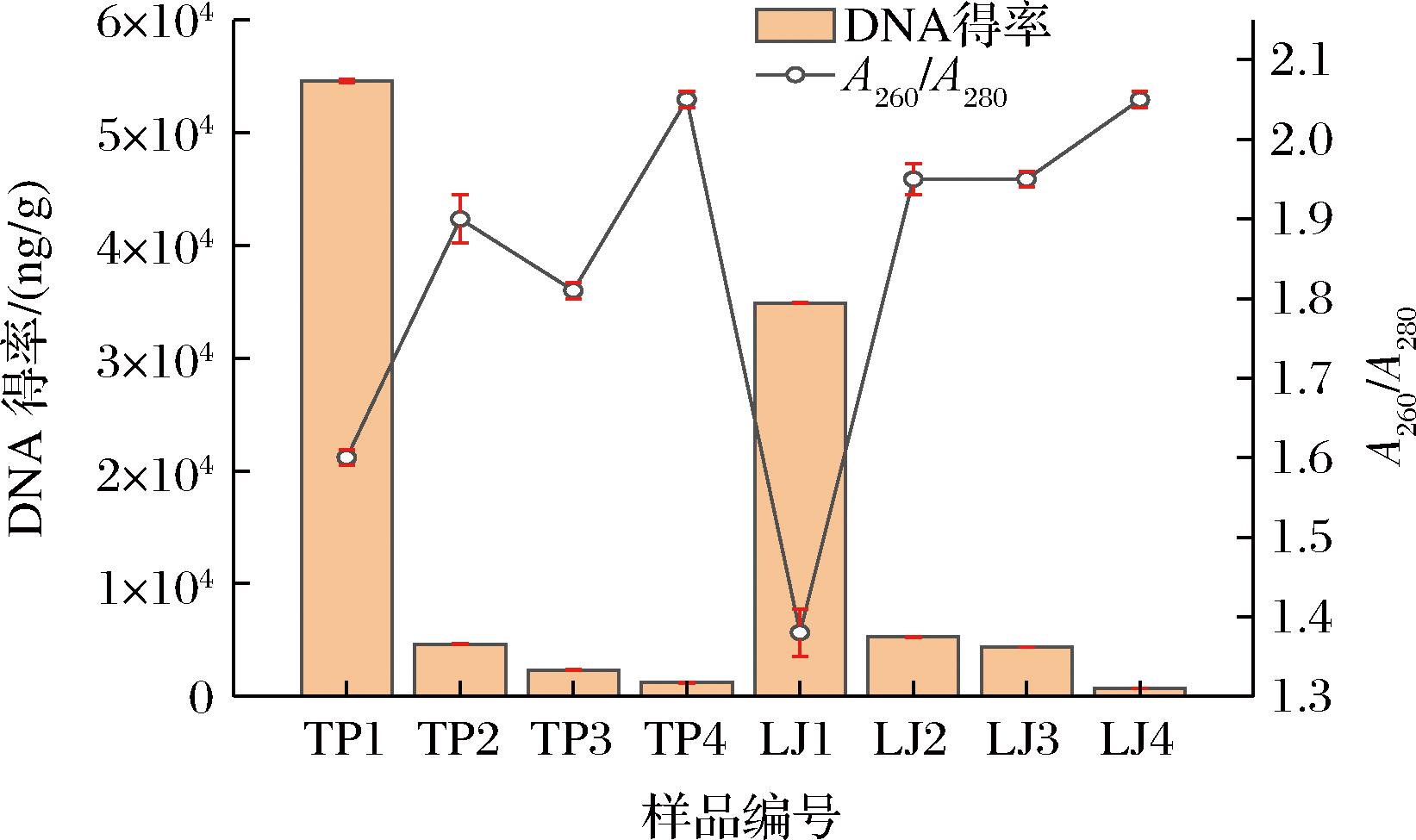
图1 不同DNA提取方法下的DNA提取得率、纯度
Fig.1 DNA extraction yield and purity under different Daqu DNA extraction methods
注:TP为浓香型大曲样本,LJ为酱香型大曲样本;样品编号中1表示原位SDS-based提取法,2表示洗脱加SDS-based提取法,3表示原位试剂盒法,4表示洗脱加试剂盒法。
从DNA得率柱状图的比较可以看出,DNA提取得率的排序分别是原位SDS-based提取法>洗脱加SDS-based提取法>原位试剂盒法>洗脱加试剂盒法。总体而言,SDS-based提取法的DNA提取得率高于试剂盒提取法,DNA提取得率在它们二者之间存在10~102的量级差异。原位SDS-based提取法的DNA得率明显高于其他3种DNA提取方法,LJ样本和TP样本原位SDS-based提取法的DNA提取得率分别为3.49×104 ng/g和5.46×104 ng/g,其他3种方法的DNA提取得率为7.44×102~5.27×103 ng/g。从DNA纯度折线图的比较也可以看出,纯度变化从高到低依次为洗脱加试剂盒法>洗脱加SDS-based提取法>原位试剂盒法>原位SDS-based提取法,经洗脱处理得到的DNA的纯度高于原位处理的DNA纯度。原位SDS-based提取法的DNA纯度较低,A260/A280为1.4~1.6,说明提取的DNA中有较多的蛋白质污染;而其余3种DNA提取法的A260/A280值均为1.81~2.05,说明洗脱和试剂盒提取能够提高基因组DNA的纯度。由于上述4种DNA提取方法提取的DNA中不仅含有大曲微生物的DNA,也可能含有制曲原料如小麦的DNA,因此上述结果并不一定意味着该方法对大曲微生物DNA实现了有效提取,后续需要结合qPCR定量结果和扩增子文库加以评估。原位SDS-based提取法提取的DNA中包含非微生物来源的DNA,这一点在后续的qPCR定量结果中得到了验证。
2.2 不同DNA提取方法下的微生物生物量的比较
不同DNA提取方法对大曲基因组DNA提取的微生物生物量,结果如表1所示。
表1 不同DNA提取方法下的DNA定量结果
Table 1 DNA quantitative results under different Daqu DNA extraction methods
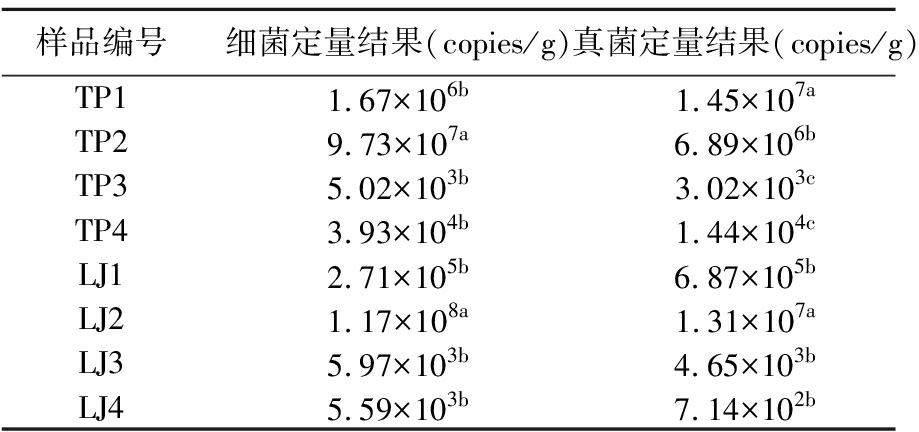
样品编号细菌定量结果(copies/g)真菌定量结果(copies/g)TP11.67×106b1.45×107aTP29.73×107a6.89×106bTP35.02×103b3.02×103cTP43.93×104b1.44×104cLJ12.71×105b6.87×105bLJ21.17×108a1.31×107aLJ35.97×103b4.65×103bLJ45.59×103b7.14×102b
注:不同小写字母表示差异显著(P<0.05)。
从表1的qPCR定量结果可以看出,SDS-based提取法的定量结果明显高于试剂盒提取法,微生物生物量存在101~106的量级差异。SDS-based提取法定量的细菌和真菌的生物量达到105~108 copies/g,但洗脱加SDS-based提取法的微生物定量结果要高于或略等于原位SDS-based提取法,这二者之间的生物量差异在101~103量级;试剂盒提取法的定量结果最低,细菌和真菌的生物量只有102~104 copies/g。对TP样本的细菌/真菌以及LJ样本的细菌/真菌分别进行显著性分析,结果显示在LJ样本中,细菌和真菌在洗脱加SDS-based 提取法中的微生物定量结果与其他3种DNA提取方法的微生物定量结果差异均显著,而3种方法之间差异不显著。在TP样本中,原位SDS-based提取法、原位试剂盒法、洗脱加试剂盒法的细菌微生物定量结果差异不显著,这三者与洗脱加SDS-based提取法中的微生物定量结果差异显著;真菌微生物定量结果显示,原位SDS-based提取法和洗脱加SDS-based 提取法微生物定量结果之间差异显著,原位试剂盒法和洗脱加试剂盒法的微生物定量结果之间差异不显著。对比图1的数据可以看出,虽然原位SDS-based提取法可以提取大量的基因组DNA,但其qPCR定量结果显示的微生物生物量不总是比洗脱加SDS-based提取法的定量结果高,说明原位SDS-based提取法获取的DNA中含有大量在选定的真菌引物对条件下无法扩增的DNA,如原料小麦来源的植物DNA[23]。
2.3 不同DNA提取方法下的微生物扩增子文库的比较
通过扩增子序列分析,并对原始序列进行质控和去除嵌合体处理,细菌16S rDNA基因扩增子文库的有效序列条数为34 955~101 081,共计1 396 580条,鉴定出平均63 480个高质量序列,并且获得5 102个具有97%的相似性操作分类单元(operational taxonomic units,OTU);真菌ITS基因扩增子文库的有效序列条数为35 141~109 107,共计1 532 883条,鉴定出平均63 870个高质量序列,并且获得2 508个OTU。对扩增子文库进行ANOSIM分析,采用weighted_unifrac距离算法,除去LJ4_3细菌16S rDNA扩增子文库和LJ3_1细菌16S rDNA扩增子文库以及ZLJ4_2真菌ITS扩增子文库后(不参与后续分析):TP样本细菌文库ANOSIM=1.000;LJ样本细菌文库ANOSIM=0.872;TP样本真菌文库ANOSIM=0.867;LJ样本真菌文库 ANOSIM=0.878;不同DNA提取方法下的微生物扩增子文库具有显著性差异(P=0.001)。所有样本按最小样本序列数抽平处理,无论是细菌还是真菌,基于OTU数的稀疏曲线接近饱和平台,表明本研究中的测序深度足以代表样品中的微生物多样性,足够反映群落的真实性[24],能够满足后续的分析需求。TP4中2个细菌文库样本TP4_1和TP4_3测序失败,仅获得TP4_2数据。
2.3.1 不同DNA提取方法下扩增子文库的Shannon多样性指数的比较
不同DNA提取方法构建的细菌16S rDNA扩增子文库和真菌ITS扩增子文库的Shannon多样性指数见图2。
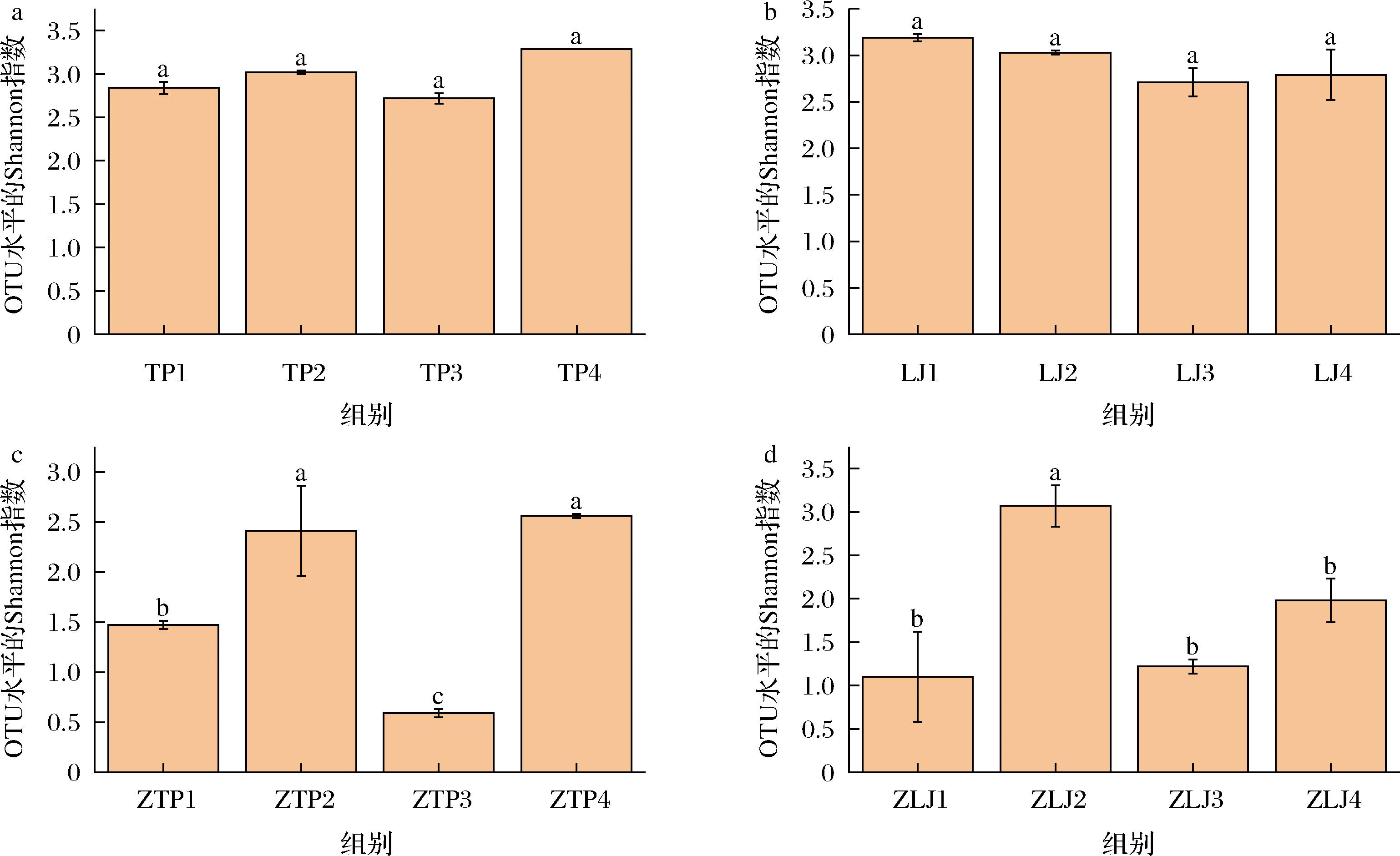
a-TP样本细菌16S rDNA扩增子文库指数组间差异检验;b-LJ样本细菌16S rDNA扩增子文库指数组间差异检验;c-TP样本真菌ITS扩增子文库指数组间差异检验;d-LJ样本真菌ITS扩增子文库指数组间差异检验
图2 不同扩增子文库的Shannon指数的比较
Fig.2 Comparison of Shannon index of different amplicon libraries
注:不同小写字母表示差异显著(P<0.05)为与细菌扩增子文库的标注区分,真菌扩增子文库标注加上首字母Z。
图2中,对不同扩增子文库的Shannon指数指数进行分析,整体而言,细菌16S rDNA扩增子文库的Shannon指数均高于相同处理的真菌ITS扩增子文库的Shannon指数。对细菌16S rDNA扩增子文库来说,TP样本(Shannon指数为2.72~3.29)和LJ样本的(Shannon指数为2.71~3.19)细菌16S rDNA扩增子文库内Shannon指数差异不显著(P>0.05),说明4种DNA提取方法对2种大曲样本的细菌DNA提取均实用。对真菌ITS扩增子文库来说,洗脱处理的Shannon指数(1.98~3.07)明显高于原位处理的Shannon指数(0.59~1.47),除TP样本的洗脱加SDS-based提取法与洗脱加试剂盒法Shannon指数差异不显著外,洗脱加SDS-based提取法与其他方法间的Shannon指数差异显著(P<0.05)。还可观察到不同DNA提取方法对真菌扩增子文库指数组间影响较大,表明洗脱处理对真菌文库的构建有益。
2.3.2 不同DNA提取方法下扩增子文库的物种差异分析
图3多物种差异检验柱形图展示了不同DNA提取方法间对OTUs提取的平均相对丰度的差异情况,不同颜色的柱子表示不同的DNA提取方法。如图3所示,采用Kruskal-Wallis H test检验方法,找到了丰度均值总和前20且具有显著性差异的OTUs。对这些丰度均值总和前20的OTUs来说,在不同DNA提取方法下它们的平均相对丰度参差不齐,说明不同的DNA提取方法对不同OTUs的提取程度不同。对细菌16S rDNA扩增子文库来说,大多数OTUs在不同DNA提取方法下存在丰度差异,多数OTUs的4种颜色的柱形图都有出现,说明它们大多数能在不同DNA提取方法中得到一定程度的有效提取,这和Shannon指数的趋势是相验证的。如图3-a,对TP样本的OTU674来说,原位试剂盒法获得的平均相对丰度为30.80%,远超原位SDS-based提取法的获得的平均相对丰度(1.03%);同时,在图3-b中,对OTU307来说,原位SDS-based提取法获取的平均相对丰度为18.08%,该比例为LJ样本中原位SDS-based提取法中丰度最高的存在,其次为原位试剂盒法(10.86%),最低的是洗脱加试剂盒法,平均相对丰度仅为1.44%;在图3-a和图3-b中,OTU207在洗脱加SDS-based提取法中平均相对丰度都是最高的(TP样本:5.18%,LJ样本:5.75%),而原位SDS-based提取法和原位试剂盒法获得的平均相对丰度分别为0.05%、0.38%(TP样本)和0.80%、0.19%(LJ样本)。以上结果表明,相同的DNA提取方法对不同微生物DNA的提取存在差异,如原位试剂盒法对不同OTUs获取的平均相对丰度,虽波动范围很大,但能提取到。
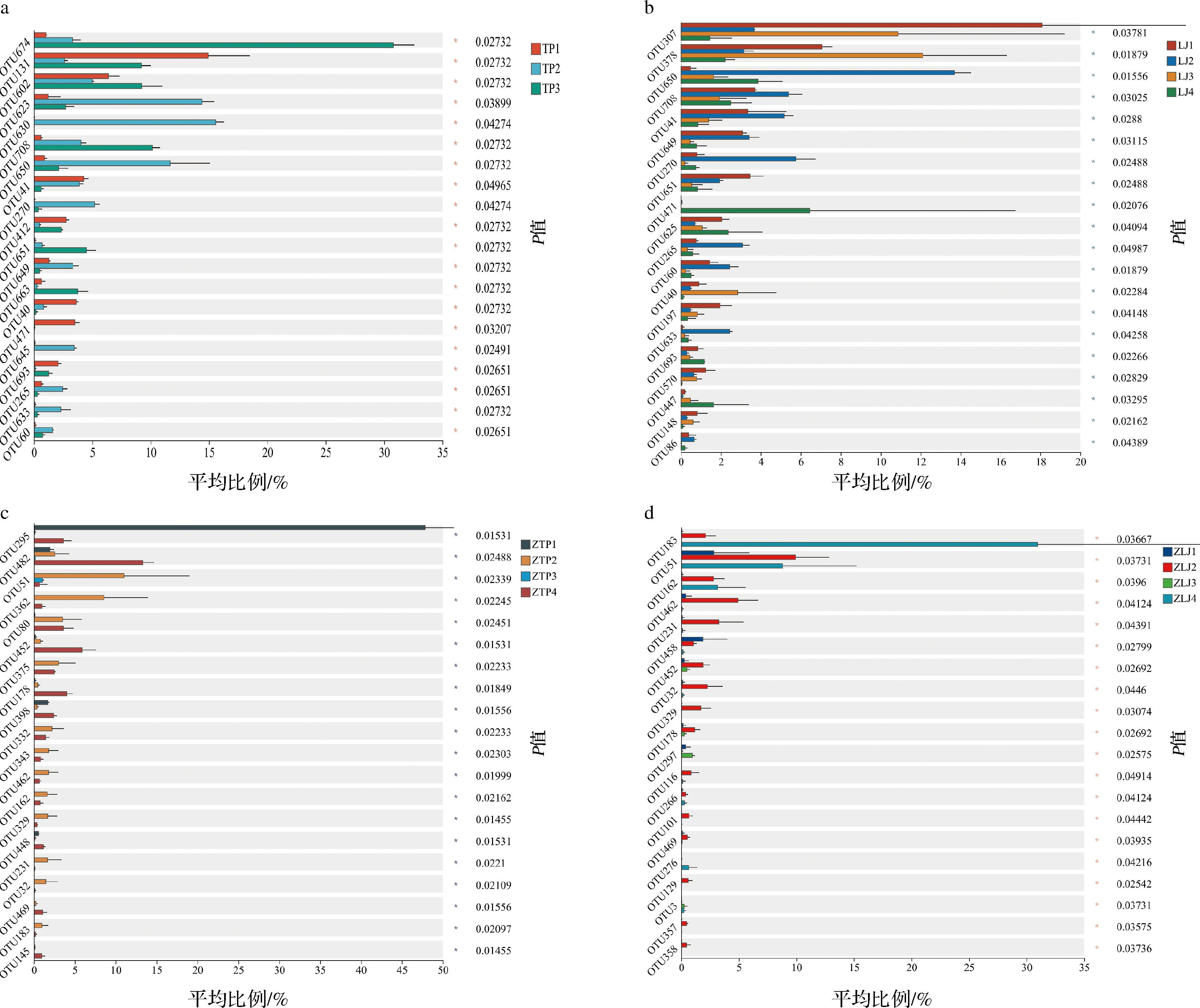
a-TP样本细菌16S rDNA扩增子文库OTU级多物种差异检验柱形图物种组成图;b-LJ样本细菌16S rDNA扩增子文库OTU级多物种差异检验柱形图;c-TP样本真菌ITS扩增子文库OTU级多物种差异检验柱形图;d-LJ样本真菌ITS扩增子文库OTU级多物种差异检验柱形图
图3 不同DNA提取方法下的扩增子文库多物种差异检验柱形图
Fig.3 Multi-species difference test of amplicon library under different DNA extraction methods column diagram
注:TP4中2个细菌文库样本TP4_1和TP4_3测序失败,仅获得TP4_2数据;*表示 0.01<P<0.05。
真菌ITS扩增子文库呈现出与细菌16S rDNA扩增子文库不一样的情况,对这些丰度均值总和前20的真菌OTUs来说,洗脱处理能提高它们的平均相对丰度,并在不同DNA提取方法下它们的平均相对丰度还呈现有无的趋势。在图3-c和图3-d中,可以明显观察到,多数真菌OTUs经由洗脱处理获得的平均相对丰度高于原位处理获得的平均相对丰度。对真菌OTUs的提取,如图3-c所示,TP样本中,OTU332、OTU343以及OTU462等,洗脱处理获得的平均相对丰度依次为:3.62%、2.57%以及2.42%,而原位处理的平均相对丰度在这几个OTU中,可以直接忽略不计;在图3-d的LJ样本中,还能观察到,洗脱加SDS-based提取法优于洗脱加试剂盒法,如OTU32,洗脱加SDS-based提取法的平均相对丰度为2.27%,洗脱加试剂盒法的平均相对丰度为0.14%,以上由多物种差异检验柱形图对真菌ITS扩增子文库的分析与图2-c和图2-d的Shannon指数的变化趋势也是相验证的,这也再次说明了洗脱处理对真菌的提取有益。
3 结论与讨论
在白酒生产过程中,高通量测序技术为微生物的研究带来了极大的方便,但微生物群落的表征会受到样品收集过程中的重复性和代表性、DNA提取方法、引物的选择性、建库过程的PCR偏差、测序平台、数据处理和统计分析的不同而产生一定的差异[25],其中高效的DNA提取方法是准确表征样本中微生物群落结构的关键。在过去的研究中,倪峥飞[15]在提取醋醅时采用了洗脱处理,提高了基因组DNA的纯度和浓度;宋培勇等[26]和洪桂云等[27]都利用PBS磷酸缓冲液对活性污泥进行洗脱,提高了土壤的基因组DNA纯度,其他的一些研究表也都表明针对样本的洗脱前处理能提高基因组DNA的纯度和质量[28-30],所以本研究在对大曲基因组DNA提取时也进行了洗脱前处理的尝试。本研究采用4种大曲DNA提取方法(原位SDS-based提取法、洗脱加SDS-based提取法、原位试剂盒法和洗脱加试剂盒法),比较其对大曲微生物基因组的提取得率、微生物生物量、群落多样性指数及群落结构的影响,从评估结果来看,4种DNA提取方法对大曲微生物基因组的提取存在差异。
从DNA得率和微生物生物量角度,原位SDS-based提取法DNA得率最高,虽然DNA纯度低于A260/280值的正常范围,但是其细菌和真菌的生物量能达到105~107 copies/g,说明该方法仍可用于大曲微生物DNA的提取。洗脱加SDS-based提取法和原位SDS-based提取法的DNA得率存在10倍差距,但是洗脱加SDS-based提取法的纯度明显高于原位SDS-based提取法,并且洗脱加SDS-based提取法的微生物定量结果为106~108 copies/g,要高于或略等于原位SDS-based提取法,说明该方法对大曲微生物DNA的提取有利。原位试剂盒法和洗脱加试剂盒法,实验流程规范且操作简便,提取的DNA也能够满足分子实验要求,但是其微生物定量结果只有102~104 copies/g,与原位SDS-based提取法和洗脱加SDS-based 提取法之间存在10~106的量级差异,说明原位试剂盒法和洗脱加试剂盒法会损失部分大曲微生物的DNA信息。总体看来,上述4种DNA提取方法既存在一致性,也存在差异性。例如,洗脱处理和试剂盒提取法对基因组DNA纯度的提高呈现出了相似的结果;而原位SDS-based提取法提取的DNA中包含了大量小麦原料来源的DNA,致使DNA得率和qPCR微生物定量的结果则呈现出差异。总之,本研究表明,要实现对大曲微生物DNA的有效提取,既要保证DNA得率和纯度,也要兼顾对大曲微生物基因组DNA的有效提取,这样才能更真实地反映大曲中微生物的信息。
为了对DNA提取方法展开深入的认识,本研究还关注了不同DNA提取方法间细菌16S rDNA扩增子文库和真菌ITS扩增子文库的Shannon多样性指数和多物种差异检验的结果。Shannon多样性指数经过q检验法的统计分析,推断出细菌16S rDNA扩增子文库的Shannon指数在4种DNA提取方法之间差异不显著,也推断出洗脱处理对真菌ITS扩增子文库的多样性结果是存在显著影响的,这些结果和多物种差异检验柱形图呈现的多物种DNA提取情况也都是相符合的。在本研究中,Shannon多样性指数是各DNA提取方法总体OTUs多样性的展示,而多物种差异检验是对丰度均值总和前20且具有显著性差异的OTUs提取情况的展示,这表征的虽是2个方面,但都说明了大多数细菌OTUs能在原位SDS-based提取法、洗脱加SDS-based提取法、原位试剂盒法和洗脱加试剂盒法中提取出来,表明4种DNA提取方法对细菌微生物DNA的提取均适用;也说明了洗脱处理对真菌微生物DNA信息的表征有益,且洗脱加SDS-based提取法优于洗脱加试剂盒法,表明洗脱加SDS-based提取法适用于大曲真菌微生物DNA的提取。
根据本研究的结果与过去的研究[15,26-30]提供的经验证据,在综合考虑大曲微生物基因组的提取得率、微生物生物量、群落多样性指数及群落结构等结果,不建议将试剂盒法用于大曲微生物定量分析,因为该方法会严重低估大曲中微生物的生物量;建议将原位SDS-based提取法和洗脱加SDS-based提取法用于大曲细菌微生物定量和群落结构分析;建议洗脱加SDS-based提取法用于大曲真菌微生物定量和群落结构分析;综合对比4种DNA提取方法,洗脱加SDS-based提取法最适合用于大曲微生物的定量和群落结构分析。
[1] 沈才洪, 张良, 应鸿, 等. 大曲质量标准体系设置的探讨[J]. 酿酒科技, 2005(11): 19-24.
SHEN C H, ZHANG L, YING H, et al. Study on daqu quality standards(Ⅴ)—Investigation on the setting of daqu quality judgment standards system[J]. Liquor-making Science &Technology, 2005(11): 19-24.
[2] 张宿义. 白酒酒体设计工艺学[M]. 北京: 中国轻工业出版社, 2020: 1-483.
ZHANG S Y. Liquor Body Design Technology[M]. Beijing: China Light Industry Press, 2020.
[3] 车路萍, 邓杰, 卫春会, 等. 基于PLFA分析不同地域浓香型大曲及曲房空气细菌群落相关性[J]. 食品与发酵工业, 2019, 45(24):33-39.
CHE L P, DENG J, WEI C H, et al. The correlation of bacterial communities between Luzhou-flavored Daqu and its workshop air based on PLFA technology in different areas[J]. Food and Fermentation Industries, 2019, 45(24):33-39.
[4] MAOLONI A, ![]() V, CARDINALI F, et al. Bacterial and fungal communities of gioddu as revealed by PCR-DGGE analysis[J]. Indian Journal of Microbiology, 2020, 60(1):119-123.
V, CARDINALI F, et al. Bacterial and fungal communities of gioddu as revealed by PCR-DGGE analysis[J]. Indian Journal of Microbiology, 2020, 60(1):119-123.
[5] KONSTANTINOS K V, PANAGIOTIS P, ANTONIOS V T, et al. PCR-SSCP: A method for the molecular analysis of genetic diseases[J]. Molecular Biotechnology, 2008, 38(2):155-163.
[6] 王彩虹. 基于克隆文库法研究不同香型大曲微生物群落结构[D]. 自贡: 四川理工学院, 2014.
WANG C H. Study of microbial community structure involved in different types of Daqu based on gene clone libraries method[D]. Zigong: Sichuan University of Science &Engineering, 2014.
[7] METHÉ B A, NELSON K E, POP M, et al. A framework for human microbiome research[J]. Nature, 2012, 486:215-221.
[8] ROSENBAUM J, USYK M, CHEN Z G, et al. Evaluation of oral cavity DNA extraction methods on bacterial and fungal microbiota[J]. Scientific Reports, 2019, 9(1):1531.
[9] 山其木格, 唐平, 王丽, 等. 微生物分析技术及其在酱香型白酒中的应用进展[J]. 中国酿造, 2021, 40(5):18-25.
SHAN Q, TANG P, WANG L, et al. Microbial analytical techniques and its application in sauce-flavor Baijiu[J]. China Brewing, 2021, 40(5):18-25.
[10] 闫亮珍, 李晓然, 全哲学, 等. 汾酒大曲和酒醅样品DNA提取方法的优化[J]. 食品与发酵工业, 2011, 37(3):32-36.
YAN L Z, LI X R, QUAN Z X, et al. Method optimization for DNA extraction from Fenjiu production and fermentation samples[J]. Food and Fermentation Industries, 2011, 37(3):32-36.
[11] 葛媛媛, 姚粟, 刘洋, 等. 芝麻香型白酒高温大曲微生物总DNA提取的改良方法[J]. 酿酒, 2012, 39(4):33-37.
GE Y Y, YAO S, LIU Y, et al. A modified method for DNA extraction from high temperature Daqu of Zhima-flavor Chinese liquor[J]. Liquor Making, 2012, 39(4):33-37.
[12] 张春辉, 赵树欣, 梁慧珍. 高温大曲中细菌总DNA提取方法比较[J]. 酿酒科技, 2008(10):54-56.
ZHANG C H, ZHAO S X, LIANG H Z. Comparison of DNA extraction methods from high temperature Daqu[J]. Liquor-Making Science &Technology, 2008(10):54-56.
[13] 潘明, 袁城金, 王世宽. 浓香型大曲中微生物总DNA的PCR优化[J]. 中国酿造, 2012, 31(3):108-111.
PAN M, YUAN C J, WANG S K. Optimization of PCR reaction system in Luzhou-flavor Daqu microbial DNA[J]. China Brewing, 2012, 31(3):108-111.
[14] TESKE A, SØRENSEN K B. Uncultured Archaea in deep marine subsurface sediments: Have we caught them all?[J]. The ISME Journal, 2008, 2(1):3-18.
[15] 倪峥飞. 镇江香醋固态发酵过程中酿造微生物强化及醋醅总DNA提取方法的初步研究[D]. 无锡: 江南大学, 2009.
NI Z F. Research on bioaugmentation of brewing microbes and total DNA extraction during solid-state fermentation of Zhenjiang vinegar[D]. Wuxi: Jiangnan University, 2009.
[16] 叶光斌, 王彩虹, 乔晓艳, 等. 超高温大曲细菌微生物群落结构的研究[J]. 酿酒科技, 2014(4):5-8.
YE G B, WANG C H, QIAO X Y, et al. Study on the structure of bacterial communities of ultrahigh-temperature daqu[J]. Liquor-Making Science &Technology, 2014(4):5-8.
[17] LIAO X B, CHEN C, ZHANG J X, et al. Operational performance, biomass and microbial community structure: Impacts of backwashing on drinking water biofilter[J]. Environmental Science and Pollution Research, 2015, 22(1):546-554.
[18] 王彩虹, 罗惠波, 卓毓崇, 等. 基于ITS基因文库法研究中高温大曲和超高温大曲真菌群落结构[J]. 食品科技, 2014, 39(7):41-46.
WANG C H, LUO H B, ZHUO Y C, et al. The fungal community structure in medium-high temperature Daqu and ultrahigh temperature Daqu based on ITS gene clone libraries[J]. Food Science and Technology, 2014, 39(7):41-46.
[19] XU N, TAN G C, WANG H Y, et al. Effect of biochar additions to soil on nitrogen leaching, microbial biomass and bacterial community structure[J]. European Journal of Soil Biology, 2016, 74:1-8.
[20] ADAMS R I, MILETTO M, TAYLOR J W, et al. Dispersal in microbes: Fungi in indoor air are dominated by outdoor air and show dispersal limitation at short distances[J]. The ISME Journal, 2013, 7(7):1262-1273.
[21] LIU J W, ZHU S Q, LIU X Y, et al. Spatiotemporal dynamics of the archaeal community in coastal sediments: Assembly process and co-occurrence relationship[J]. The ISME Journal, 2020, 14(6):1463-1478.
[22] XIE M W, LV F X, MA G X, et al. High throughput sequencing of the bacterial composition and dynamic succession in Daqu for Chinese sesame flavour liquor[J]. Journal of the Institute of Brewing, 2020, 126: 98-104.
[23] LI S Z, DENG Y, WANG Z J, et al. Exploring the accuracy of amplicon-based internal transcribed spacer markers for a fungal community[J]. Molecular Ecology Resources, 2020, 20(1):170-184.
[24] LI H F, LI Z J, QU J H, et al. Bacterial diversity in traditional Jiaozi and sourdough revealed by high-throughput sequencing of 16S rRNA amplicons[J]. LWT-Food Science and Technology, 2017, 81:319-325.
[25] WINTZINGERODE F V, GÖBEL U B, STACKEBRANDT E. Determination of microbial diversity in environmental samples: Pitfalls of PCR-based rRNA analysis[J]. FEMS Microbiology Reviews, 1997, 21(3):213-229.
[26] 宋培勇, 马莉莉. TENP-PBS预处理在提取土壤DNA中的应用[J]. 安徽农业科学, 2010, 38(24):12978-12980.
SONG P Y, MA L L. Application of pretreatment with TENP-PBS in the extraction of DNA from soil[J]. Journal of Anhui Agricultural Sciences, 2010, 38(24):12978-12980.
[27] 洪桂云, 鲍立宁, 魏兆军. 同时提取土壤微生物DNA和RNA方法的研究[J]. 环境科学与技术, 2011, 34(6):24-27.
HONG G Y, BAO L N, WEI Z J. Simultaneous recovery of DNA and RNA from soil microbe[J]. Environmental Science &Technology, 2011, 34(6):24-27.
[28] MARDAN-NIK M, SAFFAR SOFLAEI S, BIABANGARD-ZAK A, et al. A method for improving the efficiency of DNA extraction from clotted blood samples[J]. Journal of Clinical Laboratory Analysis, 2019, 33(6): e22892.
[29] FATIMA F, PATHAK N, RASTOGI VERMA S. An improved method for soil DNA extraction to study the microbial assortment within rhizospheric region[J]. Molecular Biology International, 2014, 2014:518960.
[30] 陈竹, 陈元彩, 詹怀宇, 等. 从颗粒污泥中提取DNA的几种不同方法比较[J]. 造纸科学与技术, 2008, 27(5):23-26.
CHEN Z, CHEN Y C, ZHAN H Y, et al. Comparison of different DNA extraction methods from granular sludge[J]. Paper Science &Technology, 2008, 27(5):23-26.