木聚糖是植物半纤维素的主要成分[1-2],在自然界中含量仅次于纤维素,是重要的可再生资源[3]。木聚糖主要由阿拉伯糖和木糖组成[4-5],因其结构的复杂性,需要包括β-1,4-内切木聚糖酶、β-1,4-木糖苷酶、α-L-呋喃阿拉伯糖苷酶等在内的木聚糖酶参与才能完全分解[6],其中起主要作用的是β-1,4-内切木聚糖酶[7],其能够内切木聚糖主链中的木糖苷键[8]。木聚糖酶在需要降解木聚糖的行业中已经得到广泛的应用,如造纸、饲料、能源、食品和医药等行业[9]。
现阶段,木聚糖酶的主要来源是毛霉、曲霉和木霉等真菌[10],虽然其来源广泛,但活性不高,且耐酸性和热稳定性大多不能满足工业生产;除真菌外,芽孢杆菌、节杆菌、肠杆菌和放线菌等细菌也能产生木聚糖酶[11-12],但该来源的木聚糖酶也很少能够兼具高活性和pH/热稳定性[13]。相比于其他催化特性的如耐热木聚糖酶,酸性木聚糖酶在酿酒、果汁、饲料等行业中具有更大的应用前景[14-16]。
越来越多的学者进行分子层面的改造来获得酶学性质更加优质的酸性木聚糖酶[17-18]。本文中突变所替换的序列均来自川内曲霉酸性木聚糖XynC(Aspergillus kawachii),是一种酸性木聚糖酶,在pH 1~9时稳定,其最适pH值为2.0,该木聚糖酶产生各种低聚木糖,包括来自木聚糖的木糖,主要产物是所有木聚糖酶中的木二糖。酸性木聚糖酶(XynC)的产生随着培养基的低初始pH值而增强[19]。本文将酸性木聚糖酶XynH基因克隆后在大肠杆菌BL21(DE3)宿主中成功表达,基于生物信息学的分析结果,对其N端、C端及“Thumb”结构进行分子改造,分析XynH及其突变体的酶学性质,探究其结构与催化特性的关联,对酸性木聚糖酶的研究具有一定的理论补充意义。
1 材料与方法
1.1 主要试剂
大肠杆菌(Escherichia coli)感受态细胞DH5α、BL21(DE3)、质粒提取试剂盒,天根生化科技有限公司;表达质粒pET-28a(+),武汉淼灵生物科技有限公司;限制性内切酶(Nco I和Not I)、T4 DNA连接酶,New England Biolabs公司;榉木木聚糖,Megazyme公司;胶回收试剂盒,Omega公司;十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfatepolyacrylamide gel electrophoresis, SDS-PAGE)试剂盒、聚氧基丙烯酸正丁酯(butyleyanoacrylate, BCA),碧云天生物技术有限公司。
LB液体培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 5。
LB固体培养基:1 L LB液体培养基中添加琼脂20 g。
1.2 仪器与设备
PCR扩增仪,美国Bio-Rad公司;恒温摇床,上海智诚实验设备有限公司;5424R型高速冷冻离心机,德国Eppendorf公司;Scientz-ⅡD超声波细胞粉碎机,宁波新芝生物科技有限公司;UV-2102型紫外可见分光光度计,尤尼柯(上海)仪器有限公司。
1.3 实验方法
1.3.1 重组质粒的构建与表达
利用NCBI数据库获得木聚糖酶XynH(ID:FJ860893.1)的基因序列,设计特异性引物(附表1,https://doi.org/10.13995/j.cnki.11-1802/ts.036616,下同),获得目的基因。目的基因由华大基因合成并整合在PUC57质粒载体,转化到大肠杆菌DH5α。基于载体上携带的抗性基因,在添加氨苄抗生素的LB培养基中培养并测序验证,测序正确的基因在Nco I和Not I酶切位点与pET28a(+)连接,然后转化至大肠杆菌BL21(DE3)。
表1 XynH及突变体的动力学参数
Table 1 Kinetic parameters of XynH and mutants
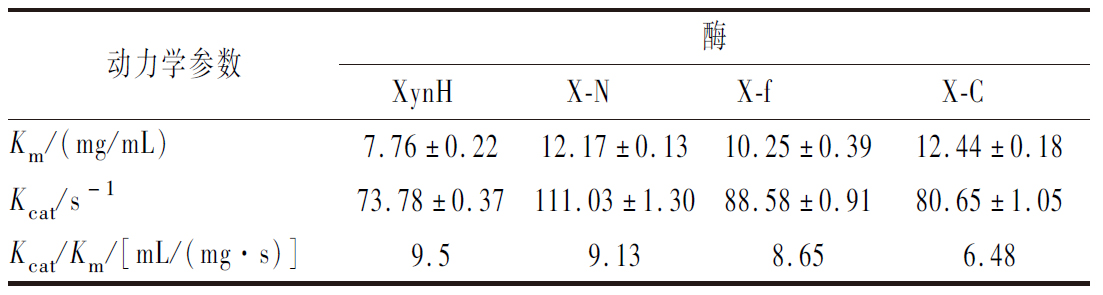
动力学参数酶XynHX-NX-fX-CKm/(mg/mL)7.76±0.2212.17±0.1310.25±0.3912.44±0.18Kcat/s-173.78±0.37111.03±1.3088.58±0.9180.65±1.05Kcat/Km/[mL/(mg·s)]9.59.138.656.48
将含有重组质粒的大肠杆菌BL21(DE3)转移到含卡那霉素的LB培养基中,在150 r/min、37 ℃条件下培养3.5 h,然后添加200 μL 500 mmol/L异丙基β-D-1-硫代半乳吡喃糖苷(ispropyl-β-D-thinogalactoside, IPTG)溶液诱导表达16 h。
1.3.2 重组木聚糖酶的纯化
将诱导表达后的发酵液在4 ℃,8 000 r/min状态下离心5 min,菌体加入10 mL 50 mmol/L Tris-HCl缓冲液(pH 7.0),超声处理15 min,再离心得到上清液即粗酶液。使用上海sangon镍琼脂糖凝胶柱(C600292/C600029,1 cm×10 cm)来纯化粗酶液,通过SDS-PAGE凝胶电泳测定分析蛋白质的分子质量,用BCA蛋白浓度测定试剂盒测定蛋白浓度。
1.3.3 木聚糖酶活性测定
将0.9 mL的1%榉木木聚糖与0.1 mL适当稀释的木聚糖酶(50 mmol/L乙酸缓冲液,pH 5.5)在55 ℃、反应5 min下测定,并用3,5-二硝基水杨酸法(3,5-dinitrosalicylic acid, DNS)计算释放的还原糖量[20]。1个单位的木聚糖酶被定义为1 min水解木聚糖底物形成1 μmol还原糖(以木糖计)所需的酶量[21]。
1.3.4 生物信息学分析
1.3.4.1 XynH的一级结构预测
利用在线软件(附表2)对XynH的理化性质、亲疏水性质、跨膜区域、信号肽、结构域及催化位点进行预测和分析。通过MEGA 11.0软件构建XynH的系统发育树,对其遗传进化的分析。
1.3.4.2 XynH的二级结构预测
通过在线软件PSIPRED和SOPMA对XynH的α-螺旋、β-折叠、延伸链及无规则卷曲等进行预测和分析。
1.3.4.3 XynH的三级结构预测
利用在线软件SWISS-MODEL构建XynH三维模型,同时使用PyMOL软件将预测模型与模板进行比对。
1.3.5 突变体的构建
将含有目的基因的大肠杆菌DH5α菌株在LB培养基中进行培养,利用质粒提取试剂盒提取质粒。以该质粒为模板PCR扩增,引物如附表3所示。将扩增的目的基因进行双酶切(Nco I和Not I),与pET-28a(+)连接并转化至大肠杆菌BL21(DE3),培养并测序验证。将构建成功的突变体在LB液体培养基中进行诱导表达[22]。
1.3.6 突变体的酶学性质
1.3.6.1 最适pH和pH稳定性
用不同pH(2.0~7.0)的缓冲液稀释酶液,以1%榉木木聚糖为底物分别测定各个酶的酶活力,最高酶活力定义为100%,每组3个平行确定其最适pH。用不同pH的缓冲液稀释酶液,37 ℃保温1 h,并在最适pH下测量酶活力,未处理组的酶活力定义为100%,每组设置3个平行。
1.3.6.2 最适温度和温度稳定性
以1%榉木木聚糖作为底物使酶液在不同温度(40~90 ℃)下进行反应,分别测定各个酶的酶活力,最高酶活力定义为100%,每组3个平行确定其最适温度。将酶液在不同温度下保温1 h后,并在最适温度下测量酶活力,未处理的酶活力定义为100%,每组设置3个平行。
1.3.6.3 动力学参数
以6种不同质量浓度(5~30 mg/mL)的榉木木聚糖为底物,将木聚糖酶置于最佳的反应条件下测定其酶活力,使用GraphPad Prism 8.2.1软件通过非线性拟合计算酶的动力学参数,包括Km、Kcat和Kcat/Km,每组设置3个平行。
2 结果与分析
2.1 克隆与表达
以含有木聚糖酶目的基因的载体PUC57为模板,进行PCR扩增,用1%(质量分数)琼脂糖凝胶电泳检验扩增产物。电泳结果如图1所示,产物的电泳条带为657 bp,编码的氨基酸218个。经双酶切、与载体pET28a(+)连接,转化至大肠杆菌BL21(DE3)进行表达。经诱导表达后,通过超声波破壁处理,离心取上清液进行SDS-PAGE检验。目标蛋白的表达量较高,分子质量大约为23 kDa左右。该重组木聚糖酶命名为XynH。
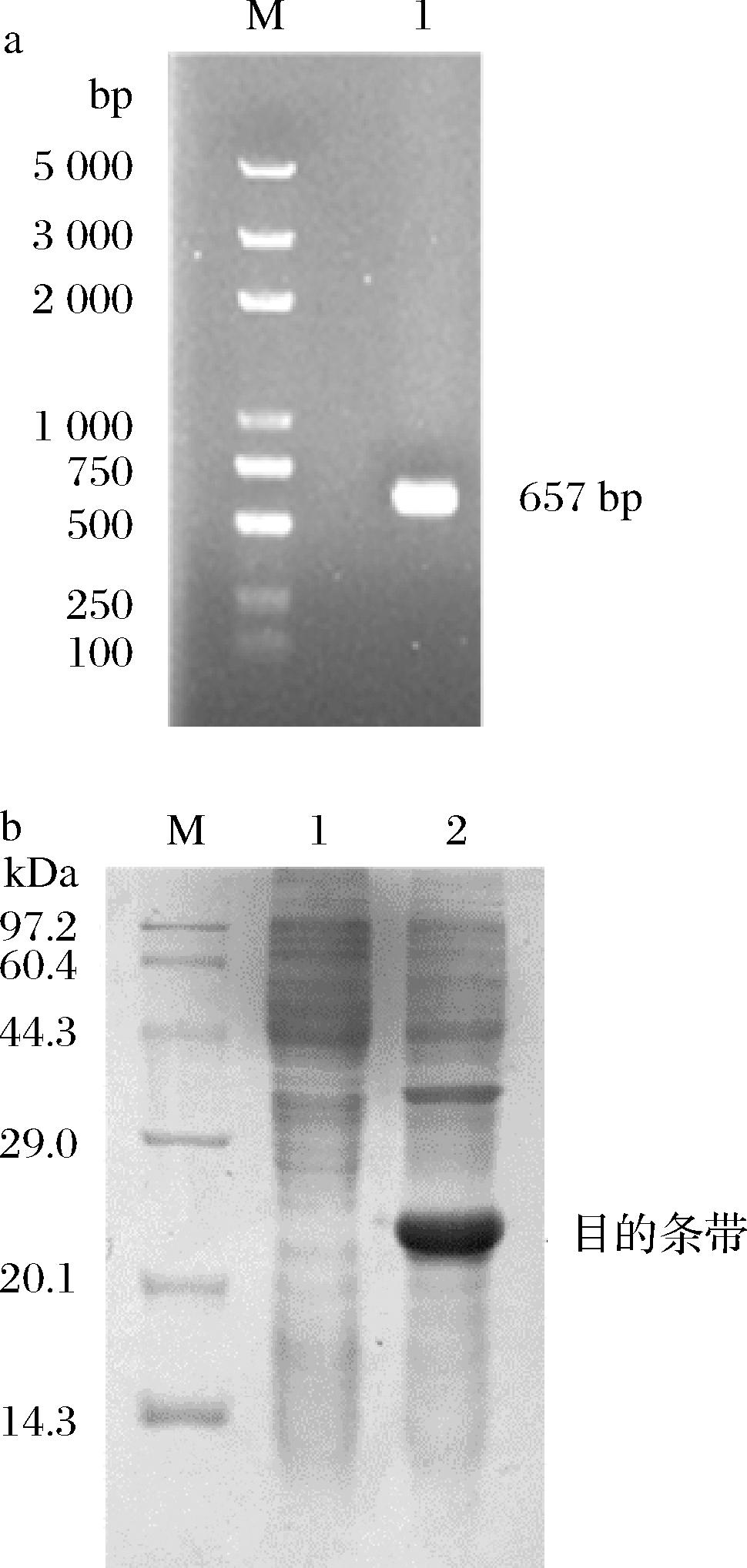
a-DNA(M-5 000 bp DNA Maker;1-目标DNA);b-木聚糖酶(M-蛋白Maker,1-空载体空白对照;2-粗酶液)
图1 DNA琼脂糖凝胶电泳和木聚糖酶的SDS-PAGE电泳
Fig.1 Results of DNA agarose gel electrophoresis and SDS-PAGE analysis of xylanase
2.2 生物信息学分析
2.2.1 同源序列比对
将XynH的氨基酸序列与NCBI数据库中已报道的木聚糖酶进行多序列比对分析如附图1所示,XynH和源于Penicillium rolfsii、Paecilomyces variotii No.5[23]、Penicillium sp.40[24]聚糖酶相似性分别为90.55%、83.58%、80.88%。XynH具有GH11家族木聚糖酶的特点,其序列含有保守区域VYGWT、PLVEYYI、SDGATYDIYE、HFNAWAKLGMNLG。
2.2.2 理化性质
通过Expasy-Protparam在线软件分析XynH的理化性质如附表4所示,XynH序列的氨基酸总数为218个,预测该氨基酸分子质量23.5 kDa(<30 kDa),与大多数酸性木聚糖酶的分子量一致[25];理论等电点pI 6.81;组成中最多的氨基酸为丝氨酸(Ser),有32个,占序列的14.7%;带负电荷的残基总数:天冬氨酸(Asp)和谷氨酸(Glu)9个,带正电荷的残基总数:精氨酸(Arg)和赖氨酸(Lys)9个;预测XynH的分子式为C1044H1531N275O339S4,脂肪系数为53.72,不稳定系数为21.28(<40),则该蛋白是结构稳定的蛋白[26]。
2.2.3 亲疏水性质分析
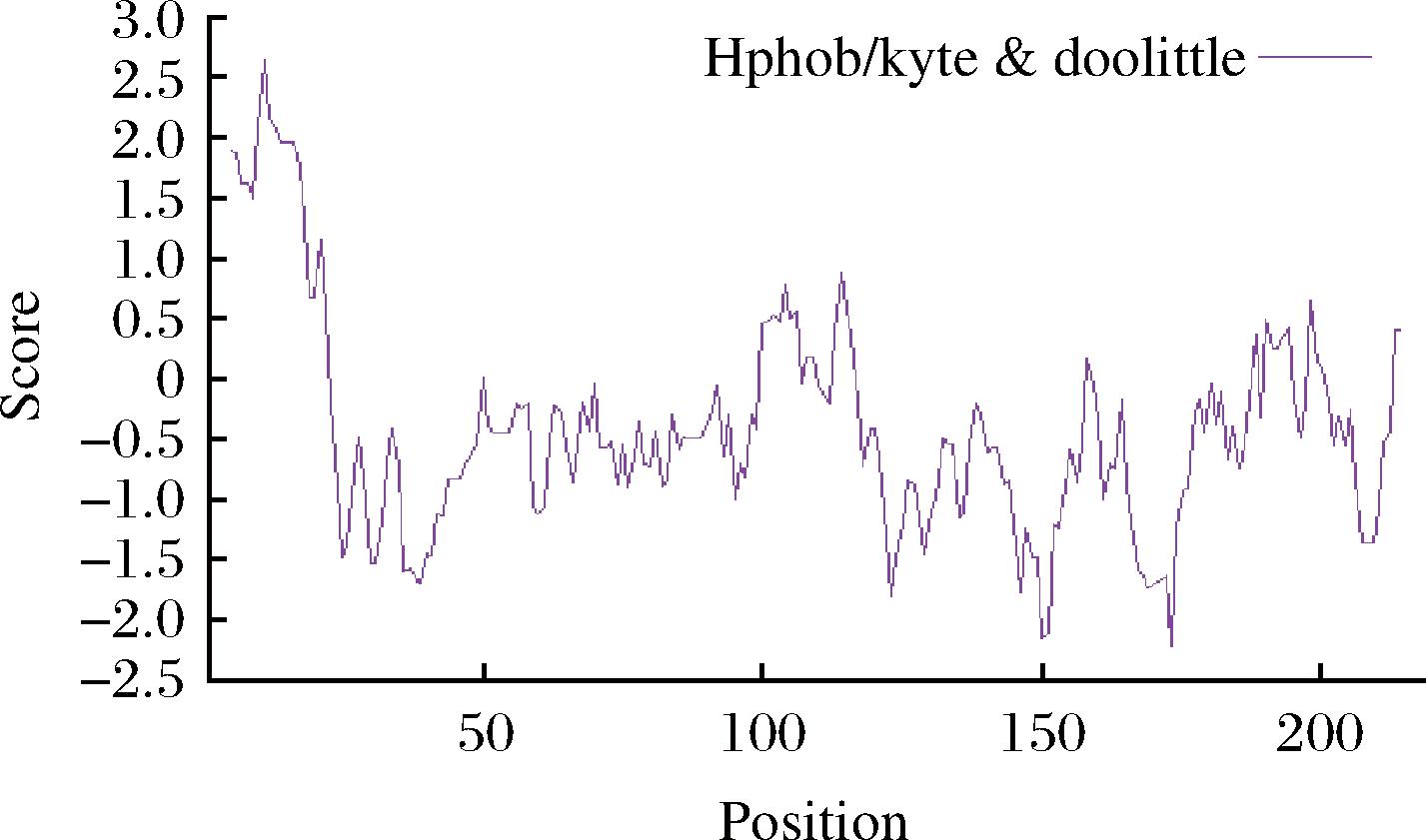
利用Expasy-Protscale在线软件分析XynH亲疏水性如附图2所示,图中是该序列各个氨基酸位点对应的分值,XynH蛋白亲疏水指数最小值为-2.211(173位),亲疏水指数最大值2.633(11位)。计算得到的平均亲水系数为-0.362<0,表明该XynH为亲水性蛋白。
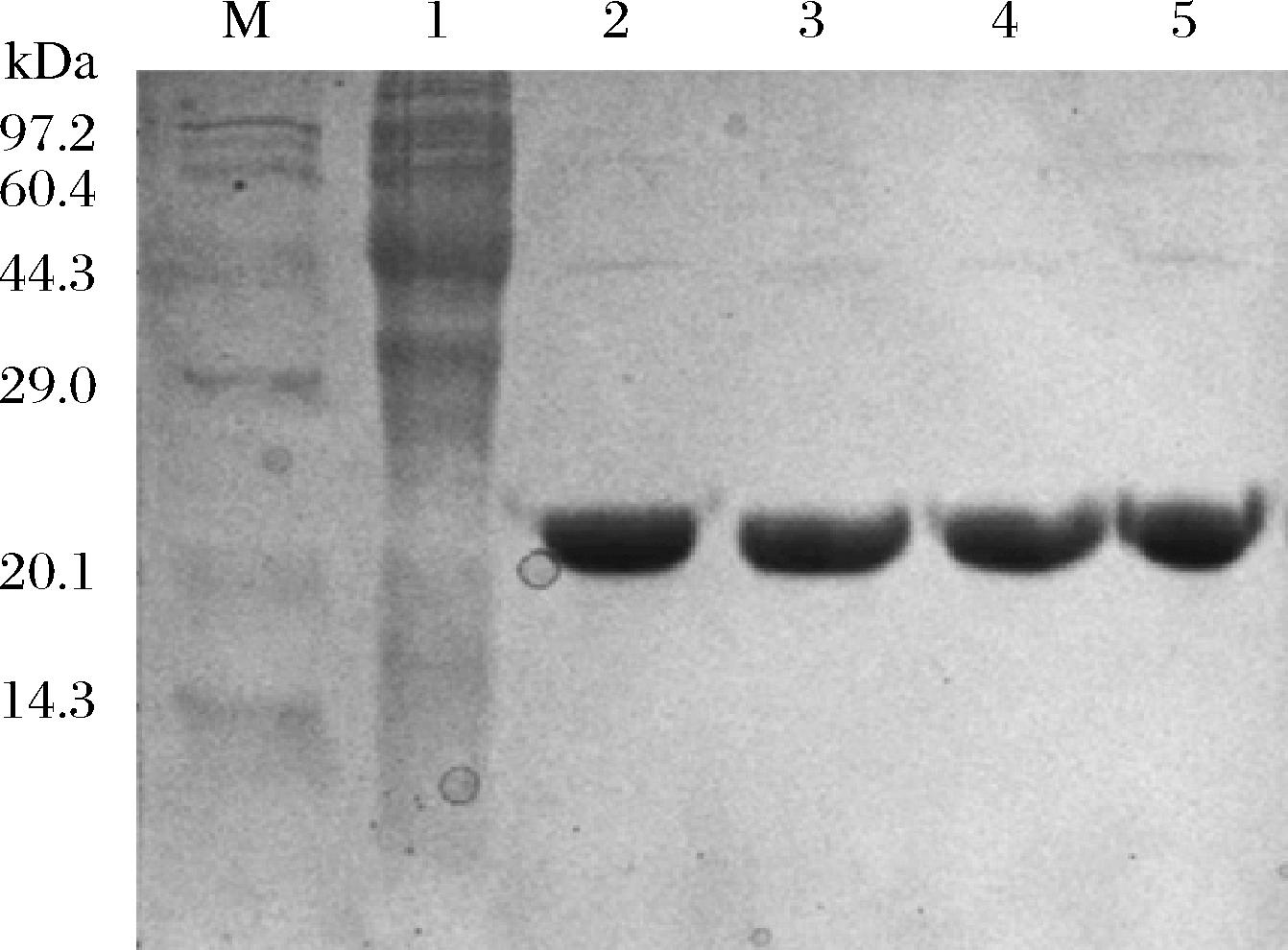
M-Marker;1-空载体空白对照;2-纯化的XynH;3-纯化的X-N;4-纯化的X-f;5-纯化的X-C
图2 纯化XynH及突变体的SDS-PAGE图
Fig.2 SDS-PAGE of purified XynH and its mutants
2.2.4 跨膜区域预测
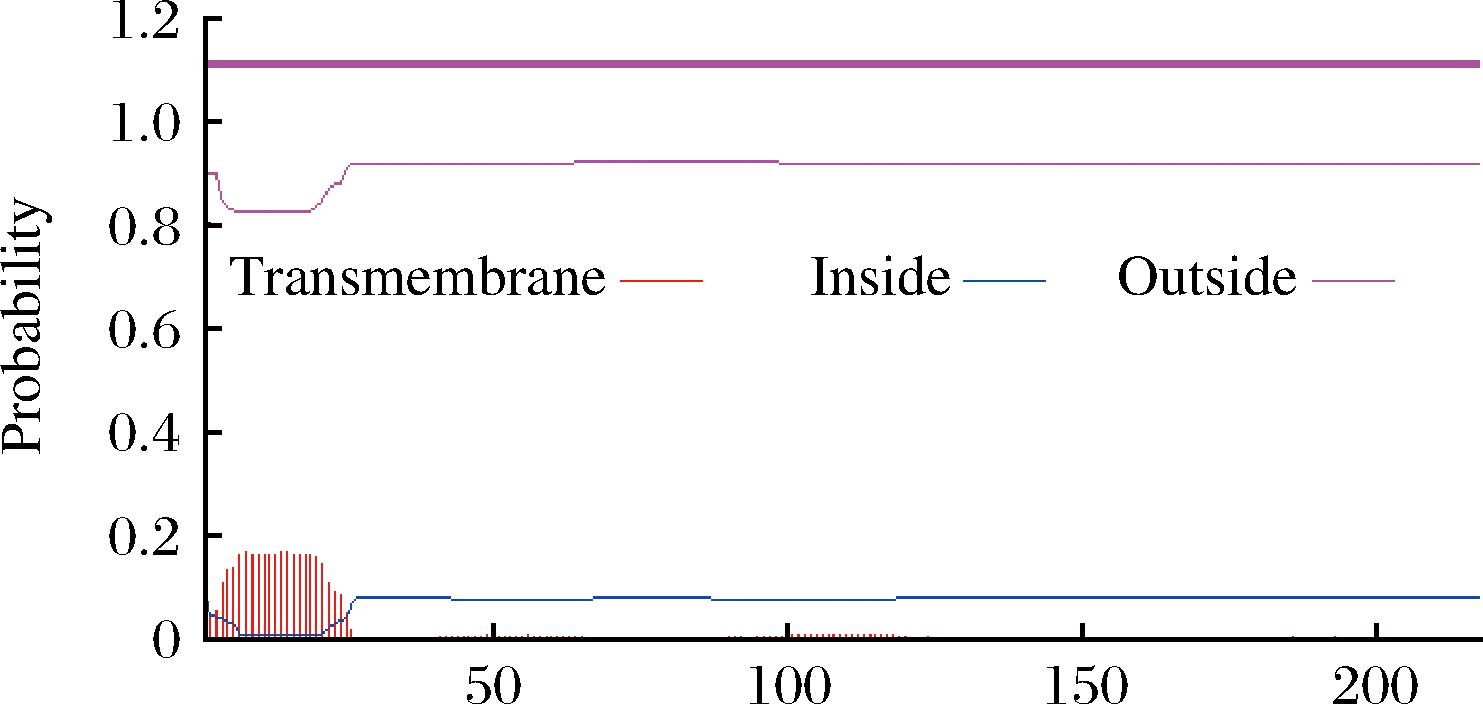
除了作为膜受体外,含有跨膜区的蛋白质也可以定位到膜蛋白或离子通道蛋白[27]。TMHMM软件会用红色标记出检测到的跨膜区,并且对具体位置作出估测。对XynH的分析结果如附图3所示,可知该蛋白中不存在显著的跨膜区域,定位在膜外,因此该蛋白不属于跨膜蛋白。
2.2.5 信号肽预测
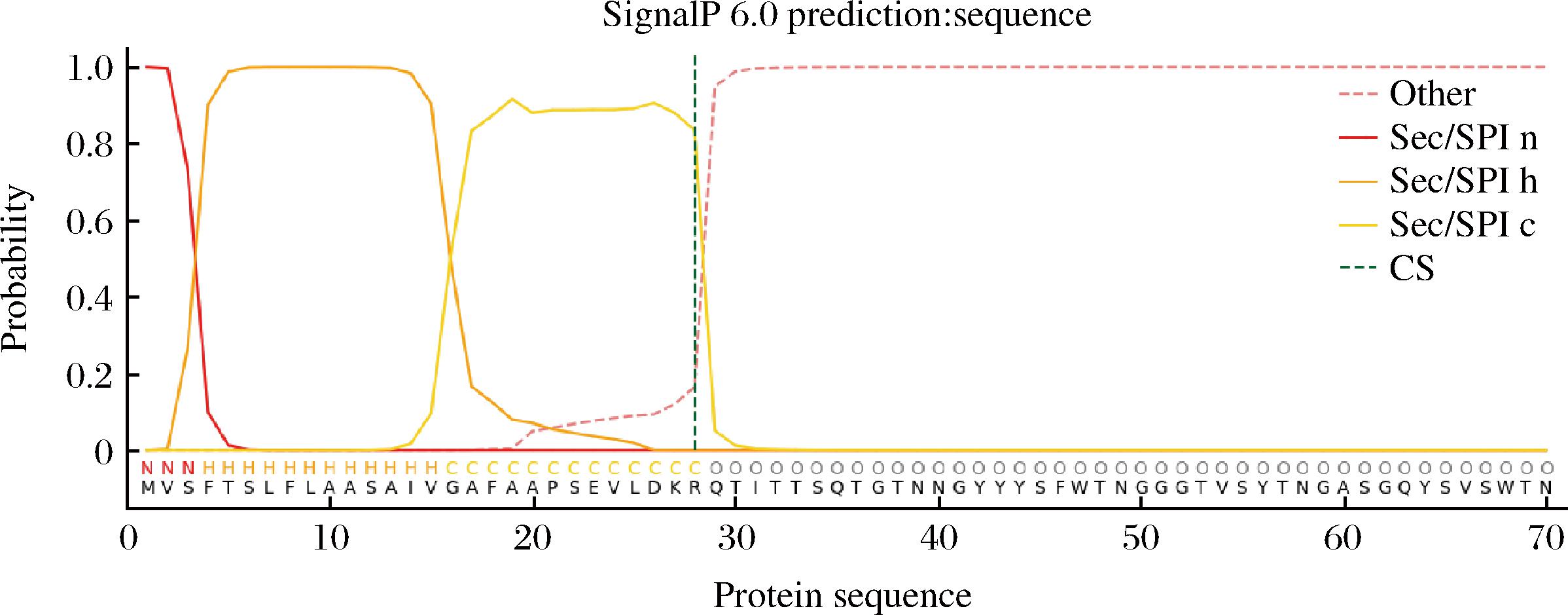
信号肽是由5~30个氨基酸组成,通常存在于分泌蛋白的N端。SignalP软件可以预测氨基酸序列中是否存在信号肽的剪切位点,并作出标记。如附图4所示,预测该蛋白的信号肽有28个氨基酸,为“MVSFTSLFLAASAIVGAFAAPSEVLDKR”1~28位氨基酸。

a-最适温度;b-温度稳定性
图4 XynH和突变体的最适温度和温度稳定性
Fig.4 Optimal temperature and temperature stability of XynH and mutants
2.2.6 结构域预测
在NCBI数据库中结构域的预测如附图5-a所示:第40~208位氨基酸为Glyco-hydro11的结构域,E值为6.33e-104<10-6,说明预测是具有可靠性,没有其他有效结构域。
在Exprosite数据库中催化位点的预测如附图5-b所示:该蛋白具有功能区域G11-3糖苷水解酶家族位于30~218位,与NCBI预测结构接近。同时,预测出亲核体为催化位点第114位谷氨酸,质子供体为第205位谷氨酸。
2.2.7 XynH二级结构预测
PSIPRED软件对XynH二级结构的预测如附图6-a所示,XynH蛋白中有3个α-螺旋(2个处于信号肽中),有16个β-折叠。同时利用SOPMA软件的预测如附图6-b所示,其中α-螺旋和无规则卷曲两种结构交替存在,并且延伸链均匀分布,这种结构有利于蛋白结构的稳定[28]。α-螺旋占14.22%;β-折叠占5.96%;延伸链占33.49%;无规则卷曲占46.33%。
2.2.8 XynH三级结构分析
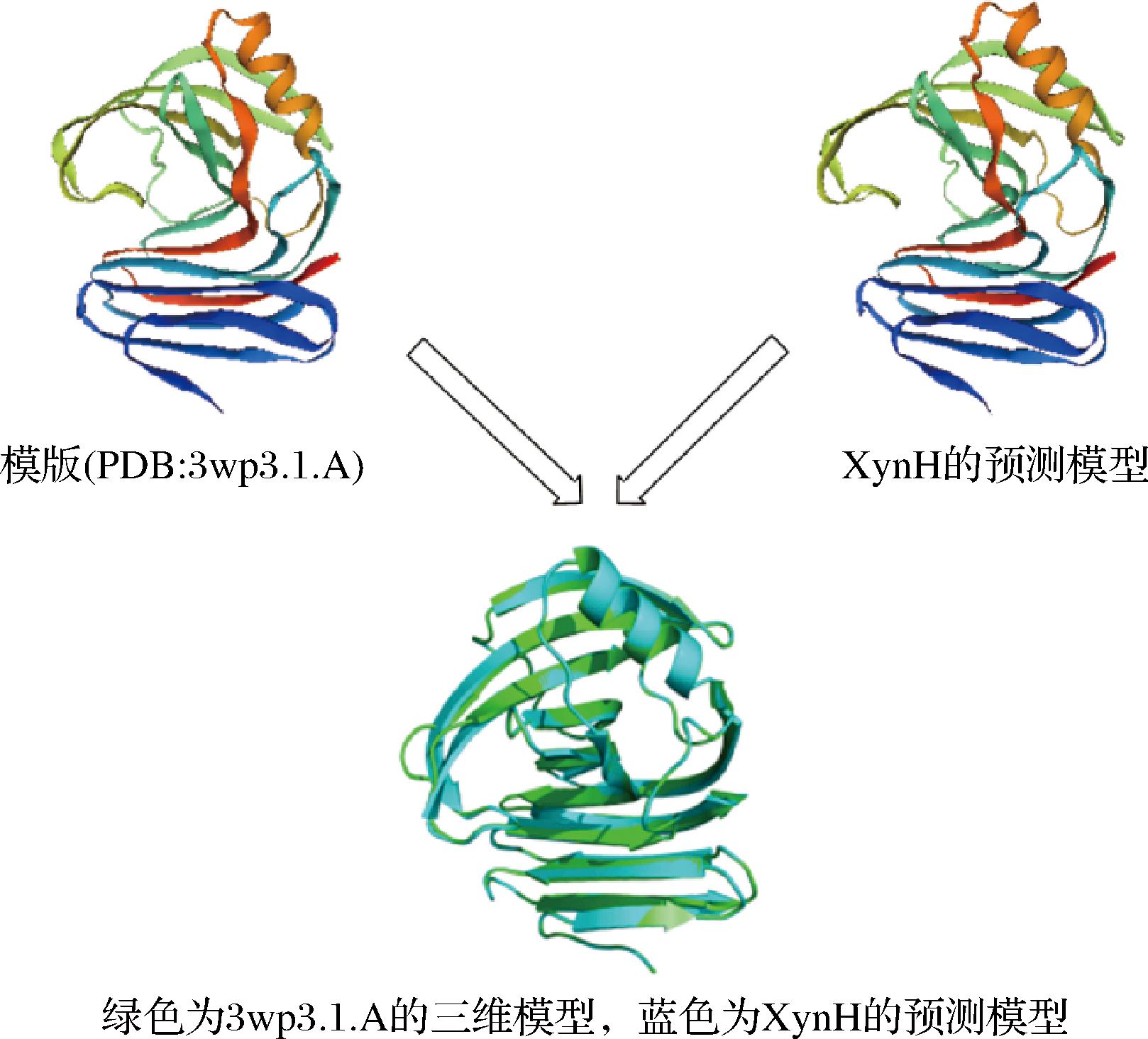
如附图7所示,XynH三级结构的预测模型与其模板(PDB:3wp3.1A)的比对结果,二者序列相似性为73.44%,属于可信的模板;同时可以看出XynH的空间结构是一个右手半握状的典型G11家族木聚糖酶结构。
2.2.9 XynH系统发育树构建
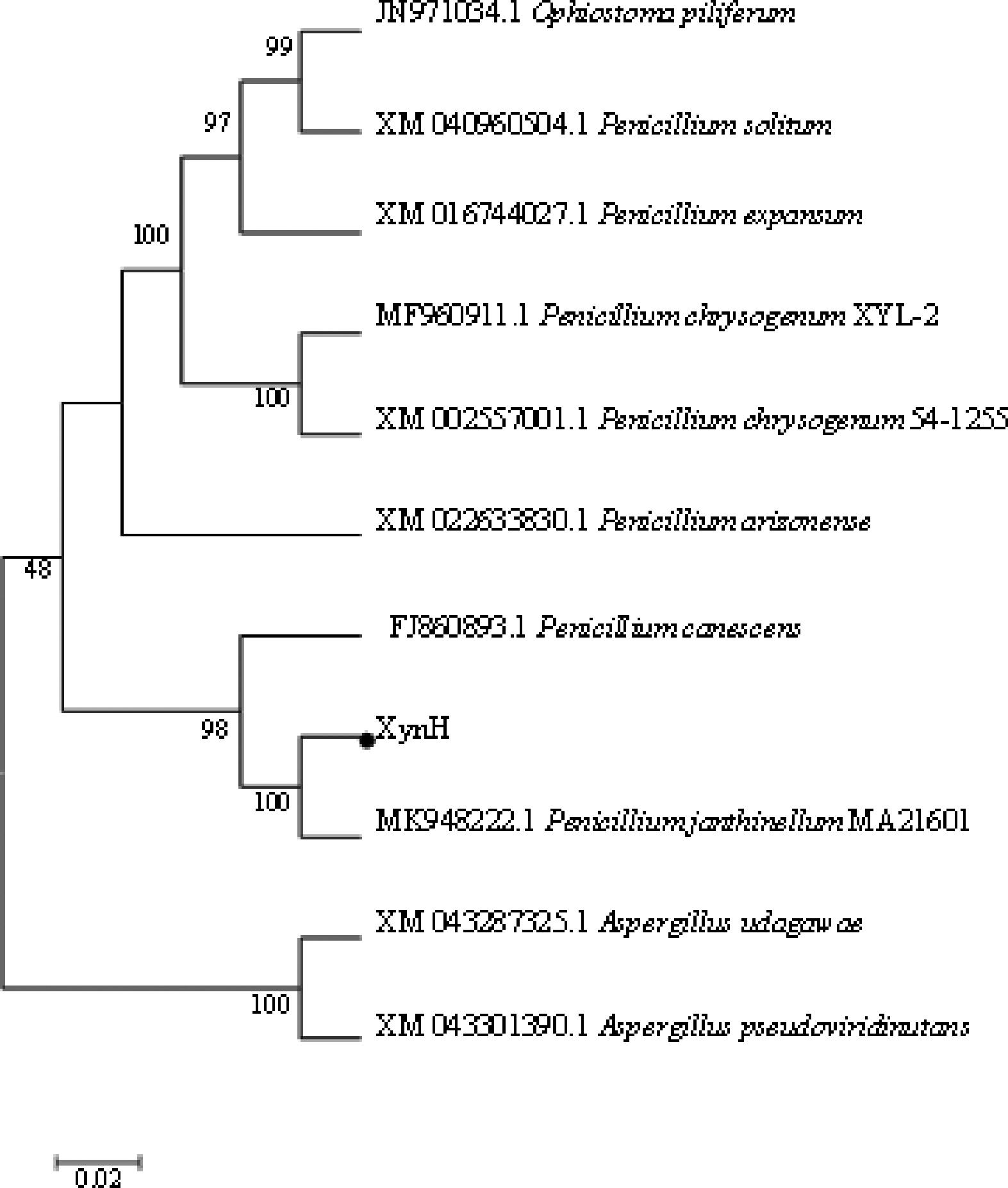
通过MEGA软件对XynH进行系统进化树的构建。如附图8所示,遗传距离为0.02,各分支处的数值越高则表示两者的亲缘关系越高,同源性越可信。XynH与Penicillium janthinellum来源的木聚糖酶基因同源性较高,与Penicillium Arizona、Penicillium chrysogenum、Penicillium expansum、Penicillium conchomyceum的亲缘性关系较远。
2.3 XynH突变体的构建
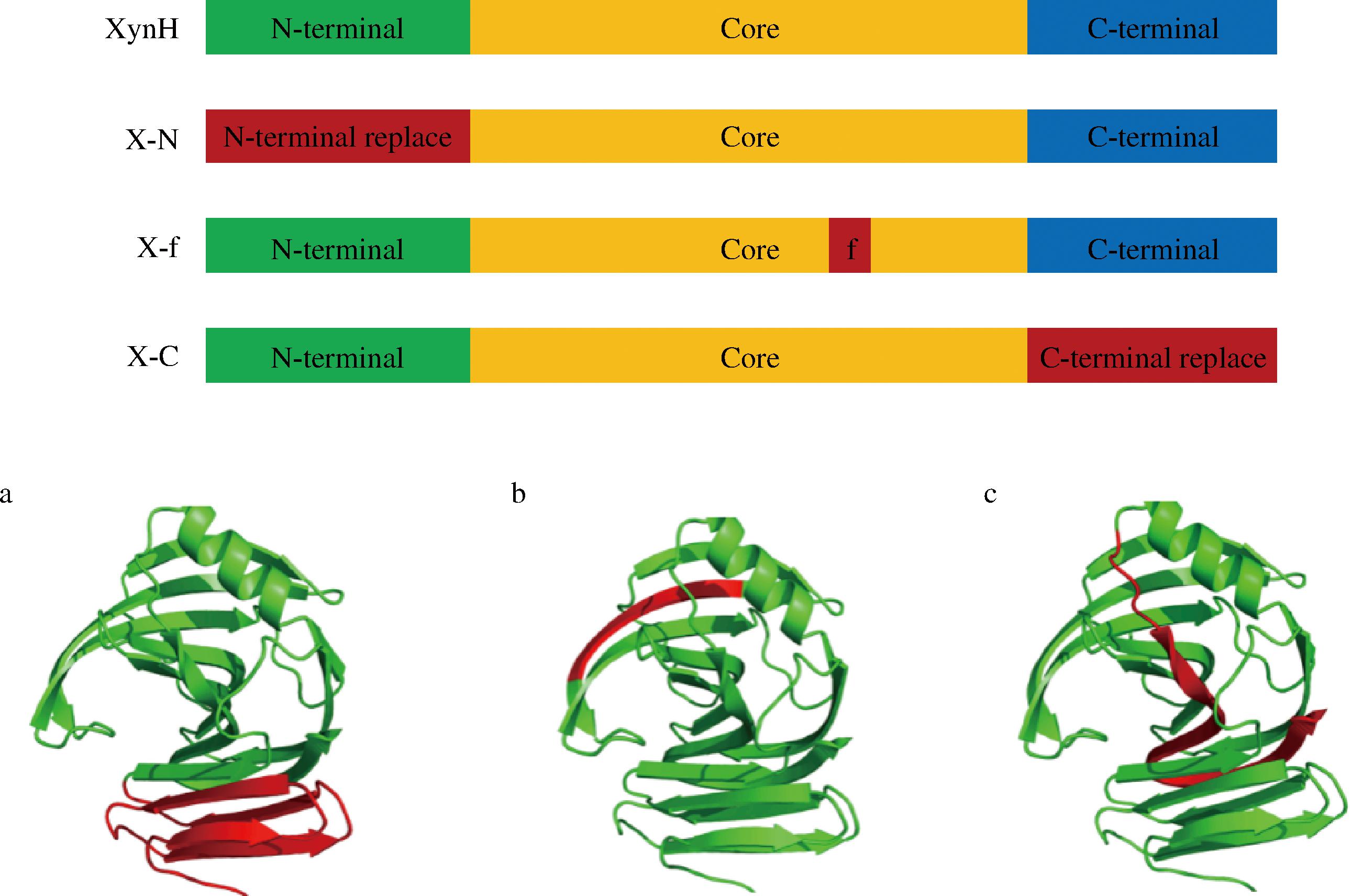
如附图9所示,X-N为XynH的N端替换,序列“Gln29-Gly72”替换成“29Ser-64Ser”;X-f为XynH“Thumb”结构的“Asp144-Val151”被“144Gln-151Thr”取代;X-C则是将XynH的C端的“Met192-Ser218”替换为“192Phe-219Ser”。同时使用PyMol软件将N端、“Thumb”结构、C端的替换在蛋白质空间结构中标记出来,可以更直观地看出各突变点的结构位置。
2.4 XynH和突变体纯化
将XynH和突变体的粗酶液通过Ni Sepharose HP柱纯化,进行SDS-PAGE凝胶电泳检验。如图2所示,目标蛋白的表达量相对较高,XynH在23.5 kDa左右有单一的蛋白质条带,与理论分子质量相同;X-N、X-f和X-C的分子质量分别约为22.8、23.2、23.6 kDa。
2.5 催化特性研究
2.5.1 最适pH及pH稳定性
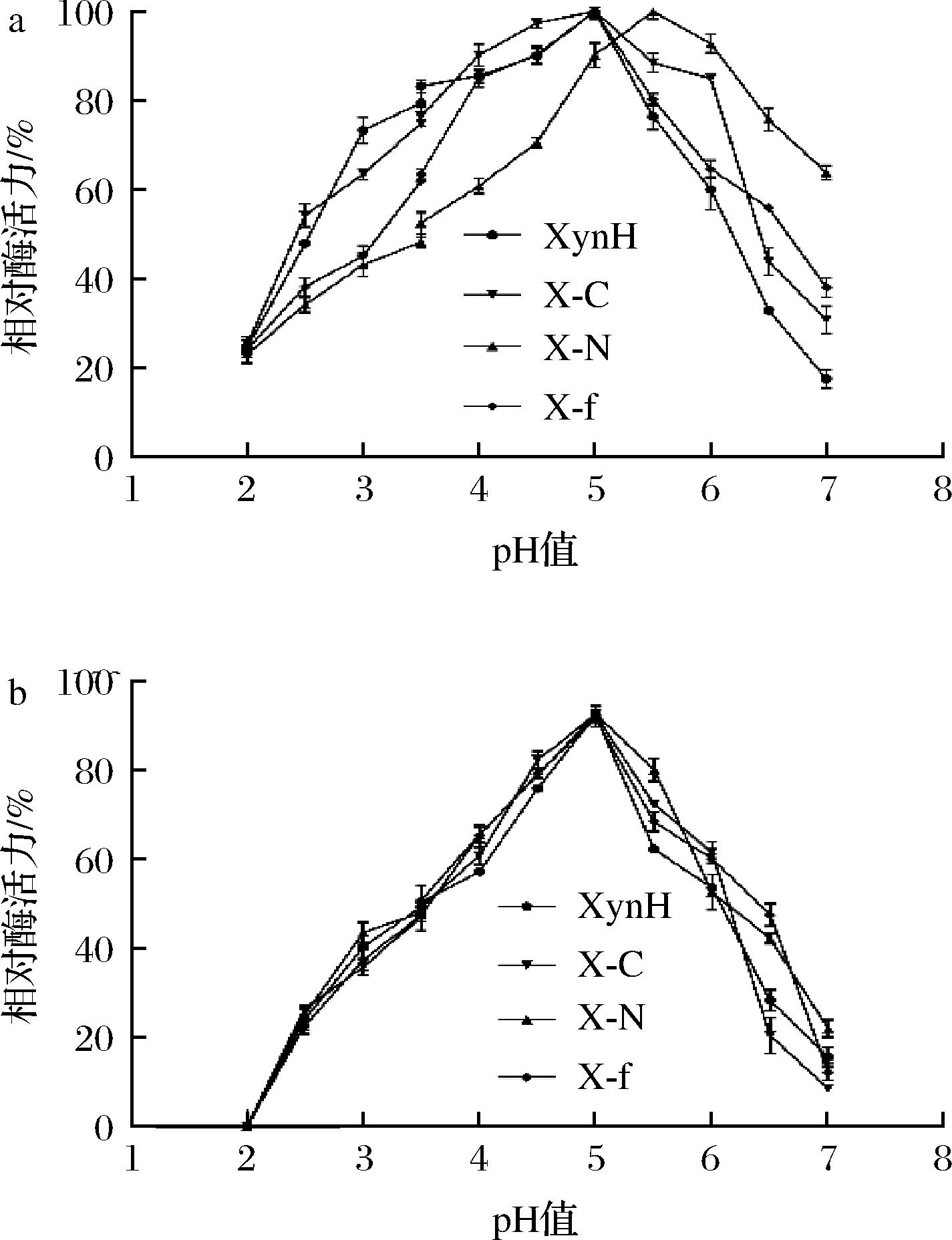
各酶在pH 2.0~7.0的条件下酶活如图3-a所示,XynH和突变体表现出相似的pH,除X-N的最适pH值为5.5,其他酶均是在pH 5.0时表现出最高酶活力,因此有利于酸性环境中生存,同时说明N端替换使酶的pH发生了向中性偏移。
将酶在不同pH(2.0~7.0)保存1 h后,各酶的pH稳定性如图3-b所示,X-N在pH 3.5和pH 6.5的残余酶活力分别为50%和42%,其中在pH 7.0时仍有22%的残余活性。X-C在pH 4.0放置1 h后残余酶活力为60%,而pH为6.5时降为20%。X-f在pH 3.5条件下的残余酶活为48%,在pH 5.0时酸度耐受性最强,为92%。综上所述,各突变体与XynH在pH 4.0~6.0的残余酶活力均在60%左右,具有良好的耐酸性。
2.5.2 最适温度及温度稳定性
各酶在40~90 ℃的反应条件酶活如图4-a所示,X-C和X-f的最适温度与XynH均为55 ℃,X-N的最适温度为60 ℃,相比其他酶高5 ℃。
在不同温度(30~60 ℃)保温1 h后,各酶的温度稳定性如图4-b所示,X-N在40 ℃和50 ℃的残余酶活力分别为76%和43%。X-C在40 ℃和50 ℃的残余酶活力分别为64%和27%,而在35 ℃时稳定性最好,残余酶活力为75%。X-f在40 ℃和50 ℃时的残余酶活力与XnyH相似,分别为73%和35%左右。综上所述,在40 ℃放置1 h后,除X-C的残余酶活力较低,其他酶均在73%以上;在50 ℃时,X-N的残余酶活力相比其他酶较高;在55 ℃下保温1 h后,X-N和X-f比XynH的残留酶活力高,其中X-N的耐热性表现更好。
2.5.3 动力学参数
木聚糖酶XynH和突变体催化榉木木聚糖时的动力学参数如表1所示,Km值越小,酶对底物亲和力越强,X-N、X-f和X-C的亲和力均低于XynH,其中X-N和X-f的Km值相近。虽然所有突变体的Kcat值均高于野生型XynH,但是最终突变体的的催化效率由Kcat/Km的比值决定。Kcat/Km的比值越大,酶的催化效率越高,突变体表现出的催化效率均小于XynH,其中X-N仅弱于XynH。
3 结论
本论文对木聚糖酶XynH进行分子改造,成功构建了N端替换,C端替换,以及“Thumb”结构氨基酸替换的突变体,分别为X-N,X-C,X-f。各突变体与原酶XynH相比,X-N的最适pH为5.5,其他酶均是在pH 5.0时表现出最高酶活力;各突变体与XynH在pH 4.0~6.0的残余酶活力均在60%左右,具有良好的耐酸性。X-N的最适温度(60 ℃)与其他酶相比提高了5 ℃;在55 ℃下保温1 h后,X-N和X-f的残余酶活力高于XynH,X-N的耐热性表现更好。覃拥灵等[29]通过对米曲霉木聚糖酶纯化得到木聚糖酶XynH1,对其酶学特性研究,得到木聚糖酶XynH1的最适pH和温度分别为pH 6.0和65 ℃。虽然本文中3种突变体的最适温度略低于木聚糖酶XynH1,但最适pH较好于XynH1,耐酸性较好。黄雪芹等[30]为获得适应于酒糟酸性环境的木聚糖酶,将内源微生物耐酸性环境的野生Bacillus velezensis P7木聚糖酶基因克隆到枯草芽孢杆菌WB800中,纯化突变得到重组B.subtilis WB800菌株,其重组酶的相对酶活力在pH为5.0时最高,在pH 3~4时,其残余酶活力是原始酶活力的80%以上,对pH有较广的耐受范围。王金华[31]研究黑曲霉(Aspergillus niger varniger strain N402)耐酸内切-β-1,4-木聚糖酶在酵母中的表达,其最适温度是37 ℃,木聚糖酶的最适作用pH值为4.0。在pH 3.0~5.0时,木聚糖酶的活性较高,当pH<2.0和pH>6.0时,木聚糖酶活性急剧下降(低于30%),说明其有较广泛的pH稳定范围。
N端与酶的稳定性有着密切的关系,改善木聚糖酶热稳定性的方法大多着手于N端结构区域[32]。突变酶X-N较原酶XynH的催化活性有所降低,但热稳定性有所提高。C端替换可能是由于木聚糖酶XynH的β折叠A4-B4结构片处的间隙引起酶结构的不稳定,同时催化结构域活性部位(Glu203)位于该区域,因此,C端改变可能涉及该木聚糖酶催化结构域的一部分,木聚糖酶活性会发生改变。X-f是在木聚糖酶XynH“Thumb”末端的β-折叠弯曲端(位于木聚糖酶β链B8的部分)进行改造,突变区域较长。然而,这些二级结构的差异并不影响“Thumb”的总体形状,“Thumb”指向催化裂缝的底部,它通过疏水相互作用以及几个氢键稳定在这个位置,但也能够移动,从而调节活性位点裂缝的宽度[33]。因此该突变可能使裂缝变窄,阻止底物进入裂缝凹槽处的活性中心。
[1] KUMAR P S, YAASHIKAA P, SARAVANAN A.Isolation, characterization and purification of xylanase producing bacteria from sea sediment[J].Biocatalysis and Agricultural Biotechnology, 2018, 13:299-303.
[2] 徐佳, 高廷, 杨彦博, 等.A152G突变对GH43家族麦氏交替单胞菌木聚糖酶XynZT-2的影响[J].食品与发酵工业, 2022, 48(3):50-55.
XU J, GAO T, YANG Y B, et al.Effect of A152G mutation on XynZT-2, xylanase from the GH43 family of Alternomonas macleodii[J].Food and Fermentation Industries, 2022, 48(3):50-55.
[3] 何敏超, 刘云云, 陈小燕.木聚糖酶产生菌的筛选、发酵及酶学性质[J].中国酿造, 2019, 38(12):107-109.
HE M C, LIU Y Y, CHEN X, et al.Screening, fermentation and enzymatic properties of xylanase-producing strain[J].China Brewing, 2019, 38(12):107-109.
[4] 陈海珊, 张国柱, 王磊, 等.木聚糖的提取分离和应用研究进展[J].广西科学, 2022, 29(2):209-220.
CHEN H S,ZHANG G Z,WANG L, et al.Research progress on extraction, separation and application of xylan[J].Guangxi Sciences, 2022, 29(2):209-220.
[5] 王文霞, 张慧敏, 张慧君, 等.阿拉伯木聚糖和壳聚糖复合膜的制备及性能研究[J].食品与发酵工业, 2021, 47(18):207-214.
WANG W X, ZHANG H M, ZHANG H J, et al.Preparation and properties of Arabinoxylan and chitosan composite film[J].Food and Fermentation Industries, 2021, 47(18):207-214.
[6] 李阳阳, 刘松, 尹小强, 等.黑曲霉木聚糖酶在大肠杆菌中的胞外表达和酶学性质研究[J].食品与发酵工业, 2022, 48(6):1-7.
LI Y Y, LIU S, YIN X Q, et al.Extracellular expression of Aspergillus niger xylanase in Escherichia coli and enzymatic property characterization[J].Food and Fermentation Industries, 2022, 48(6):1-7.
[7] OMISORE S O, FABUNMI T B, AYODEJI A O, et al.Production and biochemical characterization of partially purified cellulase-free, thermo-acidophilic endoxylanase from Lysinibacillus fusiformis strain TB7 using kolanut husk as feedstock[J].Heliyon, 2022, 8(10):e11106.
[8] 吴凤梅, 周燕霞, 姚冬生, 等.定向引入N-糖基化位点改进黑曲霉β-1,4-内切木聚糖酶热稳定性[J].微生物学通报, 2020, 47(7):2128-2139.
WU F M, ZHOU Y X, YAO D S, et al.Improvement of thermal stability of endo-β-1,4-xylanase from Aspergillus niger by introducing N-glycosylation site[J].Microbiology China, 2020, 47(7):2128-2139.
[9] 李吉萍, 包昌杰, 陈光, 等.木聚糖酶异源表达的研究进展[J].中国生物工程杂志, 2019, 39(7):91-99.
LI J P, BAO C J, CHEN G, et al.Research advances in heterologous expression of xylanase[J].China Biotechnology, 2019, 39(7):91-99.
[10] LONG C N, LIU J, GAN L H, et al.Optimization of xylanase production by Trichoderma orientalis using corn cobs and wheat bran via statistical strategy[J].Waste and Biomass Valorization, 2019, 10(5):1 277-1 284.
[11] DEVI S, DWIVEDI D, BHATT A K.Utilization of agroresidues for the production of xylanase by Bacillus safensis XPS7 and optimization of production parameters[J].Fermentation, 2022, 8(5):221.
[12] TIWARI S, SINGH R, YADAV J, et al.Three-step purification and characterization of organic solvent-tolerant and alkali-thermo-tolerant xylanase from Bacillus paramycoides T4 [MN370035][J].Catalysts.2022;12(7):749.
[13] 刘艳, 张婷, 阚婷婷, 等.饱和突变改善米曲霉11家族木聚糖酶AEx11A的催化特性[J].食品与生物技术学报, 2020, 39(5):31-37.
LIU Y, ZHANG T, KAN T T, et al.Improvement in the catalytic properties of A GH 11 xylanase AEx11A, from Aspergillus oryzae by saturated mutagenesis[J].Journal of Food Science and Biotechnology, 2020, 39(5):31-37.
[14] 郑亚伦, 夏瑛, 李良, 等.源于解淀粉芽孢杆菌酸性木聚糖酶酶学性质的研究[J].食品与发酵工业, 2020, 46(24):58-65. `
ZHENG Y L, XIA Y, LI L, et al.Enzymatic properties of acid resistant xylanase from Bacillus amyloliquefaciens[J].Food and Fermentation Industries, 2020, 46(24):58-65.
[15] LI C J, KUMAR A, LUO X, et al.Highly alkali-stable and cellulase-free xylanases from Fusarium sp.21 and their application in clarification of orange juice[J].International Journal of Biological Macromolecules, 2020, 155:572-580.
[16] 田文卓, 王国栋, 马俊, 等.碱性木聚糖酶研究进展[J].中国生物工程杂志, 2022, 42(3):124-131.
TIAN W Z, WANG G D, MA J, et al.Research progress of alkaline xylanase[J].China Biotechnology, 2022, 42(3):124-131.
[17] 张晓龙, 王晨芸, 刘延峰, 等.基于合成生物技术构建高效生物制造系统的研究进展[J].合成生物学, 2021, 2(6):863-875.
ZHANG X L, WANG C Y, LIU Y F, et al.Research progress of constructing efficient biomanufacturing system based on synthetic biotechnology[J].Synthetic Biology Journal, 2021, 2(6):863-875.
[18] PAGOLU R, SINGH R, SHANMUGAM R, et al.Site-directed lysine modification of xylanase for oriented immobilization onto silicon dioxide nanoparticles[J].Bioresource Technology, 2021, 331:125063.
[19] ITO K, OGASAWARA H, SUGIMOTO T, et al.Purification and properties of acid stable xylanases from Aspergillus kawachii[J].Bioscience, Biotechnology, and Biochemistry, 1992, 56(4):547-550.
[20] MILLER G L.Use of dinitrosalicylic acid reagent for determination of reducing sugar[J].Analytical Chemistry, 1959, 31(3):426-428.
[21] LI Q, WU Q H, SUN B G, et al.Effect of disulfide bridge on hydrolytic characteristics of xylanase from Penicillium janthinellum[J].International Journal of Biological Macromolecules, 2018, 120(Part A):405-413.
[22] ITO K, IWASHITA K, IWANO K.Cloning and sequencing of the xynC gene encoding acid xylanase of Aspergillus kawachii[J].Bioscience, Biotechnology, and Biochemistry, 1992, 56(8):1338-1340.
[23] OKA T, EKINO K, FUKUDA K, et al.Draft genome sequence of the formaldehyde-resistant fungus Byssochlamys spectabilis No.5 (Anamorph Paecilomyces variotii No.5) (NBRC109023)[J].Genome Announcements.2014, 2(1):e01162-e01113.
[24] KIMURA T, ITO J, KAWANO A, et al.Purification, characterization, and molecular cloning of acidophilic xylanase from Penicillium sp.40[J].Bioscience, Biotechnology, and Biochemistry, 2000, 64(6):1230-1237.
[25] LUNIN V V, WANG H T, BHARADWAJ V S, et al.Molecular mechanism of polysaccharide acetylation by the Arabidopsis xylan O-acetyltransferase XOAT1[J].The Plant Cell, 2020, 32(7):2367-2382.
[26] 李晓凯, 刘霞, 常红娟, 等.拟南芥START结构域亚家族生物信息学分析及功能[J].河北农业大学学报, 2022, 45(2):76-85.
LI X K, LIU X, CHANG H J, et al.Bioinformatics analysis of the subfamily containing START domain and study on its function in Arabidopsis[J].Journal of Hebei Agricultural University, 2022, 45(2):76-85.
[27] 兰玉婷, 王双蕾, 李征珍, 等.沙冬青属植物响应非生物胁迫的蛋白质组学研究进展[J].生物技术通报, 2019, 35(1):112-119.
LAN Y T, WANG S L, LI Z Z, et al.Research advances in proteomics of Ammopiptanthus in responses to abiotic stresses[J].Biotechnology Bulletin, 2019, 35(1):112-119.
[28] GUO Z H, LIU M, XIANG X W, et al.Effects of inulins with various molecular weights and added concentrations on the structural properties and thermal stability of heat-induced gliadin and glutenin gels[J].LWT, 2021, 149:111891.
[29] 覃拥灵, 何海燕.米曲霉耐热木聚糖酶纯化及酶学特性研究[J].工业微生物, 2011, 41(4):77-82.
QIN Y L, HE H Y.Purification and characterization of a thermostable xylanase from Aspergillus oryzae[J].Industrial Microbiology, 2011, 41(4):77-82.
[30] 黄雪芹, 左勇, 张强, 等.耐酸Bacillus velezensis P7木聚糖酶基因在枯草芽孢杆菌中的表达及优化[J].现代食品科技, 2021, 37(12):40-48;32.
HUANG X Q, ZUO Y, ZHANG Q, et al.Expression of the acid-resistant xylanase gene from Bacillus velezensis P7 in Bacillus subtilis and optimization of its production[J].Modern Food Science and Technology, 2021, 37(12):40-48;32.
[31] 王金华. 黑曲霉酸性木聚糖酶基因在毕赤酵母中的高效表达及其表达产物性质及应用特性研究[D].昆明:云南师范大学, 2003.
WANG J H.Cloning and overexpression of gene encoding acidic endo-β-1, 4-xylanase from Aspergillus niger (N402)in Pichia pastoris and analysis of properties of the recombinant xylanase[D].Kunming:Yunnan Normal University,2023.
[32] GIVER L, GERSHENSON A, FRESKGARD P O, et al.Directed evolution of a thermostable esterase[J].Proceedings of the National Academy of Sciences, 1998, 95(22):12 809-12 813.
[33] JUGE N, PAYAN F, WILLIAMSON G.XIP-I, a xylanase inhibitor protein from wheat:A novel protein function[J].Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2004, 1696(2):203-211.