凝乳酶(milk-clotting enzyme,MCE)是一种广泛应用于干酪乳制品加工的酶类,其主要功能是通过特异性切割酪蛋白分子中的特定肽键,从而破坏乳蛋白稳定的胶体结构,形成凝块并释放乳清,使干酪形成独特的固体结构和质地[1]。传统使用的凝乳酶是主要来源于小牛胃部的皱胃酶(calf rennet,EC3.4.23.4),该酶属于天冬氨酸蛋白酶类,能特异性水解κ-酪蛋白中的特定肽键(Phe105-Met106),导致牛奶凝结[2]。动物凝乳酶由于制备过程复杂、原料来源受限且成本较高,这导致了对于其替代品的迫切需求。因此,寻找制备简单、价格较低、凝乳活性良好的动物凝乳酶替代酶制剂变得十分重要。
目前其他来源的替代品主要有重组小牛凝乳酶、微生物凝乳酶和植物凝乳酶。重组凝乳酶是利用重组DNA技术,将小牛皱胃酶的mRNA反转录为cDNA,然后克隆后转化到食品安全的微生物中进行表达,通过发酵后获得酶制剂产品。由于一些国家对转基因技术用于食品加工的严格管控,限制了重组小牛凝乳酶在干酪加工中的应用。微生物产凝乳酶具有制备过程相对简单、生产成本低廉和可实现规模化生产等优点,较植物凝乳酶有生产成本上优势,是传统小牛皱胃酶的较理想替代品。目前已报道的微生物凝乳酶主要包括细菌凝乳酶和真菌凝乳酶。细菌凝乳酶的研究主要集中在芽孢杆菌属(Bacillus)细菌中,如枯草芽孢杆菌(B.subtilis)[3-4]和解淀粉芽孢杆菌(B.amyloliquefaciens)[5]等。丝状真菌具有广谱的产酶能力,并且能够将酶分泌到细胞外,便于酶的分离和纯化,因此是较理想的凝乳酶产生菌来源。已报道的丝状真菌包括有米黑根毛霉(Rhizomucor miehei)[6]、微小毛霉(Mucor pusillus)[7]和米曲霉(Aspergillus oryzae)[8]等。然而,目前微生物凝乳酶的主要问题是许多微生物凝乳酶表现出较高的蛋白酶活性,只有少部分的微生物凝乳酶适合用于干酪的生产。蛋白水解活性高导致在干酪成熟过程中酪蛋白的水解,引起干酪质地不良、产量降低,并可能形成苦味的多肽,影响干酪风味口感[9]。因此,如何从微生物中寻找和发现具有良好的凝乳特性和低蛋白水解活性,并适用于干酪生产的凝乳酶,是当前微生物凝乳酶研究的热点问题。
传统腐乳发酵是在半开放的体系中进行的,利用环境中的微生物作用将大豆蛋白降解为多肽和氨基酸等化合物,从而形成独特的滋味和香气。传统腐乳发酵菌群中含有丰富的具有蛋白水解和降解多肽活性的微生物[10],因此从腐乳来源菌群中筛选是获得产凝乳酶微生物的有效途径之一。目前从腐乳发酵菌群中筛选凝乳酶产生菌株的研究较少。为寻找优良的凝乳酶产生菌,本研究以从传统腐乳分离的霉菌为出发菌株,筛选具有高凝乳活性和低蛋白水解活性的菌株;通过分子生物学方法对筛选到的菌株进行鉴定,并分离纯化其所产的凝乳酶,研究该酶的酶学性质。本研究旨在拓展产凝乳酶的菌种资源,为微生物凝乳酶的研究和应用提供理论和实践的依据。
1 材料与方法
1.1 材料与试剂
马铃薯葡萄糖琼脂(potato dextrose agar,PDA)培养基,杭州微生物试剂有限公司;真菌基因组DNA快速抽提试剂盒、Bradford法蛋白质定量检测试剂盒,上海生工生物工程有限公司;ExTaq酶,日本Takara公司;其余试剂为均为分析纯。
1.2 菌种和培养基
1.2.1 菌种
从传统发酵法制备腐乳中分离的霉菌菌株共28株。
1.2.2 培养基
PDA培养基用于霉菌的斜面培养与保藏。筛选培养基:葡萄糖20 g,酵母提取物0.5 g,酪蛋白2 g,磷酸二氢钾1 g,蒸馏水1 000 mL,pH 6.0,121 ℃灭菌15 min,用于产凝乳酶菌株的筛选。发酵培养基:葡萄糖30 g,蛋白胨0.5 g,酵母提取物0.5 g,酪蛋白2 g,磷酸二氢钾1 g,氯化钙0.5 g,硫酸锰0.5 mg,吐温80 1 mL,蒸馏水1 000 mL,pH 6.0,121 ℃灭菌15 min,用于凝乳酶的发酵。
1.3 实验方法
1.3.1 产凝乳酶菌株的筛选与鉴定
挑取保藏于PDA斜面的菌种接种到发酵培养基,于28 ℃,160 r/min的摇床上振荡培养72 h,发酵培养物经抽滤后得到上清液。测定上清液的凝乳酶和蛋白酶活性。
1.3.2 菌株的分子鉴定
斜面菌种接种于发酵培养基中,28 ℃,160 r/min的摇床上振荡培养72 h后离心收集菌丝体,用无菌生理盐水洗涤2次,放入研钵内加入液氮后研磨成粉,按照真菌基因组DNA快速抽提试剂盒描述的方法提取基因组DNA。
采用通用引物ITS1(5′-TCCGTAGGTGAACCTGCGG -3′)和ITS4(5′-CCTCCGCTTATTGATATGC-3′)PCR扩增ITS序列。ITS扩增体系(25 μL):10×buffer 2.5 μL,dNTPs 2 μL,ITS1和ITS4各1 μL,模板DNA 2.0 μL,ExTaq 0.1 μL。扩增条件:94 ℃预变性4 min;94 ℃变性1 min,55 ℃退火40 s,在72 ℃延伸90 s,35个循环;72 ℃延伸10 min。
采用引物BMS05(5′-TTAATTTTGGTTCNGATTGAACG-3′)和BMS173(5′-TGCTATGACTTTTGAGATGTTAC-3′)PCR扩增线粒体核糖体小亚基基本全长序列(mt SSU rDNA)[11]。扩增体系(25 μL):10×buffer 2.5 μL,dNTPs 2 μL,BMS05和BMS173各2 μL,模板DNA 3.0 μL,ExTaq 0.1 μL。扩增条件:94 ℃预变性4 min;94 ℃变性30 s,50 ℃退火30 s,72 ℃延伸2 min,35个循环;72 ℃延伸10 min。
PCR产物经1.0%琼脂糖凝胶电泳验证为清晰唯一的条带后,送上海生工生物工程有限公司测序。测序结果提交NCBI数据库进行BLAST比对分析。采用MEGA11.0软件构建进化树。
1.3.3 凝乳酶活力测定
凝乳酶活力测定参考ARIMA等[12]描述的方法稍作修改。脱脂奶粉配制成100 g/L质量浓度制成复原乳,作为凝乳酶活性测定底物。将装有2 mL复原乳的试管放入35 ℃水浴中预热5 min,然后取0.2 mL待测酶液加入试管中,混匀后开始反应后计时。观察凝乳状况,当管壁上可观察到刚出现清晰的絮状沉淀时即为凝乳终点,并记录凝乳时间,以蒸馏水代替酶液作为空白对照。凝乳酶活力定义为40 min内凝结2 mL 100 g/L脱脂乳的酶量为一个活力单位(U/mL)。以单位蛋白浓度的酶液所测得的酶活力力表示待测样品的凝乳酶比活力(U/mg蛋白)。酶学性质试验中,在不同条件下处理时,用相对酶活力表示不同条件下凝乳酶活性的差异:以最高的凝乳酶活力定义为 100%,计算其他不同条件下的相对酶活力。凝乳酶活力的计算如公式(1)所示:
凝乳酶活力/(U/mL)=2 400/凝乳时间(s)×2/0.2
(1)
式中:2 400表示40 min换算成秒,s;2表示所用底物(脱脂乳)的体积,mL;0.2表示加入的酶液的体积,mL。
1.3.4 蛋白酶活力测定
参考GB/T 23527.1—2023《酶制剂质量要求 第1部分:蛋白酶制剂》中的描述的蛋白水解活力的测定方法进行。
1.3.5 蛋白含量测定
采用Bradford法对蛋白进行定量测定。
1.3.6 凝乳酶分离纯化
1.3.6.1 粗酶液的发酵制备
挑取斜面菌丝接种于发酵培养基,于30 ℃,180 r/min摇床上培养84 h,液体培养物经抽滤后收集上清液,即得到粗酶液。
1.3.6.2 硫酸铵分级沉淀盐析
将装有1 L粗酶液的烧杯置于冰水浴中预冷后,分别加入硫酸铵至饱和度为20%、60%、80%、100%,依次离心并收集沉淀,得到相应饱和度的沉淀。各沉淀组分分别溶解于20 mL 50 mmol/L pH 6.5磷酸盐缓冲液中,转入3 kDa透析袋采用相同磷酸盐缓冲液透析脱盐。测定透析后各沉淀组分的蛋白质含量和凝乳酶活力,凝乳酶活力高的组分冷冻干燥后-20 ℃保藏备用。
1.3.6.3 DEAE-Sepharose Fast Flow阴离子交换层析
采用50 mmol/L pH 6.5磷酸盐缓冲液预平衡装好的DEAE-Sepharose Fast Flow层析柱(2.6×15 cm),脱盐冻干盐析组分溶解于50 mmol/L pH 6.5磷酸缓冲液,并经0.45 μm微孔滤膜过滤后上样。用150 mL 50 mmol/L pH 6.5磷酸缓冲液清洗除去未吸附的蛋白。用含有0 mol/L到1 mol/L NaCl的50 mmol/L pH 6.5磷酸盐缓冲液进行梯度洗脱,合并各个280 nm波长下有蛋白吸收峰洗脱液测定凝乳酶活力。收集具有凝乳酶活性的组分,冷冻干燥后-20 ℃保藏。
1.3.6.4 Sephadex G100凝胶过滤层析
采用含有0.2 mol/L NaCl的50 mmol/L pH 6.5磷酸盐缓冲液预平衡Sephadex G100凝胶过滤柱(1.6×60 cm),经上一步纯化后的冻干粉溶解于0.2 mol/L NaCl的50 mmol/L pH 6.5磷酸盐缓冲液,上样后用含有0.2 mol/L NaCl的50 mmol/L pH 6.5磷酸盐缓冲液进行洗脱,流速为0.5 mL/min,收集具有凝乳酶活力的洗脱液,冷冻干燥后-20 ℃保存。
1.3.6.5 凝乳酶纯度检测与分子质量测定
采用SDS-PAGE凝胶电泳进行纯度检测和分子质量测定。采用浓缩胶浓度为5%,分离胶浓度为12%,电压80 V,电泳后采用考马斯亮蓝染色,凝胶成像仪拍照,并采用Quantity One 4.6软件估算分子质量大小。
1.3.7 酶学性质试验
1.3.7.1 最适温度与热稳定性
纯化后的凝乳酶冻干粉配制成0.2 mg/mL质量浓度,以100 g/L脱脂牛奶为底物,在20~65 ℃的不同温度下测定凝乳酶活力,考察酶的最适温度。为评价酶的热稳定性,将酶液在35~70 ℃下孵育30 min,在45 ℃下测定凝乳酶活力;以将酶液置于4 ℃ 30 min后测定作为空白对照。
1.3.7.2 最适pH与pH稳定性
以100 g/L脱脂牛奶为底物,在pH 5.5~8.0的不同pH值、45 ℃下测定凝乳酶活力,考察酶的最适pH。分别用缓冲液将酶液的pH值调整为4.0~8.0的不同pH值条件,在4 ℃下放置6 h后,45 ℃下测定凝乳酶活力,评价酶的pH稳定性。
1.3.7.3 金属离子的影响
分别在100 g/L脱脂牛奶的底物中加入不同金属离子的氯化物或硫酸盐,使金属离子的终浓度达10 mmol/L,以不加金属离子的作为空白对照,于45 ℃下测定凝乳酶活力,考察金属离子的影响。
1.3.7.4 蛋白酶抑制剂的影响
在凝乳酶活力测定反应体系中,分别加入不同的蛋白酶抑制剂至其终浓度为1 mmol/L,以不加抑制剂作为空白对照,在45 ℃下测定凝乳酶活力,评价抑制剂的影响。
1.4 数据处理与分析
数据以平均值±标准差的形式表示,采用SPSS 23.0软件进行单因素方差分析(ANOVA),P<0.05表示为显著水平。
2 结果与分析
2.1 产凝乳酶菌株的筛选
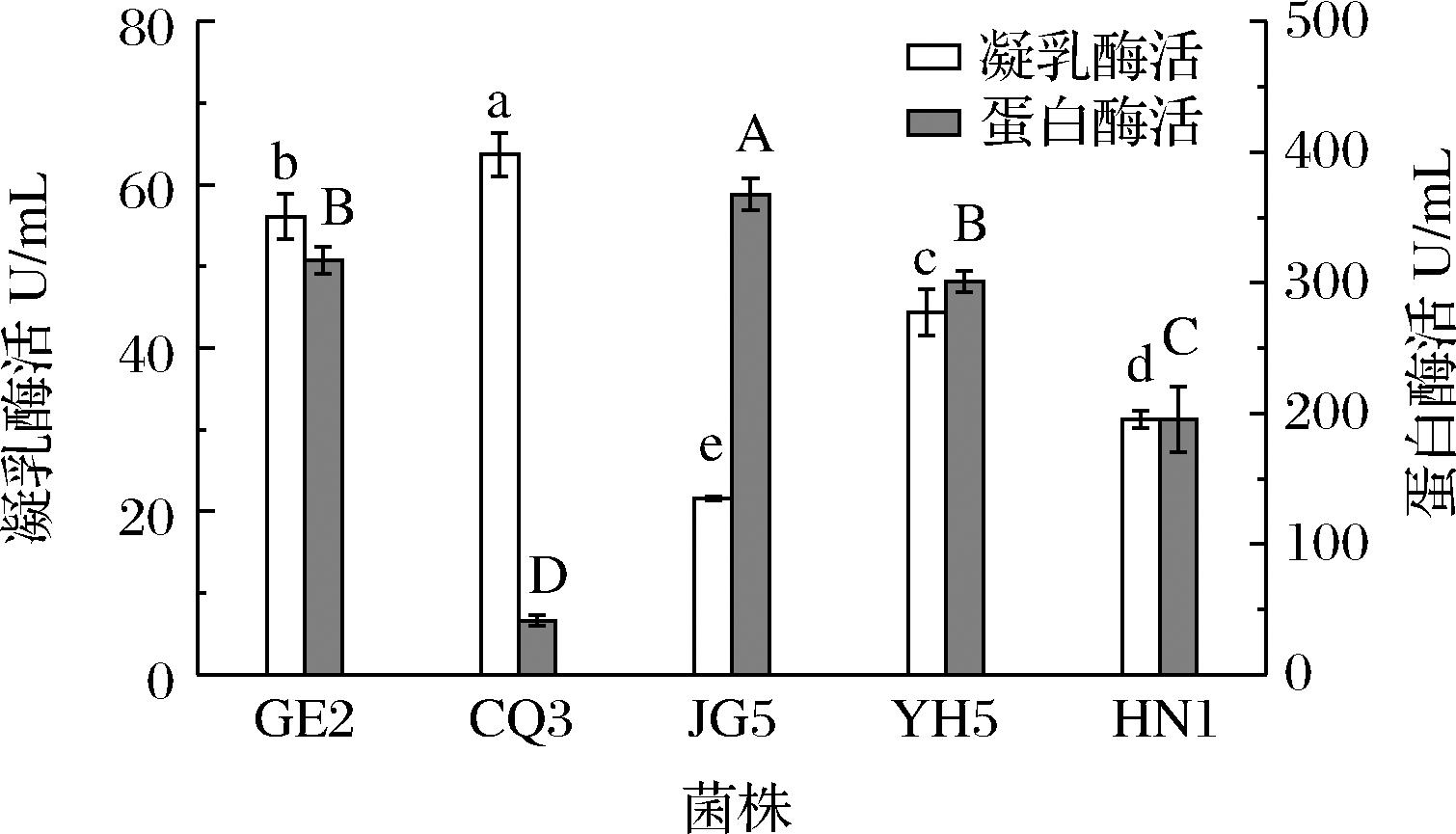
对从传统发酵法制备腐乳中分离的28株霉菌菌株进行了筛选试验,其中5株菌具有凝乳酶活性,结果如附图1所示(https://doi.org/10.13995/j.cnki.11-1802/ts.036810)。在这5株菌中,菌株CQ3展现出了最强的凝乳酶活力力,达到了63.1 U/mL,而菌株JG5的凝乳酶活力力最弱,为21.6 U/mL。在蛋白水解活力方面,菌株JG5的蛋白酶活性最强,而CQ3的蛋白水解能力最弱。CQ3凝乳酶活力高而蛋白水解能力较弱,在筛选出的5株霉菌中表现最好,符合产凝乳酶菌株的筛选要求。
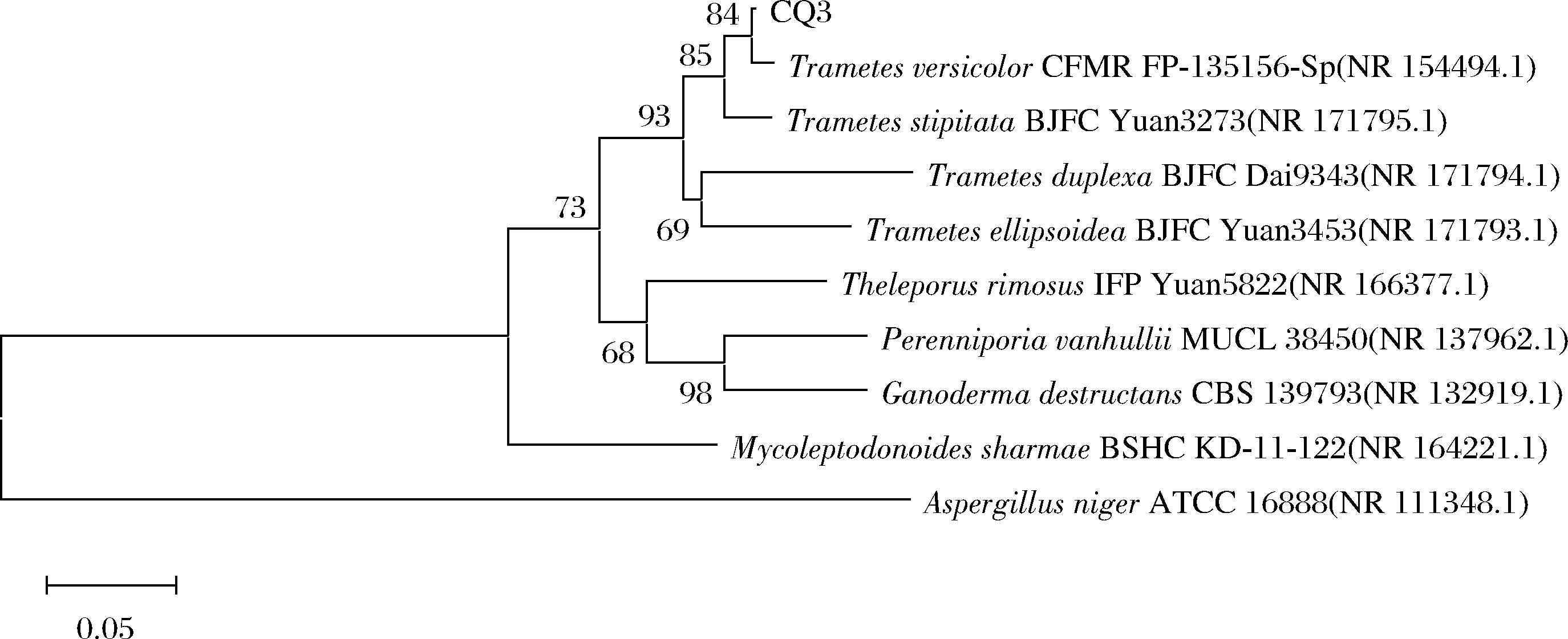
图1 菌株CQ3基于ITS序列的系统发育进化树
Fig.1 Phylogenetic tree based on ITS sequence of strain CQ3
2.2 产凝乳酶菌株的鉴定
菌株CQ3接种于PDA平板28 ℃培养7~8 d后可铺满整个平板,菌落形态呈白色绒毛状,较薄,质地较紧密贴于培养基上,菌落背面呈浅灰白色,未发现分泌色素。挑去菌丝体经石碳酸棉兰染色后的显微镜下观察,菌丝体分支繁茂,菌丝体无隔,无假根,未发现有孢子囊及常见霉菌(如曲霉和青霉等)的分生孢子头形态。
采用特征引物分别对菌株CQ3的内源转录间隔区(internally transcribed spacer,ITS)和线粒体小亚基核糖体DNA(mitochondrial small subunit ribosomal DNA,mt SSU rDNA)序列进行扩增,经测序后分别得到572 bp和1 402 bp长度的核酸序列。CQ3的ITS序列提交NCBI经过BLAST进行比对,结果显示与Trametes versicolor(NR_154494.1)的相似度(identity)最高,达98.77%,与该目标序列的覆盖度(query coverage)达99%。CQ3的mt SSU rDNA序列提交NCBI经过BLAST进行比对,结果显示与Trametes versicolor(AF042324.2)的相似度最高,达98.78%,与该目标序列的覆盖度达98%。以黑曲霉(Aspergillus niger)为外群,选取BLAST比对后与CQ3序列相似度排名前列的物种序列分别构建基于ITS和mt SSU rDNA序列的系统发育进化树。由图1可知,CQ3与Trametes versicolor CFMR FP-135156-Sp(NR 154494.1)的遗传距离最近。由图2可知,CQ3与Trametes versicolor CBS 292.33(AF042324.2)的遗传距离最近。综合以上结果,ITS和mt SSU rDNA测序后BLAST比对结果与进化树分析结果相吻合,表明CQ3属于栓菌属(Trametes)的变色栓菌(Trametes versicolor)。
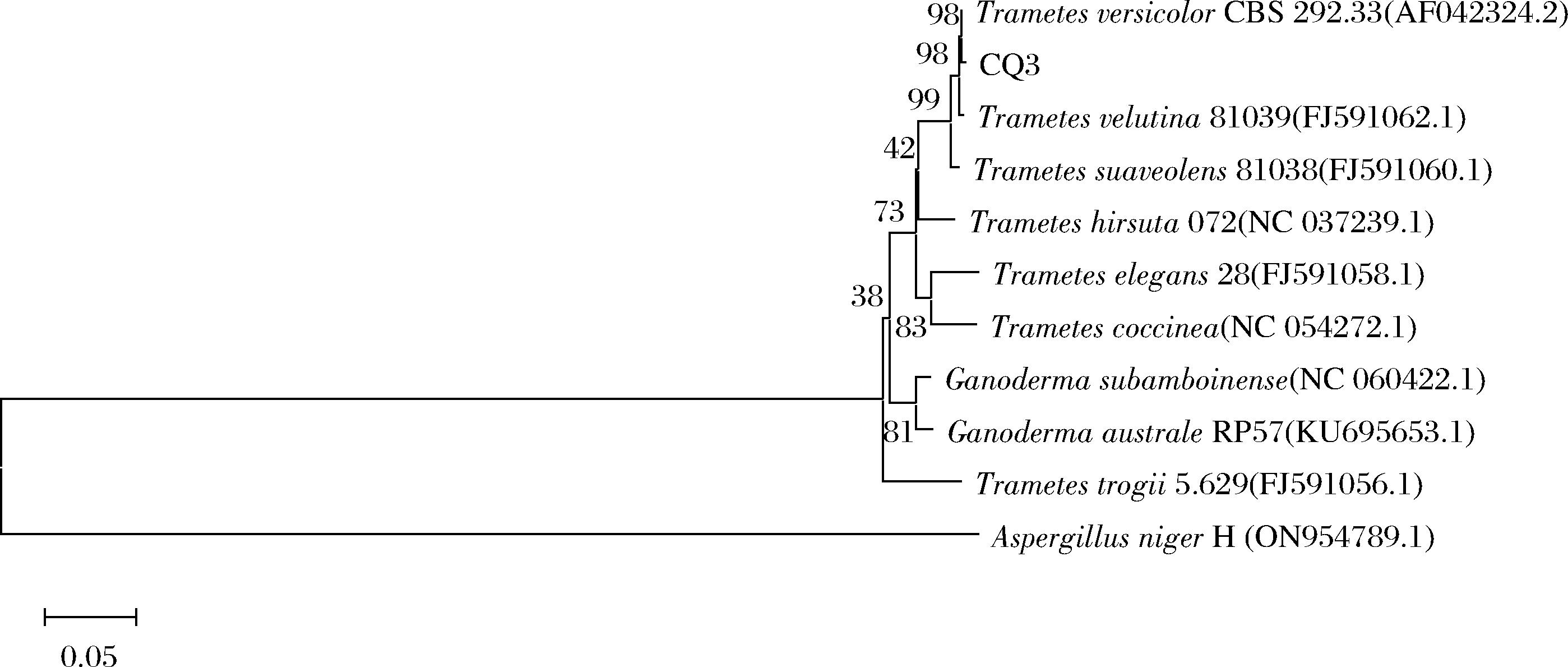
图2 菌株CQ3基于mt SSU rDNA序列的系统发育进化树
Fig.2 Phylogenetic tree based on mt SSU rDNA sequence of strain CQ3
2.3 栓菌凝乳酶的分离与纯化
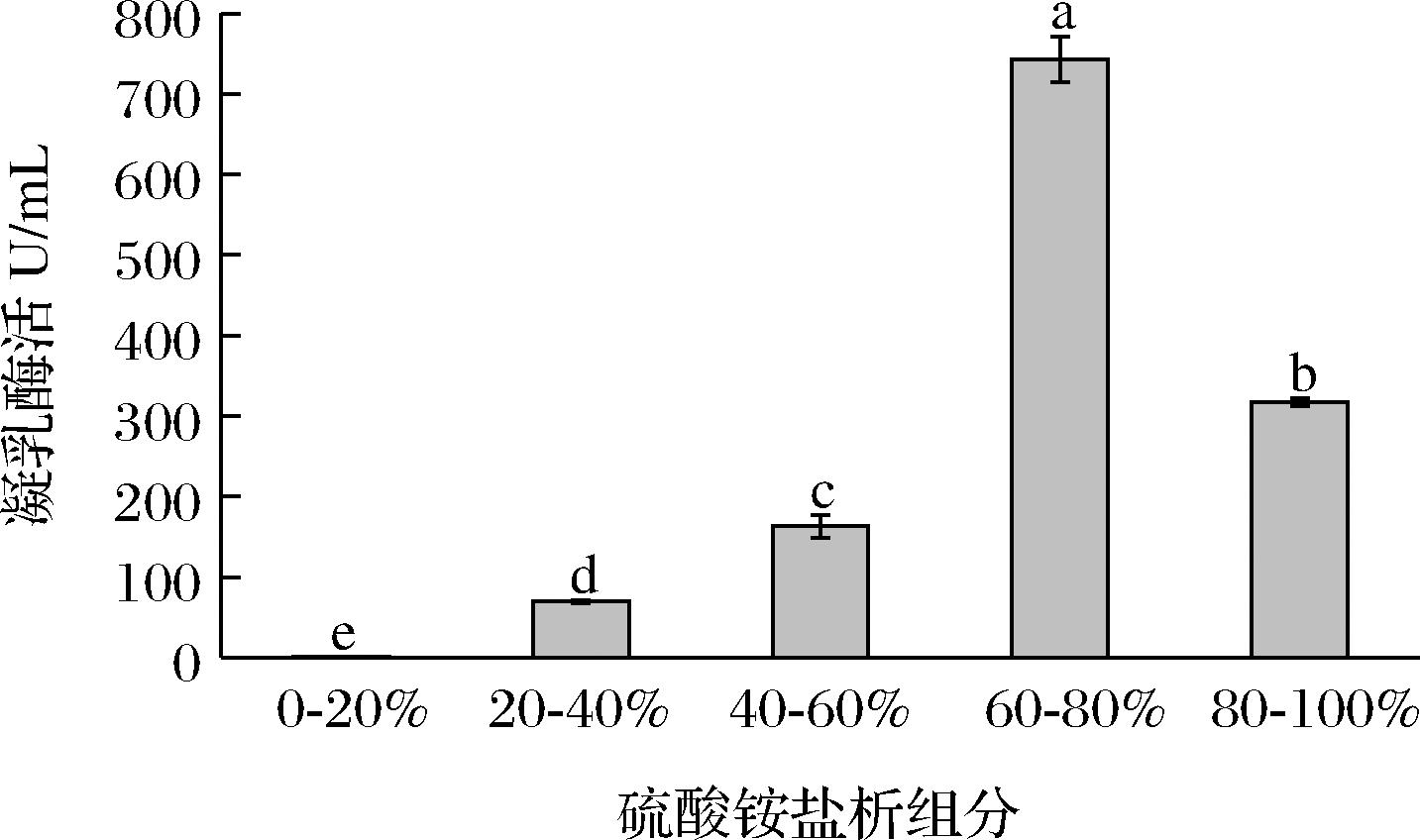
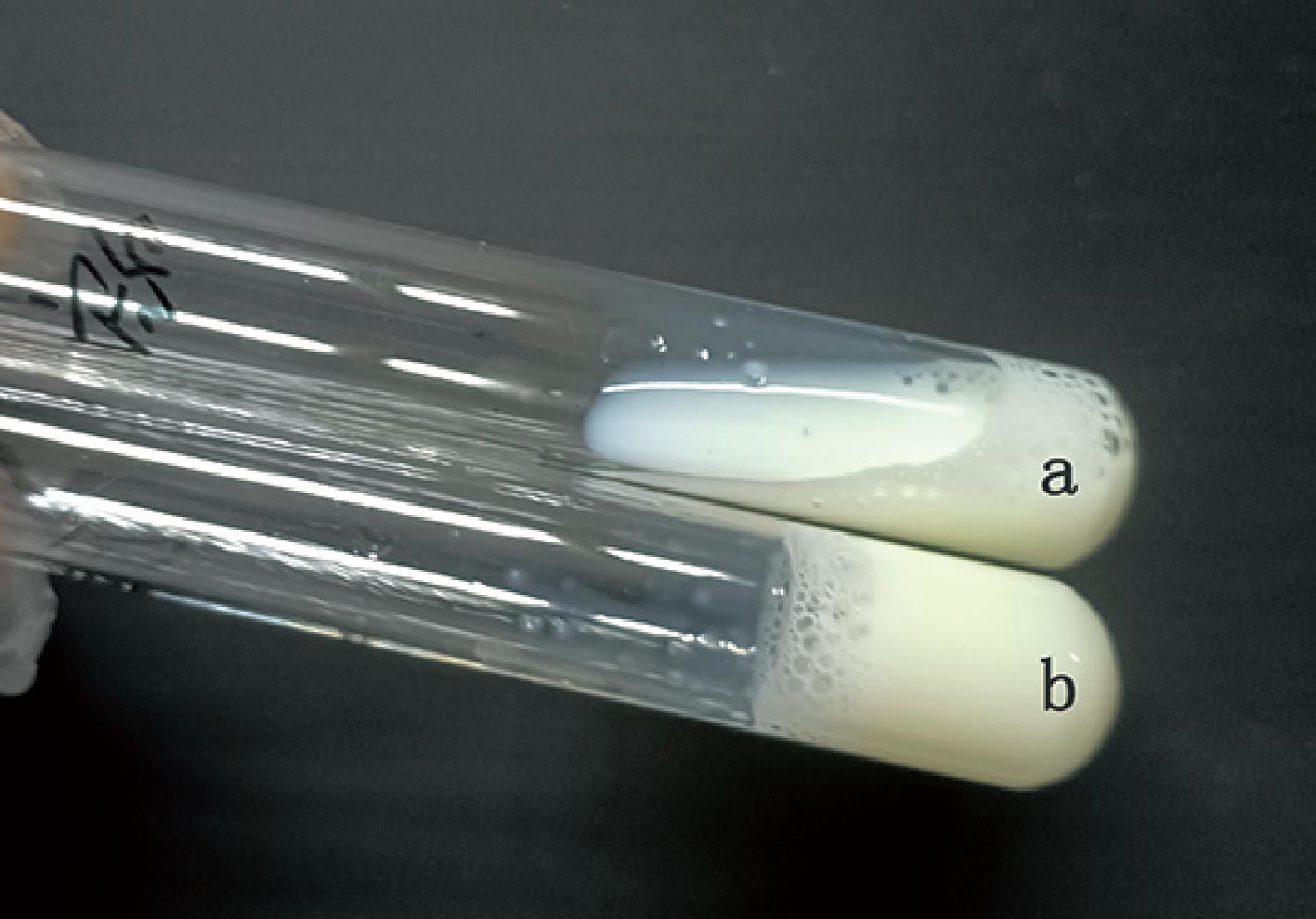
粗酶液经硫酸铵分级沉淀后,各级沉淀组分溶解后透析脱盐和浓缩后测定凝乳酶活力,结果如附图2(https://doi.org/10.13995/j.cnki.11-1802/ts.036810)。除饱和度为0~20%的组分外,其他各组分均能测定到凝乳酶活力,其中饱和度60%~80%组分凝乳酶活力显著高于其他组分(P<0.05),表明目标酶蛋白主要集中于该饱和度下的盐析沉淀中。将60%~80%饱和度分离组分上样于DEAE-Sepharose Fast Flow阴离子交换柱,采用NaCl浓度梯度洗脱后收集到3个蛋白峰D1、D2和D3(图3-a),其中D3峰组分的凝乳酶活性最高,显著高于其余的2个峰组分(图3-b)。将收集的D3组分冻干并再次溶解后上样于Sephadex G100凝胶过滤层析柱,经分离后收集到4个蛋白峰S1、S2、S3、S4(图4-a),凝乳酶活性检测结果显示蛋白峰S3活性最高(图4-b),表明目标凝乳酶集中在该组分中。为检测纯化后酶的纯度,将纯化后的酶进行SDS-PAGE分析,结果如图5所示,酶蛋白呈现出清晰锐利的单一蛋白染色条带,表明获得的酶纯度高。根据该蛋白条带对应于标准蛋白marker的位置估算其分子质量约为61 kbp(图5)。表1中总结了栓菌凝乳酶的分离与纯化过程,经过硫酸铵分级盐析、透析脱盐、DEAE-Sepharose Fast Flow阴离子交换柱和Sephadex G100凝胶过滤层析柱分离纯化后,得到高纯度的凝乳酶蛋白,纯化倍数达18.47,回收率为7.6%。纯化后的CQ3凝乳酶冻干粉溶于50 mmol/L的pH 6.0磷酸盐缓冲液后进行凝乳试验并检测凝乳酶活力,结果如附图3(https://doi.org/10.13995/j.cnki.11-1802/ts.036810)所示;整个乳液都得到了充分的凝结,形成均匀分布且比较紧密的凝块,有一定的结实度,试管倒置后无流出,未观察到乳中有小的颗粒形成;凝乳酶比活力达(822.1±12.3) U/mg。
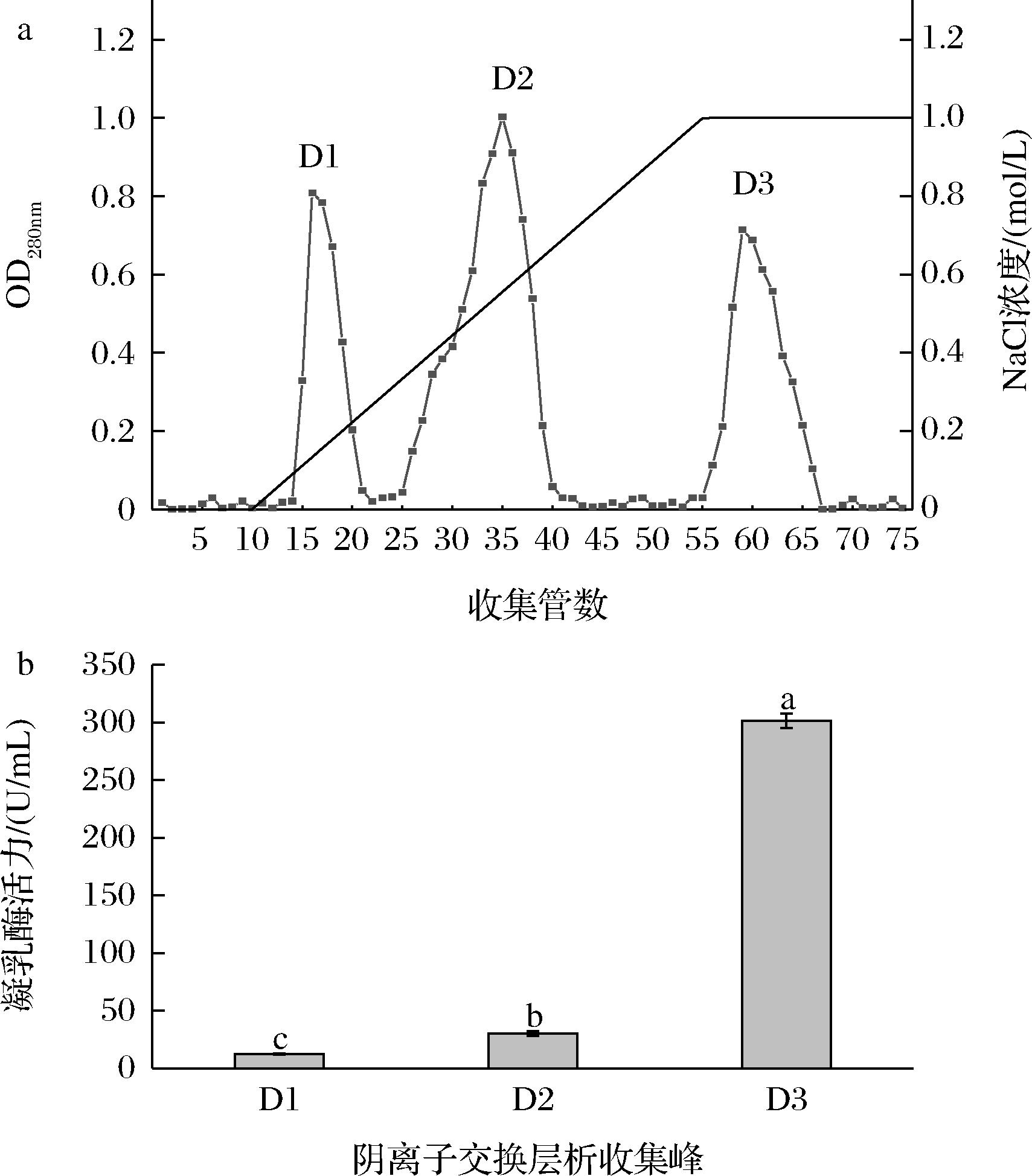
a-收集管数;b-阴离子交换层析收集峰
图3 DEAE-Sepharose Fast Flow阴离子交换层析
Fig.3 Anion exchange chromatography on DEAE-Sepharose Fast Flow column
注:不同小写字母表示凝乳酶活力的显著性差异(P<0.05)(下同)。
表1 变色栓菌CQ3产凝乳酶纯化过程
Table 1 Summery of purification process of milk-clotting enzyme produced by strain CQ3
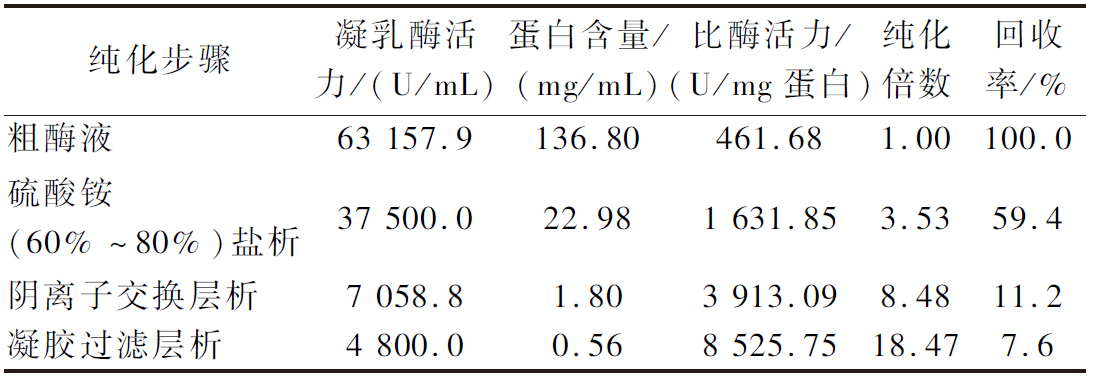
纯化步骤凝乳酶活力/(U/mL)蛋白含量/(mg/mL)比酶活力/(U/mg蛋白)纯化倍数回收率/%粗酶液63 157.9136.80461.681.00100.0硫酸铵(60%~80%)盐析37 500.022.981 631.853.5359.4阴离子交换层析7 058.81.803 913.098.4811.2凝胶过滤层析4 800.00.568 525.7518.477.6

a-收集管数;b-凝胶过滤层析收集峰
图4 Sephadex G100凝胶过滤层析
Fig.4 Gel filtration chromatography on Sephadex G100 column
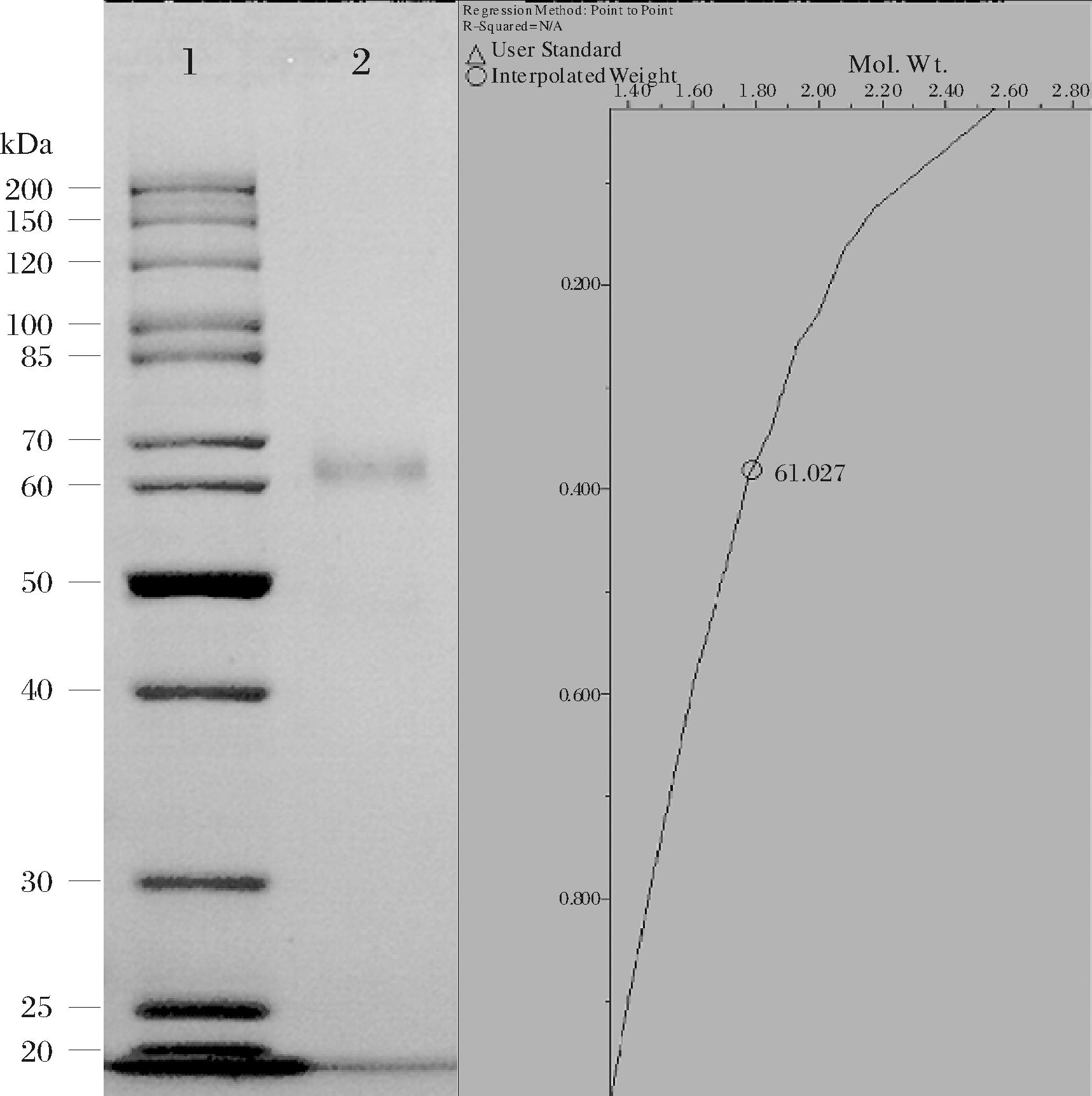
泳道1-蛋白质Marker;泳道2-组分S3
图5 纯化后的栓菌凝乳酶SDS-PAGE图与分子质量分析
Fig.5 SDS-PAGE and molecular weight analysis of purified milk-clotting enzyme
2.4 栓菌凝乳酶的酶学性质
2.4.1 最适温度与热稳定性
变色栓菌CQ3凝乳酶受温度影响和热稳定性的结果如图6所示。从25 ℃开始,凝乳酶活力随温度的升高而增加,到45 ℃达到最大;超过55 ℃后凝乳酶活力损失较快,到65 ℃时凝乳酶活力仅为25%。在35~55 ℃内,酶活性能维持在较高的水平,相对酶活力保持在80%以上(图6-a)。这一结果表明,CQ3凝乳酶最适温度为45 ℃,且能在较宽的温度范围保持高的酶活力,最佳温度使用范围为35~55 ℃。CQ3凝乳酶在35~70 ℃下保温30 min,测定残留凝乳酶活力(图6-b)。在温度低于50 ℃条件下,凝乳酶对热较稳定,处理后的残留相对酶活力随温度下降较慢,50 ℃保温30 min仍然能保持在80%以上相对酶活力。当温度超过55 ℃后,凝乳酶的热稳定性随温度的升高下降较快,65 ℃时30 min残留相对酶活力为20%,70 ℃时30 min凝乳酶活力完全丧失。

a-凝乳酶活力;b-酶的热稳定性
图6 温度对凝乳酶活力和热稳定性的影响
Fig.6 Effect of temperature on activity and thermal stability of milk-clotting enzyme
2.4.2 最适pH与pH稳定性
最适pH和pH稳定性结果如图7所示。CQ3凝乳酶的最适作用pH值为6.5,在pH 5.5~7.5,相对酶活力保持在80%以上,其中在pH 6.0~7.0相对酶活力可维持在90%,表明该凝乳酶在较宽的弱酸性到中性和偏弱碱性pH具有较好的酶活力(图7-a)。在pH值为4.5~7.0,凝乳酶活力稳定性较好,残留活力都在80%以上;而当pH值超过7.5时,残留相对酶活力随pH降低很快,在pH 8.0时残留相对酶活力仅为24%(图7-b)。以上结果表明,CQ3凝乳酶在pH 4.5~7.0有较好的稳定性,能耐受弱酸性和中性环境。
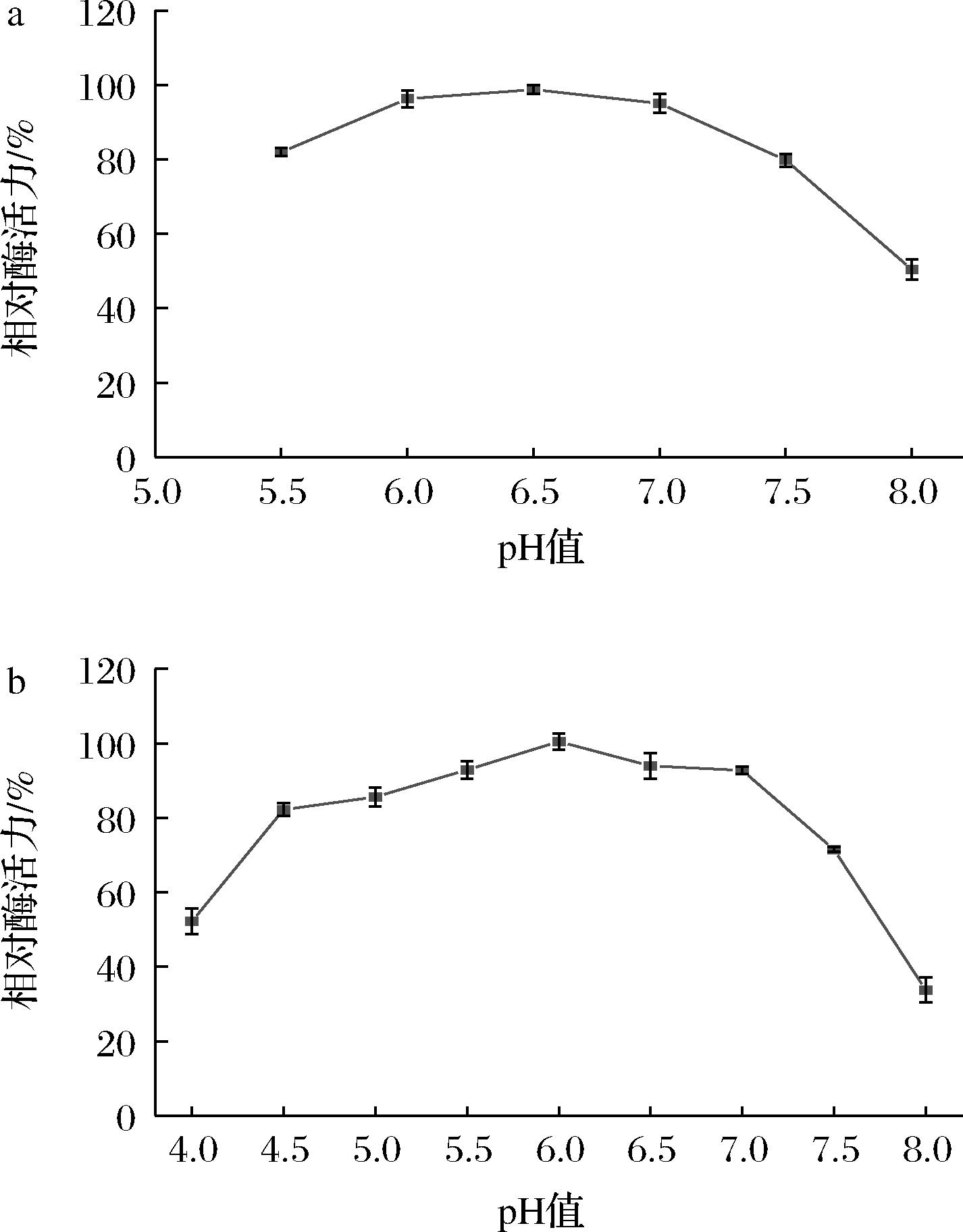
a-凝乳酶活力;b-酶的pH稳定性
图7 pH对凝乳酶活力和pH稳定性的影响
Fig.7 Effect of pH on activity and pH stability of milk-clotting enzyme
2.4.3 金属离子的影响
结果如图8所示。与不添加金属离子的对照(C.K.)相比,在10 mmol/L的浓度下,Ca2+、Mg2+和Ba2+对凝乳酶活力有显著的促进作用(P<0.05),其中Ca2+的促进作用最高,相对酶活力可达到124.4%,而Ba2+对凝乳酶活力有较弱的促进作用。K+、Na+和Zn2+对凝乳酶活力无显著影响(P>0.05)。Cu2+、Mn2+、Fe2+和Pb2+对凝乳酶活力具有显著抑制作用(P<0.05),其中Cu2+的Pb2+抑制作用最强,作用后的相对酶活力分别为对照的6.7%和10.8%,Mn2+和Fe2+的抑制作用较弱,作用后的相对酶活力分别仍能保持在85.7%和87.7%。

图8 金属离子对凝乳酶活力的影响
Fig.8 Effect of metal ions on activity of milk-clotting enzyme produced by Trametes versicolor CQ3
2.4.4 蛋白酶抑制剂的影响
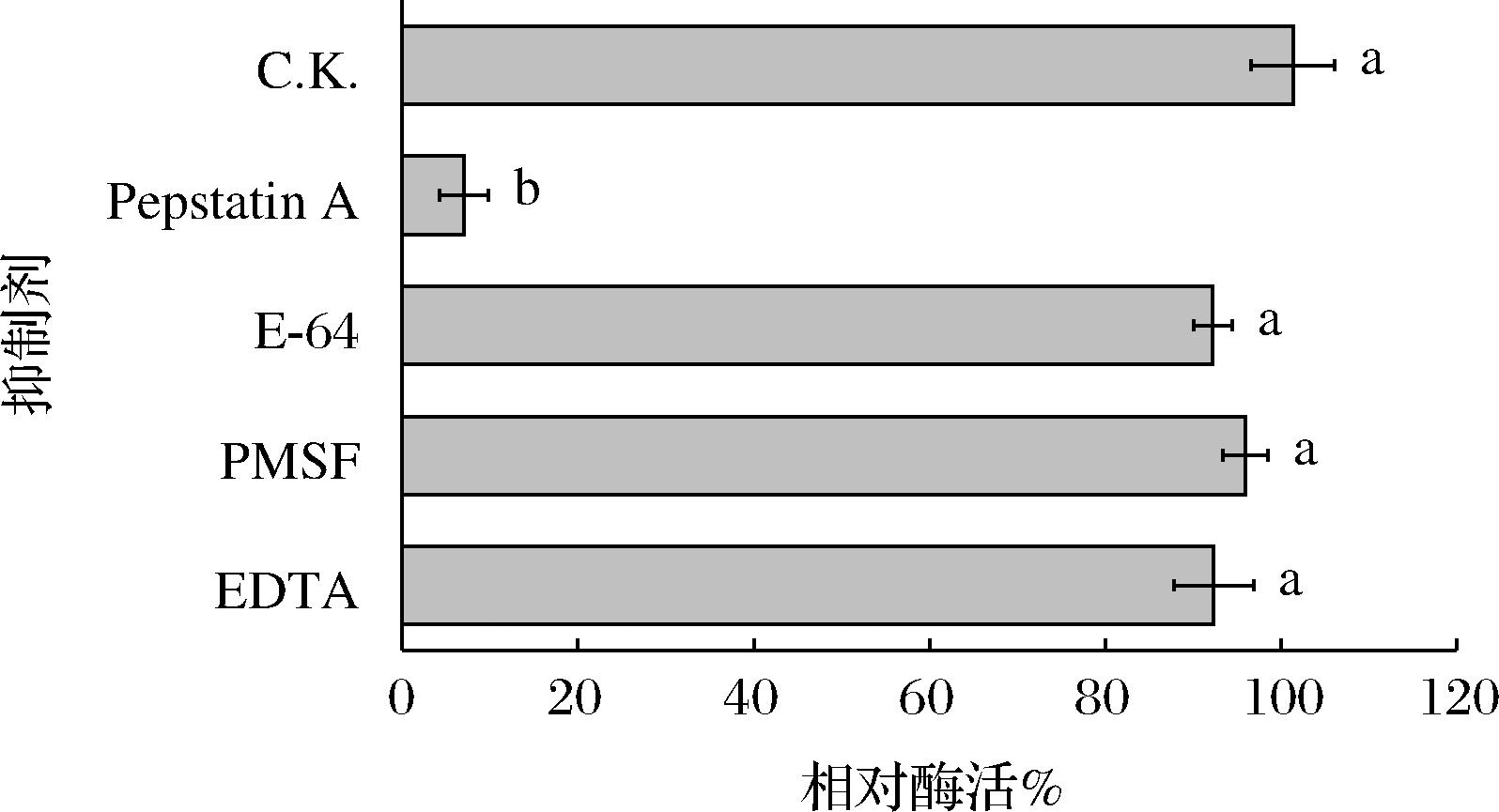
为了验证CQ3凝乳酶所属于的蛋白酶类型,采用丝氨酸蛋白酶、半胱氨酸蛋白酶、天冬氨酸蛋白酶和金属蛋白酶抑制剂进行抑制试验,结果如附图4所示(https://doi.org/10.13995/j.cnki.11-1802/ts.036810)。半胱氨酸蛋白酶抑制剂(E-64)、丝氨酸蛋白酶抑制剂和金属蛋白酶抑制剂对凝乳酶活力无显著性影响(P>0.05),而天冬氨酸蛋白酶抑制剂(pepstatin A)能显著抑制凝乳酶活力(P<0.05),残存凝乳酶活力仅为7%。因此,CQ3凝乳酶属于天冬氨酸蛋白酶。
3 讨论
丝状真菌的传统鉴定方法常根据菌落形态、显微形态以及生理生化反应等具有代表性的典型表型特征,来确定具体的分类属种。由于丝状真菌种类繁多,不同种真菌的表型特征可能很接近而难以区分,同时许多真菌在实验室培养阶段较难完成其完整的生活史,导致无法观察到特有的繁殖体结构(如子实体)特征[13]。因此,大多数情况下仅仅依赖传统方法很难鉴定到种的水平,需要借助于分子系统分类学的方法鉴定。ITS常用于真菌鉴定的分子标记序列,具有较高的遗传稳定性,较为复杂的多态性以及PCR扩增和测序的成功率高等优点[14]。然而单纯使用ITS作为分子标记鉴定真菌有一定的局限性,由于ITS序列较短,采用通用引物ITS1和ITS4扩增后的产物长度常为600 bp左右,因此在一些情况下无法提供足够多的基因序列信息用于比对和系统发育分析。此外,在鉴定某一些亲缘较近的种时其区分度较小[15],仅仅以ITS序列比对和发育分析的方法做鉴定,常会得出不准确甚至错误的结果。mt SSU rDNA是一种适合在家族水平进行系统发育分析的分子标记,在一些真菌如灵芝(Ganoderma)的分子鉴定中,与ITS序列相比,mt SSU rDNA序列能更有效地解析同属内各种的系统发育关系[16]。本研究基于ITS和mt SSU rDNA序列来鉴定菌株CQ3,通过BLAST比对和研究CQ3和比对相似度高的物种之间的系统发育关系,2种序列得到的结果可相互印证,表明鉴定结果有很好的可靠性。
栓菌属(Trametes)真菌在自然界中广泛分布,属内许多种具有较高的药用价值和生物学活性,在生物制药及食品工业等领域具有重要的应用价值。变色栓菌(Trametes versicolor),也称为云芝,是一种具有药用价值的大型真菌,其主要活性成分是多糖,具有免疫调节功能和抑制癌细胞增殖的作用[17-18]。此外,变色栓菌还具有丰富的产酶能力,目前研究主要集中其产漆酶方面。漆酶能够催化酚类和芳香胺类化合物的氧化反应,在食品工业中可以用于改善食品的色度,并降解去除导致啤酒、果汁混浊的酚类物质[19]。变色栓菌属于白腐真菌,常生长于木桩和树木枝干等木制基质上,其孢子随空气传播,而传统的腐乳制作通常利用环境中的霉菌进行自然发酵,因此本研究从传统发酵腐乳中分离出的霉菌中筛选到了能够产生凝乳酶的变色栓菌。目前还没有关于变色栓菌产凝乳酶、酶的分离纯化和酶学性质的相关报道。优良微生物凝乳酶需具备有高凝乳酶活性和低蛋白酶活性。如果凝乳酶制剂的蛋白水解活性较高,可能会在干酪成熟过程中持续水解酪蛋白,释放出有苦味的多肽,从而影响干酪成品的口感和风味[20]。在本研究中,筛选鉴定的变色栓菌CQ3表现出良好的凝乳酶活性和低蛋白酶活性,显示出在实际中应用的潜力。凝乳酶的最佳生化反应条件对于其在实际应用中的价值至关重要,能够在凝乳加工的pH和温度条件下保持活性和稳定性是保证其实际应用的关键。本研究报道的CQ3所产的凝乳酶在pH值为4.5~7.0和温度低于50 ℃条件下能够保持较高酶活性,并且在Ca2+的存在下,对凝乳酶活力有刺激作用,这符合干酪实际加工的工作条件,表明该酶有一定的应用潜力。
4 结论
本研究从腐乳分离的霉菌菌株中筛选到一株具有良好凝乳酶活性和低蛋白酶活性的菌株CQ3,经ITS和mt SSU rDNA分子鉴定为变色栓菌(Trametes versicolor)。CQ3凝乳酶经硫酸铵分级沉淀,DEAE-Sepharose Fast Flow阴离子交换层析和Sephadex G100凝胶过滤层析纯化后,纯化倍数为18.47,回收率为7.6%。经凝乳试验表明,纯化的酶有较好的凝乳效果,凝乳酶比活力达(822.1±12.3) U/mg。经SDS-PAGE分析表明,纯化后的CQ3凝乳酶为唯一的蛋白条带,分子质量为61 kDa。该酶为一种天冬氨酸蛋白酶,其最适温度为45 ℃,最适pH为6.5,Ca2+对凝乳酶活力有显著的促进作用,该酶在pH值为4.5~7.0和温度低于50 ℃条件下有良好的稳定性。变色栓菌CQ3是一株能产凝乳酶的菌株,其凝乳酶具备高凝乳活性和低蛋白水解活性,酶的作用条件符合干酪加工的工作条件,具有进一步研究其在干酪加工中实际应用性的价值。
[1] JACOB M, JAROS D, ROHM H.Recent advances in milk clotting enzymes[J].International Journal of Dairy Technology, 2011, 64(1):14-33.
[2] BRITTEN M, GIROUX H J.Rennet coagulation of heated milk:A review[J].International Dairy Journal, 2022, 124:105179.
[3] MENG F Q, CHEN R, ZHU X Y, et al.Newly effective milk-clotting enzyme from Bacillus subtilis and its application in cheese making[J].Journal of Agricultural and Food Chemistry, 2018, 66(24):6162-6169.
[4] SHIEH C J, PHAN THI L A, SHIH I L.Milk-clotting enzymes produced by culture of Bacillus subtilis natto[J].Biochemical Engineering Journal, 2009, 43(1):85-91.
[5] ZHAO X, CAI M, YANG Z J, et al.Purification and characterization of a novel milk-clotting enzyme produced by Bacillus amyloliquefaciens GSBa-1[J].European Food Research and Technology, 2019, 245(11):2447-2457.
[6] CELEBI M, TOPUZOGULLARI M, KUZU H.Thermal destabilization of Rhizomucor miehei rennet with aldehyde dextran sulfate:Purification, bioconjugation and milk-clotting activities[J].Applied Biochemistry and Biotechnology, 2016, 180(2):261-273.
[7] NOUANI A, BELHAMICHE N, SLAMANI R, et al.Extracellular protease from Mucor pusillus:Purification and characterization[J].International Journal of Dairy Technology, 2009, 62(1):112-117.
[8] TAKYU Y, ASAMURA T, OKAMOTO A, et al.A novel milk-clotting enzyme from Aspergillus oryzae and A.luchuensis is an aspartic endopeptidase PepE presumed to be a vacuolar enzyme[J].Bioscience, Biotechnology, and Biochemistry, 2022, 86(3):413-422.
[9] AN Z G, HE X L, GAO W D, et al.Characteristics of miniature Cheddar-type cheese made by microbial rennet from Bacillus amyloliquefaciens:A comparison with commercial calf rennet[J].Journal of Food Science, 2014, 79(2):M214-M221.
[10] WEI G M, CHITRAKAR B, REGENSTEIN J M, et al.Microbiology, flavor formation, and bioactivity of fermented soybean curd (furu):A review[J].Food Research International, 2023, 163:112183.
[11] 崔岱宗. 栓菌及其近缘属的分子系统学研究[D].哈尔滨:东北林业大学, 2009.
CUI D Z.Study on molecular systematics of Trametes and relative genera[D].Harbin:Northeast Forestry University, 2009.
[12] ARIMA K, YU J, IWASAKI S.Milk-clotting enzyme from Mucor pusillus var.Lindt[J].Methods in Enzymology, 1970,19:446-459.
[13] HIBBETT D S, OHMAN A, GLOTZER D, et al.Progress in molecular and morphological taxon discovery in Fungi and options for formal classification of environmental sequences[J].Fungal Biology Reviews, 2011, 25(1):38-47.
[14] SCHOCH C L, SEIFERT K A, HUHNDORF S, et al.Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi[J].Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(16):6241-6246.
[15] KOV CS G M, JANKOVICS T, KISS L.Variation in the nrDNA ITS sequences of some powdery mildew species:Do routine molecular identification procedures hide valuable information?[J].European Journal of Plant Pathology, 2011, 131(1):135-141.
CS G M, JANKOVICS T, KISS L.Variation in the nrDNA ITS sequences of some powdery mildew species:Do routine molecular identification procedures hide valuable information?[J].European Journal of Plant Pathology, 2011, 131(1):135-141.
[16] HONG S G, JUNG H S.Phylogenetic analysis of Ganoderma based on nearly complete mitochondrial small-subunit ribosomal DNA sequences[J].Mycologia, 2004, 96(4):742-755.
[17] HE Z C, LIN J, HE Y Y, et al.Polysaccharide-peptide from Trametes versicolor:The potential medicine for colorectal cancer treatment[J].Biomedicines, 2022, 10(11):2841.
[18] HABTEMARIAM S.Trametes versicolor (Synn.Coriolus versicolor) polysaccharides in cancer therapy:Targets and efficacy[J].Biomedicines, 2020, 8(5):135.
[19] 王科峰. 变色栓菌产漆酶和多糖的发酵过程调控[D].北京:中国科学院大学(中国科学院过程工程研究所), 2017.
WANG K F.Fermentation process regulation of laccase and polysaccharides from Trametes versicolor[D].Beijing:University of Chinese Academy of Sciences (Institute of Process Engineering, Chinese Academy of Sciences), 2017.
[20] AMIRA B, BESBE S, ATTIA H, et al.Milk-clotting properties of plant rennets and their enzymatic, rheological, and sensory role in cheese making:A review[J].International Journal of Food Properties, 2017, 20(s1):S76-S93.