副溶血性弧菌(Vibrio parahaemolyticus)俗称“嗜盐菌”,是一种广泛存在于河口和海洋环境中的革兰氏阴性菌,是引起养殖虾类急性肝胰腺坏死综合症(acute hepatopancreas necrosis syndrome, AHPNS)并导致大规模死亡的重要病原菌之一,给全球的水产养殖业带来了巨大的经济损失[1]。人们摄入受副溶血性弧菌污染的海产品会导致严重的胃肠炎、败血症等疾病。副溶血性弧菌的毒力机制涉及多种因素,包括蛋白酶、溶血素(hemolysin)、铁摄取系统、外膜蛋白和黏附因子等,其中溶血素是最主要的毒力因子。
溶血素是一种能够溶解红细胞的外毒素[2]。副溶血性弧菌可分泌3种类型的溶血素:耐热直接溶血素(thermostable direct hemolysin, TDH)、耐热直接相关溶血素(TDH-related hemolysin, TRH)和不耐热溶血素(thermolabile hemolysin, TLH),一株副溶血性弧菌可表达一种或多种溶血素[3]。目前认为,TDH和TRH是导致人类疾病的主要毒力因子,分别由tdh和trh基因编码[3-4]。然而,一些缺乏这些基因的临床分离株仍表现出毒力,这表明副溶血性弧菌的致病性存在其他因素的参与[4]。TLH由tlh基因编码,已有研究证明tlh基因在模拟人体肠道环境中表达量会明显升高,但在肠道致病机制中的作用尚不明确,需进一步研究[5-6]。tlh基因在不同的弧菌中广泛存在,部分弧菌如哈维弧菌(Vibrio harveyi)和溶藻弧菌(V.alginolyticus)产生的TLH对鱼类具有高致病力[6-8]。
本研究通过分析TLH家族蛋白的氨基酸序列和多序列比对,探究了TLH在弧菌和非弧菌物种中的演化关系,进一步了解了TLH蛋白的关键生物学特征;使用同源建模预测了TLH的三维结构,并通过50 ns 的分子动力学模拟对其结构稳定性和残基波动情况进行了分析,为揭示TLH毒力的关键因素及开发治疗副溶血性弧菌病及其相关疾病的新方法提供理论参考。
1 材料与方法
1.1 材料与试剂
供试菌:副溶血性弧菌,本实验室从蛤蜊中分离、鉴定,于-80 ℃保藏。
LB营养琼脂、LB肉汤、2216E琼脂培养基、2216E液体培养基,青岛高科园海博生物技术有限公司;细菌基因组DNA快速抽提试剂盒、PCR引物、DNA分子质量标准Marker(100~5 000 bp),生工生物工程(上海)股份有限公司;StarPrep DNA纯化/胶回收试剂盒,北京康润诚业生物科技有限公司;异丙基硫代半乳糖苷(isopropyl thiogalactoside,IPTG)、X-Gal、氨苄青霉素(ampicillin,Amp),北京索莱宝科技有限公司;PrimeSTAR R Max DNA Polymerase,北京宝日医生物技术有限公司。
1.2 仪器与设备
DL-CJ-2 N超级洁净工作台,北京市东联哈尔仪器制造有限公司;凝胶电泳及配套设备、Quantity One凝胶成像系统,美国Bio-Rad公司;防蒸发梯度PCR仪,德国Eppendorf公司。
1.3 副溶血性弧菌tlh基因的克隆与测序
将本实验室-80 ℃超低温冰箱保藏的tlh阳性副溶血性弧菌接种到2216E固体培养基中复苏培养后提取总DNA。
根据NCBI数据库中副溶血性弧菌tlh基因序列(GenBank登录号:GU971655.1)设计了一对扩增引物(tlh-F:5′-ATGATGAAAAAAACAATCACACTATTAACTGC-3′;tlh-R:5′-TTAGAAACGGTACTCGGCTAAG TTG-3′),并委托生工生物工程(上海)股份有限公司合成。
制备了50 μL的PCR反应体系,包括1 μL DNA模板、25 μL PrimeSTAR® Max DNA聚合酶、每个引物(20 pmol)各1 μL、22 μL ddH2O。PCR扩增反应条件如下:94 ℃预变性2 min,随后94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸75 s,重复30次;最后72 ℃延伸10 min。扩增产物经1%琼脂糖凝胶电泳检测。
PCR扩增产物纯化回收后与pMD18-T载体连接,转化入E.coli TOP10感受态细胞中,挑取阳性克隆送至北京天一辉远生物科技有限公司测序。
1.4 基于tlh基因序列的生物信息学分析
测序的tlh序列首先使用SnapGene软件转化为氨基酸序列,然后利用多种在线工具进行分析:利用ProtParam在线工具[9](http://web.expasy.org/protparam/)预测TLH蛋白的基本理化性质,包括等电点、分子质量和脂肪指数等;使用ProtScale服务器[9](http://ca.expasy.org/tools/protscale.html)选择Hphob./Kyte &Doolittle和Hphob./Eisenberg et al.算法,对蛋白质的亲疏水性进行分析;使用DEEP TMHMM服务器[10](https://services.healthtech.dtu.dk/service.php?DeepTMHMM)对TLH蛋白的跨膜区域进行预测;通过NetNGlyc(https://services.healthtech.dtu.dk/service.php?NetNGlyc-1.0)和NetPhos服务器[11](https://services.healthtech.dtu.dk/service.php?NetPhos-3.1)检测糖基化和磷酸化位点;使用CD-Search在线工具[12](https://www.ncbi.nlm.nih.gov/cdd/)和Interpro蛋白结构域数据库[13](http://www.ebi.ac.uk/interpro/)分析TLH蛋白的结构域、基序和超家族,并使用PSIPRED在线软件[14](http://bioinf.cs.ucl.ac.uk/psipred/)预测二级结构。
1.5 多序列比对和系统发育树分析
使用BLAST工具(https://blast.ncbi.nlm.nih.gov/Blast.cgi)对TLH蛋白氨基酸序列进行同源性分析,并将其与来自不同弧菌(如副溶血性弧菌、霍乱弧菌、溶藻弧菌、拟态弧菌、河流弧菌)以及非弧菌(如帕鲁迪菌)的多个TLH蛋白质序列用ClustalX 2.1软件[15]进行多序列比对。用MEGA11软件[16]的邻接法(Neighbor-Joining, NJ)构建系统发育树。
1.6 TLH蛋白结构的同源建模和结构质量评价
使用同源建模法构建TLH蛋白的三维结构。首先,使用BLASTP搜索PDB数据库,选择与副溶血性弧菌TLH蛋白具有最高相似性的创伤弧菌(V.vulnificus)的TLH(PDB:6 JL1)[17],用Moderller 9.11软件[18]比较目标蛋白和模板蛋白的序列,确定它们具有相同的保守结构后,使用默认参数进行同源建模。从生成的10个模型中选择DOPE评分最低的模型进行目标-模板比较和结构分析。最后,使用PROCHECK[19]和ProSA[20]两种模型验证工具来评估建立的TLH蛋白模型的质量;前者用于验证模型的立体化学质量,后者则分析模型整体质量Z分数。
1.7 分子动力学模拟
使用GROMACS 2020.6软件[21]对TLH及其结合4-硝基苯基月桂酸酯(p-nitrophenylaurate, PNPL)底物的复合物进行分子动力学模拟。首先使用Autodock Vina算法将PNPL底物对接到TLH的活性位点上,形成TLH-PNPL复合物结构,对接后选择最优构象。使用PRODRG服务器生成其配体拓扑结构,CHARMM36力场用于所有仿真。每个蛋白质系统全部浸入充满显式SPC(单点电荷)水分子的菱形十二面体盒子中,蛋白质表面在各个方向上相对边界的距离为10 Å。添加Na+/Cl-离子中和系统总电荷至0。使用Parrinello-Rahman控压器保持气压在1.01×105 Pa大气压下,使用改进的Berendsen控温器将温度保持在300 K。最后,对系统进行50 ns的分子动力学模拟,记录能量和原子坐标轨迹,设置记录间隔为20 ps。利用GROMACS软件包中提供的工具和VMD软件对模拟得到的轨迹进行分析。
2 结果与分析
2.1 副溶血性弧菌tlh基因的PCR扩增、克隆和测序
使用总基因组DNA作为PCR扩增模板,扩增副溶血性弧菌的tlh基因。PCR产物经1%琼脂糖凝胶电泳(附图1-A,https://doi.org/10.13995/j.cnki.11-1802/ts.037068)。PCR扩增产物及转化后筛选的阳性菌(附图1-B)的扩增片段(附图1-C)在1 000~1 500 bp处可见清晰单一的特异性条带,与目的基因大小相符。经提取阳性质粒测序表明,获得了1 257 bp的tlh核苷酸序列。
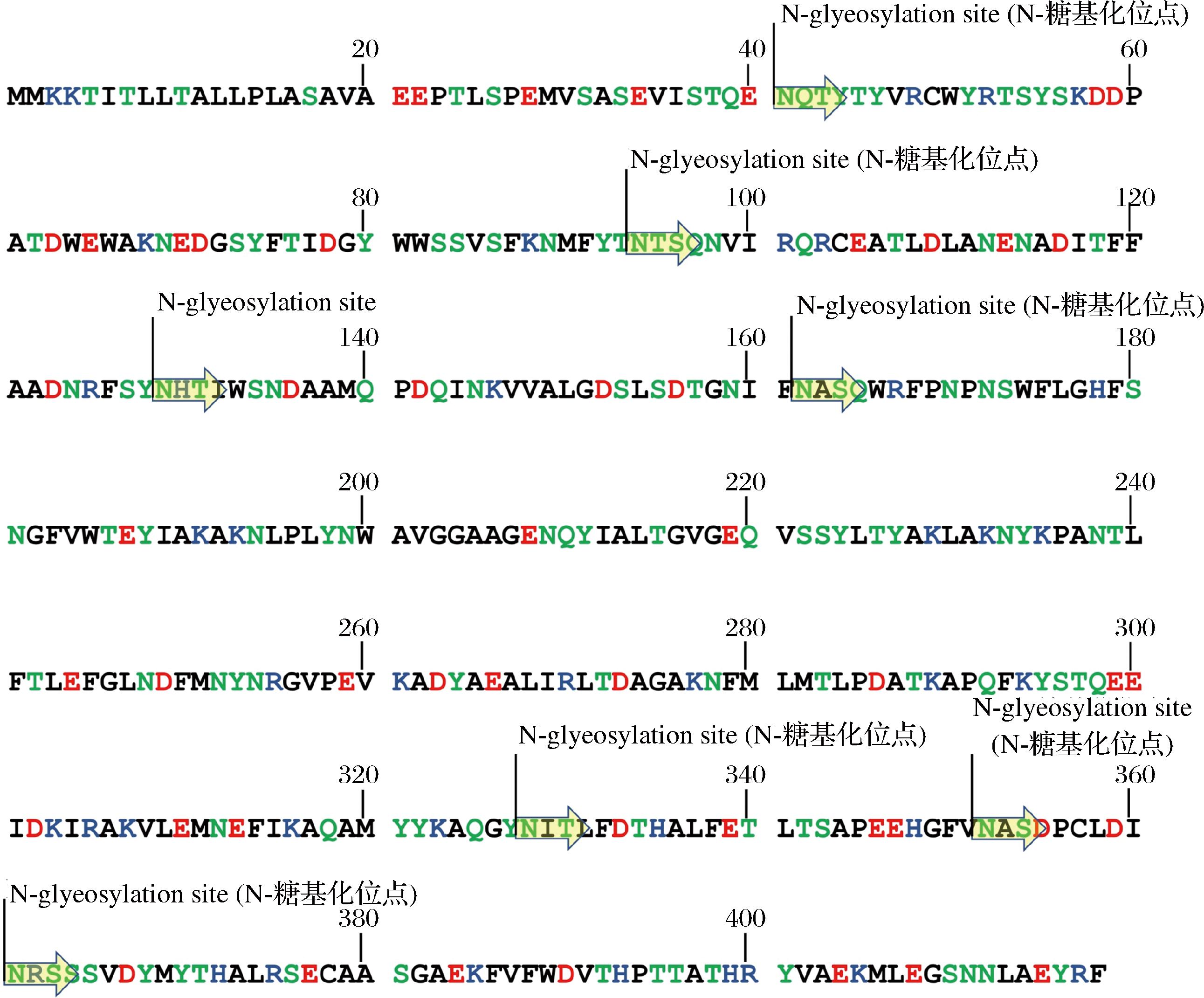
图1 TLH蛋白中预测的N-糖基化位点
Fig.1 Predicted N-glycosylation sites in TLH protein
2.2 副溶血性弧菌全长tlh基因的生物信息学分析
副溶血性弧菌tlh基因编码418个氨基酸组成的蛋白质序列。PortParam预测TLH的分子质量为47.36 kDa,理论等电点4.97,脂肪指数68.73,消光系数2.032。
预测结果(附图2-A,https://doi.org/10.13995/j.cnki.11-1802/ts.037068)表明,TLH蛋白为亲水蛋白,N末端可能含有一个跨膜区域,其疏水性得分高于1.6,Kyte-Doolittle与Eisenberg疏水性曲线得出的结果相同。Deep TMHMM对TLH蛋白跨膜结构的预测结果显示,N端的跨膜区域为20个氨基酸的信号肽,表明TLH定位为分泌蛋白(附图2-B)。
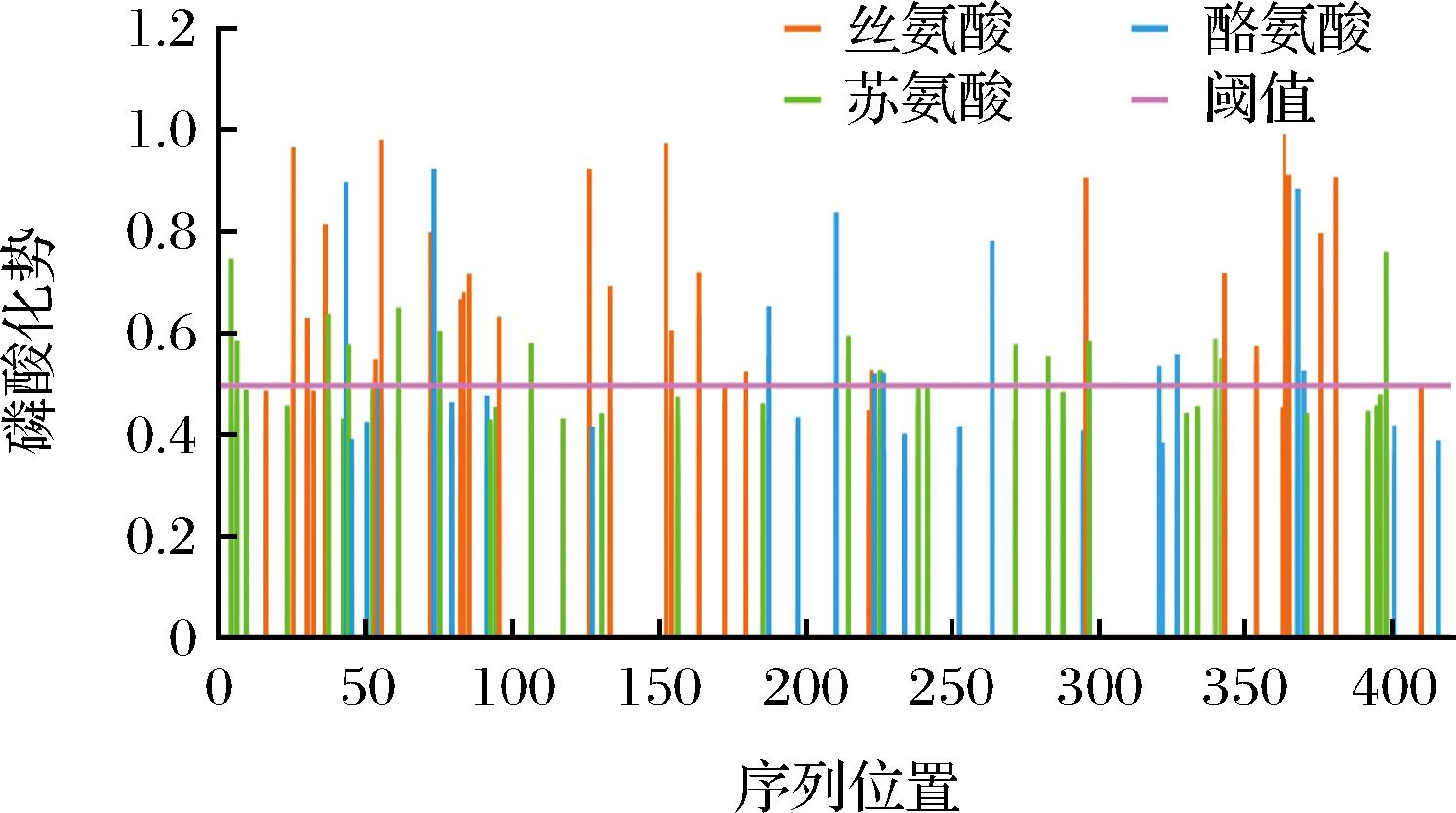
图2 TLH蛋白中预测的磷酸化位点
Fig.2 Predicted phosphorylation sites in TLH protein
翻译后修饰(post-translational modification,PTM)是指对已完成翻译的蛋白质进行共价加工的过程,通过在一个或多个氨基酸残基上添加修饰基团,可改变蛋白质的物理化学性质,影响其空间结构、活性状态、亚细胞定位、折叠稳定性以及蛋白质间相互作用等[22]。常见的PTM类型包括泛素化、磷酸化、糖基化、甲基化、乙酰化和酯基化[23]。NetNGlyc检测发现,在TLH蛋白中有7个N-糖基化位点,分别位于第41、94、129、162、328、352、361个氨基酸位置上(图1中的黄色箭头所示)。糖基化是通过酶的作用将糖链添加到蛋白质中,与许多细菌的致病性有关。在流感嗜血杆菌中,HMW1的糖基化对其粘附于宿主细胞至关重要[24];而在空肠弯曲杆菌中,N-连接蛋白的糖基化与其在鸡和小鼠的胃肠道中的定殖和在人类上皮细胞上的黏附有关,表明其在病原体中的潜在作用[25]。TLH中糖基化位点的作用机制仍待深入研究。
磷酸化也是细菌的一种重要的调节修饰方式。例如,结核分枝杆菌(Mycobacterium tuberculosis)可以将丝氨酸/苏氨酸蛋白激酶PknG分泌到宿主巨噬细胞中,从而避免宿主的免疫反应[26]。此外,分枝杆菌分泌的酪氨酸磷酸酶PtpA也有助于病原体避免被宿主的免疫系统攻击[27]。NetPhos对TLH蛋白可能存在的磷酸化位点的预测表明共有53个潜在的磷酸化位点(图2),其中包括26个丝氨酸、16个苏氨酸和11个酪氨酸磷酸化位点,这些位点可能在TLH的溶血和细胞毒性中发挥作用。
CD-search结构域预测表明,TLH蛋白的146~408氨基酸与属于SGNH水解酶超家族的cd01846 SGNH水解酶的脂肪酰基转移酶样亚家族结构域高度保守(图3-A)。SGNH水解酶超家族的三级结构与α/β水解酶家族的三级结构差异较大,并且在所有已知的水解酶中比较独特,其活性位点与其他丝氨酸水解酶中发现的Ser-His-Asp(Glu)三联体类似。通过InterProScan在线程序,使用多个数据库分析了TLH蛋白的保守域,并成功匹配了6个蛋白质(图3-B)。其中,最成功的匹配是PF00657:GDSL脂肪酶/酯酶,该亚类脂肪酶具有独特的GDSL序列模式。
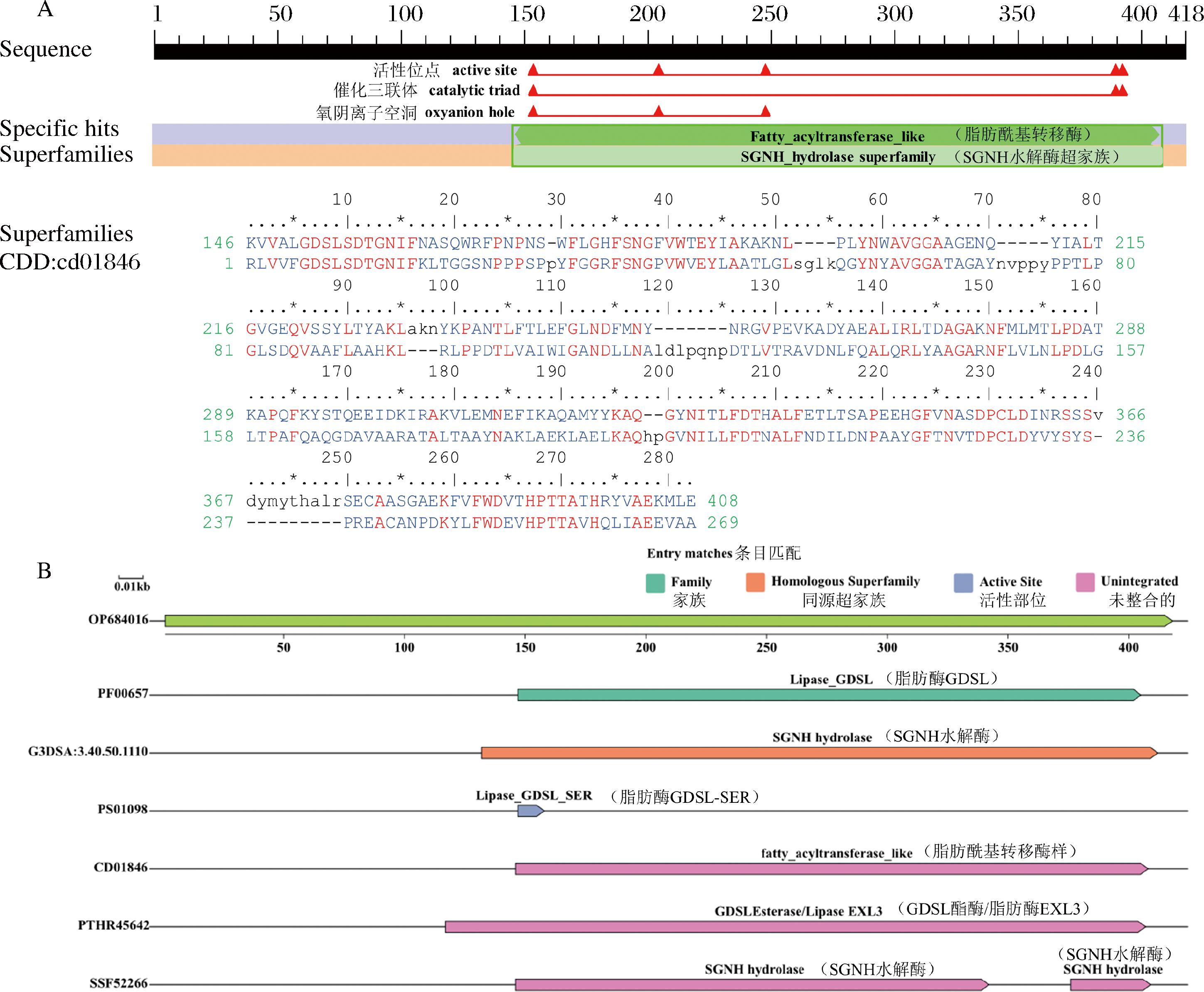
A-CD-search;B-InterProScan
图3 CD-search和InterProScan对TLH蛋白的保守结构域分析鉴定
Fig.3 Conserved domain analysis of TLH Protein by CD-search and InterProScan
PSIPRED预测显示,副溶血性弧菌TLH蛋白的二级结构由α-螺旋、β-折叠、β-转角、γ-转角和二硫键组成(图4),分别为20个α-螺旋(30.30%)、13个β-折叠(19.70%)、26个β-转角(39.39%)、5个γ-转角(7.58%)和2个二硫键(3.03%)。

图4 TLH蛋白二级结构组成
Fig.4 Topology of the secondary structure composition and secondary structure of TLH protein
注:α-螺旋由紫色螺旋表示,β-折叠由粉色箭头表示。β-转角、γ-转角和β-发夹标记为β、γ和⊃,二硫键由黄色连接线表示。
2.3 多序列比对和系统进化树分析
为了研究TLH蛋白在不同弧菌和非弧菌中的进化保守性,选择了多种细菌的21个TLH蛋白序列进行比较(附图3,https://doi.org/10.13995/j.cnki.11-1802/ts.037068),结果表明,大多数氨基酸残基在进化过程中高度保守。在与SGNH水解酶家族对应的5个残基模块中,TLH类蛋白表现出很高的保守性,这些残基包括I模块的Ser、II模块的Gly、III模块的Asn、V模块的His,以及在GDSL脂肪酶中保守的GDSL基序。催化氨基酸残基的高度保守表明这些区域可能对维持TLH蛋白的正常结构和功能至关重要。副溶血性弧菌TLH的SGNH水解酶结构域包含经典的催化三联体Ser153-His390-Asp393,这对于丝氨酸蛋白酶、金属蛋白酶和其他蛋白质的活性起着关键作用[28]。此外,其N端结构域包含一个由保守残基组成的β-折叠结构,可能在TLH蛋白的功能中发挥未知但重要的作用。
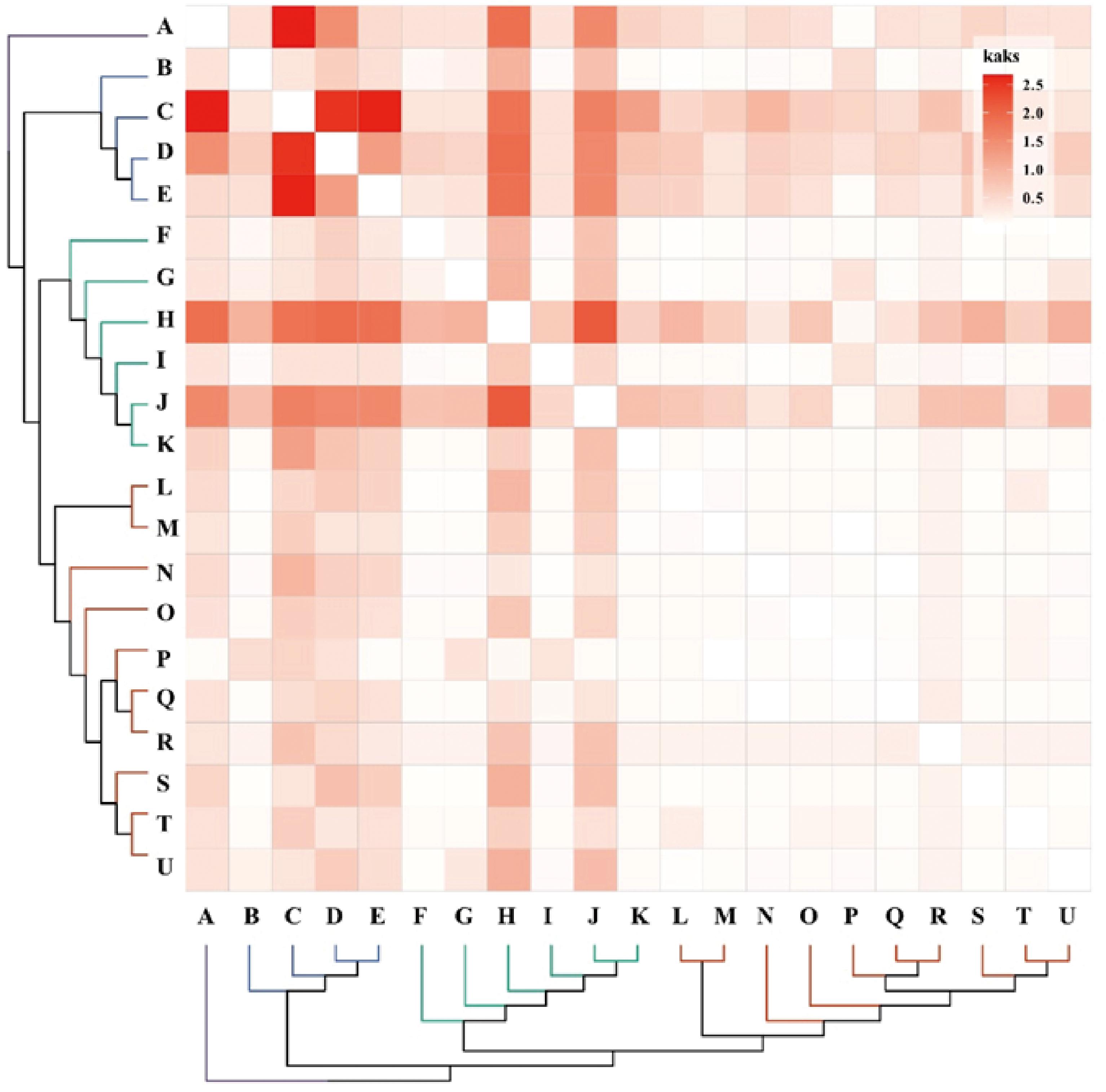
由上述21个TLH蛋白序列系统进化树和Ka/Ks热图(附图4,https://doi.org/10.13995/j.cnki.11-1802/ts.037068)可知,这些序列被分为3个不同的簇。最大的簇是第三簇(红色),由10个序列组成,其中包括7个不同的副溶血性弧菌序列(包括本研究的序列)以及来自溶藻弧菌、坎氏弧菌(V.campbellii)、哈维弧菌等弧菌的序列。在第二簇(绿色)中,几个致病性弧菌的TLH蛋白序列表现出高度的相似性,而第一簇(蓝色)由4个非致病性弧菌的TLH蛋白组成。因此,TLH类蛋白在弧菌物种中表现出高度保守性,而非弧菌的TLH蛋白则表现出一定程度的进化分歧。此外,tlh通常被认为是副溶血性弧菌的独特分子标记[29]。然而,系统发育分析表明,副溶血性弧菌、溶藻弧菌、坎氏弧菌和哈维弧菌之间存在密切相关性。因此,tlh基因缺乏特异性,将其作为单一分子标记使用可能会在其他弧菌物种中产生假阳性结果,这与MOHAMAD等[30]的研究结论一致。
2.4 同源建模和结构质量评估
使用PDB数据库中6 JL1模板进行TLH蛋白的三维结构预测,该模板是创伤弧菌的TLH晶体结构,分辨率为2.29 Å,与目标蛋白的序列相似性超过了73%。利用6 JL1模板构建了10个TLH蛋白模型,并从中选择DOPE评分最低的模型(-48 588.781 25)作为最终模型。图5展示了构建的TLH蛋白模型与6 JL1模板的结构比较,二者的RMSD为0.218,TM-score为0.744。
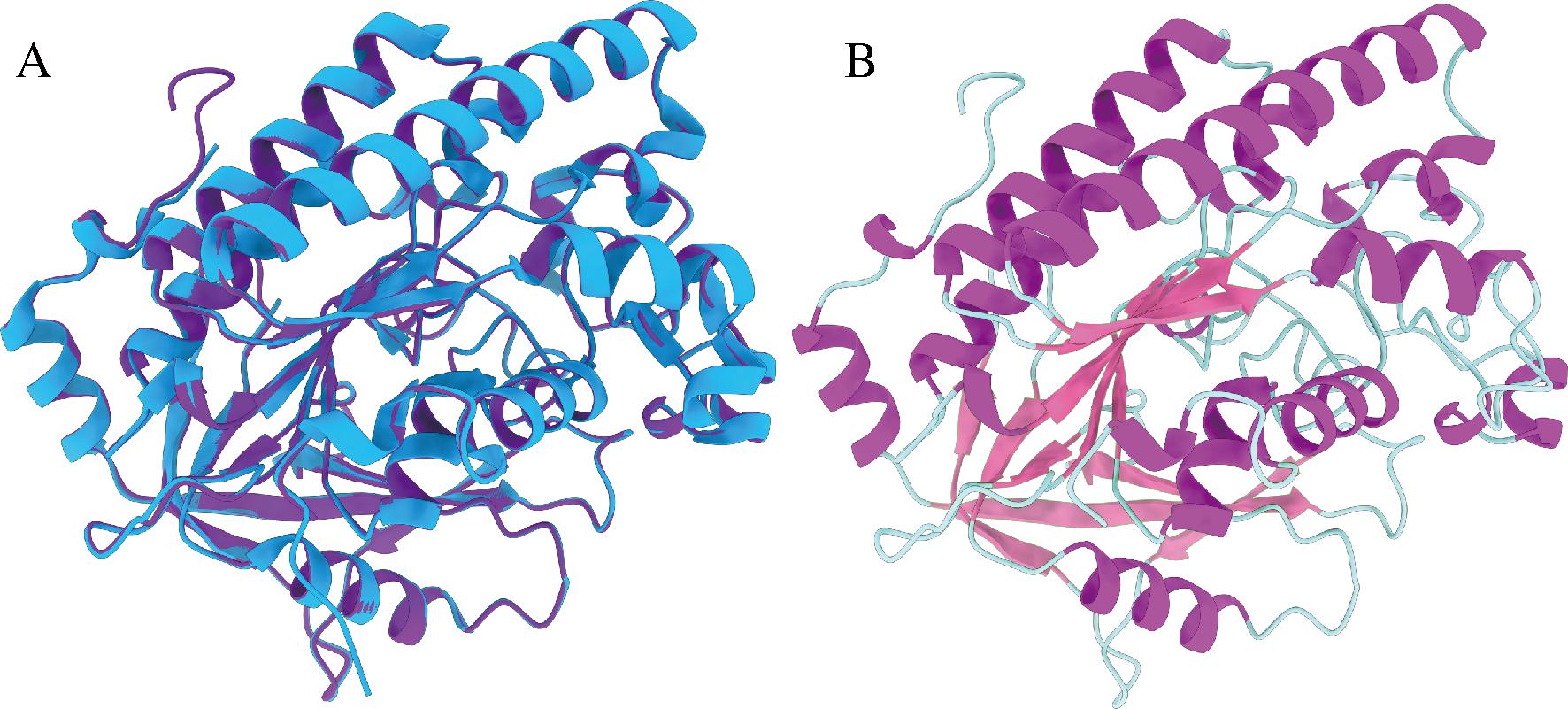
A-TLH同源模型和模板蛋白6 JL1的结构叠加(紫色为6 JL1, 蓝色为TLH);B-TLH同源模型的整体结构(紫色为α-螺旋,粉色为β-折叠)
图5 TLH的同源建模
Fig.5 Homology modeling of TLH
为了验证构建的TLH蛋白质量,使用了多种蛋白质结构验证工具,以确保建模结构的可靠性。通过SAVES 6.0服务器生成的Ramachandran图(附图5-A,https://doi.org/10.13995/j.cnki.11-1802/ts.037068),显示91.4%的残基位于允许区域内,7.6%位于最合理的允许区域内,只有0.3%位于不合理的区域内,且不允许区域内的残基均不是关键残基。使用ProSA程序估计了模型的总体质量,结果用Z-score表示。模型的Z-score为-6.32,表明与类似大小的天然蛋白结构相比,TLH蛋白的同源模型具有相似的质量(附图5-B)。
2.5 分子动力学模拟
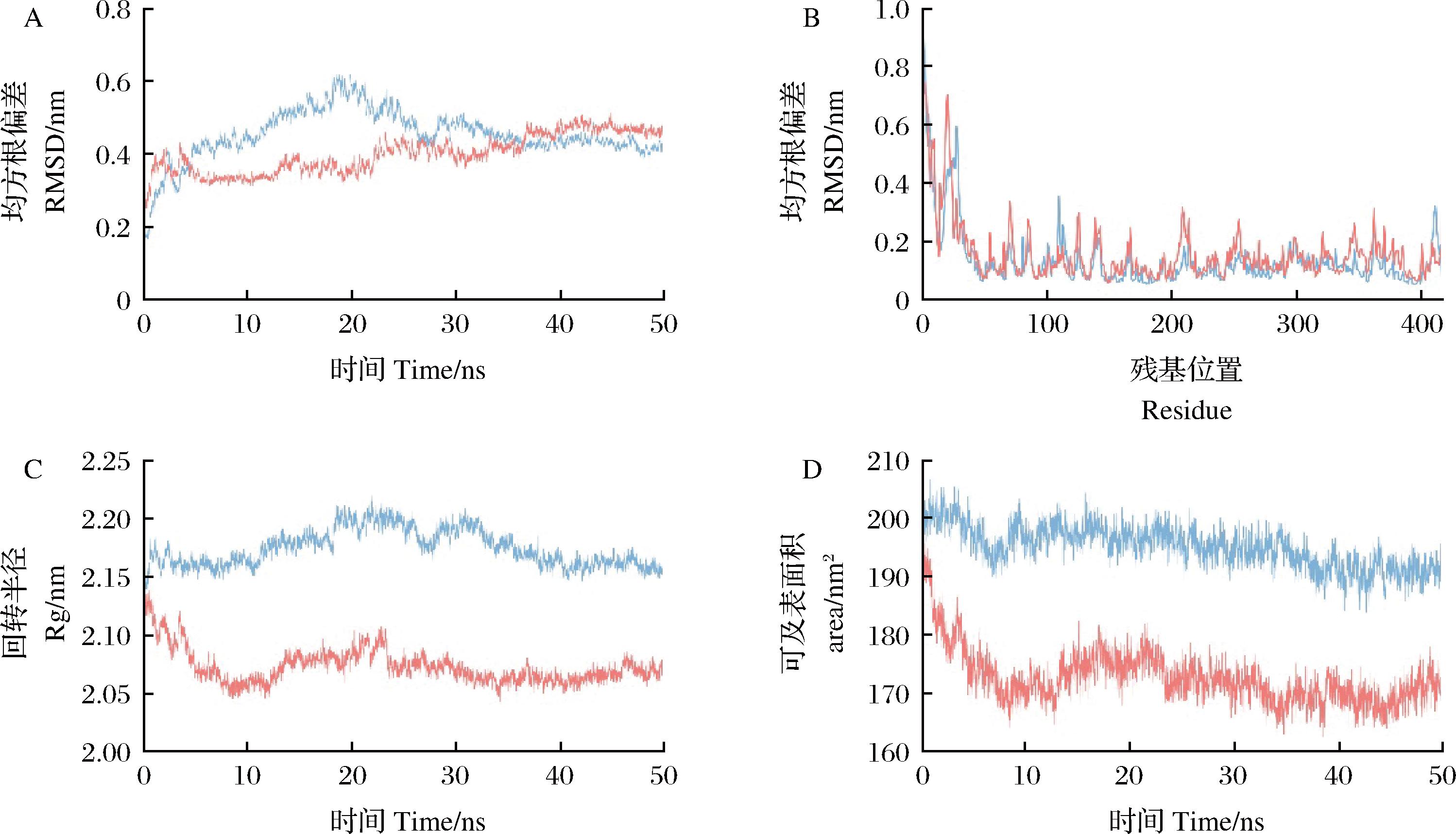
本研究使用了50 ns的分子动力学模拟,评估了TLH蛋白在结合底物时的整体稳定性和构象变化。计算了TLH蛋白和TLH-PNPL复合物系统的均方根偏差(root-mean-square deviation,RMSD)、均方根波动(root mean square fluctuation,RMSF)、回转半径(radius of gyration,Rg)和溶剂可及表面积(solvent-accessibility surface area,SASA)。如附图6-A(https://doi.org/10.13995/j.cnki.11-1802/ts.037068)所示,TLH蛋白和TLH-PNPL复合物的平均RMSD值分别为0.4、0.45 nm,且TLH-PNPL复合物比TLH蛋白更快地达到平衡状态。此外,TLH和TLH-PNPL复合物的RMSF曲线相似,但在15~20、25~30、110~115、200~215、250~260、340~360和405~415这些残基之间存在差异,这些高度波动的残基可能参与了构象变化,以确保TLH和底物之间的稳定结合(附图6-B)。Rg与模拟时间的关系能够评估系统的紧密程度或扩张性。与单独TLH的Rg值相比,复合物的较低Rg值表示了更紧密的分子排列(附图6-C),这反映出TLH和配体之间的相互作用导致了更紧密的结构。此外,SASA(附图6-D)的结果也表明相同结果。TLH的SASA值高于TLH-PNPL复合物,说明TLH与底物结合后增加了蛋白表面的疏水性,具有更紧密的构象。TLH-PNPL复合物的SASA相对较大的波动表明蛋白固有的灵活性。

图6 TLH与PNPL复合后的波动残基
Fig.6 Fluctuating residues of TLH after binding with PNPL
注:催化三联体结构以紫色标记。
对分子模拟过程中波动较大的残基(图6)进行分析发现,主要的波动残基均位于活性中心,即催化三联体的周边。分子动力学分析显示该区域的β-折叠和长α-螺旋是刚性的,而催化三联体周边的残基表现出较高的结构可变性,从而使底物结合口袋可以通过构象变化结合多种底物并与之产生相互作用。VAZQUEZ-ARMENTA等[31]的研究表明,TLH的酶活力受金属离子和化学修饰剂的调节,可能是由于与催化氨基酸残基(如Ser153和/或His393)的相互作用。VAZQUEZ-MORADO等[32]发现,类黄酮如槲皮素和表儿茶素能够抑制TLH的活性,分子对接结果显示它们可能与TLH的活性位点氨基酸相互作用,从而抑制了其酶活力和溶血活性。ZHAO等[33-34]发现,白藜芦醇和天麻素能够有效地对抗由副溶血性弧菌溶血素介导的溶血和细胞毒性效应,分子对接和突变残基L247A和G246A的实验结果表明,其作用机制涉及阻断蛋白质活性中心的氨基酸残基。因此,可以将催化三联体周边残基的活性中心作为抑制剂结合的潜在靶点,有助于顺利且有效地与该口袋的化合物或肽结合,抑制TLH蛋白的酶活力,并进一步减弱溶血活性。
3 结论
本研究从实验室保存的一株蛤蜊源副溶血性弧菌中成功克隆得到全长为1 257 bp的tlh基因,对其进行了生物信息学分析及分子动力学模拟。结果表明,TLH蛋白的SGNH水解酶结构域包含的经典的Ser153-His390-Asp393催化三联体是一个有潜力的药物结合活性口袋,可以作为潜在的药物作用靶点。本研究通过对TLH蛋白与其底物结合模式的分析,更全面地了解了TLH在副溶血性弧菌中的功能和影响,进一步评估了其对水产品的安全性的影响,为开发控制水产品中副溶血性弧菌感染的抑菌物质提供参考。
[1] PRITHVISAGAR K S, KUMAR B K, KODAMA T, et al.Whole genome analysis unveils genetic diversity and potential virulence determinants in Vibrio parahaemolyticus associated with disease outbreak among cultured Litopenaeus vannamei (Pacific white shrimp) in India[J].Virulence, 2021, 12(1):1936-1949.
[2] ZHANG L L, ORTH K.Virulence determinants for Vibrio parahaemolyticus infection[J].Current Opinion in Microbiology, 2013, 16(1):70-77.
[3] JONES J L, LÜDEKE C H M, BOWERS J C, et al.Biochemical, serological, and virulence characterization of clinical and oyster Vibrio parahaemolyticus isolates[J].Journal of Clinical Microbiology, 2012, 50(7):2343-2352.
[4] BROBERG C A, CALDER T J, ORTH K.Vibrio parahaemolyticus cell biology and pathogenicity determinants[J].Microbes and Infection, 2011, 13(12-13):992-1001.
[5] GOTOH K, KODAMA T, HIYOSHI H, et al.Bile acid-induced virulence gene expression of Vibrio parahaemolyticus reveals a novel therapeutic potential for bile acid sequestrants[J].PLoS One, 2010, 5(10):e13365.
[6] LI L Z, MENG H M, GU D, et al.Molecular mechanisms of Vibrio parahaemolyticus pathogenesis[J].Microbiological Research, 2019, 222:43-51.
[7] JIA A R, WOO N Y S, ZHANG X H.Expression, purification, and characterization of thermolabile hemolysin (TLH) from Vibrio alginolyticus[J].Diseases of Aquatic Organisms, 2010, 90(2):121-127.
[8] BAI F F, SUN B G, WOO N Y S, et al.Vibrio harveyi hemolysin induces ultrastructural changes and apoptosis in flounder (Paralichthys olivaceus) cells[J].Biochemical and Biophysical Research Communications, 2010, 395(1):70-75.
[9] DUVAUD S, GABELLA C, LISACEK F, et al.Expasy, the Swiss Bioinformatics Resource Portal, as designed by its users[J].Nucleic Acids Research, 2021, 49(W1):W216-W227.
[10] HALLGREN J, TSIRIGOS K D, PEDERSEN M D, et al.DeepTMHMM predicts alpha and beta transmembrane proteins using deep neural networks[J/OL].bioRxiv, 2022 (2022-04-10).https://www.biorxiv.org/content/10.1101/2022.04.08.487609v1.
[11] BLOM N, SICHERITZ-PONTÉN T, GUPTA R, et al.Prediction of post-translational glycosylation and phosphorylation of proteins from the amino acid sequence[J].Proteomics, 2004, 4(6):1633-1649.
[12] MARCHLER-BAUER A, DERBYSHIRE M K, GONZALES N R, et al.CDD:NCBI′s conserved domain database[J].Nucleic Acids Research, 2015, 43(D1):D222-D226.
[13] JONES P, BINNS D, CHANG H Y, et al.InterProScan 5:Genome-scale protein function classification[J].Bioinformatics, 2014, 30(9):1236-1240.
[14] BUCHAN D W A, JONES D T.The PSIPRED protein analysis workbench:20 years on[J].Nucleic Acids Research, 2019, 47(W1):W402-W407.
[15] LARKIN M A, BLACKSHIELDS G, BROWN N P, et al.Clustal W and Clustal X version 2.0[J].Bioinformatics, 2007, 23(21):2947-2948.
[16] TAMURA K, STECHER G, KUMAR S.MEGA11:Molecular evolutionary genetics analysis version 11[J].Molecular Biology and Evolution, 2021, 38(7):3022-3027.
[17] WAN Y, LIU C S, MA Q J.Structural analysis of a Vibrio phospholipase reveals an unusual Ser-His-chloride catalytic triad[J].Journal of Biological Chemistry, 2019, 294(30):11391-11401.
[18]  ALI A, POTTERTON L, YUAN F, et al.Evaluation of comparative protein modeling by MODELLER[J].Proteins:Structure, Function, and Bioinformatics, 1995, 23(3):318-326.
ALI A, POTTERTON L, YUAN F, et al.Evaluation of comparative protein modeling by MODELLER[J].Proteins:Structure, Function, and Bioinformatics, 1995, 23(3):318-326.
[19] LASKOWSKI R A, MACARTHUR M W, THORNTON J M.PROCHECK:Validation of Protein-Structure Coordinates[M].Hoboken:John Wiley &Sons Inc., International Tables for Crystallography, 2006(F), 25.2:722-725.
[20] WIEDERSTEIN M, SIPPL M J.ProSA-web:Interactive web service for the recognition of errors in three-dimensional structures of proteins[J].Nucleic Acids Research, 2007, 35:W407-W410.
[21] ABRAHAM M J, MURTOLA T, SCHULZ R, et al.GROMACS:High performance molecular simulations through multi-level parallelism from laptops to supercomputers[J].SoftwareX, 2015, 1-2:19-25.
[22] HASAN M M, KHATUN M S.Prediction of protein post-translational modification sites:An overview[J].Annals of Proteomics and Bioinformatics, 2017, 2(1):49-57.
[23] MACEK B, FORCHHAMMER K, HARDOUIN J, et al.Protein post-translational modifications in bacteria[J].Nature Reviews Microbiology, 2019, 17(11):651-664.
[24] GROSS J, GRASS S, DAVIS A E, et al.The Haemophilus influenzae HMW1 adhesin is a glycoprotein with an unusual N-linked carbohydrate modification[J].Journal of Biological Chemistry, 2008, 283(38):26010-26015.
[25] CAIN J A, L.DALE A L, SUMER-BAYRAKTAR Z, et al.Identifying the targets and functions of N-linked protein glycosylation in Campylobacter jejuni[J].Molecular Omics, 2020, 16(4):287-304.
[26] RIECK B, DEGIACOMI G, ZIMMERMANN M, et al.PknG senses amino acid availability to control metabolism and virulence of Mycobacterium tuberculosis[J].PLoS Pathogens, 2017, 13(5):e1006399.
[27] WANG J, GE P P, QIANG L H, et al.The mycobacterial phosphatase PtpA regulates the expression of host genes and promotes cell proliferation[J].Nature Communications, 2017, 8(1):244.
[28] AKOH C C, LEE G C, LIAW Y C, et al.GDSL family of serine esterases/lipases[J].Progress in Lipid Research, 2004, 43(6):534-552.
[29] WANG R Z, HUANG J D, ZHANG W, et al.Detection and identification of Vibrio parahaemolyticus by multiplex PCR and DNA-DNA hybridization on a microarray[J].Journal of Genetics and Genomics, 2011, 38(3):129-135.
[30] MOHAMAD N, AMAL M N A, SAAD M Z, et al.Virulence-associated genes and antibiotic resistance patterns of Vibrio spp.isolated from cultured marine fishes in Malaysia[J].BMC Veterinary Research, 2019, 15(1):176.
[31] VAZQUEZ-ARMENTA F J, VALDEZ-OLMOS U F, ARVIZU-FLORES A A, et al.Metal ions and chemical modification reagents inhibit the enzymatic activity of lecithin-dependent hemolysin from Vibrio parahaemolyticus[J].Toxins, 2022, 14(9):609.
[32] VAZQUEZ-MORADO L E, ROBLES-ZEPEDA R E, OCHOA-LEYVA A, et al.Biochemical characterization and inhibition of thermolabile hemolysin from Vibrio parahaemolyticus by phenolic compounds[J].PeerJ, 2021, 9:e10506.
[33] ZHAO X R, GUO Y, HE J L, et al.Gastrodin relieves Vibrio harveyi infection by blocking hemolysin active centers[J].Aquaculture, 2021, 544:737056.
[34] ZHAO X R, GUO Y, NI P, et al.Resveratrol inhibits the virulence of Vibrio harveyi by reducing the activity of Vibrio harveyi hemolysin[J].Aquaculture, 2020, 522:735086.