贻贝作为海产品的主要品种,营养价值丰富。由于贻贝以藻类为食,有害的藻类会导致贻贝体内的毒素积累,这些毒素主要包括腹泻贝类毒素(diarrheal shellfish toxins, DST)和麻痹性贝类毒素[1]。DST由冈田酸等毒素组成,是一些常见食物中毒事件发生的主要原因[2]。食用被DST污染的贻贝会导致明显的胃肠功能障碍,如腹泻、腹部绞痛、恶心、呕吐等,严重者可危及生命。食用被毒素污染的贻贝导致的食物中毒会对休闲、旅游和贝类产业产生负面的经济影响[3]。
许多贝类养殖国家已经采取了一些检测方法减少中毒的风险,确保海鲜的质量,如液相色谱法[4-5]、气相色谱法[6]、等离子谱法[7]、电泳法[8]等。这些方法所需耗材昂贵、耗时,并且需要训练有素的技术人员才可以完成检测。此外,生物测定法检测毒素也是一种广泛使用的检测方法,但该方法只能检测样品中的总毒性,而不能检测样品中的单一毒素,其缺点是不可重复、耗材昂贵,并需要较长的检测周期[9]。
近红外光谱具有快速、低成本可定性[10]或定量检测[11]的优点,已广泛应用于食品质量安全检测。一些研究者利用近红外光谱鉴别掺假的羊肉、牛肉[12]、牛奶[13]和鸡蛋[14]等。近红外光谱在水产品检测中也有应用,GHIDINI等[15]将近红外光谱作为测定鱼类中组胺含量的分析手段,CHENG等[16]研究了在400~1 000 nm进行可见光和近红外光谱测定活菌总数和评价鱼片微生物腐败的可行性,TITO等[17]研究了近红外光谱方法检测和预测大西洋鲑鱼微生物腐败的潜力。由于仪器灵敏度低以及食品中水分的干扰,使用近红外光谱进行痕量成分的定量分析往往变得复杂,然而,适当的实验设计和光谱处理技术可以获得基于痕量量化校准的最佳近红外光谱。
DST的积累会导致贻贝组织的物理和化学变化,它能促进贝类解毒阳离子蛋白的合成,引起分子化学键的变化[3],当用光谱检测系统光源照射被DST污染的贻贝时,贻贝的组织会吸收或反射不同波长的光,近红外光谱中的分子振动信息可以反映出正常贻贝和受DST污染贻贝的差异,因此,应用近红外光谱信息可间接检测贻贝体内的DST。但DST污染贻贝和健康贻贝的光谱曲线的差异无法用肉眼区分,需通过分类器识别。在机器学习领域许多分类器,如支持向量机、决策树分类器、极限学习机等[18]在区分两类或多类数据都表现出良好的分类性能。然而,这些分类器在区分高维数据的光谱数据时表现一般。本团队对基于支持向量机的贻贝DST污染检测方法[19]进行了评价。
近年来,稀疏表示分类(sparse representation classification, SRC)在人脸识别中得到了相当大的关注[20],但SRC在光谱数据分类中还比较少见。ZHANG等[21]认为类别相关表示分类(class-specific representation classification, CRC)采用范数最小化来获得系数比SRC性能更好,但是SRC和CRC包含负的系数可能导致误分类。如果每个样本的系数都是非负的,则CRC将演变为非负表示分类(non-negative representation classification, NRC)[22]。NRC意味着来自正确类别的样本具有正系数,而其他类别样本的系数为0。NRC在SRC和CRC之间取得了平衡,但由于缺乏正则化项,NRC在对高维和非线性数据进行分类时,其分类精度表现不佳。
本研究将近红外光谱与类别相关残差约束的非负表示分类(class-specific residual constraint nonnegative representation classification, CRNRC)模型相结合,用于检测受DST污染贻贝。首先分析DST污染与健康贻贝近红外光谱特征的差异,阐述利用光谱曲线间接鉴定DST污染贻贝的可行性。通过实验比较CRNRC、CRC和NRC的编码向量,获得CRNRC模型最优参数。实验结果表明:对受DST污染贻贝和健康贻贝分类时,在平均准确度、F-measure和1-specificity指标上CRNRC性能优于CRC和NRC。
1 材料与方法
1.1 样本的培养和制备
贻贝来自广东省湛江市的东风海鲜市场,用干净的海水冲洗贻贝表面的沉积物和杂质,选择生命力强的贻贝进行培养实验,这些贻贝具有膜瓣运动,并能分泌足丝。将贻贝随机分成2组,并放在2个塑料箱中,大小为119 cm×108 cm×32 cm。用于DST富集的贻贝每天喂食浓度为7.3×109 个细胞/L的利玛原甲藻1 L。利玛原甲藻是我国近海四大海域广泛分布的底栖有毒赤潮藻,是产生DST毒素的主要藻类之一,因其易于培育且分布广,常作为研究DST毒素的重要材料。在摄食过程中,贻贝积累了冈田酸和甲藻毒素,并与脂肪酸酯化,大量的冈田酸转化为酯,在贻贝的消化腺中积累。对照组贻贝每天饲喂浓度为109个细胞/L的光合细菌0.5 L。实验过程中,水温保持在26 ℃,并为贻贝提供足够的氧气,每天更换海水,清洗容器内表面。培养6 d后,采集所有贻贝样品的近红外光谱数据。
1.2 光谱收集和预处理
采用SW2520-050-NIRA,OtO Photonics公司生产这款近红外光谱仪采集贻贝样品光谱。该光谱系统主要由近红外光谱仪、光纤、卤素光源和可调位移平台组成[18],如图1所示。使用近红外光谱仪采集了950~1 700 nm的光谱,光学分辨率为6.5 nm。在采集贻贝样品的光谱前,对近红外光谱仪进行黑白校正以降低噪声,白色聚四氟乙烯板的反射率约为100%,以获得白色参考强度,通过关闭光源获得暗参考强度。

图1 近红外光谱采集系统
Fig.1 NIRS measurement system
为使研究更接近现场检测要求,贻贝样品未被干燥研磨。而是打开壳后,将贝肉放在一半贝壳上后直接采集光谱。贻贝光谱采集的条件与黑白校正的条件相同。所有样品的光谱数据集使用Spectra Smart软件处理。贻贝受到DST污染,贻贝体内的一些酶活性会发生变化,这种变化可被近红外光谱仪捕捉到。这种光谱上的差异可用于区分受DST污染贻贝和健康贻贝。
在获取贻贝光谱时,不可避免地会受到一些环境影响,如光散射、光强的变化和随机噪声等。本研究采用多元散射校正[23]对光谱进行预处理,消除散射效应提高光谱质量。贻贝样品的光谱图2所示,在950~1 700 nm的波长范围内有114 个波长。在这些光谱曲线中,肉眼无法区分某条曲线是DST污染贻贝光谱曲线还是健康贻贝光谱曲线,因此,需要一种能够区分DST污染贻贝和健康贻贝光谱曲线的分类方法。
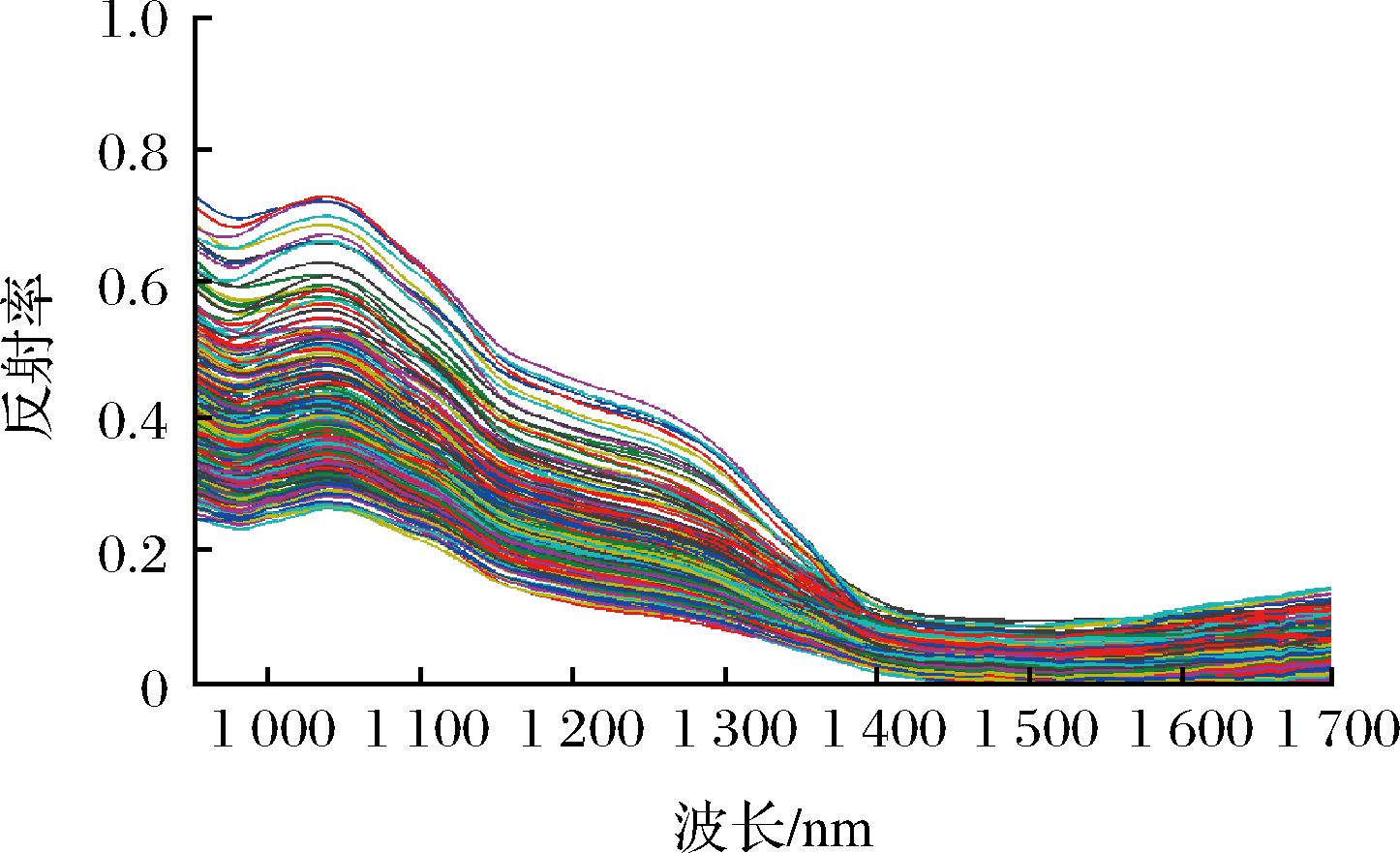
图2 所有贻贝样品的光谱
Fig.2 The spectra of all mussel samples
1.3 分类模型
1.3.1 CRC 模型
CRC的思想是一个类的测试样本可以用所有类的训练样本来表示,这种表示的前提是两个类的样本必须具有相似性。由图2可以看出健康贻贝样品和受DST污染贻贝样品的光谱具有相似性。
假设K个类中有n个训练样本,训练样本矩阵如公式(1)所示:
A=[A1,A2,…,AK]=[a1,a2,…,an]∈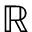 d×n
d×n
(1)
式中:Ai是第i个类的训练样本,d是样本的维数,![]() 是第i类训练样本的编号。
是第i类训练样本的编号。
CRC的目标函数如公式(2)所示:
(2)
式中:第1项是协同表示。测试样本q∈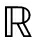 d表示为所有训练样本的稀疏线性叠加,平衡参数λ>0,c是系数向量,如公式(3)所示:
d表示为所有训练样本的稀疏线性叠加,平衡参数λ>0,c是系数向量,如公式(3)所示:
c=(ATA+λI)-1ATq
(3)
式中:I是单位阵,(ATA+λI)-1AT只与训练矩阵A相关,因此,在得到所有的训练样本后,可以提前计算(ATA+λI)-1AT,这使得CRC模型高效,CRC通过使用公式(4)正则化残差对样本进行分类:
(4)
式中:系数向量ci属于第i类。
1.3.2 NRC模型
在CRC模型中的编码向量包含负项,NRC模型采用的样本表示具有非负代表系数的测试样本,NRC模型如公式(5)所示:
(5)
NRC对测试样本的分类,类似于公式(3),但NRC模型没有其他正则化项。由于缺乏正则化项,NRC模型可能导致误分类。此外,NRC忽略了编码与分类阶段之间的关系,在NRC中引入类别相关残差约束来缓解这些问题,就得到CRNRC模型。
1.3.3 CRNRC模型
CRNRC模型是NRC和CRC的综合模型,CRNRC模型目标函数表示公式(6):
(6)
式中:平衡参数λ≥0,第1项是协同表示,第2项是类别相关残差约束。如果λ=0,CRNRC就退化为NRC。
为求解CRNRC模型,在公式(6)中引入一个辅助变量z,可有如公式(7)的等价形式:
(7)
使用交替方向乘子法[24]求解该模型,公式(7)对应的增广Lagrangian函数为公式(8):
(8)
式中:δ和μ分别为Lagrangian乘子和惩罚系数。
求解公式(8)中z和c的方法参见文献[25],CRNRC算法步骤如下:
第1步:初始化参数,包括训练样本矩阵A、测试样本q和平衡参数λ;
第2步:归一化矩阵A和测试样本q;
第3步:通过求解式(6),得到测试样本q在矩阵A上的表示系数c;
第4步:计算每类样本的残差ri=‖q-Aici‖2;
第5步:输出![]()
2 结果与分析
将贻贝样品的近红外光谱数据按7∶3的比例分为训练数据集和测试数据集。训练样本数量为168个,测试样本数量为72个。准确性、F-measure和1-specificity作为评价指标,定义如公式(9)~公式(11):
(9)
(10)
(11)
式中:TP是预测为健康样本的健康样本数,FN是预测为DST污染样本的健康样本数量,FP是预测为健康样本的受DST污染样本的数量,TN为预测为DST污染样本的DST污染样本数量。
2.1 协同表示编码向量
为直观地显示协同表示的含义,本研究做如下实验。测试集中将受DST污染的训练样本编号为1~84,健康样本编号为85~168。同样,测试集中将受DST污染的测试样本编号为1~36,健康样本编号为37~72。在CRNRC、NRC和CRC中,分别协同表示第40个测试样本,其编码向量如图3所示,横坐标为样本序列号,纵坐标为编码向量的系数值。
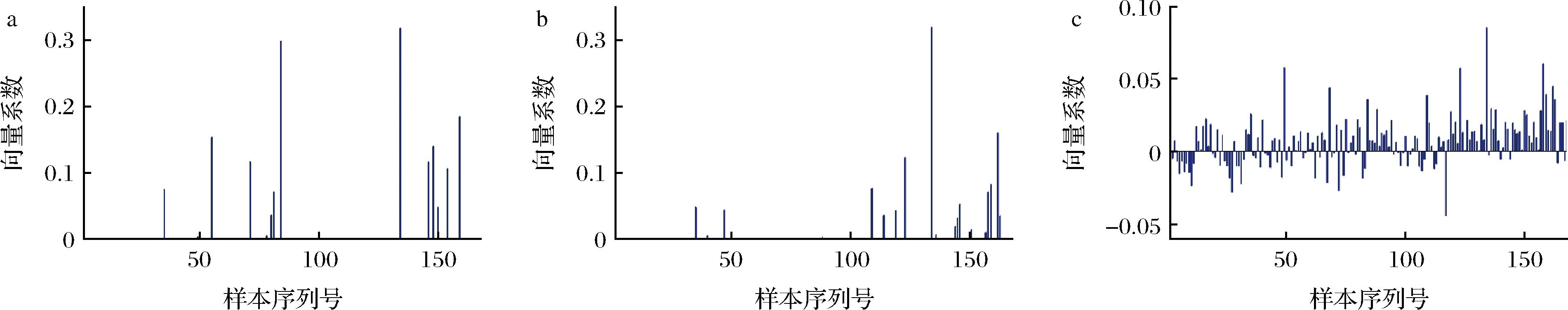
a-CRNRC;b-NRC;c-CRC
图3 由CRNRC、NRC、CRC得到的编码向量
Fig.3 The coding vectors obtained by CRNRC, NRC, and CRC
由图3可知,对于CRNRC、NRC和CRC模型,第40个测试样本的编码向量除了健康样本表示外,还由训练集中受DST污染样本表示。CRC使用更多的训练集样本来表示测试集的第40个样本,主要原因是CRC包含负项。CRNRC比NRC使用更多的训练数据集中的受DST污染样本来表示第40个样本,因此,协同表示使用更多的其他类训练集样本表示测试样本,以解决实践中样本不足问题。
2.2 CRNRC模型的最优参数
利用主成分分析法对贻贝光谱数据集进行降维后,维数在1~20,评价指标为CRNRC在不同参数λ和维度值下的平均准确率。30次重复实验的平均准确率如图4所示。

图4 参数λ和维度对平均准确率的影响
Fig.4 Impact of parameter λ and dimension on accuracy
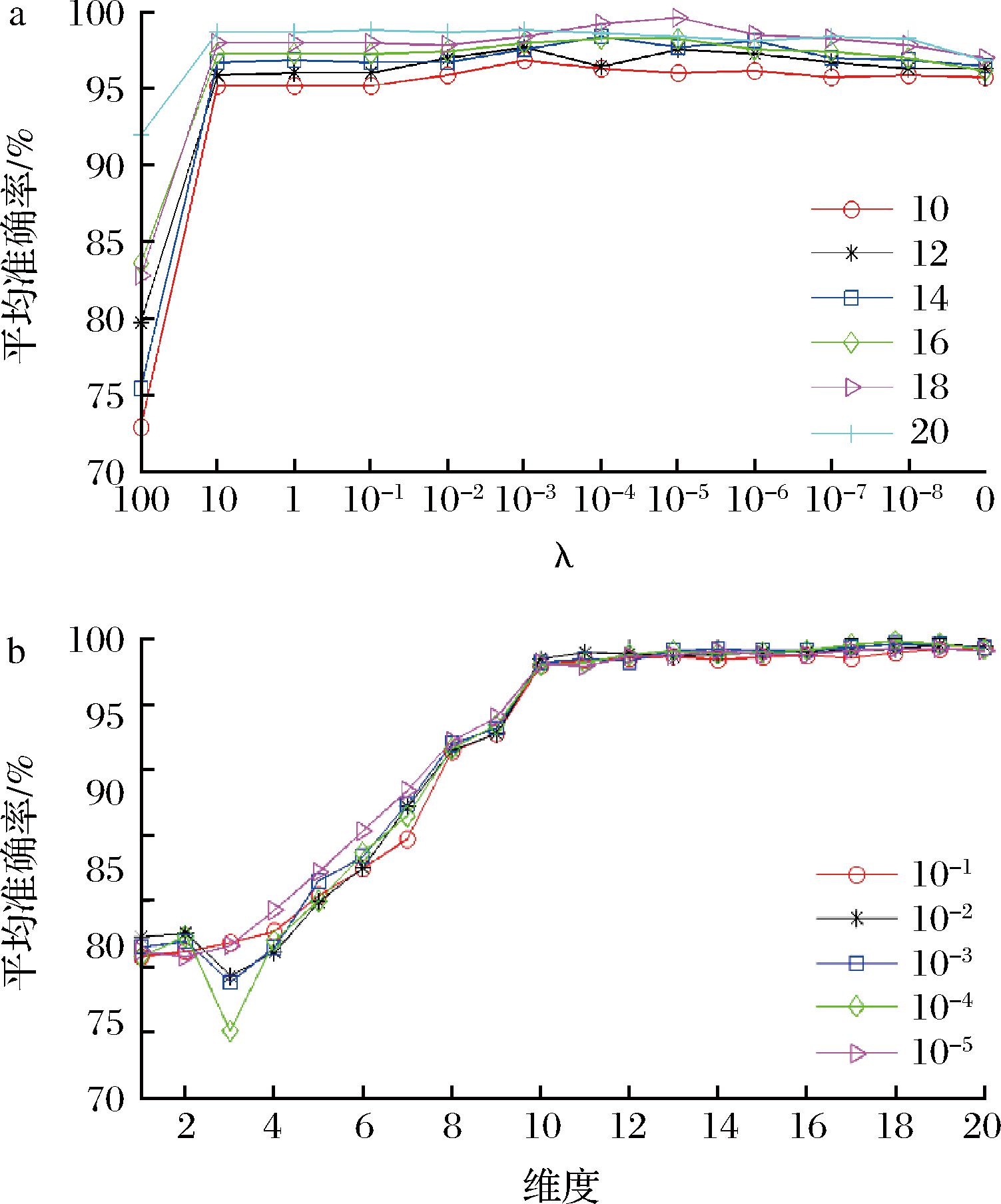
当维度小于10时,平均准确率随维度的减小而急剧变化。当维度大于10时,准确率随着参数λ的减小而缓慢上升。为了探究参数λ、维度和平均准确率之间的具体关系。将图4中的三维图形投影到参数λ和平均准确率的平面上,以精确地表示它们之间的关系,如图5-a所示。
a-参数λ对平均准确率的影响;b-维度对平均准确率的影响
图5 图4的2个投影图
Fig.5 Two projections of figure 4
平均准确率随参数λ的变化如图5-a所示。这6条曲线分别表示了维度为10、12、14、16、18和20时的平均准确率变化情况。当参数λ从100减小到10时,平均准确率曲线迅速上升。当该参数λ=10时,除维度10和12外,其余维度对应的平均准确率均大于96%。当参数λ在10~0范围内变化时,平均准确率略有波动。当参数为λ=10-5时,各维度的平均准确率达到最大值。因此,参数λ=10-5为最优值。
将图4中的三维图形投影到维度和平均准确率的平面上,平均准确率随不同维度的变化情况如图5-b所示。5条曲线分别表示参数为10-1、10-2、10-3、10-4、10-5时的精度变化情况。从总体趋势看,除维度3外,平均准确率随维数的增加而上升。当维度在10~20时,平均准确率均在97%以上。当维度为18时,平均准确率达到最大值,因此,维度的最优值为18。
惩罚参数μ对平均准确率也有影响。图6为参数λ为10-5时,平均准确率随各惩罚参数μ的变化情况。这6条曲线分别表示了维度为10、12、14、16、18和20时的平均准确率变化情况,精度随惩罚参数μ的变化而波动。惩罚参数μ为10-5时,除维度10外,其他维度的准确率达到最大值,维度18平均准确率达到99.58%,因此,惩罚参数μ为10-5是最优值。
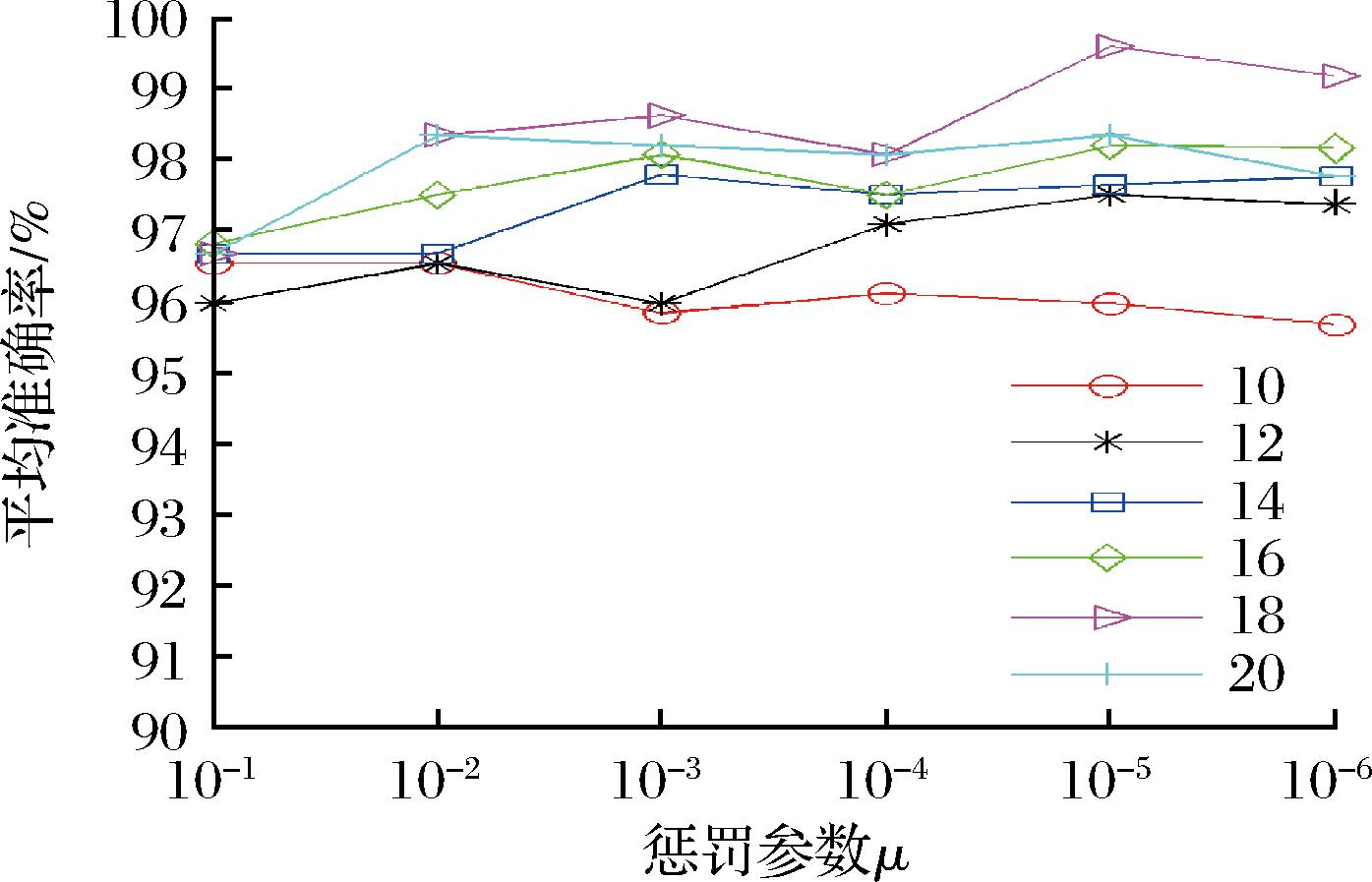
图6 惩罚参数μ对准确率的影响
Fig.6 Influence of the penalty parameter μ on accuracy
2.3 分类模型性能比较
在确定CRNRC模型最佳参数后,对CRNRC、CRC和NRC的准确性、F-measure和1-specificity指标进行评估。30次重复实验的平均见表1、表2和表3。
表1 三种分类模型的平均准确率 单位:%
Table 1 The average accuracy of three classification models
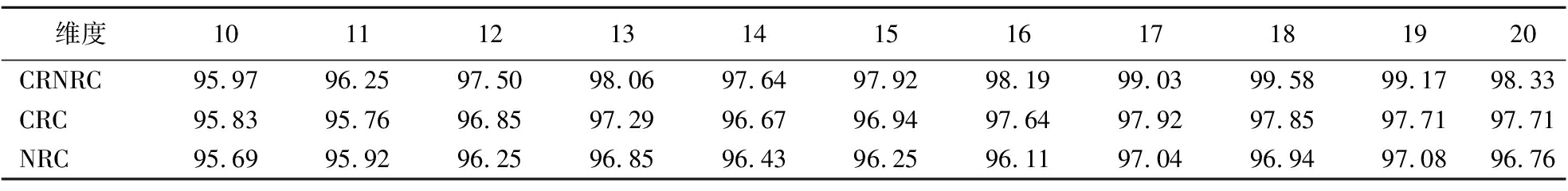
维度1011121314151617181920CRNRC95.9796.2597.5098.0697.6497.9298.1999.0399.5899.1798.33CRC95.8395.7696.8597.2996.6796.9497.6497.9297.8597.7197.71NRC95.6995.9296.2596.8596.4396.2596.1197.0496.9497.0896.76
表2 三种分类模型的F-measure值
Table 2 The F-measurement of three classification models
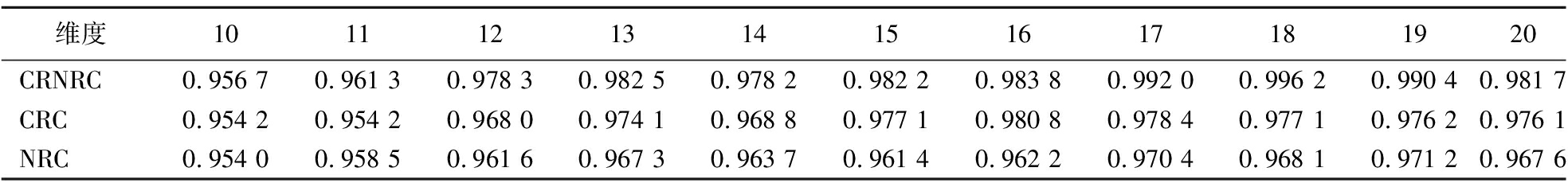
维度1011121314151617181920CRNRC0.956 70.961 30.978 30.982 50.978 20.982 20.983 80.992 00.996 20.990 40.981 7CRC0.954 20.954 20.968 00.974 10.968 80.977 10.980 80.978 40.977 10.976 20.976 1NRC0.954 00.958 50.961 60.967 30.963 70.961 40.962 20.970 40.968 10.971 20.967 6
表3 三种分类模型的1-specificity值
Table 3 The 1-specificity of three classification models
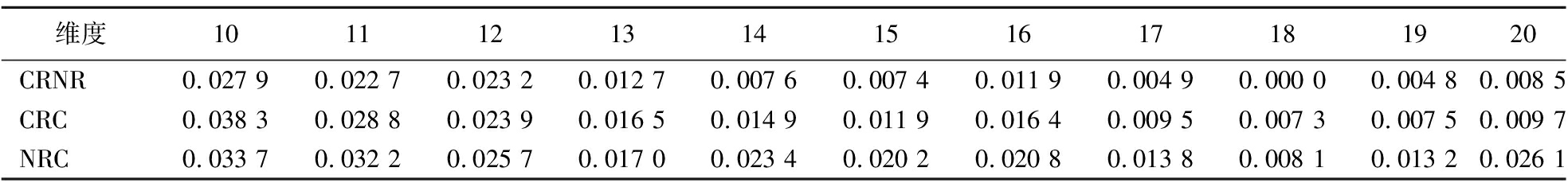
维度1011121314151617181920CRNR0.027 90.022 70.023 20.012 70.007 60.007 40.011 90.004 90.000 00.004 80.008 5CRC0.038 30.028 80.023 90.016 50.014 90.011 90.016 40.009 50.007 30.007 50.009 7NRC0.033 70.032 20.025 70.017 00.023 40.020 20.020 80.013 80.008 10.013 20.026 1
表1给出了3种模型的平均准确率实验结果。在各个维度上,CRNRC的平均准确率均高于CRC和NRC。对于CRNRC,当维度为18时,平均准确率达到99.58%。当维度为17和19时,CRC和NRC的平均准确率分别达到97.92%和97.08%。在维度18时,3种算法的分类性能存在明显差异。CRNRC、CRC和NRC的准确率分别为99.62%、97.84%和96.81%,因此,在平均准确率方面,CRNRC的性能在各个维度上都优于CRC和NRC。
表2给出了3种模型的F-measure的实验结果。在各维度上,CRNRC的F-measure值均高于CRC和NRC。对于CRNRC,F-measure在维度18上达到最大值0.996 2。对于CRC和NRC,F-measure的最大值分别出现在第16维和第19维,分别为0.980 8和0.971 2,因此,CRNRC的分类质量优于CRC和NRC。
在本研究中,1-specificity的值代表了被DST污染的贻贝被预测为健康贻贝的概率,本研究期望在某些维度上发现较低的1-specificity值。表3给出了3种算法1-specificity的实验结果。可见,在各维度上,CRNRC的1-specificity均低于CRC和NRC。3种算法的1-specificity在维数为18时降至最低,CRC、NRC和CRNRC的1-specificity最小值分别为0.007 5、0.008 1和0。由于CRC和NRC的1-specificity在每个维度上都不为零,因此,CRC和NRC会导致将受DST污染的贻贝预测为健康贻贝的问题。由于1-specificity值在第18维度上减小为零,则CRNRC模型不会出现将受DST污染的贻贝预测为健康贻贝的问题,因此,利用CRNRC模型对贻贝进行识别,可以保证人类食用健康的贻贝。
总之,CRNRC在平均准确率、F-measure和1-specificity方面优于CRC和NRC,CRNRC比CRC和NRC更适合于检测DST污染的贻贝。特别是当参数λ=10-5、惩罚参数μ=10-5,且维度为18时,CRNRC的1-specificity为零,表明CRNRC模型可以准确区分受DST污染的贻贝。
比较CRNRC与极限梯度增强(eXtreme gradient boosting,XGBoost)、多层感知器(multilayer perceptron,MLP)和偏最小二乘判别分析(partial least squares discriminant analysis,PLS-DA)在精度、F-measure和1-specificity方面的性能,实验结果见表4。CRNRC的参数来源于上面的实验结果,其他3个经典算法使用默认参数执行。可以看出,CRNRC在准确率、F-measure和1-specificity方面明显优于其他经典算法。
表4 比较CRNRC和几个经典算法
Table 4 Comparsion of CRNRC and several classical algorithms
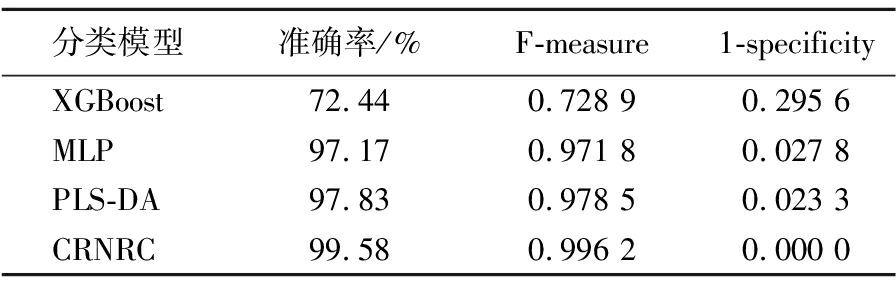
分类模型准确率/%F-measure1-specificityXGBoost72.440.728 90.295 6MLP97.170.971 80.027 8PLS-DA97.830.978 50.023 3CRNRC99.580.996 20.000 0
3 结论
本文研究了CRNRC与近红外光谱相结合检测受DST污染贻贝的可行性,这是一种比传统化学方法的检测技术更出色的检测手段,其通过识别光谱曲线间接检测了受DST污染贻贝。在确定了CRNRC模型的最优参数后,评估了CRNRC、NRC和CRC的平均准确率、F-measure和1-specificity指标。CRNRC的检测性能最佳,其平均准确率为99.58%,F-measure值为0.996 2,1-specificity值为0。
研究表明,采用近红外光谱技术的CRNRC可以准确地识别出DST污染的贻贝,该检测方法具有快速、智能和准确等优点。特别值得注意的是,CRNRC不会将受DST污染的贻贝鉴别为健康贻贝,因此,应用CRNRC方法检测贻贝可以保证人类食用健康的贻贝。
在收集含水贻贝光谱数据的过程中,水分会使近红外光谱分析复杂。在未来的工作中,将研究如何提高检测模型的鲁棒性,以消除水对贻贝近红外光谱的影响。同时可将近红外光谱检测贻贝的方法扩展到受到的其他污染贝类的检测,如检测贝类受到核污染等。
[1] REGUERA B, RIOB P, RODR
P, RODR GUEZ F, et al.Dinophysis toxins:Causative organisms, distribution and fate in shellfish[J].Marine Drugs, 2014, 12(1):394-461.
GUEZ F, et al.Dinophysis toxins:Causative organisms, distribution and fate in shellfish[J].Marine Drugs, 2014, 12(1):394-461.
[2] FARABEGOLI F, BLANCO L, RODR GUEZ L P, et al.Phycotoxins in marine shellfish:Origin, occurrence and effects on humans[J].Marine Drugs, 2018, 16(6):188.
GUEZ L P, et al.Phycotoxins in marine shellfish:Origin, occurrence and effects on humans[J].Marine Drugs, 2018, 16(6):188.
[3] GERSSEN A, POL-HOFSTAD I E, POELMAN M, et al.Marine toxins:Chemistry, toxicity, occurrence and detection, with special reference to the dutch situation[J].Toxins, 2010, 2(4):878-904.
[4] HOLLOWAY T, RIOS A, OKORO C, et al.Replacing high-performance liquid chromatography (HPLC) with high-resolution thin layer chromatography (TLC) for rapid radiopharmaceutical analysis[J].Nuclear Medicine and Biology, 2021, 96:S63.
[5] RODELL R, NING T S, GANGULY A, et al.DNA Damage Responses[M].New York:Springer, 2022:125-140.
[6] ![]() M, JEROMEL A, et al.Analysis of varietal thiols in sauvignon blanc wines—Optimization of a solid-phase extraction gas chromatography Tandem mass spectrometry method[J].Food Analytical Methods, 2022, 15(6):1591-1605.
M, JEROMEL A, et al.Analysis of varietal thiols in sauvignon blanc wines—Optimization of a solid-phase extraction gas chromatography Tandem mass spectrometry method[J].Food Analytical Methods, 2022, 15(6):1591-1605.
[7] MOLNAROVA K, DURIS A, JECMEN T, et al.Comparison of human IgG glycopeptides separation using mixed-mode hydrophilic interaction/ion-exchange liquid chromatography and reversed-phase mode[J].Analytical and Bioanalytical Chemistry, 2021,413 (16):4321-4328.
[8] KARGI F, TOPRAK H.Rational design of metal mesh particles for biological fluidized bed reactors[J].Journal of Chemical Technology &Biotechnology, 1994, 59 (2):201-204.
[9] KUMAR R, SHARMA V.A novel combined approach of diffuse reflectance UV-Vis-NIR spectroscopy and multivariate analysis for non-destructive examination of blue ballpoint pen inks in forensic application[J].Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2017, 175(3):67-75.
[10] GUO X H, CAI R, WANG S S,et al.Non-destructive geographical traceability of sea cucumber (Apostichopus japonicus) using near infrared spectroscopy combined with chemometric methods[J].Royal Society Open Science, 2018,5(1):170714.
[11] SHIM K, JEONG Y.Freshness evaluation in chub mackerel (Scomber japonicus) using near-infrared spectroscopy determination of the cadaverine content[J].Journal of Food Protection, 2019, 82(5):768-774.
[12] HUAI Q Y, GAO C L, MIAO J L, et al.Fast detection of saxitoxin using laser tweezers surface enhanced Raman spectroscopy[J].Analytical Methods, 2013, 5(23):6870-6873.
[13] VARR M O, FASOLATO L, SERVA L, et al.Use of near infrared spectroscopy coupled with chemometrics for fast detection of irradiated dry fermented sausages[J].Food Control, 2020, 110(4):107009.
M O, FASOLATO L, SERVA L, et al.Use of near infrared spectroscopy coupled with chemometrics for fast detection of irradiated dry fermented sausages[J].Food Control, 2020, 110(4):107009.
[14] PUERTAS G, V ZQUEZ M.Fraud detection in hen housing system declared on the eggs′ label:An accuracy method based on UV-VIS-NIR spectroscopy and chemometrics[J].Food Chemistry.2019, 288(2):8-14.
ZQUEZ M.Fraud detection in hen housing system declared on the eggs′ label:An accuracy method based on UV-VIS-NIR spectroscopy and chemometrics[J].Food Chemistry.2019, 288(2):8-14.
[15] GHIDINI S, CHIESA L M, PANSERI S, et al.Histamine control in raw and processed tuna:A rapid tool based on NIR spectroscopy[J].Foods, 2021, 10(4):885.
[16] CHENG J H, SUN D W.Rapid and non-invasive detection of fish microbial spoilage by visible and near infrared hyperspectral imaging and multivariate analysis[J].LWT - Food Science and Technology.2015, 62 (2):1060-1068.
[17] TITO N B, RODEMANN T, POWELL S M.Use of near infrared spectroscopy to predict microbial numbers on Atlantic salmon[J].Food Microbiology,2012, 32(2):431-436.
[18] LIU Y, XU L L, ZENG S G, et al.Rapid detection of mussels contaminated by heavy metals using near-infrared reflectance spectroscopy and a constrained difference extreme learning machine[J].Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2022, 269(3):120776.
[19] LIU Y, QIAO F, XU L L, et al.Fast detection of diarrhetic shell sh poisoning toxins in mussels using NIR spectroscopy and improved twin support vector machines[J].Frontiers in Marine Science, 2022, 9:907378.
sh poisoning toxins in mussels using NIR spectroscopy and improved twin support vector machines[J].Frontiers in Marine Science, 2022, 9:907378.
[20] YIN H F, WU X J.Class-specific residual constraint non-negative representation for pattern classification[J].Journal of Electronic Imaging.2020, 29 (2):1.
[21] ZHANG L, YANG M, FENG X C. Sparse representation or collaborative representation: Which helps face recognition?[C]. Barcelona: International Conference on Computer Vision, 2011: 6-13.
[22] XU J, AN W P, ZHANG L, et al.Sparse, collaborative, or nonnegative representation:Which helps pattern classification?[J].Pattern Recognition, 2019, 88 (4):679-688.
[23] MISHRA P, LOHUMI S, Improved prediction of protein content in wheat kernels with a fusion of scatter correction methods in NIR data modelling[J].Biosystems Engineering, 2021, 203:93-97.
[24] BOYD S, PARIKH N, CHU E, et al.Distributed optimization and statistical learning via the alternating direction method of multipliers[J].Foundations and Trends® in Machine Learning, 2010, 3(1):1-122.
[25] HU K J, YIN H F, SUN J.Discriminative non-negative representation based classifier for image recognition[J].Journal of Algorithms &Computational Technology.2021, 15:174830262110449.