生物传感器作为一种新型、便捷、高效的将生物信号转化为电信号、化学信号等可视化信号或易检测信号的一种接收与信息转化器[1],其与目标代谢物特异性结合后将改变核酸或蛋白质的空间构象,并将胞内目标代谢物的浓度转化为荧光信号或代谢通路表达水平信号等[1]。基于上述信号的变化可实时监测出胞内目标代谢物的浓度,进而广泛用于生物、医学等领域。然而,生物传感器本身的结构特征(如启动子,核糖体结合位点等元件)使其与目标代谢物无法特异性结合导致输出信号有偏差;输出信号无法与胞内的目标代谢物浓度的响应形成一定的数学关系,这些均是构建性能优良的生物传感器的难点。转录因子的传感器(transcription-factor-based biosensors,TFBBs)因转录因子的多样性和特异性而广泛应用。因此,本文对TFBBs进行了分类,也介绍了优化生物传感器性能的策略,及总结了生物传感器在高通量筛选等方面的应用。
1 TFBBs的种类及其原理
根据转录因子的来源,可以将基于TFBBs分为基于天然转录因子的生物传感器和基于人工合成转录因子的生物传感器两类。
1.1 基于天然转录因子的生物传感器
转录因子在自然界中无处不在,天然转录因子即在各类生物中本身存在的可调控靶配体的转录因子,直接将其提取用于同种或者导入不同种的生物中构建生物传感器。近年,将天然转录因子导入不同生物中的技术越发成熟,通过这种方法可以使基因多样化,为生物传感器的构建创造更多的可能性。当其直接构建的生物传感器性能不佳时,采用定向进化、易错PCR等方式进行改造,从而可获得更好的性能。天然转录因子中,变构转录因子是重要的一部分[2]。其原理是变构转录因子的结构会受到效应分子的诱导发生改变,从而与结合位点结合或从结合位点脱落,影响基因的表达(图1)。例如NI等[3]在大肠杆菌中过量表达枯草芽孢杆菌的ExuR转录因子,构建了具有异源转录因子和同源杂交启动子的半乳糖醛酸生物传感器。BEABOUT等[4]通过在大肠杆菌中表达转录因子BreR(一种来自胆管弧菌的类TetR抑制因子)来设计胆汁酸传感器。XIAO等[5]利用来自铜绿假单胞菌PAO1的变构转录因子LldR开发了D,L-乳酸生物传感器,并将其导入大肠杆菌进行性能调节。ASEMOLOYE等[6]利用细菌转录因子MarR和PdhR,设计了酵母生物传感器,其中,基于MarR的酵母生物传感器可响应苯甲酸盐和水杨酸盐,基于PdhR的生物传感器可响应丙酮酸。
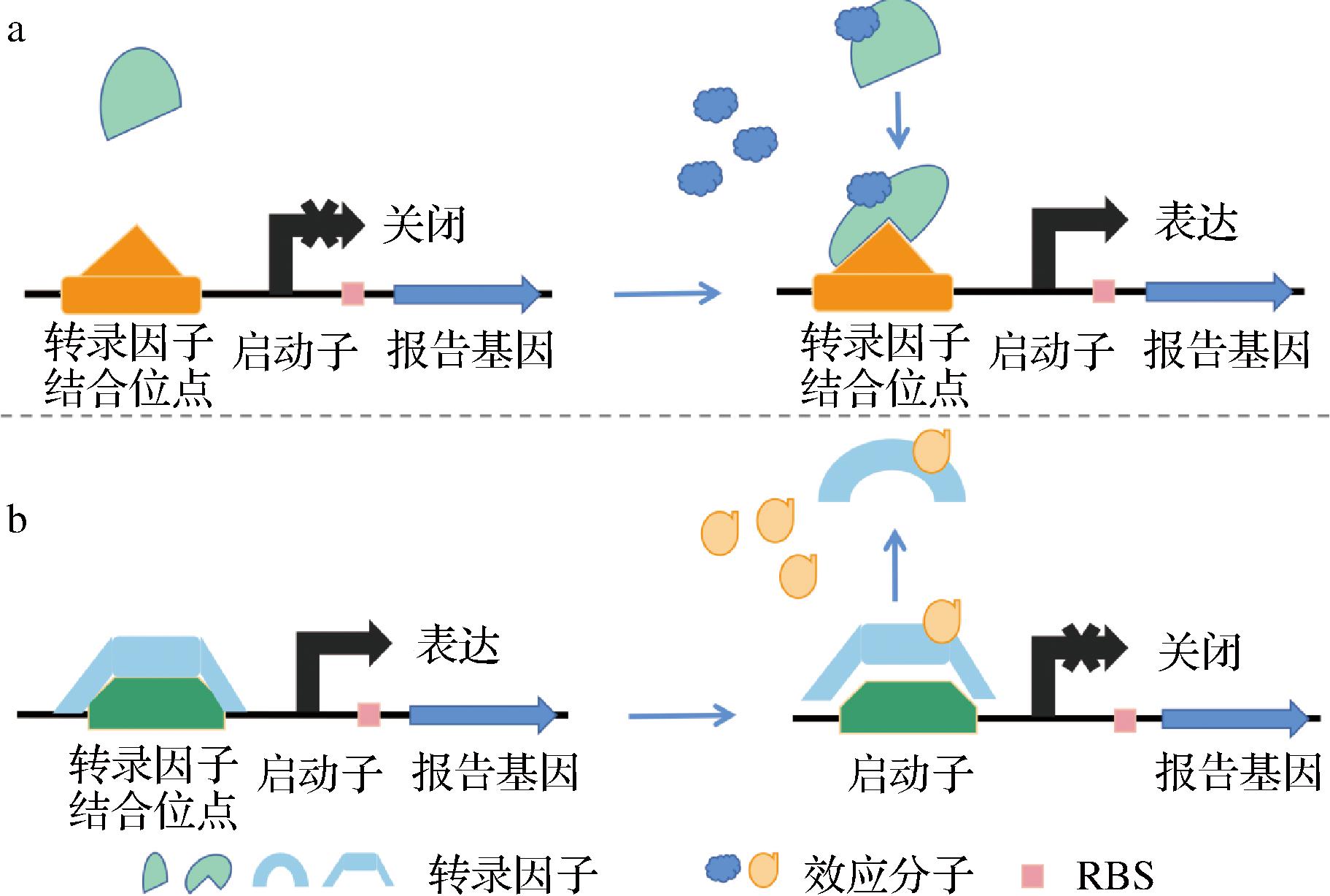
a-基于转录激活因子的生物传感器;b-基于转录抑制因子的生物传感器
图1 变构转录因子的调控原理
Fig.1 Regulation of allosteric transcription factors
1.2 基于人工合成转录因子的生物传感器
自然界中已知的转录因子效应范围、性能往往存在一定的局限性,开发高效的生物传感器来检测代谢物仍具挑战性。当今,随着合成生物学的快速发展,当环境中没有直接可用的天然转录因子时,可以利用模块化[7],或者应用定向进化创建生物传感器[8],满足人类的应用需求。例如CHEN等[9]构建了一种基于合成转录因子的酿酒酵母S-腺苷蛋氨酸(S-mdenosyl methionine,SAM)检测生物传感器,利用大肠杆菌衍生的DNA结合结构域MetJ、GS接头、人的雌激素受体结合域hER和病毒激活域VP16合成转录因子,构建的生物传感器能够以荧光强度输出SAM的剂量,受到诱导剂SAM和β-雌二醇的严格调节。YOUNGERA等[10]将锌指结合序列与麦芽糖结合蛋白融合,采用分裂蛋白的方法,将麦芽糖结合蛋白分裂成两半,然后将其融合到完整的锌指结合序列的N端和C端,从而将配体结合蛋白转化为配体响应转录因子,既能结合配体麦芽糖,又能与DNA相互作用,通过人工合成转录因子构建了能对麦芽糖响应的生物传感器。
2 生物传感器的性能调节
生物传感器在医学检测、环境监测、发酵工业、制药工程等行业都具有广阔的前景[11-14],然而天然的TFBBs由于其响应时间慢、检测范围窄以及选择性不专一等,可能会产生假阳性[7]等不利反应,往往不能满足实时检测的要求[15],无法适应现代工业需求。因此,需要对生物传感器的性能进行改善、调节,以发挥出生物传感器的最大功能。生物传感器的性能主要包括灵敏性、检测范围、动态范围和特异性4个方面[16](图2)。可以通过改变生物传感器中的启动子的强度和数目、调节转录因子和结合位点的亲和力[17]、改变质粒拷贝数、利用文库模拟寻找最优元件组合[17]等方法来调节TFBBs的性能。
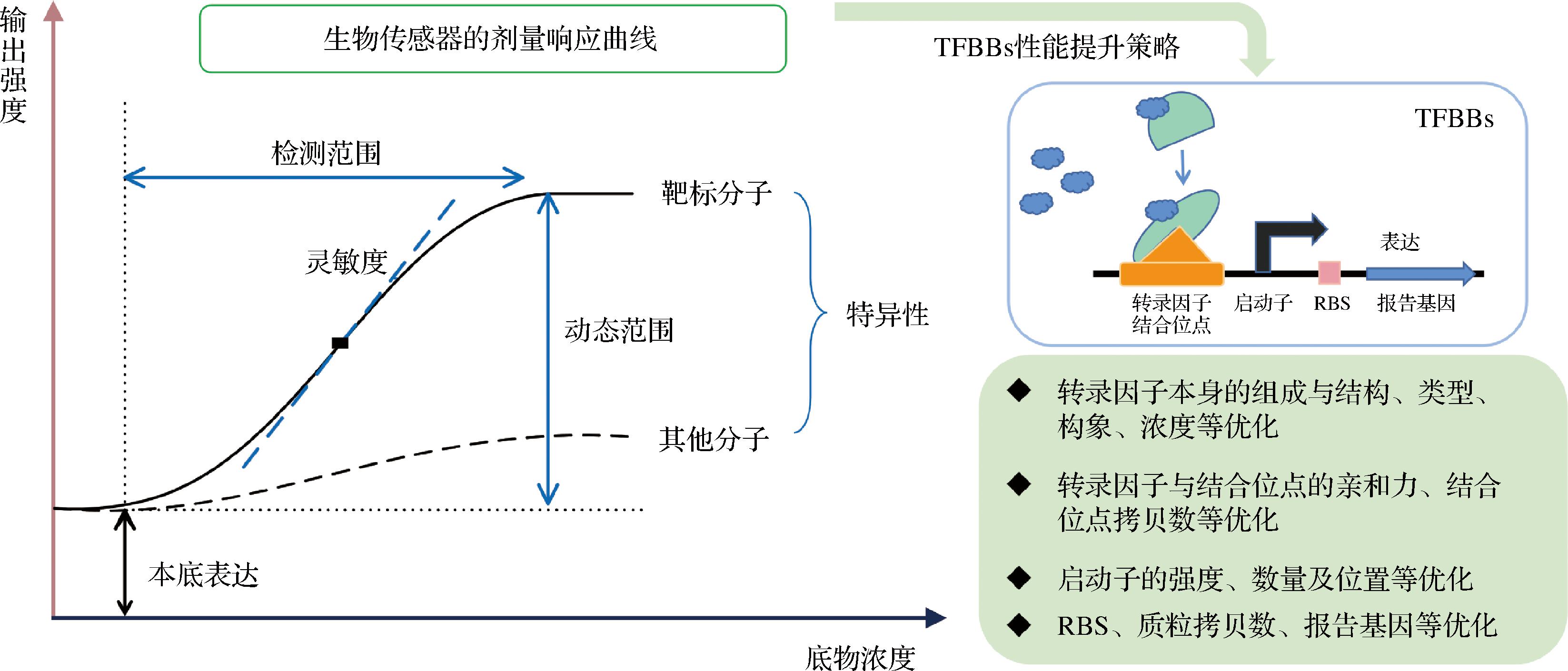
图2 生物传感器的剂量响应曲线及性能调节策略示意图
Fig.2 Dose-response curve and schematic diagram of biosensor perfoemance tuning
2.1 灵敏性
生物传感器的灵敏性通常是指目标检测物浓度发生变化时,传感器输出信号的变化大小[18],同样的目标检测物浓度变化能引起传感器输出信号的变化越大,灵敏性就越好。
转录因子浓度的高低可以影响传感器对靶分子的敏感性,同时,传感器转录因子的类型是激活剂还是阻遏因子,会决定可得到最佳响应对应的转录因子细胞内浓度是需要增加还是减少[19]:对于激活剂,增加转录因子的浓度可以增加反应启动子的输出,使其对靶分子更敏感;而对于阻遏因子则与之相反[20]。TSAI等[17]构建了基于转录因子CupR的生物传感器,用于检测Au3+的浓度,比较了不同CupR浓度下基因表达强度的差异,发现CupR浓度越低时,报告基因表达强度越高。另外,转录因子本身的组成和结构,也会在不同程度上影响传感器响应的灵敏程度,改变转录因子本身,对提高传感器的灵敏性也有帮助。LEBOVICH等[21]对于大肠杆菌(Escherichia coli)Nissle 1917(EcN)丙酸盐传感器,调整了PrpR转录激活剂的表达,将传感器的灵敏度提高了>500倍,使得传感器能够被人体肠道中普遍的丙酸盐浓度(5~30 mmol/L)激活。
转录因子的结合位点拷贝数、构象等,能够改变基因表达的强度,如图3所示,从而对传感器的灵敏度产生一定的影响;不同启动子、不同核糖体结合位点(ribosome binding site,RBS)下报告基因的表达会存在一定的强度差异,合适的启动子和RBS组合能够增强基因的表达,从而提高传感器的灵敏性,得到更加高效的生物传感器。ZHAO等[22]将在初步构建的基于转录因子CgmR和启动子Ptrc的生物传感器,进行了启动子的更换和通过易错PCR设计转录因子CgmR,最终获得的基于启动子PaspB的转录因子CgmR突变体I152T的生物传感器,相较于野生型,改进后的生物传感器对腐胺的检测限由3.2 mmol/L降为了0.2 mmol/L,生物传感器灵敏性得到了提高。NU EZ等[23]在构建检测岩藻糖的生物传感器时,将转录因子FucR分别克隆到低拷贝质粒和高拷贝质粒中,发现在高拷贝质粒中,岩藻糖检测值提高了近50%,且具有良好的分离度,灵敏性得到了提高,部分TFBBs的性能调节实例如表1所示。
EZ等[23]在构建检测岩藻糖的生物传感器时,将转录因子FucR分别克隆到低拷贝质粒和高拷贝质粒中,发现在高拷贝质粒中,岩藻糖检测值提高了近50%,且具有良好的分离度,灵敏性得到了提高,部分TFBBs的性能调节实例如表1所示。
表1 部分TFBBs的性能调节实例
Table 1 Some performance adjustment examples of TFBBs

转录因子检测物性能调节策略调节后性能参考文献PrpR丙酸盐通过更换启动子调节PrpR的表达背景噪音降至(0.04±0.005) RPU,灵敏性提高500倍,动态范围提高59倍,检测范围为5~30 mmol/L[21]BmoR正丁醇、异丁醇对BmoR进行易错PCR检测范围提升至0~200 mmol/L[24]FucRL-岩藻糖拷贝数、诱导物浓度及启动子优化L-岩藻糖浓度在0~45 mmol/L范围内线性响应,检测范围相比对照提高50%,分离度及灵敏性均得到提升[23]ExuRD-半乳糖醛酸通过优化不同来源的ExuR、启动子及宿主传感器的动态调控范围提高29.8倍,渗漏表达降至1.8[3]BreR胆汁酸通过优化启动子、报告基因表达动态范围增加470倍[4]GabRγ-氨基丁酸通过更换启动子探究GabR的表达空间首次建立γ-氨基丁酸传感器,动态范围相比对照高138倍[21]CgmR腐胺更换启动子和通过易错PCR改造CmgR生物传感器检测限由3.2 mmol/L降为0.2 mmol/L[22]ArsR亚砷酸盐转录因子结合位点拷只数及启动子优化亚砷酸盐在0.1~4 μmol/L范围内线性响应,检测限在10 nmol/L[25]PsiRD-阿洛酮糖理性设计转录因子PsiR生物传感器最低检测浓度由50 mmol/L下降至10 mmol/L[26]BenM顺,顺-黏康酸对转录因子BenM进行易错PCR及改造实现对己二酸的特异性响应,0~15 mmol/L的响应范围[27]NodD1-FdeR类黄酮、柚皮素、芹菜素、木犀草素开发定制配体并嵌合启动子调控传感器性能实现对木犀草素的特异性响应,对其响应特异性从27.5%提高到95.3%[28]
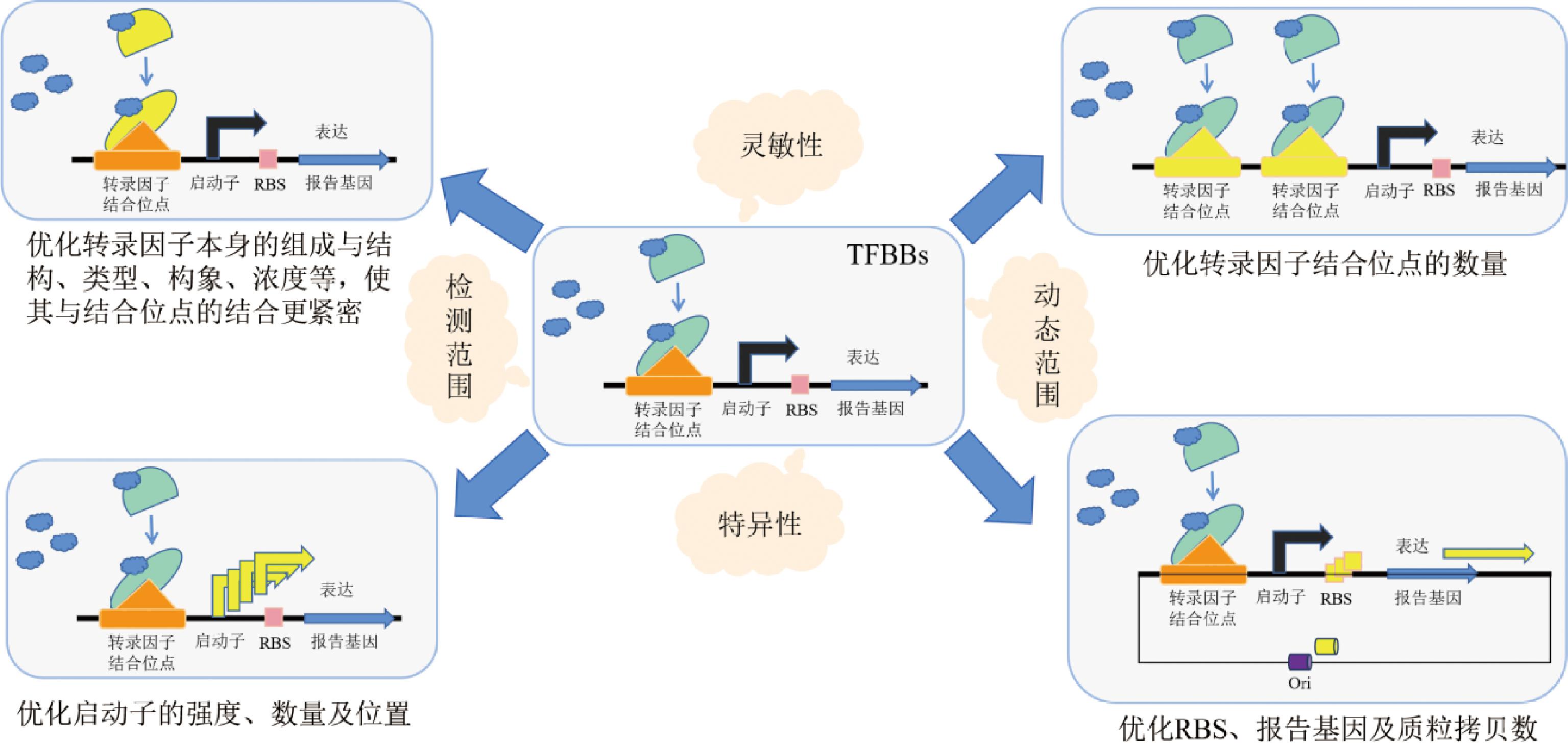
图3 TFBBs性能调节的策略举例
Fig.3 Examples of TFBBs performance tuning strategies
2.2 检测范围
检测范围表示可检测到的最低代谢物浓度和最高代谢物浓度间的范围[29]。适宜的可检测范围对于日常生产非常重要,检测范围过高或过低都会导致检测不准确甚至无法检测出目的代谢物,因此,需要优化生物传感器以获得更适宜的检测范围来满足实际需求。
转录因子与结合位点的结合能力以及调节转录的能力都会随构象改变而发生变化,当天然转录因子不足以满足传感器设计的需求时,可采用蛋白质工程对天然转录因子进行突变[30],从而得到与天然转录因子构象不同的突变转录因子,再利用计算机和文库结合进行模拟,从突变中筛选出优质的转录因子,从而得到更加宽广的检测范围。HUO等[31]通过改造天然转录因子BmoR,获得BmoR F276A/E627N的生物传感器突变体,经与野生型BmoR比较发现,突变体BmoR对底物正丁醇和异丁醇的响应范围增加到了0~100 mmol/L(0~7.4 g/L)。利用易错PCR构建随机诱变文库,同样也能够筛选出宽检测范围的突变体。WU等[24]利用易错PCR构建了基于BmoR 转录因子N末端的随机诱变文库,并获得了与野生型BmoR相比具有更宽检测范围(0~200 mmol/L)的突变体(T12 N)。由此可以得出,通过构建突变文库这一方法来筛选、构建更佳性能的生物传感器,是可行且高效的。
2.3 动态范围
动态范围表示在存在和缺乏代谢物时,基因表达得到的信号输出的倍数变化[16],是泄漏表达与最大表达之间的比率。最大化动态范围对于得到可靠的结果非常重要,为了改善动态范围,需要尽量减少泄漏表达,同时尽可能增加最大表达,从而确保良好的信噪比[19],增大动态范围。
通过调整负责控制输出表达的启动子,改造优化启动子的-35 区和-10 区,从而改变泄漏表达和最大表达,可以达到优化和改善动态范围的目的[32]。LEBOVICH等[21]构建了一种E.coli Nissle 1917(EcN)基于神经递质γ-氨基丁酸(γ-aminobutyric acid,GABA)和GabR 转录抑制因子的生物传感器,通过一种合成的GabR转录抑制因子调控的EcN启动子,调整启动子的活性,最终使得生物传感器具有138倍的动态范围。
改变转录因子自身的结构,可以获得能引起高动态范围的生物传感器,如图3所示。GONG等[33]通过对TrpR1-PtrpO1生物传感器中的TrpR1转录因子进行诱变,得到多种突变体,相较于野生型,V58G突变体中色氨酸的动态范围增大17.7倍,5-羟基色氨酸(5-hydroxytryptophan,5-HTP,一种色氨酸类似物)的动态范围增大11.1倍;V58Q突变体对色氨酸和5-HTP的动态范围分别增大10.5倍和23.7倍。
RBS可以控制蛋白质的翻译水平和折叠,高翻译率将增加最大表达但会导致更高的泄漏表达,而低翻译率将减少泄漏表达[19],因此,RBS的强度可以影响蛋白的表达从而改善生物传感器的动态范围[34]。丁娜娜等[16]和SNOEK等[27]依托RBS大数据文库,通过随机选择不同来源的RBS替换初始的RBS,构建了81种cRBS生物传感器,从而调节所构建的葡萄糖二酸生物传感器的动态范围,经方差分析发现调节 RBS 对改善生物传感器动态范围贡献最大。
2.4 特异性
特异性表示生物传感器对靶分析物的响应与其他干扰物的响应有显著差异,从而能够实现对靶分析物的专一响应,或指生物传感器仅通过与目标生物分析物的相互作用才能产生阳性结果的能力。特异性较差的生物传感器往往具有较高的假阳性率,阻碍了生物传感器的可靠应用[35]。
转录因子的本质是蛋白质,它本身的结构以及与转录因子结合位点的亲和力就决定了它的特异性,提高转录因子的特异性通常也会提高生物传感器的灵敏度,因为配体与转录因子的专一结合可以使得生物传感器响应加强[19]。SNOEK等[27]通过易错PCR获得了转录因子BenM(受顺,顺-黏康酸诱导)的突变体,再通过定向进化,使用己二酸作为诱导剂进行了筛选,成功获得了己二酸特异性的BenM突变体,使得BenM的特异性由顺,顺-粘康酸进化为己二酸。DE PAEPE等[28]通过在生物传感器效应模块上构建嵌合启动子区域和在生物传感器检测器模块上构建嵌合转录因子两种策略,成功构建了一种嵌合生物传感器,把传感器对木犀草素的特异性从27.5% 提高到95.3%。由此可见,改造转录因子以及转录因子结合位点是改善生物传感器特异性的有效策略。
3 生物传感器的应用
TFBBs作为检测工具,具有许多优点:首先,其特异性强,能实现精准识别某一配体,大大降低了错误率;再者,其检测操作简单,能快速检测响应物质;同时其灵敏性强,微量的响应物质也能实现快速的检测;除此之外,将其与报告基因相偶联还能实现细胞内响应物的实时监测等。鉴于TFBBs的这些特点,生物传感器在如今已广泛应用于高通量筛选、适应性进化、动态调控等领域[36-37](图4)。

a-利用转录因子高通量筛选目的菌株;b-利用转录因子适应性进化高产菌株;c-利用转录因子FapR动态调控脂肪酸合成
图4 TFBBs在高通量筛选、适应性进化及动态调控上的应用
Fig.4 Application of TFBBs in high throughput screening, adaptive evolution, and dynamic regulation
3.1 高通量筛选
当TFBBs结合流式细胞荧光分选技术(fluorescence activated cell sorting,FACS)便可实现高活性酶及高产菌株的快速、批量筛选[8,38-39],大大提高了工作效率。XU等[38]开发了一种L-精氨酸生物传感器,在该传感器调控下,L-精氨酸产量高的菌株才可以在10%蔗糖筛选中存活,进而实现了高产突变体的高通量筛选,获得了产量增加35%的高产菌株。GAO等[39]利用L-半胱氨酸响应型转录调节因子CcdR开发了一种生物传感器,FACS偶联建立了高通量筛选平台,用于筛选高产L-半胱氨酸菌株。LI等[26]设计了一种基于转录因子PsiR的生物传感器,可响应D-阿洛酮糖,将其与酮糖3-差向异构酶表达系统偶联,构建了D-阿洛酮糖检测酶筛选系统,实现对酮糖3-差向异构酶进行超高通量筛选。CHEN等[40]首次开发和表征了一种基于转录因子AlkS的新型烷烃生物传感器,可用于实时监测长链烷烃产量。他们利用此生物传感器作为筛选工具,结合FACS,从酰基-ACP醛还原酶和醛变形加氧酶随机突变文库中高通量筛选高烷烃产菌株,成功将烷烃产量提高13倍。
3.2 适应性进化
当TFBBs结合外界环境给细胞施加一定的筛选压力便可实现对菌株或酶的适应性进化。MAHR等[41]在TFBBs、FACS及人工的选择压力下对谷氨酸棒杆菌菌株进行适应性进化,含有7个位点的突变体比亲本菌株的生长效率更高,L-缬氨酸产量增加25%,副产物减少3~4倍。SEOK等[42]基于C4-lysR转录因子的TFBBs结合四环素抗性压力对3-羟基丙酸生产菌株进行适应性进化,在重新分配细胞生长与化合物生产的碳通量后3-羟基丙酸的产量提高至7.7 g/L,约为最大理论产量的93%。CHA等[43]开发了基于XylR的生物传感器,结合FACS,在高浓度木糖的筛选压力下对生产2, 3-丁二醇(2,3-Butanediol,2,3-BDO)的克雷伯菌进行适应性进化,经筛选得到一株OmpR突变菌,其能够在短时间(47 h)内消耗高浓度木糖(238.13 g/L),进而合成57.5 g/L的2, 3-BDO。ZHANG等[44]为实现L-半胱氨酸菌株的高效生产,在培养基中额外添加L-半胱氨酸前体丝氨酸,结合大肠杆菌中构建的基于NCgl0581转录调节因子的丝氨酸TFBBs,经适应性进化该菌株丝氨酸的耐受性及产量均得到提升,其中丝氨酸的产量相比对照提高了275%。RAMAN等[45]利用TFBBs将代谢物浓度与细胞适应性相结合,并开发了一个阴性选择方案来排除假阳性菌株,经四轮适应性进化柚皮素及葡萄糖二酸产量分别提高了36倍、22倍。随后ZHENG等[46]使用上述葡萄糖二酸的TFBBs筛选肌醇加氧酶进化突变体,最终发现D82Y、S173N突变体的酶活力及合成葡萄糖二酸的产量均得到提升。除此之外,HAN等[47]基于转录因子BsNadR所构建的高性能烟酸生物传感器对腈水解酶适应性进化,筛选后酶活力相比野生型提高6.6倍。
3.3 动态调控
当TFBBs与细胞生长、细胞内化合物的合成等结合便可调控代谢途径,实现相关代谢产物的动态调控。XU等[48]基于FapR转录因子的TFBBs通过调控胞内丙二酰辅酶A浓度激活或抑制杂合启动子,实现对丙二酰辅酶A的动态调控,脂肪酸的产量提升2.1倍。LO等[49]基于FadR转录因子的TFBBs通过动态调控大肠杆菌的细胞生长和香草酸的合成,使得代谢的负担降低2倍,生产率提高5倍。WU等[50]基于LuxR转录因子、TetR转录因子的TFBBs通过理论建模与具体实验研究并重,调节细胞生长与代谢的平衡,酪醇和红景天苷的产量得到了一定的提升。LIU等[51]将基于红假单胞菌的转录抑制因子CouR构建p-香豆酰辅酶A生物传感器,并与基于转录因子FapR的丙二酰辅酶A生物传感器结合使用,用于柚皮素生产途径内对香豆酰辅酶A合成的双重动态调节,最终获得了47.3 mg/L的柚皮素,比非调节系统柚皮素产量增加了15倍。ZHANG等[52]构建了一个基于HrtR的血红素生物传感器,并与CRISPRi结合构建了基因表达与细胞内血红素的响应调节系统,经优化将血红素前体5-氨基乙酰丙酸产量提高到了5.35 g/L,为目前大肠杆菌分批发酵的最高产量。
4 结论与展望
TFBBs相较于传统的检测方法,具有灵敏性高、检测操作简单等显著优点,如果能够将生物传感器应用于生产,将会极大地提高效率,创造更大的价值。然而,基于天然转录因子的TFBBs还存在一定的局限性,其灵敏性、检测范围、动态范围、特异性虽然相较于传统代谢物检测手段有一定的优越性,但对于生产来说,通常还是无法满足需求,因此需要对传感器的各个部件,例如启动子、RBS等进行调换、重组,以获得最优的组合,得到高灵敏度、高特异性、宽检测范围、宽动态范围的优异生物传感器;也需要借助于现代科学手段,利用文库、模型等,简化测试,从而高效地筛选出适用的组合,再做进一步优化得到理想的结果;同时,随着合成生物学的高速发展,已经有人工合成的转录因子应用到生物传感器中,并表现出良好的性能,利用好合成生物学,将极大提高转录因子的丰富性,从而为构建具有优良性状的生物传感器创造可能。在不远的未来,随着生物元件的不断挖掘及科学技术的不断发展,有望构造更加灵敏、高效的生物传感器,实现胞内代谢物的实时监测及微生物细胞工厂的高通量筛选,进一步促进工业技术的发展。
[1] 廖佳敏, 杨华, 孙鹏宇, 等.生物传感器发展研究综述[J].中国高新科技, 2022(12):118-120.
LIAO J M, YANG H, SUN P Y, et al.Review of biosensor development research[J].China High and New Technology, 2022(12):118-120.
[2] MITCHLER M M, GARCIA J M, MONTERO N E, et al.Transcription factor-based biosensors:a molecular-guided approach for natural product engineering[J].Current Opinion in Biotechnology, 2021, 69:172-181.
[3] NI C, FOX K J, PRATHER K L J.Substrate-activated expression of a biosynthetic pathway in Escherichia coli[J].Biotechnology Journal, 2022,17(3), e2000433.
[4] BEABOUT K, EHRENWORTH BREEDON A M, BLUM S M, et al.Detection of bile acids in complex matrices using a transcription factor-based biosensor[J].ACS Biomaterials Science &Engineering, 2023,9(9):5151-5162.
[5] XIAO D, HU C X, XU X Z, et al.A D,L-lactate biosensor based on allosteric transcription factor LldR and amplified luminescent proximity homogeneous assay[J].Biosensors and Bioelectronics, 2022, 211:114378.
[6] ASEMOLOYE M D, MARCHISIO M A.Synthetic metabolic transducers in Saccharomyces cerevisiae as sensors for aromatic permeant acids and bioreporters of hydrocarbon metabolism[J].Biosensors and Bioelectronics, 2023, 220.
[7] CARPENTER A C, PAULSEN I T, WILLIAMS T C.Blueprints for biosensors:Design, limitations, and applications[J].Genes, 2018, 9(8): 375.
[8] LIANG Y, LUO J, YANG C, et al.Directed evolution of the PobR allosteric transcription factor to generate a biosensor for 4-hydroxymandelic acid[J].World Journal of Microbiology and Biotechnology, 2022, 38(6):104.
[9] CHEN Y W, ZHENG H J, YANG J J, et al.Development of a synthetic transcription factor-based S-adenosylmethionine biosensor in Saccharomyces cerevisiae[J].Biotechnology Letters, 2023, 45(2):255-262.
[10] YOUNGER A K D, DALVIE N C, ROTTINGHAUS A G, et al.Engineering modular biosensors to confer metabolite-responsive regulation of transcription[J].ACS Synthetic Biology, 2017, 6 (2):311-325.
[11] LIU K, ZHANG Y S, LIU K, et al.De novo design of a transcription factor for a progesterone biosensor[J].Biosensors and Bioelectronics, 2022, 203:113897.
[12] YEOM S J, KIM M, KWON K K, et al.A synthetic microbial biosensor for high-throughput screening of lactam biocatalysts[J].Nature Communications, 2018, 9(1):5053.
[13] ZHANG J W, BARAJAS J F, BURDU M, et al.Development of a transcription factor-based lactam biosensor[J].ACS Synthetic Biology, 2017, 6 (3):439-445.
[14] SCOTT L H, WIGGLESWORTH M J, SIEWERS V, et al.Genetically encoded whole cell biosensor for drug discovery of HIF-1 interaction inhibitors[J].ACS Synthetic Biology, 2022, 11(10):3182-3189.
[15] YANG H Q, YANG X Y, LU Y B, et al.Engineering a fumaric acid-responsive two-component biosensor for dynamic range improvement in Escherichia coli[J].Systems Microbiology and Biomanufacturing, 2022, 2(3):533-541.
[16] 丁娜娜, 周胜虎, 邓禹.基于转录因子的代谢物生物传感器的研究进展[J].生物工程学报, 2021, 37(3):911-922.
DING N N, ZHOU S H, DENG Y.Progress in transcription-factor-based metabolite biosensors[J].Chinese Journal of Biotechnology, 2021, 37(3):911-922.
[17] TSAI S T, CHENG W J, ZHANG Q X, et al.Gold-specific biosensor for monitoring wastewater using genetically engineered Cupriavidus metallidurans CH34[J].ACS Synthetic Biology, 2021, 10 (12):3576-3582.
[18] PHAM C, STOGIOS P J, SAVCHENKO A, et al.Advances in engineering and optimization of transcription factor-based biosensors for plug-and-play small molecule detection[J].Current Opinion in Biotechnology, 2022, 76:102753.
[19] HICKS M, BACHMANN T T, WANG B J.Synthetic biology enables programmable cell-based biosensors[J].Chemphyschem, 2020, 21(2):132-144.
[20] WANG B J, BARAHONA M, BUCK M.Amplification of small molecule-inducible gene expression via tuning of intracellular receptor densities[J].Nucleic Acids Research, 2015, 43(3):1955-1964.
[21] LEBOVICH M, ANDREWS L B.Surveying the genetic design space for transcription factor-based metabolite biosensors:synthetic gamma-aminobutyric acid and propionate biosensors in E.coli Nissle 1917[J].Frontiers in Bioengineering and Biotechnology, 2022, 10:938056.
[22] ZHAO N N, SONG J, ZHANG H, et al.Development of a transcription factor-based diamine biosensor in Corynebacterium glutamicum[J].ACS Synthetic Biology, 2021, 10 (11):3074-3083.
[23] NU EZ S, BARRA M, GARRIDO D.Developing a fluorescent inducible system for free fucose quantification in Escherichia coli[J].Biosensors, 2023,13(3):388.
EZ S, BARRA M, GARRIDO D.Developing a fluorescent inducible system for free fucose quantification in Escherichia coli[J].Biosensors, 2023,13(3):388.
[24] WU T, CHEN Z Y, GUO S Y, et al.Engineering transcription factor BmoR mutants for constructing multifunctional alcohol biosensors[J].ACS Synthetic Biology, 2022, 11(3):1251-1260.
[25] CHEN S Y, WEI W P, YIN B C, et al.Development of a highly sensitive whole-cell biosensor for arsenite detection through engineered promoter modifications[J].ACS Synthetic Biology, 2019, 8(10):2295-2302.
[26] LI C, GAO X, QI H B, et al.Substantial improvement of an epimerase for the synthesis of D-allulose by biosensor-based high-throughput microdroplet screening[J].Angewandte Chemie International Edition, 2023, 62(10):e202216721.
[27] SNOEK T, CHABERSKI E K, AMBRI F, et al.Evolution-guided engineering of small-molecule biosensors[J].Nucleic Acids Research, 2020, 48(1):e3.
[28] DE PAEPE B, MAERTENS J, VANHOLME B, et al.Chimeric LysR-type transcriptional biosensors for customizing ligand specificity profiles toward flavonoids[J].ACS Synthetic Biology, 2019, 8(2):318-331.
[29] CHENG F, TANG X L, KARDASHLIEV T.Transcription factor-based biosensors in high-throughput screening:Advances and applications[J].Biotechnology Journal, 2018, 13(7):e1700648.
[30] WILDING M, HONG N, SPENCE M, et al.Protein engineering:The potential of remote mutations[J].Biochemical Society Transactions, 2019, 47(2):701-711.
[31] HUO Y, YU H, CHEN Z.New BmoR F276A/E627 N biosensor mutant based on the transcription factor useful for screening isobutanol-producing strains:China, CN110615832-A[P].2019-12-27.
[32] CHEN Y, HO J M L, SHIS D L, et al.Tuning the dynamic range of bacterial promoters regulated by ligand-inducible transcription factors[J].Nature Communications, 2018, 9(1):64.
[33] GONG X Y, ZHANG R H, WANG J, et al.Engineering of a TrpR-based biosensor for altered dynamic range and ligand preference[J].ACS Synthetic Biology, 2022, 11(6):2175-2183.
[34] DING N N, YUAN Z Q, ZHANG X J, et al.Programmable cross-ribosome-binding sites to fine-tune the dynamic range of transcription factor-based biosensor[J].Nucleic Acids Research, 2020, 48(18):10602-10613.
[35] MORALES M A, HALPERN J M.Guide to selecting a biorecognition element for biosensors[J].Bioconjugate Chemistry, 2018, 29(10):3231-3239.
[36] LI J W, ZHANG X Y, WU H, et al.Transcription factor engineering for high-throughput strain evolution and organic acid bioproduction:A review[J].Frontiers in Bioengineering and Biotechnology, 2020, 8.
[37] MORMINO M, SIEWERS V, NYGÅRD Y.Development of an Haa1-based biosensor for acetic acid sensing in Saccharomyces cerevisiae[J].FEMS Yeast Research, 2021, 21(6):foab049.
[38] XU M J, LIU P P, CHEN J M, et al.Development of a novel biosensor-driven mutation and selection system via in situ growth of corynebacterium crenatum for the production of L-arginine[J].Frontiers in Bioengineering and Biotechnology, 2020, 8: 175.
[39] GAO J S, DU M H, ZHAO J H, et al.Design of a genetically encoded biosensor to establish a high-throughput screening platform for L-cysteine overproduction[J].Metabolic Engineering, 2022, 73:144-157.
[40] CHEN D D, XU S M, LI S L, et al.Directly evolved AlkS-based biosensor platform for monitoring and high-throughput screening of alkane production[J].ACS Synthetic Biology, 2023, 12 (3):832-841.
[41] MAHR R, G TGENS C, G
TGENS C, G TGENS J, et al.Biosensor-driven adaptive laboratory evolution of L-valine production in Corynebacterium glutamicum[J].Metabolic Engineering, 2015, 32:184-194.
TGENS J, et al.Biosensor-driven adaptive laboratory evolution of L-valine production in Corynebacterium glutamicum[J].Metabolic Engineering, 2015, 32:184-194.
[42] SEOK J Y, HAN Y H, YANG J S, et al.Synthetic biosensor accelerates evolution by rewiring carbon metabolism toward a specific metabolite[J].Cell Reports, 2021, 36(8):109589.
[43] CHA J W, JANG S H, SON J, et al.Engineering of Klebsiella oxytoca for the production of 2,3-butanediol from high concentration of xylose[J].ACS Sustainable Chemistry &Engineering, 2021, 9 (43):14395-14404.
[44] ZHANG X M, SUN Z H, BIAN J Y, et al.Rational metabolic engineering combined with biosensor-mediated adaptive laboratory evolution for L-cysteine overproduction from glycerol in Escherichia coli[J].Fermentation-Basel, 2022, 8(7):299.
[45] RAMAN S, ROGERS J K, TAYLOR N D, et al.Evolution-guided optimization of biosynthetic pathways[J].Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(50):17803-17808.
[46] ZHENG S, HOU J, ZHOU Y, et al.One-pot two-strain system based on glucaric acid biosensor for rapid screening of myo-inositol oxygenase mutations and glucaric acid production in recombinant cells[J].Metabolic Engineering, 2018, 49:212-219.
[47] HAN L C, LIU X Y, CHENG Z Z, et al.Construction and application of a high-throughput in vivo screening platform for the evolution of nitrile metabolism-related enzymes based on a desensitized repressive biosensor[J].ACS Synthetic Biology, 2022, 11(4):1577-1587.
[48] XU P, LI L Y, ZHANG F M, et al.Improving fatty acids production by engineering dynamic pathway regulation and metabolic control[J].Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(31):11299-11304.
[49] LO T M, CHNG S H, TEO W S, et al.A two-layer gene circuit for decoupling cell growth from metabolite production[J].Cell Systems, 2016, 3 (2):133-143.
[50] WU S B, XUE Y T, YANG S J, et al.Combinational quorum sensing devices for dynamic control in cross-feeding cocultivation[J].Metabolic Engineering, 2021, 67:186-197.
[51] LIU D, SICA M S, MAO J W, et al.A p-Coumaroyl-CoA biosensor for dynamic regulation of naringenin biosynthesis in Saccharomyces cerevisiae[J].ACS Synthetic Biology, 2022, 11 (10):3228-3238.
[52] ZHANG J, WANG Z G, SU T Y, et al.Tuning the binding affinity of heme-responsive biosensor for precise and dynamic pathway regulation[J].iScience, 2020, 23 (5):101067.