L-高丝氨酸是一种重要的非蛋白质氨基酸,具有重要的生理功能,在食品、饲料和化工领域具有重要的应用[1]。在细胞中,L-高丝氨酸是合成L-苏氨酸、L-蛋氨酸等天冬氨酸家族氨基酸的重要前体,影响细胞天冬氨酸家族氨基酸的合成效率。在化工领域,L-高丝氨酸是一种重要的平台化学品,是生产1,3丙二醇、丙烯酸和草铵膦等重要化学品的中间原料。近年来,利用环境友好的微生物发酵法生产L-高丝氨酸越来越受到关注。在大肠杆菌和谷氨酸棒杆菌中L-高丝氨酸的合成途径已经阐明[2-3],天冬氨酸在天冬氨酸激酶(aspartate kinase, AK)的作用下生成天冬氨酸磷酸,然后在天冬氨酸半醛脱氢酶的催化下生成L-天冬氨酸-半醛(L-aspartate-4-semialdehyde,L-ASA),最后由高丝氨酸脱氢酶催化生成L-高丝氨酸。研究表明,天冬氨酸激酶和高丝氨酸脱氢酶是L-高丝氨酸合成途径的关键酶[4],受到L-苏氨酸、L-赖氨酸的反馈抑制作用。其中高丝氨酸脱氢酶位于代谢途径的关键节点,与L-赖氨酸合成途径竞争调控关键前体天冬氨酸半醛,其活性受到L-苏氨酸等的反馈抑制,是影响L-高丝氨酸生物合成的重要靶点。因此,挖掘和改造具有高效催化活性和反馈抑制不敏感的高丝氨酸脱氢酶,对高效发酵生产L-高丝氨酸等天冬氨酸家族氨基酸具有重要意义。
高丝氨酸脱氢酶是一种NAD(P)H依赖型氧化还原酶,催化L-ASA生成L-高丝氨酸的可逆反应,在植物和微生物中广泛存在。根据结构和功能差异高丝氨酸脱氢酶可分为两个家族,即双功能的天冬氨酸激酶-高丝氨酸脱氢酶(AK-homoserine dehydrogenase,AK-HSD)和单功能的高丝氨酸脱氢酶。双功能的天冬氨酸激酶-高丝氨酸脱氢酶通常采用四聚体排列,包含AK和HSD 2个催化结构域和一个调节结构域,例如大肠杆菌(Escherichia coli)和拟南芥(Arabidopsis thaliana)中的高丝氨酸脱氢酶(AK-HSDI和AK-HSDⅡ)。单功能的高丝氨酸脱氢酶通常以二聚体形式存在,根据结构又可分为结构亚群,即包含激活(activation,ACT)结构域的单功能高丝氨酸脱氢酶,该类酶受到L-苏氨酸的反馈抑制调节,如谷氨酸棒杆菌(Corynebacterium glutamicum)中的高丝氨酸脱氢酶(CgHSD),相反缺乏ACT结构域的高丝氨酸脱氢酶则表现出反馈抑制不敏感的特性[5-6]。近年来,各国学者在挖掘和改造高丝氨酸脱氢酶的研究上取得了一定的进展。TANG等[7]在淋病奈瑟菌(Neisseria gonorrhoeae)中鉴定了一个新的NADH依赖型高丝氨酸脱氢酶,该酶包含ACT结构域,并受到L-苏氨酸轻微的反馈抑制,随后通过对辅因子NADH结合位点氨基酸定点突变,改变了该酶的辅因子偏好性。国内学者申术霞等[8]通过对北京棒杆菌AS1.299中的高丝氨酸脱氢酶进行定向进化,获得的突变体L200F/D215K酶活性提高了1.24倍,热稳定性提高了1.12倍。REINSCHEID等[9]研究发现将CgHSD的第378位甘氨酸突变成谷氨酸能有效解除CgHSD的反馈抑制。尽管如此,目前报道的高丝氨酸脱氢酶活性仍然较低,不利于L-高丝氨酸等氨基酸的高效合成。因此,本项目通过数据库比对检索,挖掘测试不同来源的高丝氨酸脱氢酶,鉴定了一个具有较高催化活性且不受反馈抑制的高丝氨酸脱氢酶,在此基础上利用蛋白质工程技术对该酶进行定向进化,进一步提升了高丝氨酸脱氢酶的催化活性,提高了L-高丝氨酸的合成能力。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株和质粒
大肠杆菌(Escherichia coli)DH5α、BL21(DE3)、谷氨酸棒杆菌(ATCC13032),中国科学院天津工业生物技术研究所保藏。
大肠杆菌表达质粒pET21b由本实验室保藏。谷氨酸棒杆菌表达质粒pEC-XK99E由本实验室保藏。研究所用菌株与质粒见表1与表2。
表1 本研究所用菌株
Table 1 The strains used in this study
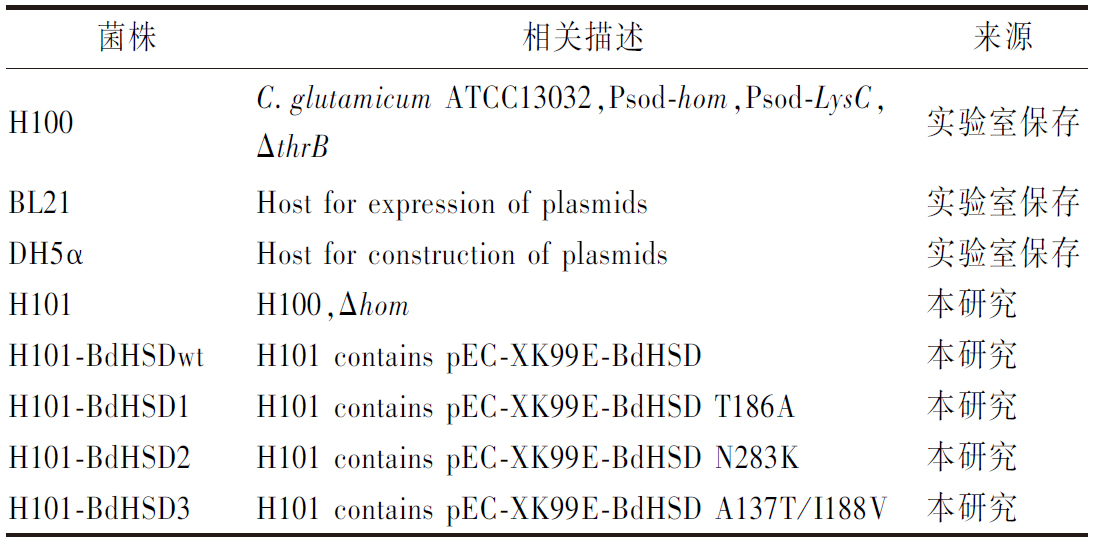
菌株相关描述来源H100C.glutamicum ATCC13032,Psod-hom,Psod-LysC,ΔthrB实验室保存BL21Host for expression of plasmids实验室保存DH5αHost for construction of plasmids实验室保存H101H100,Δhom本研究 H101-BdHSDwtH101 contains pEC-XK99E-BdHSD 本研究 H101-BdHSD1H101 contains pEC-XK99E-BdHSD T186A本研究 H101-BdHSD2H101 contains pEC-XK99E-BdHSD N283K本研究 H101-BdHSD3H101 contains pEC-XK99E-BdHSD A137T/I188V本研究
注:BdHSD为二穗短柄草(Brachypodium distachyon)来源的高丝氨酸脱氢酶。
表2 本研究所用质粒
Table 2 The plasmids used in this study

质粒相关描述来源pET21b-BdHSDpET21b contains hom gene from Brachypodium distachyon本研究pET21b-CgHSDpET21b contains hom gene from C.glutamicum本研究pET21b-ScHSDpET21b contains hom gene from Saccharomyces cerevisiae本研究pET21b-NgHSDpET21b contains hom gene from N.gonorrhoeae本研究pET21b-ZmHSDpET21b contains hom gene from Zea mays本研究pET21b-BsHSDpET21b contains hom gene from Bacillus subtilis本研究pET21b-EcHSDpET21b contains hom gene from E.coli本研究pET21b-TdHSDpET21b contains hom gene from Thiobacillus denitrificans本研究pEC-XK99E-WTpEC-XK99E contains hom gene本研究pEC-XK99E-T186ApEC-XK99E contains homT186Agene本研究pEC-XK99E-N283KpEC-XK99E contains homN283K gene本研究pEC-XK99E-A137T/I188VpEC-XK99E contains homA137T/I188V gene本研究pCRD206-ΔhompCRD206 contains upstream and downstream homology arms本研究
注:Saccharomyces cerevisiae为酿酒酵母;Zea mays为玉米;Bacillus subtilis为枯草芽孢杆菌;Thiobacillus denitrificans为脱氮硫杆菌。
1.1.2 试剂
无缝克隆酶,苏州宇恒生物科技有限公司;Dpn I消化酶,北京擎科生物公司;磷酸氢二钠、盐酸、氯化钠(均为国产分析纯);L-高丝氨酸、烟酰胺腺嘌呤二核苷磷酸(NADP+)和烟酰胺腺嘌呤二核苷酸(NAD+),上海源叶生物试剂有限公司;苏氨酸、赖氨酸、柠檬酸亚铁、维生素B1、烟酰胺、硫酸铵和异丙基-β-D-硫代半乳吡喃糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG),索莱宝生物科技有限公司;0.1% BSA,NEB公司;限制性内切酶FastDigest Eco31I、T4 DNA连接酶、蛋白Marker、Phusion DNA聚合酶,Thermo Fisher公司;质粒提取试剂盒、胶回收试剂盒,天根生物公司。
1.1.3 培养基
LB培养基(g/L):酵母粉5.0,氯化钠10.0,胰蛋白胨10.0(固体培养基加2%的琼脂粉)。
LBHIS培养基(g/L):酵母粉2.5,氯化钠5.0,胰蛋白胨5.0,山梨醇91,脑心浸液18.75(固体培养基加2%的琼脂粉)。
种子培养基(g/L):玉米浆30,尿素5,硫酸镁 0.5,硫酸铵5,磷酸二氢钾0.25,葡萄糖25,苏氨酸0.4。
发酵培养基(g/L):玉米浆70,FeSO4-柠檬酸盐溶液0.55 mL/L(FeSO4·7H2O 20 g/L,柠檬酸18.14 g/L),硫酸铵35,硫酸镁0.5,85%磷酸0.225 mL/L,磷酸二氢钾0.25,一水葡萄糖50,维生素溶液7 mL/L(含生物素300 mg/L,维生素B1 500 mg/L,泛酸钙盐2 g/L,烟酰胺600 mg/L),尿素5,苏氨酸0.4。
1.2 仪器与设备
Synergy NEO2多功能酶标仪,美国Biotek;恒温摇床,美国精骐;Scientz-IID超声破碎仪,宁波新芝公司;高速冷冻离心机,德国Eppendoff;Agilent 1260高效液相色谱仪,美国Agilent公司。
1.3 实验方法
1.3.1 高丝氨酸脱氢酶重组表达质粒的构建
来源于B.distachyon,Z.mays,N.gonorrhoeae,T.denitrificans的高丝氨酸脱氢酶基因片段由金唯智合成。来源于E.coli,C.glutamicum,B.subtilis和S.cerevisiae的高丝氨酸脱氢酶基因均以各基因组为模板,通过PCR方法获得。将获得的基因通过Golden gate的方式连接至pET21b骨架上,构建不同高丝氨酸脱氢酶重组表达质粒。将构建的质粒转化到E.coli BL21感受态细胞中。
1.3.2 高丝氨酸脱氢酶序列分析
本研究从NCBI和BRENDA数据库中挖掘了不同来源的高丝氨酸脱氢酶。使用在线软件InterPro分析预测了高丝氨酸脱氢酶的结构域和所属家族。使用在线软件ClustalW和ESpript 3.0对高丝氨酸脱氢酶进行蛋白序列比对。蛋白进化树分析使用软件MEGA7.0完成。
1.3.3 高丝氨酸脱氢酶诱导表达和纯化
将构建完成的质粒转化到E.coli BL21感受态细胞中,涂布于含有100 μg/mL Amp的LB固体平板上,37 ℃过夜培养,待长出单菌落接种至含100 μg/mL氨苄青霉素(ampicillin,Amp)的4 mL的LB培养基的试管中过夜培养,将过夜培养的重组菌按照1%接种量接种含有100 μg/mL Amp的LB液体培养基中,37 ℃、200 r/min培养至菌液OD600=0.6,冰浴10 min,加入终浓度为0.4 mmol/L的IPTG,16 ℃、200 r/min诱导培养16~20 h,4 ℃、6 000 r/min离心收集菌体。加入适量裂解缓冲液(20 mmol/L Na2HPO4,200 mmol/L NaCl,pH 7.5),冰水浴超声破碎处理20 min,随后在4 ℃、6 000 r/min条件下离心10 min,收集上清液即为粗酶液。采用镍柱亲和层析的方法对高丝氨酸脱氢酶进行纯化,用保存缓冲液(20 mmol/L Na2HPO4溶液,200 mmol/L NaCl,5%甘油,pH值调至7.5)进行超滤,分装保存于-80 ℃冰箱中。用十二烷基硫酸钠-聚丙烯酰胺(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE)方法检测粗酶液和纯化的高丝氨酸脱氢酶,并用Bradford方法测定蛋白质含量。
1.3.4 高丝氨酸脱氢酶酶活测定方法
高丝氨酸脱氢酶酶活是通过检测340 nm处吸光度的变化测定的[6],摩尔消光系数为6 220/(mol·cm)。反应体系为100 mmol/L Tris缓冲液(pH 7.5),80 mmol/L L-高丝氨酸,400 mmol/L NaCl和1 mmol/L NAD(P)+。高丝氨酸脱氢酶的酶活单位定义为单位时间内每生成1 μmol NAD(P)H所消耗的酶量。
1.3.5 高丝氨酸脱氢酶筛选
采用易错PCR技术[10]构建BdHSD突变体文库。通过调整Mg2+和Mn2+浓度将突变率控制到每个kbp片段有1~3个突变。易错PCR反应体系包括(50 μL):10×buffer 5 μL,25 mmol/L Mg2+ 3.5 μL,1 mmol/L Mn2+ 3 μL,2.5 mmol/L脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)4 μL,二甲基亚砜(dimethyl sulfoxide,DMSO)2.5 μL,Primers 2 μL,Template 1 μL,TaKaRa rTaq 0.5 μL,ddH2O 26.5 μL。
将获得的菌落挑取至96孔板中,37 ℃过夜培养后转接到新的含有LB培养基的96孔板中,加入0.4 mmol/L IPTG,在16 ℃,200 r/min条件下诱导16~20 h。离心收集菌体,加入裂解缓冲液(含有3 mg/mL的溶菌酶)重悬起来,37 ℃孵育3 h后,离心收集上清获得粗酶液。通过酶活性测定分析,选择具有较高催化能力的酶突变体。
1.3.6 BdHSD及其突变体对谷氨酸棒杆菌中L-高丝氨酸合成的影响
谷氨酸棒杆菌H100是本实验室保藏的一株高丝氨酸生产菌,为消除自身高丝氨酸脱氢酶的干扰,敲除掉H100中CgHSD的编码基因hom。采用Phusion DNA聚合酶,以C.glutamicum基因组为模板,通过PCR方法获得CgHSD的上下游同源区域,利用无缝克隆试剂盒的方法连接至敲除质粒pCRD206上,构建pCRD206-Δhom。将构建的敲除质粒电转化进入H100感受态细胞中,按照同源重组方法对H100中hom基因进行敲除,构建底盘菌株H101。
为探究BdHSD及其突变体对谷氨酸棒杆菌中L-高丝氨酸合成的影响,利用Phusion DNA聚合酶PCR扩增野生型BdHSD及其突变体基因片段,利用无缝克隆试剂盒标准方法,分别连接至pEC-XK99E质粒骨架上,构建高丝氨酸脱氢酶表达质粒pEC-XK99E-WT、pEC-XK99E-T186A、pEC-XK99E-N283K和pEC-XK99E-A137T/I188V。随后将上述表达质粒分别电转化至底盘菌株H101中,构建高丝氨酸生产菌株H101-BdHSDwt,H101-BdHSD1,H101-BdHSD2,H101-BdHSD3。
1.3.7 L-高丝氨酸生产菌株摇瓶发酵研究
本研究采用的谷氨酸棒杆菌L-高丝氨酸发酵方法按照文献报道的方法进行[11]。挑取上述构建的L-高丝氨酸合成菌株,置于含有25 μg/mL Kanamycin的LBHIS液体培养基中,30 ℃过夜活化培养。按照1%的接种量,接种到含有Kanamycin的种子培养基中,30 ℃,200 r/min条件下培养14 h至对数中期。然后按照初始OD600=1接种到含有25 μg/mL Kanamycin的发酵培养基中,30 ℃,200 r/min培养4 h后,加入0.2 mmol/L IPTG,继续发酵36 h,使用分光光度计测定菌株的生长情况OD600,使用HPLC测定发酵液中L-高丝氨酸的含量[11]。
2 结果与分析
2.1 HSD的挖掘与序列分析
高丝氨酸脱氢酶是L-高丝氨酸和L-苏氨酸等氨基酸生物合成的关键酶,其活性受到L-苏氨酸等的反馈抑制[12]。因此,挖掘鉴定具有高效催化活性并且不受反馈抑制的高丝氨酸脱氢酶对实现L-高丝氨酸的高效生产尤为重要。因此,本研究根据文献调研和数据库比对,选择了8个不同来源的高丝氨酸脱氢酶,分别为来源于E.coli的EcHSD、C.glutamicum的CgHSD、B.subtilis的BsHSD、S.cerevisiae的ScHSD、B.distachyon的BdHSD,Z.mays的ZmHSD,N.gonorrhoeae的NgHSD,T.denitrificans的TdHSD。这8种高丝氨酸脱氢酶在各自最适条件下的催化性能见表3。
表3 不同来源高丝氨酸脱氢酶性能分析
Table 3 Performance analysis of homoserine dehydrogenase from different sources

来源底物酶活性/(U/mg)Km/(mmol/L)底物抑制参考文献Z.maysL-天冬氨酸半醛3600.15-[13]S.cerevisiaeL-天冬氨酸半醛570.186-[14]E.coliL-天冬氨酸半醛-0.17Thr、Lys、Met[15]C.glutamicumL-天冬氨酸半醛990.94Thr、Lys、Met[12]B.distachyonL-高丝氨酸61~70--本研究N.gonorrhoeaeL-高丝氨酸18--[7]T.denitrificansL-高丝氨酸----B.subtilisL-高丝氨酸1.48~2.01Thr、Lys、Met[16]
注:表中“-”表示未有报道。
随后本研究利用Interpro在线分析软件对上述挖掘的HSD蛋白序列进行分析,鉴定其蛋白家族和包含的蛋白结构域。结果表明,除了EcHSD属于双功能的高丝氨酸脱氢酶外,其余7个蛋白均属于单功能的高丝氨酸脱氢酶,其中CgHSD、TdHSD和NgHSD包含有ACT结构域,而ScHSD、ZmHSD和BdHSD不包含有ACT结构域。为了更为细致的鉴定和比较高丝氨酸脱氢酶的保守序列,本研究额外添加了4个双功能的高丝氨酸脱氢酶进行序列比对分析,分别为来源于A.thaliana的AtHSD,来源于大豆(Glycine max)的高丝氨酸脱氢酶GmHSD,来源于水稻白叶枯病菌(Xanthomonas oryzae)的高丝氨酸脱氢酶XoHSD和来源于产气克雷伯氏菌(Klebsiella aerogenes)的高丝氨酸脱氢酶KaHSD。
结果表明,高丝氨酸脱氢酶N端的保守基序GXGXXG对辅因子NAD(P)H的结合十分重要,而在150~240处酶活性中心区域的氨基酸具有较高的保守性(图1)。进化树分析结果表明,ScHSD、ZmHSD和BdHSD 3个蛋白具有更亲缘的进化关系,而CgHSD、TdHSD和NgHSD 3个蛋白具有更亲缘的进化关系(图2),该结果与结构域分析结果基本一致。

图1 不同来源高丝氨酸脱氢酶的序列比对分析
Fig.1 Sequence alignment analysis of homoserine dehydrogenase from different sources
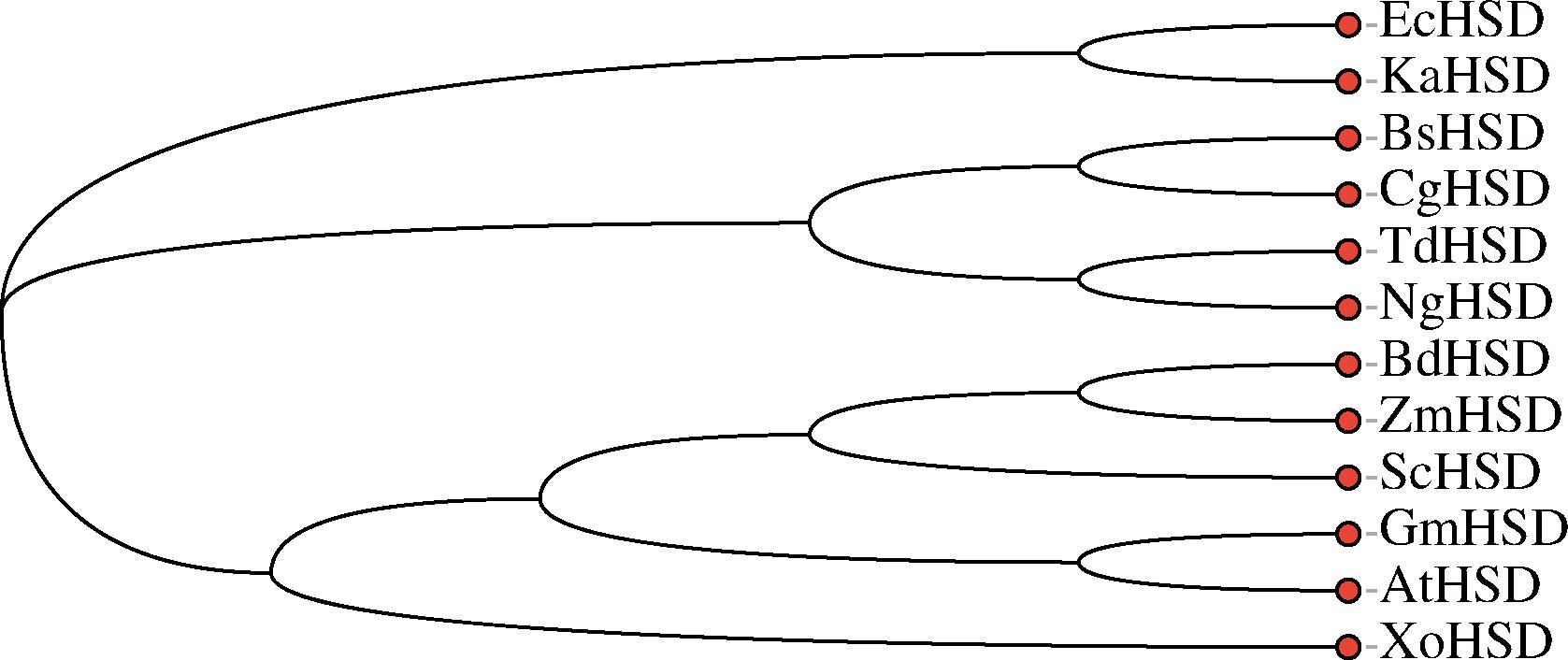
图2 不同来源高丝氨酸脱氢酶的系统进化树分析
Fig.2 Phylogenetic tree analysis of homoserine dehydrogenase from different sources
2.2 高丝氨酸脱氢酶的表达纯化与催化活性分析
BdHSD、ZmHSD、NgHSD、TdHSD基因由金斯瑞生物科技有限公司进行密码子优化和基因合成。EcHSD、CgHSD、BsHSD和ScHSD基因均以各种群的基因组为模板,采用PCR扩增方法获得。采用Golden Gate方法将上述基因分别克隆至pET21b表达载体上,构建表达质粒并转化至E.coli BL21(DE3)菌株中。在37 ℃条件下培养至OD600=0.6左右,加入0.4 mmol/L IPTG在16 ℃条件下继续诱导20 h,离心收集菌体。采用镍柱亲和层析方法分别对上述8个高丝氨酸脱氢酶进行蛋白纯化,SDS-PAGE分析结果表明,纯化后HSD蛋白条带单一,蛋白分子质量普遍在35~47 kDa,而来源于大肠杆菌的双功能高丝氨酸脱氢酶分子质量约为85 kDa(图3-A),与预期结果基本一致。
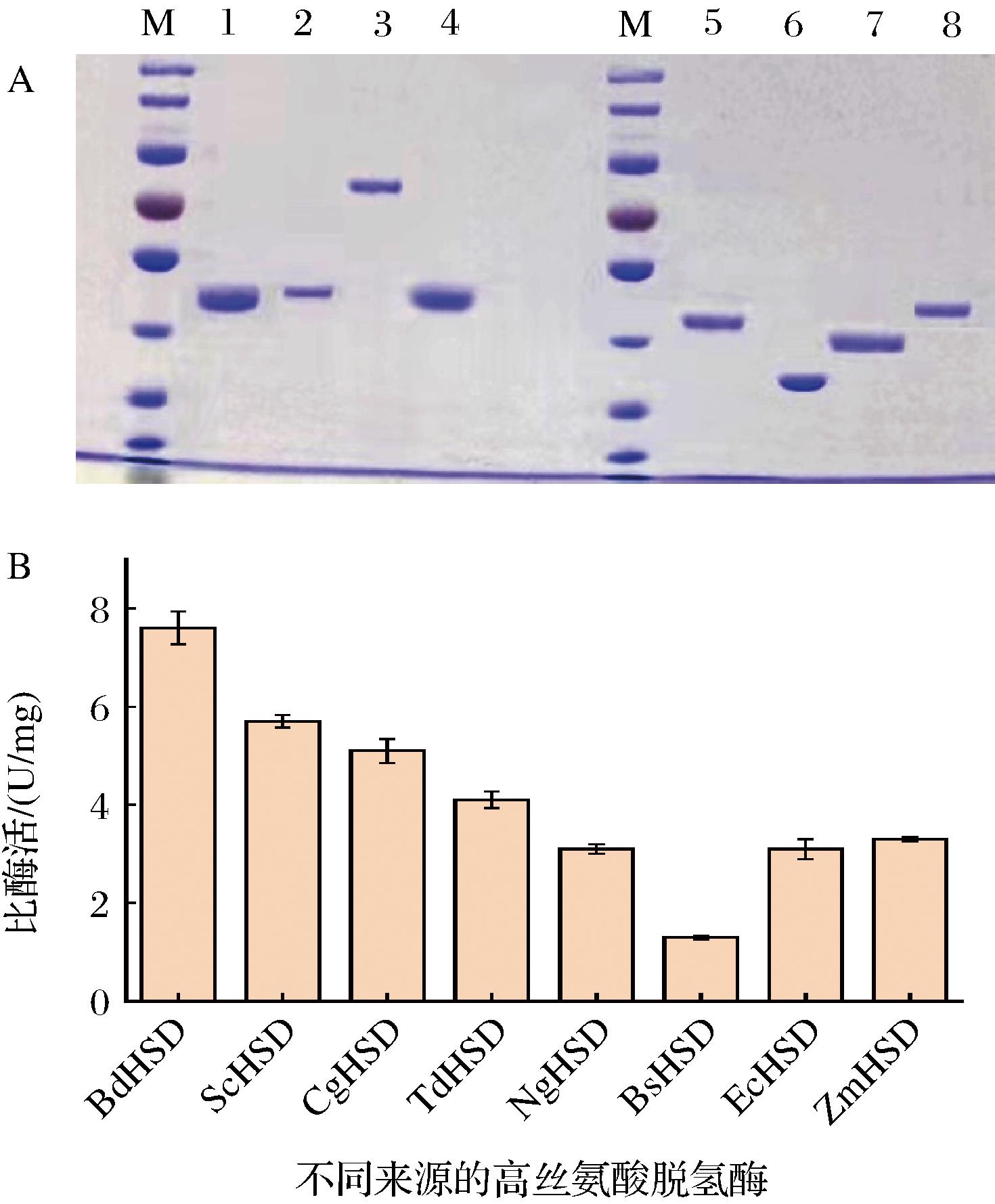
A-不同来源的HSD蛋白表达和SDS-PAGE分析(1~8分别代表CgHSD、BsHSD、EcHSD、ZmHSD、NgHSD、ScHSD、BdHSD、TdHSD);B-不同来源的HSD的酶活性测定
图3 不同来源的HSD的酶活性比较分析
Fig.3 Comparative analysis of enzyme activity of HSD from different sources
随后本研究分别对纯化的8个高丝氨酸脱氢酶进行酶活比较分析。如图3-B所示,在37 ℃,pH=7.5条件下,BdHSD表现出最高的催化活性,比酶活达到7.6 U/mg,显著高于其他高丝氨酸脱氢酶的酶活性。值得注意的是,NgHSD和TdHSD的辅因子偏好性为NAD+,比酶活达到4.1 U/mg和3.1 U/mg,而其余的高丝氨酸脱氢酶的辅因子偏好性为NADP+。多辅因子偏好性的研究为解析高丝氨酸脱氢酶催化调控机制提供了重要的研究基础和依据。
2.3 BdHSD的催化特性研究
根据上述研究结果,BdHSD具有最高的酶催化活性,并且不包含ACT结构域,从而避免了L-苏氨酸的反馈抑制作用,是理想的高丝氨酸脱氢酶候选蛋白。因此,本研究进一步探究了BdHSD的催化特性,探究了其催化最适pH值和最适温度。本研究测定了BdHSD在不同pH下的酶活性,如图4-A所示,BdHSD及其突变体最适pH为10.5。在pH 5.0~7.0范围内,BdHSD及其突变体酶催化活性较低,而在pH 8.5~11.0,BdHSD具有较好的催化能力,说明BdHSD在碱性条件下表现出较高的高丝氨酸氧化活性,与文献报道的NgHSD的结果基本一致,高于BsHSD[7,16]。通过在不同温度下测定BdHSD的酶活性发现,BdHSD最适催化温度为38 ℃(图4-B),并具有较好的温度稳定性。
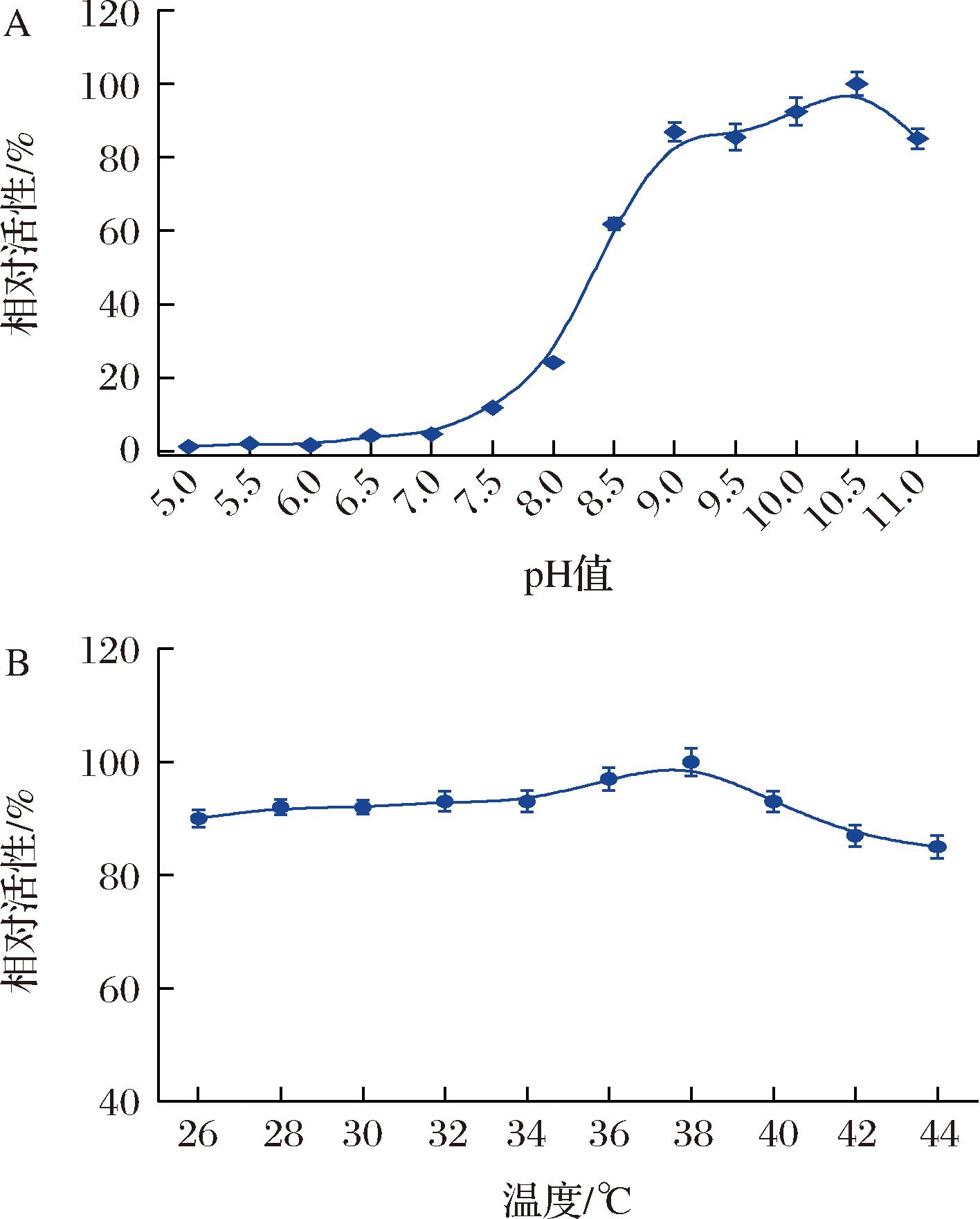
A-pH值对BdHSD酶活性的影响;B-温度对BdHSD酶活性的影响
图4 BdHSD的催化特性研究
Fig.4 Catalytic characteristics of BdHSD
2.4 BdHSD的定向进化和高通量筛选
本研究为进一步提高BdHSD的催化活性,对BdHSD进行定向进化。采用易错PCR策略构建突变体文库。利用低保真性rTaq聚合酶扩增BdHSD基因片段,通过调整Mg2+和Mn2+浓度将突变率控制到每kbp片段有1~3个突变。将获得的基因片段通过Golden Gate的方法整合至pET21b表达质粒上,构建BdHSD突变体文库,并转化至E.coli BL21(DE3)中。挑取单菌落至96孔板中37 ℃培养过夜,转接至新的LB培养基中加入0.4 mmol/L IPTG,16 ℃诱导16~20 h。离心后加入含溶菌酶的裂解缓冲液孵育3 h后,离心收集上清液获得粗酶液。通过测定反应体系中NADPH含量变化速率对酶突变体进行高通量评价和筛选。通过多轮筛选,共获得了10个粗酶活显著提升的突变体(图5-A)。随后对10个突变体进行蛋白纯化和酶活性测定,结果表明mut-5、mut-8和mut-10 表现出较高的酶活性,比酶活达到10.3、9.7、9.6 U/mg,分别比野生型提高了35.5%,27.6%,26.3%(图5-B)。
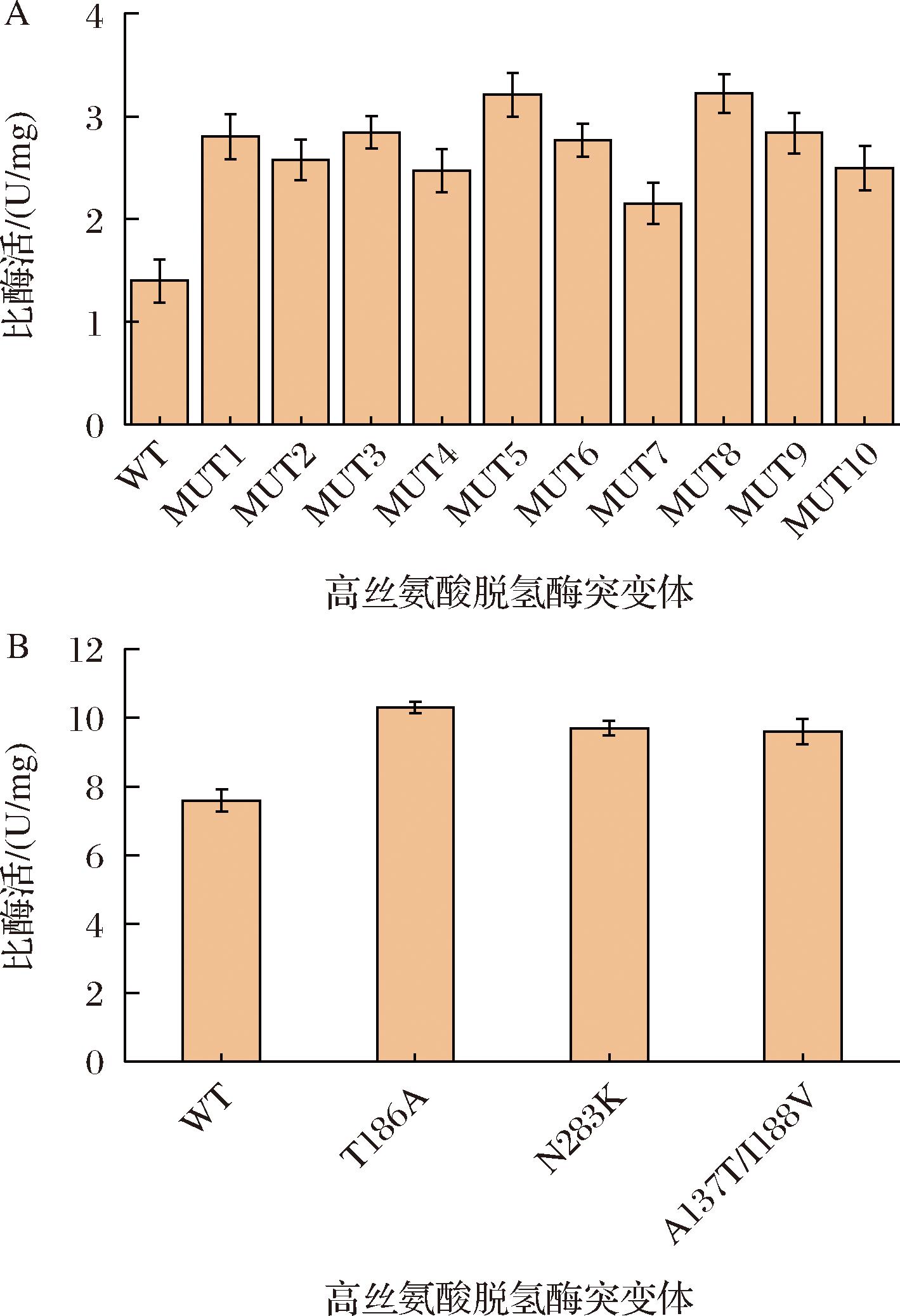
A-BdHSD及其突变体粗酶液酶活性测定;B-BdHSD及其突变体蛋白纯化与酶活性测定
图5 BdHSD及其突变体的催化活性分析
Fig.5 Analysis of catalytic activity of BdHSD and its mutants
经测序mut-5、mut-8和mut-10突变分别为T186A、N283K和A137T/I188V。为进一步探究BdHSD及其突变体对蛋白结构的影响,本研究对BdHSD及其突变体进行蛋白结构分析。利用AlphaFold在线分析软件预测BdHSD及其突变体的三维结构,通过结构分析发现,如图6-A所示,186位苏氨酸突变为疏水的丙氨酸后,与周围的疏水环境更为匹配,有利于该区域的结构稳定。N283周围有几个可以成盐桥的残基,2个电负性的E287,D284,一个电正性的K227,当283位氨基酸突变成K后,能与D284形成盐桥,从而增强该区域的稳定(图6-B)。如图6-C所示,A137所在的位置是一个疏水性的区域,但是由于A比较小,造成这个区域存在空隙,可以被水分子深入,从而造成失稳,当A283突变为T后,填补了该部分的空隙。有趣的是,I188所在的位置也是一个疏水中心,但是当I188突变为V后,形成了间隙,反而不利于催化反应的进行。因此,我们推测A137T/I188V双突变对酶活的提升可能主要是由A137T贡献的。
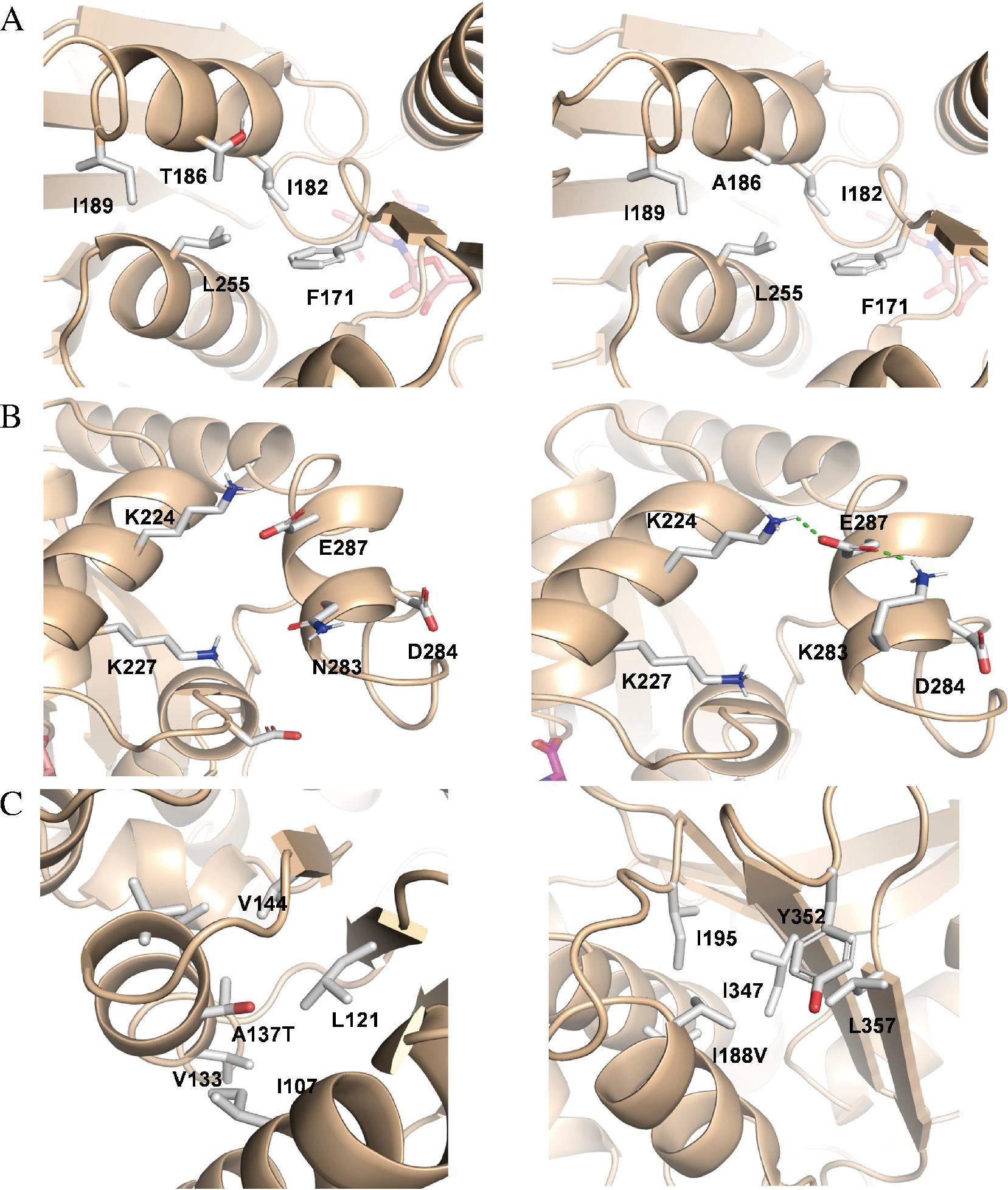
A-T186A突变对BdHSD结构的影响;B-N283K突变对BdHSD结构的影响;C-A137T/I188V双突变对BdHSD结构的影响
图6 BdHSD及其突变体蛋白结构分析
Fig.6 Protein structure analysis diagram
2.5 探究BdHSD过表达对谷氨酸棒杆菌高丝氨酸合成的影响
本研究为探究BdHSD对L-高丝氨酸合成能力的影响,将BdHSD及其突变体mut-5、mut-8和mut-10分别连接至表达载体pEC-XK99E上,构建表达质粒pEC-XK99E-WT、pEC-XK99E-T186A、pEC-XK99E-N283K和pEC-XK99E-A137T/I188V。随后将上述表达质粒分别电转化至谷氨酸棒杆菌合成菌株H101中,构建高丝氨酸生产菌株H101-BdHSDwt、H101-BdHSD1、H101-BdHSD2、H101-BdHSD3。
按照1.3.6节所述方法,对构建的L-高丝氨酸合成菌株进行摇瓶发酵测试,结果如图7所示。表达BdHSD突变体能有效提升菌株、L-高丝氨酸的合成能力,其中含有BdHSD T186A突变体的重组菌株H101-BdHSD1的L-高丝氨酸产量最高,达到3.2 g/L,比对照菌株H101-BdHSDwt的L-高丝氨酸含量提高了28%,且野生型和突变体菌株生长情况基本一致。结果表明,高丝氨酸脱氢酶的催化活性是影响L-高丝氨酸合成的关键因素,提升高丝氨酸脱氢酶活性能显著提升L-高丝氨酸的合成水平。本研究通过对高丝氨酸脱氢酶的挖掘和改造,获得催化活性高且不受反馈抑制的BdHSD及其突变体,为L-高丝氨酸等氨基酸的高效生物合成提供了有效的催化元件。
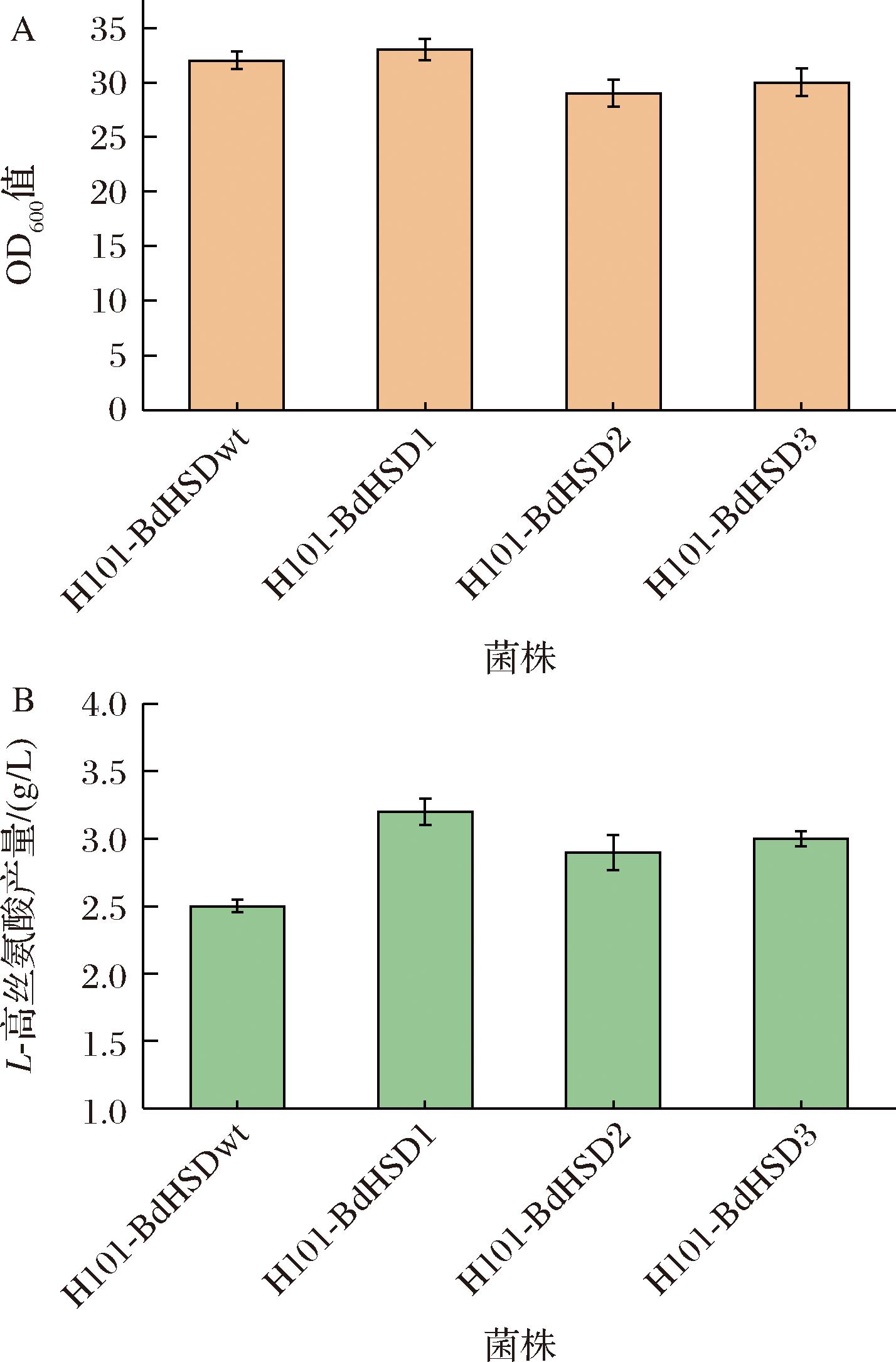
A-高丝氨酸合成菌株的生长情况;B-工程菌株L-高丝氨酸的产量
图7 BdHSD及其突变体对谷氨酸棒杆菌L-高丝氨酸合成的影响
Fig.7 Effects of BdHSD and its mutants on L-homoserine synthesis by Corynebacterium glutamate
3 结论
高丝氨酸脱氢酶是L-高丝氨酸、L-苏氨酸等天冬氨酸家族氨基酸生物合成途径的关键酶。本研究通过数据库比对检索,挖掘了8个不同来源的高丝氨酸脱氢酶。通过酶活性比对分析,发现BdHSD具有更高的催化活性,并且不受L-苏氨酸和L-赖氨酸的反馈抑制,是极具潜力的高丝氨酸脱氢酶候选蛋白。通过对BdHSD催化特性分析发现,其催化最适pH值为10.5,最适催化温度为38 ℃。为进一步提高BdHSD的催化活性,本研究对其进行定向进化和高通量筛选,获得了3个具有更高催化活性的BdHSD突变体T186A、N283K、A137T/I188V。其中突变体T186A酶活性比野生型提高了1.35倍,并且过表达突变体T186A能有效提升谷氨酸棒杆菌L-高丝氨酸的合成水平。相关研究成果为进一步探究高丝氨酸脱氢酶的催化机制奠定了重要的研究基础,为L-高丝氨酸、L-苏氨酸和L-蛋氨酸等天冬氨酸家族氨基酸的高效生物合成提供了有力的催化元件。
[1] MU Q X, ZHANG S S, MAO X J, et al.Highly efficient production of L-homoserine in Escherichia coli by engineering a redox balance route[J].Metabolic Engineering, 2021, 67:321-329.
[2] CAI M M, ZHAO Z Q, LI X F, et al.Development of a nonauxotrophic L-homoserine hyperproducer in Escherichia coli by systems metabolic engineering[J].Metabolic Engineering, 2022, 73:270-279.
[3] LI N, LI L H, YU S Q, et al.Dual-channel glycolysis balances cofactor supply for L-homoserine biosynthesis in Corynebacterium glutamicum[J].Bioresource Technology, 2023, 369:128473.
[4] SUN B Y, WANG F Q, ZHAO J, et al.Engineering Escherichia coli for L-homoserine production[J].Journal of Basic Microbiology, 2023, 63(2):168-178.
[5] SCHROEDER A C, ZHU C M, YANAMADALA S R, et al.Threonine-insensitive homoserine dehydrogenase from soybean:Genomic organization,kinetic mechanism, and in vivo activity[J].The Journal of Biological Chemistry, 2010, 285(2):827-834.
[6] LIANG X X, DENG H X, BAI Y J, et al.Characterization of a novel type homoserine dehydrogenase with high oxidation activity from Arthrobacter nicotinovorans[J].Process Biochemistry, 2022, 114:102-110.
[7] TANG W G, DONG X, MENG J, et al.Biochemical characterization and redesign of the coenzyme specificity of a novel monofunctional NAD+-dependent homoserine dehydrogenase from the human pathogen Neisseria gonorrhoeae[J].Protein Expression and Purification, 2021, 186:105909.
[8] 申术霞, 朱运明, 闵伟红, 等.北京棒杆菌AS1.299高丝氨酸脱氢酶突变体L200F/D215K的异源表达及酶学性质[J].微生物学报, 2014, 54(10):1178-1184.
SHEN S X, ZHU Y M, MIN W H, et al.Heterologous expression and characterization of L200F/D215K mutant of homoserine dehydrogenase from Corynebacterium pekinense AS1.299[J].Acta Microbiologica Sinica, 2014, 54(10):1178-1184.
[9] REINSCHEID D J, EIKMANNS B J, SAHM H.Analysis of a Corynebacterium glutamicum hom gene coding for a feedback-resistant homoserine dehydrogenase[J].Journal of Bacteriology, 1991, 173(10):3228-3230.
[10] 郑婉, 刘振杨, 郑金珠, 等.易错PCR技术改造β-环糊精葡萄糖基转移酶的催化特性[J].现代食品科技, 2023, 39(5):25-31.
ZHENG W, LIU Z Y, ZHENG J Z, et al.Modification of the catalytic properties of β-cyclodextrin glucosyltransferase through error-prone PCR[J].Modern Food Science and Technology, 2023, 39(5):25-31.
[11] 赵金花, 魏亮, 徐宁, 等.双酶偶联法测定发酵液中L-高丝氨酸的含量[J].食品与发酵工业, 2023, 49(2):239-245.
ZHAO J H, WEI L, XU N, et al.Determination of L-homoserine concentration in fermentation broth using double-enzyme coupling method[J].Food and Fermentation Industries, 2023, 49(2):239-245.
[12] CHEN Z, RAPPERT S, ZENG A P.Rational design of allosteric regulation of homoserine dehydrogenase by a nonnatural inhibitor L-lysine[J].ACS Synthetic Biology, 2015, 4(2):126-131.
[13] WALTER T J, CONNELLY J A, GENGENBACH B G, et al.Isolation and characterization of two homoserine dehydrogenases from maize suspension cultures[J].The Journal of Biological Chemistry, 1979, 254(4):1349-1355
[14] YUMOTO N, KAWATA Y, NODA S, et al.Rapid purification and characterization of homoserine dehydrogenase from Saccharomyces cerevisiae[J].Archives of Biochemistry and Biophysics, 1991, 285(2):270-275.
[15] WEEDLER F C, LEY B W.Kinetic and regulatory mechanisms for (Escherichia coli) homoserine dehydrogenase-I.Equilibrium isotope exchange kinetics[J].Journal of Biological Chemistry, 1993, 268(7):4880-4888.
[16] KIM D H, NGUYEN Q T, KO G S, et al.Molecular and enzymatic features of homoserine dehydrogenase from Bacillus subtilis[J].Journal of Microbiology and Biotechnology, 2020, 30(12):1905-1911.