ε-聚赖氨酸一般是由25~35个L-赖氨酸单体,在非核糖体肽合成酶催化作用下,以α-COOH和ε-NH2缩合方式形成的一种天然同型氨基酸聚合物。由于具有抑菌谱广、水溶性好、热稳定性强、安全性高等特点,在食品防腐、化妆品保质、饲料替抗等领域具有广泛用途[1]。小白链霉菌(Streptomyces albulus)是ε-聚赖氨酸的主要生产菌[2]。过去几十年,为了提高S.albulus的ε-聚赖氨酸合成能力,诸多理化诱变和抗性筛选策略被广泛用于S.albulus改造,并实现了ε-聚赖氨酸发酵产量的显著提升[3]。然而,随着S.albulus 对不同诱变剂和抗性“筛子”的敏感度降低,传统育种方法提高产生菌的ε-聚赖氨酸合成能力陷入了停滞[4]。近些年,随着基因组学的发展,S.albulus的基因组信息逐渐被解析,使得利用分子生物学手段继续改造S.albulus成为新的选择。例如,XU等[5]通过过表达atmB基因强化S.albulus PD-1的铵离子利用能力,实现ε-聚赖氨酸产量提高了17%;汪泽等[6]通过对ε-聚赖氨酸合成酶基因pls的过表达结合前体流加将S.albulus M-Z18的ε-聚赖氨酸产量提高了20.8%。然而,上述对于ε-聚赖氨酸生产菌株的改造效果还十分有限,甚至不及传统育种效果。究其原因,主要是仅局限在个别基因的过表达,还缺乏在代谢途径层面的支路弱化甚至阻断。然而,由于链霉菌基因组庞大、GC含量高、同源重组困难,菌株生长周期长等因素,导致其基因敲除操作周期长且成功率低[7]。因此,在S.albulus中建立一种高效的基因编辑方法对于利用代谢工程手段改造新一代ε-聚赖氨酸生产菌具有重要价值。
成簇的规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats ,CRISPR)-CRISPR关联(CRISPR associated,CAS)系统是在原核生物中发现的一种特异性防御外来DNA天然免疫系统,通过人为设计可以利用CRIPSR系统对各种细胞的遗传物质进行编辑[8]。近几年,CRISPR-Cas系统被用于链霉菌基因组的编辑。例如,2015年,COBB等[9]率先构建了适用于链霉菌基因编辑的CRISPR-Cas系统,并使用质粒pCRISPRomyces-2分别在变铅青链霉菌、白色链霉菌和绿色产色链霉菌中实现了不同基因的敲除。与此同时,TONG等[10]构建了tipA诱导型启动子启动Cas9的质粒pCRISPR-Cas9,在硫链丝菌素诱导下,使用此质粒成功敲除了天蓝色链霉菌中的actIORF1和actVB基因。他们利用tipA诱导型启动子控制了Cas9的表达强度,降低了Cas9的表达强度,使得链霉菌结合转移效率得以显著提高。为了进一步控制Cas9对链霉菌产生的毒性问题,WANG等[11]于2019年分别使用了诱导型启动子tipA、茶碱诱导的核糖开关、以及蓝光诱导来控制Cas9毒性[11]。随后,该团队又于2021年将蓝光诱导替换为同源抑制因子AcrIIA4,构建出了pTRIA质粒,进一步抑制了Cas9的表达强度,并且保留了足够的编辑效率(65%~90%)[12]。由此可见,基于CRISPR-Cas系统的基因编辑方法是改造链霉菌的一种十分有效手段。然而,由于链霉菌基因组的复杂性,已报道的质粒并不能直接应用于不同种的链霉菌中。例如,YE等[13]的研究发现,在Streptomyces lividans TK24中无法使用质粒pCRISPRomyces-2获得结合子,导致无法实现目标基因的敲除。因此,有必要在S.albulus中建立基于CRISPR-Cas9系统的基因编辑方法。
本文以实验室前期依靠传统育种方法获得的一株工业菌株S.albulus GS114为研究对象,以ε-聚赖氨酸合成酶基因pls为靶基因,尝试构建基于CRISPR-Cas9系统的基因敲除体系,并对影响获得敲除菌株(结合子)数量的结合转移条件进行系统优化,以期开发一种高效编辑S.albulus基因组的CRISPR-Cas9系统,为后续S.albulus的代谢工程改造提供重要工具,也为其他工业链霉菌构建CRISPR-Cas系统提供有价值的参考。
1 材料与方法
1.1 菌株与质粒
本研究所使用的菌株和质粒如表1所示。菌株S.albulus GS114,大肠杆菌(Eschericbia coli) ET12567/pUZ8002和E.coli DH5α由本实验室保藏。
表1 本研究所用菌株和质粒
Table 1 Strains and plasmids used in this study
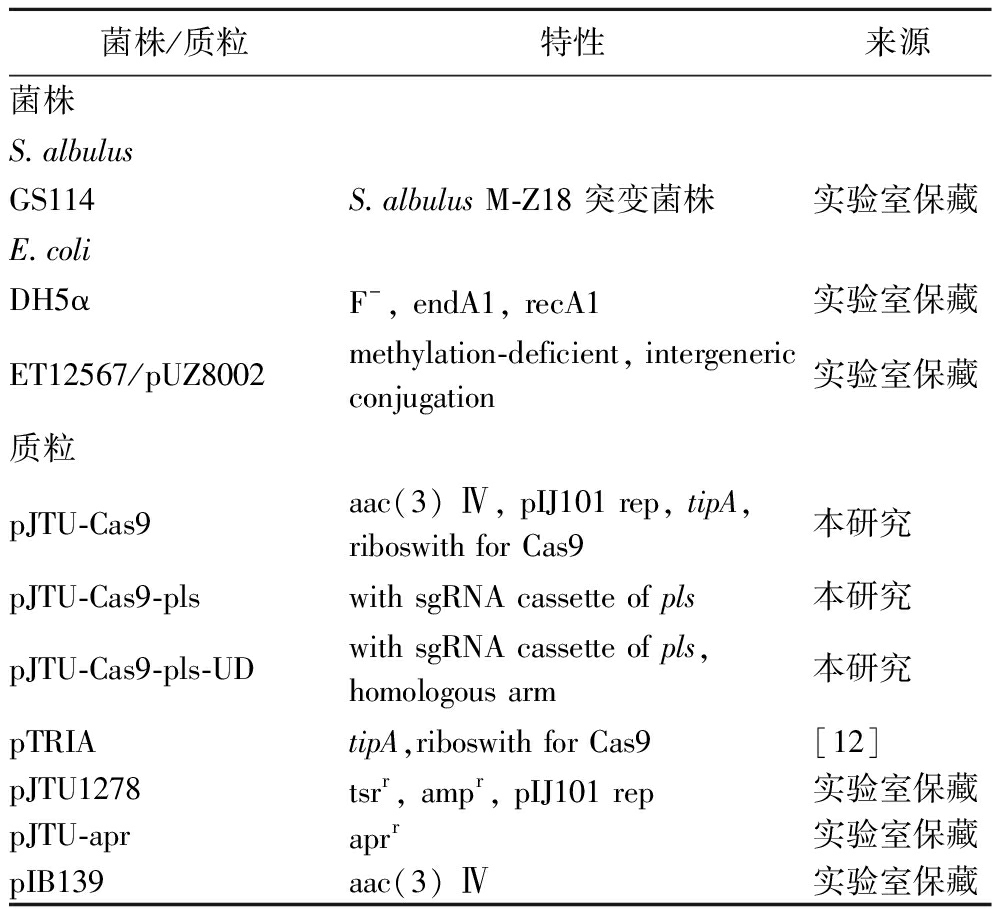
菌株/质粒特性来源菌株S.albulusGS114S.albulus M-Z18 突变菌株实验室保藏E.coliDH5αF-, endA1, recA1实验室保藏ET12567/pUZ8002methylation-deficient, intergeneric conjugation实验室保藏质粒pJTU-Cas9aac(3) Ⅳ, pIJ101 rep, tipA, riboswith for Cas9本研究pJTU-Cas9-plswith sgRNA cassette of pls本研究pJTU-Cas9-pls-UDwith sgRNA cassette of pls, homologous arm本研究pTRIAtipA,riboswith for Cas9[12]pJTU1278tsrr, ampr, pIJ101 rep实验室保藏pJTU-apraprr实验室保藏pIB139aac(3) Ⅳ实验室保藏
1.2 引物
本研究所使用引物及其序列如表2所示。
表2 本研究所用引物
Table 2 Sequence of primers used in this study

引物名称引物序列 (5′-3′)Cas9-FCTATGACATGATTACGAATTCTAATACGACTCACTA-TAGGGGAATTGTGCas9-RGGTTGGTAGGATCCACATATGAACCGAGCGTTCTG-AACAAATCSC-FTCGTGCCGGTTGGTAGGATCCAGCGCTCGAAGGAC-GATTCGAGAGTTTTAGAGCTAGAAATAGCAAGTTA-AAATAASC-RCGCTCTAGAACTAGTGGATCCTAATACGACTCACT-ATAGGGGAATTGTGUHA-FGTTGAATTCGATATCAAGCTTCTTGTCGTACAGCCA-GGTGTGCUHA-RAAGGATCTTCAGCAGAACGGGAAGCTGTACCDHA-FGCGGCTAAAAAAGGGTCTGTTCCGCAATTCGAADHA-RGTCGACGGTATCGATAAGCTTTGAGCATG-AGCCG-CATGTCApr-FCCGTTCTGCTGAAGATCCTTTGATCTTTTCTACGGApr-RACAGACCCTTTTTTAGCCGCTAAAACGGCY-spacer-test-FTAATACGACTCACTATAGGGGAATTGTGY-spacer-test-RATCTTGACGGCTGGCGAGAY-arm-test-FTTCTCTGACGCCGTCCACGY-arm-test-RCAGGGTTTTCCCAGTCACGACY-pls-KO-FCGTTCCTGGAGCAGTGCGY-pls-KO-RCGATCGGGATCACGGTCC
1.3 试剂与仪器
一步克隆试剂盒、PCR预混酶、纯化试剂盒、质粒提取试剂盒及基因组提取试剂盒,诺唯赞生物科技有限公司;快速限制性核酸内切酶(BamHⅠ、EcoRⅠ、HindⅢ),TaKaRa有限公司;抗生素及缓冲液,上海生物工程股份有限公司。
BIOTECH-5BG-7000A PCR仪,Bio-Rad公司;Gel Doc EZ 凝胶成像仪,Bio-Rad公司;DYCP-31DN 电泳仪,北京市六一仪器厂;UV-2100 微量分光光度计,优尼科仪器有限公司;AB204-N 分析天平,瑞士梅特勒公司;GNP-9160恒温培养箱,上海光都仪器设备有限公司;HYL-C 组合式摇床,太仓市强乐设备有限公司。
1.4 培养基和培养条件
本研究所用培养基和培养条件和表3所示。
表3 本研究所用培养基和培养条件
Table 3 Culture media and conditions used in this study

培养基用途培养条件大肠杆菌用培养基LB培养基培养大肠杆菌37 ℃摇床(220 r/min)或者37 ℃培养箱链霉菌用培养基BTN固体培养基培养链霉菌30 ℃培养箱MS固体培养基结合转移的平板30 ℃培养箱YP液体培养基用于链霉菌发酵30 ℃摇床(200 r/min)2×YT液体培养基用于链霉菌孢子预萌发30 ℃摇床(200 r/min)
LB培养基(g/L):氯化钠10.0,胰蛋白胨10.0,酵母粉5.0。
BTN固体培养基(g/L):葡萄糖10.0,鱼粉蛋白胨2.0,酵母提取物1.0,琼脂条20。
MS固体培养基(g/L):D-甘露醇20.0,熟黄豆粉20.0,琼脂条20.0。
YP液体培养基(g/L):葡萄糖60.0,酵母粉10.0,硫酸铵10.0,磷酸二氢钾1.36,磷酸氢二钾0.8,硫酸镁0.5,硫酸锌0.04,硫酸亚铁0.03。
2×YT液体培养基(g/L):胰蛋白胨16.0,酵母粉10.0,氯化钠5.0。
1.5 质粒构建
使用诺唯赞一步克隆试剂盒(C113)连接线性化质粒和片段,将连接产物通过化学转化法导入E.coli DH5α感受态中,涂布于对应抗性的LB固体平板,37 ℃培养过夜。通过菌落PCR获得目标菌株,接种至 5 mL LB液体中培养,添加相应抗生素,37 ℃摇床(220 r/min)培养16~18 h。获得的菌液通过诺唯赞质粒提取试剂盒(DC201)提取质粒。
1.6 结合转移
结合转移参考张重阳等[14]。混合热激后的孢子悬液与含有目的质粒的大肠杆菌菌液并涂布于MS固体平板上,培养一定时间后,覆盖对应抗生素及萘啶酮酸溶液,30 ℃培养4 d。
1.7 发酵方法
发酵方法参考张重阳等[14]论文。使用接种环挑取3环孢子,接种至装有40 mL YP培养基的500 mL锥形瓶中,于200 r/min、30 ℃摇床培养96 h。
1.8 检测方法
HPLC检测ε-聚赖氨酸参照刘洋等[15]。进样量为10 μL,检测波长为210 nm,柱温设置为30 ℃,流动相使用92 mL磷酸缓冲液混合8 mL乙腈。
1.9 计数及结合转移效率计算方法
通过稀释涂布孢子悬液获得单个平板上的菌落数在30~300内的梯度,在此梯度下,涂布3个平板,计算3个平板上菌落数的平均值乘以稀释倍数作为使用的孢子数。结合转移效率(η)计算如公式(1)所示:
(1)
2 结果与分析
2.1 基于pJTU1278质粒构建含诱导型Cas9的质粒
以pJTU1278质粒作为骨架,使用限制性核酸内切酶BamHⅠ和EcoRⅠ对该质粒进行双酶切,获得线性化质粒pJTU1278;为了控制Cas9的毒性,以质粒pTRIA为模板,通过引物Cas9-F/Cas9-R进行PCR获得含有诱导型启动子和核糖开关调控的Cas9片段;最后,通过一步克隆试剂盒将线性化质粒pJTU1278和Cas9片段进行连接,获得重组质粒pJTU-Cas9(图1-a)。为验证重组质粒pJTU-Cas9是否构建成功,使用BamH Ⅰ和EcoR Ⅰ对该质粒进行双酶切,经核酸凝胶电泳,获得的2个条带长度分别为9 089、4 526 bp,与理论DNA片段大小一致(图1-b),表明质粒pJTU-Cas9构建成功。
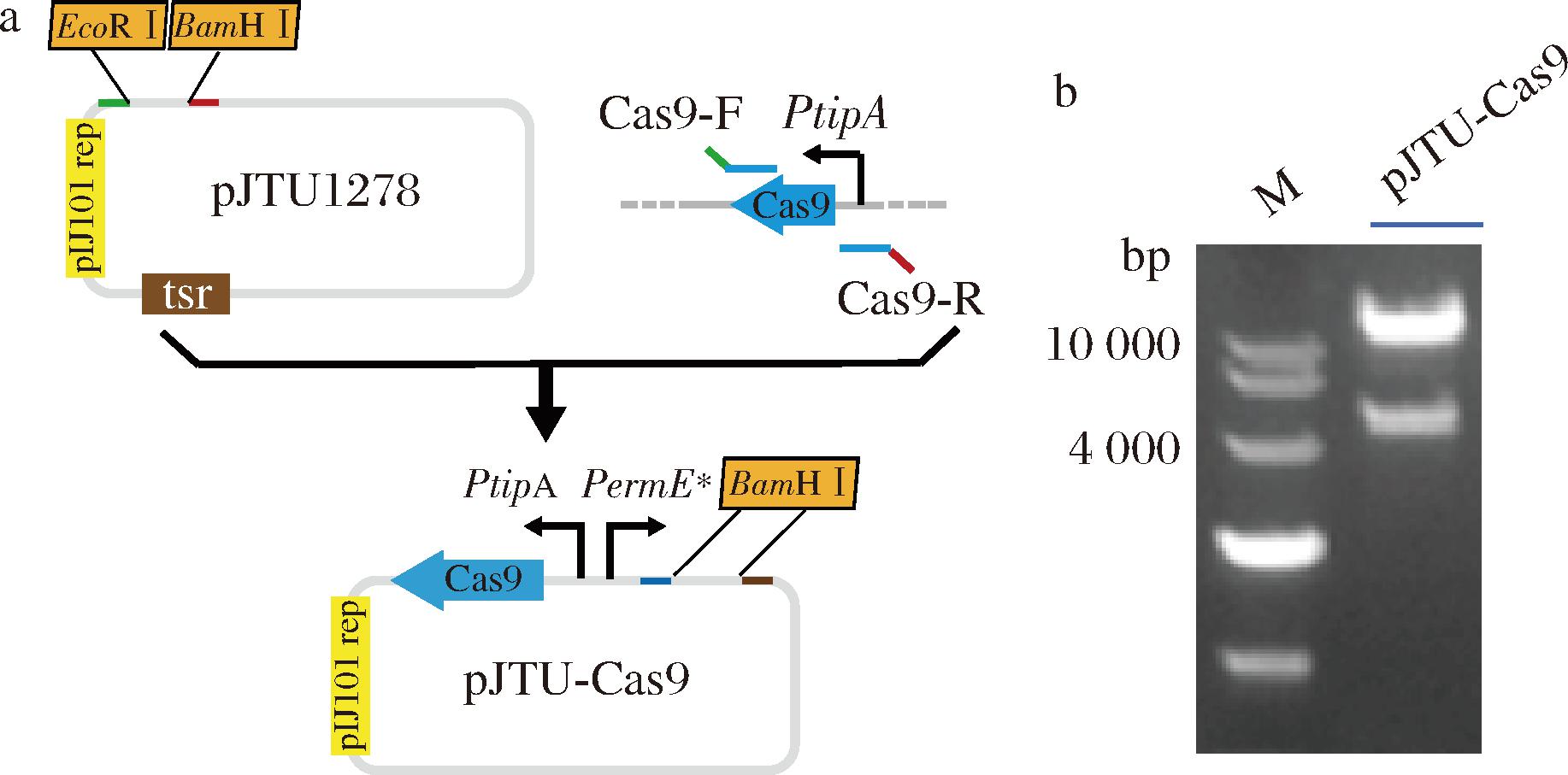
a-质粒pJTU-Cas9构建流程; b-质粒pJTU-Cas9双酶切核酸凝胶电泳胶图
图1 基于pJTU1278构建含诱导型Cas9的质粒
Fig.1 Construction of inducible Cas9 plasmid based on pJTU1278
2.2 靶向pls的敲除质粒构建
为便于验证敲除菌株的表型变化,本文选择S.albulus GS114基因组中编码ε-聚赖氨酸合成酶的基因pls作为靶基因。靶向pls的敲除质粒构建分为4步。
2.2.1 sgRNA的基因盒的构建
使用CRISPy-web网站[16]选择原间隔序列S1。为降低脱靶效应[17],选择的原间隔序列S1与基因组仅一个结合位点,且大于一个碱基错配时不存在结合位点。为节约时间和成本,通过在引物尾部添加碱基的方式引入原间隔序列,通过PCR来获得完整的sgRNA基因盒。基因盒片段SC通过引物SC-F/SC-R使用质粒pJTU-Cas9作为模板PCR获得。
2.2.2 质粒pJTU-Cas9-pls的构建
使用BamHⅠ酶切以线性化质粒pJTU-Cas9,将sgRNA表达盒与线性化质粒通过一步克隆试剂盒连接,获得重组质粒pJTU-Cas9-pls。使用引物Y-spacer-test-F/Y-spacer-test-R验证,产物条带正确(293 bp),与对照(273 bp)相对位置正确(图2-b),表明质粒pJTU-Cas9-pls构建成功。

a-质粒pJTU-Cas9-pls-UD构建流程;b-验证质粒pJTU-pJTU-Cas9-pls的核酸凝胶电泳胶图; c-验证质粒pJTU-Cas9-pls-UD的核酸凝胶电泳胶图
图2 靶向pls敲除质粒的构建
Fig.2 Construction of knockout plasmids targeting pls
2.2.3 同源臂及安普霉素抗性基因盒的获取
为降低对基因组的影响,使用HDR修复,同源臂片段UHA和DHA通过引物UHA-F/UHA-R和引物DHA-F/DHA-R使用S.albulus GS114的基因组作为模板PCR获得。同时,为了提高筛选的效率,选择将安普霉素基因盒置于上下游同源臂中间,其可以随着重组的进行整合至基因组上[18],安普霉素抗性基因盒由引物Apr-F/Apr-R使用质粒pIB139作为模板PCR获得。
2.2.4 质粒pJTU-Cas9-pls-UD的构建
通过HindⅢ酶切以线性化质粒pJTU-Cas9-pls,将对应的同源臂、安普霉素抗性基因盒和线性化质粒通过一步克隆试剂盒连接,获得重组质粒pJTU-Cas9-pls-UD。使用引物Y-arm-test-F/Y-arm-test-R验证,如图2-c所示,产物条带正确(2 878 bp),表明质粒pJTU-Cas9-pls-UD构建成功。
2.3 pls敲除菌株的获取和验证
将上述构建成功的pJTU-Cas9-pls-UD质粒导入感受态E.coil ET12567/pUZ8002中,再通过结合转移导入S.albulus GS114,经安普霉素筛选后,在MS平板上共长出7个结合子(图3-b)。JIANG等[12]研究表明,Cas9表达强度足够时无需诱导,泄露表达的Cas9即可实现基因编辑。因此,使用引物Y-pls-KO-f/Y-pls-KO-R对结合子的基因组进行PCR,发现7个结合子的目的片段大小均为2 868 bp(图3-c),与对照差异明显且与预期一致,表明安普霉素抗性基因成功插入。
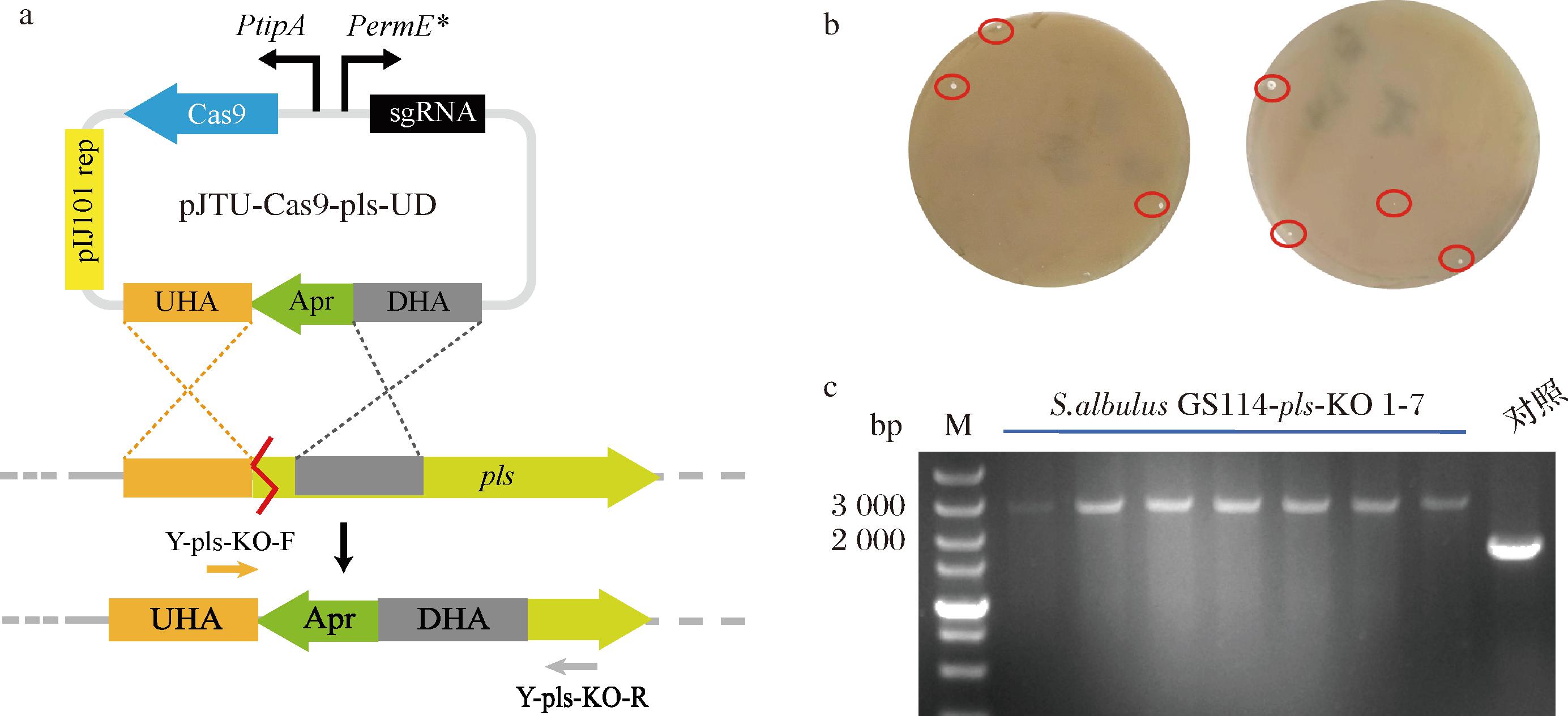
a-pls敲除示意图;b-结合转移平板;c-验证pls敲除菌株的核酸凝胶电泳胶图
图3 pls敲除菌株的获取
Fig.3 Acquisition of pls knockout strains
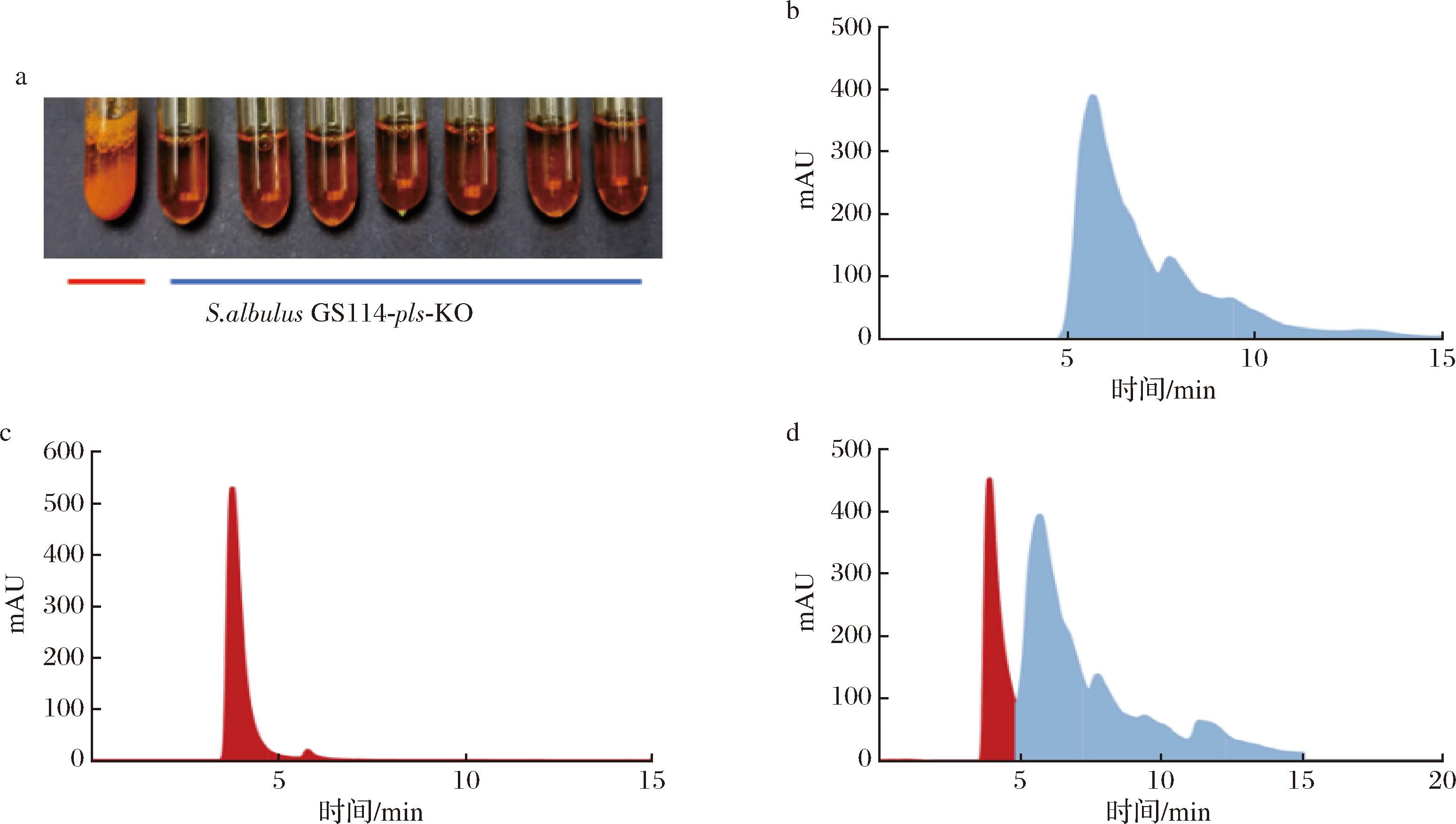
a-德拉道夫试剂检测发酵液;b-敲除菌株(S.albulus GS114-pls-KO)发酵上清液HPLC图;c-ε-聚赖氨酸标品HPLC图; d-原始菌株(S.albulus GS114)发酵上清液HPLC图
图4 pls敲除菌株的验证
Fig.4 Validation of pls knockout strains
为进一步验证pls基因是否被成功敲除,将获得的7株S.albulus GS114-pls-KO和对照菌株S.albulus GS114分别接入YP培养基进行发酵。取发酵上清液与德拉道夫试剂反应,发现S.albulus GS114-pls-KO发酵上清液与德拉道夫试剂反应后均无明显沉淀,而S.albulus GS114的发酵上清液与德拉道夫试剂反应后有明显的沉淀(图4-a),表明7株敲除菌均丧失了ε-聚赖氨酸合成能力。同时,利用HPLC对S.albulus GS114-pls-KO和S.albulus GS114的发酵上清液进行ε-聚赖氨酸检测。与原始菌株相比(图4-d),pls敲除菌株发酵上清液在ε-聚赖氨酸标准品(图4-c)所对应的保留时间上无特征峰(图4-b),这表明pls敲除菌株发酵液中不含ε-聚赖氨酸,意味着pls基因已成功从S.albulus GS114中敲除,且敲除效率为100%。
2.4 小白链霉菌结合转移条件优化
尽管上述建立的基于CRISPR-Cas9系统的基因敲除方法能够高效的实现目的基因敲除(敲除率100%),但在结合转移过程中获得的结合子数量较少(两个平板仅有7个结合子,结合转移效率为1.4×10-8)。为了提高结合子数量,有必要对结合转移条件进行系统优化,以期提高结合转移效率。据文献报道,影响大肠杆菌和链霉菌结合转移效率的条件主要包括供受体比例、覆盖抗生素时间、金属离子浓度和链霉菌孢子热激温度[19]。为此,本文对上述影响结合转移效率的条件进行系统优化。
2.4.1 供受体比例对结合转移的影响
大肠杆菌和链霉菌的结合转移是大肠杆菌作为供体、链霉菌作为受体发生的DNA物质交换的过程。链霉菌获得大肠杆菌中穿梭质粒的效率受到供受体比例的影响。将供体菌E.coil ET12567/pUZ8002(含有质粒pJTU-apr,安普霉素抗性)与受体菌S.albulus GS114的比例设置为100∶1至1∶10,分别进行结合转移,结果如表4所示。当供受体比例从100∶1降低至1∶1时,结合转移效率从7.6×10-7增加至4.0×10-5,提高了2个数量级。然而,将供受体比例继续降低至1∶10时,结合转移效率却下降了约3倍,表明供受体比例是影响E.coil ET12567和S.albulus GS114结合转移效率的重要因素。因此,选择供受体比例为1∶1,此时获得的最大结合转移效率为4.0×10-5。
表4 供受体比例对结合转移的影响
Table 4 Effect of donor receptor ratio on conjugation frequency

S.albulus供受体比例MgCl2浓度/(mmol/L)热冲击温度结合转移效率3.7×106100∶1(108∶106)10507.6×10-73.7×10710∶1(108∶107)10504.5×10-63.7×1081∶1(108∶108)10504.0×10-53.7×1081∶10(108∶107)10501.2×10-5
2.4.2 覆盖抗生素时间对结合转移的影响
在结合转移过程中,质粒由大肠杆菌转入链霉菌需要一定时间,因此,在结合转移中后期需要在平板上覆盖与质粒抗性相对应的抗生素,以杀死未能获得质粒的受体菌(S.albulus GS114);同时通过覆盖萘啶酮酸杀死所有的供体菌(E.coil ET12567/pUZ8002),从而筛选出成功导入质粒的受体菌。适当延长覆盖抗生素的时间上可以增加转移的时间,提高结合转移的效率,但是过长的培养时间会导致未获得质粒的受体菌也生长为单菌落,不利于筛选。为此,考察了E.coil ET12567/pUZ8002(含质粒pJTU-apr)与S.albulus GS114进行结合转移的抗生素覆盖时间,结果如表5所示。培养8~12 h后覆盖抗生素,无法获得结合子。14~20 h培养后覆盖抗生素,结合转移的效率逐渐增大。然而,培养超过20 h覆盖抗生素,无法起到筛选作用。因此,选择培养20 h时覆盖抗生素,可以在保证有筛选作用的情况下,获得最大的结合转移效率7.3×10-5。
表5 覆盖抗生素时间对结合转移的影响
Table 5 Effect of overlaying time of antibotics on conjugation frequency
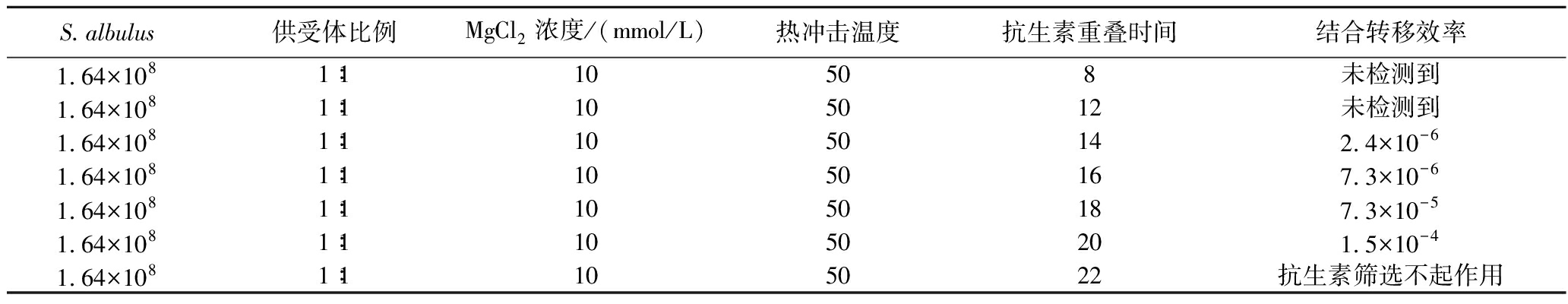
S.albulus供受体比例MgCl2浓度/(mmol/L)热冲击温度抗生素重叠时间结合转移效率1.64×1081∶110508未检测到1.64×1081∶1105012未检测到1.64×1081∶11050142.4×10-61.64×1081∶11050167.3×10-61.64×1081∶11050187.3×10-51.64×1081∶11050201.5×10-41.64×1081∶1105022抗生素筛选不起作用
2.4.3 镁离子和钙离子浓度对结合转移的影响
根据文献报道,在MS固体平板中添加一定浓度钙离子和镁离子可以提高结合转移的效率[20]。因此,考察了不同浓度MgCl2、CaCl2对E.coil ET12567/pUZ8002(含质粒pJTU-apr)与S.albulus GS114结合转移的影响,结果如表6和表7所示。同等浓度下,MgCl2的结合转移效率均大于CaCl2。由于高浓度金属离子对菌体生长有显著抑制作用,最终选择30 mmol/L的MgCl2作为最适浓度,此时结合转移效率为3.0×10-5。
表6 镁离子浓度或钙离子浓度对结合转移影响
Table 6 Effect of concentration of MgCl2and CaCl2on conjugation frequency
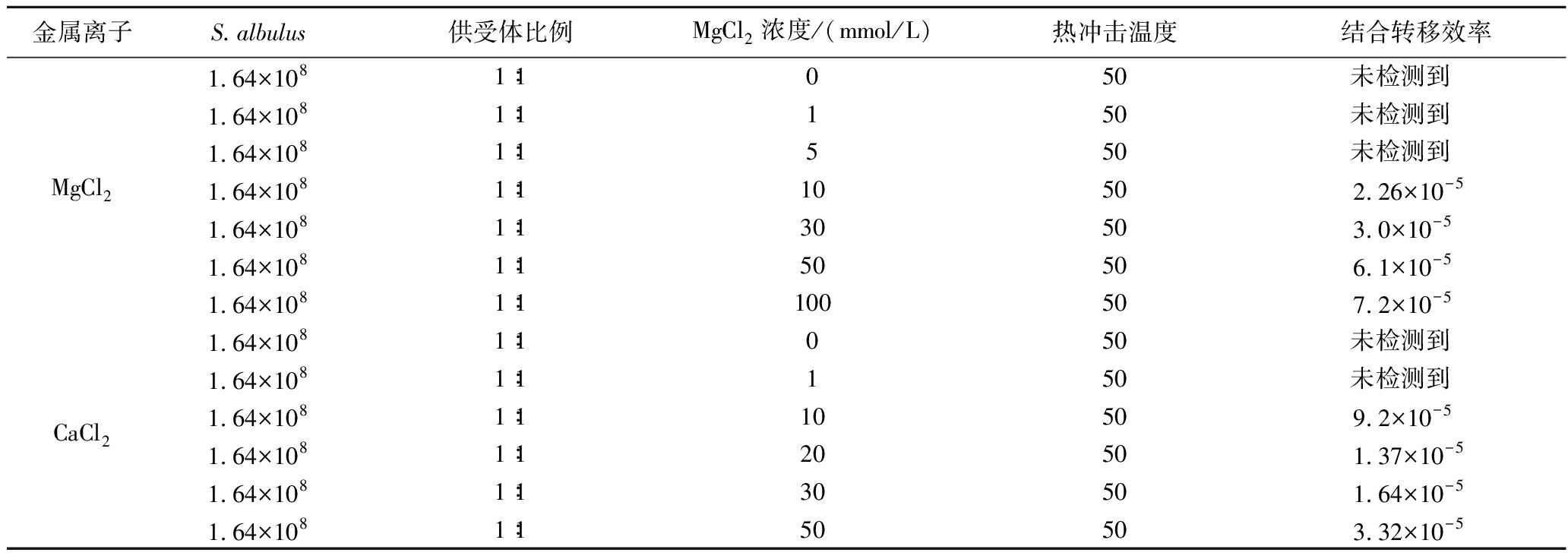
金属离子S.albulus供受体比例MgCl2浓度/(mmol/L)热冲击温度结合转移效率MgCl21.64×1081∶1050未检测到1.64×1081∶1150未检测到1.64×1081∶1550未检测到1.64×1081∶11050 2.26×10-51.64×1081∶13050 3.0×10-51.64×1081∶15050 6.1×10-51.64×1081∶110050 7.2×10-5CaCl21.64×1081∶1050未检测到1.64×1081∶1150未检测到1.64×1081∶11050 9.2×10-51.64×1081∶12050 1.37×10-51.64×1081∶13050 1.64×10-51.64×1081∶15050 3.32×10-5
表7 热激温度对结合转移
Table 7 Effect of temperature of heat-shock on conjugation frequency

S.albulus供受体比例MgCl2浓度/(mmol/L)热冲击温度结合转移效率0.92×1081∶110未加热3.15×10-50.92×1081∶110454.2×10-50.92×1081∶110509.08×10-50.92×1081∶110551.01×10-40.92×1081∶11060未检测到
2.4.4 热激温度对结合转移的影响
热激链霉菌孢子可以改变细胞膜组分比例,提高细胞的通透性,该操作对结合转移影响显著。其中热激的温度是影响转化效率最重要的影响因素之一。将受体菌S.albulus GS114孢子悬液使用不同的温度热激后与供体菌ET12567/pUZ8002(pJTU-apr)进行结合转移,结果如表7所示。当温度低于55 ℃时,随着温度的提高,结合转移效率不断增加;当温度为60 ℃时,结合转移几乎无法发生。因此,热激链霉菌孢子悬液时的温度选择55 ℃,此时获得最高结合转移效率为1.01×10-4。
2.5 最优条件下小白链霉菌中CRISPR-Cas9系统结合转移效率评估
通过上述实验,获得的最优结合转移条件为:供受体比例1∶1,在MS平板中添加30 mmol/L镁离子,使用55 ℃热激S.albulus GS114孢子,于20 h时覆盖抗生素。在此最适条件下,使用含有pJTU-Cas9-pls-UD的E.coil ET12567/pUZ8002重复对S.albulus GS114中pls进行敲除,实验结果如图5所示。单个结合转移平板长出10个结合子,结合转移效率达到2.5×10-8,较优化前提高了78%。
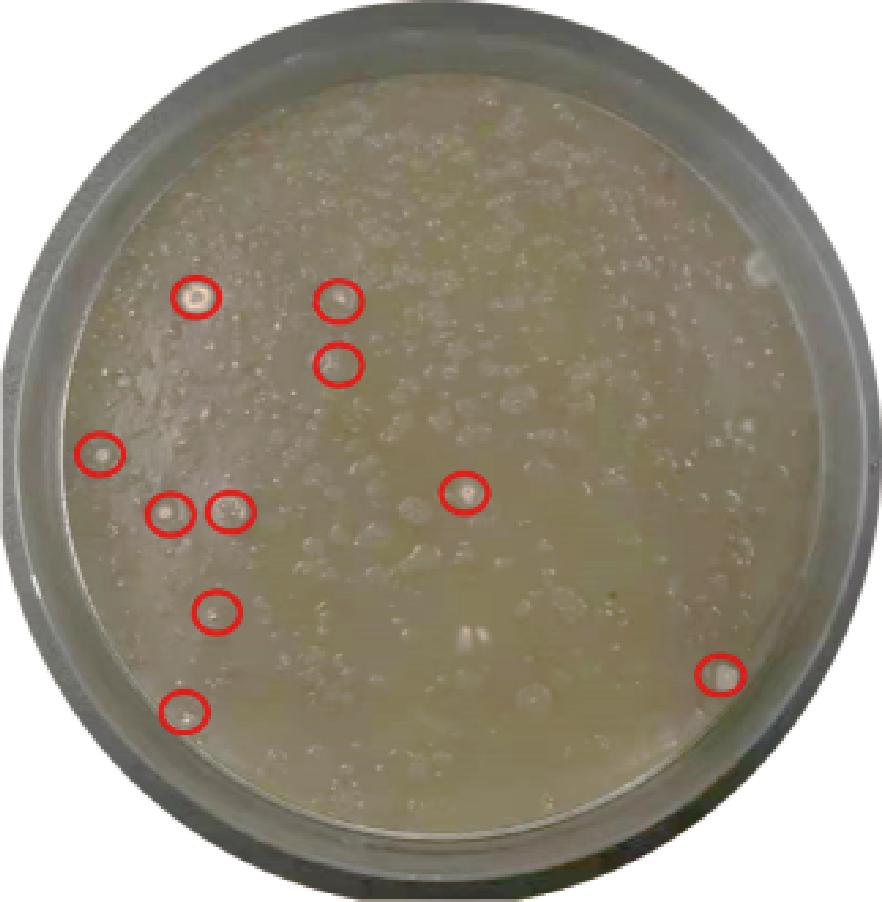
图5 最优条件下小白链霉菌中CRISPR-Cas9系统 转移效率评估
Fig.5 Evaluation of CRISPR-Cas9 system conjugation efficiency in S.albulus under optimal conditions
3 结论
本文以pJTU1278作为质粒骨架,通过一步克隆技术,构建了包含sgRNA、同源臂、带有tipA和核糖开关调控的Cas9的敲除质粒pJTU-Cas9-pls-UD。通过结合转移,将上述敲除质粒成功导入S.albulus GS114,获得了7个结合子,结合转移效率为1.4×10-8;进一步PCR和发酵验证,实现了S.albulus GS114中靶向敲除了pls基因,敲除成功率为100%。为进一步提高敲除株数量,通过系统优化结合转移条件,实现结合转移效率,达到2.5×10-8,较优化前提高了78%。基于以上研究,初步在S.albulus中建立了一种基于CRISPR-Cas9系统的基因敲除方法,为后续利用代谢工程方法系统改造新一代ε-聚赖氨酸生产菌种奠定了坚实基础。
[1] TUERSUNTUOHETI T, WANG Z H, WANG Z Y, et al.Review of the application of ε-poly-L-lysine in improving food quality and preservation[J].Journal of Food Processing and Preservation, 2019, 43(10):e14153
[2] WANG L, ZHANG C Y, ZHANG J H, et al.Epsilon-poly-L-lysine:Recent advances in biomanufacturing and applications[J].Frontiers in Bioengineering and Biotechnology, 2021, 9:748976.
[3] XIANG J H, YANG Y, DABBOUR M, et al.Metabolomic and genomic profiles of Streptomyces albulus with a higher ε-polylysine production through ARTP mutagenesis[J].Biochemical Engineering Journal, 2020, 162:107720.
[4] YANG H, ZHU D J, KAI L, et al.Engineering Streptomyces albulus to enhance ε-poly-L-lysine production by introducing a polyphosphate kinase-mediated ATP regeneration system[J].Microbial Cell Factories, 2023, 22(1):51.
[5] XU D L, YAO H Q, CAO C H, et al.Enhancement of ε-poly-L-lysine production by overexpressing the ammonium transporter gene in Streptomyces albulus PD-1[J].Bioprocess and Biosystems Engineering, 2018, 41(9):1337-1345.
[6] 汪泽, 王开方, 胡扬帆, 等.过表达pls基因结合前体流加提高小白链霉菌ε-聚赖氨酸产量[J].食品与发酵工业, 2020, 46(14):1-6.
WANG Z, WANG K F, HU Y F, et al.Enhanced ε-poly-L-lysine production through overexpression of pls gene combined with precursor feeding in Stretomyces albulus[J].Food and Fermentation Industries, 2020, 46(14):1-6.
[7] BU Q T, LI Y P, XIE H, et al.Rational engineering strategies for achieving high-yield, high-quality and high-stability of natural product production in actinomycetes[J].Metabolic Engineering, 2021, 67:198-215.
[8] HSU P D, LANDER E S, ZHANG F.Development and applications of CRISPR-Cas9 for genome engineering[J].Cell, 2014, 157(6):1262-1278.
[9] COBB R E, WANG Y J, ZHAO H M.High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system[J].ACS Synthetic Biology, 2015, 4(6):723-728.
[10] TONG Y J, CHARUSANTI P, ZHANG L X, et al.CRISPR-Cas9 based engineering of actinomycetal genomes[J].ACS Synthetic Biology, 2015, 4(9):1020-1029.
[11] WANG K, ZHAO Q W, LIU Y F, et al.Multi-Layer controls of Cas9 activity coupled with ATP synthase over-expression for efficient genome editing in Streptomyces[J].Frontiers in Bioengineering and Biotechnology, 2019, 7:304.
[12] JIANG Y H, LIU Y F, WANG K, et al.Fine-tuning Cas9 activity with a cognate inhibitor AcrIIA4 to improve genome editing in Streptomyces[J].ACS Synthetic Biology, 2021, 10(11):2833-2841.
[13] YE S H, ENGHIAD B, ZHAO H M, et al.Fine-tuning the regulation of Cas9 expression levels for efficient CRISPR-Cas9 mediated recombination in Streptomyces[J].Journal of Industrial Microbiology &Biotechnology, 2020, 47(4-5):413-423.
[14] 张重阳, 杨昊, 朱道君, 等.过表达L-赖氨酸合成途径关键基因和ε-聚赖氨酸合成酶基因对ε-聚赖氨酸合成的影响[J].食品与发酵工业, 2023, 49(6):1-9.
ZHANG C Y,YANG H,ZHU D J, et al.Effect of overexpression of key genes in L-lysine biosynthesis pathway and ε-poly-L-lysine synthase gene on ε-poly-L-lysine production[J].Food and Fermentation Industries, 2023, 49(6):1-9.
[15] 刘洋, 王靓, 张宏建, 等. 大孔强酸树脂膨胀床吸附提取ε-聚赖氨酸的研究[J]. 食品与发酵工业, 2022, 48(22):9-17.
LIU Y, WANG L, ZHANG H J, et al. Extraction of ε-poly-L-lysine from fermentation broth by expanded bed adsorption using macroporous strong acid resin as adsorbent[J]. Food and Fermentation Industries, 2022, 48(22):9-17.
[16] TONG Y J, WHITFORD C M, BLIN K, et al.CRISPR-Cas9, CRISPRi and CRISPR-BEST-mediated genetic manipulation in streptomycetes[J].Nature Protocols, 2020, 15(8):2470-2502.
[17] 张晓婷, 张妍, 戴剑漉, 等.利用CRISPR-Cas9系统构建新型异戊酰螺旋霉素Ⅰ产生菌[J].生物工程学报, 2019, 35(3):472-481.
ZHANG X T,ZHANG Y,DAI J L, et al.Construction of a new isovalerylspiramycin Ⅰ producing strain by CRISPR-Cas9 system[J].Chinese Journal of Biotechnology, 2019, 35(3):472-481.
[18] ZHANG S M, CHEN T S, JIA J, et al.Establishment of a highly efficient conjugation protocol for Streptomyces kanamyceticus ATCC12853[J].Microbiologyopen, 2019, 8(6):e00747.
[19] 王一琇. 基于CRISPR: cBEST技术的三株放线菌遗传操作体系构建[D]. 哈尔滨: 东北农业大学, 2023.
WANG Y X. Construction of genetic operation system of three actinomycetes based on CRISPR—cBEST technology[D].Harbin: Northeast Agricultural University, 2023.
[20] PANJA S, SAHA S, JANA B, et al.Role of membrane potential on artificial transformation of E.coli with plasmid DNA[J].Journal of Biotechnology, 2006, 127(1):14-20.