N-糖基化修饰是蛋白质糖基化最主要的修饰方式之一,在蛋白质的折叠、稳定性以及功能等方面发挥关键作用。N-糖基化修饰的相关研究在食品领域中具有重要意义,如鉴别掺假食品[1]、改善食品品质[2]、揭发食物过敏机制[3]等。N-聚糖结构和功能分析是N-糖基化修饰研究中的关键步骤,N-糖酰胺酶(N-glycosidase, PNGase)是释放N-糖链的重要工具,此类酶可以催化水解天冬酰胺残基与N-乙酰葡萄糖胺之间的酰胺键,释放完整的糖链和蛋白质或多肽[4]。
常用的商业PNGase主要有PNGase A和PNGase F,这2种酶在实际应用过程中存在一定的局限性。PNGase F可以在大肠杆菌中重组表达但是无法酶切植物或昆虫来源的含有核心α-1,3岩藻糖的结构的糖肽或糖蛋白,适用于动物来源的糖蛋白上的N-糖链的释放[5-6];PNGase A由TAKAHASHI等[7]在甜杏仁中提取,该酶可以酶解释放有核心α-1,3岩藻糖结构的N-糖链,但无法使用大肠杆菌异源表达系统进行重组制备,并且对大分子糖蛋白的水解效率要劣于小分子糖肽,更适用于植物源糖蛋白上N-链寡糖的释放[8-10]。根据已有报道,PNGase F-II可以在大肠杆菌中进行重组表达并可以释放所有类型的N-糖链,但该酶在酸性环境中缺乏活性,无法应用于氢-氘交换质谱[11];PNGase H+为WANG等[12]开发的新型PNGase,该酶可以在酸性条件下释放N-糖链,但此酶纯化过程复杂,且稳定性较差,在纯化过程中容易发生降解。因此,发掘性能优良的PNGase具有研究意义。
本研究从NCBI数据库中得到了细菌(Dyella caseinilytica)来源的新型PNGase(该酶命名为PNGase Dc)基因,将其在大肠杆菌E.coli BL21(DE3)中进行表达,并对表达、纯化后的PNGase Dc进行酶学特性测定和功能鉴定,以期为N-链寡糖的释放提供一个新的工具酶,并为PNGase Dc的开发利用奠定理论基础。
1 材料与方法
1.1 材料与试剂
本研究所用的BL21(D3)感受态细胞原始菌株购自天根生化科技有限公司;突变引物以及含有PNGase基因的重组质粒pET30a(+)-PNGase Dc由南京擎科生物科技有限公司合成。
FastPAGETM蛋白预制胶,南京擎科生物科技有限公司;PNGase A,南京梅林学海生物科技有限公司;PNGase F,南京伟沃生物科技有限公司;高保真PrimeSTAR HS DNA聚合酶、dNTP Mixture(2.5 mmol/L),TaKaRa公司;野生型 Col-0拟南芥由本实验室保存并繁种;C57BL/6 野生型小鼠,南京市青龙山动物繁殖场;辣根过氧化物酶(horseradish peroxidase,HRP),南京都莱生物技术有限公司;牛乳铁蛋白,上海惠诚生物科技有限公司。
1.2 仪器与设备
超声波细胞破碎仪Soniprep 150,MSE公司;Thermo Multiscan FC型酶标仪,赛默飞世尔科技有限公司;超高效液相色谱LC-30AD-RF20Axs荧光检测器,Shimadzu Nexera公司。
1.3 实验方法
1.3.1 重组PNGase Dc的同源性分析
从NCBI数据库(http://www.ncbi.nlm.nih.gov/blast/)中调取若干PNGase Dc的氨基酸序列,并用Protein Blast功能进行同源性分析;使用软件MEGA X通过邻接法(Neighbor-Joining,NJ)构建系统发育树,Bootstrap值为1 000;使用软件GeneDoc进行多重序列比对。
1.3.2 重组PNGase Dc的体外表达及纯化
将重组质粒pET30a(+)-PNGase Dc转入E.coli BL21(DE3),挑取单菌落接种至5 mL含卡那霉素(50 μg/mL)的 LB 培养基中于37 ℃过夜培养,然后转至400 mL LB 培养基中扩大培养。待菌液OD600达到0.5~0.8时,加入200 μL 1 mol/L的IPTG,18 ℃诱导18 h。将菌液离心,收集菌体沉淀并加入10 mL细胞裂解液(100 mmol/L Nacl,50 mmol/L Tris-HCl pH 8.0,1% Triton X-100)重悬。超声功率为200 W进行细胞破碎。将细胞破碎液离心,取上清液利用亲和层析镍柱进行纯化。取PNGase Dc诱导前菌体、诱导后菌体、裂解上清液及纯化后的粗酶液,使用12%质量分数的SDS-PAGE进行凝胶电泳分析。
1.3.3 重组PNGase Dc的酶学特性测定
1.3.3.1 最适pH的测定
采用SDS-PAGE凝胶迁移法测定PNGase Dc的最适pH。反应体系中含有1 μL 底物HRP(10 μg/μL),6 μL磷酸钠-柠檬酸盐缓冲液(1 mol/L,pH 分别为2.0、2.5、3.0、3.5、4.0、4.5、5.0),2 μL PNGase Dc(1 μg/μL)和11 μL超纯水,于37 ℃下过夜孵育。反应结束后用10 mol/L NaOH调节溶液的pH值至7.0,然后使用12% SDS-PAGE凝胶电泳进行分析。
1.3.3.2 最适温度的测定
参考WANG等[13]建立的PNGase酶活性测定方法,以HRP为底物,采用比色法测定PNGase Dc的最适温度。在最适pH下,将重组PNGase Dc的反应体系(与1.3.3.1节反应体系一致)置于不同温度下,反应温度依次为4、25、37、45、55、70 ℃,过夜孵育。反应结束后加入20 μL 2.5 mol/L三氯乙酸终止反应并沉淀蛋白,12 000 r/ min离心30 min。离心结束后,每组取10 μl的反应上清液加入384孔板中,然后依次加入5 μL NaOH(4 mol/L)和10 μL WST-1(1.7 mM,水溶性四唑盐-1),与反应上清液充分混合,于50 ℃烘箱中反应1 h。使用酶标仪测定每组样品在584 nm下的吸光值。每组反应设置3个平行,将最大酶活性定义为100%。
1.3.3.3 金属离子对酶活性的影响测定
参考1.3.3.2节的酶活性测定方法,配制20 mmol/L的Ca2+、Co2+、Fe3+、Mg2+、Zn2+、Cu2+、Mn2+、EDTA溶液,将PNGase Dc分别置于含有不同金属离子的反应体系中,在最适温度及pH条件下过夜孵育,每组反应设置3个平行,将未添加任何金属离子及EDTA的样品作为阴性对照。
1.3.3.4 4 ℃贮藏稳定性测定
参考1.3.3.2节的酶活性测定方法,将PNGase Dc的酶液预先分装在不同的离心管中并在4 ℃冰箱中贮藏,贮藏期间每隔5 d进行1次酶活性检测。每组反应设置3个平行,将贮藏0 d的样品作为阴性对照并定义其酶活性为100%。
1.3.4 重组PNGase Dc对牛乳铁蛋白去糖基化能力的的测定
参考1.3.3.1节的酶活测定方法,使用FastPAGE预制梯度胶检测PNGase Dc对牛乳铁蛋白的去糖基化能力。以不添加酶的样品作为阴性对照,以添加商业酶PNGase F的样品作为阳性对照。
1.3.5 重组PNGase Dc与N,N′-二乙酰壳二糖的分子对接
使用AlphaFold2(https://github.com/deepmind/alphafold#readme)对PNGase Dc进行三维结构预测;使用Chem3D软件绘制底物N,N′-二乙酰壳二糖;使用AutoDock Vina软件进行半柔性对接,以Asp102和Glu236这两个位点的空间位置作为活性空腔,设置对接盒子大小为(24×24×24)Å3,中心为(13.1,-3.9,-6.3),使用软件Pymol和Discover studio 2021进行可视化分析。
1.3.6 定点突变
利用Quick-change定点突变的方法,将PNGase Dc的Asp102和Glu236分别突变为丙氨酸,突变引物设计如表1所示。突变反应体系:0.5 μL pET30a(+)-PNGase Dc质粒,0.5 μL高保真PrimeSTAR HS DNA聚合酶,0.5 μL正向引物,0.5 μL反向引物,4 μL dNTP和34 μL超纯水。PCR反应程序:98 ℃预变性5 min;98 ℃变性10 s,55 ℃退火15 s,72 ℃延伸1 min,循环30次;72 ℃终延伸10 min,降温至 4 ℃保存。D102A和E236A 2个突变质粒经DNA测序验证后分别转化E.coli BL 21(DE3)感受态细胞进行重组表达。表达纯化方法参考1.3.2节,酶活性测定方法参考1.3.3.1节。
表1 实验所用引物
Table 1 Primers used in this study
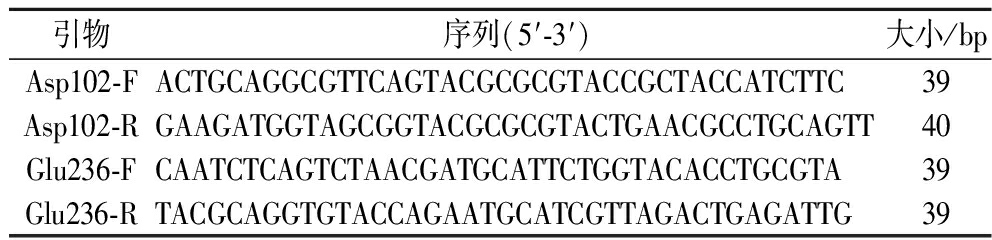
引物序列(5′-3′)大小/bpAsp102-FACTGCAGGCGTTCAGTACGCGCGTACCGCTACCATCTTC39Asp102-RGAAGATGGTAGCGGTACGCGCGTACTGAACGCCTGCAGTT40Glu236-FCAATCTCAGTCTAACGATGCATTCTGGTACACCTGCGTA39Glu236-RTACGCAGGTGTACCAGAATGCATCGTTAGACTGAGATTG39
1.4.7 重组PNGase Dc的应用研究
参考DU等[14]提取植物糖蛋白的方法,使用40%的三氯乙酸溶液提取拟南芥中的糖蛋白。PNGase Dc和PNGase A分别以拟南芥为底物,在最适温度和pH下过夜孵育;小鼠血浆由摘眼球采血法制得,PNGase Dc和PNGase F分别以小鼠血浆为底物,在最适温度和pH下孵育过夜。反应结束后,使用SupelcleanTM ENVI-CarbTM SPE柱纯化反应液中的N-链寡糖,之后使用2-氨基苯甲酰胺对N-糖链进行衍生化标记,利用HPLC对N-糖链进行检测。检测所用的色谱柱为BEH Glycan column(1.7 μm×2.1 mm×150 mm,水相),流动相A为50 mmol/L甲酸铵溶液,pH值为4.5,流动相B为乙腈,激发波长为330 nm,检测波长为420 nm,柱温设为60 ℃。
2 结果与分析
2.1 重组PNGase Dc的同源性分析
系统发育进化树表明,PNGase Dc、PNGase H+、PNGase A、AtPng1p共源于同一进化分支,且PNGase Dc与PNGase H+均为细菌来源,具有更近的进化关系(图1-A)。PNGase Dc与PNGase H+的多重序列比对结果表明,这二者氨基酸序列具有44.72%的相似性(图1-B)。在PNGase H+[13]中,Asp97和Glu231是影响该酶活性的关键催化位点,且这2个位点在氨基酸序列中具有保守性,因此,推测在PNGase Dc中相对应的Asp102和Glu236可能对该酶的活性起关键作用。

A-不同物种间PNGase系统进化树;B-多重序列比对
图1 重组PNGase Dc的同源性分析
Fig.1 Homology analysis of recombinant PNGase Dc
2.2 重组PNGase Dc的SDS-PAGE分析
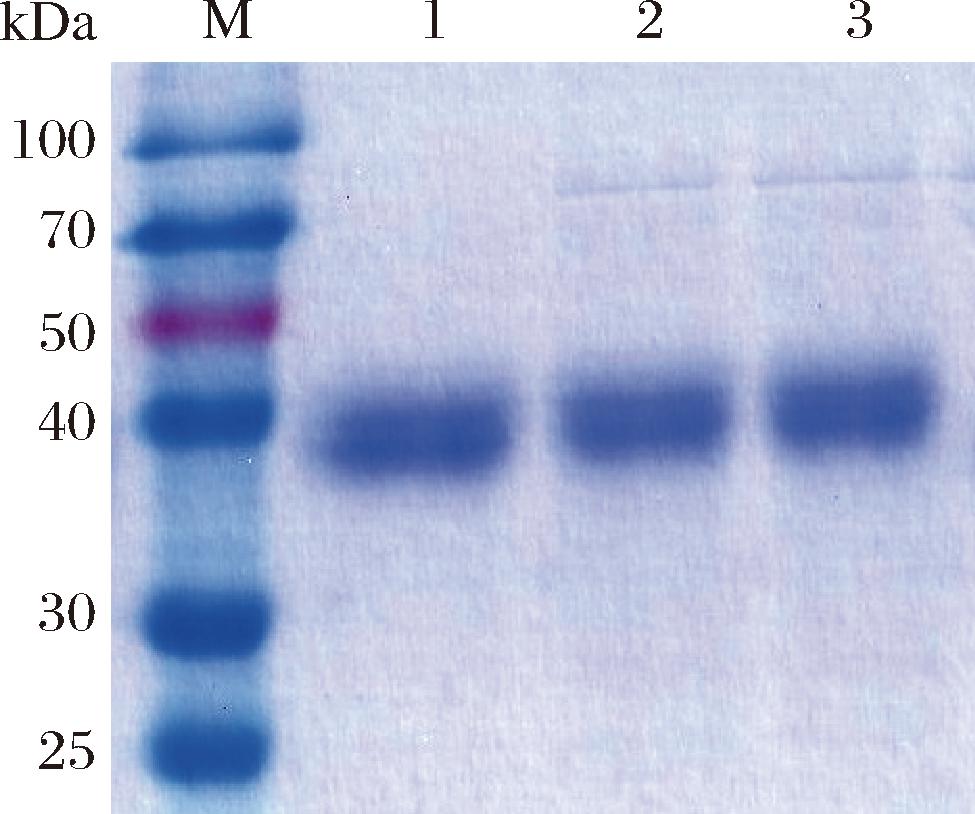
将重组质粒pET30a(+)-PNGase Dc转化到E.coli BL21(DE3)进行诱导表达,采用SDS-PAGE的方法检测其分子质量及蛋白纯度(图2)。PNGase Dc经纯化后,其蛋白条带单一,表观分子质量为63.01 kDa,与理论分子量相符。
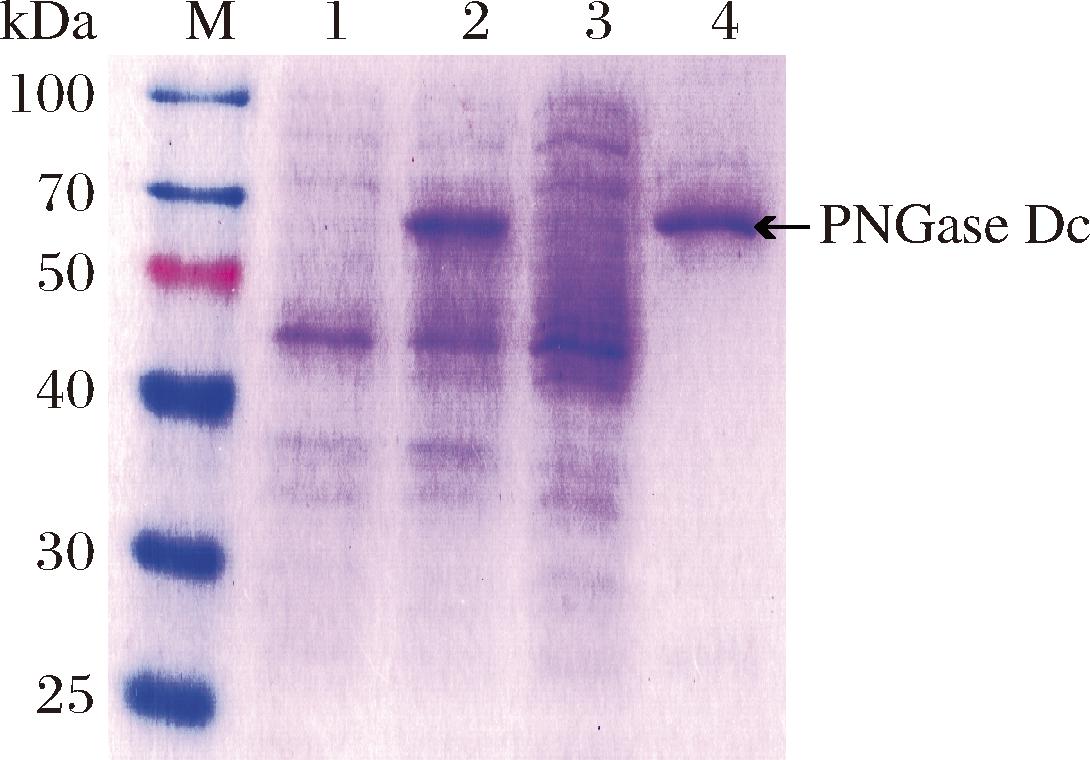
M-蛋白Marker标准品;1-诱导前菌体;2-诱导后菌体; 3-裂解后上清;4-纯化后蛋白
图2 重组PNGase Dc的SDS-PAGE分析
Fig.2 SDS-PAGE analysis of recombinant PNGase Dc
注:PNGase Dc理论分子质量为63.01 kDa;箭头指向目标蛋白的条带。
2.3 重组PNGase Dc的最适pH
HRP作为植物源标准糖蛋白,含有核心α-1,3岩藻糖,结构为(Xyl)1(Man)3(Fuc)1(GlcNAc)2,这是典型的植物型N-聚糖结构[15-17]。以HRP为底物测定pH对PNGase Dc酶活性的影响(图3-A),与对照相比,当pH为2.0~4.0时,经PNGase Dc作用的HRP蛋白分子质量变小,蛋白条带在SDS-PAGE上产生明显位移,且pH为2.0时,HRP蛋白条带位移最大;当pH为4.5时,HRP的蛋白条带仅出现部分位移;当pH为5.0时,HRP的蛋白条带未发生位移,酶几乎失去活性,无法切除HRP上的N-糖链。因此,PNGase Dc的最适pH值为2.0。与之前报道的PNGase H+[12]相比,PNGase Dc具有更广泛的pH作用范围;与PNGase A[10,18]相比,PNGase Dc对底物HRP具有更好的去糖基化效果。
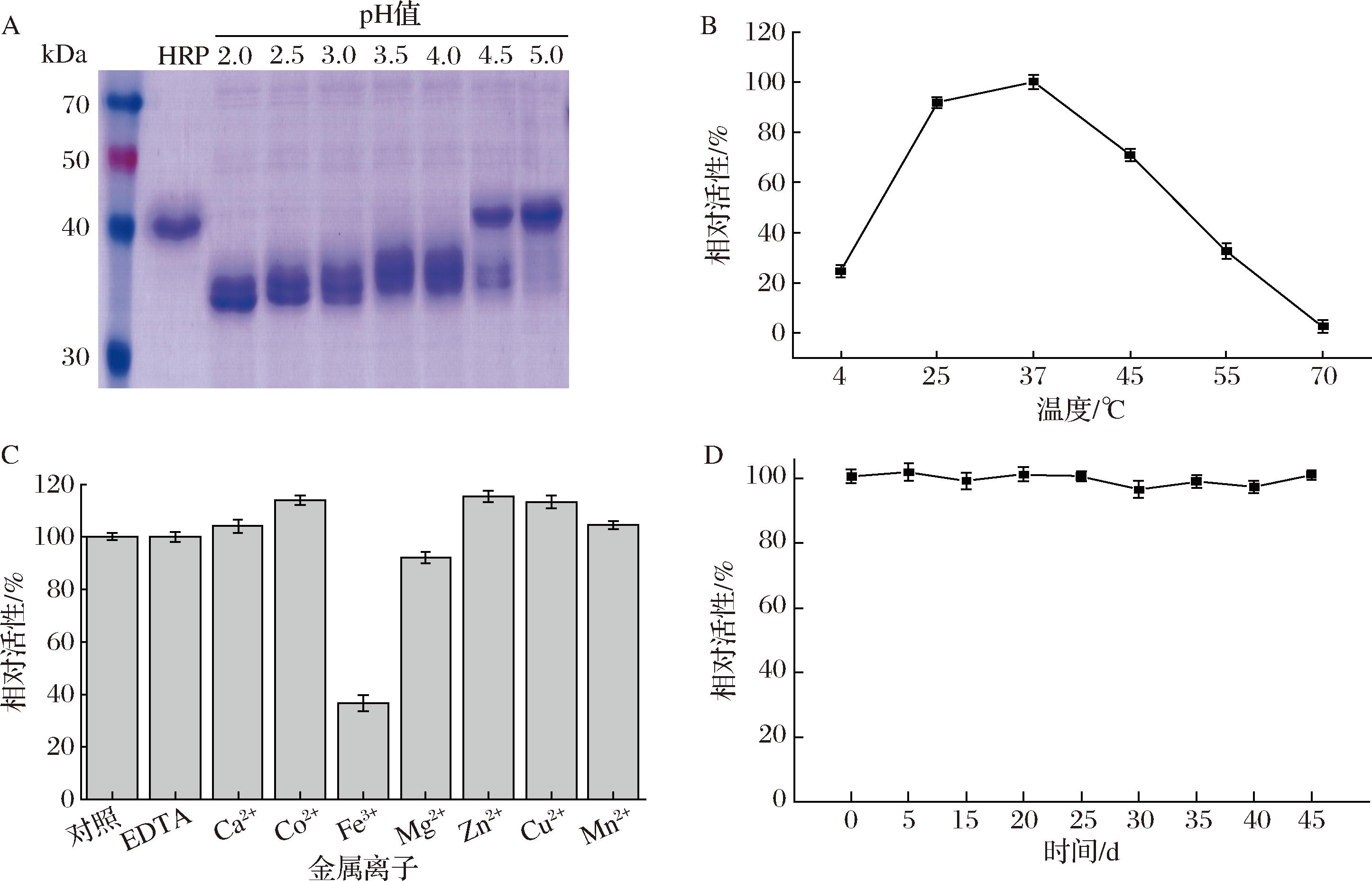
A-最适pH;B-最适温度;C-金属离子依赖性;D-4 ℃贮藏稳定性
图3 重组PNGase Dc的酶学特性测定
Fig.3 Determination of enzymatic characterization of recombinant PNGase Dc
2.4 重组PNGase Dc的最适温度
为探究温度对PNGase Dc酶活性的影响,将PNGase Dc分别置于温度范围为4~70 ℃的反应体系中进行测活。图3-B显示,PNGase Dc的最适反应温度为37 ℃。当温度为45 ℃时,PNGase Dc仍具有70.96%的酶活性,当温度高于45 ℃时,酶活性迅速下降。
2.5 金属离子对PNGase Dc活性的影响
为探究金属离子对PNGase Dc酶活性的影响,将PNGase Dc分别置于含有不同金属离子及EDTA的反应体系中进行测活。图3-C显示,PNGase Dc经EDTA处理后,酶活性并未受到影响;PNGase Dc经Co2+、Zn2+和Cu2+处理后,酶活性轻微上升;PNGase Dc经Fe3+处理后,酶活性显著下降,仅为36.55%。因此,PNGase Dc不属于金属离子依赖型的酶。
2.6 重组PNGase Dc在4 ℃环境下的稳定性测定
为探究PNGase Dc在 4 ℃环境下的稳定性,将PNGase Dc的酶液预先贮藏在4 ℃冰箱中,期间每隔5 d进行测活。将贮藏0 d的样品作为阴性对照。图3-D显示,PNGase Dc在4 ℃冰箱中可以稳定保存至少45 d,且贮藏期间几乎没有活性下降。与PNGase H+[12]相比,PNGase Dc具有更优良的稳定性。
2.7 重组PNGase Dc对底物牛乳铁蛋白的去糖基化能力测定
为探究PNGase Dc对动物源糖蛋白的去糖基化能力,我们以牛乳铁蛋白作为底物进行研究。图4显示,牛乳铁蛋白经PNGase Dc和PNGase F处理后,蛋白条带均发生位移,且位移位置相同,这表明PNGase Dc对底物牛乳铁蛋白具有较好的去糖基化效果。
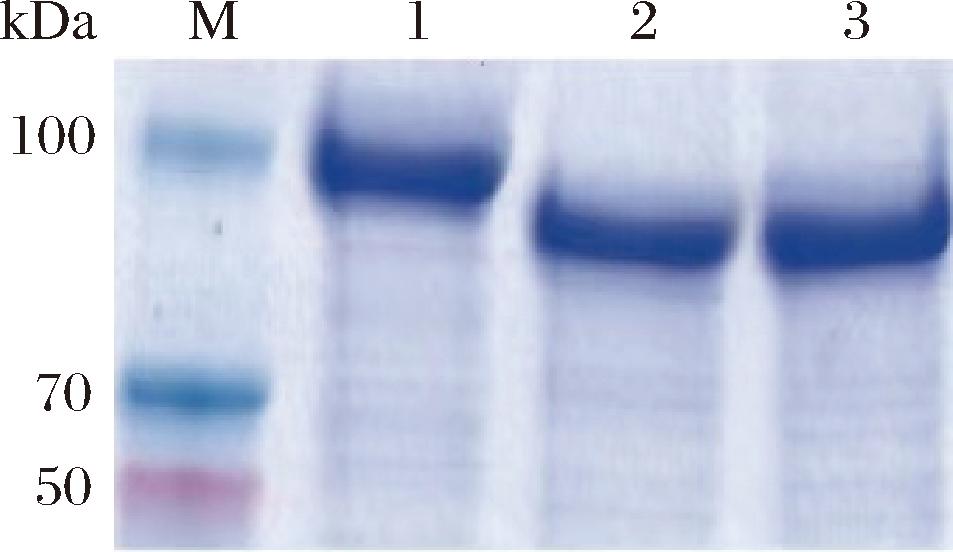
M-蛋白Marker标准品;1-牛乳铁蛋白过夜孵育;2-牛乳铁蛋白与 重组PNGase Dc的过夜反应产物;3-牛乳铁蛋白与商业酶 PNGase F的过夜反应产物
图4 SDS-PAGE检测重组PNGase Dc和商业酶PNGase F 对Lactoferrin的去糖基化
Fig.4 SDS-PAGE analysis of the deglycosylation of Lactoferrin by recombinant PNGase Dc and commercial PNGase F
2.8 重组PNGase Dc与N,N′-二乙酰壳二糖的分子对接分析
PNGase Dc的三维结构模型(图5-A)整体由2个相似的结构域和1个β-片层构成。上部分包含结构域a和结构域b,2个结构域之间由无规则卷曲连接。结构域a由9个反向平行的β-折叠和4个α-螺旋构成,结构域b由8个反向平行的β-折叠和1个α-螺旋构成。下部分为一个巨大的β-片层c,由14个反向平行的β-折叠和1个α-螺旋构成,结构域a和β-片层由无规则卷曲连接。
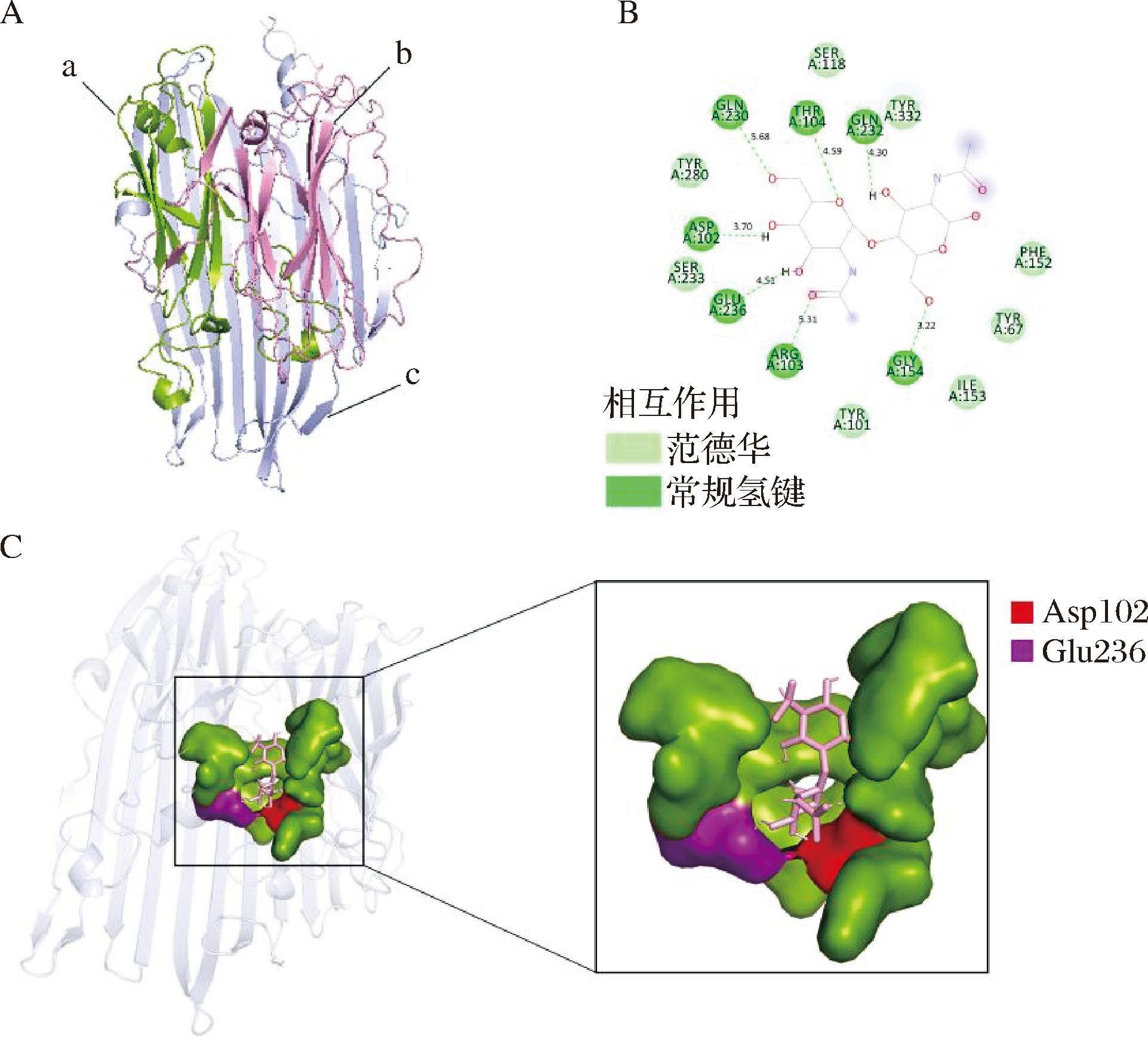
A-PNGase Dc蛋白的三级结构;B-相互作用图;C-对接效果图
图5 重组PNGase Dc与N,N′-二乙酰壳二糖的分子对接
Fig.5 Molecular docking of recombinant PNGase Dc and N,N′-diacetylchitobiose
N-糖链含有五糖核心结构(Man3GlcNAc2),PNGase Dc的识别位点为天冬酰胺残基与近端N-乙酰葡萄糖胺(GlcNAc)之间的酰胺键[19-20]。因此,为探究PNGase Dc与底物的结合模式,本研究选用N,N′-二乙酰壳二糖作为配体与PNGase Dc的三维结构模型进行分子对接。亲和力最强的结合位点的自由能为-7.5 kcal/mol,选择此构象进行分析。图5-C显示,PNGase Dc的活性口袋由结构域a和结构域b上的氨基酸构成。图5-B显示,Tyr67、Tyr102、Ser118、Phe152、Ile153、Ser233、Tyr280、Tyr332与底物N,N′-二乙酰壳二糖形成了范德华力,这些作用力使底物远离活性口袋,其中Ser233、Tyr280、Tyr332来自于结构域a,Tyr67、Tyr102、Ser118、Phe152、Ile153来自于结构域b。Asp102、Arg103、Thr104、Gly154、Gln232、Glu236、Gln230与底物N,N′-二乙酰壳二糖形成了氢键,这些氢键作用使底物更加紧密地靠近活性口袋内部,其中Gln232、Glu236、Gln230来自于结构域a,Asp102、Arg103、Thr104、Gly154来自于结构域b。Asp102和Glu236处于活性口袋最内部位置,Asp102和Glu236作为酸性氨基酸,侧链基团带有羧基基团呈现负电性,更易于N,N′-二乙酰壳二糖上的羟基结合而形成氢键。该结果进一步提高了对PNGase Dc活性位点预测的准确性。之后研究将Asp102和Glu236突变为丙氨酸并进行测活。
定点突变结果(图6)表明,突变体D102A和E236A分别作用于底物HRP时,二者HRP的蛋白条带均未发生迁移,这表明突变体D102A和E236A都失去了活性,无法酶解释放HRP上的N-链寡糖。因此Asp102和Glu236是PNGase Dc的关键活性位点。
注:M-蛋白 Marker;1-HRP;2-HRP与突变体D102 A的过夜 反应产物;3-HRP突变体E236 A的过夜反应产物
图6 SDS-PAGE检测PNGase Dc突变体的活性
Fig.6 Detection of activity of PNGase Dc mutants by SDS-PAGE
2.9 重组PNGase Dc的应用分析
将PNGase Dc应用于复杂样品中,以分析PNGase Dc的应用潜能以及对不同类型N-糖链的释放能力。小鼠血浆和拟南芥的糖组学背景清晰,这两者糖类化合物的种类、结构和功能等方面已得到广泛的研究和描述。因此,选取小鼠血浆和拟南芥作为底物进行研究。图7-A显示,PNGase Dc与PNGase A的色谱图一致,表明PNGase Dc以拟南芥为底物所释放的N-糖链类型与PNGase A相同。根据已有报道,拟南芥包含的N-链寡糖类型丰富,还含有核心α-1,3岩藻糖结构的N-糖链[21]。因此,PNGase Dc适用于酶解释放植物源糖蛋白上的N-糖链。图7-B显示,PNGase Dc与PNGase F色谱峰基本一致,表明PNGase Dc以小鼠血浆为底物所释放的N-糖链类型与PNGase F相同。多项研究表明,小鼠血浆中包含多种类型的N-糖链,包括高甘露糖型、杂合型和复杂型的结构[22-24]。因此,PNGase Dc适用于酶解释放动物源糖蛋白上的N-糖链。综上所述,PNGase Dc具有广泛的底物特异性,在动植物源糖蛋白的研究中具有一定的应用前景。

A-拟南芥N-链寡糖HPLC图谱;B-小鼠血浆N-链寡糖HPLC图谱
图7 重组PNGase Dc的应用分析
Fig.7 Application analysis of Recombinant PNGase Dc
3 结论与讨论
本研究从细菌Dyella caseinilytica中获得了一种编码PNGase的基因,并通过大肠杆菌原核表达系统成功实现了异源表达,得到了有活性的重组PNGase Dc。酶学特性结果表明,该酶的最适pH值为2.0,且在pH值为2.0~4.0均具有较高活性,最适温度为37 ℃,无金属离子依赖性,该酶在4 ℃冰箱中贮藏45 d时,仍能维持最高活性。相较于前人开发的PNGase H+[12],PNGase Dc在pH作用范围和稳定性两方面展现了更为优良的性能。在底物特异性方面,PNGase Dc对于植物来源的HRP及动物来源的牛乳铁蛋白均有良好的去糖基化效果;分子对接结果表明,PNGase Dc与底物N,N′-二乙酰壳二糖的相互作用力以氢键和范德华力为主,其中Asp102和Glu236处于口袋的最内部位置,这种结构可能有助于酶与底物的结合并发生催化作用。定点突变的结果最终验证了Asp102和Glu236是对PNGase Dc的催化活性起关键作用的氨基酸。将PNGase Dc应用于复杂样品,该结果进一步证实了PNGase Dc能够有效地酶解释放动植物源糖蛋白或糖肽上的N-糖链。综上所述,PNGase Dc具有良好的稳定性及广泛的pH作用范围,该酶结合了现有商业酶PNGase A与PNGase F的共同优势,能够在大肠杆菌中进行重组表达,并且可以有效释放动植物源糖蛋白上的N-链寡糖。
[1] SHI Z H, YIN B R, LI Y Q, et al. N-glycan profile as a tool in qualitative and quantitative analysis of meat adulteration[J]. Journal of Agricultural and Food Chemistry, 2019, 67(37):10543-10551.
[2] WANG X Y, ZHONG P Y, HUANG W Q, et al. Qualitative and quantitative mass spectrometry comparison of characteristic galactosyl lactose isomers from goat milk at different lactation stages[J]. Journal of Dairy Science, 2022, 105(9):7203-7215.
[3] LI C, TIAN Y, HAN J L, et al. An innovative method used for the identification of N-glycans on soybean allergen β-conglycinin[J]. Food Science and Human Wellness, 2023, 12(3):842-850.
[4] TAKAHASHI N, MURAMATSU T. CRC Handbook of Endoglycosidases and Glycoamidases [M]. CRC Press, 1992.
[5] PLUMMER T H Jr, ELDER J H, ALEXANDER S, et al. Demonstration of peptide: N-glycosidase F activity in endo-beta-N-acetylglucosaminidase F preparations[J]. The Journal of Biological Chemistry, 1984, 259(17):10700-10704.
[6] TRETTER V, ALTMANN F, M RZ L. Peptide-N4-(N-acetyl-beta-glucosaminyl)asparagine amidase F cannot release glycans with fucose attached alpha 1:: 3 to the asparagine-linked N-acetylglucosamine residue[J]. European Journal of Biochemistry, 1991, 199(3):647-652.
RZ L. Peptide-N4-(N-acetyl-beta-glucosaminyl)asparagine amidase F cannot release glycans with fucose attached alpha 1:: 3 to the asparagine-linked N-acetylglucosamine residue[J]. European Journal of Biochemistry, 1991, 199(3):647-652.
[7] TAKAHASHI N. Demonstration of a new amidase acting on glycopeptides[J]. Biochemical and Biophysical Research Communications, 1977, 76(4):1194-1201.
[8] ISHIMIZU T. Plant N-glycans and Their Degrading Enzymes[M]//Glycoscience: Biology and Medicine. Tokyo: Springer Japan, 2014:1111-1117.
[9] TAGA E M, WAHEED A, VAN ETTEN R L. Structural and chemical characterization of a homogeneous peptide N-glycosidase from almond[J]. Biochemistry, 1984, 23(5):815-822.
[10] ALTMANN F, SCHWEISZER S, WEBER C. Kinetic comparison of peptide: N-glycosidases F and A reveals several differences in substrate specificity[J]. Glycoconjugate Journal, 1995, 12(1):84-93.
[11] SUN G , YU X , BAO C ,et al.Identification and characterization of a novel prokaryotic peptide: N-glycosidase from Elizabethkingia meningoseptica.[J].Journal of Biological Chemistry, 2015, 290.DOI:10.1074/jbc.M114.605493.
[12] WANG T, CAI Z P, GU X Q, et al. Discovery and characterization of a novel extremely acidic bacterial N-glycanase with combined advantages of PNGase F and A[J]. Bioscience Reports, 2014, 34(6): e00149.
[13] WANG T, ZHENG S L, LIU L, et al. Development of a colorimetric PNGase activity assay[J]. Carbohydrate Research, 2019, 472:58-64.
[14] DU Y M, ZHENG S L, LIU L, et al. Analysis of N-glycans from Raphanus sativus cultivars using PNGase H[J]. Journal of Visualized Experiments: JoVE, 2018(136):57979.
[15] YANG B Y, GRAY J S, MONTGOMERY R. The glycans of horseradish peroxidase[J]. Carbohydrate Research, 1996, 287(2):203-212.
[16] STAUDACHER E, ALTMANN F, WILSON I B, et al. Fucose in N-glycans: From plant to man[J]. Biochimica et Biophysica Acta, 1999, 1473(1):216-236.
[17] WILSON I B H, ZELENY R, KOLARICH D, et al. Analysis of Asn-linked glycans from vegetable foodstuffs: Widespread occurrence of Lewis a, core α1, 3-linked fucose and xylose substitutions[J]. Glycobiology, 2001, 11(4):261-274.
[18] 王媛, 贾鹏, 李学俊, 等. 水稻N-糖酰胺酶基因(OsPNGase A)的克隆及在毕赤酵母中的分泌表达[J]. 生物工程学报, 2018, 34(3):421-428.
WANG Y, JIA P, LI X J, et al. Cloning of Oryza sativa N-glycanase gene(OsPNGase A) and its expression in Pichia pastoris[J]. Chinese Journal of Biotechnology, 2018, 34(3):421-428.
[19] MITRA N, SINHA S, RAMYA T N C, et al. N-linked oligosaccharides as outfitters for glycoprotein folding, form and function[J]. Trends in Biochemical Sciences, 2006, 31(3):156-163.
[20] SUZUKI T, PARK H, HOLLINGSWORTH N M, et al. PNG1, a yeast gene encoding a highly conserved peptide: N-glycanase[J]. The Journal of Cell Biology, 2000, 149(5):1039-1052.
[21] QIN S S, QIN S D, TIAN Z X. Comprehensive site- and structure-specific characterization of N-glycosylation in model plant Arabidopsis using mass-spectrometry-based N-glycoproteomics[J]. Journal of Chromatography. B, Analytical Technologies in the Biomedical and Life Sciences, 2022, 1198:123234.
[22] GU Y, HAN J, LIU X, et al. Dynamic alterations in serum IgG N-glycan profiles in the development of colitis-associated colon Cancer in mouse model[J]. Biochimica et Biophysica Acta. General Subjects, 2020, 1864(10):129668.
[23] GUSTAFSSON O J R, BRIGGS M T, CONDINA M R, et al. Raw N-glycan mass spectrometry imaging data on formalin-fixed mouse kidney[J]. Data in Brief, 2018, 21:185-188.
[24] OTAKI M, HIRANE N, NATSUME-KITATANI Y, et al. Mouse tissue glycome atlas 2022 highlights inter-organ variation in major N-glycan profiles[J]. Scientific Reports, 2022, 12(1):17804.