β-葡萄糖苷酶是在动物、植物、微生物中广泛存在的一种糖苷水解酶(glycoside hydrolysase, GH),其能够特异性的水解底物非还原末端的β-D-葡萄糖苷键释放葡萄糖和相应配基[1]。纤维素农业废弃物的有效利用对于产生可持续的生物燃料生产方法至关重要。纤维素降解为葡萄糖通常是由3种糖苷水解酶协同作用完成的,如内切葡聚糖酶(endoglucanase,EG, EC 3.2.1.4)、纤维素生物水解酶(cellulose biohydrolase,CBHs, EC 3.2.1.91)和β-葡萄糖苷酶(β-glucosidase,BGLs, EC 3.2.1.21)[2-3]。目前,真菌是纤维素酶的主要生产者,也有很多研究构建了相应的适用于工业生产的菌株[4-6]。然而,正如之前报道所述,内源性β-葡萄糖苷酶的缺乏一直是目前用于纤维素降解的工业微生物的致命缺陷,如里氏木霉作为纤维素酶的主流生产菌株,可以同时生产上述3种纤维素酶,但该菌株生产的β-葡萄糖苷酶只占这3种酶总酶量的1%[7-8]。纤维素二糖既是纤维素降解的中间产物,同样也是β-葡萄糖苷酶的天然底物,然而β-葡萄糖苷酶的活性较低,导致纤维素二糖积累。然而,纤维素二糖的积累会显著抑制CBHs的催化活性,因此β-葡萄糖苷酶是纤维素降解过程中的重要的限速酶[9-10]。所以,高酶活的β-葡萄糖苷酶对于提高纤维素农业废弃物的利用效率至关重要[11-12]。
β-葡萄糖苷酶主要催化水解各种双糖、低聚糖以及烷基和芳基β-d-葡萄糖苷中的β-1,4-糖苷键,根据其氨基酸序列将其分为GH家族1、3、5、9、30和116[13-14]。不同的β-葡萄糖苷酶在酶的性质上有很大的不同,比如反应的最佳条件、底物的特异性、催化效率、对不利条件的耐受性和对葡萄糖的抑制常数。目前大多数用于纤维素水解的微生物β-葡萄糖苷酶属于GH家族3[15]。其中,bgl3A是一种来自该家族的重要的纤维素水解酶,具有较好的热稳定性,已得到广泛研究。目前已有研究在大肠杆菌宿主表达bgl3A,但是酶活力较低[16]。巴斯德毕赤酵母具有在翻译后修饰、分泌表达、高密度培养及产物纯化回收等方面均展现了诸多优势,XIA等[17]用毕赤酵母表达bgl3A,但研究重点探究糖基化对酶的影响,而缺乏对该酶分泌表达策略的探究。
由于β-葡萄糖苷酶在生物燃料工业中的实际效益,已经有很多研究来改善现有GH3酶的酶学性质。分子对接技术已广泛用于构建酶-底物的复合物模型,用于预测结合模式和催化机制的探究[18]。XIA等[16]通过分子对接和饱和突变,获得了催化活性上升的bgl3A突变体,但该研究仅以活性位点距离为依据设计突变体,这使得突变及筛选的工作量大幅增加。结合能扫描也是目前常用的改造酶活力的策略,它可以提前预测结合能的变化,使突变的选择更理性,且成功实现多种工业酶的活性改造[19-20]。因此本研究基于柔性对接和结合能扫描的策略,有望为β-葡萄糖苷酶的活性提升提供新的靶点。
本研究基于柔性对接和结合能分析对bgl3A的酶活力进行改造,并使用毕赤酵母重组表达。首先,通过Discovery Studio 2022(DS)柔性对接模块获得了bgl3A和底物pNPG的复合物结构。其次,通过DS的结合能预测模块对所选择的结合能扫描评分前20的残基进行模拟饱和突变,筛选结合能显著下降的bgl3A突变体。对上述突变体进行表达、纯化和酶学性质表征。接下来,基于分子动力学模拟解析了突变体酶活提升的机制。最后,最优突变体使用毕赤酵母进行异源表达,通过信号肽优化和分子伴侣共表达提高了重组菌的酶活力。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
毕赤酵母(Komagatella phaffii )GS115、大肠杆菌(Escherichia coli) JM109、大肠杆菌(Escherichia coli)BL21(DE3)由实验室保藏,质粒 pET-28a(+)、pPIC9K由研究室保藏,质粒pET28a(+)/bgl3A由上海生工合成。
1.1.2 主要试剂
柱回收试剂盒、蛋白质marker,Thermo公司;质粒提取试剂盒、大肠杆菌感受态制备试剂盒购自上海生工公司,一步克隆试剂盒、PrimerSTAR Max DNA聚合酶,Sal I快切酶、Sac I 消化酶均从TaKaRa公司购得;无氨基酵母氮源(不含氨基酸)、卡纳青霉素、异丙基-β-D-硫代半乳糖苷(isopropyl β-D-thiogalactoside,IPTG),MCE公司;蛋白胨和酵母粉,Oxoid公司;Bradford蛋白浓度检测试剂盒,碧云天公司;蛋白电泳loading buffer和Bis-Tris预制凝胶,Invitrogen公司。其他试剂均购自MCE公司。
1.1.3 培养基及缓冲液
LB液体培养基(g/L):蛋白胨10,酵母粉5,NaCl 10。
TB培养基(g/L):蛋白胨12,酵母粉24,KH2 PO4·3H2O 16.4,KH2 PO4 2.31,甘油5。
YPD培养基(g/L):酵母粉10、蛋白胨20、葡萄糖20。固体培养基在液体培养基基础上添加2%琼脂粉。
BMGY培养基(g/L):酵母粉10、蛋白胨20、KH2PO4 3.01、K2HPO4 11.81、生物素4×10-4、甘油10、YNB 13.4。
BMMY培养基(g/L):酵母粉10、蛋白胨20、KH2PO4 3.01、K2HPO4 11.81、生物素4×10-4、YNB 13.4、甲醇10。
MD固体培养基(g/L):YNB 13.4、葡萄糖20,在此基础上添加1.5%~2%琼脂粉。
pH 5.0的乙酸-乙酸钠缓冲液配制:0.2 mol/L醋酸:11.55 mL冰醋酸定容1 000 mL。0.2 mol/L醋酸钠:16.4 g无水醋酸钠溶于1 000 mL水中。0.2 mol/L醋酸6.8 mL与0.2 mol/L醋酸钠43.2 mL,混匀,测定pH值。
500 μmol/L的pNP溶液:对硝基苯酚13.91 g溶于1 000 mL pH 5.0的乙酸-乙酸钠缓冲液中,继续用缓冲液稀释2 000倍。
1 mol/L Na2CO3溶液:105.99 g Na2CO3溶于1 000 mL水中。
10 mmol/L对硝基苯基-β-D-葡萄糖苷(4′-nitrophenyl-beta-D-glucopyranoside,pNPG)溶液:10 mmol 3.013 g pNPG溶于1 000 mL pH 5.0的乙酸-乙酸钠缓冲液中。
1.2 实验方法
1.2.1 bgl3A及突变体表达载体的构建
研究共选择20个突变位点,通过单点突变的方式进行质粒构建。以pET28a(+)/bgl3A为模板,突变的引物如表1所示,利用DNA聚合酶PrimerStar Max进行PCR。对PCR产物进行柱回收,将回收后的片段稀释合适浓度后,转入E.coli JM109感受态中,37 ℃培养12~16 h。挑取转化子进行测序验证。测序正确的质粒转入E.coli BL21(DE3)获得重组菌。
表1 本研究使用的引物
Table 1 Primers used in this study
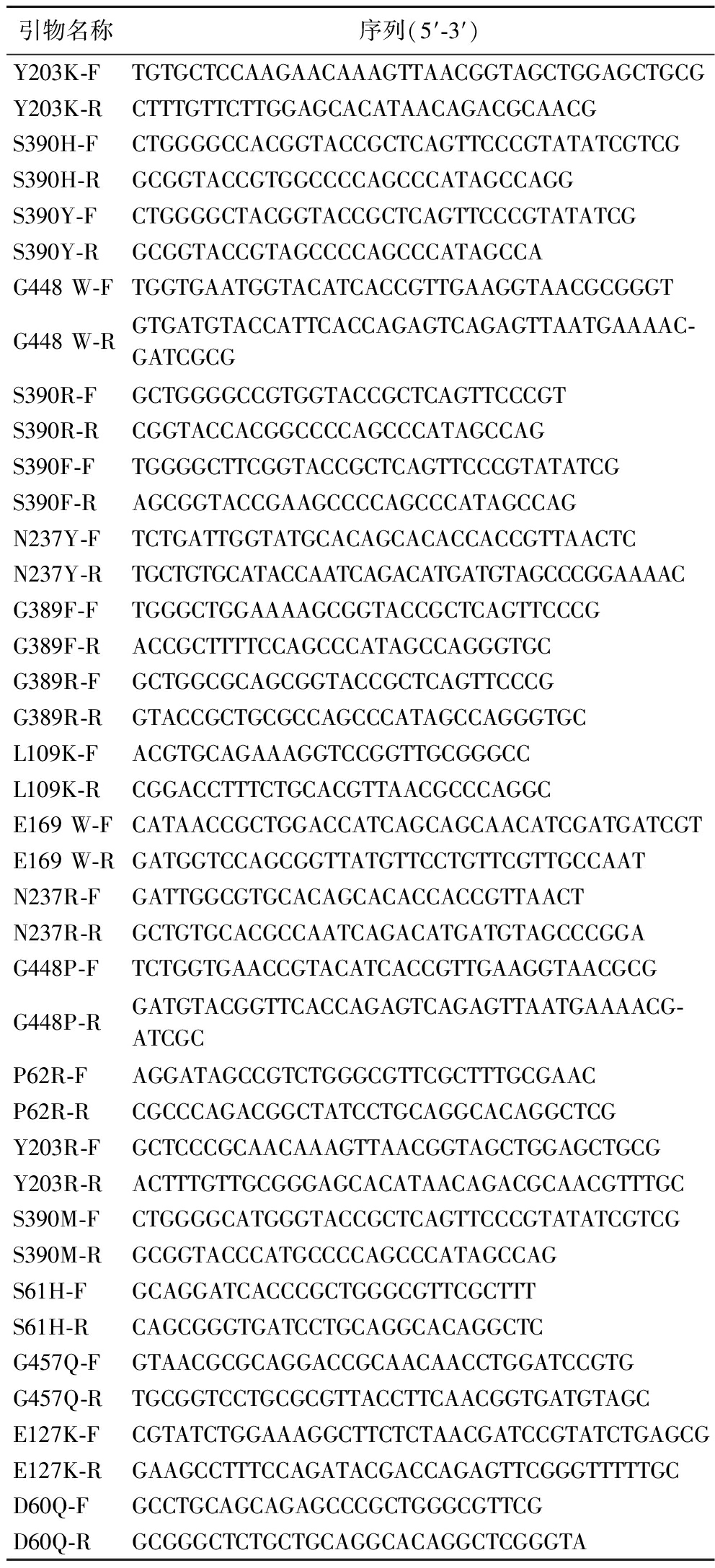
引物名称序列(5′-3′)Y203K-FTGTGCTCCAAGAACAAAGTTAACGGTAGCTGGAGCTGCGY203K-RCTTTGTTCTTGGAGCACATAACAGACGCAACGS390H-FCTGGGGCCACGGTACCGCTCAGTTCCCGTATATCGTCGS390H-RGCGGTACCGTGGCCCCAGCCCATAGCCAGGS390Y-FCTGGGGCTACGGTACCGCTCAGTTCCCGTATATCGS390Y-RGCGGTACCGTAGCCCCAGCCCATAGCCAG448 W-FTGGTGAATGGTACATCACCGTTGAAGGTAACGCGGGTG448 W-RGTGATGTACCATTCACCAGAGTCAGAGTTAATGAAAAC-GATCGCGS390R-FGCTGGGGCCGTGGTACCGCTCAGTTCCCGTS390R-RCGGTACCACGGCCCCAGCCCATAGCCAGS390F-FTGGGGCTTCGGTACCGCTCAGTTCCCGTATATCGS390F-RAGCGGTACCGAAGCCCCAGCCCATAGCCAGN237Y-FTCTGATTGGTATGCACAGCACACCACCGTTAACTCN237Y-RTGCTGTGCATACCAATCAGACATGATGTAGCCCGGAAAACG389F-FTGGGCTGGAAAAGCGGTACCGCTCAGTTCCCGG389F-RACCGCTTTTCCAGCCCATAGCCAGGGTGCG389R-FGCTGGCGCAGCGGTACCGCTCAGTTCCCGG389R-RGTACCGCTGCGCCAGCCCATAGCCAGGGTGCL109K-FACGTGCAGAAAGGTCCGGTTGCGGGCCL109K-RCGGACCTTTCTGCACGTTAACGCCCAGGCE169 W-FCATAACCGCTGGACCATCAGCAGCAACATCGATGATCGTE169 W-RGATGGTCCAGCGGTTATGTTCCTGTTCGTTGCCAATN237R-FGATTGGCGTGCACAGCACACCACCGTTAACTN237R-RGCTGTGCACGCCAATCAGACATGATGTAGCCCGGAG448P-FTCTGGTGAACCGTACATCACCGTTGAAGGTAACGCGG448P-RGATGTACGGTTCACCAGAGTCAGAGTTAATGAAAACG-ATCGCP62R-FAGGATAGCCGTCTGGGCGTTCGCTTTGCGAACP62R-RCGCCCAGACGGCTATCCTGCAGGCACAGGCTCGY203R-FGCTCCCGCAACAAAGTTAACGGTAGCTGGAGCTGCGY203R-RACTTTGTTGCGGGAGCACATAACAGACGCAACGTTTGCS390M-FCTGGGGCATGGGTACCGCTCAGTTCCCGTATATCGTCGS390M-RGCGGTACCCATGCCCCAGCCCATAGCCAGS61H-FGCAGGATCACCCGCTGGGCGTTCGCTTTS61H-RCAGCGGGTGATCCTGCAGGCACAGGCTCG457Q-FGTAACGCGCAGGACCGCAACAACCTGGATCCGTGG457Q-RTGCGGTCCTGCGCGTTACCTTCAACGGTGATGTAGCE127K-FCGTATCTGGAAAGGCTTCTCTAACGATCCGTATCTGAGCGE127K-RGAAGCCTTTCCAGATACGACCAGAGTTCGGGTTTTTGCD60Q-FGCCTGCAGCAGAGCCCGCTGGGCGTTCGD60Q-RGCGGGCTCTGCTGCAGGCACAGGCTCGGGTA
使用一步克隆法完成目的基因bgl3A、bgl3A-N237Y和载体pPIC9K的连接,测序验证。将重组载体pPIC9K/bgl3A-N237Y、pPIC9K/bgl3A载体线性化后通过电转方式转入毕赤酵母GS115感受态中,经验证得到正确的重组菌。
不同分子伴侣的表达质粒由本实验室构建[21],用SacI酶进行线性化其次将线性化后的质粒通过电转方式转入毕赤酵母GS115感受态中,涂布100 μg/mL博来霉素抗性平板,经验证得到正确的重组菌。共表达菌株转化子后转接至高浓度博来霉素抗性平板进行复筛,博来霉素工作浓度为1 000 μg/mL。
1.2.2 bgl3A及突变体的表达
将带有正确质粒的大肠杆菌划线与相应抗性平板上活化,将重组菌株单菌落接种于卡那霉素终浓度为50 μg/mL的LB培养基,于37 ℃培养10 h。以2%的接种量将上述培养物接种TB培养基,于37 ℃培养至OD600≈0.6~0.8后加入IPTG(终浓度为0.3 mmol/mL),并于20 ℃,220 r/min诱导培养24 h。
将毕赤酵母的重组菌株划线于YPD平板活化,后挑取YPD平板上的单菌落接种于YPD液体培养基,30 ℃ 培养 18~22 h,以2%的转接量转接至BMGY培养基中,30 ℃,220 r/min培养至OD600≈0.4~0.6后,离心收集酵母细胞,使用生理盐水洗涤细胞2次。最后用BMMY培养基将菌体重悬,30 ℃培养132 h,每24 h添加1%的甲醇。
1.2.3 bgl3A及突变体的纯化
将发酵液在4 ℃,10 000 r/min下离心10 min去除上清液,用缓冲液重悬细胞后,用高压均质机破碎细胞,后12 000 r/min,4 ℃下离心20 min。收集离心后的上清液进行镍柱亲和层析。使用ATKA蛋白纯化仪器,进样完成后先用A液(50 mmol/L Tris-HCl,0.5 mol/L氯化钠,pH 7.6)平衡,再用不同浓度的B液(50 mmol/L Tris-HCl,0.5 mol/L氯化钠,0~300 mmol/L咪唑,pH 7.6)洗脱目的蛋白。
1.2.4 SDS-PAGE分析和蛋白浓度测定
将发酵液8 000 r/min,4 ℃离心10 min后去除上清液,细胞用等体积PBS溶液重悬后,采用高压均质机在90 MPa的压力下破碎细胞悬液3 min,离心收集上清液。蛋白样品与上样缓冲液按照3∶1的体积比混合,95 ℃加热10 min,在恒压120 V下进行SDS-PAGE。蛋白浓度采用碧云天Bradford蛋白浓度测定试剂盒。
1.2.5 bgl3A酶活力测定
酶活力的定义为:50 ℃和pH 5.0下,1 mL酶液在1 min内水解pNPG底物产生1 μmol pNP定义为一个酶活力单位。
酶活力测定:取一定稀释倍数的125 μL的酶液,加入125 μL 10 mmol/L pNPG(使用0.2 mol/L,pH 5.0 的乙酸-乙酸钠缓冲液配制),于70 ℃水浴中准确反应10 min,加入0.75 mL 1 mol/L Na2CO3溶液以终止反应,用Synergy NEO2 HTS全功能酶标仪测定OD400的吸光度。
1.2.6 突变体的动力学参数测定
配制0、0.4、0.8、1.2、1.6、1.8、2.4、2.8、3.2、3.6、4、5、10 mmol/L的pNPG,测定不同底物浓度下bgl3A及突变体的酶活力。利用Origin 2019b进行非线性拟合,计算Km和kcat。
1.2.7 生物信息分析
采用ColabFold构建bgl3A及其突变体模型,并使用Rosetta_Relax进行侧链构象优化,使用Rosetta Cartesian_ddG预测bgl3A及其突变体结合自由能。
动力学分析:用GROMACS-2020[22]程序进行动力学分析:用程序在300 K条件下进行MD模拟,力场为FF14 sb[23],时间为50 ns。建模系统采用SPC/E建立水模型的立方盒子,其中溶剂边缘到酶的最小距离为12 Å。然后,通过添加的Cl-和Na+对系统进行电荷中和。并采用最陡下坡法对系统进行能量最小化;等容等温系综和等温等体积系综各进行200 ps后解除限制。计算模拟过程中均方根偏差(root mean square deviation,RMSD)和均方根波动(root mean square fluctuation,RMSF),并选择最终平衡构象。
2 结果与分析
2.1 bgl3A在大肠杆菌中的初步表达及纯化
本研究中,首先构建了bgl3A的大肠杆菌重组菌株,将bgl3A基因克隆至pET28a中,得到重组表达质粒pET28a-bgl3A(图1-a)。上述表达质粒序列均经上海生工测序验证。
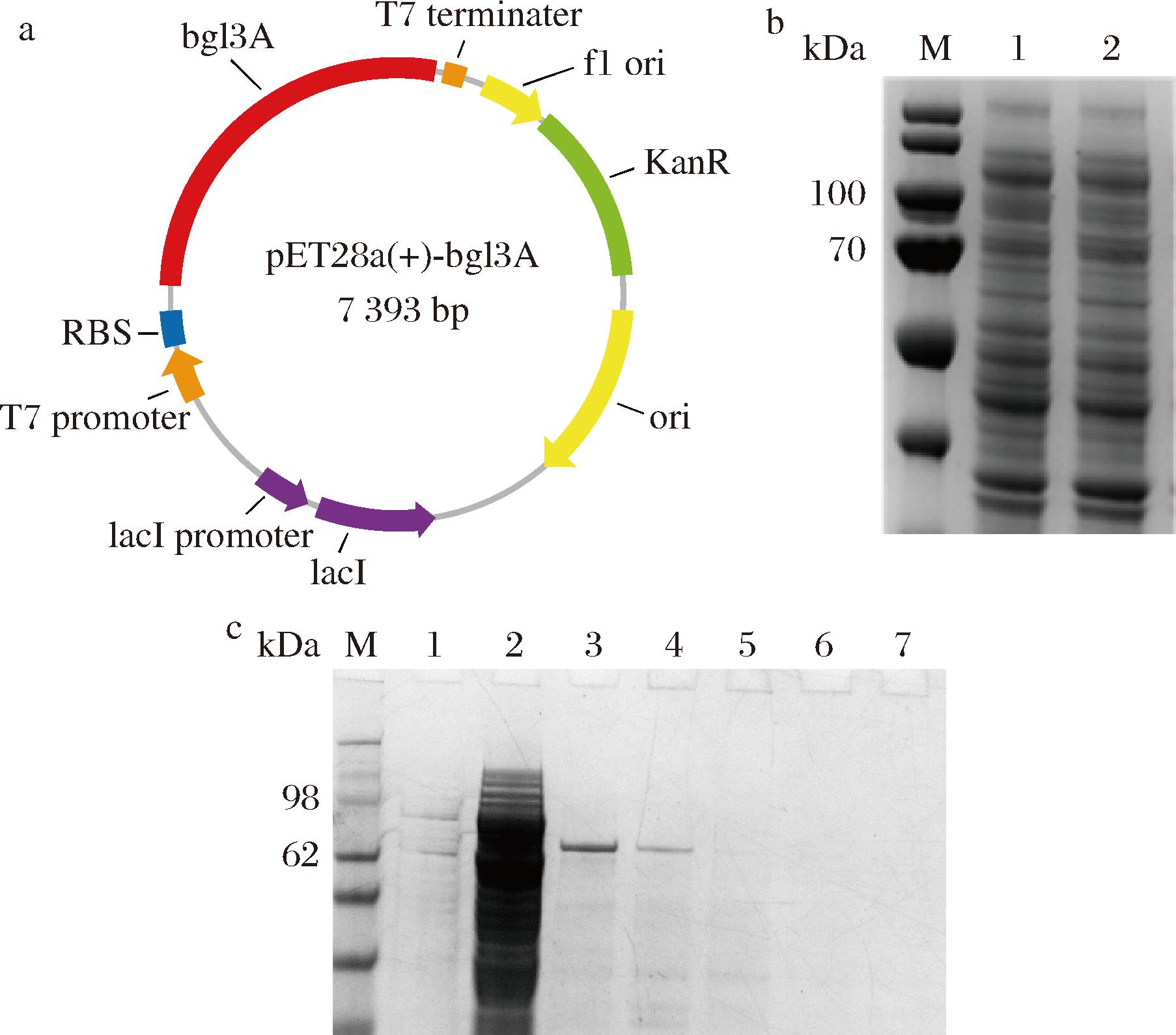
a-pET28a-bgl3A质粒示意图;b-诱导24 h后BL21/pET28a和 BL21/pET28a-bgl3A的胞内可溶部分SDS-PAGE分析(M-Marker; 1-BL21/pET28a胞内可溶部分;2-BL21/pET28a-bgl3A胞内可溶部分); c-不同浓度B液洗脱出的蛋白样品SDS-PAGE验证(M-Marker; 1-5% B液洗脱;2-10% B液洗脱;3-20% B液洗脱;4-40% B 液洗脱;5-60% B 液洗脱;6-80% B液洗脱;6-100% B液洗脱)
图1 重组bgl3A的表达情况分析和SDS-PAGE分析
Fig.1 Expression analysis and SDS-PAGE analysis of recombinant bgl3A
为探究bgl3A的表达情况,测定了重组菌发酵24 h后的发酵液上清液、胞内可溶部分和不溶部分的酶活,并对胞内可溶部分进行SDS-PAGE分析(图1-b)。结果显示,重组菌BL21/pET28a-bgl3A仅胞内可溶部分可以检测到bgl3A酶活力,为2.7 U/mL,而空载质粒菌株的发酵液上清液、胞内可溶部分和不溶部分均检测不到酶活力。重组菌发酵液经纯化后可观察到明显条带(理论分子质量为76.3 kDa)(图1-c)。这些结果表明,bgl3A在E.coli BL21(DE3)中实现可溶性表达。将重组菌BL21/pET28a-bgl3A的胞内可溶部分进行镍柱亲和层析,分别用5%、10%、20%、40%、60%、80%、100% B液进行梯度洗脱,收集洗脱样品并进行SDS-PAGE分析。如图1-c所示,10% B液去除杂蛋白,20% B液洗脱出目标蛋白,以上结果表明,镍柱亲和层析可以获得单一的目标蛋白。
2.2 突变体的设计
采用ColabFold构建bgl3A模型结构。同时使用Rosetta_Relax进行侧链优化,基于DS的柔性对接模块构建了bgl3A和pNPG的复合物结构(图2-a),随后通过结合能预测模块对的活性位点Trp36、Arg66、Arg168(图2-b)6 Å以内的残基模拟饱和突变。最后,选择预测结合能下降排行在前20个突变体(图2-c)。基于构建的E.coil表达系统,表达上述20个突变体。
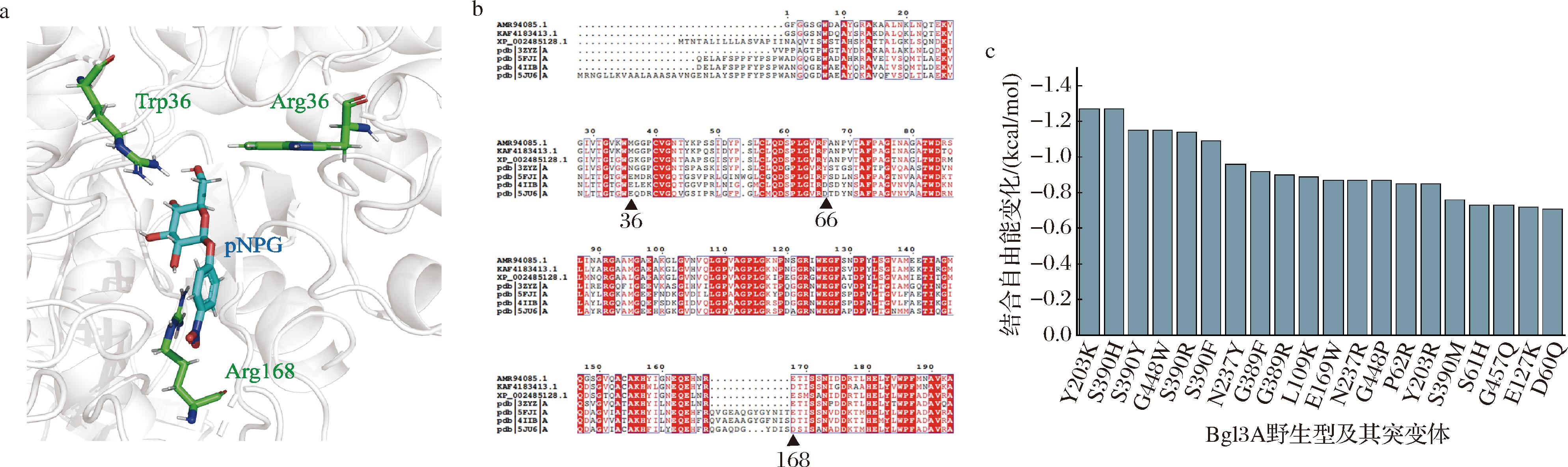
a-同源建模的可视化结构;b-bgl3A的多序列比对(活性位点由蓝色箭头标出);c-突变体预测结合能排序
图2 突变体设计与突变选点依据
Fig.2 Mutant design and mutation site selection criteria
2.3 野生型bgl3A及其突变体的催化性能
如图3-a、图3-b所示,除了E169 W以外的突变体均被纯化为单一蛋白条带。对纯化后的bglA及其突变体进行酶学性质表征。在野生型和所有突变型酶的最佳条件下,对于底物pNPG,bgl3A-N237Y突变体Km值的减少了16.7%,kcat/Km上升了25%,比酶活提升了23%(图3-c),bgl3A-N237Y突变体的底物亲和力提升了(表2)。在20个突变体中,仅bgl3A-N237Y对pNPG的催化效率提高了25%,其余突变体的催化效率均有不同程度的降低。bgl3A-N237Y的kcat值相较于野生型的提升并不明显(表2),但突变体Km值降低,经计算N237Y突变体的催化效率仍有一定的提升。证实突变体bgl3A-N237Y对pNPG的底物亲和力和催化效率高于野生型bgl3A。

a、b-bgl3A及突变体纯化后SDS-PAGE验证;c-CALB及突变体纯化后的比酶活
图3 bgl3A及突变体的SDS-PAGE分析及比酶活
Fig.3 SDS-PAGE analysis and specific enzyme activity of bgl3A and its mutants
注:对样本之间的差异采用Excel(Microsoft Office 365)中的Student′s t-test进行评估;**P<0.01。
表2 bgl3A和bgl3A-N237Y的性质比较
Table 2 Properties comparison of bgl3A and bgl3A-N237Y
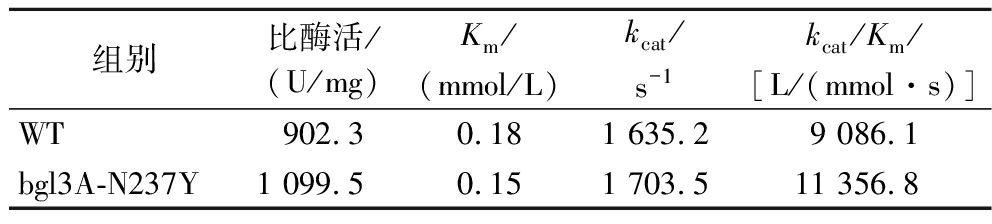
组别比酶活/(U/mg)Km/(mmol/L)kcat/s-1kcat/Km/[L/(mmol·s)]WT902.30.181 635.29 086.1bgl3A-N237Y1 099.50.151 703.511 356.8
2.4 bgl3A-N137Y突变体酶活提升机制解析
基于bgl3A野生型-底物复合物预测结构,通过Pymol的mutagenesis模块构建突变体bgl3A-N237Y-底物的复合物结构模型,并在300 K的条件下进行分子动力学模拟。结果显示,bgl3A-N237Y突变体的平均(0.121 nm)和最终(0.124 nm)RMSD值与bgl3A的平均(0.103 nm)和最终(0.098 nm)RMSD值相比有所增加(图4-a),表明酶整体的柔性增加。相较于bgl3A野生型,N237Y突变体的Trp36、Phe259、Tyr449、Arg66和Arg168残基RMSF值上升。值得注意的是,这些残基空间位置上靠近结合位点(Trp36、Phe259、Tyr449、Arg66和Arg168),可能降低底物进入活性口袋的空间位阻(图4-b)。有研究显示,活性中心附近残基柔性增加导致催化活性提高[21]。因此,酶口袋区域柔性的增加可能是提高该酶的催化效率重要原因之一。
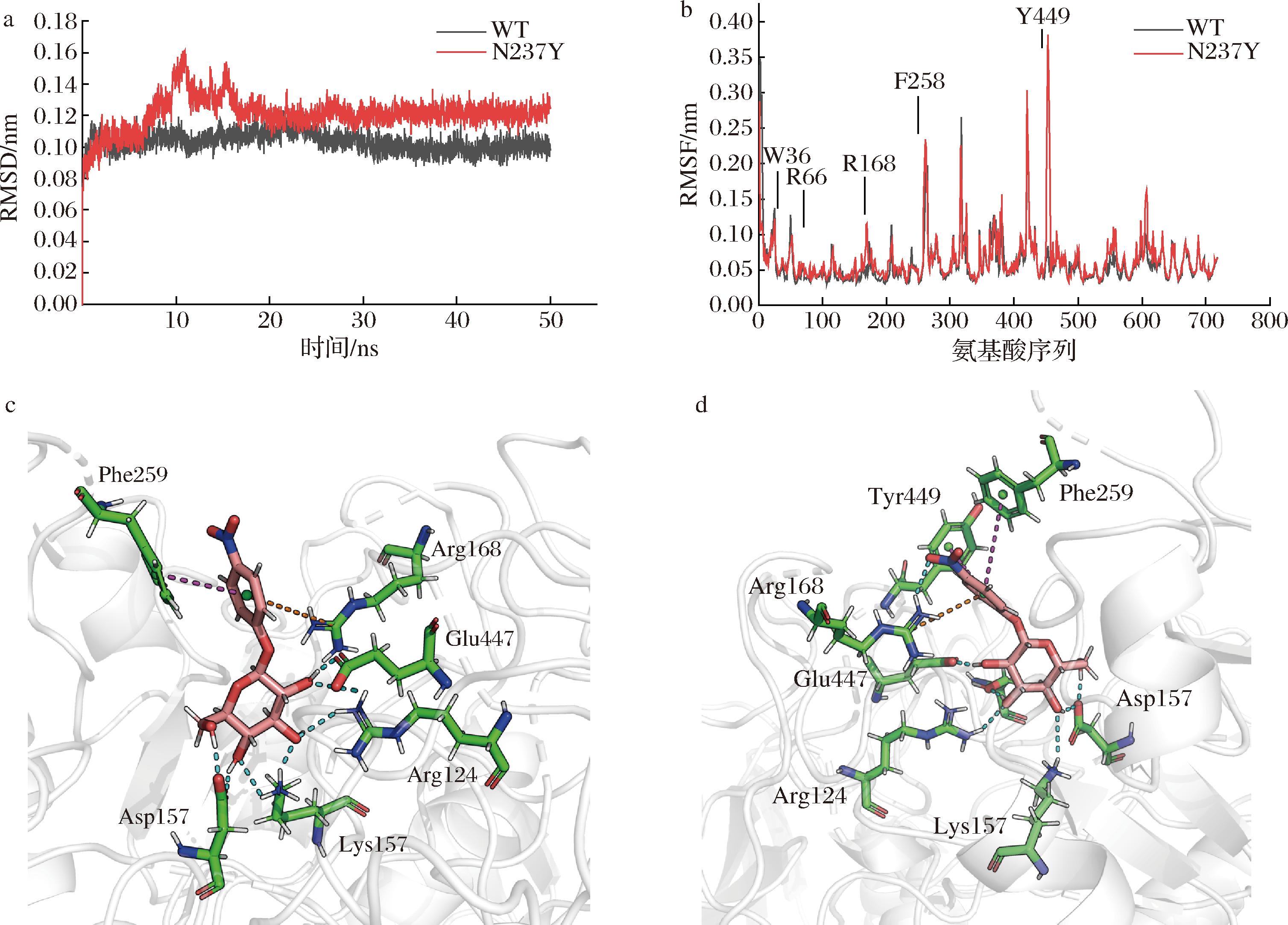
a-RMSD分析;b-RMSF分析;c-bgl3A和pNPG相互作用力;d-bgl3A-N237Y和pNPG相互作用力
图4 bgl3A和bgl3A-N237Y的RMSD和RMSF分析及突变位点的相互作用力变化
Fig.4 RMSD and RMSF analysis of bgl3A and bgl3A-N237Y and interaction changes in mutation sites
本研究对分子动力学模拟最后的20 ns进行聚类,并提取了占比最高的构象进行局部作用力分析(图4-c、图4-d)。结果显示,bgl3A-N237Y突变体生成了1个新的芳香相互作用。有研究显示,新增的芳香相互作用有利于底物和酶的相互作用从而提高酶活[16]。因此,新的芳香相互作用力的形成有助于酶从溶剂中捕捉底物,从而导致酶催化活性的提高。
2.5 bgl3A及突变体在毕赤酵母的表达及优化
2.5.1 bgl3A及突变体在毕赤酵母宿主表达
为提升酶的表达量,将基因bgl3A、bgl3A-N237Y和载体pPIC9K连接(图5-a),将重组载体pPIC9K/bgl3A、pPIC9K/bgl3A-N237Y载体经过sal I酶线性化后电转入毕赤酵母GS115。将重组菌甲醇诱导表达96 h后测定发酵液上清液的酶活力,并进行SDS-PAGE分析。重组菌GS115/bgl3A-N237Y的胞外上清中酶活力为62.78 U/mL,相较于重组菌GS115/bgl3A酶活力提升了27%,可观察到明显条带(图5-b)。这些结果表明,bgl3A、bgl3A-N237Y可在毕赤酵母中实现分泌表达,且突变带来了酶活力的提升,经测定,在摇瓶发酵水平。
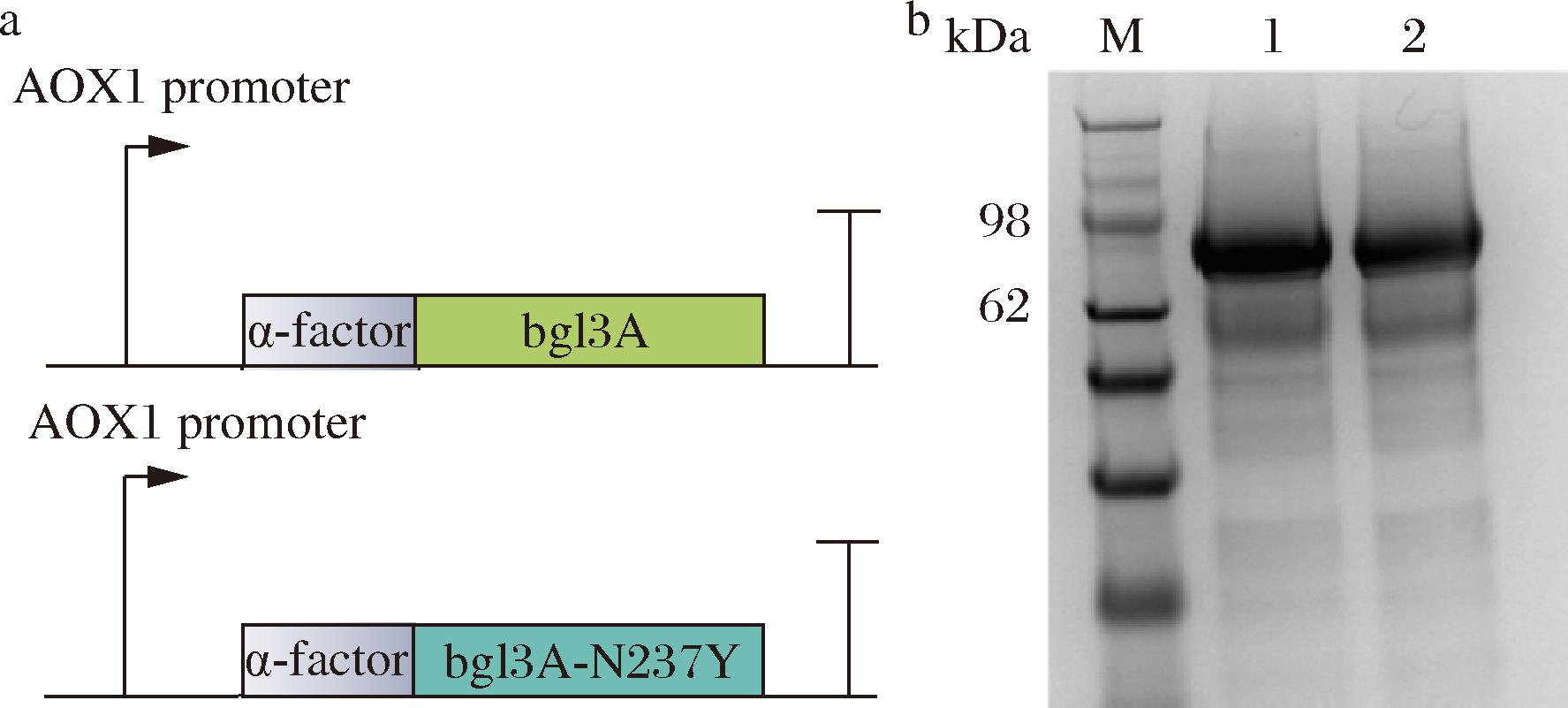
a-bgl3A及突变体的表达框;b-GS115/bgl3A和GS115/ bgl3A-N237Y的胞外上清液部分SDS-PAGE分析(M-Marker; 1-GS115/bgl3A胞外上清液;2-GS115/bgl3A-N237Y胞外上清液)
图5 bgl3A及突变体在毕赤酵母里分泌表达的 SDS-PAGE分析
Fig.5 SDS-PAGE analysis of secretory expression of bgl3A and mutants in Pichia pastoris
2.5.2 信号肽替换提高bgl3A的分泌效率
分泌信号肽通常位于N末端并包含3个部分,决定新生多肽向内质网的易位和分泌到细胞外培养基中的转移。在过去研究里,除了常用的酿酒酵母α因子前体肽,很少有已知的信号肽序列被表征并应用于异源蛋白的分泌表达。经文献调研分析,选择4条已报道分泌蛋白效率优于信号肽α-factor,即chr3[24]、pho1[25]、exg1[26]以及mab2[27]作为候选信号肽并对其分泌bgl3A的效果进行表征(图6-a)。结果表明信号肽chr3、pho1、exg1以及mab2均可以实现bgl3A胞外分泌(图6-b),其中用Pho1信号肽引导蛋白分泌的重组菌酶活力最高,120 h达到79.3 U/mL(图6-c),相较于用信号肽α-factor引导分泌的菌株,发酵上清液酶活力提升了29%。
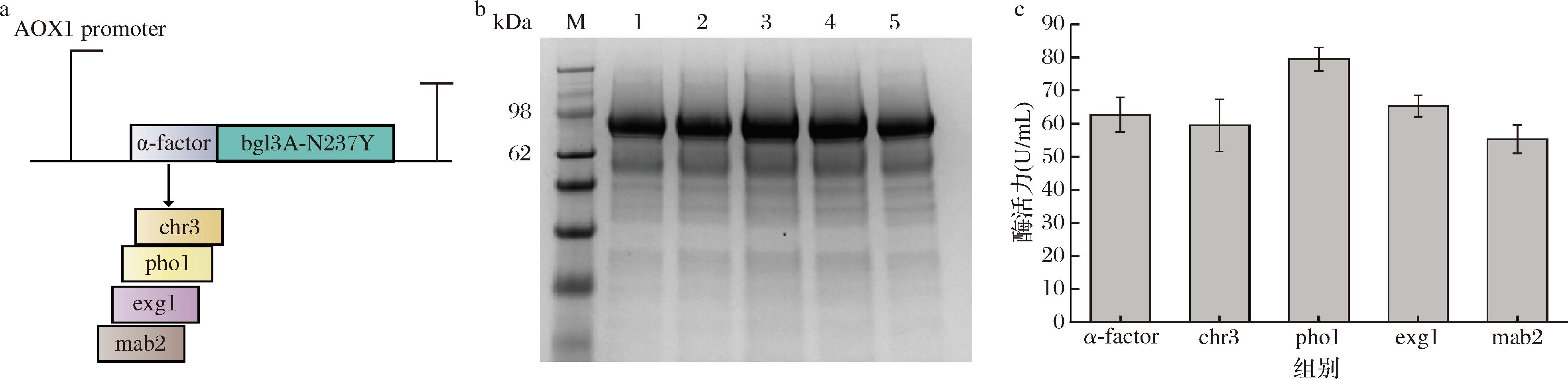
a-替换了不同信号肽的表达框;b-胞外上清液部分SDS-PAGE分析(M-Marker;泳道1~5重组菌的上清分别为α-bgl3A、chr3-bgl3A、 Pho1-bgl3A、exg1-bgl3A和mab2-bgl3A);c-不同信号肽影响蛋白分泌效率
图6 信号肽替换对bgl3A分泌表达的影响
Fig.6 The effect of signal peptide replacement on the secretory expression of bgl3A
注:蓝色虚线:氢键;紫色虚线:pi-pi芳香相互作用;黄色虚线:pi作用力。
2.5.3 蛋白质折叠分子伴侣共表达提升bgl3A分泌表达
异源蛋白的表达会给酵母细胞带来负担和压力[28],使蛋白在内质网积累并引发未折叠蛋白的反应(unfolded protein response,UPR),由此触发一系列下游基因的表达,包括协助折叠的分子伴侣和辅助蛋白降解等蛋白[29]。通过调控相关蛋白,减少错误折叠积累,辅助目标蛋白折叠,提升分泌效率。因此,我们选择内质网上辅助蛋白折叠的分子伴侣进行共表达(表3),分子伴侣表达框如图7-a所示,以达到缓解异源蛋白表达带来的内质网压力,进而实现目标蛋白分泌表达量进一步提高。
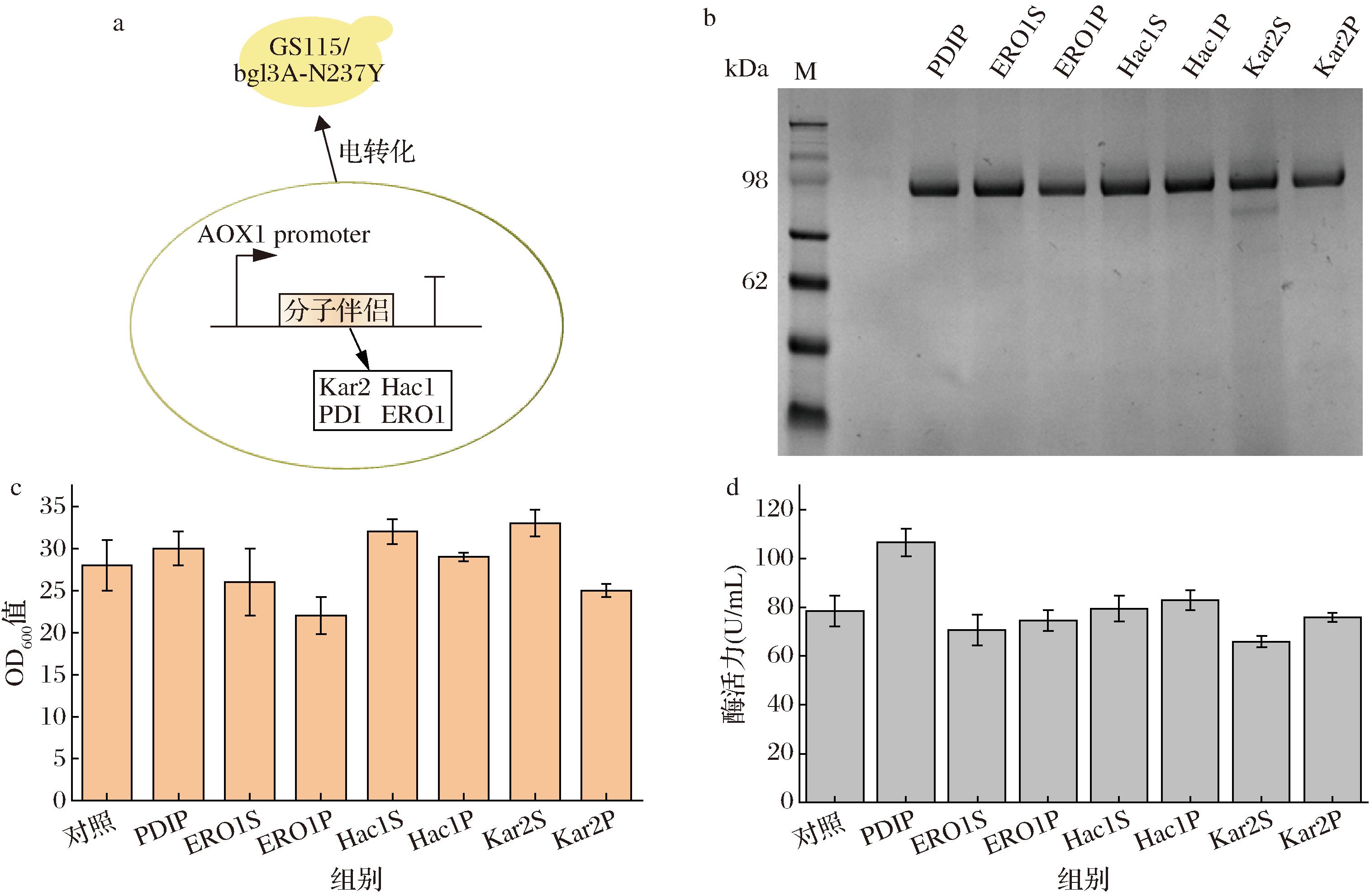
a-不同分子伴侣的表达框;b-胞外上清部分SDS-PAGE分析(M-Marker);c-不同分子伴侣菌株摇瓶发酵生长情况;d-不同分子伴侣菌株的酶活力
图7 共表达分子伴侣对bgl3A分泌表达的影响
Fig.7 The effect of co-expression of molecular chaperones on the secretion and expression of bgl3A
表3 本研究过表达的分子伴侣
Table 2 The overexpressed molecular chaperones in this study
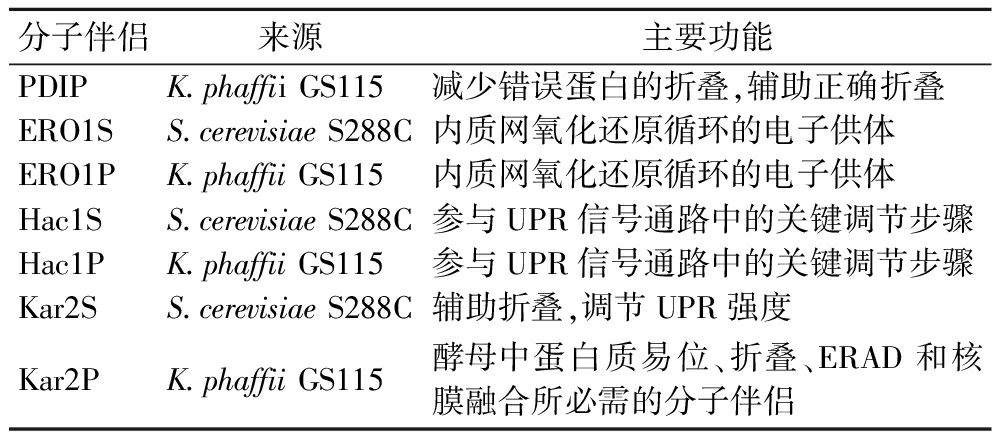
分子伴侣来源主要功能PDIPK.phaffii GS115 减少错误蛋白的折叠,辅助正确折叠ERO1SS.cerevisiae S288C内质网氧化还原循环的电子供体ERO1PK.phaffii GS115内质网氧化还原循环的电子供体Hac1SS.cerevisiae S288C参与UPR信号通路中的关键调节步骤Hac1PK.phaffii GS115参与UPR信号通路中的关键调节步骤Kar2SS.cerevisiae S288C辅助折叠,调节UPR强度Kar2PK.phaffii GS115酵母中蛋白质易位、折叠、ERAD和核膜融合所必需的分子伴侣
注:ERAD:内质网关联降解(endoplasmic reticulum associated degradation)。
通过摇瓶发酵的结果表明,分子伴侣共表达菌株均能实现bgl3A的分泌表达(图7-b)。发酵过程中仅有分子伴侣ERO1共表达引起了菌体OD600的下降,而其他分子伴侣共表达菌株菌体OD600并没明显变化,这说明其他伴侣共表达没有影响菌体的正常生长(图7-c)。共表达Hac1的菌株的酶活力并没有明显的提升,可能是因为重组蛋白表达没有诱发菌株的UPR响应。ERO1和Kar2的共表达菌株的酶活有不同程度的下降(图7-d),可能过表达ERO1破坏了内质网上氧化还原反应的平衡;而过量表达Kar2有可能会抑制分子伴侣活性,导致部分目标蛋白通过内质网降解途径被降解,不利于异源蛋白的表达。共表达PDI使bgl3A酶活力提高到106.6 U/mL(较出发菌株提高了35%)。我们推测PDI通过2种不同方式提高了bgl3A的胞外酶活力:(1)在折叠过程的早期阶段,阻止了错误折叠中间体的聚集,并在一定程度上促进链的折叠;(2)当肽链已经折叠到一定程度时,它又发挥异构酶的功能来辅助折叠,减轻了错误折叠聚集所带来的内质网压力,有利于目标蛋白的分泌表达。
3 结论
本研究通过分子对接和结合能预测,得到对bgl3A催化活性有潜在影响的点进行突变,获得比酶活提升的突变体bgl3A-N237Y。和bgl3A相比,bgl3A-N237Y在最适反应条件下测定的比酶活提升了22.9%。Km减少了16.7%,对底物的亲和力增加,kcat/Km也获得一定程度的提升。分子动力学模拟结果表明,bgl3A比酶活提升的原因可能是由于突变后在活性位点引入了新的芳香作用力使蛋白与底物的结合能力增强,使得突变体表现出更高的催化活性。为便于生产,将最佳突变体转入毕赤酵母中表达,获得摇瓶水平酶活力62.7 U/mL的重组菌株,酶活力远高于大肠杆菌宿主,后经表达策略优化使酶活力提升了约75%,达到106.6 U/mL。本研究所使用的相关策略将为类似功能酶的改造提供技术方法上的借鉴,同时为该酶的工业化生产提供参考。
[1] BOGATI K, WALCZAK M.The Impact of drought stress on soil microbial community, enzyme activities and plants[J].Agronomy-Basel, 2022, 12(1):189.
[2] DADWAL A, SHARMA S, SATYANARAYANA T.Thermostable cellulose saccharifying microbial enzymes:Characteristics, recent advances and biotechnological applications[J].International Journal of Biological Macromolecules, 2021, 188:226-244.
[3] RAJNISH K N, SAMUEL M S, JOHN J A, et al.Immobilization of cellulase enzymes on nano and micro-materials for breakdown of cellulose for biofuel production-a narrative review[J].International Journal of Biological Macromolecules, 2021, 182:1793-1802.
[4] POIDEVIN L, FELIU J, DOAN A, et al.Insights into Exo- and Endoglucanase activities of family 6 glycoside hydrolases from Podospora anserina[J].Applied and Environmental Microbiology, 2013, 79(14):4220-4229.
[5] VLASENKO E, SCHÜLEIN M, CHERRY J, et al.Substrate specificity of family 5, 6, 7, 9, 12, and 45 endoglucanases[J].Bioresource Technology, 2010, 101(7):2405-2411.
[6] SONG J Z, LIU B D, LIU Z H, et al.Cloning of two cellobiohydrolase genes from Trichoderma viride and heterogenous expression in yeast Saccharomyces cerevisiae[J].Molecular Biology Reports, 2010, 37(4):2135-2140.
[7] BISCHOF R H, RAMONI J, SEIBOTH B.Cellulases and beyond:The first 70 years of the enzyme producer Trichoderma reesei[J].Microbial Cell Factories, 2016, 15(1):106.
[8] PAUL M, MOHAPATRA S, KUMAR DAS MOHAPATRA P, et al.Microbial cellulases-An update towards its surface chemistry, genetic engineering and recovery for its biotechnological potential[J].Bioresource Technology, 2021, 340:125710.
[9] HUANG Q, WANG K, LI H, et al.Enhancing cellulosic ethanol production through coevolution of multiple enzymatic characteristics of β-glucosidase from Penicillium oxalicum 16[J].Applied Microbiology and Biotechnology, 2020, 104(19):8299-8308.
[10] SRIVASTAVA N, RATHOUR R, JHA S, et al.Microbial beta glucosidase enzymes:Recent advances in biomass conversation for biofuels application[J].Biomolecules, 2019, 9(6):220.
[11] TEUGJAS H, VAELJAMAEE P.Selecting β-glucosidases to support cellulases in cellulose saccharification[J].Biotechnology for Biofuels, 2013, 6(1):105.
[12] HAVEN M O, JØRGENSEN H.Adsorption of β-glucosidases in two commercial preparations onto pretreated biomass and lignin[J].Biotechnology for Biofuels, 2013, 6(1):165.
[13] HENRISSAT B, DAVIES G.Structural and sequence-based classification of glycoside hydrolases[J].Current Opinion in Structural Biology, 1997, 7(5):637-644.
[14] LIMA R A T, DE OLIVEIRA G, SOUZA A A, et al.Functional and structural characterization of a novel GH3 β-glucosidase from the gut metagenome of the Brazilian Cerrado termite Syntermes wheeleri[J].International Journal of Biological Macromolecules, 2020, 165(Pt A):822-834.
[15] XIA W, BAI Y G, CUI Y, et al.Functional diversity of family 3 β-glucosidases from thermophilic cellulolytic fungus Humicola insolens Y1[J].Scientific Reports, 2016, 6:27062.
[16] XIA W, BAI Y G, SHI P J.Improving the Substrate Affinity and Catalytic Efficiency of β-Glucosidase Bgl3A from Talaromyces leycettanus JCM12802 by Rational Design[J].Biomolecules, 2021, 11(12):1882.
[17] XIA W, XU X X, QIAN L C, et al.Engineering a highly active thermophilic β-glucosidase to enhance its pH stability and saccharification performance[J].Biotechnoloy for Biofuels, 2016, 9:147.
[18] MUSHTAQ A, AZAM U, MEHREEN S, et al.Synthetic α-glucosidase inhibitors as promising anti-diabetic agents:Recent developments and future challenges[J].European Journal of Medicinal Chemistry, 2023, 249:115119.
[19] LEWIS D F V, JACOBS M N, DICKINS M.Compound lipophilicity for substrate binding to human P450 s in drug metabolism[J].Drug Discovery Today, 2004, 9(12):530-537.
[20] REDDY M R, VISWANADHAN V N, ERION M D.Rapid estimation of relative binding affinities of enzyme inhibitors[J].Perspectives in Drug Discovery and Design, 1998, 9-11:85-98.
[21] SONG W Y, LI Y Y, TONG Y, et al.Improving the catalytic efficiency of Aspergillus fumigatus glucoamylase toward raw starch by engineering its N-glycosylation sites and saturation mutation[J].Journal of Agricultural and Food Chemistry, 2022, 70(39):12672-12680.
[22] ABRAHAM M J, MURTOLA T, SCHULZ R, et al.GROMACS:High performance molecular simulations through multi-level parallelism from laptops to supercomputers[J].SoftwareX, 2015, 1-2:19-25.
[23] MAIER J A, MARTINEZ C, KASAVAJHALA K, et al.ff14SB:Improving the accuracy of protein side chain and backbone parameters from ff99SB[J].Journal of Chemical Theory and Computation, 2015, 11(8):3696-3713.
[24] SHEN Q, ZHOU X T, GUO Q, et al.Potential of the signal peptide derived from the PAS_chr3_0030 gene product for secretory expression of valuable enzymes in Pichia pastoris [J].Applied and Environmental Microbiology, 2022, 88(9):e0029622.
[25] MURASUGI A, TOHMA-AIBA Y.Comparison of three signals for secretory expression of recombinant human midkine in Pichia pastoris[J].Bioscience Biotechnology and Biochemistry, 2001, 65(10):2291-2293.
[26] LIANG S L, LI C, YE Y R, et al.Endogenous signal peptides efficiently mediate the secretion of recombinant proteins in Pichia pastoris[J].Biotechnology Letters, 2013, 35(1):97-105.
[27] DUAN G D, DING L M, WEI D S, et al.Screening endogenous signal peptides and protein folding factors to promote the secretory expression of heterologous proteins in Pichia pastoris[J].Journal of Biotechnology, 2019, 306:193-202.
[28] HUANG H, LIANG Q X, WANG Y, et al.High-level constitutive expression of leech hyaluronidase with combined strategies in recombinant Pichia pastoris[J].Applied Microbiology and Biotechnology, 2020, 104(4):1621-1632.
[29] HWANG J, QI L.Quality control in the endoplasmic reticulum:Crosstalk between ERAD and UPR pathways[J].Trends in Biochemical Sciences, 2018, 43(8):593-605.