假小链双歧杆菌于1979年首次在婴儿粪便中分离得到,因为与已知的其他双歧杆菌表现出不同的形态和发酵特征,所以被命名为假小链双歧杆菌[1]。至今为止,已发现假小链双歧杆菌具有多种益生特性,例如缓解肝损伤[2]、改善结肠炎[3]和儿童肥胖[4]等。此外,假小链双歧杆菌可转化生物活性物质,具有开发功能性食品和益生菌的潜力。部分菌株可分解木脂素,具备抗氧化和潜在抗癌功效[5];假小链双歧杆菌INIA P815能够从鞣花酸中产生尿石素[6];复配发酵豆奶可提升糖苷配基形式的类黄酮含量[7]。该物种具有潜在益生功效的菌株主要分离自人类粪便,但益生特性在菌株间存在差异。
作为高通量测序发展衍生的技术,比较基因组学可以依据不同菌株基因组信息进行比对,寻找相似点和差异点。有研究人员利用双歧杆菌基因组探究其代谢机制[8],通过耐药基因的可转移性和携带毒力因子数量评估菌株安全性[9]。了解菌株携带功能基因的种类有助于识别有益及风险菌株,但关于假小链双歧杆菌的遗传进化及功能基因差异研究较少。
本研究以NCBI数据库中公布的104株假小链双歧杆菌全基因组为研究对象,采用比较基因组学方法探究不同国家假小链双歧杆菌携带功能基因的差异。为解析假小链双歧杆菌基因组多样性和功能基因差异提供依据。
1 材料与方法
1.1 菌株基因组数据
104株假小链双歧杆菌包括52个中国分离株、41个日本分离株、10个美国分离株和1个模式株。所有菌株的基因组均下载至NCBI RefSeq 数据库 (National Center for Biotechnology Information, https://www.ncbi.nlm.nih.gov/)。筛选标准为:分离源为人类;有明确的分离地;测序深度>30 X;Scaffolds数量<100。具体菌株信息见附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.036839)。
表1 假小链双歧杆菌泛-核心基因功能占比
Table 1 Functional categories of representative core and pan genes of Bifidobacterium pseudocatenulatum
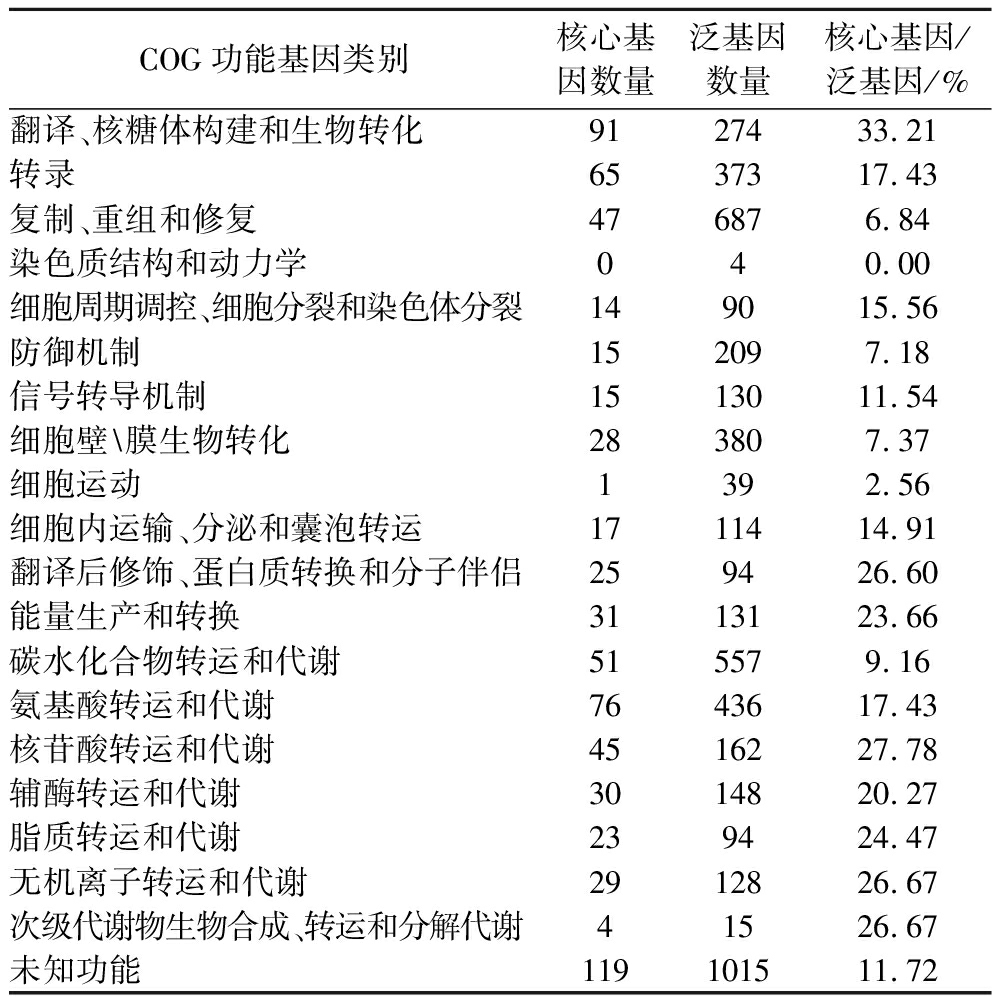
COG功能基因类别核心基因数量泛基因数量核心基因/泛基因/%翻译、核糖体构建和生物转化9127433.21转录6537317.43复制、重组和修复476876.84染色质结构和动力学040.00细胞周期调控、细胞分裂和染色体分裂149015.56防御机制152097.18信号转导机制1513011.54细胞壁\膜生物转化283807.37细胞运动1392.56细胞内运输、分泌和囊泡转运1711414.91翻译后修饰、蛋白质转换和分子伴侣259426.60能量生产和转换3113123.66碳水化合物转运和代谢515579.16氨基酸转运和代谢7643617.43核苷酸转运和代谢4516227.78辅酶转运和代谢3014820.27脂质转运和代谢239424.47无机离子转运和代谢2912826.67次级代谢物生物合成、转运和分解代谢41526.67未知功能119101511.72
注:使用eggNOG-mapper在线注释基因功能。
1.2 基因预测与注释
利用 Prokka(v1.14)[10]软件进行基因预测,将预测到的CDS使用eggNOG-mapper功能注释数据库(v2.1.9)[11]在线注释。
1.3 泛-核心基因集构建
利用Roary(v3.6.1)[12]软件以氨基酸序列相似性大于0.9识别基因家族,以检出率100%为筛选标准构建核心基因集,以检出到的所有基因家族构建泛基因集。
1.4 系统发育分析
利用FastANI(https://github.com/ParBLiSS/FastANI)计算菌株两两之间的平均核苷酸一致性(average nucleotide identity, ANI),将整理后的ANI值使用TBtools[13]绘制热图并聚类。利用TreeBeST软件(https://github.com/Ensembl/treebest)对104株假小链双歧杆菌的核心基因基于最大似然法经1 000次迭代构建系统发育树,利用ITOL[14]在线网站对系统发育树进行可视化。
1.5 碳水化合物活性酶注释
利用HMMER(3.3.2)[15](http://hmmer.org/)软件将104株假小链双歧杆菌氨基酸序列与碳水化合物活性酶数据库比对进行碳水化合物活性酶注释,比对标准与dbCAN3碳水化合物活性酶注释网站一致(https://bcb.unl.edu/dbCAN2/blast.php)。
1.6 耐药基因注释
将所分析菌株氨基酸序列与耐药基因数据库(v3.2.2)(Comprehensive Antibiotic Resistance Database,CARD;http://arpcard.mcmaster.ca)进行 BLAST(v2.2.26)[16]比对,以相似度>40%,E值
1.7 毒力因子注释
将所分析菌株氨基酸序列与毒力因子数据库(版本:2022年7月15日)(Virulence Factor Database,VFDB;http://www.mgc.ac.cn/VFs/main.htm)中的Protein sequences of full dataset进行BLAST(v2.2.26)比对,以相似度>60%,E值
1.8 可移动遗传元件注释
将所分析菌株的氨基酸序列与MobileOG[19]数据库进行BLAST(v2.2.26)比对,将比对结果中相似度>60%,E值 104株假小链双歧杆菌基因组平均大小为(2.27±0.09) Mb;平均GC含量为(56.43±0.18)%;编码序列(coding sequence,CDS)数量为1 318~2 095个,平均为(1 747.28±135.67)个。通过比较菌株的基因组特征,发现日本分离株的基因组大小显著高于美国分离株(P<0.05),其他组别间没有显著差异;不同国家分离株的GC含量均无显著差异;菌株CDS数量范围跨度较大,中国分离株TM01-4拥有最低的CDS(1318),日本分离株高于中国分离株(P<0.001)和美国分离株(P<0.01),而在中国和美国间,菌株的CDS数量无显著差异(图1)。 图1 假小链双歧杆菌的基因组特征比较 为探究假小链双歧杆菌的平均核苷酸一致性,本研究对包括模式菌株DSM20438T的104株假小链双歧杆菌进行分析。结果显示,104株假小链双歧杆菌的ANI值为97.39%~99.99%,菌株之间较高的ANI值表明基因组差异较小,参考CIUFO等[20]依据ANI值判断细菌分类学地位的研究结果可判断本研究中所选用分析菌株均为假小链双歧杆菌。基于ANI聚类热图可发现大部分假小链双歧杆菌具有地域相似性,相同分离地菌株表现出更高的ANI值(图2-A)。 A-104株假小链双歧杆菌两两之间平均核苷酸一致性热图;B-基于Roary获得的核心基因使用TreeBeST软件采用最大似然法 经1 000次迭代构建的系统发育树 图2 假小链双歧杆菌的平均核苷酸一致性和系统发育树 为探讨假小链双歧杆菌功能基因的多样性对假小链双歧杆菌进行泛-核心基因计算(表1)。104株假小链双歧杆菌核心基因有772个,泛基因有8 247个,核心基因仅占泛基因的9.36%,表明假小链双歧杆菌基因组多样性较高。对泛基因组进行功能注释发现与复制,重组和修复(13.50%)、碳水化合物转运和代谢(10.95%)、氨基酸运输和代谢(8.57%)相关的基因占比较多。而核心基因主要是维持基本生命活动的功能基因。 如图2-B所示,基于772个核心基因构建的系统发育树形成两个混合簇,中国、日本和美国分离株在两簇中均存在。一簇包含14/52的中国分离株、28/41的日本分离株和8/10的美国分离株以及模式菌株DSM20438T,另一大簇包含所分析的剩余菌株。分布于两个大簇中的中国和日本分离株能够按分离国家较好相聚,美国分离株1001275B_160808_D7、1001099B_141217_G10、1001271B_151109_H7、1001271 st1_F3和J1101437_171009_A10与模式株聚为一小簇,其余5株美国分离株与中国和日本分离株聚在一起。 如图3所示,104株假小链双歧杆菌共注释到57种碳水化合物活性酶,其中包括31种糖基水解酶家族(glycoside hydrolases,GHs)、10种糖基转移酶家族(glycosyl transferases, GTs)、12种碳水化合物结合模块 (carbohydrate-binding modules,CBMs)和4种碳水化合物酯酶家族(carbohydrate esterases,CEs)。存在21个检出率为100%的碳水化合物活性酶,对于糖基水解酶家族,GH13家族、GH43家族和GH3家族占据主导地位,分别占全部碳水化合物酶的13.70%、11.62%和5.45%。假小链双歧杆菌丰富的碳水化合物活性酶有助于其在多种碳源环境下生长。 A-假小链双歧杆菌携带碳水化合物活性酶热图;B-不同国家分离株携带碳水化合物活性酶数量差异箱线图 图3 不同国家分离株携带碳水化合物活性酶数量差异 A-假小链双歧杆菌携带抗生素抗性基因热图;B-分离自不同国家假小链双歧杆菌携带耐药基因数量差异;C-分离自不同国家假小链双杆菌 携带特有耐药基因数量 图4 假小链双歧杆菌携带耐药基因的种类、数量热图 不同国家分离株携带的碳水化合物活性酶有15个家族在拷贝数量上存在差异。中国分离株携带碳水化合物活性最丰富,携带CBM67、GH25、GH27、GH30、GH78和GH121等家族的碳水化合物活性酶较多,主要是分解半乳糖、鼠李糖和β-L-阿拉伯糖等植物及乳制品来源的多糖。2簇中国分离株在携带GH25、GH31、GH51和GH95家族数量存在显著差异(P<0.05)。日本分离株携带CBM67、GH1、GH2和GH95等家族的碳水化合物活性酶较多,主要是分解β-半乳糖、β-D-葡萄糖和鼠李糖等乳制品和植物来源的多糖。美国分离株极大部分碳水化合物活性酶的拷贝数量相比中国和日本分离株数量较少,GH3和GH51家族为其携带相对较多的碳水化合物活性酶,可分解β-D-葡萄糖和α-L-阿拉伯呋喃糖等。 对日本婴儿分离株和成人分离株之间差异比对发现6个碳水化合物活性酶(GH1、GH5、GH30、GH51、GT14和GT32)在二者之间存在显著差异(P<0.05)。但值得注意的是,与母乳低聚糖利用相关的糖基水解酶在成人和婴儿间并不存在显著差异。在婴儿源菌株中与母乳低聚糖利用相关的糖基水解酶家族,GH2和GH20检出率为100%,GH95检出率为47.62%(10/21),GH16、GH18、GH29、GH33检出率为0。 菌株携带耐药基因可帮助菌株抵抗抗生素环境,提升菌株存活率。通过与耐药基因数据库CARD比对注释,在104株假小链双歧杆菌基因组中共找到13个大类,32种抗生素耐药基因(图4-A)。其中有7个耐药基因的检出率为100%,分别是莫匹罗星抗性基因Bbif_ileS_MUP、利福平抗性基因Bado_rpoB_RIF和RbpA、磷霉素抗性基因Ctra_murA_FOF、抗生素外排泵基因efrA、macB和mtrA。依图4-B可知,中国分离株携带耐药基因的数量显著高于日本分离株(P<0.000 1)和美国分离株(P<0.01),且2簇中国分离株所携带耐药基因不存在显著差异(P=0.43)。中国分离株耐药基因的高携带量表现在携带其他国家分离株未携带或少量携带的vanRF、vanRL、vanUG、tetW、kdpE、dfrF、ErmX、msbA等耐药基因,赋予菌株万古霉素、四环素、氨基糖苷类、大环内酯类和林可酰胺类等抗生素抗性。 一般认为毒力因子为致病菌入侵、黏附、攻击宿主免疫系统的功能基因,近年也发现黏附类型的毒力因子可帮助益生菌定植人类肠道[21]。如图5所示,与VFDB数据库比对注释所分析菌株携带毒力因子状况,104株假小链双歧杆菌携带隶属6种类型的35种毒力因子,22.75%的毒力因子与黏附相关。与黏附相关的毒力因子可帮助菌株更好地定植于人体肠道,在所分析菌株中检测出9个该类型的毒力因子,其中groEL2和tufA在所有菌株中均存在,其余7种黏附类型毒力因子在中国分离株中均检测到,而在其他国家分离株中仅检测到部分毒力因子。 图5 假小链双歧杆菌携带毒力因子的种类、数量热图 如图6所示,经过MobileOG数据库注释,104株假小链双歧杆菌共注释到10 613个可移动遗传元件。其中,中国分离株和日本分离株携带的5种类型可移动遗传元件在数量上均无显著差异,并且两国与美国分离株相比,在整合/切除、稳定性/转移/防御和转移相关可移动遗传元件上也无显著差异。美国相对中国分离株和日本分离株在复制/重组/修复和噬菌体生物过程相关的可移动遗传元件数量上存在显著差异(P<0.05),其携带2种可移动遗传元件数量最低。两簇中国分离株携带噬菌体相关可移动遗传元件差异较大(P<0.05),中国两簇分离株携带更多。 A-复制、重组和修复;B-噬菌体;C-整合、切除;D-稳定性、转移和防御;E-转移;F-噬菌体 图6 不同分离地假小链双歧杆菌携带可移动遗传元件比对 已有的研究大多集中关注假小链双歧杆菌菌株水平的功能,特别是益生功能,但对其携带功能基因的差异研究较少。本研究表明假小链双歧杆菌基因组存在地理性差异,这种差异主要表现在携带不同种类和数量的碳水化合物活性酶、耐药基因、毒力因子和可移动遗传元件等相关基因,这个结果对假小链双歧杆菌食品工业用菌及益生菌的筛选具有借鉴意义。由于研究中不同国家分离株数量不同,不能排除菌株数量造成的偏差。本研究利用有限的数据为假小链双歧杆菌的系统发育和地理特征提供新的依据。 对所分析菌株的泛基因组进行功能注释发现与碳水化合物利用相关的基因占比较高,且不同国家分离株的该类功能基因在拷贝数量上存在较大差异,这对不同国家分离株的功能基因构成带来一定影响。有研究表明碳水化合物活性酶的拷贝数会受到饮食习惯的影响[22],假小链双歧杆菌相比其他双歧杆菌携带水解植物来源聚糖的相关酶更丰富[23],本研究也发现相似结果。中国和日本饮食中植物性食物占比较大[24-25],在相关分离株中利用植物源多糖的碳水化合物活性酶拷贝数量相对较高。而美国人饮食以肉类为主,蔬菜摄入量较少[26],其相关分离株携带分解植物源多糖的碳水化合物活性酶拷贝数量则相对较低。 新生儿饮食以母乳为主,母乳低聚糖是母乳中第三大固体成分,这使得具有母乳低聚糖代谢能力的双歧杆菌更容易定植。前人研究表明母乳喂养婴儿的双歧杆菌群以短双歧杆菌、两歧双歧杆菌和长双歧杆菌为主[27],并有研究发现两歧双歧杆菌对2’-岩藻糖基乳糖的利用能力强于假小链双歧杆菌[28]。本研究发现假小链双歧杆菌携带水解岩藻糖基化母乳低聚糖的糖基水解酶(GH95和GH29)过少,这或是其对2’-岩藻糖基乳糖利用能力不如两歧双歧杆菌的原因。 细菌的抗生素耐药性是由其基因组中抗生素耐药基因所赋予的[29],中国人群假小链双歧杆菌携带万古霉素、四环素和硝基咪唑等抗生素耐药基因,虽然该基因型可提升菌株暴露于抗生素环境中的生存能力,但抗生素抗性基因在一定条件下可通过转化、转导和接合转移等方式在肠道微生物之间转移,从而降低抗生素在临床治疗中的有效性。推测中国分离株的抗生素抗性基因高携带量的原因是早期对于抗生素使用管控不严,被广泛用于人类和家畜的疾病治疗[30]。抗生素的过度使用会对人体肠道菌群多样性和丰度造成影响,甚至使原本不耐药细菌获得耐药性。假小链双歧杆菌中国分离株耐药基因的高携带量为抗生素的不规范使用敲响警钟。 可移动遗传元件介导菌株间的水平基因转移在细菌进化过程起到极大作用,有研究发现致病菌可通过可移动遗传元件快速获得耐药基因[31]。中国和日本分离株携带的复制、重组和修复及噬菌体相关的可移动遗传元件数量均高于美国分离株,表明中国和日本分离株的基因组存在较多外源基因片段插入。系统发育树中的中国分离株形成两簇,两簇菌株仅在携带噬菌体相关可移动遗传元件上变现出显著差异,表明噬菌体相关可移动遗传元件是造成中国分离株聚类不同的原因之一。 对于毒力因子,更多的研究者关注其造成宿主细菌感染的可能性,毒力因子通常促进致病菌附着于肠黏膜细胞,进而分泌毒素引起疾病,甚至可以逃避免疫反应[32]。最终能否对宿主引起危害,主要在于菌株是否存在致病性,而在假小链中为未检测到可对宿主产生危害的外毒素型毒力因子。协助细菌黏附宿主细胞主要是通过黏附型毒力因子发挥作用,所分析的全部假小链双歧杆菌均携带的tuf基因可编码EF-Tu[33],前人发现EF-Tu可帮助益生菌Lactobacillus johnsonii NCC533附着肠上皮细胞[21]。GroEL有助于细菌对于黏蛋白的黏附[34],groEL2为GroEL的分子伴侣。中国分离株携带的更多种类黏附类型毒力因子或可提升其定植于人类肠道的能力。 在本研究中,104株假小链双歧杆菌的基因组序列为该物种的进化和功能基因解析提供了基础。研究发现菌株的分离地差异对菌株所携带的功能基因产生较大影响,特别体现在碳水化合物活性酶拷贝数量差异上,其次是耐药基因、毒力因子和可移动遗传元件的种类和数量。中国分离株携带上述基因的种类和数量最多,日本和美国携带相关基因的种类和数量相对较少。研究结果拓宽了对该物种的了解,并为进一步研究该菌株的功能和筛选具备益生功效的菌株提供了参考。 [1] SCARDOVI V, TROVATELLI L D, BIAVATI B, et al.Bifidobacterium cuniculi, Bifidobacterium choerinum, Bifidobacterium boum, and Bifidobacterium pseudocatenulatum:Four new species and their deoxyribonucleic acid homology relationships[J].International Journal of Systematic Bacteriology, 1979, 29(4):291-311. [2] FANG D Q, SHI D, LYU L X, et al.Bifidobacterium pseudocatenulatum LI09 and Bifidobacterium catenulatum LI10 attenuate D-galactosamine-induced liver injury by modifying the gut microbiota[J].Scientific Reports, 2017, 7(1):8770. [3] CHEN Y, YANG B, STANTON C, et al.Bifidobacterium pseudocatenulatum ameliorates DSS-induced colitis by maintaining intestinal mechanical barrier, blocking proinflammatory cytokines, inhibiting TLR4/NF-κB signaling, and altering gut microbiota[J].Journal of Agricultural and Food Chemistry, 2021, 69(5):1496-1512. [4] SANCHIS-CHORD [5] PEIROTÉN [6] GAYA P, PEIROTÉN [7] DI GIOIA D, STRAHSBURGER E, LOPEZ DE LACEY A M, et al.Flavonoid bioconversion in Bifidobacterium pseudocatenulatum B7003:A potential probiotic strain for functional food development[J].Journal of Functional Foods, 2014, 7:671-679. [8] 姜天翌, 吴欢, 陈峰, 等.基于基因组的假小链双歧杆菌与卷曲乳杆菌代谢互作分析[J].基因组学与应用生物学, 2021, 40(S3):3044-3054. [9] YU D S, JEONG H, LEE D H, et al.Complete genome sequence of the probiotic bacterium Bifidobacterium bifidum strain BGN4[J].Journal of Bacteriology, 2012, 194(17):4757-4758. [10] SEEMANN T.Prokka:Rapid prokaryotic genome annotation[J].Bioinformatics, 2014, 30(14):2068-2069. [11] CANTALAPIEDRA C P, HERN [12] PAGE A J, CUMMINS C A, HUNT M, et al.Roary:Rapid large-scale prokaryote pan genome analysis[J].Bioinformatics, 2015, 31(22):3691-3693. [13] CHEN C J, CHEN H, ZHANG Y, et al.TBtools:An integrative toolkit developed for interactive analyses of big biological data[J].Molecular Plant, 2020, 13(8):1194-1202. [14] LETUNIC I, BORK P.Interactive Tree Of Life (iTOL) v5:An online tool for phylogenetic tree display and annotation[J].Bioinformatics, 2007, 23(1): 127-128. [15] POTTER S C, LUCIANI A, EDDY S R, et al.HMMER web server:2018 update[J].Nucleic acids research, 2018, 46(W1):W200-W204. [16] YE J, MCGINNIS S, MADDEN T L.BLAST:Improvements for better sequence analysis[J].Nucleic Acids Research, 2006, 34(Web Server issue):W6-W9. [17] 刘倩. 假小链双歧杆菌遗传多样性及其对健康小鼠肠道微生态的影响[D].无锡:江南大学, 2020. [18] D [19] BROWN C L, MULLET J, HINDI F, et al.MobileOG-db:A manually curated database of protein families mediating the life cycle of bacterial mobile genetic elements[J].Applied and Environmental Microbiology, 2022, 88(18):e0099122. [20] CIUFO S, KANNAN S, SHARMA S, et al.Using average nucleotide identity to improve taxonomic assignments in prokaryotic genomes at the NCBI[J].International Journal of Systematic and Evolutionary Microbiology, 2018, 68(7):2386-2392. [21] GRANATO D, BERGONZELLI G E, PRIDMORE R D, et al.Cell surface-associated elongation factor Tu mediates the attachment of Lactobacillus johnsonii NCC533 (La1) to human intestinal cells and mucins[J].Infection and immunity, 2004, 72(4):2160-2169. [22] PAJIC P, PAVLIDIS P, DEAN K, et al.Independent amylase gene copy number bursts correlate with dietary preferences in mammals[J].eLife, 2019, 8:e44628. [23] WATANABE Y, SAITO Y, HARA T, et al.Xylan utilisation promotes adaptation of Bifidobacterium pseudocatenulatum to the human gastrointestinal tract[J].ISME Communications, 2021,1(1):62. [24] 赵霖, 鲍善芬.中华民族传统膳食结构的特点和优势[J].中国食品学报, 2004, 4(4):1-6. [25] MURAKAMI K, SASAKI S.Meal-specific dietary patterns and their contribution to overall dietary patterns in the Japanese context:Findings from the 2012 National Health and Nutrition Survey, Japan[J].Nutrition, 2019, 59:108-115. [26] HOY M K, CLEMENS J C, MARTIN C L, et al.Fruit and vegetable consumption of US adults by level of variety, what we eat in America, NHANES 2013-2016[J].Current Developments in Nutrition, 2020, 4(3):nzaa014. [27] O′CALLAGHAN A, VAN SINDEREN D.Bifidobacteria and their role as members of the human gut microbiota[J].Frontiers in Microbiology, 2016, 7:925. [28] 李刘若兰, 张程程, 刘秉书, 等.不同假小链双歧杆菌对碳水化合物利用能力的比较研究[J].食品与发酵工业, 2023,49(14):94-102. [29] WRIGHT G D.The antibiotic resistome:The nexus of chemical and genetic diversity[J].Nature Reviews. Microbiology, 2007, 5(3):175-186. [30] YIN X, SONG F J, GONG Y H, et al.A systematic review of antibiotic utilization in China[J].The Journal of Antimicrobial Chemotherapy, 2013, 68(11):2445-2452. [31] JIANG X L, ELLABAAN M M H, CHARUSANTI P, et al.Dissemination of antibiotic resistance genes from antibiotic producers to pathogens[J].Nature Communications, 2017, 8:15784. [32] WEBB S A R, KAHLER C M.Bench-to-bedside review:Bacterial virulence and subversion of host defences[J].Critical Care, 2008, 12(6):234. [33] HARVEY K L, JAROCKI V M, CHARLES I G, et al.The diverse functional roles of elongation factor tu (EF-tu) in microbial pathogenesis[J].Frontiers in Microbiology, 2019, 10:2351. [34] HAGEMANN L, GRÜNDEL A, JACOBS E, et al.The surface-displayed chaperones GroEL and DnaK of Mycoplasma pneumoniae interact with human plasminogen and components of the extracellular matrix[J].Pathogens and Disease, 2017, 75(3):10.1093/femspd/ftx017.2 结果与分析
2.1 不同国家的假小链双歧杆菌基因组特征

Fig.1 Comparison of genomic characteristics of Bifidobacterium pseudocatenulatum2.2 平均核苷酸一致性分析

Fig.2 Average nucleotide identity and phylogenetic tree of Bifidobacterium pseudocatenulatum2.3 泛-核心基因分析
2.4 系统发育分析
2.5 碳水化合物活性酶比对

Fig.3 The differences in the number of carbohydrate-active enzymes carried by different countries′ isolates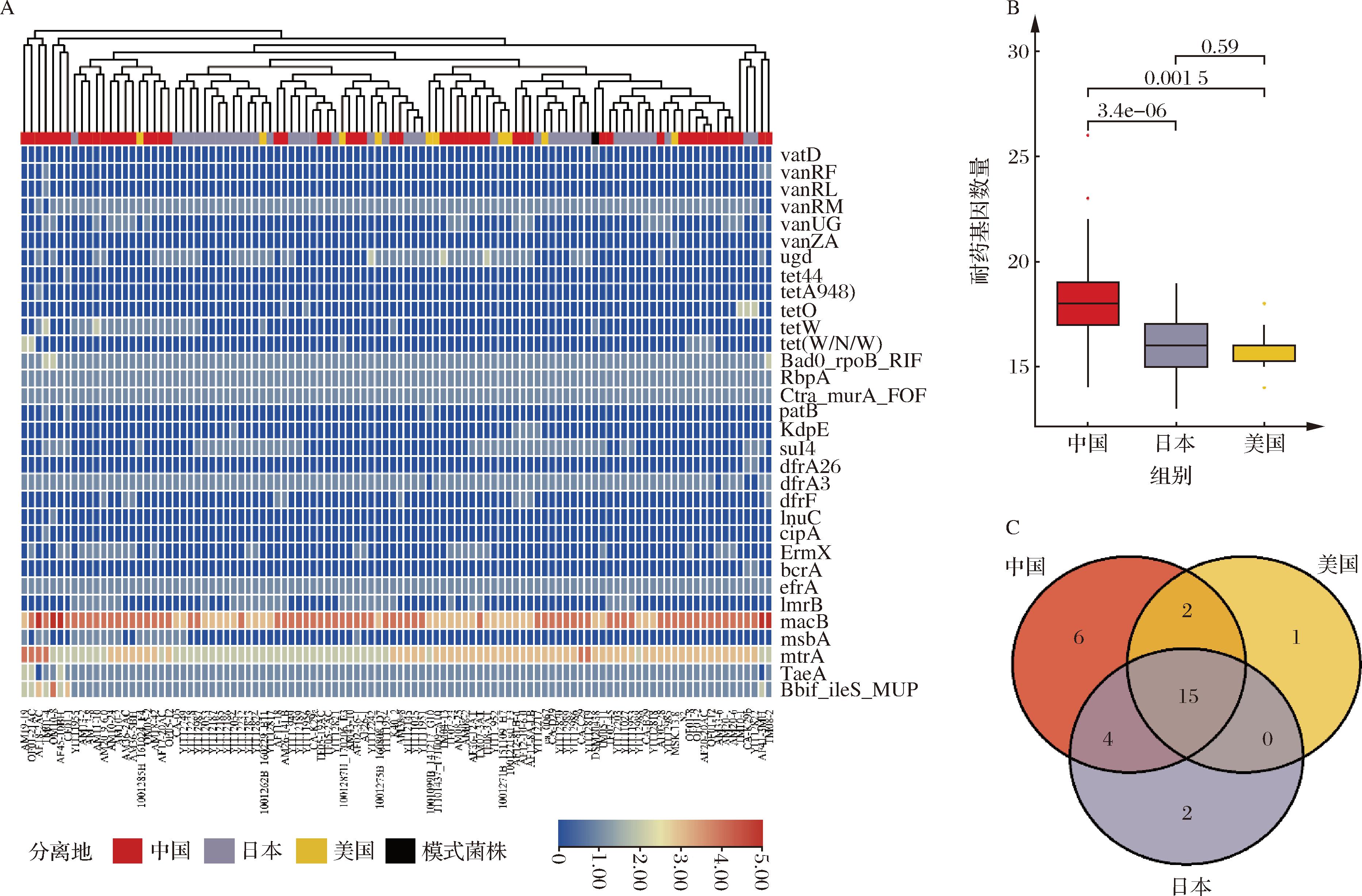
Fig.4 Heatmaps of species and number of antibiotic resistance genes carried by Bifidobacterium pseudocatenulatum2.6 耐药基因比对
2.7 毒力因子比对

Fig.5 Heatmaps of species and number of virulence factors carried by Bifidobacterium pseudocatenulatum2.8 可移动遗传元件比对
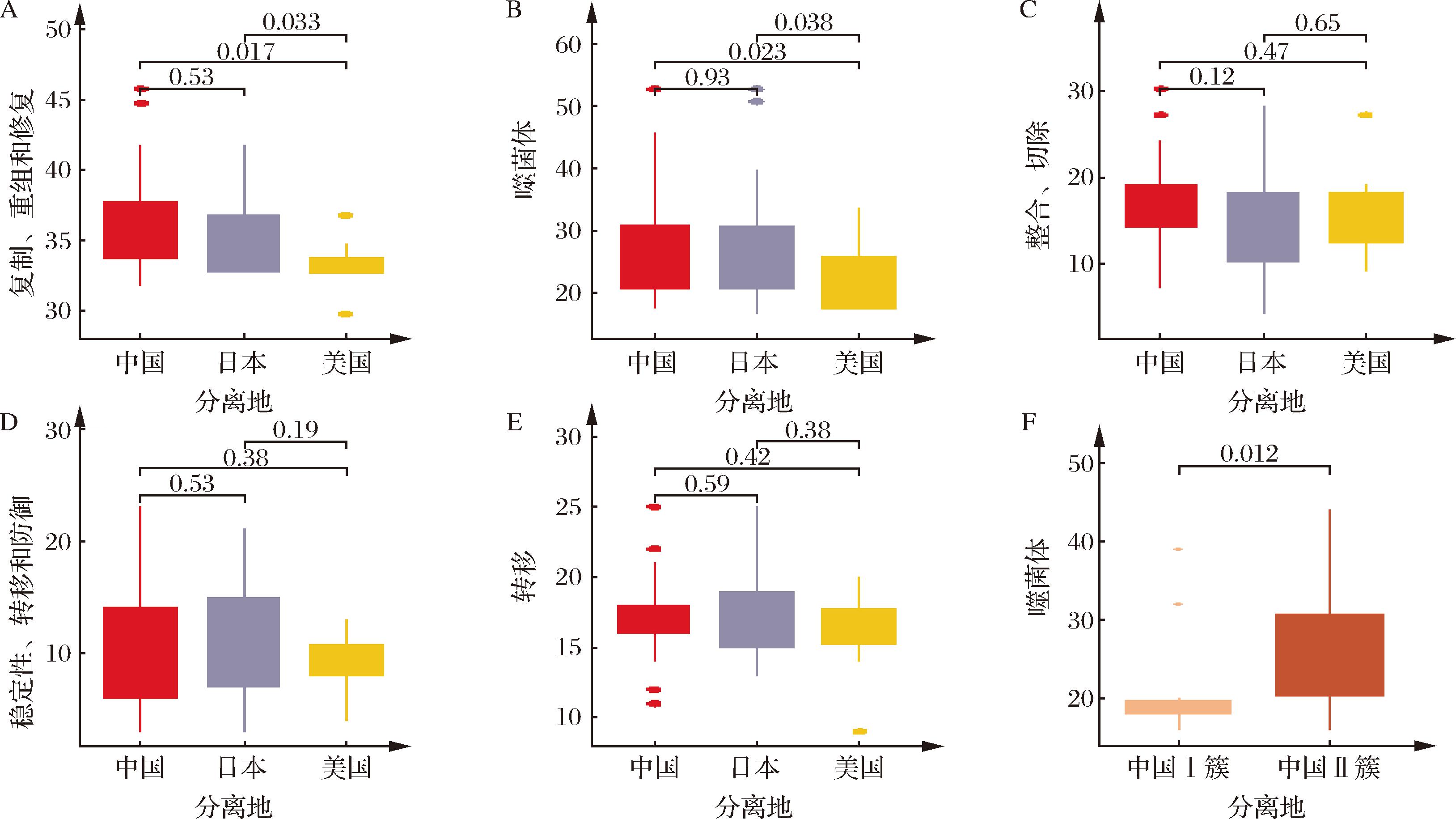
Fig.6 Comparison of mobile genetic elements carried by different isolates of Bifidobacterium pseudocatenulatum3 讨论
4 结论
 J, DEL PULGAR E M G, CARRASCO-LUNA J, et al.Bifidobacterium pseudocatenulatum CECT 7765 supplementation improves inflammatory status in insulin-resistant obese children[J].European Journal of Nutrition, 2019, 58(7):2789-2800.
J, DEL PULGAR E M G, CARRASCO-LUNA J, et al.Bifidobacterium pseudocatenulatum CECT 7765 supplementation improves inflammatory status in insulin-resistant obese children[J].European Journal of Nutrition, 2019, 58(7):2789-2800. , GAYA P,
, GAYA P,  LVAREZ I, et al.Influence of different lignan compounds on enterolignan production by Bifidobacterium and Lactobacillus strains[J].International Journal of Food Microbiology, 2019, 289:17-23.
LVAREZ I, et al.Influence of different lignan compounds on enterolignan production by Bifidobacterium and Lactobacillus strains[J].International Journal of Food Microbiology, 2019, 289:17-23. , MEDINA M, et al.Bifidobacterium pseudocatenulatum INIA P815:The first bacterium able to produce urolithins A and B from ellagic acid[J].Journal of Functional Foods, 2018, 45:95-99.
, MEDINA M, et al.Bifidobacterium pseudocatenulatum INIA P815:The first bacterium able to produce urolithins A and B from ellagic acid[J].Journal of Functional Foods, 2018, 45:95-99.
JIANG T Y, WU H, CHEN F, et al.The genome-scale analysis of metabolic interaction between Bifidobacterium pseudominor and Lactobacillus crispatus based on genome[J].Genomics and Applied Biology,2021, 40(S3):3044-3054. NDEZ-PLAZA A, LETUNIC I, et al.EggNOG-mapper v2:Functional annotation, orthology assignments, and domain prediction at the metagenomic scale[J].Molecular Biology and Evolution, 2021, 38(12):5825-5829.
NDEZ-PLAZA A, LETUNIC I, et al.EggNOG-mapper v2:Functional annotation, orthology assignments, and domain prediction at the metagenomic scale[J].Molecular Biology and Evolution, 2021, 38(12):5825-5829.
LIU Q.Genomic diversity of Bifidobacterium pseudocatenulatum and its effect on the gut microbiota of healthy mice [D].Wuxi:Jiangnan University, 2020. AZ R, TORRES-MIRANDA A, ORELLANA G, et al.Comparative genomic analysis of novel Bifidobacterium longum subsp.longum strains reveals functional divergence in the human gut microbiota[J].Microorganisms, 2021, 9(9):1906.
AZ R, TORRES-MIRANDA A, ORELLANA G, et al.Comparative genomic analysis of novel Bifidobacterium longum subsp.longum strains reveals functional divergence in the human gut microbiota[J].Microorganisms, 2021, 9(9):1906.
ZHAO L, BAO S F.The advantages and characteristics of Chinese traditional dietary structure[J].Journal of Chinese Institute of Food Science and Technology, 2004, 4(4):1-6.
LI L R L, ZHANG C C, LIU B S, et al.Comparative study on the carbohydrate utilization capacity of different Bifidobacterium pseudocatenulatum[J].Food and Fermentation Industries, 2023, 49(14):94-102.