酸胁迫限制了工业微生物的生长,提高微生物耐酸性有利于维持生产过程的稳定性并降低发酵成本[1]。生物膜由胞外多糖和蛋白质构成,还含有少量腐殖质、核酸和脂类等物质,能够增强微生物对环境压力、抗生素和宿主免疫的抵抗能力[2]。环二鸟苷酸(cyclic diguanylate monophosphate,c-di-GMP)作为细菌的第二信使,由二鸟苷酸环化酶(diguanylate cyclases, DGCs)催化2分子GTP合成。Escherichia coli K12中多个DGCs参与生物膜形成[3]。如DgcE产生的c-di-GMP可以解除二鸟苷酸环化酶M(diguanylate cyclase,DgcM)和MlrA(a DNA-binding transcriptional activator)之间的相互作用,从而激活生物膜主调节因子CsgD,而参与纤维素合成的关键酶DgcC又受到CsgD的控制[4]。DgcQ产生的c-di-GMP能够降低细菌运动性,促进细菌聚集[5]。过表达DgcO可以促进卷曲菌毛的形成[6]。探索控制这些基因表达的工具对于研究E.coli生物膜形成至关重要。
光遗传学因高特异性和低毒性成为诱导表达的理想策略[7]。例如,LEVSKAYA等[8]基于光敏蛋白Cph1和大肠杆菌EnvZ/OmpR双组分信号通路,首次在细菌中创建了黑暗条件下激活,而红光下抑制目标基因表达的光控基因表达系统。WANG等[9]开发的响应蓝光的LightOn系统成功调控了胰岛素的表达与分泌,有效地降低了I型糖尿病小鼠的血糖水平。PU等[10]将c-di-GMP降解蛋白PA21333整合到光遗传系统中,通过蓝光刺激抑制铜绿假单胞菌生物膜的形成,导致生物膜更加稀疏。
来自Erythrobacter litoralis HTCC2594的EL222蓝光响应蛋白在蓝光照射(450 nm)下,光氧电压结构域受到刺激发生二聚化,特异性地结合到启动子的确定序列从而启动报告基因的转录和翻译[11]。本研究基于EL222的光学特性,开发了双质粒调控系统,用于精确调控生物膜形成关键基因csgD和dgcM,增加了E.coli的生物膜含量和耐酸性。探究其内源性机制发现,耐酸性提高可以归因于生物膜中多糖和蛋白质增多、可溶性微生物代谢产物(soluble microbial products,SMP)减少,结合实时荧光定量逆转录PCR(quantitative reverse transcription PCR,RT-qPCR)结果显示,CsgD和DgcM过表达菌株中纤维素和卷曲菌毛形成相关基因的转录水平显著改变,这些作用共同提高了生物膜结构的稳定性。通过光遗传调控系统调节生物膜形成关键基因不仅增强了E.coli的耐酸性,也为微生物有机酸发酵的应用提供了重要的参考和指导。
1 材料和方法
1.1 实验材料
E.coli JM109,本实验室保藏。Luria-Bertani(LB)培养基,中国国药化学试剂有限公司;高保真酶、一步克隆试剂盒、胶回收试剂盒、质粒提取试剂盒、总RNA提取试剂盒、RT-qPCR试剂盒、蛋白浓度试剂盒,南京诺唯赞生物科技有限公司;c-di-GMP检测试剂盒,上海恒远生物科技有限公司;引物设计、基因合成和Sanger测序,苏州金唯智生物科技有限公司。
1.2 实验仪器
LGJ-12A真空冷冻干燥机,北京四环科技有限公司;ReadMax 1500型光吸收全波长酶标仪,上海闪谱生物科技有限公司;SB-80DTD超声波清洗器、SCIENTZ-IID超声波破碎机,宁波新芝生物科技有限公司;AF-7000荧光分光光度计,日本日立公司;SU8220扫描电子显微镜(scanning electron microscope,SEM),日本日立公司;QuantStudio 3D 数字PCR系统,美国赛默飞公司;Synergy H4酶标仪,美国伯腾仪器有限公司。KM-2FL200200蓝光面板(波长450 nm),东莞科麦视觉技术有限公司;QNano超微分光光度计,上海翌圣生物有限公司。
1.3 实验方法
1.3.1 质粒构建
本研究使用一步克隆法分别构建了单质粒和双质粒蓝光调控系统。单质粒调控系统将el222(Gene ID:3868928)和荧光报告基因mKate克隆到pACYCDuet-1中。双质粒系统将el222和mKate分别克隆到pACYCDuet-1和pCDFDuet-1中。为了调控生物膜相关基因的表达,从E.coli JM109中扩增dgcC、csgD、dgcE、dgcM、dgcO和dgcQ,并通过一步克隆法替换mKate。
1.3.2 验证蓝光诱导系统的有效性
携带单质粒和双质粒系统的E.coli JM109被分别接种到含有相应抗生素的LB培养基中,37 ℃,200 r/min活化12 h。为了测定最适光照强度,将种子液接种于12孔细胞培养板中于不同光照强度下培养12 h后检测荧光表达量(激发波长和发射波长分别为588 nm和645 nm)。为了测定最佳调控倍数,37 ℃、200 r/min进行蓝光/黑暗培养。第4 h时开始用酶标仪测量荧光表达量与OD600,之后每隔2 h测量1次,总共培养12 h。
1.3.3 过表达菌株耐酸生长和结晶紫染色
在pH 7.0、5.0和4.5的LB培养基中检测E.coli JM109的生长表现(用1 mol/L柠檬酸溶液调节培养基的pH值)。将携带有不同基因的E.coli JM109活化后转移至12孔细胞培养板中,37 ℃、200 r/min蓝光条件下培养12 h,测量OD600。参考KIM等[12]的方法利用结晶紫染色测定菌株生物膜形成能力,以OD595来反映生物膜形成情况。
1.3.4 胞外聚合物(extracellular polymeric substances,EPS)组成成分分析
EPS是生物膜的主要成分。将活化后的种子液按1%的接种量接种于pH 5.0的LB培养基中,37 ℃、200 r/min蓝光/黑暗条件下培养36 h。一组样品冷冻干燥后测量细菌干重。另一组样品用超声加热法提取EPS。EPS中多糖含量用苯酚-硫酸法测定[13],蛋白质含量用Bradford法测定,使用三维荧光技术对EPS组成进行分析,得到的荧光光谱用积分区域法进行计算以得到EPS含量变化[14]。
1.3.5 刚果红实验
将E.coli JM109接种到12孔细胞培养板培养至对数生长期。4 ℃、3 000 r/min离心5 min收集细胞,PBS清洗2次调整OD600值为3,于pH 5.0的刚果红培养基中心滴加6 μL菌体,28 ℃培养48 h后观察菌落形态。
1.3.6 SEM观察细胞形态
将过夜培养的细菌稀释至1×107 CFU/mL,于pH 5.0的培养基中37 ℃蓝光照射培养48 h。取1 mL菌液经固定、脱水、干燥和喷金后使用SEM观察细胞形态。
1.3.7 检测胞内c-di-GMP浓度
将种子液接种到pH 5.0的培养基中,在蓝光/黑暗条件下37 ℃、200 r/min培养12 h后收集细胞。PBS清洗重悬后,用超声波破碎机8 000 r/min、10 min裂解细胞,得到上清液。用c-di-GMP检测试剂盒测定胞内c-di-GMP水平,同时用Bradford蛋白检测试剂盒检测蛋白质浓度。c-di-GMP浓度以pmol/mg总蛋白表示。
1.3.8 RT-qPCR
将种子液接种于pH 5.0的培养基中,37 ℃、200 r/min蓝光照射培养至对数生长期。取1.0 mL样品离心,4 ℃、10 000 r/min离心3 min弃上清液,提取细菌总RNA于-80 ℃保存。使用HiScript IV RT SuperMix for qPCR反转录试剂盒进行gDNA去除和反转录,产物于-20 ℃保存。以16S rRNA为内参,用ChamQ Universal SYBR qPCR Master Mix进行RT-qPCR以评估pH 5.0下基因的相对表达量。使用QuantStudio 3D数字PCR仪,将Ct值换算为2-ΔΔCt以计算基因相对表达量。
2 结果与讨论
2.1 蓝光调控系统的构建与功能验证
不同光照强度下荧光表达量随光照强度增加先升后降,15 mW/cm2时达到最佳(图1-a)。荧光蛋白表达量未随光照强度增加而持续增加,可能是由于蓝光诱导产生的活性氧损伤了胞内含巯基的生物大分子[15]。图1-b和图1-c分别展示了在15 mW/cm2光照强度下单质粒与双质粒系统的调控效果。蓝光照射12 h后,单质粒和双质粒系统的荧光表达量分别是黑暗条件的4.5倍和8.6倍。因此后续实验均使用双质粒系统调控目标基因。
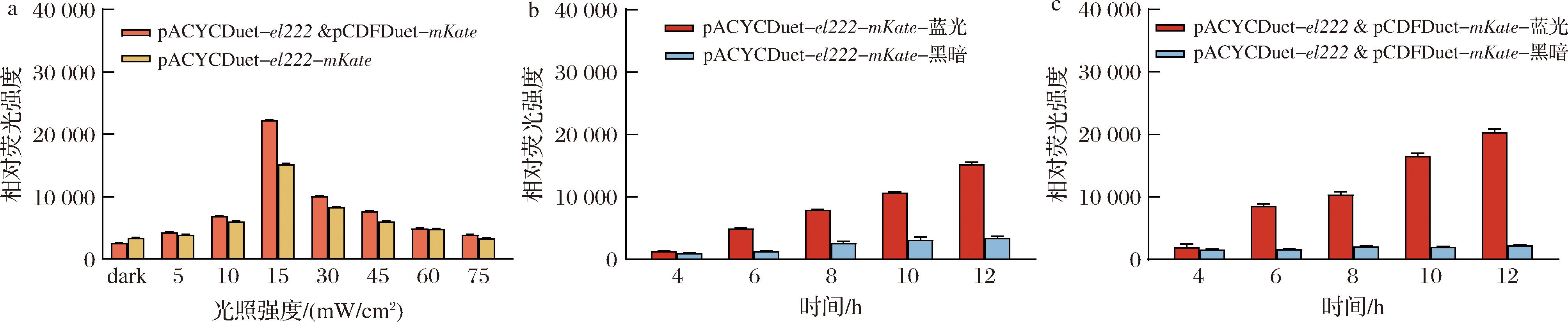
a-不同光照强度对蓝光调控系统的影响;b-最优光强度下单质粒调控系统在蓝光/黑暗条件下的调控效果;c-最优光强度下双质粒调控系统在蓝光/黑暗条件下的调控效果
图1 蓝光调控系统的诱导表达效果
Fig.1 Induced expression effect of blue light-regulated system
2.2 蓝光调控生物膜相关基因过表达
2.2.1 不同pH下过表达菌株的生长性能
如图2所示,在pH 7.0,所有菌株生长无显著差异,表明中性环境下基因表达对细菌影响小。在pH 5.0,CsgD、DgcM和DgcE过表达菌株的OD600比对照组分别高出22.3%、16.4%和9.0%。而在pH 4.5,所有菌株OD600约为0.6,表明此时的酸性环境不利于菌株生长[16]。但是CsgD和DgcM过表达菌株的OD600相比于对照组仍提高了13.0%和9.3%。以上结果表明CsgD和DgcM过表达能够提高E.coli的耐酸性。类似地,HU等[17]发现在BL101和BL102菌株中过表达A.caldu来源的cfa能够使菌株的耐酸性分别提高9%和18%。
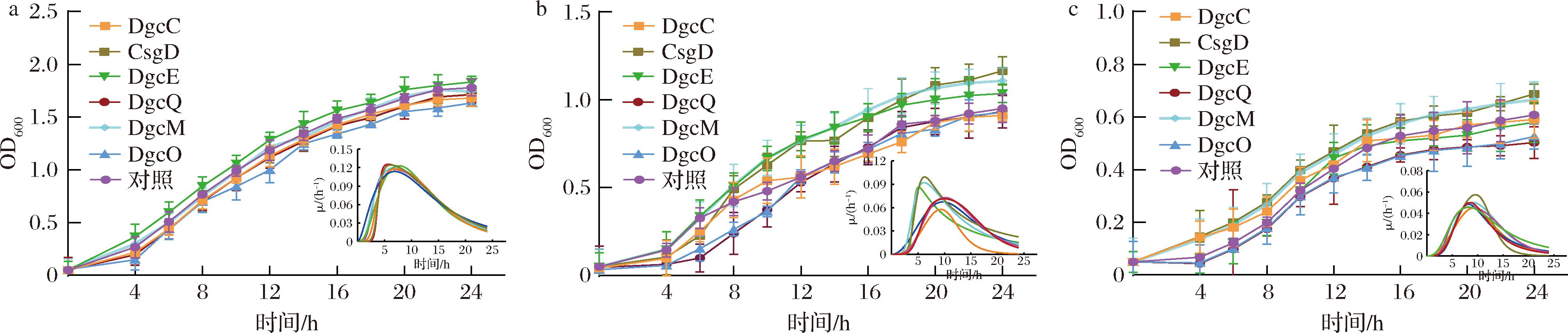
a-pH 7.0;b-pH 5.0;c-pH 4.5
图2 不同pH条件下诱导表达不同基因后大肠杆菌的生长曲线和比生长速率
Fig.2 Growth curves and specific growth rate of E.coli after inducing the expression of different genes using a blue light-regulated system under varying pH
2.2.2 过表达菌株生物膜含量分析
图3显示蓝光诱导后所有菌株的生物膜含量均增加,且在pH 5.0时,CsgD、DgcE和DgcM的OD595分别是黑暗条件下的3.5、2.4和3.3倍。CsgD作为生物膜的主调节因子,受到DgcM与MlrA复合物的激活,而DgcM-MlrA复合物又受到DgcE的调控作用[18]。因此,推测上述3个生物膜含量明显增加的过表达菌株是通过影响CsgD的表达量来促进生物膜形成。
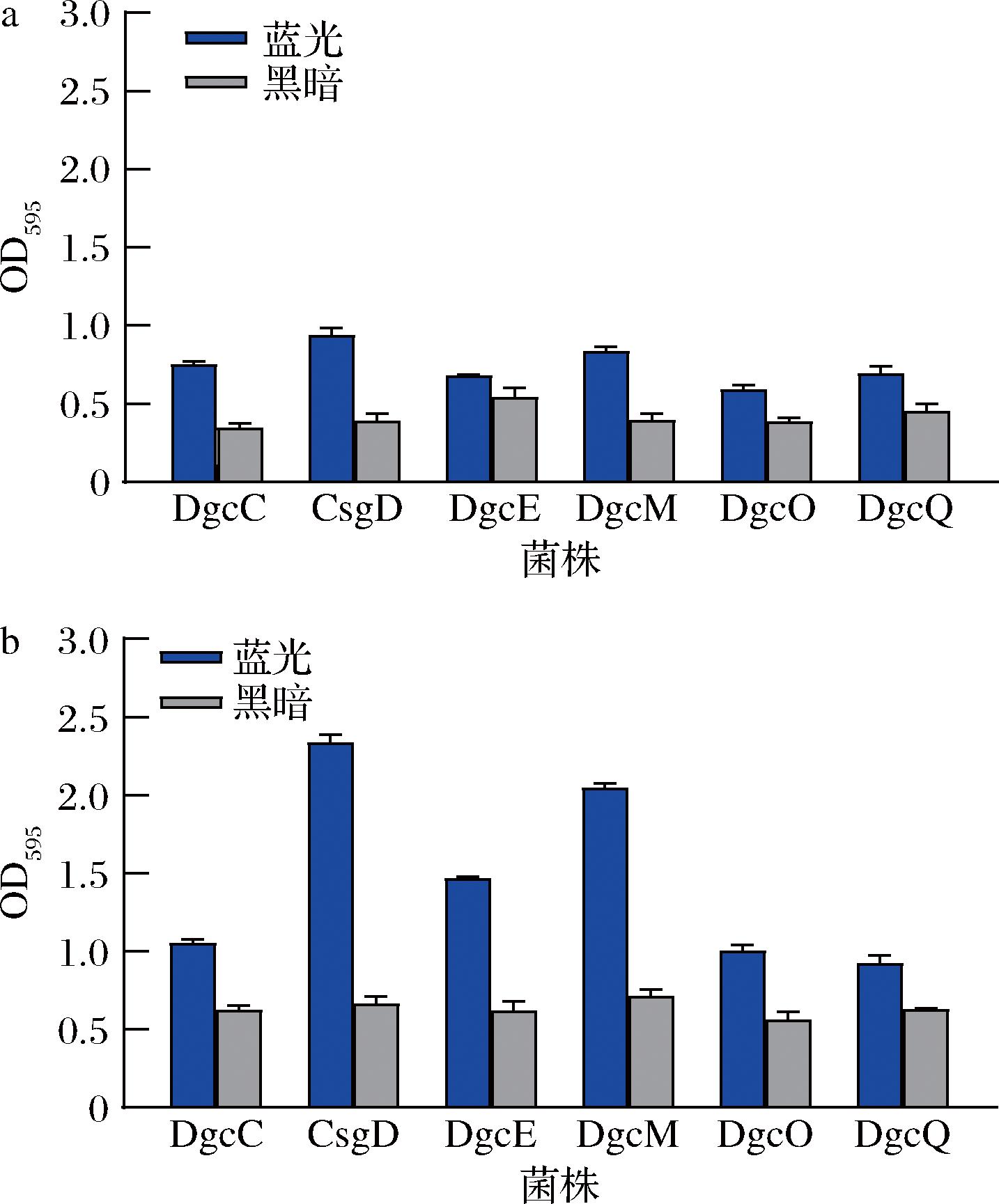
a-pH 7.0;b-pH 5.0
图3 不同表达菌株在蓝光/黑暗条件下生物膜形成能力差异比较
Fig.3 Biofilm formation ability of different expression strain under bluelight irradiation or dark conditions
2.2.3 EPS中蛋白质和多糖含量分析
多糖和蛋白质是EPS主要成分,对生物膜结构至关重要。如图4所示,在蓝光条件下,CsgD和DgcM菌株的多糖和蛋白质总量分别达到23.0和21.6 mg/g-细胞干重,是黑暗条件下的2.2倍和1.9倍,且CsgD(363.5%)和DgcM(327.1%)中蛋白质/多糖比值最低。以上结果表明蓝光诱导CsgD和DgcM过表达促进细胞产生更多的多糖和蛋白质。海洋假交替单胞菌生物膜形式与游离态相比,EPS增加了149 mg/L,且多糖和蛋白质含量均有提升,这有助于增强细胞黏附并减少活性氧损伤[19]。
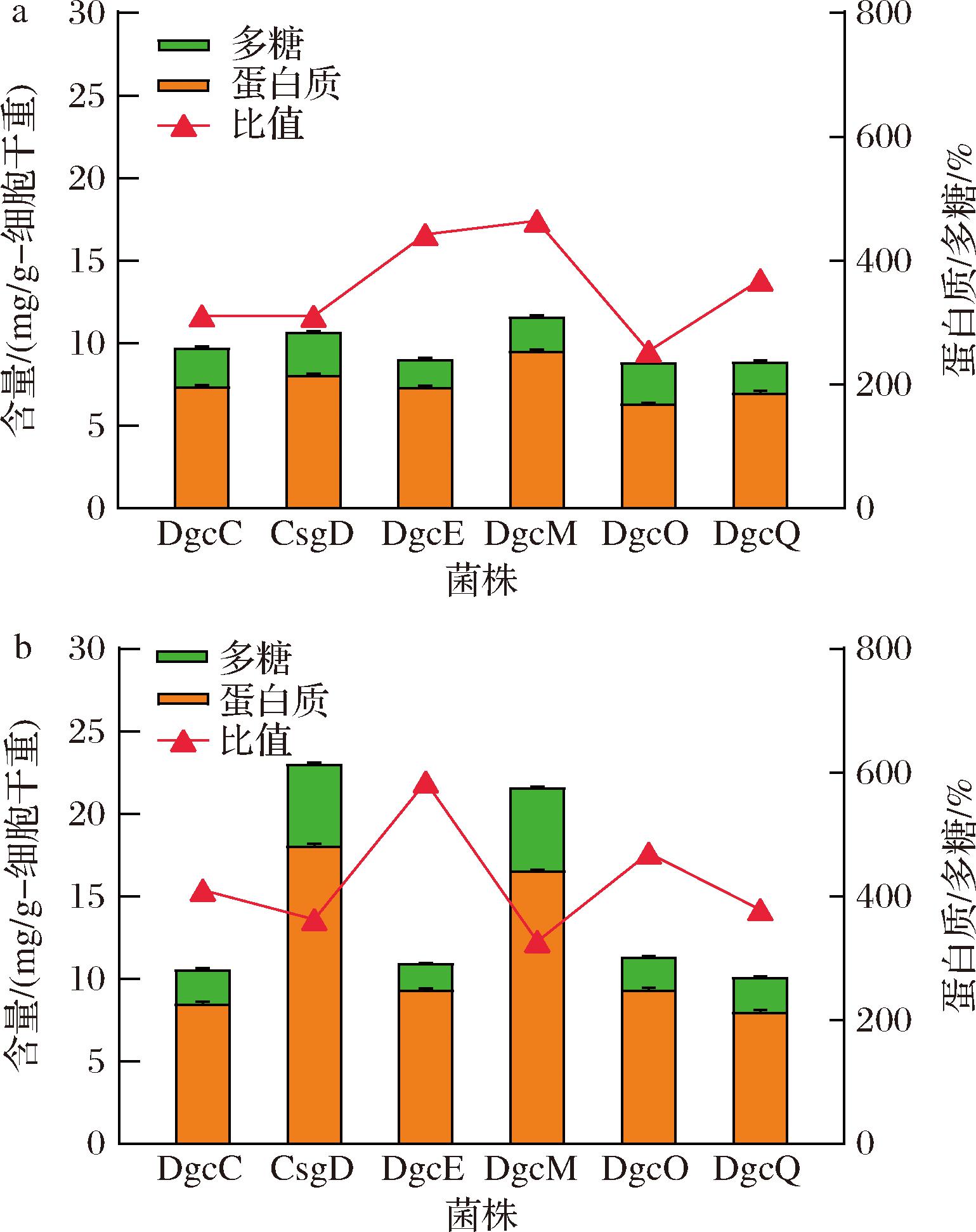
a-黑暗;b-蓝光
图4 pH 5.0条件下诱导表达不同基因后EPS中多糖和蛋白质组成
Fig.4 The composition of polysaccharide and protein in EPS after inducing expression of different genes under pH 5.0 conditions
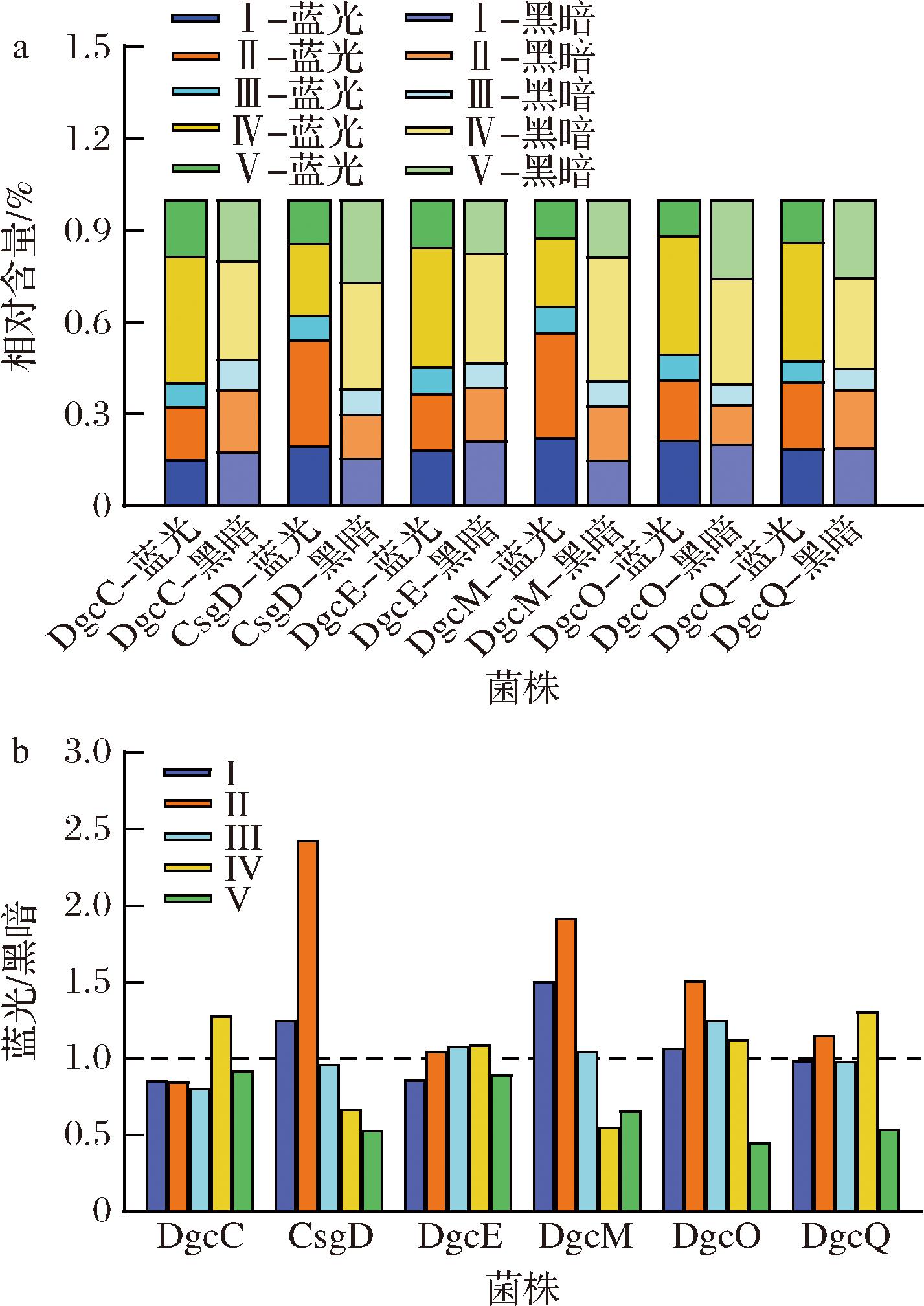
a-蓝光或黑暗条件下EPS中各种物质占比;b-同种物质蓝光照射组与黑暗组的比值
图5 基于积分域法分析蓝光或黑暗条件下EPS中各种物质的组成
Fig.5 Analysis of the composition of various substances in EPS under blue light irradiation or dark conditions based on the integration region method
注:I-V分别为酪氨酸类物质、色氨酸类物质、富里酸类物质、SMP和腐殖酸类有机物
2.2.4 EPS组成成分/含量变化分析
图5-a是通过积分区域法而得到的五类物质的相对含量。蓝光照射后,CsgD和DgcM中蛋白质总量(54.3%,57.6%)显著高于其他组(32.2%~41.3%)、IV(23.1%,21.4%)的含量显著低于其他组(38.7%~42.2%)。图5-b展示了同种物质蓝光照射组与黑暗组的比值,直观地反映了各物质的增减情况。CsgD中II和IV的含量分别是黑暗下的2.4倍和0.7倍,DgcM中I、II和IV的含量分别是黑暗下的1.5倍、1.9倍和0.6倍。芳香族氨基酸类物质增加主要是通过提高细胞表面疏水性以促进细胞聚集,从而增强EPS的总量和持久性。HSUEH等[20]证明向低营养培养基中添加1%(质量分数)的酪氨酸可以延长生物膜的持续期。SMP的产生与基质利用率、细胞衰亡和EPS水解有关,过多的SMP会抑制微生物代谢活性[21]。因此,SMP减少意味着菌株可能通过增强自身代谢活动以抵御酸胁迫。
2.3 蓝光调控CsgD和DgcM过表达菌株对菌落和细胞形态的影响
2.3.1 蓝光调控系统对菌落形态的影响
图6展示了pH 5.0下,CsgD和DgcM菌株在刚果红培养基上的菌落形态差异。蓝光照射下,CsgD菌落边缘形成密集的短皱纹,菌落中心向外部发散形成径向脊;DgcM菌落边缘形成了长而高的脊,中间区域出现点状凸起。相反,黑暗条件下,CsgD菌落边缘皱纹减少,径向脊消失;DgcM菌落边缘皱纹变得密集短小,中心凸起减少。菌落的不同形态取决于卷曲菌毛和纤维素的不同组合,二者共存会出现长而高的脊、同时缺乏则无形态分化[22]。以上结果表明蓝光照射后CsgD和DgcM菌株产生了更多卷曲菌毛和纤维素。
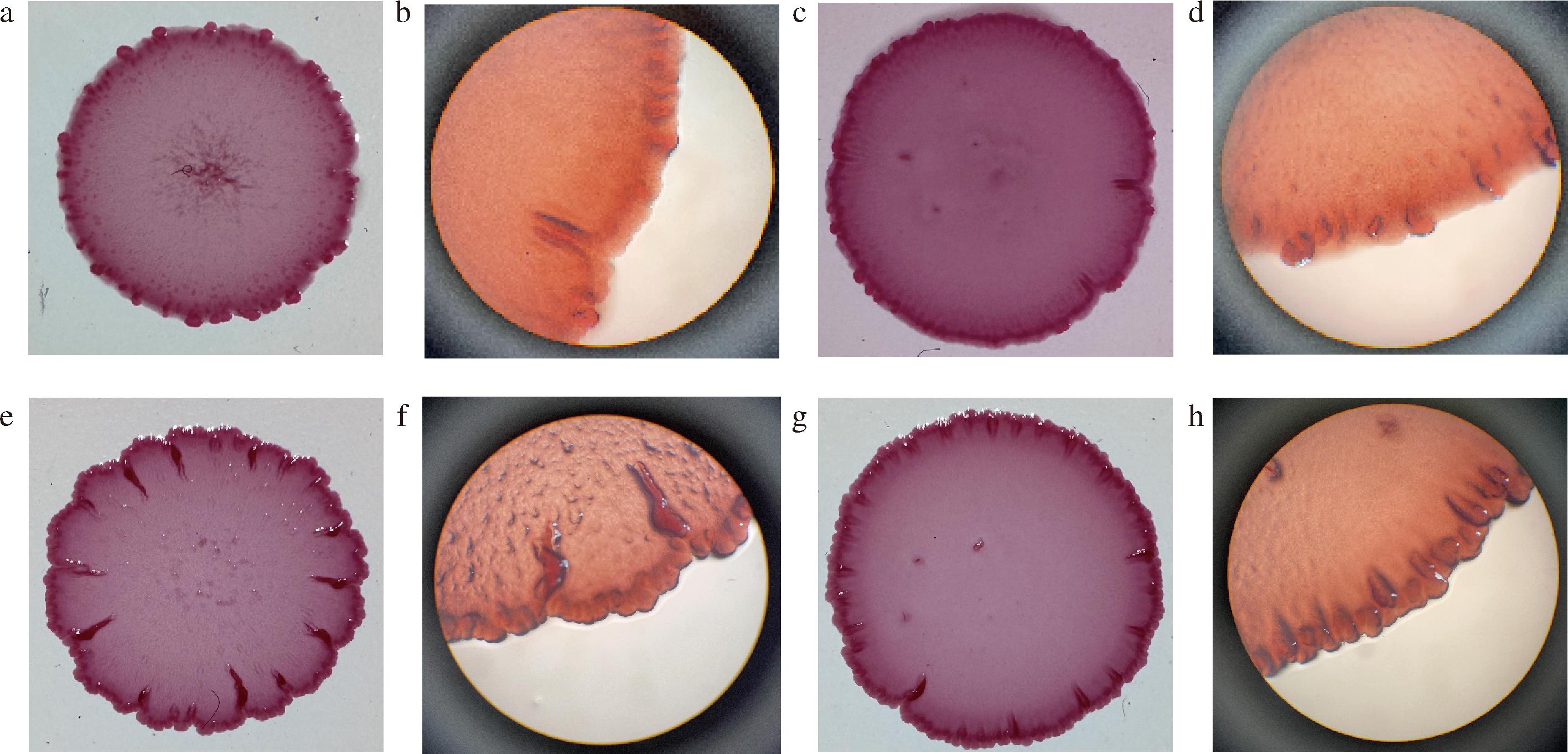
a-CsgD 蓝光1×;b-CsgD 蓝光4×;c-CsgD 黑暗1×;d-CsgD 黑暗 4×;e-DgcM 蓝光1×;f-DgcM 蓝光 4×;g-DgcM 黑暗1×;h-DgcM 黑暗 4×
图6 pH 5.0时CsgD和DgcM表达菌株在蓝光/黑暗条件下的菌落形态
Fig.6 Colony morphology of CsgD/DgcM overexpression strains at pH 5.0 under blue light irradiation or dark conditions
2.3.2 SEM观察生物膜形成和细胞形态变化
如图7所示,在黑暗条件下,CsgD和DgcM细胞形状都为规则的杆状,且生物膜形成较少。蓝光照射后,CsgD出现个别细胞形态变长,两者生物膜形成均明显增多。说明在酸胁迫下,蓝光诱导基因表达会使菌株细胞形态发生改变,并且产生更多的生物膜来抵御酸胁迫。张爱静等[23]发现当E.coli ATCC 43889受到热胁迫时,也会出现细胞形态变长、生物膜增强,还会伴随外膜蛋白和饱和脂肪酸含量升高,这些表现有利于菌株增强耐热性,提高其生存能力。
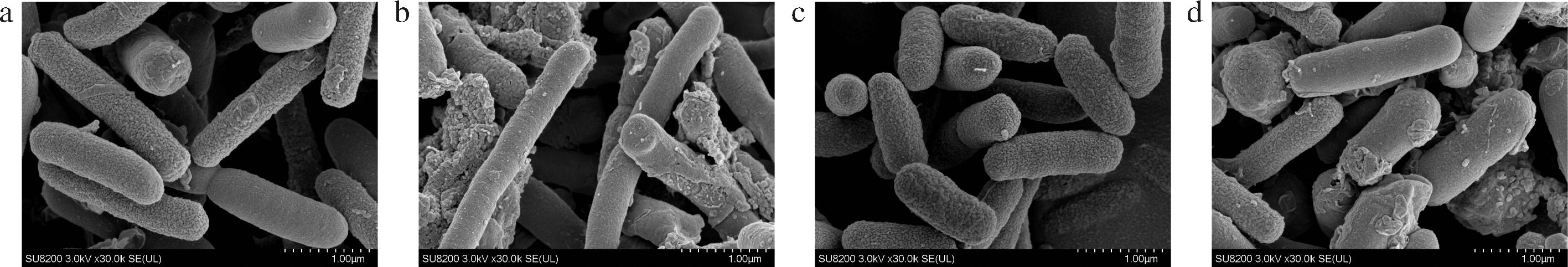
a-CsgD 黑暗;b-CsgD 蓝光;c-DgcM 黑暗;d-DgcM 蓝光
图7 pH 5.0时SEM下CsgD和DgcM过表达菌株在蓝光/黑暗条件下的细胞形貌
Fig.7 Cell morphology of CsgD and DgcM overexpression strains under blue light irradiation or dark conditions by SEM at pH 5.0
2.4 CsgD和DgcM过表达菌株生物膜形成和耐酸性增强的内在机制
c-di-GMP能够调节与生物膜形成相关的基因表达,促进胞外多糖和蛋白质的合成与释放。如图8所示,不同表达菌株在蓝光/黑暗条件下胞内c-di-GMP浓度都与JM109相当,pH 7.0和pH 5.0时胞内c-di-GMP分别在0.8和0.6 pmol/mg总蛋白左右。这表明细胞内c-di-GMP浓度保持在相对稳定的范围。这一结果与SARENKO等[3]研究结果一致,即几乎所有控制c-di-GMP的酶都平行表达,不同的DGCs或PDEs控制着细胞不同的生命活动,并不会影响胞内c-di-GMP水平。
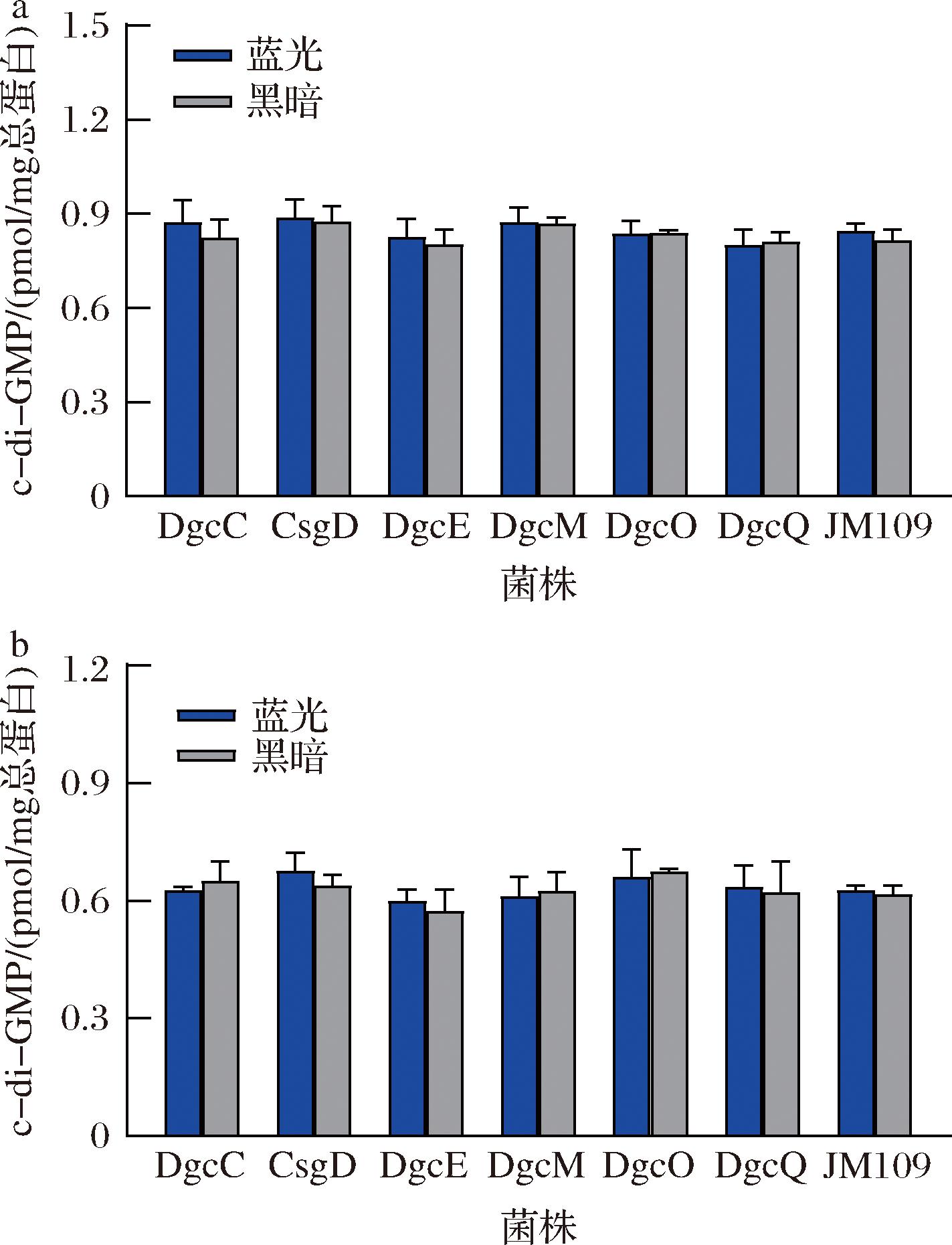
a-pH 7.0;b-pH 5.0
图8 在不同pH条件下诱导表达不同基因后胞内c-di-GMP浓度差异
Fig.8 Effects of overexpression different genes on intracellular c-di-GMP
如图9所示,CsgD过表达菌株中ropS和mlrA分别提高2.0和1.9倍,DgcM过表达菌株中ropS和mlrA分别提高2.2和1.5倍。RpoS能够诱导RNAP识别并结合到特定的启动子区域,MlrA能够增强RpoS对启动子的亲和力,因此在生物膜的形成过程中,rpoS和mlrA上调有利于卷曲菌毛和纤维素合成。flhD和flhC在CsgD中分别减少55.2%、46.3%,在DgcM中分别减少44.5%、33.2%。FlhDC操纵子是鞭毛生成的主调控因子[24]。TEPLITSKI等[25]发现,在沙门氏菌中敲除flhD和flhC提高了生物膜形成能力,故flhDC下降有利于减缓鞭毛运动,促进细胞定植。CsgD和DgcM菌株中pdeR分别减少47.7%、27.8%。WEBER等[26]报道PdeR的表达减少有利于局部c-di-GMP浓度升高,起到正向调控卷曲菌毛表达的作用。CsgD菌株中csgA和csgB分别提高2.7和1.8倍,DgcM菌株中csgA和csgB分别提高1.6和1.3倍,CsgA和CsgB是构成卷曲菌毛的2个亚单位,因此csgA和csgB表达上调有利于形成卷曲菌毛。DgcM菌株中bcsA和bcsB分别提高2.0和1.9倍,c-di-GMP变构激活BcsA-BcsB复合体,最终促进纤维素合成[27]。总之,对EPS组成和细胞形态分析表明,提高耐酸性的内在机制可以归因于卷曲菌毛和纤维素的增加,这反映在多糖和蛋白质的增加以及SMP的减少。RT-qPCR进一步证实了上述机制,即CsgD和DgcM在蓝光刺激后,卷曲菌毛和纤维素相关基因转录水平显著上调。同时,与鞭毛旋转相关的基因表达水平下调。
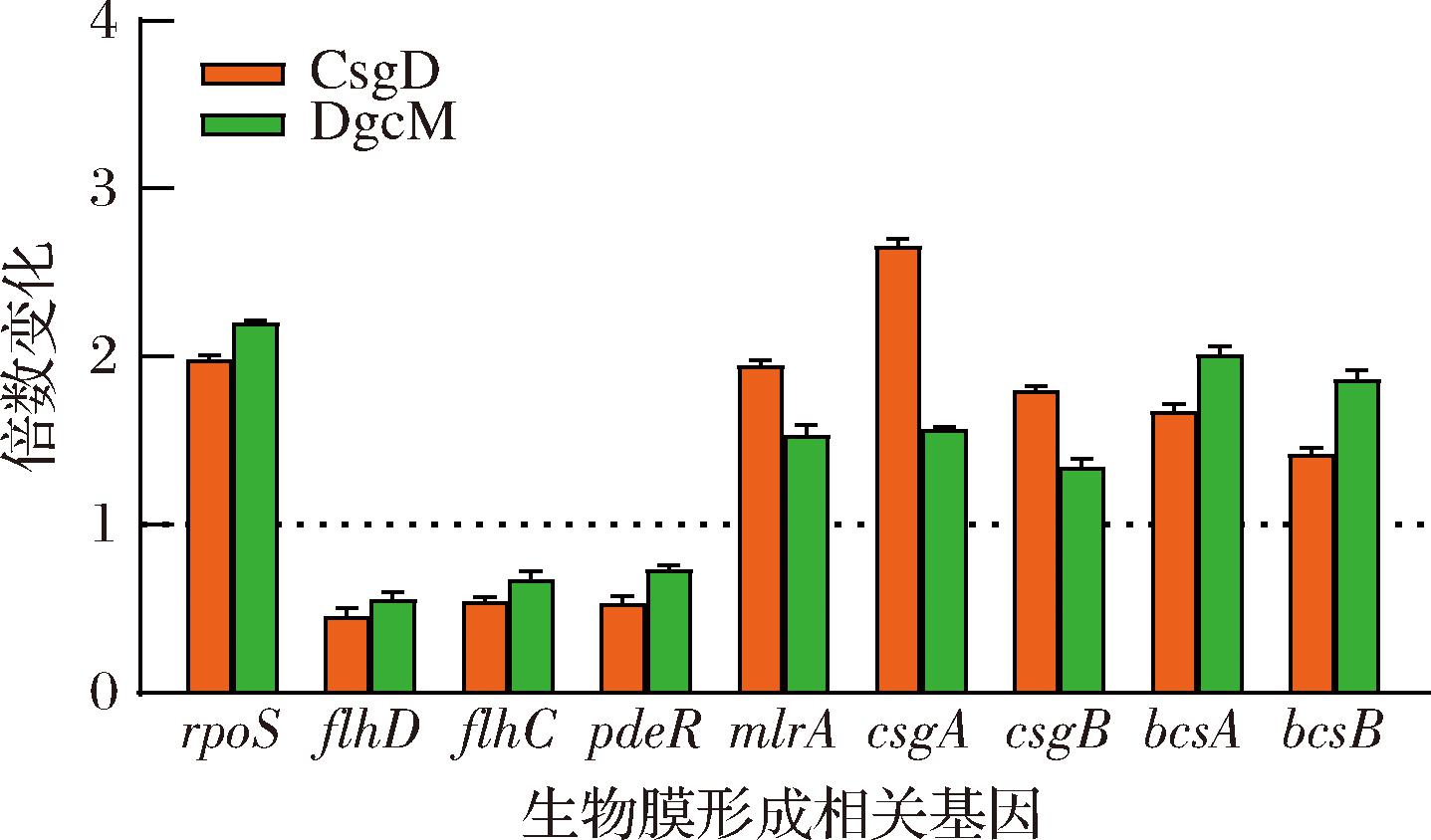
图9 RT-qPCR分析蓝光/黑暗条件下CsgD和DgcM菌株生物膜形成相关基因转录水平的差异
Fig.9 The transcription levels of different biofilm-related genes in CsgD/DgcM overexpression strains under blue light/dark conditions via RT-qPCR analysis
3 结论
本研究基于蓝光诱导蛋白EL222构建了双质粒调控系统,通过调控生物膜形成关键基因csgD和dgcM提高了大肠杆菌的耐酸性。通过分析EPS组成、细胞形态以及生物膜相关基因转录水平,证实了该调控系统通过促进生物膜形成以增强大肠杆菌耐酸性方面的有效性。为未来利用光遗传学提高工业微生物耐酸性研究提供了有价值的参考。为了更有效地利用光遗传学系统调节细菌的生命活动,未来应考虑将蓝光调控系统与其他光调控组分结合,开发出多组分共同调控的光遗传学系统。
[1] GUAN N Z, LI J H, SHIN H D, et al.Microbial response to environmental stresses:From fundamental mechanisms to practical applications[J].Applied Microbiology and Biotechnology, 2017, 101(10):3991-4008.
[2] HALL-STOODLEY L, COSTERTON J W, STOODLEY P.Bacterial biofilms:From the natural environment to infectious diseases[J].Nature Reviews.Microbiology, 2004, 2(2):95-108.
[3] SARENKO O, KLAUCK G, WILKE F M, et al.More than enzymes that make or break cyclic di-GMP-local signaling in the interactome of GGDEF/EAL domain proteins of Escherichia coli[J].mBio, 2017, 8(5):e01639-17.
[4] LINDENBERG S, KLAUCK G, PESAVENTO C, et al.The EAL domain protein YciR acts as a trigger enzyme in a c-di-GMP signalling cascade in E.coli biofilm control[J].The EMBO Journal, 2013, 32(14):2001-2014.
[5] FANG X, GOMELSKY M.A post-translational, c-di-GMP-dependent mechanism regulating flagellar motility[J].Molecular Microbiology, 2010, 76(5):1295-1305.
[6] BROMBACHER E, BARATTO A, DOREL C, et al.Gene expression regulation by the Curli activator CsgD protein:Modulation of cellulose biosynthesis and control of negative determinants for microbial adhesion[J].Journal of Bacteriology, 2006, 188(6):2027-2037.
[7] TAN P, HE L, HUANG Y, et al.Optophysiology:Illuminating cell physiology with optogenetics[J].Physiological Reviews, 2022, 102(3):1263-1325.
[8] LEVSKAYA A, CHEVALIER A A, TABOR J J, et al.Synthetic biology:Engineering Escherichia coli to see light[J].Nature, 2005, 438(7067):441-442.
[9] WANG X, CHEN X J, YANG Y.Spatiotemporal control of gene expression by a light-switchable transgene system[J].Nature Methods, 2012, 9(3):266-269.[10] PU L, YANG S, XIA A G, et al.Optogenetics manipulation enables prevention of biofilm formation of engineered Pseudomonas aeruginosa on surfaces[J].ACS Synthetic Biology, 2018, 7(1):200-208.
[11] JAYARAMAN P, DEVARAJAN K, CHUA T K, et al.Blue light-mediated transcriptional activation and repression of gene expression in bacteria[J].Nucleic Acids Research, 2016, 44(14):6994-7005.
[12] KIM M J, LIM E S, KIM J S.Enzymatic inactivation of pathogenic and nonpathogenic bacteria in biofilms in combination with chlorine[J].Journal of Food Protection, 2019, 82(4):605-614.
[13] DUBOIS M, GILLES K A, HAMILTON J K, et al.Colorimetric method for determination of sugars and related substances[J].Analytical Chemistry, 1956, 28(3):350-356.
[14] JONES E R, VAN VLIET M T H, QADIR M, et al.Country-level and gridded estimates of wastewater production, collection, treatment and reuse[J].Earth System Science Data, 2021, 13(2):237-254.
[15] DAI T H, GUPTA A, MURRAY C K, et al.Blue light for infectious diseases:Propionibacterium acnes, Helicobacter pylori, and beyond?[J].Drug Resistance Updates, 2012, 15(4):223-236.
[16] XU Y, ZHAO Z, TONG W H, et al.An acid-tolerance response system protecting exponentially growing Escherichia coli[J].Nature Communications, 2020, 11(1):1496.
[17] HU W B, TONG Y J, LIU J J, et al.Improving acid resistance of Escherichia coli base on the CfaS-mediated membrane engineering strategy derived from extreme acidophile[J].Frontiers in Bioengineering and Biotechnology, 2023, 11:1158931.
[18] PFIFFER V, SARENKO O, POSSLING A, et al.Genetic dissection of Escherichia coli’s master diguanylate cyclase DgcE:Role of the N-terminal MASE1 domain and direct signal input from a GTPase partner system[J].PLoS Genetics, 2019, 15(4):e1008059.
[19] 吕菁萍, 李泽龙, 张鹤睿, 等.海洋假交替单胞菌生物膜产ROS特性研究[J].大连理工大学学报, 2021, 61(6):569-575.LYU J P, LI Z L, ZHANG H R, et al.Study of ROS production characteristics of biofilm by marine Pseudoalteromonas sp[J].Journal of Dalian University of Technology, 2021, 61(6):569-575.
[20] HSUEH Y H, SOMERS E B, WONG A C.Characterization of the CodY gene and its influence on biofilm formation in Bacillus cereus[J].Archives of Microbiology, 2008, 189(6):557-568.
[21] KUNACHEVA C, STUCKEY D C.Analytical methods for soluble microbial products (SMP) and extracellular polymers (ECP) in wastewater treatment systems:A review[J].Water Research, 2014, 61:1-18.
[22] SERRA D O, RICHTER A M, HENGGE R.Cellulose as an architectural element in spatially structured Escherichia coli biofilms[J].Journal of Bacteriology, 2013, 195(24):5540-5554.
[23] 张爱静, 李琳琼, 王鹏杰等. 热胁迫对大肠杆菌细胞膜和膜蛋白的影响[J]. 中国农业科学, 2020, 53(5): 1046-1057.ZHANG A J, LI L Q, WANG P J, et al. Effects of heat stress on cell membrane and membrane protein of Escherichia coli[J]. Journal of Integrative Agriculture, 2020, 53(5): 1046-1057.
[24] WANG S Y, FLEMING R T, WESTBROOK E M, et al.Structure of the Escherichia coli FlhDC complex, a prokaryotic heteromeric regulator of transcription[J].Journal of Molecular Biology, 2006, 355(4):798-808.
[25] TEPLITSKI M, AL-AGELY A, AHMER B M M.Contribution of the SirA regulon to biofilm formation in Salmonella enterica serovar Typhimurium[J].Microbiology, 2006, 152(11):3411-3424.
[26] WEBER H, PESAVENTO C, POSSLING A, et al.Cyclic-di-GMP-mediated signalling within the σ network of Escherichia coli[J].Molecular Microbiology, 2006, 62(4):1014-1034.
[27] MORGAN J L W, STRUMILLO J, ZIMMER J.Crystallographic snapshot of cellulose synthesis and membrane translocation[J].Nature, 2013, 493(7431):181-192.