葡萄酒是以葡萄或葡萄汁为原料,经酒精发酵(alcoholic fermentation, AF)酿造而成的酒精饮料[1]。AF是由酵母菌主导的将葡萄汁转化为葡萄酒的生物化学过程[2](如图1)。涉及主要真菌为酵母菌(酿酒酵母与非酿酒酵母),酿酒酵母在葡萄汁的AF中起关键作用。近年来,由于全球变暖,导致葡萄中糖含量的积累过多,造成葡萄酒中的平均乙醇含量升高[3]。
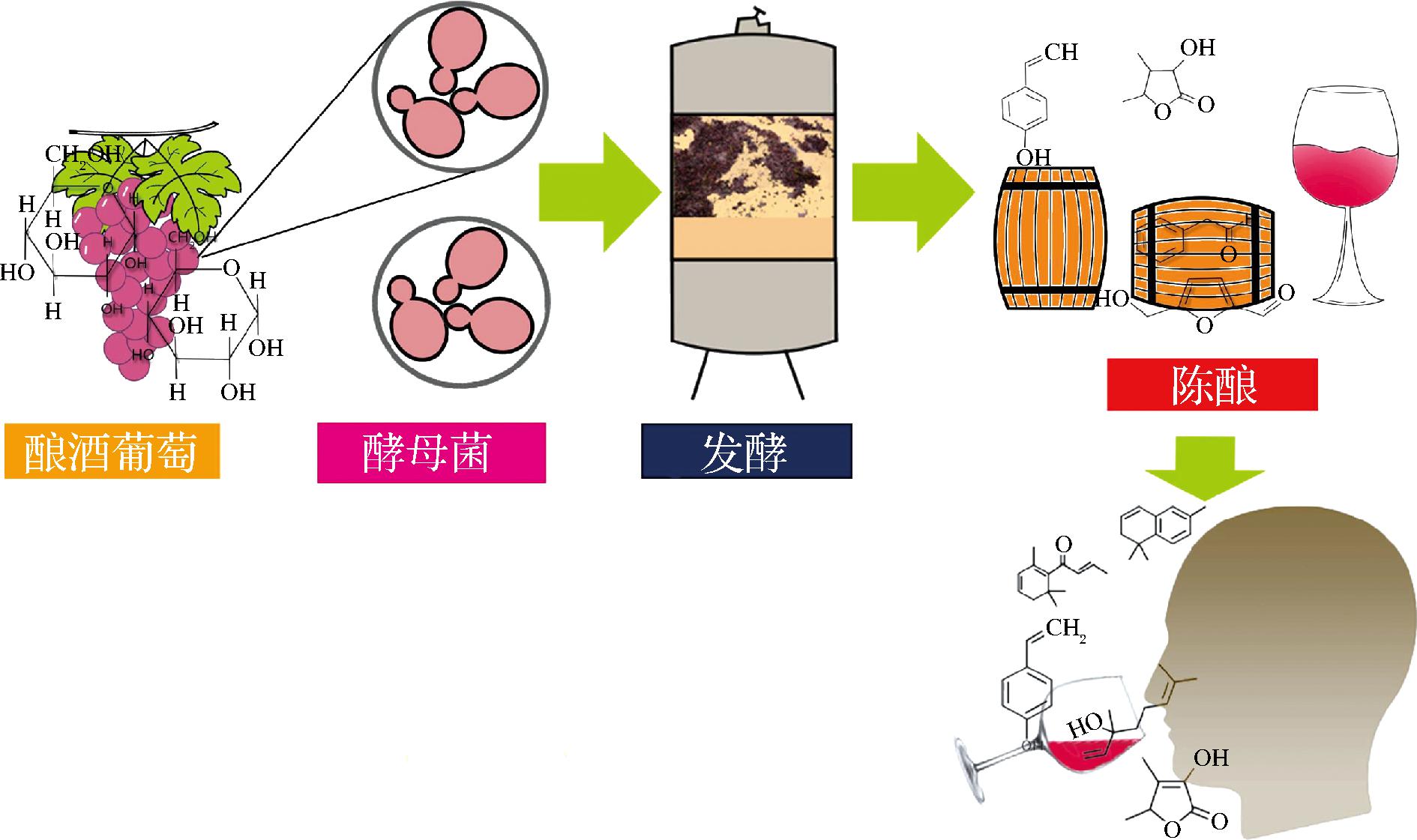
图1 酿酒酵母酒精发酵
Fig.1 Alcoholic fermentation of Saccharomyces cerevisiae
葡萄酒中过高浓度的乙醇会产生许多负面影响。如,增加葡萄酒的苦味和灼热感,掩盖某些重要挥发性芳香化合物(如酯类和萜类),严重影响葡萄酒的感官品质[4];此外,还会增加肝脏负担,损害消化系统等[5],危害人体健康[6]。在葡萄酒发酵中,高浓度酒精会抑制酵母细胞的生长和发酵活力(破坏细胞结构并削弱细胞活力)[7],导致发酵缓慢甚至停滞,影响葡萄酒发酵进程及质量[8]。为了解决葡萄酒中高乙醇含量引发的感官、健康、技术和经济等方面的问题,开发低酒度葡萄酒已势在必行。在降低葡萄酒中酒精含量的各种方法中,易于实施且不需要专用设备的微生物学方法有望在保持葡萄酒的感官特性和质量中发挥重要作用[9]。
葡萄酒中的乙醇主要来源于酵母菌利用葡萄中的糖发酵生成,葡萄原料的含糖量是影响酒精含量的重要因素。在葡萄酒发酵过程中,大约92%的糖(葡萄糖和果糖)用于合成乙醇和CO2,每消耗一克糖乙醇的产量为0.47 g[10],剩余的糖用于合成生物质和各种次级代谢产物。低醇葡萄酒是指将酵母菌的碳通量从乙醇代谢转移到其他代谢途径,但这种转移过程具有一定挑战性,不仅需要维持酵母菌内的碳平衡,而且必须维持细胞内氧化还原平衡[11]。由于从自然资源中分离酵母菌株的传统方法时间成本高,分离效率低,且具随机性。因此,有必要研究酿酒酵母乙醇代谢途径及调控基因,使用生物信息学和系统生物学的方法,更有助于发现酵母改良靶基因的遗传——表型关系[12]。从酿酒酵母乙醇代谢调控机制出发,选育优良的低产乙醇酿酒酵母菌株,从而从根本上解决葡萄酒乙醇产量过高的问题。
本文重点介绍了酿酒酵母酿造过程中乙醇代谢途径及其调控基因,以及低产乙醇菌株的选育方法,重点介绍了基于诱变育种、适应性进化、基因工程和合成生物学方法选育低产乙醇菌株的最新进展。
1 酿酒酵母乙醇代谢途径及调控基因
1.1 乙醇的合成代谢关键酶及其调控基因
在酿酒酵母中,乙醇生产的关键途径是糖酵解途径。如图2所示,主要步骤如下:(1)葡萄糖通过糖酵解途径分解成丙酮酸;(2)丙酮酸被脱羧酶催化生成CO2和乙醛;(3)乙醛通过ADH进一步催化成乙醇[13]。目前,对于低产乙醇的菌株筛选和代谢机理研究,国内外研究者主要以酿酒酵母作为研究对象,探究其通过各种生化反应消耗葡萄糖生产生物乙醇路径。
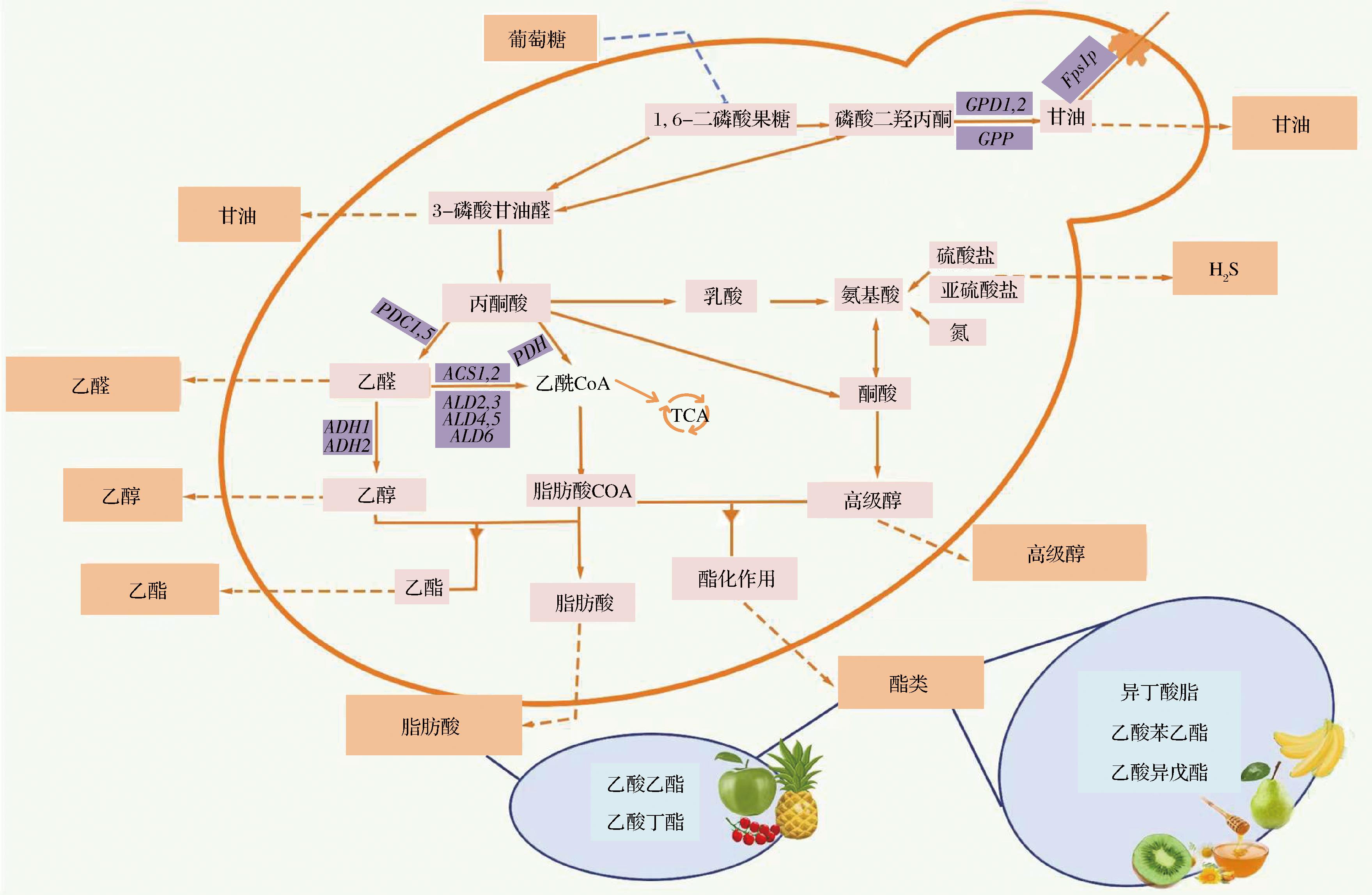
图2 酿酒酵母菌代谢途径及产乙醇相关基因
Fig.2 Metabolic pathway and related ethanol-producing genes of S. cerevisiae
注:PDC:丙酮酸脱羧酶编码基因;PDH:丙酮酸脱氢酶编码基因;GPD:甘油-3-磷酸脱氢酶编码基因;GPP:甘油-3-磷酸酶编码基因;Fps1p:甘油通道蛋白;ALD:乙醛脱氢酶编码基因;ADH:乙醇脱氢酶编码基因;ACS:乙酰辅酶A合成酶编码基因。
1.1.1 糖酵解途径对细胞乙醇产生的影响
在酿酒酵母中,糖酵解过程中3-磷酸甘油醛脱氢酶的NADH再生被乙醇生物合成过程中乙醇脱氢酶的NADH消耗所平衡。乙醇合成的减少将导致NADH再生/消耗的不平衡,并严重影响糖酵解的能量产生,进而影响正常细胞生长。
1.1.1.1 PDC和ADH基因
在糖酵解过程中,丙酮酸是重要的中间产物[14]。在低O2浓度下,酵母菌进行酒精发酵,丙酮酸转化为乙醇和CO2,而在较高的O2浓度下,酵母菌发生好氧发酵,丙酮酸转化为乙酰辅酶A和CO2,葡萄糖消耗量较低(此时酿酒酵母菌表现出Crabtree效应)[15]。主要由PDH和PDC对丙酮酸的代谢途径进行调节,乙醇是由包含PDC和ADH的内源性途径产生[16]。有研究提出通过好氧发酵来降低葡萄酒的乙醇含量,这会产生高水平含量的乙酸,严重影响葡萄酒质量[16]。
糖酵解途径含有由ADH1基因编码的PDH,在发酵过程中能催化乙醛还原为乙醇[14],但由于酿酒酵母中的Crabtree效应,大多数其他前体磷酸烯醇式丙酮酸被丙酮酸激酶转化为丙酮酸[15],然后通过PDC和ADH进一步代谢为乙醇[17]。有相关研究表明,编码PDC主要亚型是PDC1,该基因的缺失使PDC活性降低了约30%[18]。但PDH和PDC也是与细胞适应性有关的重要酶,消除或降低其活性会导致细胞生长和生产力严重降低。研究发现,通过过度表达MTH1[19]来降低糖的摄取活性能够消除这些不良结果。
最初关于低醇酵母菌的研究中,有研究试图删除PDC编码PDC基因,即在乙醇生物合成途径中完全删除Crabtree效应[20]。然而,删除了PDC的菌株在过量的葡萄糖中生长过程中,其生长速率很低。接着,DAI等[21]研究中通过敲除菌株PDC基因构建了一条PDH旁路途径,以提供胞质乙酰辅酶A的合成,并且进化后的菌株获得了较原菌株更快的生长速率。尽管合成的Crabtree阴性酵母可以在葡萄糖代谢过程中不产生乙醇,但其生长性能远低于野生型酵母。YU等[22]研究中将酵母的代谢过程的基因进行调控,从乙醇发酵到脂肪生成,这导致游离脂肪酸(free fatly acids,FFA)的产量升高,而没有积聚乙醇。同时,在OUD等[19]的研究中表明,缺乏PDC的酿酒酵母综合了稳健性和高糖酵解能力,并且没有酒精发酵。这使得这类酿酒酵母将葡萄糖有效转化为丙酮酸衍生产品,而不会形成乙醇。然而,该菌株不能在高葡萄糖浓度下正常生长,这也证明了消除或减少PDH和PDC活性会导致细胞在高渗透胁迫下无法生长或生产力严重降低。
1.1.1.2 甘油-3-磷酸脱氢酶(GPD1、GPD2、FPS1和ADH2基因)
在糖酵解途径中也会产生大量副产物,如甘油、有机酸等[23]。甘油是葡萄糖偏离乙醇形成的首选代谢物。甘油占酿酒酵母乙醇发酵中高达5%的碳通量,其产量仅次于乙醇和CO2,不仅对葡萄酒的感官特性具有积极作用,在酵母渗透调节和氧化还原平衡中也起着关键作用[24]。甘油在酒精发酵中能够作为乙醇的碳竞争者,因此,提高甘油产量有利于发酵饮料的乙醇含量的减少和风味改变[25]。
甘油-3-磷酸脱氢酶是甘油生产中的关键酶,由GPD1和GPD2编码。对于GPD1和GPD2敲除后的酿酒酵母突变体不能产生甘油,且细胞具有高度渗透敏感性,并且在缺氧条件下无法生长[25]。研究表明,增加甘油形成的最直接方法是过表达GPD1,其编码甘油-3-磷酸脱氢酶(GPDH)亚型Gpd1p。在甘油代谢过程中,甘油3-磷酸脱氢酶(GPD2)负责甘油合成。此外,GPD2在厌氧菌状态下的氧化还原平衡中也起着至关重要的作用[26]。酿酒酵母GPD2的过表达导致甘油合成效率提高[27]。因此,GPD2基因修饰是碳通量重定向合成甘油的有效途径。此外,甘油通道蛋白FPS1是通道蛋白主要内在蛋白家族的成员,是酿酒酵母细胞外变化响应甘油摄取和外排的促进剂,FPS1的过表达增强了甘油的产生[24]。乙醇通常通过醇脱氢酶(ADH2)催化生成乙醛。ADH2过表达可以降低酿酒酵母发酵过程中的乙醇浓度和产量,而将碳通量转向甘油。因此,通过GPD1、GPD2、FPS1和ADH2等与甘油平衡相关的关键基因的调控,对通过代谢途径的重定向来降低酿酒酵母的乙醇产量有重要意义。
1.1.1.3 高渗透压甘油途径(HOG)、甘油激酶、线粒体甘油-3-磷酸脱氢酶(HOG1基因、GUTI基因、GUT2基因)
由于甘油过多也会对葡萄酒的风味产生不良的影响,因此对于酿酒酵母菌产甘油过量的问题也应考虑在内,并进行适当的调控。REMIZE等[28]研究发现,葡萄酒发酵过程中HOG1基因的缺失导致生长速率略有下降,甘油产量下降20%,表明HOG途径在葡萄酒发酵条件下起作用,但所起到的作用有限。此外,研究发现[29]胞质甘油激酶(Gut1p)和线粒体甘油-3-磷酸脱氢酶(Gut2p)构成了酿酒酵母的甘油利用途径。因此,对于甘油产量过量的调控可以通过甘油利用的相关基因GUT1、GUT2及通路等进行调控。
1.1.2 三羧酸(TCA)循环和醇乙酰转移酶(AAT)途径对细胞乙醇产生的影响
乙醇代谢的转移途径除了增加甘油产量外,还可以将碳转移到有机酸的合成上,例如葡萄糖酸和参与TCA循环的有机酸[30]。参与TCA循环的几个基因的缺失和过表达已被证明,不仅影响有机酸的形成,也影响乙醇生产[31],但在酿酒环境中的真正影响尚未得到探索。减少生成乙醇的另一种方法是在发酵过程中将碳通量转移到酯类的合成上。发酵产生的典型果味主要归因于乙酸己酯、己酸乙酯(苹果味香气)、乙酸异戊酯(香蕉味香气)、辛酸乙酯(苹果味香气)和2-醋酸苯乙酯(果香、花香和蜂蜜味)[32]。近年来,构建高产酯的酿酒酵母菌株成为研究热点[33]。AAT途径是酵母中产生酯的代谢途径中最重要的途径,乙酰转移酶基因(如ATF1)的过表达可以显著影响缺乏香气的葡萄酒和馏出物的风味特征,在发酵结束装瓶后保持更长时间的果味特征[34]。同时,需要指出的是,ATT基因的表达水平对乙酸盐的合成有显著影响[35]。有研究合成了EAT1(一种新型AAT基因),作为酯合成途径的关键限速酶,催化乙酰CoA和乙醇在酵母中产生乙酸乙酯[36]。
1.2 乙醇的分解代谢及其调控基因
目前,在酿酒酵母细胞质中乙醇可以通过ADH2、ALD2、ALD3、ALD6和ACS1转化为乙酰辅酶A[37]。乙酰辅酶A是酿酒酵母柠檬酸合成的重要前体。已经采用了各种方法来加速乙醇-乙酰CoA途径的代谢通路,以加速乙醇的利用。研究发现[38]酿酒酵母ADH2和ALD6的过表达以及ACS1基因乙酰化结合位点的突变改善了乙酰辅酶A及其衍生物的产生。也有相关研究[39]通过过表达AAC2(AAC2是酿酒酵母线粒体内膜的主要ADP/ATP载体)来加速乙醇利用和乙酰辅酶A产生的速度。这都为酿酒酵母提供了一种将乙醇或乙酸生产乙酰辅酶A,用以参与其他风味物质(如上述提到的乙酸乙酯)的合成途径的研究思路。
2 低产乙醇酿酒酵母菌的选育方法
低产乙醇酿酒酵母选育方法包括适应性进化、诱变育种、基因工程、合成生物学,各研究方法中所使用的菌株及选育后的降醇效果如表1所示。
表1 低产乙醇酿酒酵母选育方法
Table 1 Selection and breeding of low-ethanol S. cerevisiae

菌株选育方法效果参考文献Lalvin EC1118适应性进化适应性进化200代之后,菌株具有比出发菌株更高的甘油和更低的乙醇产量(乙醇含量降低1.3%,甘油产量增加41%)[40]Ho C32-2-72诱变育种利用ARTP诱变育种技术,将低产乙醇且酿造特性优良的非酿酒酵母菌株进行诱变育种,得到了一株发酵速度快于出发菌株,乙醇产率降低10.26%[41]Y1-2基因工程通过构建得到了丙酮酸脱氢酶基因敲除重组酿酒酵母Y1-2,经过发酵获得酒度为3.7%(v/v)的低醇葡萄酒[42]YPH499合成生物学通过对酿酒酵母中参与糖酵解和异源d-LDH基因的12个基因表达的优化,得到稳定的高d-乳酸生产菌株,且具有高糖代谢和低乙醇产量的特点[43]
2.1 诱变育种
由于酿酒酵母代谢和调控网络的复杂性,很难通过合理的设计和基因工程策略获得稳定的表型[40]。在CHATSURACHAI等[44]研究中,鉴定分离得到的菌株(MP11、MP15)和商业酵母菌株参与乙醇生产的基因的DNA变异,并与NCBI数据库中检索到的其他菌株进行比较发现,分离菌株和商业菌株的底物结合位点ADH1发生变异,并得出DNA序列变异最大的是生产乙醇的初级酶(ADH1基因)。这表明酵母菌株之间的遗传变异对乙醇途径中的关键酶有影响。但是,由于遗传变异的周期长,不能准确地得到理想的变异菌株。因此,越来越多的研究趋向于通过诱变的方式,加快基因突变的速度。诱变育种方法包括:物理诱变、化学诱变、生物诱变以及复合诱变技术[45]。在近年的研究中,冯文倩[41]通过利用ARTP诱变育种技术,将低产乙醇非酿酒酵母菌株进行诱变育种,并得到了一株发酵速度快于出发菌株,乙醇产率降低10.26%的菌株Ho C32-2-72。
目前,诱变和遗传育种实践已被成功地应用于优良酿酒酵母的开发,以改善更适合某些酿酒实践和葡萄酒风格的特定性状(例如稳健性、发酵性能和感官属性)[46]。人工诱变育种技术为低醇葡萄酒生产提供了大量具有应用潜力的优质菌株,也为利用全基因组学、转录组学、蛋白质组学等组学技术研究酿酒酵母乙醇代谢调控网络提供了较为理想的实验菌株。但是,这些诱变方法在迭代突变过程中,一种诱变方法引发较单一的突变类型,复合诱变方法能引起的突变效率通常优于单一诱变方法[47]。因此,通常需要利用两种及两种以上的诱变手段来达到令人满意的突变效果。此外,大多数诱变因素,特别是化学诱变剂和辐射,都是强毒致癌物。因此,随着基因组工程和合成生物学的发展和广泛使用,这些内在缺陷导致物理或化学诱变方法的使用次数越来越少。
2.1 适应性进化
适应性实验室进化(adaptive laboratory evolution,ALE),也称为进化工程,利用生物体自身选定适应的环境条件以改善菌株性状以维持生命活力[47]。ALE通过提高育种中的突变率和筛选效率来模拟并加速自然进化[48]。通过筛选快速获得具有稳定性状的菌株,在构建稳定的微生物细胞工厂中发挥着重要作用[49]。近年来,ALE的快速发展加快了基因突变的效率,并已广泛应用于改善与生物技术应用相关的各种酵母性状[48],提高耐受性及氧化应激能力[50]或改善或扩大底物利用率的菌株。在TILLOY等[40]研究中,菌株进化200代之后,KCl的实验进化导致菌株具有比出发菌株更高的甘油和更低的乙醇产量(乙醇含量降低1.3%,甘油产量增加41%)。但是,ALE存在周期过长、效率低的缺点,这一过程中的关键步骤是提高遗传多样性和筛选效率。在适应性进化之前,可以采用诱变育种方式,加快菌株基因组的突变,再结合实验室进化进行低产乙醇酿酒酵母菌的选育,这将是必要且有效的选育方法。如,ZHAO等[23]通过将代谢工程与ALE相结合,降低了乙醇积累,提高了其他代谢产物,如高脂肪酸产量的合成酵母。研究发现,适应性进化实验最大的优势是通过作用于酵母的固有PYK1和PYK2基因以达到恢复氧化还原平衡的作用,产生所需的特性菌株[51]。随着全自动高通量微生物液滴培养仪(microbial microdroplet culture system, MMC)的开发,其在酵母菌适应性进化方面得到了广泛的应用(如图3)。MMC平台能够通过添加化学因子进行适应性进化实现自动化传代培养,更高效、经济的培育出理想菌株[52]。有研究采用MMC系统对酿酒酵母YN81进行ALE,以提高其对渗透压和乙醇胁迫的耐受性。同时得到了用于酿造烈性啤酒中进化的突变体[53]。但目前在低产乙醇酿酒酵母的适应性进化方面还未采用MMC平台进行研究,研究者有望在此方向进行探索。这些重大进展表明,实验室进化是完全重塑生物体遗传和代谢表型的有力手段,为探索生物体的代谢潜力、了解其复杂的代谢途径以及开发用于工业应用的稳健突变体提供机会。
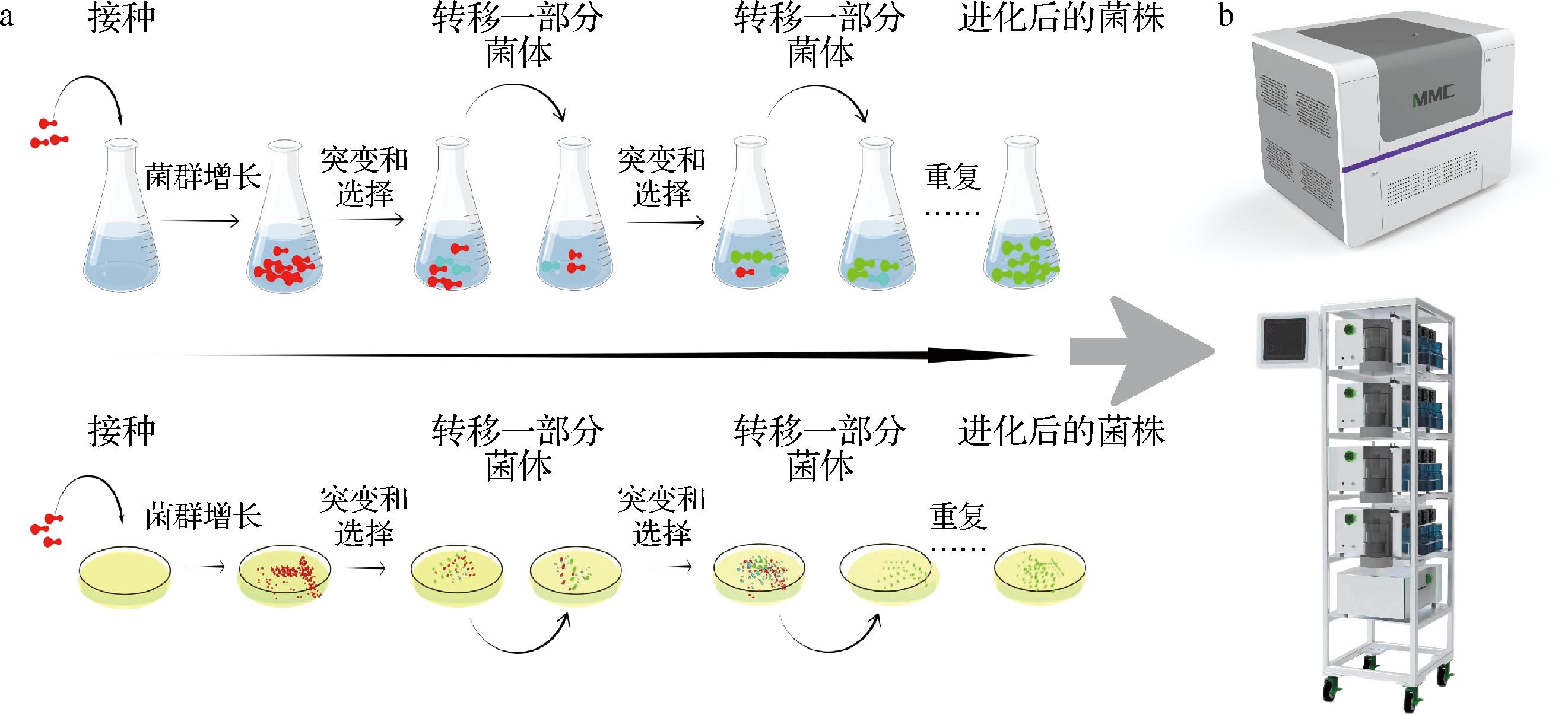
图3 适应性实验室进化方法的改进
Fig.3 Improvements in adaptive laboratory evolutionary methods
2.3 基因工程改造
随着酿酒酵母全基因组序列的公开以及乙醇代谢途径研究的不断深入,运用基因工程技术定向改造乙醇代谢途径成为研究的热点[54]。基因工程改造在酿酒酵母中的应用中较为新颖,为增强或改变葡萄酒品质的生产工艺开辟了新的可能性[55],复杂的基因工程技术在近年来受到广泛的关注。
2.3.1 基因敲除
基因敲除是基因工程中的一种策略,相应基因的敲除可以适当改变乙醇发酵性能[35]。目前,常用的是CRISPR-Cas9技术,其利用菌株天然存在的“适应性免疫”机制来构建精确的基因组编辑工具[56]。近年来,有研究利用CRISPR-Cas9方法构建酿酒酵母工程菌株,通过敲除GPD2、FPS1和ADH2来提高乙醇产量[57]。结果表明,通过同时敲除GPD2、FPS1和ADH2改造酿酒酵母菌SCGFA产生23.1 g/L的乙醇,与以50 g/L葡萄糖为底物的野生型菌株相比增加了0.18%[58]。而通过敲除编码乙醇脱氢酶基因(ADH1、ADH2、ADH3)[43]以及丙酮酸脱羧酶基因(PDC1、PDC5)[59],达到了减少乙醇生成量的目的,且甘油转化率增加,其他代谢产物浓度均有明显变化。但研究发现PDC酶的敲除会形成酿酒酵母菌的Crabtree效应,产生诸多不良的后果,例如C2营养不良,需要补充乙酸盐或乙醇,并降低生长速度和发酵速度[60]。因此,期望开发一种能够不用敲除PDC,就将丙酮酸有效代谢为非乙醇化合物的酵母菌株很有必要。
2.3.2 同源重组
基于基因组测序数据,对不同发酵特性的酿酒酵母相关调控基因差异进行分析。待改造菌株的调控基因利用同源重组将优良的发酵性状与待改造菌株整合[61]。通过构建表达载体,再将携带特定基因的表达载体转入酵母细胞内进行表达即可以获得所需的性状[6]。WANG等[34]研究通过采用同源重组的原理构建了过表达AAT基因EAT的重组酿酒酵母菌株W303-EAT。重组菌株W303-EAT与其亲本W303-1A相比,生长性能良好且减少了酒精生成。同时,CUELLO等[62]通过对PDC2基因编码的酿酒酵母Pdc2p转录因子的遗传修饰开发了一种低产乙醇菌株。该转录因子参与调节PDC同工酶,并在乙醇生物合成途径中催化丙酮酸与乙醛的反应,通过同源重组,该菌株可将乙醇浓度降低至1.89%(体积分数),而不影响整体发酵动力学。但这种方法主要依靠独立于核基因组外的表达,稳定性较差。
2.3.3 基因修饰
目前,已有相关基因工程技术研究实现了将酵母的乙醇代谢转移到增加其他代谢产物,从而减少乙醇形成。例如,YAMADA等[63]优化了酿酒酵母中参与糖酵解和异源d-LDH基因的12个基因的表达,以产生稳定的乳酸生产菌株,具有高糖代谢和低乙醇产量。另一种导致乙醇产量降低的转基因修饰是降低PDC水平或损害ADH表达活性[64]。研究发现[65],将编码粟酒裂殖酵母苹果酸转运蛋白的MAEL基因引入酿酒酵母中,能够降低乙醇含量,同时也有助于增加琥珀酸的产量。
虽然使用基因工程技术将酵母代谢重定向到增加甘油产量相对容易实现,但世界许多地方不允许使用转基因菌株进行酿酒,消费者对转基因生物仍具争议。因此,必须依靠非转基因方法实现低产乙醇菌株的选育。但是,仅仅通过合理的工程设计很难将乙醇积聚的通量完全重定向到目标产物,需要联系其他代谢途径进行综合研究。
2.4 合成生物学
酿酒酵母的合成生物学方法可用于开发基因组规模代谢模型和分子工具,例如Cas9、Cas12a、Cpf1和Csy4基因组编辑工具[66]。微生物细胞工厂通常是通过建立新的生物合成途径而不破坏天然代谢特征来构建的,这可以防止通量重定向到所需产物[67]。目前,在开发微生物细胞工厂中,基因组整合由于长期稳定性、同质性和拷贝数控制等优点,对生物合成途径的表达具有重要意义[66]。例如,对于低产乙醇酿酒酵母菌细胞的构建,由于葡萄糖转化为其他化学物质的过量生产通常受到其自身发酵代谢的阻碍,其中大多数葡萄糖分解代谢为乙醇,合成生物学的最新进展集中在重新设计ADH基因以提高底物特异性和改善催化活性,以及用蛋白质编码基因改造酵母基因组[68],从而降低乙醇产量生成甘油、有机酸、酯等其他副产物。DELLOMONACO等[43]研究发现,通过优化酿酒酵母中参与糖酵解和异源d-LDH基因的12个基因的表达,可以产生稳定的高D-乳酸生产菌株,具有高糖代谢和低乙醇产量。生物合成基因在复杂途径中的多重整合可以加速微生物细胞工厂的工程设计[63]。与此同时,合成生物学的快速发展将为我们如何构建和设计用于新生物系统(例如降低酿酒酵母低产乙醇)提供新的视角和革命。
3 结论与展望
目前,酿酒酵母的乙醇代谢途径中的酶系及其编码基因已基本明确,但存在调控效果不理想,利用人工智能等计算机辅助手段来提升改造的可靠性和效率不高等诸多问题。葡萄酒中乙醇含量的调控,更要兼顾葡萄酒的风味特征,不应单纯降低乙醇的含量。因此,在之后的研究中,酿酒酵母乙醇代谢的精细化调控需要关注酵母菌代谢活动的整体性,尤其是对酒精含量降低后,所释放的香气和风味化合物含量的监测。此外,在研究中还需注意对单个基因调控机制的研究,应同时构建基因敲除和过量表达重组菌株并结合重组菌株在不同营养条件下代谢流的变化,研究该基因在乙醇代谢途径中的功能。采用组学技术对低产乙醇的关键基因进行深度研究,与人工智能学科交叉融合,实现低产乙醇代谢的高效调控。综上所述,实现酿酒酵母低产乙醇代谢的精细化调控是一项艰难、复杂的系统工程,单对乙醇的代谢途径进行研究很难完成。因此,需研究酿酒酵母的发酵过程和其内部生理生化特征,并且,在此基础上建立低产乙醇代谢调控系统和定向育种自动化技术体系,最终实现酿酒酵母风味物质比例协调的目标。
[1] GUINDAL A M, MORALES P, TRONCHONI J, et al.Reduction of ethanol content in wine with an improved combination of yeast strains and process conditions[J].Food Microbiology, 2023, 115:104344.
[2] ABREU T, PERESTRELO R, BORDIGA M, et al.The flavor chemistry of fortified wines-a comprehensive approach[J].Foods, 2021, 10(6):1239.
[3] JONES-MOORE H R, JELLEY R, MARANGON M, et al.The polysaccharides of winemaking:From grape to wine[J].Trends in Food Science and Technology, 2021, 111:731-740.
[4] ENGLEZOS V, CRAVERO F, TORCHIO F, et al.Oxygen availability and strain combination modulate yeast growth dynamics in mixed culture fermentations of grape must with Starmerella bacillaris and Saccharomyces cerevisiae[J].Food Microbiology, 2018, 69:179-188.
[5] LAHTINEN H, MOUSTGAARD H, RIPATTI S, et al.Association between genetic risk of alcohol consumption and alcohol-related morbidity and mortality under different alcohol policy conditions:Evidence from the Finnish alcohol price reduction of 2004[J].Addiction, 2023, 118(4):678-685.
[6] CANONICO L, COMITINI F, CIANI M.Metschnikowia pulcherrima selected strain for ethanol reduction in wine:Influence of cell immobilization and aeration condition[J].Foods, 2019, 8(9):378.
[7] MULERO-CEREZO J, TU
 N-MOLINA A, CANO-VICENT A, et al.Alcoholic and non-alcoholic rosé wines made with Saccharomyces cerevisiae var.boulardii probiotic yeast[J].Archives of Microbiology, 2023, 205(5):201.
N-MOLINA A, CANO-VICENT A, et al.Alcoholic and non-alcoholic rosé wines made with Saccharomyces cerevisiae var.boulardii probiotic yeast[J].Archives of Microbiology, 2023, 205(5):201.
[8] HENDERSON C M, BLOCK D E.Examining the role of membrane lipid composition in determining the ethanol tolerance of Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 2014, 80(10):2966-2972.
[9] ROLLE L, ENGLEZOS V, TORCHIO F, et al.Alcohol reduction in red wines by technological and microbiological approaches:A comparative study[J].Australian Journal of Grape and Wine Research, 2018, 24(1):62-74.[10] TILLOY V, CADI RE A, EHSANI M, et al.Reducing alcohol levels in wines through rational and evolutionary engineering of Saccharomyces cerevisiae[J].International Journal of Food Microbiology, 2015, 213:49-58.
RE A, EHSANI M, et al.Reducing alcohol levels in wines through rational and evolutionary engineering of Saccharomyces cerevisiae[J].International Journal of Food Microbiology, 2015, 213:49-58.
[11] DUNCAN J D, SETATI M E, DIVOL B.Redox cofactor metabolism in Saccharomyces cerevisiae and its impact on the production of alcoholic fermentation end-products[J].Food Research International, 2023, 163:112276.
[12] SABATER-MU OZ B, MATTENBERGER F, FARES M A, et al.Transcriptional rewiring, adaptation, and the role of gene duplication in the metabolism of ethanol of Saccharomyces cerevisiae[J].mSystems, 2020, 5(4):e00416-e00420.
OZ B, MATTENBERGER F, FARES M A, et al.Transcriptional rewiring, adaptation, and the role of gene duplication in the metabolism of ethanol of Saccharomyces cerevisiae[J].mSystems, 2020, 5(4):e00416-e00420.
[13] HA S J, GALAZKA J M, KIM S R, et al.Engineered Saccharomyces cerevisiae capable of simultaneous cellobiose and xylose fermentation[J].Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(2):504-509.
[14] SHARMA S, TAURO P.Control of ethanol production by yeast:Role of pyruvate decarboxylase and alcohol dehydrogenase[J].Biotechnology Letters, 1986, 8(10):735-738.
[15] PRONK J T, YDE STEENSMA H, VAN DIJKEN J P.Pyruvate metabolism in Saccharomyces cerevisiae[J].Yeast, 1996, 12(16):1607-1633.
[16] TRONCHONI J, GONZALEZ R, GUINDAL A M, et al.Exploring the suitability of Saccharomyces cerevisiae strains for winemaking under aerobic conditions[J].Food Microbiology, 2022, 101:103893.
[17] 郭伟. 代谢工程改造酿酒酵母生产酪醇及红景天苷的研究[D].济南:山东大学, 2020.GUO W.Studies on metabolic engineering of Saccharomyces cerevisiae for the production of tyrosol and salidroside[D].Jinan:Shandong University, 2020.
[18] BROCHADO A R, MATOS C, MØLLER B L, et al.Improved vanillin production in baker’s yeast through in silico design[J].Microbial Cell Factories, 2010, 9:84.
[19] OUD B, FLORES C L, GANCEDO C, et al.An internal deletion in MTH1 enables growth on glucose of pyruvate-decarboxylase negative, non-fermentative Saccharomyces cerevisiae[J].Microbial Cell Factories, 2012, 11:131.
[20] ZHANG Y M, LIU G D, ENGQVIST M K M, et al.Adaptive mutations in sugar metabolism restore growth on glucose in a pyruvate decarboxylase negative yeast strain[J].Microbial Cell Factories, 2015, 14:116.
[21] DAI Z J, HUANG M T, CHEN Y, et al.Global rewiring of cellular metabolism renders Saccharomyces cerevisiae Crabtree negative[J].Nature Communications, 2018, 9(1):3059.
[22] YU T, ZHOU Y J, HUANG M T, et al.Reprogramming yeast metabolism from alcoholic fermentation to lipogenesis[J].Cell, 2018, 174(6):1549-1558.
[23] ZHAO X D, PROCOPIO S, BECKER T.Flavor impacts of glycerol in the processing of yeast fermented beverages:A review[J].Journal of Food Science and Technology, 2015, 52(12):7588-7598.
[24] LUYTEN K, ALBERTYN J, SKIBBE W F, et al.Fps1, a yeast member of the MIP family of channel proteins, is a facilitator for glycerol uptake and efflux and is inactive under osmotic stress[J].The EMBO Journal, 1995, 14(7):1360-1371.
[25] ANSELL R, GRANATH K, HOHMANN S, et al.The two isoenzymes for yeast NAD+-dependent glycerol 3-phosphate dehydrogenase encoded by GPD1 and GPD2 have distinct roles in osmoadaptation and redox regulation[J].The EMBO Journal, 1997, 16(9):2179-2187.
[26] HUBMANN G, GUILLOUET S, NEVOIGT E.Gpd1 and Gpd2 fine-tuning for sustainable reduction of glycerol formation in Saccharomyces cerevisiae[J].Applied and Environmental Microbiology, 2011, 77(17):5857-5867.
[27] REMIZE F, BARNAVON L, DEQUIN S.Glycerol export and glycerol-3-phosphate dehydrogenase, but not glycerol phosphatase, are rate limiting for glycerol production in Saccharomyces cerevisiae[J].Metabolic Engineering, 2001, 3(4):301-312.
[28] REMIZE F, CAMBON B, BARNAVON L, et al.Glycerol formation during wine fermentation is mainly linked to Gpd1p and is only partially controlled by the HOG pathway[J].Yeast, 2003, 20(15):1243-1253.
[29] GRAUSLUND M, RØNNOW B.Carbon source-dependent transcriptional regulation of the mitochondrial glycerol-3-phosphate dehydrogenase gene, GUT2, from Saccharomyces cerevisiae[J].Canadian Journal of Microbiology, 2000, 46(12):1096-1100.
[30] VARELA C, KUTYNA D R, SOLOMON M R, et al.Evaluation of gene modification strategies for the development of low-alcohol-wine yeasts[J].Applied and Environmental Microbiology, 2012, 78(17):6068-6077.
[31] SELECK R,
R,  MOGROVI
MOGROVI OV
OV D, SULO P.Beer with reduced ethanol content produced using Saccharomyces cerevisiae yeasts deficient in various tricarboxylic acid cycle enzymes[J].Journal of the Institute of Brewing, 2008, 114(2):97-101.
D, SULO P.Beer with reduced ethanol content produced using Saccharomyces cerevisiae yeasts deficient in various tricarboxylic acid cycle enzymes[J].Journal of the Institute of Brewing, 2008, 114(2):97-101.
[32] LILLY M, LAMBRECHTS M G, PRETORIUS I S.Effect of increased yeast alcohol acetyltransferase activity on flavor profiles of wine and distillates[J].Applied and Environmental Microbiology, 2000, 66(2):744-753.
[33] KRUIS A J, GALLONE B, JONKER T, et al.Contribution of Eat1 and other alcohol acyltransferases to ester production in Saccharomyces cerevisiae[J].Frontiers in Microbiology, 2018, 9:3202.
[34] WANG X D, SHEN K, TANG S P, et al.Study on the construction and aroma-producing characteristics of the recombinant Saccharomyces cerevisiae strain W303-EAT[J].European Food Research and Technology, 2022, 248(2):447-456.
[35] PARK Y C, SHAFFER C E H, BENNETT G N.Microbial formation of esters[J].Applied Microbiology and Biotechnology, 2009, 85(1):13-25.
[36] KRUIS A J, LEVISSON M, MARS A E, et al.Ethyl acetate production by the elusive alcohol acetyltransferase from yeast[J].Metabolic Engineering, 2017, 41:92-101.
[37] LI T, LIU G S, ZHOU W, et al.Metabolic engineering of Saccharomyces cerevisiae to overproduce squalene[J].Journal of Agricultural and Food Chemistry, 2020, 68(7):2132-2138.
[38] LIU Y Q, BAI C X, LIU Q, et al.Engineered ethanol-driven biosynthetic system for improving production of acetyl-CoA derived drugs in Crabtree-negative yeast[J].Metabolic Engineering, 2019, 54:275-284.
[39] XU Y Y, LI Z M.Utilization of ethanol for itaconic acid biosynthesis by engineered Saccharomyces cerevisiae[J].FEMS Yeast Research, 2021, 21(6):foab043.
[40] TILLOY V, ORTIZ-JULIEN A, DEQUIN S.Reduction of ethanol yield and improvement of glycerol formation by adaptive evolution of the wine yeast Saccharomyces cerevisiae under hyperosmotic conditions[J].Applied and Environmental Microbiology, 2014, 80(8):2623-2632.
[41] 冯文倩. 低产乙醇本土非酿酒酵母的选育[D].杨凌:西北农林科技大学, 2021.FENG W Q.Breeding of native Non-Saccharomyces cerevisiae with low ethanol yield[D].Yangling:Northwest A &F University, 2021.
[42] 于淼, 李洁, 李凯, 等.丙酮酸脱氢酶基因敲除酿酒酵母发酵低醇葡萄酒工艺条件的研究[J].井冈山大学学报(自然科学版), 2016, 37(6):36-41;47.YU M, LI J, LI K, et al.Study of fermentation process conditions of low-alcohol wine by the pyruvate dehydrogenase gene knock-out mutant Saccharomyces cerevisiae[J].Journal of Jinggangshan University (Natural Science), 2016, 37(6):36-41;47.
[43] DELLOMONACO C, FAVA F, GONZALEZ R.The path to next generation biofuels:Successes and challenges in the era of synthetic biology[J].Microbial Cell Factories, 2010, 9:3.
[44] CHATSURACHAI S, WATANAROJANAPORN N, PHAENGTHAI S, et al.Genetic variation in genes involved in ethanol production among Saccharomyces cerevisiae strains[J].Sugar Tech, 2020, 22(2):250-258.
[45] NAGHSHBANDI M P, TABATABAEI M, AGHBASHLO M, et al.Progress toward improving ethanol production through decreased glycerol generation in Saccharomyces cerevisiae by metabolic and genetic engineering approaches[J].Renewable and Sustainable Energy Reviews, 2019, 115:109353.
[46] XIA X K, ZHANG Y E, LEI S J, et al.Identification and iterative combinatorial mutagenesis of a new naringinase-producing strain, Aspergillus tubingensis MN589840[J].Letters in Applied Microbiology, 2021, 72(2):141-148.
[47] SUN X M, REN L J, BI Z Q, et al.Adaptive evolution of microalgae Schizochytrium sp.under high salinity stress to alleviate oxidative damage and improve lipid biosynthesis[J].Bioresource Technology, 2018, 267:438-444.
[48] WAGNER J M, LIU L Q, YUAN S F, et al.A comparative analysis of single cell and droplet-based FACS for improving production phenotypes:Riboflavin overproduction in Yarrowia lipolytica[J].Metabolic Engineering, 2018, 47:346-356.
[49] JIA Y L, LI J, NONG F T, et al.Application of adaptive laboratory evolution in lipid and terpenoid production in yeast and microalgae[J].ACS Synthetic Biology, 2023, 12(5):1396-1407.
[50] PATIL K R, RALSER M.Freeing yeast from alcohol addiction (just) to make (it) fat instead[J].Cell, 2018, 174(6):1342-1344.
[51] CAKAR Z P, TURANLI-YILDIZ B, ALKIM C, et al.Evolutionary engineering of Saccharomyces cerevisiae for improved industrially important properties[J].FEMS Yeast Research, 2012, 12(2):171-182.
[52] WANG Q, JIN W B, HAN W, et al.Enhancement of DHA production from Aurantiochytrium sp.by atmospheric and room temperature plasma mutagenesis aided with microbial microdroplet culture screening[J].Biomass Conversion and Biorefinery, 2023, 13(18):16807-16818.
[53] YANG T Y, ZHANG S S, PAN Y R, et al.Breeding of high-tolerance yeast by adaptive evolution and high-gravity brewing of mutant[J].Journal of the Science of Food and Agriculture, 2024, 104(2):686-697.
[54] 陈碧燕, 文李.酿酒酵母全基因组学及其应用研究进展[J].食品与机械, 2020, 36(11):223-227;232.CHEN B Y, WEN L.Progress in Saccharomyces cerevisiae genome research and relative application[J].Food &Machinery, 2020, 36(11):223-227;232.
[55] MOLINA-ESPEJA P.Next generation winemakers:Genetic engineering in Saccharomyces cerevisiae for trendy challenges[J].Bioengineering, 2020, 7(4):128.
[56] JESSOP-FABRE M M, JAKO I
I NAS T, STOVICEK V, et al.EasyClone-MarkerFree:A vector toolkit for marker-less integration of genes into Saccharomyces cerevisiae via CRISPR-Cas9[J].Biotechnology Journal, 2016, 11(8):1110-1117.
NAS T, STOVICEK V, et al.EasyClone-MarkerFree:A vector toolkit for marker-less integration of genes into Saccharomyces cerevisiae via CRISPR-Cas9[J].Biotechnology Journal, 2016, 11(8):1110-1117.
[57] 高莹莹. 低醇葡萄酒酵母菌株的选育[D].天津:天津科技大学, 2020.GAO Y Y.Breeding of low-ethanol-wine yeasts[D].Tianjin:Tianjin University of Science &Technology, 2020.
[58] YANG P Z, JIANG S Y, JIANG S W, et al.CRISPR-Cas9 approach constructed engineered Saccharomyces cerevisiae with the deletion of GPD2, FPS1, and ADH2 to enhance the production of ethanol[J].Journal of Fungi, 2022, 8(7):703.
[59] ZHANG W, KANG J, WANG C L, et al.Effects of pyruvate decarboxylase (pdc 1, pdc 5) gene knockout on the production of metabolites in two haploid Saccharomyces cerevisiae strains[J].Preparative Biochemistry &Biotechnology, 2022, 52(1):62-69.
[60] LANE S, TURNER T L, JIN Y S.Glucose assimilation rate determines the partition of flux at pyruvate between lactic acid and ethanol in Saccharomyces cerevisiae[J].Biotechnology Journal, 2023, 18(4):e2200535.
[61] ROUSHAN M R, SHAO S, POLEDRI I, et al.Increased Agrobacterium-mediated transformation of Saccharomyces cerevisiae after deletion of the yeast ADA2 gene[J].Letters in Applied Microbiology, 2022, 74(2):228-237.
[62] CUELLO R A, FLORES MONTERO K J, MERCADO L A, et al.Construction of low-ethanol-wine yeasts through partial deletion of the Saccharomyces cerevisiae PDC2 gene[J].AMB Express, 2017, 7(1):67.
[63] YAMADA R, WAKITA K, MITSUI R, et al.Enhanced d-lactic acid production by recombinant Saccharomyces cerevisiae following optimization of the global metabolic pathway[J].Biotechnology and Bioengineering, 2017, 114(9):2075-2084.
[64] DREWKE C, THIELEN J, CIRIACY M.Ethanol formation in adh0 mutants reveals the existence of a novel acetaldehyde-reducing activity in Saccharomyces cerevisiae[J].Journal of Bacteriology, 1990, 172(7):3909-3917.
[65] ITO Y, HIRASAWA T, SHIMIZU H.Metabolic engineering of Saccharomyces cerevisiae to improve succinic acid production based on metabolic profiling[J].Bioscience, Biotechnology, and Biochemistry, 2014, 78(1):151-159.
[66] TING T Y, LI Y D, BUNAWAN H, et al.Current advancements in systems and synthetic biology studies of Saccharomyces cerevisiae[J].Journal of Bioscience and Bioengineering, 2023, 135(4):259-265.
[67] MENG J, QIU Y, ZHANG Y P, et al.CMI:CRISPR/Cas9 based efficient multiplexed integration in Saccharomyces cerevisiae[J].ACS Synthetic Biology, 2023, 12(5):1408-1414.
[68] DARVISHI F, RAFATIYAN S, ABBASPOUR MOTLAGH MOGHADDAM M H, et al.Applications of synthetic yeast consortia for the production of native and non-native chemicals[J].Critical Reviews in Biotechnology, 2024, 44(1):15-30.