食源性致病菌是引起食源性疾病的主要原因之一,其中沙门氏菌、副溶血性弧菌、志贺氏菌、单核细胞增生李斯特氏菌(单增李斯特菌)、金黄色葡萄球菌等较为常见。食源性致病菌的感染对象涉及不同年龄段人群。大多数食源性致病菌感染人体后主要表现为肠胃症状,一般可自行恢复,但针对特殊人群可能会导致败血症、神经系统疾病以及全身感染等严重疾病,甚至死亡[1]。
体外细胞系和体内动物模型是研究食源性致病菌感染能力的主要研究工具。食源性致病菌通过被污染的食物进入人体后首先需要突破肠道屏障,基于体内外肠道模型,可以研究食源性致病菌的毒性,包括评估可感染细胞的最低菌数,分析菌株的感染周期和感染进程,分析不同菌株毒性的强弱以及潜在的感染能力,并深入解析致病菌毒力因子的作用机制,以此预测食源性致病菌感染人体的潜力。动物模型作为一种传统的实验模型,主要用于研究食源性致病菌感染后各组织器官细菌的负荷、宿主免疫情况以及在动物体内的感染进程等。然而由于动物模型所产生的结果不完全适用于人体,在病原体与宿主相互作用的研究上存在局限性。近年来,应用体外人源细胞或者人体组织构建的肠道模型,在研究食源性致病菌方面取得了较多进展。除了体外细胞模型,肠道类器官的发展为研究食源性致病菌的毒力因子、感染机制、抗菌药物开发以及疫苗测试提供了新的模型系统。虽然这些类器官模型仍处于初级阶段,但在研究食源性致病菌方面潜力巨大。目前国内外已有研究者基于体内外模型研究食源性致病菌的感染机制并进行综述,但其对各种模型的优劣并未进行比较,也缺少对体内外肠道模型研究进展的系统总结。
本文对近年来研究食源性致病菌的体内外肠道模型进行了系统阐述。以期为研究食源性致病菌的感染机制及最优模型的选择提供理论支撑,从而为进一步预防和控制食源性致病菌提供科学参考。
1 食源性致病菌感染的体内模型
体内模型一般是指动物模型,是利用致病因子作用于小鼠、豚鼠以及兔子等实验动物,构建具有模拟人类疾病表现的动物实验对象。人体肠道内部结构复杂,肠道内微生物种类繁多且数量巨大,因此,动物模型一直以来都是研究食源性致病菌的重要工具,可用于探究食源性致病菌的致病机制、探索控制食源性致病菌的防治方法,以及评估治疗方案的有效性。啮齿类动物、非人类灵长类动物、兔、猪等动物(表1)由于与人有相似性,因此常被作为致病菌感染人类的实验模型。
表1 研究食源性致病菌感染的不同模型
Table 1 Different models of studying foodborne pathogen infection

模型特点优点缺点参考文献啮齿类动物成本低、繁殖快可基因编辑、相关研究多、遗传背景明确静脉注射感染、不是天然宿主、临床表现不符[7]非人类灵长类动物肠道与人类具有高度相似性在进化树上与人类接近、病理及免疫反应与人类相似不经济、伦理问题、相关研究少,饲养技术和环境要求高[13]兔体型中等、性格温顺易于操作、遗传背景明确、解剖结构与人类相似胆小、易产生应激反应[14]猪肠道器官重量及功能特征与人类相似适合研究人畜共患病、免疫反应与人类相似价格较高、体型大,饲养技术和环境要求高[17-18]2D细胞模型简单、可重复性强操作简单、技术成熟、简化生物系统、可大规模培养缺乏3D结构、细胞类型单一[23]肠类器官具有隐窝-绒毛结构增殖分化、更好模拟人体结构、可实时观察缺乏神经支配、静态、无微生物组成、封闭系统不易感染[24]肠道芯片动态模拟人体肠道内液体流动及肠蠕动、便携处于起步阶段需要更多研究[25]
1.1 啮齿类动物
啮齿类动物由于易饲养、成本低等特点被广为应用于实验研究。近年来食源性致病菌的抗生素替代疗法是一个研究热点,一些基于啮齿类动物肠道模型的研究为深入了解生物活性物质治疗食源性疾病提供了初步的结论。覃兰迁等[2]利用沙门氏菌诱导肠道损伤的BALB/c小鼠模型发现,迷迭香提取物可能通过抑制炎症反应,有效改善沙门氏菌导致的小鼠十二指肠上皮细胞脱落,使肠绒毛结构更完整,减少小鼠肠道损伤。DONG等[3]使用鼠伤寒沙门氏菌感染BALB/c小鼠建立腹泻模型,表明黄芪多糖的治疗增加了空肠绒毛高度和隐窝深度,并减少炎症细胞浸润,诱导免疫相关因子的表达,抑制核因子κB(nuclear factor kappa-B,NF-κB)的活化。王兆雨等[4]利用C57BL/6小鼠建立产肠毒素大肠杆菌诱导的肠道感染模型,发现了生物活性肽可以通过上调转录因子核受体77的表达,抑制小肠上皮细胞焦亡,降低炎症细胞因子释放,缓解肠道炎症。以上研究显示,小鼠模型可用于评估化合物是否具有预防和减缓细菌感染肠道的作用,为继续开展临床试验提供重要的数据支持。
普通的啮齿类动物与人体存在一定差距,因此,需对实验动物进行人源化改造。人与鼠类的肠道中仅有4%肠道微生物基因具有较大的同一性[5]。为了研究食源性致病菌与人体肠道菌群的相互作用,可以建立人源化菌群动物模型。FAURE等[6]使用接种了人粪混悬液的人源化菌群C3H大鼠模型,发现CXT-M-9耐药基因能从动物来源的沙门氏菌转移到人的肠道菌群中。由于啮齿类动物与人类之间的基因差异,有时会导致无法建立合适的动物感染模型。例如单增李斯特菌的内化素蛋白A(internalinA, InlA)只能识别人的肠道上皮细胞钙黏蛋白(E-cadherin),但对于大鼠或小鼠存在识别问题[7],因此大鼠或小鼠仅能通过静脉感染建立单增李斯特菌感染模型,而不能通过口-肠道感染建立模型。为了解决这一问题,LECUIT等[8]建立了一种在小鼠肠细胞中表达人E-cadherin的转基因小鼠模型,发现细菌经口感染转基因小鼠会导致85%的死亡,且单增李斯特菌毒力因子内化蛋白InlA缺失菌株经口感染转基因小鼠死亡率为0,证明了InlA与肠上皮细胞E-cadherin的相互作用在单增李斯特菌穿过肠道屏障中的关键作用。这些研究显示,相比野生型小鼠,人源化菌群或转基因小鼠模型可以更好地模拟食源性致病菌感染人体的情况,然而由于食源性致病菌种类较多,不能针对每一种致病菌开发相匹配的人源化小鼠,在使用上仍然受到限制。
啮齿类动物模型存在一些局限性。如传统饲养的啮齿类动物胃肠道原有菌群对胃肠道感染具有抵抗力,可能出现无法感染的情况。此外虽然超过95%的基因在小鼠和人类之间是同源的,但在基因家族成员、基因冗余、基因表达水平的精细调控等方面也存在差异[9],基因的差异会导致生理的差异,一些在啮齿类动物身上获得的结果不能在人体研究中得到证实[10]。为了提高实验结果的可重复性和实验的统计能力,实验室小鼠已被培育成具有高度同质遗传构成的近亲系。这导致在一个品系实验中得到的结果,用另一个品系实验可能会得到不一样的临床表现[11]。
1.2 非人类灵长类动物
啮齿类动物虽可作为研究食源性致病菌的肠道模型,但大多数模型在生理解剖结构、肠道菌群和基因等方面与人类有较大差异。非人灵长类动物与人类在进化上相近,肠道特征具有高度的相似性,作为研究人类食源性疾病具有独特的优势。
灵长类动物被食源性致病菌感染后表现出人类的许多临床症状,因此非人类灵长类模型被认为是多种食源性致病菌感染的金标准动物模型,可用于在人体临床试验前期对食源性致病菌疫苗安全性、免疫原性以及有效性的评估。HIGGINSON等[12]利用2~3岁雌性恒河猴(Macaca mulatt)模型来检验优化的鼠伤寒沙门氏菌减毒活疫苗CVD1926的反应原性和免疫原性,以及疫苗对沙门氏菌肠胃炎的功效。ROLLENHAGEN等[13]使用秘鲁夜猴(Aotus nancymaae)建立了一种有效、可重复的产肠毒素大肠埃希氏菌(enterotoxigenic Escherichia coli,ETEC)疾病模型,显示秘鲁夜猴在经口感染ETEC后出现与人类相似的腹泻症状和发病概率。
此外,非人类灵长类动物也可用来研究食源性致病菌的感染机制。TAYLOR等[14]将狒狒作为一种研究志贺毒素1(Shiga toxin-1,Stx-1)的动物模型,接受低剂量Stx-1的狒狒出现的病变与人类相似;注射高剂量Stx-1的狒狒大肠和小肠绒毛尖端上皮细胞丢失。SANTOS等[15]使用2~4岁雄性恒河猴建立回肠环模型对沙门氏菌感染肠道进行形态学表征,感染野生型沙门氏菌2 h能在肠道绒毛尖端的上皮及固有层检测到致病菌,感染5~8 h后能检测到致病菌向固有层更深处侵袭;此外,实验中发现沙门氏菌能诱导巨噬细胞焦亡。
灵长类动物与其他动物模型相比,在进化树上与人类更加接近,是目前最理想的研究食源性致病菌感染的肠道模型,但减毒活疫苗对宿主肠道微生物的影响以及通过灵长类动物实验得到的数据是否能直接应用于临床等问题仍需进一步探索。尤其是,由于灵长类动物实验成本高、审查条件严格、自然感染率高、不可控因素多等缺点一定程度上限制了灵长类动物模型的使用。
1.3 其他动物
除啮齿类动物和非人类灵长类动物,兔也是研究中常用到的动物之一。实验兔具有体型中等、性格温顺、繁殖率高的特征,易于在实验室中操作和繁殖,且实验室培育的兔有明确的遗传背景[16]。此外实验兔解剖结构和病理反应与人类相似[17],在对食源性致病菌的研究中,可以建立兔的肠道感染模型来研究食源性致病菌的发病机制及防治方法。HECZKO等[18]使用30~55日龄新西兰白兔建立肠道模型,发现分节丝状菌可能通过竞争的方式预防致病性大肠埃希氏菌(enteropathogenic Escherichia coli,EPEC)导致的肠道疾病。RITCHIE等[19]使用灌胃感染的幼兔(2日龄新西兰白兔)模型发现,副溶血性弧菌黏附并定植在幼兔的远端小肠上,这里也是副溶血性弧菌引起组织损伤、上皮屏障功能受损和形成炎症的主要部位,同时,副溶血性弧菌也能诱导小肠绒毛显著增长并在上皮表面形成内部充满细菌的空腔,并最终导致绒毛破裂。RUI等[20]使用3日龄家兔模型发现反应性腹泻可能是宿主对霍乱弧菌毒力因子鞭毛蛋白炎症反应的结果。然而,由于实验兔易产生应激反应,导致实验结果易产生误差,因此在使用上也存在一定的局限性。
除以上动物外,猪的基因组大小和组成与人类相当,生理解剖结构也与人类非常相似,肠道器官的重量及功能特征都与人类极为相似,易用于研究人畜共患病,是实验室常用的哺乳动物模型[21]。与灵长类动物相比,使用猪作为实验动物更加经济;与啮齿类动物等相比猪的免疫系统更具有代表性,其免疫系统超过80%的分析参数与人类相似[22],因此猪的肠道模型更加适合研究致病菌感染后宿主的免疫反应。MEURENS等[23]建立了猪体内肠道循环模型,研究发现鼠伤寒沙门氏菌感染使促炎和T辅助因子1的mRNA表达水平更高,NOD2基因的转录产物增多,Toll样受体(Toll-like receptors, TLR)5 mRNA、趋化因子CCL25及其受体mRNA表达下降,这表明沙门氏菌能减少淋巴细胞归巢到肠道。猪模型是一种很有前景的大型实验动物模型,然而,其体型较大、价格较贵以及所需饲养环境较特殊等问题限制了这类模型的使用和发展。
当前,各种动物模型的使用为研究人员更加深入探索食源性致病菌肠道感染途径以及致病机制提供有效工具,除了无法避免伦理问题外,仍具有以下几方面的局限性:一是不同种类的动物对于同一种致病菌感染有时会出现不同表现,甚至同种属、同品种的动物在相同实验条件下也会表现出差异性,这影响了模型的一致性;二是动物和人类之间的物种差异,导致一些动物实验得到的结果与临床结果不符,由普通动物模型得出的结论不能直接应用到人体上;三是成本高、实验周期长且有些问题也无法用动物建立合适的模型。此外,有些国家和国际上出台了相关法律法规限制了科学实验中动物的使用,并且3R原则(减少、替代、优化)也旨在倡导减少动物实验。另外,未来应进一步探索优化食源性致病菌感染的体内模型的解决策略,包括开发更符合真实情况的经口感染动物模型、运用生物学技术构建与人体肠道更相似的动物模型、以及模拟人体肠道菌群等方面,为研究者深入了解食源性致病菌致病机制及有效防控食源性致病菌提供更适合的动物模型。
2 食源性致病菌感染的体外模型
尽管体内肠道模型已广泛用于食源性致病菌的研究中,但致病菌导致的症状以及致病机制在人与动物之间存在一定差异,在使用上具有一定的局限性,并且国际上以及个别国家对动物实验的规定和限制,推进了代替动物实验方法的发展。近年,在传统二维(two-dimensional,2D)细胞模型的基础上,三维(three-dimensional,3D)细胞模型、类器官和器官芯片等新型体外模型(表1)也被开发出来,并在食源性致病菌研究领域取得了一定的进展。几种不同类型的细胞模型如图1所示。
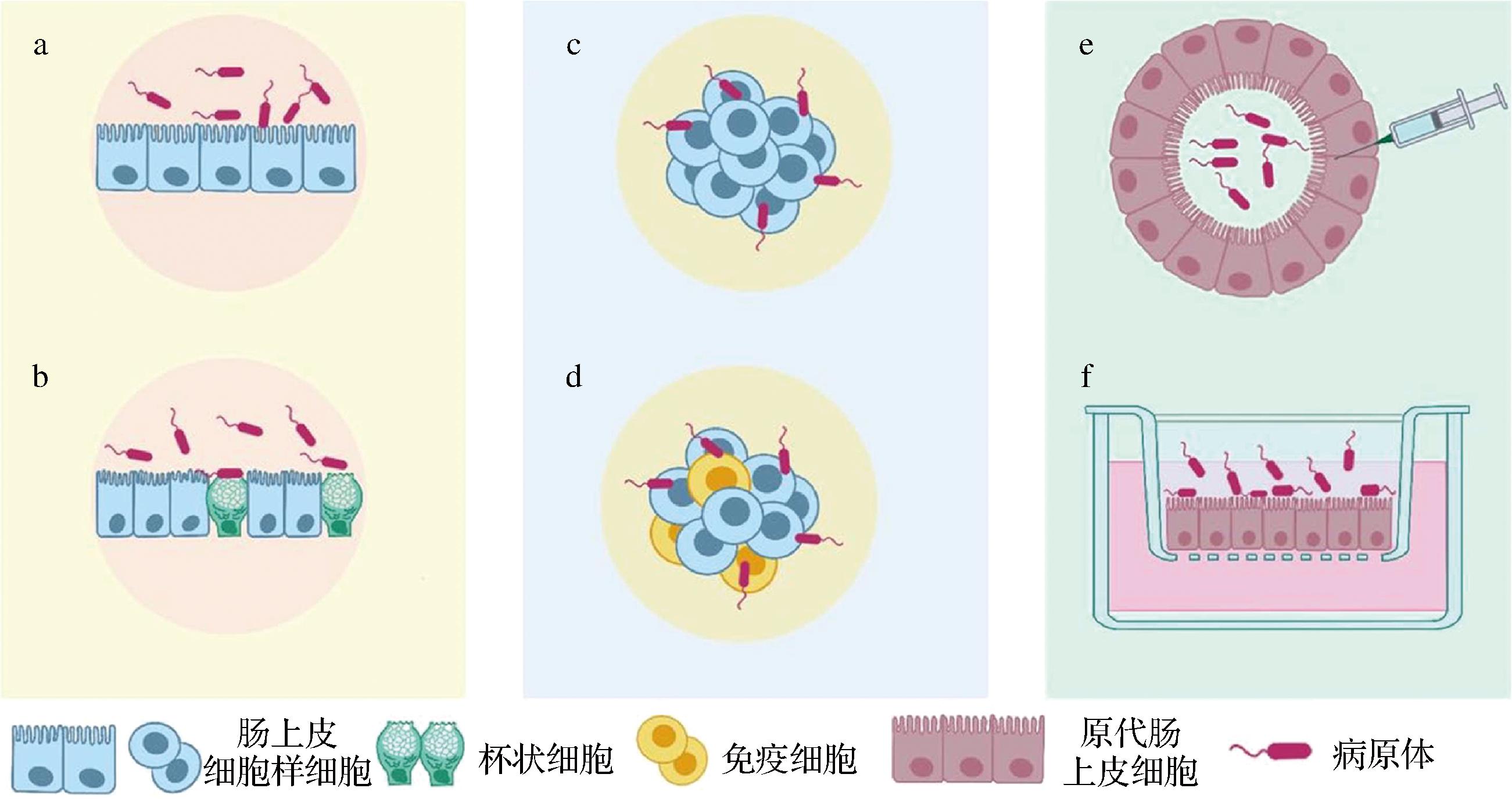
a-具有肠细胞样表型的永生化细胞系形成的同种极化细胞单层;b-模拟基本肠道特征的多种细胞共培养细胞单层;c-由一种永生化细胞系开发的3D细胞球;d-由多种永生化细胞系开发的3D细胞球;e-顶端表面朝内的肠道类器官(病原体通常被注射到类器官内部);f-类器官衍生的单层细胞系统
图1 模拟肠道食源性疾病的细胞模型(根据文献[24]改绘)
Fig.1 Cell models mimicking intestinal foodborne diseases[24]
2.1 2D细胞模型
与动物模型相比,细胞模型能够大规模培养和筛选,简化了生物系统,相较于动物实验更经济且符合3R原则,因此细胞模型成为了动物模型首选的替代方案。最常用的2D细胞肠道模型,是使用单一的人类肿瘤细胞由来细胞系或原代细胞系等,将其培养于不预先涂层细胞外基质的培养皿上,形成的细胞单层或细胞多层的模型系统(图1-a)。在2D肠道模型中常用的肠道上皮细胞有人结直肠腺癌细胞(Caco-2)、人结肠癌细胞(HT29)、人结肠腺癌肺转移细胞(T84)以及人结肠腺癌细胞(SW480)等人类肠上皮癌细胞系,其中Caco-2是目前应用最广泛、最成熟的细胞系。研究人员基于上述2D细胞系统从微观层面探究了食源性致病菌感染肠上皮细胞的作用规律。MATHIPA等[26]使用表达单增李斯特菌内化素蛋白InlA和内化素蛋白B(internalin B,InlB)的干酪乳杆菌(LbcinlAB)与单增李斯特菌同时感染Caco-2单层细胞,结果显示LbcinlAB可以通过与单增李斯特菌竞争结合位点及生长繁殖所需养分降低单增李斯特菌对细胞单层的黏附、侵袭及穿透。TAKAHASHI等[27-28]使用Caco-2体外细胞模型,证明了副溶血性弧菌耐热直接溶血素引起生物腹泻的机制,其能引起Cl-分泌增加进而升高细胞内Ca2+。MCNAMARA等[29]用T84细胞模型证明EPEC通过Ⅲ型分泌系统分泌的EspF蛋白能转移到宿主细胞的细胞质中,造成跨上皮电阻消失、肠上皮单层细胞旁途径通透性增加、以及Occludin蛋白损伤,从而破坏肠道的屏障功能。
为使2D系统更接近人体肠道微环境,研究人员建立了肠道免疫共培养模型(图1-b)。ZAMORA等[30]将人肠上皮细胞C2BBe1与树突状细胞及能产生黏液的HT29-MTX细胞共培养,建立了肠道免疫共培养模型,发现N-连接糖基化途径受损的空肠弯曲菌菌株黏附和侵入该模型的能力比野生型降低了100倍。RODRIGUES等[31]使用HT29细胞和HT29-MTX细胞的共培养模型,显示肿瘤坏死因子α(tumor necrosis factor α, TNFα)能刺激人体细胞产生促炎细胞因子,降低空肠弯曲菌对细胞的黏附和侵袭的能力;与不产生黏液的HT29细胞相比,HT29-MTX细胞产生的黏液能起到保护上皮细胞的作用,TNFα对空肠弯曲菌黏附侵袭能力的影响更显著。
由于2D细胞模型简化了复杂的生物系统,因此2D系统可以方便比较不同菌株对同一细胞或同一菌株对不同细胞的作用,从而初步筛选出符合需求的菌株或细胞模型。GERARDI等[32]基于Caco-2细胞和SW480细胞单层细胞模型,发现红酒渣产品能通过改变细菌与受体细胞作用的连接蛋白E-cadherin及Claudin进而抑制单增李斯特菌侵入肠道上皮细胞。此外,也发现claudin基因只参与单增李斯特菌对SW480细胞的入侵,因此,SW480细胞系可能是研究单增李斯特菌感染时受体claudin基因表达的有效模型。GHARBI等[33]基于HT29 2D细胞模型,发现人类粪便分离的发酵乳杆菌88和植物乳杆菌9显出对HT29细胞的高黏附,并能通过竞争作用降低大肠杆菌对HT29细胞的黏附,但不能抑制阪崎克罗诺杆菌和沙门氏菌对HT29细胞的黏附。
2D系统操作简单、可重复性强,在研究细胞促炎因子、细菌与宿主细胞的分子相互作用、单一益生菌与致病菌的拮抗作用,细菌侵袭肠道上皮细胞的结合位点等方面获得了较多结果,但是大部分2D肠道模型,多使用肿瘤由来的细胞,在培养中细胞会出现扁平的形态、异常分裂、丧失分化表型等情况[34],与人类原代肠道上皮细胞之间存在显著差异,不能很好地表征人体宿主细胞的实际特征。即使使用原代细胞构建2D细胞模型,建立的多是在培养皿接触表面的单层或多层结构,不能模拟体内细胞与细胞之间、以及细胞与细胞外基质之间的通讯和连接作用,因此不能获得体内细胞的生理特征。另外,由一类细胞系建立的2D细胞模型,不能模拟体内肠道屏障多类型细胞的复杂生理结构和特征,在研究致病菌如何靶向和跨越肠道屏障方面存在局限性。
2.2 3D细胞模型
近年来,随着生物工程以及纳米技术的出现,在3D限定的结构中培养不同类型的细胞已经取得成效,其不仅允许两种或多种细胞类型相互作用,还允许细胞在生理相关的环境中生长,并在所有3D结构中与相邻细胞接触,促进细胞活性相关的蛋白表达量以及激素分泌,与人类生理环境都更为接近(图1-c和图1-d)。BARRILA等[35]发现由人组织细胞淋巴瘤细胞U937与人结肠癌细胞HT29构建的一种新型3D共培养感染系统(图1-d),与仅由HT29一种细胞系构成的3D系统(图1-c)相比,具有免疫活性,能抑制沙门氏菌的黏附、侵袭及存活。该结果更接近人体内巨噬细胞能保护宿主免受肠道病原体侵害的事实,强调了免疫细胞在宿主与病原体早期相互作用中的重要性。与源自同一细胞系的单层2D细胞模型相比,3D球体模型与天然肠道上皮组织更接近,细胞间不再是单纯的物理接触,形成了细胞间生物通信,对病原菌的反应也与2D培养的细胞不同。在研究鼠伤寒沙门氏菌对人类的感染时,NICKERSON等[36]发现,使用人小肠上皮细胞INT407构建的3D培养物比2D培养物更准确地模拟了人体内分化的组织,与2D模型相比,沙门氏菌对3D模型INT407细胞的黏附、侵袭和诱导凋亡的能力显著降低。陈晓清等[37]利用Caco-2细胞与正常混合菌(含有乳双歧杆菌、植物乳杆菌、大肠杆菌及粪球杆菌)建立了一个稳定的3D体外模拟肠道共生系统,并证实了正常肠道菌群对入侵的沙门氏菌有拮抗作用。
综上,在研究食源性致病方面,与2D细胞肠道模型体系相比,3D细胞模型具有优越性,也取得了诸多进展,但仍面临许多问题和挑战。如3D培养价格昂贵,培养技术较为复杂没有统一标准,不同细胞系模拟人类身体微环境的程度存在差异等。3D细胞模型在研究食源性致病菌方面,仍处于起步阶段,在构建应用于食源性致病菌研究的3D模型体系,在解析致病菌毒性机制、毒力因子作用、趋向性等方面仍需进行更多的验证和评估。另外,3D细胞模型作为动物模型等体内模型的替代模型,其优越性仍有待进一步证实。
2.3 肠道类器官
肠道类器官一般指通过3D培养技术,利用从肠道组织中获取的干细胞或多能干细胞,通过自我更新和分化能力,在体外培养出结构及功能都与肠道组织类似的细胞团(图1-e和图2)。由于类器官在形成过程中有自组装的特性,能在体外模拟人体组织的复杂结构。SATO等[38]利用小鼠Lgr5+干细胞首次在体外培养出小肠隐窝-绒毛类器官(图2-a)。ZIETEK等[39]利用野生型C57BL/6小鼠小肠隐窝组织制备小肠类器官模型(图2-b),该模型保留了小肠营养吸收及屏障功能等特征,可用于研究物质运输、传感和激素分泌。WANG等[40]使用3T3-J2细胞作为滋养层,利用胎儿肠道干细胞制备出肠道类器官模型(图2-a),该模型能精确再现肠上皮结构和肠道屏障功能,并能在体外长期维持。与传统2D以及2.2节中所述的3D细胞模型相比,类器官能增殖分化,维持基因组稳定;能较好模拟人体内的细胞的状态,再现病原菌与肠道细胞间的相互作用和生物过程;并且体外操作降低了伦理风险,更加直观、经济。随着肠类器官培养技术的发展,肠类器官用于食源性致病菌的研究将成为一个趋势,例如,用于研究食源性致病菌感染的细胞和组织趋向性、食源性感染后宿主的免疫反应、食源性致病菌的毒力机制以及益生菌抗食源性疾病的活性等。
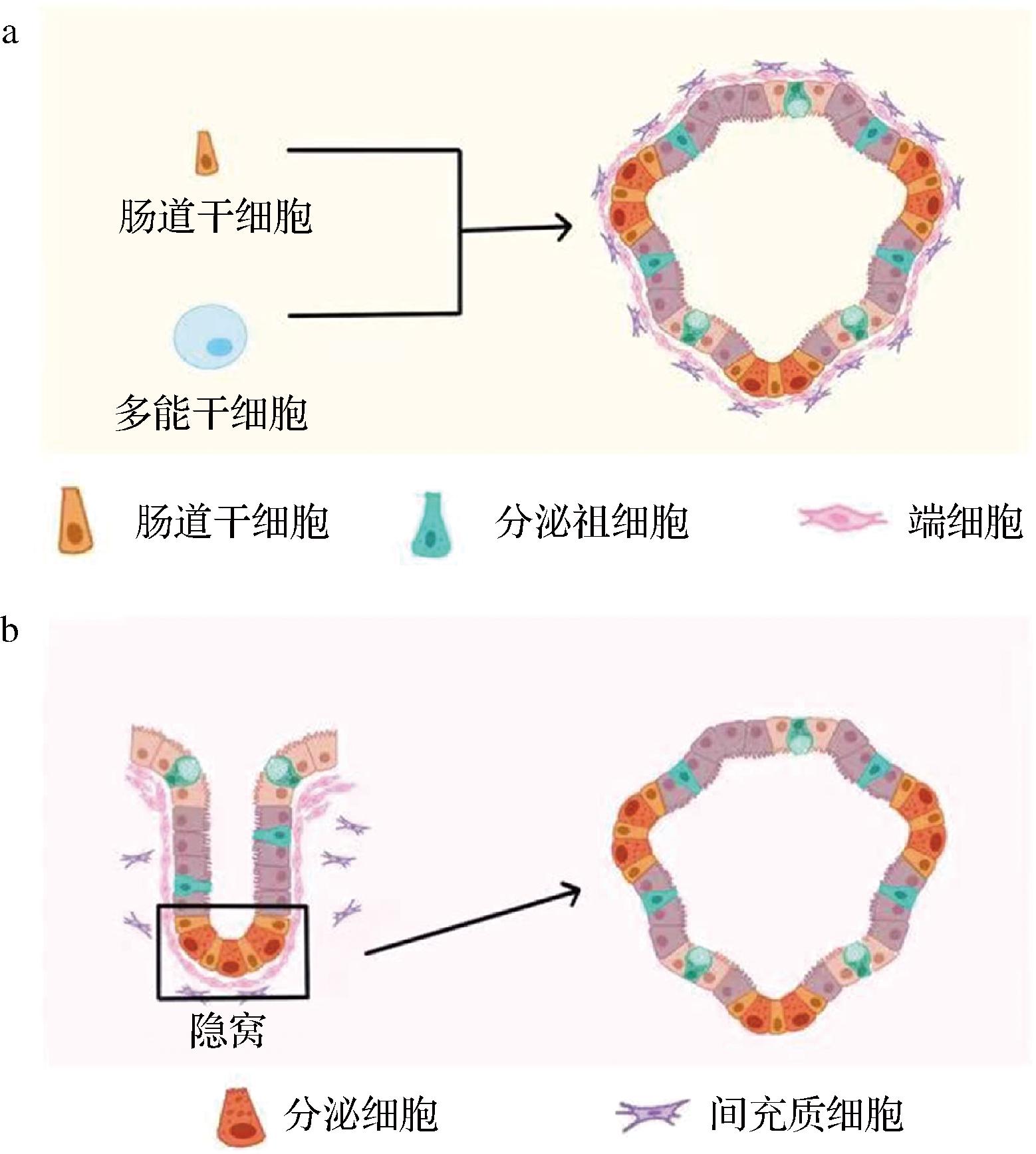
a-由肠道干细胞、多能干细胞衍生的肠道类器官;b-由隐窝分离的成体干细胞衍生的肠道类器官
图2 肠道类器官示意图(根据文献[24]改绘)
Fig.2 Schematic diagram of intestinal organoid[24]
食源性致病菌感染肠道的部位和能力是不同的,使用类器官可以探索食源性致病菌感染的细胞和组织趋向性。IN等[41]使用成人近端结肠干细胞培养的类器官建立了肠出血性大肠埃希氏菌(enterohemorrhagic Escherichia coli,EHEC)感染模型,发现空肠类器官附着的细菌很少,产生黏液的分化的结肠类器官优先被定殖。VANDUSSEN等[42]使用人体肠类器官发现,EPEC菌株优先黏附到回肠上皮细胞上,肠聚集性大肠杆菌(enteroaggregative Escherichia coli, EAggEC)菌株则优先粘附到直肠上皮细胞上。类器官具有与体内肠道相同的遗传信息并且易于基因编辑,因此可用来研究食源性致病菌感染后宿主的免疫反应。CROWLEY等[43]建立了半光天冬酶(caspase)-1/11缺陷型Casp1/11-/-肠类器官模型,这些模型排除了肠道微生物群和免疫细胞等复杂因素,能直接反应肠道上皮细胞对鼠伤寒沙门氏菌的控制作用,发现caspase-1和caspase-11分别在感染早期和后期发挥作用。WILSON等[44]利用正常及Mmp7基因缺陷的小鼠小肠隐窝培养出类器官,模型表明正常类器官产生的α-防御素能在注射鼠伤寒沙门氏菌后一段时间内限制类器官管腔内致病菌生长;在缺陷型类器官中转基因表达人体防御素5能补偿抗菌活性。
食源性致病菌通过不同机制感染宿主,通过研究食源性致病菌的毒力机制有助于开发药物、疫苗及检测方法。类器官模型突破了传统实验模型的限制,正逐渐成为研究致病菌毒力机制的新工具。为了探究鼠伤寒沙门氏菌感染的完整周期过程,GEISER等[45]通过显微注射、延时显微镜等方式建立了一种具有高度时间和空间分辨率的肠类器官感染模型,通过追踪感染的时间变化,确定了鼠伤寒沙门氏菌Ⅲ型分泌系统1(type-three-secretion-system-1, TTSS-1)促进其入侵上皮细胞、在上皮细胞内复制并通过感染的上皮细胞排出进行循环,从而确保了病原菌的有效定植。除上述应用,肠类器官还具有探索益生菌拮抗食源性致病菌活性的潜力,以此为治疗耐药性细菌感染提供新方法。PRADHAN等[46]使用H1人胚胎干细胞分化形成肠类器官,发现大肠杆菌菌株Nissle1917(EcN)作为益生菌可以通过激活宿主的防御系统,防止大肠杆菌致病性菌株EHEC和尿路致病性大肠埃希氏菌(uropathogenic Escherichia coli, UPEC)引起的肠道上皮屏障功能丧失和细胞凋亡。
国内外应用类器官模型开展食源性致病菌的研究仍处于起步阶段。虽然类器官相比其他体外细胞模型优势显著,但仍存在一些亟需解决的问题,例如,目前培养出的类器官比较低级,没有血管、神经等参与,且大部分类器官的上皮细胞顶端向内后被包裹在管腔内(图1-e),这导致在操作过程中很难使病原体与上皮细胞顶端直接接触,且腔内物不断积累可能会对实验结果产生影响。尽管可以使用显微注射技术,但是需要精密的操作和显微设备,因此在使用上受到了一定的局限。为了改善这一问题,CO等[47]将肠道类器官包埋在基底膜提取物中,利用螯合剂EDTA破坏细胞外基质(extracellular matrix,ECM),诱导上皮细胞极性反转使顶端表面朝外,使病原体与类器官的接触更容易(图3)。有研究人员使用Transwell系统以期将球状3D细胞体系平展为多层细胞模型(图1-f),但可能会破坏原有的自组装结构,并且需要大量细胞且通过显微镜不易进行观察[42]。此外,未来可以通过在肠道类器官中引入人体肠道微生物群和各种免疫细胞使类器官模型更好地模拟人体内真实情况,以此加强肠道类器官在研究食源性致病菌中的应用。
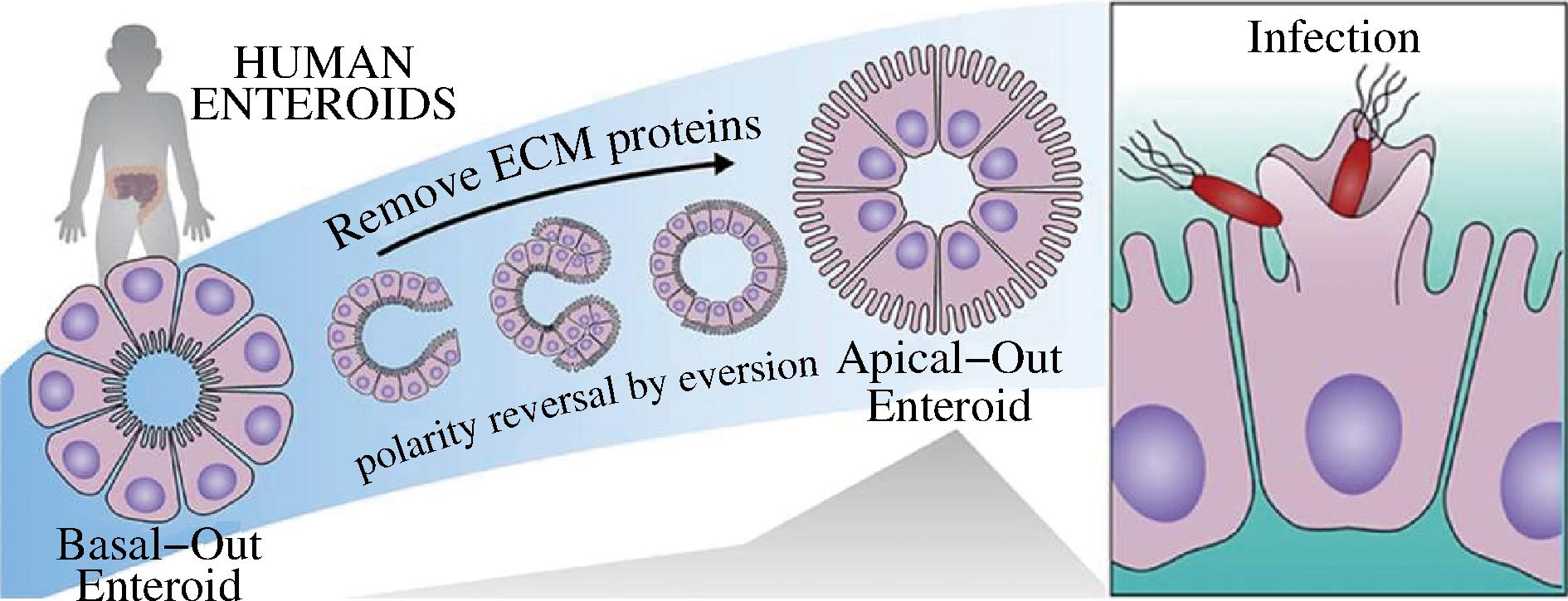
图3 肠上皮极性反转类器官示意图[47]
Fig.3 Schematic diagram of apical-out organoid[47]
2.4 器官芯片
传统体外模型都是静态模型,尽管3D肠道模型能够模拟肠上皮细胞的3D结构和生理功能,但无法模拟肠道微环境的特征,主要是液体流动和肠蠕动。近年,随着生物微工程的发展和应用,肠道芯片正成为肠道疾病建模、研究宿主与食源性致病菌相互作用、以及药物开发和测试的有力工具。如图4所示,肠道芯片是利用软光刻技术制造的聚二甲基硅氧烷芯片(图4-a),多孔膜将微流控通道分成两个腔室,上皮细胞接种在多孔膜上形成屏障(图4-b),通过介质流动可以确保营养分配和废物排出;通过循环真空诱导多孔膜拉伸从而重现蠕动状运动;还可合并多种细胞类型(图4-c)或肠道添加微环境背景微生物群从而更好地反映肠道结构和生理。目前研究人员已开发出不同种类的肠道芯片,具体信息见表2。
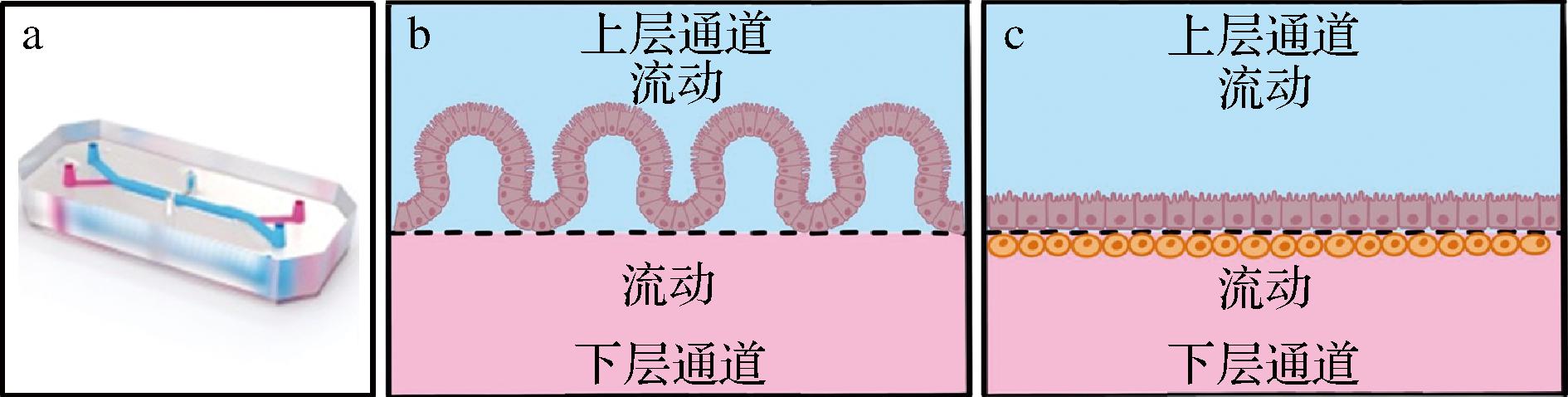
a-肠道芯片;b-在多孔膜上接种单一肠道细胞系制成的器官芯片;c-在多孔膜两侧接种不同细胞系制成的器官芯片
图4 肠道芯片示意图(根据文献[25,48]改绘)
Fig.4 Schematic diagram of the intestine-chip[25,48]
表2 不同种类的肠道芯片
Table 2 Different types of Intestine-on-a-chip

模型肠细胞或组织类型特点参考文献肠道芯片Caco-2低速液体流动、模拟生理蠕动、Caco-2细胞自发形成肠绒毛结构[49]小肠芯片十二指肠类器官可连续收集流经肠芯片内腔的液体、模拟天然十二指肠的结构和功能、具有屏障功能[50]空肠芯片空肠类器官与内皮细胞共培养、持续分化[51]结肠芯片结肠类器官与内皮细胞共培养、紧密的上皮屏障[48]OrganoPlate®Caco-2、iPSC、HT29-MTX-E12无膜、无需特定的微流体技术或专业设备[52]HMITMCaco-2有两个隔室、可与人体胃肠道连续模拟器耦合[53]HuMiXCaco-2实时监测O2浓度、同时满足细胞需氧条件及细菌厌氧条件[54]PMI ChipCaco-2非线性管腔、实时成像、多轴变形[55]GuMI结肠类器官结肠上皮细胞尖端氧浓度低可与厌氧菌长期共培养[56]
GRASSART等[25]利用肠道芯片探讨了液体流动和肠蠕动对志贺氏菌的感染性是否有影响,结果显示隐窝结构可以保护细菌免受腔内液体流动的影响,此外蠕动会明显增强志贺氏菌对上皮细胞的入侵,最小细菌负荷足以使细菌从顶端侵入肠细胞并引发屏障完整性丧失,并证实了微环境对细菌入侵肠道上皮屏障的重要性。KIM等[57]利用肠道芯片结合肠道微生物构建了肠侵袭性大肠埃希氏菌(enteroinvasive Escherichia coli,EIEC)感染模型,结果表明益生菌能够显著增强肠道屏障功能,与未加入益生菌相比,添加益生菌后EIEC感染引起破坏性损伤显著减少,并且益生菌还能抑制EIEC的过度生长,此外,通过调节参数改变肠蠕动情况,在体外模拟肠梗阻患者的情况,发现细菌会过度生长,这与临床症状相似。SUNUWAR等[58]利用空肠类器官构建器官芯片,证实与静止状态相比,机械流动增加了ETEC毒力因子热稳定肠毒素(heat-stable enterotoxin, ST)诱导的小肠上皮细胞内环二鸟苷酸(cyclic guanosine monophosphate,cGMP)分泌及含量增加,进而导致肠液和电解质损失,同时,动态肠道芯片模型,引发了肠上皮细胞的特征变化,包括机械刺激产生的绒毛结构。
器官芯片模型为致病菌致病机制的研究提供了新平台。与传统模型相比,肠道芯片具有模拟肠道功能和可实时观察的特点,但肠道芯片制作过程复杂,操作繁琐,不同实验室之间的再现性较差,亟需制定统一标准。此外,机械力的增加是肠道芯片的优势,然而,目前的肠道芯片并未完全模拟出肠道的复杂性,如体外重复拉伸不能完全模拟体内肠道不规则蠕动的复杂性,以及肠道芯片中液体流动与人体肠道内液体流动的差异等,未来还需要进一步优化肠道芯片,以更好地重现体内食源性致病菌感染的实际情况。
3 结论和展望
为深入研究食源性致病菌感染机制及控制方法,建立有效的食源性致病菌感染肠道的体内外模型至关重要。本文总结了食源性致病菌的毒性,并详细描述了常用的体内外模型的特征,介绍了它们在研究食源性致病菌侵袭肠道屏障方面的研究进展,比较了不同模型的优势和局限性,讨论了它们在研究病原菌-宿主相互作用机制方面的潜在作用。
由于天然肠道环境的复杂性,现阶段的肠道模型尚不能完全模拟真实肠道情况,不足以完全揭示致病菌在体内与上皮细胞的相互作用,亟需新型体外模型的研究与开发。因此,本文针对目前利用体内外肠道模型研究食源性致病菌感染中的不足,提出4点展望:(1)基于体内外肠道模型,对菌株异质性进行研究。同种菌株不同亚型之间存在致病性、耐受、等方面差异。在今后的研究中,进一步以不同亚型的菌株为对象开展更为精细化的毒性研究,将为未来食源性致病菌的精准风险评估提供科学依据;(2)基于致病菌感染进程及其对不同脏器的伤害,对食源性致病菌毒性的研究,也将从体内各脏器的感染周期、宏观感染过程等内容转变为更为精准地对某一宿主细胞或某一信号通路的分子机制研究方面,因此从体内外模型的选择方面,则更强调有效性和精准性;(3)运用现代生物学技术对动物模型进行改造以更好模拟人体情况。动物模型能反应食源性致病菌的总体感染症状,但仍需要进一步评估食源性致病菌菌株类型与动物模型感染的匹配性,包括动物的人源化改造等;(4)建立更有效、更标准的体外细胞模型。新兴3D细胞模型、器官芯片等未来将成为更有潜力的食源性致病菌感染模型,但是其与人体实际情况的差异,以及模型构建技术的便捷性和统一性等方面仍需要进一步验证。另外,选择多个体内外互补模型进行实验,并与人体组织表型进行比较,将会增加食源性致病菌与宿主相互作用研究的可能性。开发并验证体内外模型的有效性,进一步解析食源性致病菌的致病机制,将为防止食源性致病菌的感染提供科学依据。
[1] CHARLIER C, PERRODEAU É, LECLERCQ A, et al.Clinical features and prognostic factors of listeriosis:The MONALISA national prospective cohort study[J].The Lancet Infectious Diseases, 2017, 17(5):510-519.
[2] 覃兰迁, 刘霞, 刘诚志, 等.迷迭香水提物对沙门氏菌致小鼠肠道损伤的抗炎保护作用研究[J].饲料研究, 2023, 46(11):81-85.QIN L Q, LIU X, LIU C Z, et al.Study on anti-inflammatory and protective effect of Rosmarinus officinalis extract on intestinal injury induced by Salmonella in mice[J].Feed Research, 2023, 46(11):81-85.
[3] DONG N, LI X R, XUE C Y, et al.Astragalus polysaccharides attenuated inflammation and balanced the gut microflora in mice challenged with Salmonella typhimurium[J].International Immunopharmacology, 2019, 74:105681.
[4] 王兆雨, 吴晓宇, 裴小琪, 等.生物活性肽对产肠毒素大肠杆菌诱导的小鼠肠道损伤的缓解作用[J].动物营养学报, 2023, 35(7):4659-4667.WANG Z Y, WU X Y, PEI X Q, et al.Mitigative effects of bioactive peptide on intestinal injury of mice induced by enterotoxigenic Escherichia coli[J].Chinese Journal of Animal Nutrition, 2023, 35(7):4659-4667.
[5] HUGENHOLTZ F, DE VOS W M.Mouse models for human intestinal microbiota research:A critical evaluation[J].Cellular and Molecular Life Sciences:CMLS, 2018, 75(1):149-160.
[6] FAURE S, PERRIN-GUYOMARD A, DELMAS J M, et al.Transfer of plasmid-mediated CTX-M-9 from Salmonella enterica serotype Virchow to Enterobacteriaceae in human flora-associated rats treated with cefixime[J].Antimicrobial Agents and Chemotherapy, 2010, 54(1):164-169.
[7] LECUIT M, DRAMSI S, GOTTARDI C, et al.A single amino acid in E-cadherin responsible for host specificity towards the human pathogen Listeria monocytogenes[J].The EMBO Journal, 1999, 18(14):3956-3963.
[8] LECUIT M, VANDORMAEL-POURNIN S, LEFORT J, et al.A transgenic model for listeriosis:Role of internalin in crossing the intestinal barrier[J].Science, 2001, 292(5522):1722-1725.
[9] BARRÉ-SINOUSSI F, MONTAGUTELLI X.Animal models are essential to biological research:Issues and perspectives[J].Future Science OA, 2015, 1(4):FSO63.[10] SEOK J, WARREN H S, CUENCA A G, et al.Genomic responses in mouse models poorly mimic human inflammatory diseases[J].Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9):3507-3512.
[11] RASMUSSEN A L, OKUMURA A, FERRIS M T, et al.Host genetic diversity enables Ebola hemorrhagic fever pathogenesis and resistance[J].Science, 2014, 346(6212):987-991.
[12] HIGGINSON E E, PANDA A, TOAPANTA F R, et al.Immunogenicity and efficacy of live-attenuated Salmonella Typhimurium vaccine candidate CVD 1926 in a Rhesus macaque model of gastroenteritis[J].Infection and Immunity, 2021, 89(10):e0008721.
[13] ROLLENHAGEN J E, JONES F, HALL E, et al.Establishment, validation, and application of a new world primate model of enterotoxigenic Escherichia coli disease for vaccine development[J].Infection and Immunity, 2019, 87(2):e00634-e00618.
[14] TAYLOR F B Jr, TESH V L, DEBAULT L, et al.Characterization of the baboon responses to Shiga-like toxin:Descriptive study of a new primate model of toxic responses to Stx-1[J].The American Journal of Pathology, 1999, 154(4):1285-1299.
[15] SANTOS R L, ALMEIDA A P, XAVIER M N, et al.Enteric pathology and Salmonella-induced cell death in healthy and SIV-infected rhesus macaques[J].Veterinary Pathology, 2011, 48(5):933-941.
[16] MATSUHISA F, KITAJIMA S, NISHIJIMA K, et al.Transgenic rabbit models:Now and the future[J].Applied Sciences, 2020, 10(21):7416.
[17] KUEHL C J, D’GAMA J D, WARR A R, et al.An oral inoculation infant rabbit model for Shigella infection[J].mBio, 2020, 11(1):e03105-e03119.
[18] HECZKO U, ABE A, FINLAY B B.Segmented filamentous bacteria prevent colonization of enteropathogenic Escherichia coli O103 in rabbits[J].The Journal of Infectious Diseases, 2000, 181(3):1027-1033.
[19] RITCHIE J M, RUI H P, ZHOU X H, et al.Inflammation and disintegration of intestinal villi in an experimental model for Vibrio parahaemolyticus-induced diarrhea[J].PLoS Pathogens, 2012, 8(3):e1002593.
[20] RUI H P, RITCHIE J M, BRONSON R T, et al.Reactogenicity of live-attenuated Vibrio cholerae vaccines is dependent on flagellins[J].Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(9):4359-4364.
[21] MEURENS F, SUMMERFIELD A, NAUWYNCK H, et al.The pig:A model for human infectious diseases[J].Trends in Microbiology, 2012, 20(1):50-57.
[22] DAWSON H D, SMITH A D, CHEN C, et al.An in-depth comparison of the porcine, murine and human inflammasomes;lessons from the porcine genome and transcriptome[J].Veterinary Microbiology, 2017, 202:2-15.
[23] MEURENS F, BERRI M, AURAY G, et al.Early immune response following Salmonella enterica subspecies enterica serovar Typhimurium infection in porcine jejunal gut loops[J].Veterinary Research, 2009, 40(1):5.
[24] AGUIRRE GARCIA M, HILLION K, CAPPELIER J M, et al.Intestinal organoids:New tools to comprehend the virulence of bacterial foodborne pathogens[J].Foods, 2022, 11(1):108.
[25] GRASSART A, MALARDÉ V, GOBAA S, et al.Bioengineered human organ-on-chip reveals intestinal microenvironment and mechanical forces impacting Shigella infection[J].Cell Host &Microbe, 2019, 26(3):435-444.
[26] MATHIPA M G, THANTSHA M S, BHUNIA A K.Lactobacillus casei expressing internalins A and B reduces Listeria monocytogenes interaction with Caco-2 cells in vitro[J].Microbial Biotechnology, 2019, 12(4):715-729.
[27] TAKAHASHI A, SATO Y, SHIOMI Y, et al.Mechanisms of chloride secretion induced by thermostable direct haemolysin of Vibrio parahaemolyticus in human colonic tissue and a human intestinal epithelial cell line[J].Journal of Medical Microbiology, 2000, 49(9):801-810.
[28] TAKAHASHI A, IIDA T, NAIM R, et al.Chloride secretion induced by thermostable direct haemolysin of Vibrio parahaemolyticus depends on colonic cell maturation[J].Journal of Medical Microbiology, 2001, 50(10):870-878.
[29] MCNAMARA B P, KOUTSOURIS A, O’CONNELL C B, et al.Translocated EspF protein from enteropathogenic Escherichia coli disrupts host intestinal barrier function[J].The Journal of Clinical Investigation, 2001, 107(5):621-629.
[30] ZAMORA C Y, WARD E M, KESTER J C, et al.Application of a gut-immune co-culture system for the study of N-glycan-dependent host-pathogen interactions of Campylobacter jejuni[J].Glycobiology, 2020, 30(6):374-381.
[31] RODRIGUES R C, POCHERON A L, CAPPELIER J M, et al.An adapted in vitro assay to assess Campylobacter jejuni interaction with intestinal epithelial cells:Taking into stimulation with TNFα[J].Journal of Microbiological Methods, 2018, 149:67-72.
[32] GERARDI G, RIVERO-PÉREZ M D, CAVIA-SAIZ M, et al.Wine pomace product inhibit Listeria monocytogenes invasion of intestinal cell lines Caco-2 and SW-480[J].Foods, 2021, 10(7):1485.
[33] GHARBI Y, FHOULA I, RUAS-MADIEDO P, et al.In-vitro characterization of potentially probiotic Lactobacillus strains isolated from human microbiota:Interaction with pathogenic bacteria and the enteric cell line HT29[J].Annals of Microbiology, 2019, 69(1):61-72.
[34] LANGERHOLC T, MARAGKOUDAKIS P A, WOLLGAST J, et al.Novel and established intestinal cell line models - An indispensable tool in food science and nutrition[J].Trends in Food Science &Technology, 2011, 22:S11-S20.
[35] BARRILA J, YANG J, CRABBÉ A, et al.Three-dimensional organotypic co-culture model of intestinal epithelial cells and macrophages to study Salmonella enterica colonization patterns[J].NPJ Microgravity, 2017, 3:10.
[36] NICKERSON C A, GOODWIN T J, TERLONGE J, et al.Three-dimensional tissue assemblies:Novel models for the study of Salmonella enterica serovar Typhimurium pathogenesis[J].Infection and Immunity, 2001, 69(11):7106-7120.
[37] 陈晓清, 焦红, 程树军, 等.Caco-2细胞与肠道菌共培养初建体外肠道共生模型[J].中山大学学报(医学科学版), 2012, 33(1):121-126.CHEN X Q, JIAO H, CHENG S J, et al.Co-culture of Caco-2 intestinal cell and intestinal floras to build an in vitro intestinal model[J].Journal of Sun Yat-Sen University (Medical Sciences), 2012, 33(1):121-126.
[38] SATO T, VRIES R G, SNIPPERT H J, et al.Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche[J].Nature, 2009, 459(7244):262-265.
[39] ZIETEK T, RATH E, HALLER D, et al.Intestinal organoids for assessing nutrient transport, sensing and incretin secretion[J].Scientific Reports, 2015, 5:16831.
[40] WANG X, YAMAMOTO Y, WILSON L H, et al.Cloning and variation of ground state intestinal stem cells[J].Nature, 2015, 522(7555):173-178.
[41] IN J, FOULKE-ABEL J, ZACHOS N C, et al.Enterohemorrhagic Escherichia coli reduce mucus and intermicrovillar bridges in human stem cell-derived colonoids[J].Cellular and Molecular Gastroenterology and Hepatology, 2016, 2(1):48-62.
[42] VANDUSSEN K L, MARINSHAW J M, SHAIKH N, et al.Development of an enhanced human gastrointestinal epithelial culture system to facilitate patient-based assays[J].Gut, 2015, 64(6):911-920.
[43] CROWLEY S M, HAN X, ALLAIRE J M, et al.Intestinal restriction of Salmonella Typhimurium requires caspase-1 and caspase-11 epithelial intrinsic inflammasomes[J].PLoS Pathogens, 2020, 16(4):e1008498.
[44] WILSON S S, TOCCHI A, HOLLY M K, et al.A small intestinal organoid model of non-invasive enteric pathogen-epithelial cell interactions[J].Mucosal Immunology, 2015, 8(2):352-361.
[45] GEISER P, DI MARTINO M L, SAMPERIO VENTAYOL P, et al.Salmonella enterica serovar typhimurium exploits cycling through epithelial cells to colonize human and murine enteroids[J].mBio, 2021, 12(1):e02684-e02620.
[46] PRADHAN S, WEISS A A.Probiotic properties of Escherichia coli nissle in human intestinal organoids[J].mBio, 2020, 11(4):e01470-e01420.
[47] CO J Y, MARGALEF-CATAL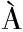 M, LI X N, et al.Controlling epithelial polarity:A human enteroid model for host-pathogen interactions[J].Cell Reports, 2019, 26(9):2509-2520.
M, LI X N, et al.Controlling epithelial polarity:A human enteroid model for host-pathogen interactions[J].Cell Reports, 2019, 26(9):2509-2520.
[48] APOSTOLOU A, PANCHAKSHARI R A, BANERJEE A, et al.A micro-engineered human colon intestine-chip platform to study leaky barrier[J].Cold Spring Harbor Laboratory, 2020. DOI: https://doi.org/10.1101/2020.08.28.271759.
[49] KIM H J, HUH D, HAMILTON G, et al.Human gut-on-a-chip inhabited by microbial flora that experiences intestinal peristalsis-like motions and flow[J].Lab on a Chip, 2012, 12(12):2165-2174.
[50] KASENDRA M, TOVAGLIERI A, SONTHEIMER-PHELPS A, et al.Development of a primary human small intestine-on-a-chip using biopsy-derived organoids[J].Scientific Reports, 2018, 8(1):2871.
[51] YIN J Y, SUNUWAR L, KASENDRA M, et al.Fluid shear stress enhances differentiation of jejunal human enteroids in Intestine-Chip[J].American Journal of Physiology.Gastrointestinal and Liver Physiology, 2021, 320(3):G258-G271.
[52] TRIETSCH S J, NAUMOVSKA E, KUREK D, et al.Membrane-free culture and real-time barrier integrity assessment of perfused intestinal epithelium tubes[J].Nature Communications, 2017, 8(1):262.
[53] MARZORATI M, VANHOECKE B, DE RYCK T, et al.The HMITM module:A new tool to study the Host-Microbiota Interaction in the human gastrointestinal tract in vitro[J].BMC Microbiology, 2014, 14:133.
[54] SHAH P, FRITZ J V, GLAAB E, et al.A microfluidics-based in vitro model of the gastrointestinal human-microbe interface[J].Nature Communications, 2016, 7:11535.
[55] SHIN Y C, SHIN W, KOH D, et al.Three-dimensional regeneration of patient-derived intestinal organoid epithelium in a physiodynamic mucosal interface-on-a-chip[J].Micromachines, 2020, 11(7):663.
[56] ZHANG J B, HUANG Y J, YOON J Y, et al.Primary human colonic mucosal barrier crosstalk with super oxygen-sensitive Faecalibacterium prausnitzii in continuous culture[J].Med, 2021, 2(1):74-98.
[57] KIM H J, LI H, COLLINS J J, et al.Contributions of microbiome and mechanical deformation to intestinal bacterial overgrowth and inflammation in a human gut-on-a-chip[J].Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(1):E7-E15.
[58] SUNUWAR L, YIN J Y, KASENDRA M, et al.Mechanical stimuli affect Escherichia coli heat-stable enterotoxin-cyclic GMP signaling in a human enteroid intestine-chip model[J].Infection and Immunity, 2020, 88(3):e00866-e00819.