诺如病毒(norovirus, NoV)是一类重要的食源性病毒,是引起人类胃肠道疾病的主要病原体之一,属于杯状病毒科(Calicivividae)诺如病毒属(Norovirus),其基因组由一条单链正义RNA组成,具有高度的多样性和变异性[1]。目前可以根据该病毒RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase, RdRp)的核酸序列和衣壳蛋白(capsid)氨基酸序列的不同,将其分为GⅠ~GⅩ等10个基因类群(genogroup),其中GⅠ、GⅡ、GⅣ、GⅧ、GⅨ等可以感染人类[2]。此外,又可以根据序列的差异,将GⅠ、GⅡ等基因类群进一步细分为更为多样的基因型(genotype),截止到2019年,国际杯状病毒工作组将其至少分为49个C型基因型(capsid type, C-type),60个P型基因型(polymerase type, P-type)[2]。
近年来,由牡蛎中诺如病毒污染引起的食品安全事件层出不穷,牡蛎作为一种常见的滤食性贝类,可以大量富集水体中的病毒颗粒,被认为是诺如病毒在环境中主要的传播载体[3]。前期研究发现,牡蛎中的诺如病毒具有高度的基因多样性,不同的基因型可能对宿主的感染性、致病性和传播能力有所差异。因此,通过研究牡蛎中诺如病毒的基因型多样性,可以揭示其遗传特征和传播规律,为疫情的预防和控制提供科学依据[4]。
目前,诺如病毒多样性的研究方法主要包括:传统的克隆测序技术和高通量测序技术。传统的克隆测序技术主要是通过反转录PCR(reverse transcription-PCR, RT-PCR)扩增、测序和序列比对,可以确定诺如病毒的基因型和亚型。然而,这种方法通常只能对有限的克隆子进行测序,往往会低估诺如病毒的基因型多样性[5-6]。扩增子测序是一种靶向的对PCR产物中所有的扩增子进行测序的方法,目前广泛应用于细菌16S rRNA基因、真菌18S rRNA基因以及ITS高度可变区等保守基因或功能基因探究群落微生物多样性[7]。而扩增子测序技术可以通过高通量测序将绝大多数甚至是全部扩增子进行测序分析,可以全面了解诺如病毒的多样性和分布情况。值得注意的是,通用引物与不同模板的匹配程度将影响扩增子的扩增效率,并进而导致目标病毒的漏检问题[8]。
本文采用经典引物(G2SKF/G2SKR),对贝类中诺如病毒多样性研究进行评估。先利用已知基因型GⅡ阳性粪便样品混合加入阴性牡蛎的消化腺组织内,模拟实际贝类中诺如病毒检测时的核酸环境,通过覆盖度,灵敏度2个方面证明扩增子测序高通量,高准确性等优势,避免了传统测序方法在检测牡蛎中诺如病毒时出现的非特异性扩增及假阴性等情况,提高了牡蛎中诺如病毒检出率及基因型多样性的准确性。
1 材料与方法
1.1 材料与仪器
1.1.1 病毒样本
本研究采用的诺如病毒阳性粪便样品,均由中国疾病预防控制中心(Chinese Center for Disease Control and Prevention,CDC)提供。随后,分别通过RT-PCR和反转录实时荧光定量PCR探针法(quantitative real-time RT-PCR,RT-qPCR)对样品中诺如病毒进行基因型和病毒浓度的确定。根据流行病学趋势,挑选6种代表性的基因型,包括GⅡ.12[P33](58415)、GⅡ.4_Den_Haag_2006b[P4]、GⅡ.2[P16]、GⅡ.3[P12](57429)、GⅡ.13[P13](57590)、GⅡ.17[P17],其病毒浓度如表1所示。
表1 诺如病毒样品及其浓度
Table 1 Norovirus samples and their concentrations
基因型GⅡ.17[P17]GⅡ.2[P16]GⅡ.12[P33]GⅡ.13[P13]GⅡ.3[P12]GⅡ.4_Den_Haag_2006b[P4]浓度/(copies/μL)3.09×1062.03×1064.40×1051.37×1057.77×1041.36×104
1.1.2 试剂与仪器
动物总RNA快速提取试剂盒,上海捷瑞生物工程有限公司;QIAamp®Viral RNA Mini Kit,上海凯杰生物技术有限公司;一步反转录试剂盒、RNA later,南京诺威赞生物科技股份有限公司;2×Taq Master Mix,近岸蛋白科技有限公司;TaqManTMRNA-to-CTTM一步法试剂盒,赛默飞世尔科技(中国)有限公司;PCR引物由上海生工生物股份有限公司合成。PCR仪、电泳仪,Bio-Rad公司;FastPrep-24MP组织均质器,美国MP生物公司。
1.2 实验方法
1.2.1 牡蛎样品的准备
在上海市芦潮港海鲜市场采集一批新鲜的牡蛎样品,置于冰上并迅速运回实验室进行解剖备用。选取牡蛎消化腺组织进行总RNA的提取。采用巢式RT-PCR方法进行GⅠ和GⅡ型诺如病毒的检测[9]。根据PCR检测结果,筛选出5只诺如病毒阴性的牡蛎,提取消化腺混合并匀浆,称取100 mg混合后的消化腺10份备用。
1.2.2 样品分组
a)为了评估扩增子测序的覆盖度,将诺如病毒阳性粪便样品人为添加至经验证阴性的牡蛎消化腺中,模拟诺如病毒阳性牡蛎的核酸环境。具体操作步骤如图1所示,将6种浓度和基因型已知的GⅡ型阳性粪便样品各取10 μL加入1.5 mL无菌离心管内漩涡振荡混匀,即为60 μL的混合基因型粪便样品(Mixed_stool);分别取10 μL以上混合基因型样品添加至2份诺如病毒阴性的牡蛎消化腺中制备人为诺如病毒污染的牡蛎样品(编号O1、O2);另再取10 μL混合基因型粪便样品(编号S3)作为阳性对照。
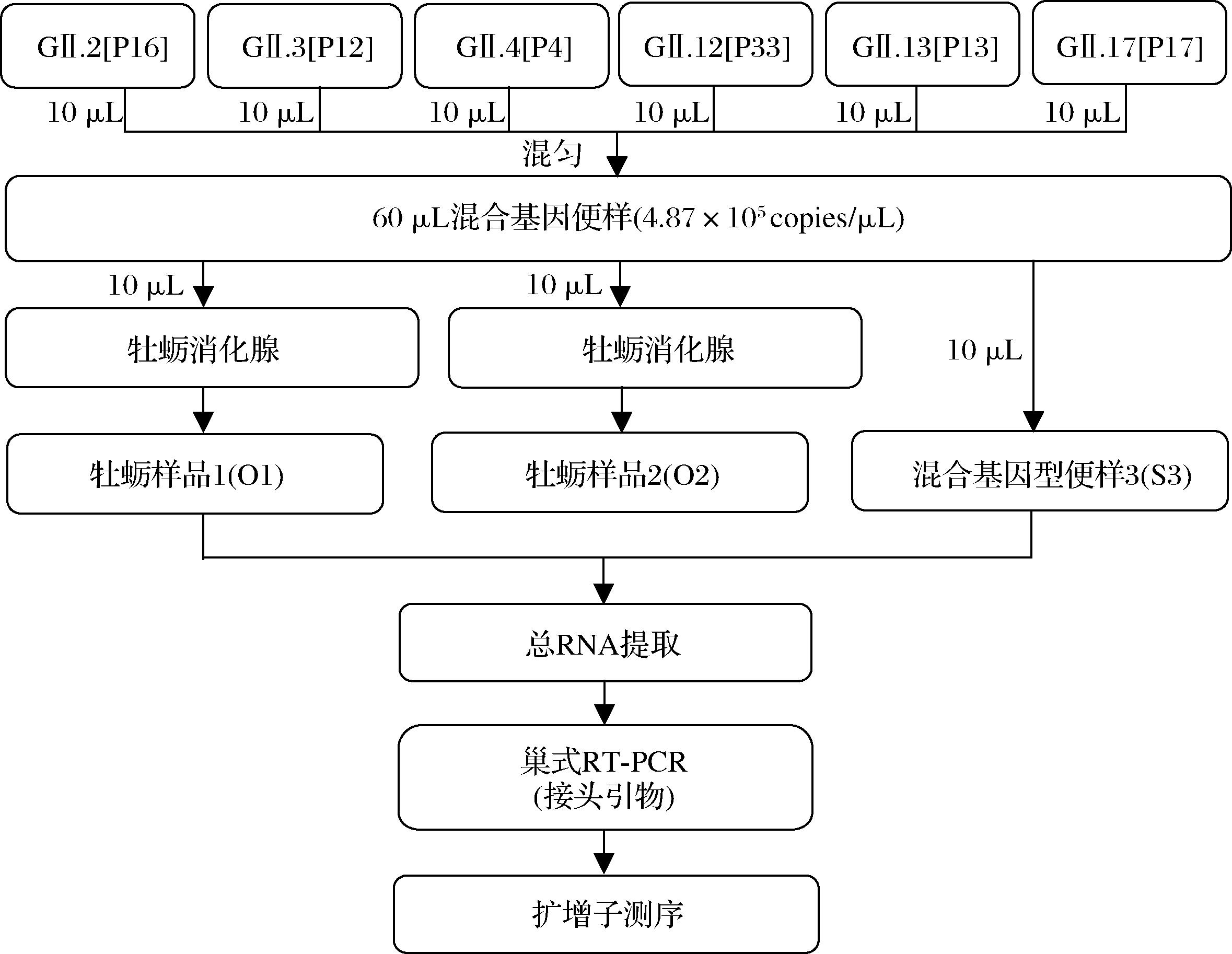
图1 扩增子测序覆盖度评估流程图
Fig.1 Flow chart for amplicon sequencing coverage assessment
注:NoV-表示诺如病毒阴性;NoV+表示GⅡ诺如病毒阳性(下同)。
b)为了评估扩增子测序的灵敏性,通过制备不同浓度梯度诺如病毒的阳性牡蛎进行实验。具体操作步骤如图2所示,取10 μL混合基因型粪便样品进行梯度稀释(101~104倍);取5种不同浓度梯度的混合基因型便样(10 μL)分别加入5份诺如病毒阴性的牡蛎中(编号A、B、C、D、E);另外取10 μL原始浓度的混合基因型便样(编号F)作为对照。

图2 扩增子测序灵敏度评估流程图
Fig.2 Flow chart for evaluating the sensitivity of amplicon sequencing
1.2.3 病毒浓度的定量
为了确定混合便样中诺如病毒的浓度,通过一步RT-qPCR进行相对定量。RT-qPCR反应体系(20 μL):10 μL的2×PCR Mix,各1.8 μL的正反向引物(10 μmol/L),0.5 μL的探针(10 μmol/L)(表2),0.5 μL的反转录酶,2 μL粪便样品RNA或RNA标准品作为模板,3.4 μL的ddH2O补足至20 μL。反应程序为:42 ℃逆转录15 min;95 ℃预变性10 min;95 ℃变性15 s,60 ℃退火60 s,45个循环;在60 ℃收集荧光信号。以标准品拷贝数的对数作为横坐标,以其对应的Ct值作为纵坐标,建立标准曲线,通过代入样品的Ct值计算出样品中诺如病毒的浓度[10]。
表2 GⅡ型诺如病毒 RT-qPCR引物及探针[11]
Table 2 Norovirus GⅡ RT-qPCR primers and probes[11]

引物引物核酸序列(5'-3')QNIF2ATGTTCAGRTGGATGAGRTTCTCWGACOG2RTCGACGCCATCTTCATTCACAQNIFs*α-AGCACGTGGGAGGGCGATCG-β
注:*表示探针序列;“α”表示5′端标记的FAM荧光基团;“β”表示3′端标记的TAMRA猝灭基团。
1.2.4 扩增子测序
将样品O1、O2、S3以及样品A~F进行动物组织总RNA提取,使用COG2F/G2SKR(经典引物中RT-PCR引物)进行RT-PCR扩增;将RT-PCR扩增产物稀释10倍作为模板,使用adaptor-G2SKF/G2SKR(经典引物被接头标记的巢式PCR引物)进行巢式PCR扩增(引物序列见表3),从而使第2轮PCR产物带有用于扩增子测序的接头序列;经2%的琼脂糖凝胶验证无误后送至上海美吉生物医药科技有限公司(简称美吉生物)完成扩增子测序。
表3 GⅡ型诺如病毒扩增子测序引物序列[12]
Table 3 Sequence of primers for sequencing amplicons of norovirus GⅡ[12]
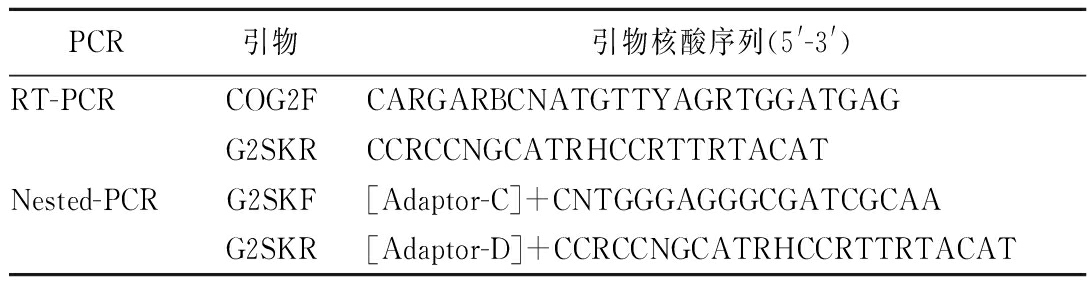
PCR引物引物核酸序列(5'-3')RT-PCRCOG2FCARGARBCNATGTTYAGRTGGATGAGG2SKRCCRCCNGCATRHCCRTTRTACATNested-PCRG2SKF[Adaptor-C]+CNTGGGAGGGCGATCGCAAG2SKR[Adaptor-D]+CCRCCNGCATRHCCRTTRTACAT
注:“Adaptor”表示引物的接头标记序列(由美吉生物提供),接头标记在引物5′端。
1.3 数据分析
扩增子测序得到序列的质控和拼接部分同样委托美吉生物完成。利用Illumina公司MiseqPE300平台进行测序。在美吉云平台将拼接后的序列以97%的相似度进行分类操作单元(operational taxonomic units,OTU)聚类,使用物种组成分析工具分析每组样品的OTU组成。将每个OTU的代表序列导入诺如病毒在线分型网站中进行在线分析得到每个OTU所代表的诺如病毒基因型[13]。使用Excel对结果进行统计,不同样品间不同基因型丰度差异使用物种组成分析工具(美吉云平台)或Rstudio作图呈现[14]。
2 结果与分析
2.1 粪便样品诺如病毒浓度的测定
将混合好的粪便样品通过RT-PCR进行定量, 6种基因型病毒混合的多基因型粪便样品的浓度为4.87×105 copies/μL。如表4所示,人为诺如病毒污染的牡蛎样品中病毒的最高浓度为每克消化腺4.87×106 copies,制备的GⅡ型诺如病毒阳性牡蛎样品中病毒浓度最低为每克消化腺487 copies。
表4 添加至每份牡蛎消化腺的诺如病毒颗粒数量
Table 4 Number of norovirus particles added to the digestive glands of each serving of oysters
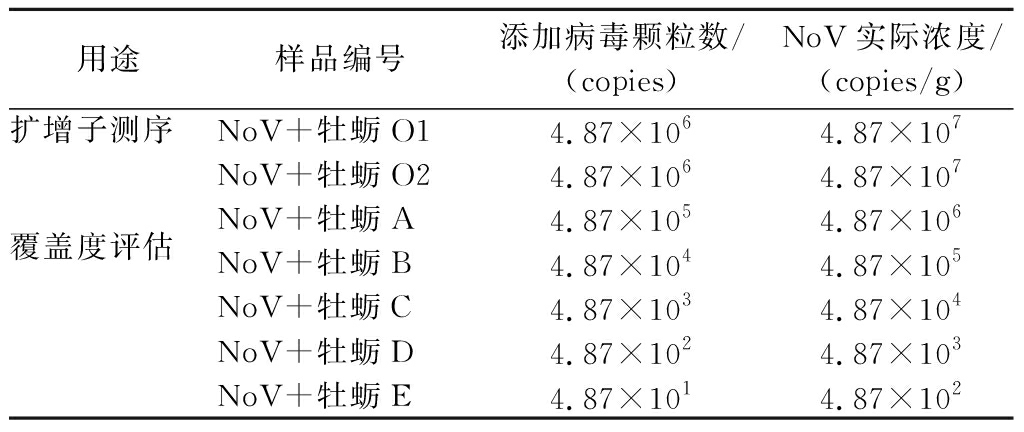
用途样品编号添加病毒颗粒数/(copies)NoV实际浓度/(copies/g)扩增子测序NoV+牡蛎O14.87×1064.87×107NoV+牡蛎O24.87×1064.87×107覆盖度评估NoV+牡蛎A4.87×1054.87×106NoV+牡蛎B4.87×1044.87×105NoV+牡蛎C4.87×1034.87×104NoV+牡蛎D4.87×1024.87×103NoV+牡蛎E4.87×1014.87×102
注:NoV+表示诺如病毒阳性。
2.2 覆盖度分析
鉴定结果如图3所示,带有接头标记的经典引物(G2SKF/G2SKR)在胶图上均有单一明亮的条带,表明PCR扩增产物满足后续的扩增子测序要求,带有接头的引物不会影响引物的特异性和扩增效率。
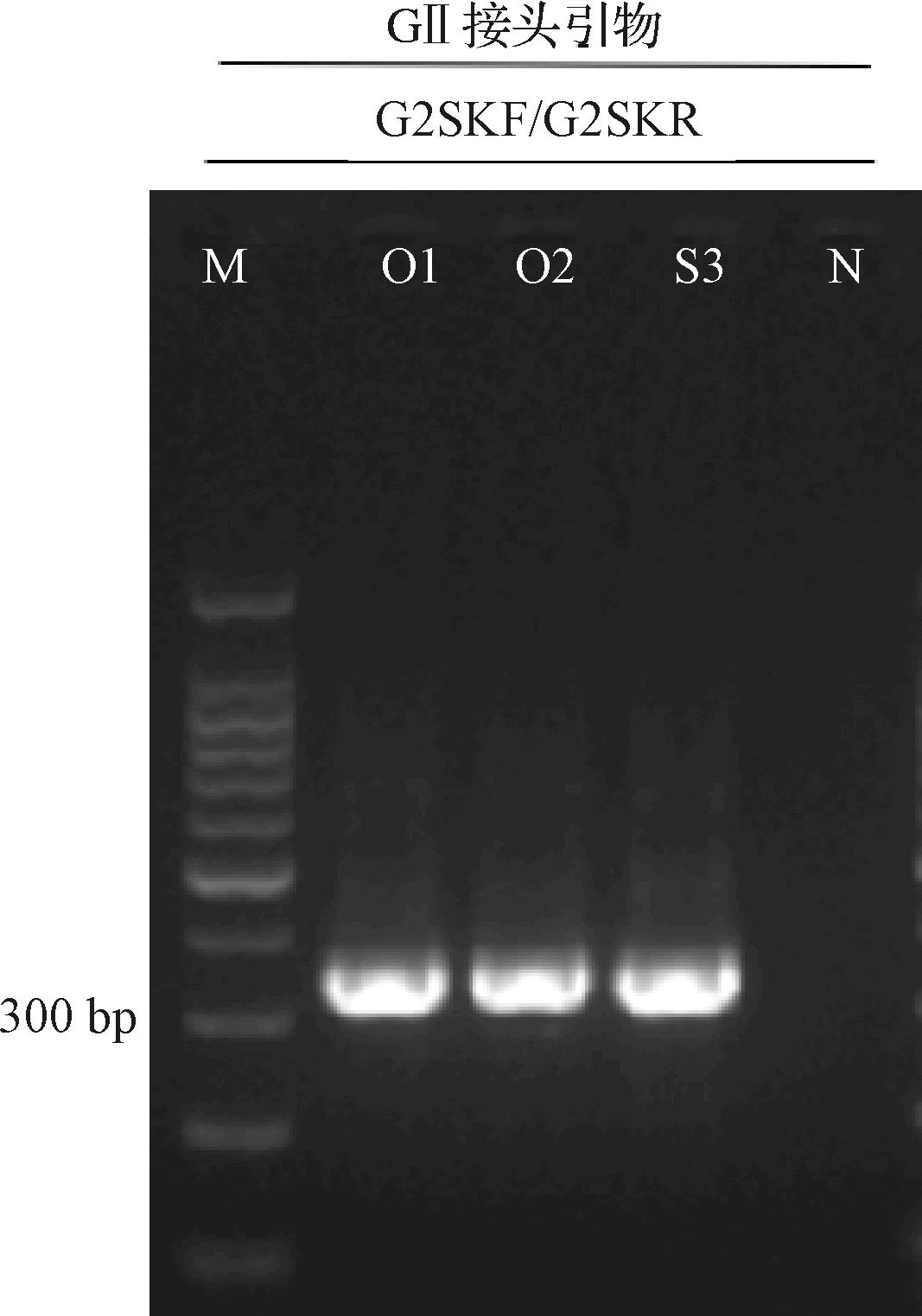
图3 经典引物对阳性样品中诺如病毒扩增结果
Fig.3 Norovirus amplification results in a positive sample with classical primers
注:O1、O2:人为GⅡ诺如病毒污染的牡蛎样品;S3:混合基因型粪便样品;M:100 bp DNA Marker;N:阴性对照。
如表5和图4所示,各基因型的序列丰度整体上与病毒浓度成正相关,经典引物(G2SKF/G2SKR)适合用于扩增子测序探究中诺如病毒的多样性,能够检测出人为添加到牡蛎中的6种GⅡ型诺如病毒(GⅡ.12[P33]、GⅡ.13[P13]、GⅡ.3[P12]、GⅡ.4_Den_Haag_2006b[P4]、GⅡ.17[P17]和GⅡ.2[P16]),覆盖度达到100%。
表5 各组扩增子测序得到的序列丰度
Table 5 Sequence abundance obtained by amplicon sequencing in each group
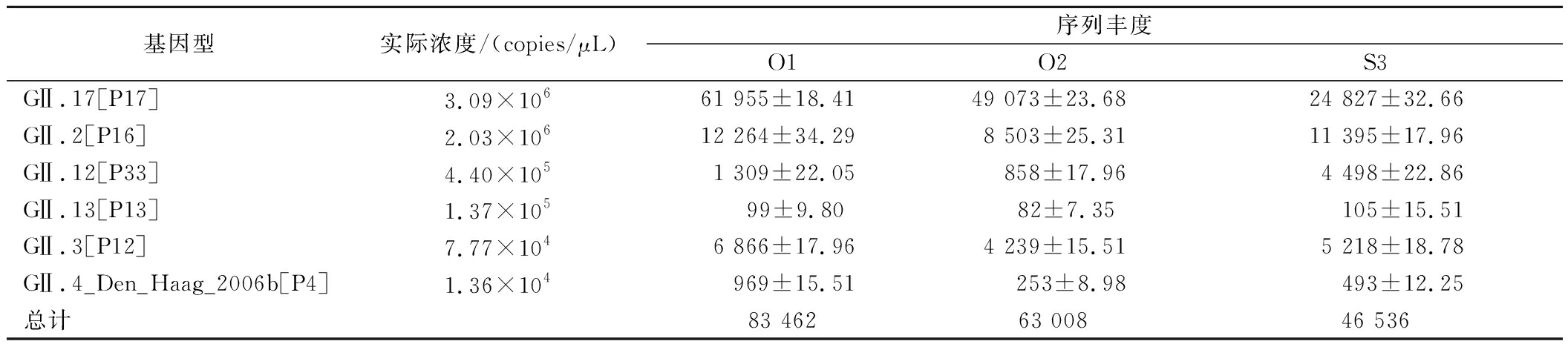
基因型实际浓度/(copies/μL)序列丰度O1O2S3GⅡ.17[P17]3.09×10661 955±18.4149 073±23.6824 827±32.66GⅡ.2[P16]2.03×10612 264±34.298 503±25.3111 395±17.96GⅡ.12[P33]4.40×1051 309±22.05858±17.964 498±22.86GⅡ.13[P13]1.37×10599±9.80 82±7.35 105±15.51GⅡ.3[P12]7.77×1046 866±17.964 239±15.515 218±18.78GⅡ.4_Den_Haag_2006b[P4]1.36×104969±15.51253±8.98493±12.25总计83 46263 00846 536
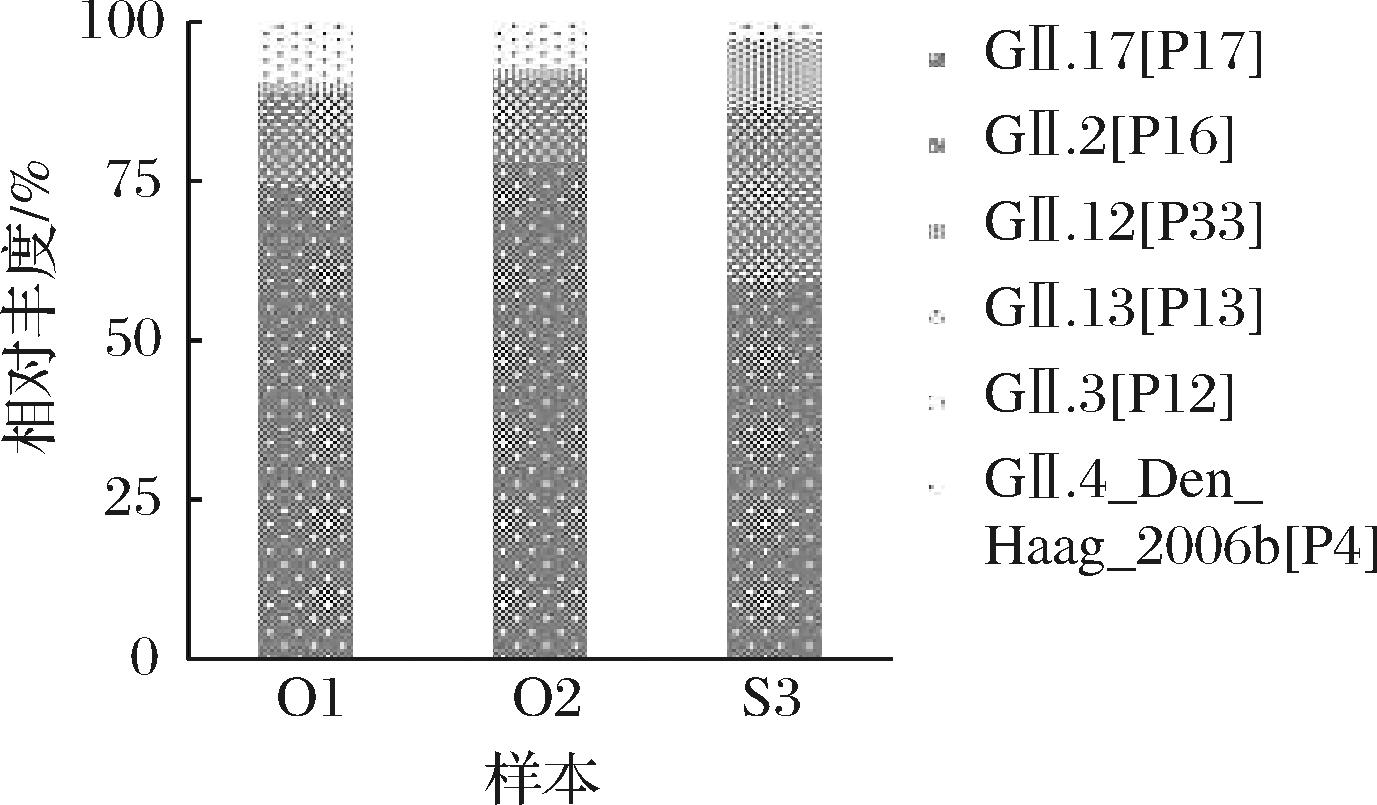
图4 扩增子测序的各基因型相对序列丰度对比图
Fig.4 Comparison of relative sequence abundance of each genotype by amplicon sequencing
2.3 灵敏度分析
采用选择的经典引物(G2SKF/G2SKR)对人为GⅡ诺如病毒污染的牡蛎样品进行扩增子测序的灵敏性评估,如图5所示,6份阳性样品中,除D和E号样品外,其余4份样品均在胶图上期望大小处呈现单一且明亮的条带。
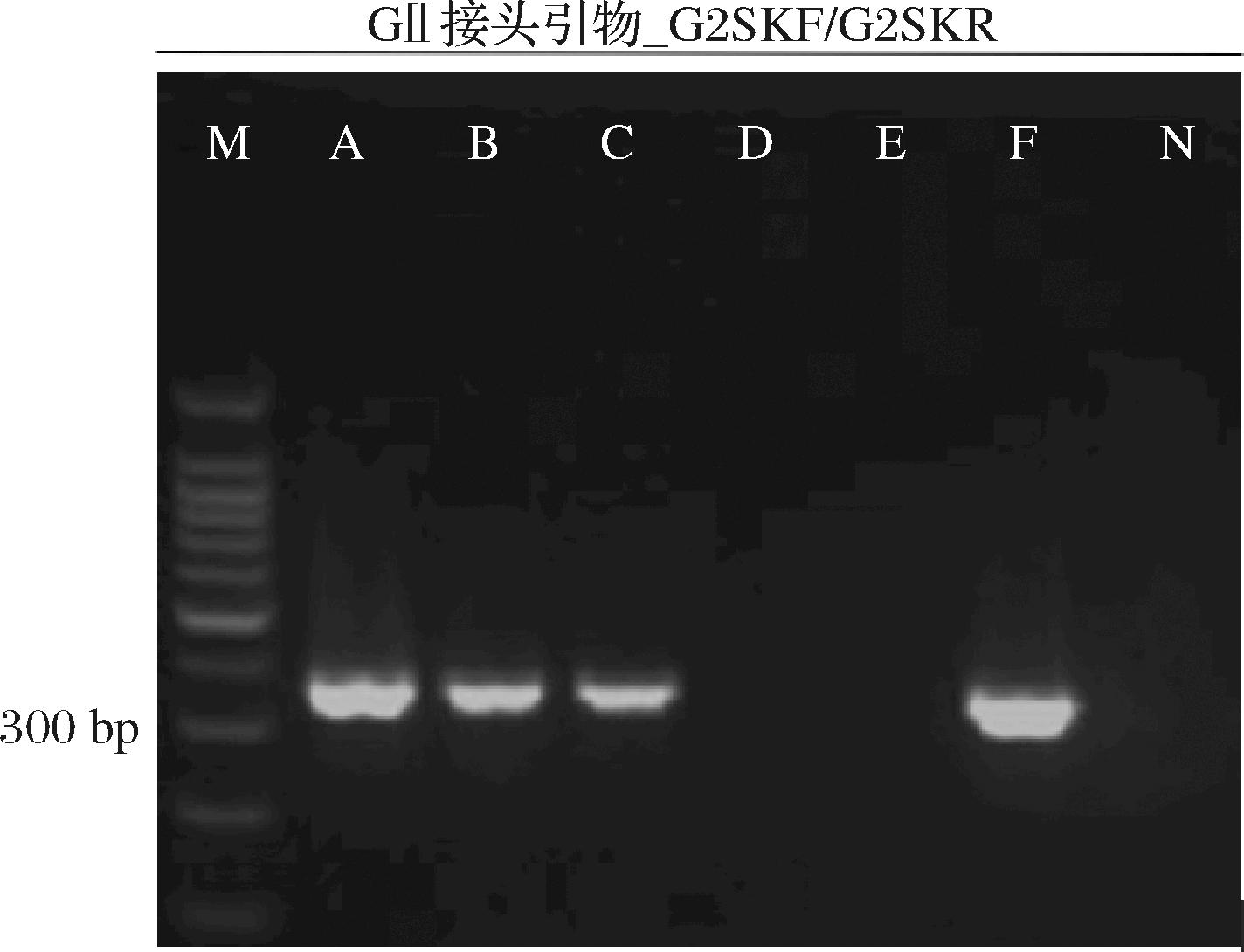
图5 浓度不同的诺如病毒阳性样品PCR产物浓度验证
Fig.5 Verification of PCR product concentration of positive norovirus samples with different concentrations
注:其总病毒粒子数为A:4.87×105、B:4.87×104、C:4.87×103、D:4.87×102、E:4.87×10个病毒、F:4.87×106个病毒,为混合基因型粪便样品;M:100 bp DNA Marker;N:阴性对照。
为了探究扩增子测序的灵敏度,决定将6份阳性样品的PCR产物进行扩增子测序,结果发现6份样品中均存在诺如病毒序列,D和E号样品的诺如病毒序列较少,牡蛎本身的序列丰度增加,这可能是因为病毒浓度相对较低。如图6和表6所示,表明虽然诺如病毒浓度较低,牡蛎相关序列越多,非特异性扩增越明显,但不会影响扩增子测序对牡蛎中诺如病毒多样性的研究,而且相比传统克隆测序技术,其灵敏度较高,低于50个病毒粒子也可以检测出,大大提高了诺如病毒检测的准确性。

a-为人为添加的6种基因型(GⅡ.12[P33]、GⅡ.13[P13]、GⅡ.3[P12]、GⅡ.4_Den_Haag_2006b[P4]、GⅡ.17[P17]和GⅡ.2[P16])以及牡蛎序列丰度占比结果;b-为GⅡ.4_Den_Haag_2006b[P4]、GⅡ.3[P12]、GⅡ.2[P16]、GⅡ.13[P13]、GⅡ.12[P33]以及牡蛎序列丰度占比结果
图6 扩增子测序的6组样品各基因型相对序列丰度对比图
Fig.6 Comparison of relative sequence abundance of each genotype in six groups of samples sequencing by amplicon sequencing
注:“oyster”表示牡蛎相关序列。
表6 六份样品扩增子测序得到的序列丰度
Table 6 Sequence abundance obtained by amplicon sequencing of six samples
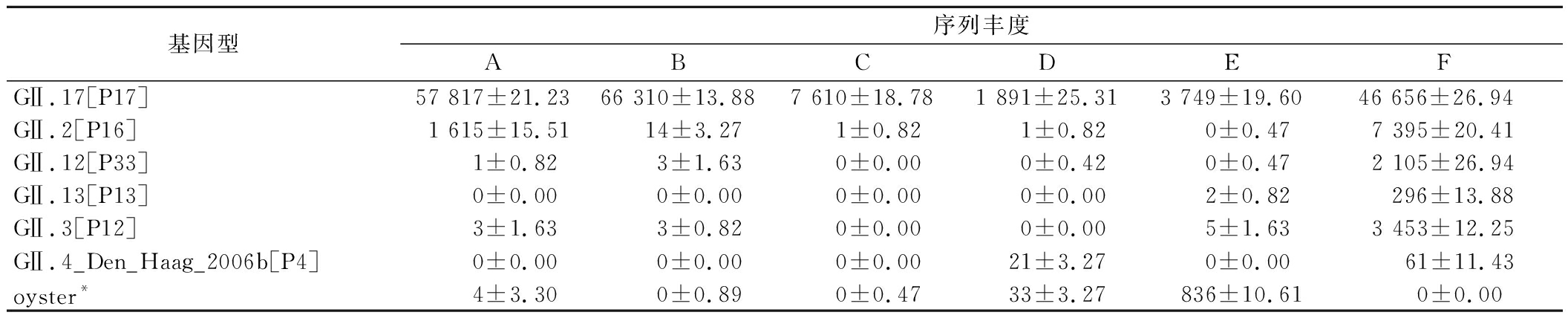
基因型序列丰度ABCDEFGⅡ.17[P17]57 817±21.2366 310±13.887 610±18.781 891±25.313 749±19.6046 656±26.94 GⅡ.2[P16] 1 615±15.51 14±3.27 1±0.82 1±0.82 0±0.477 395±20.41GⅡ.12[P33] 1±0.82 3±1.63 0±0.00 0±0.42 0±0.472 105±26.94GⅡ.13[P13] 0±0.00 0±0.00 0±0.00 0±0.00 2±0.82 296±13.88GⅡ.3[P12] 3±1.63 3±0.82 0±0.00 0±0.00 5±1.633 453±12.25GⅡ.4_Den_Haag_2006b[P4] 0±0.00 0±0.00 0±0.00 21±3.27 0±0.00 61±11.43oyster* 4±3.30 0±0.89 0±0.47 33±3.27 836±10.61 0±0.00
注:表中“oyster*”表示牡蛎相关的基因序列。
3 讨论与结论
如今,PCR技术已经基本满足研究者对牡蛎中诺如病毒检测的要求[15],但在本小组前期对牡蛎中诺如病毒检测筛查中发现,利用巢式PCR方法对贝类中诺如病毒进行检测时会因为样品本底背景的影响出现许多非特异性扩增,导致在胶图上的期望大小处出现非特异性条带影响对结果的判断[5-6]。同时贝类可能会富集周围环境多种诺如病毒基因型,PCR产物中可能会因此存在多种基因型的扩增产物,若将其直接送至测序公司进行Sanger测序,会因为测序结果出现双峰导致测序失败或低估样品中诺如病毒的基因型多样性[16-17]。若想要解决此类问题,通常会进行后续的割胶回收、PCR产物纯化、TA链接、质粒转化、蓝白斑筛选、克隆测序等繁琐的实验,耗费大量的人力物力。为了更准确更简便的探究诺如病毒多样性,通过扩增子测序的方法探究样品中诺如病毒多样性是大势所趋[18]。
本文采用了今年来比较流行且牡蛎中检出最多的6种GⅡ型诺如病毒(GⅡ.12[P33]、GⅡ.13[P13]、GⅡ.3[P12]、GⅡ.4_Den_Haag_2006b[P4]、GⅡ.17[P17]和GⅡ.2[P16])阳性的粪便样品人为添加至阴性牡蛎消化腺,用于扩增子测序引物的选择和灵敏性的评估,较真实地反映了牡蛎中诺如病毒的检测[19-20]。结果表明,经典引物较适合扩增子测序的研究且灵敏性较高;另外,在扩增子测序得到的序列中,有少数序列为牡蛎及其肠道微生物的基因组序列,进一步说明了牡蛎本低背景对诺如病毒检测时的影响,结果也显示诺如病毒的浓度越低出现的非特异性扩增越多,但是扩增子测序可以通过序列聚类的方法排除牡蛎以及其肠道微生物基因组对检测过程中的影响[21]。在实验的过程中,有意思的是S3和F的确是相同类型的样品(含有相同的病毒基因型和病毒颗粒数量,仅为不同实验过程中制备的批次不同),它们使用同一对引物检测的序列丰度存在一定的差异,推测是样品在制备、核酸提取、PCR扩增或测序等过程中误差所致。不过,本研究重点聚焦的是探索牡蛎样本中不同基因型病毒的序列比重以及相对丰度,后续研究将继续提高方法的稳定性和重复性。另外,本研究发现病毒浓度越高的基因型得到的序列丰度越高。在对其灵敏度评估的过程中发现病毒浓度越高引物对其扩增的偏好性越大,这是因为PCR是具有偏好性的,在设计引物的过程中,GⅡ.17的序列占比较多,设计出的引物与GⅡ.17的相似性更高一些,所以导致扩增子测序的结果对GⅡ.17具有偏好性,其序列丰度比较高[22-23]。对于人为诺如病毒污染的牡蛎样品中的6种诺如病毒阳性基因型没有控制到相同的浓度,可能会导致浓度较低的基因型序列丰度极低,所以还需要后续优化[3]。综上,本研究证明了通过扩增子测序探究牡蛎中诺如病毒多样性的灵敏性较高,且其可以通过序列聚类的方法排除非特异性扩增的影响,能够更加准确地探究牡蛎中诺如病毒的污染状况以及基因型多样性[24]。
本研究通过人为诺如病毒污染的牡蛎样品选取经典引物(G2SKF/G2SKR)进行扩增子测序覆盖度的评估,结果表明,该引物能检测出人为添加的6种基因型,覆盖度100%;同时利用不同诺如病毒数量的阳性牡蛎样品评估了扩增子测序的灵敏度,结果显示,其灵敏度较高,低于50个病毒粒子也可以检测出。在病毒浓度较低时,避免了巢式 RT-PCR方法在检测牡蛎中诺如病毒时出现的非特异性扩增及假阴性等情况,提高了牡蛎中诺如病毒检测的准确性。
[1] 廖巧红, 冉陆, 靳淼, 等.诺如病毒感染暴发调查和预防控制技术指南(2015版)[J].中国病毒病杂志, 2015, 5(6):448-458.LIAO Q H, RAN L, JIN M, et al.Guidelines on outbreak investigation, prevention and control of norovirus infection (2015)[J].Chinese Journal of Preventive Medicine, 2015, 5(6):448-458.
[2] CHHABRA P, DE GRAAF M, PARRA G I, et al.Updated classification of norovirus genogroups and genotypes[J].The Journal of General Virology, 2019, 100(10):1393-1406.
[3] YU Y X, CAI H, HU L H, et al.Molecular epidemiology of oyster-related human noroviruses and their global genetic diversity and temporal-geographical distribution from 1983 to 2014[J].Applied and Environmental Microbiology, 2015, 81(21):7615-7624.
[4] 张萌, 龙逸芳, 孙立梅.2013—2017年三种基因型诺如病毒感染暴发疫情的流行病学特征分析[C]//2018新发传染病研究热点研讨会,2018.ZHANG M, LONG Y F, SUN L M.Epidemiological characteristics of three genotypes of norovirus infection outbreaks from 2013 to 2017[C]//2018 Symposium on Research Hotspots in Emerging Infectious Diseases, 2018.
[5] 贾添慧, 董蕾, 王永杰, 等.牡蛎中GⅡ型诺如病毒巢式RT-PCR检测方法的优化与评价[J].上海海洋大学学报, 2021, 30(2):239-246.JIA T H, DONG L, WANG Y J, et al.Updating and evaluation of nested RT-PCR for specific detection of genogroup Ⅱ norovirus in oysters[J].Journal of Shanghai Ocean University, 2021, 30(2):239-246.
[6] GUO P, YU Y X, PAN Y J, et al.Design and evaluation of nested PCR primers for specific detection of genogroup I noroviruses in oysters[J].Molecular and Cellular Probes, 2018, 40:40-43.
[7] RANJAN R,RANI A, METWALLY A, et al.Analysis of the microbiome:Advantages of whole genome shotgun versus 16S amplicon sequencing[J].Biochemical and Biophysical Research Communications, 2016, 469(4):967-977.
[8] LI D D, ZHANG J W, LI J M.Primer design for quantitative real-time PCR for the emerging Coronavirus SARS-CoV-2[J].Theranostics, 2020, 10(16):7150-7162.
[9] 贾添慧, 王永杰, 杨明树, 等.上海市售牡蛎中诺如病毒的污染及基因型和流行趋势分析[J].生物学杂志, 2021, 38(4):38-42.JIA T H, WANG Y J, YANG M S, et al.Contamination, genotype and epidemiological trends of norovirus in oysters collected from the market in Shanghai[J].Journal of Biology, 2021, 38(4):38-42.
[10] BHADRA S, MARANHAO A C, PAIK I, et al.One-enzyme reverse transcription qPCR using Taq DNA polymerase[J].Biochemistry, 2020, 59(49):4638-4645.
[11] JOTHIKUMAR N, LOWTHER J A, HENSHILWOOD K, et al.Rapid and sensitive detection of noroviruses by using TaqMan-based one-step reverse transcription-PCR assays and application to naturally contaminated shellfish samples[J].Applied and Environmental Microbiology, 2005, 71(4):1870-1875.
[12] KAGEYAMA T, KOJIMA S, SHINOHARA M, et al.Broadly reactive and highly sensitive assay for Norwalk-like viruses based on real-time quantitative reverse transcription-PCR[J].Journal of Clinical Microbiology, 2003, 41(4):1548-1557.
[13] KRONEMAN A, VENNEMA H, DEFORCHE K, et al.An automated genotyping tool for enteroviruses and noroviruses[J].Journal of Clinical Virology, 2011, 51(2):121-125.
[14] 程弘正, 杨文华.R语言程序包依赖关系与更新情况的实证研究[J].计算机科学,2024(6):1-11.CHENG H Z, YANG W H.An empirical study on the dependency and update of R language package[J].Computer Science, 2024(6):1-11.
[15] KOJIMA S, KAGEYAMA T, FUKUSHI S, et al.Genogroup-specific PCR primers for detection of Norwalk-like viruses[J].J Virol Methods, 2002, 100(1-2):107-114.
[16] SCHWAB K J, NEILL F H, FANKHAUSER R L, et al.Development of methods to detect “Norwalk-like viruses” (NLVs) and hepatitis A virus in delicatessen foods:Application to a food-borne NLV outbreak[J].Applied and Environmental Microbiology, 2000, 66(1):213-218.
[17] NISHIDA T, KIMURA H, SAITOH M, et al.Detection, quantitation, and phylogenetic analysis of noroviruses in Japanese oysters[J].Applied and Environmental Microbiology, 2003, 69(10):5782-5786.
[18] MACKAY I M, ARDEN K E, NITSCHE A.Real-time PCR in virology[J].Nucleic Acids Research,2002, 30(6):1292-1305.
[19] MOHAMMADI M M, BAVI O.DNA sequencing:An overview of solid-state and biological nanopore-based methods[J].Biophysical Reviews,2021, 14(1):99-110.
[20] 王大鹏, 史贤明.贝类中致病微生物的检测技术及其组织分布[J].微生物学报, 2011, 51(10):1304-1309.WANG D P, SHI X M.Distribution and detection of pathogens in shellfish-A review[J].Acta Microbiologica Sinica, 2011, 51(10):1304-1309.
[21] BHAT A H, PRABHU P, BALAKRISHNAN K.A critical analysis of state-of-the-art metagenomics OTU clustering algorithms[J].Journal of Biosciences, 2019, 44(6):148.
[22] HU T S, CHITNIS N, MONOS D, et al.Next-generation sequencing technologies:An overview[J].Human Immunology, 2021, 82(11):801-811.
[23] GAZULLA C R, CABELLO A M, S NCHEZ P, et al.A metagenomic and amplicon sequencing combined approach reveals the best primers to study marine aerobic anoxygenic phototrophs[J].Microbial Ecology, 2023, 86(3):2161-2172.
NCHEZ P, et al.A metagenomic and amplicon sequencing combined approach reveals the best primers to study marine aerobic anoxygenic phototrophs[J].Microbial Ecology, 2023, 86(3):2161-2172.
[24] BOONCHAN M, MOTOMURA K, INOUE K, et al.Distribution of norovirus genotypes and subtypes in river water by ultra-deep sequencing-based analysis[J].Letters in Applied Microbiology, 2017, 65(1):98-104.