木聚糖是一种重要的半纤维素,广泛存在于自然界中。木聚糖及其寡糖具有抗肿瘤[1]、免疫调节[2]、肠道调节[3]、抗氧化[4]、抗病毒[5]等多种生理功能。木聚糖在木糖醇、低聚木糖生产[6]、生物材料制备[7-8]、药物递送[9]等方面均有良好应用。其中,β-1,3-木聚糖是由D-木糖单元以β-1,3糖苷键连接而成的多糖。β-1,3-木聚糖是紫菜等海藻中的重要结构多糖[10],对细胞壁的机械性能、细胞壁的生长调节等具有重要作用。
多糖结合蛋白是多糖研究的关键工具,常被用作探针服务于多糖的原位分析[11]、特异性检测[12]、高通量分析[13]等场景。碳水化合物结合结构域(carbohydrate-binding module,CBM)是一类重要的多糖结合蛋白[14],其被定义为碳水化合物活性酶中具有碳水化合物结合能力的连续氨基酸序列。截止到2023年9月,基于氨基酸序列的相似性,CAZy数据库中(www.cazy.org/)记载有98个CBM家族。通过实验证实的能够结合β-1,3-木聚糖的CBM仅有4个[15-17],同时它们在β-1,3-木聚糖研究中的应用潜力尚未被阐明。
本研究旨在挖掘并表征一个具有β-1,3-木聚糖结合能力的CBM,为β-1,3-木聚糖研究提供良好工具。据报道,糖苷水解酶GH26家族成员展示出β-1,3-木聚糖的水解活性[18]。前期从GH26家族中挖掘得到一条潜在的β-1,3-木聚糖酶基因序列(CP047475.1),将其命名为VaXyl。dbCAN注释结果显示(图1-a),该序列由一个GH26家族结构域和一个CBM6家族结构域组成。AlphaFold2结构预测结果表明,该CBM6家族序列的三维结构为β-sandwich折叠(图1-b)。据悉,β-sandwich是CBM的主要折叠形式,同时CBM的结合能力通常与其附属酶的水解活性相匹配。因此猜测该CBM6家族序列具有β-1,3-木聚糖结合能力。本研究将该CBM6家族序列异源表达至大肠杆菌中,重组蛋白命名为VaCBM6,系统表征了该蛋白的结合能力。同时还证实了该蛋白在β-1,3-木聚糖原位分析中的应用潜力。
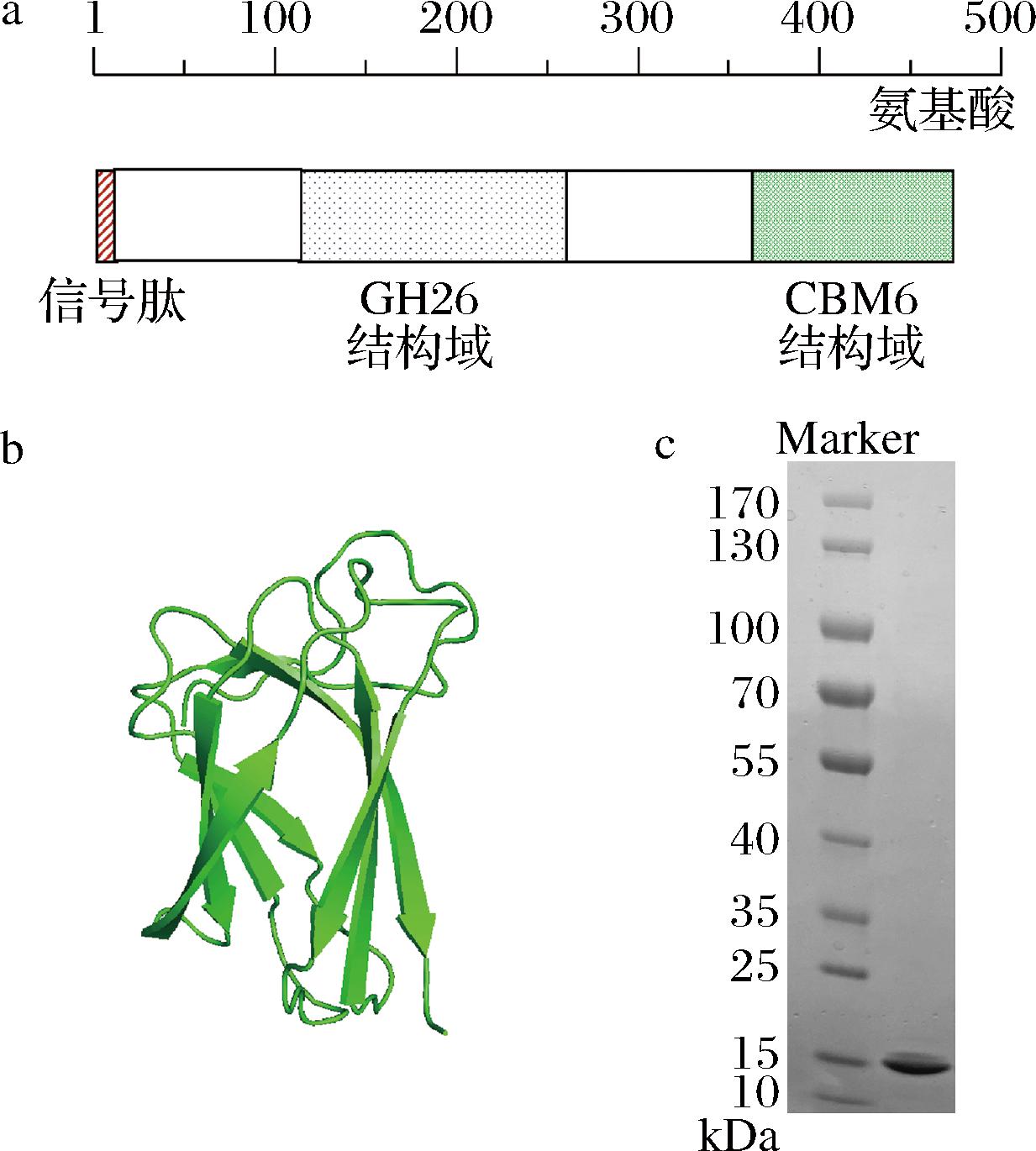
a-VaXyl结构域分布(GH26家族结构域,114~267 aa;CBM6家族结构域,370~477 aa);b-AlphaFold2对VaCBM6结构预测结果;c-纯化后VaCBM6的SDS-PAGE图
图1 VaCBM6的生物信息学分析及纯化
Fig.1 Bioinformatics analysis and purification of VaCBM6
1 材料与方法
1.1 多糖与仪器
多糖:市售干紫菜,参照IRIKI等[10]的方法提取紫菜中的β-1,3-木聚糖和β-1,4-甘露聚糖。干紫菜粉碎后过筛,经热水浸提去除紫菜多糖,次氯酸钠浸提脱色去除杂质,离心后弃去上清液。取沉淀加入高浓度NaOH溶液浸提4 h,高速离心分离浸提上清液与沉淀,沉淀经乙酸中和、超纯水洗涤后获得β-1,3-木聚糖;上清液经乙醇过夜沉淀获得β-1,4-甘露聚糖。紫菜多糖、魔芋葡甘露聚糖均由实验室自制;β-1,4-木聚糖(山毛榉木),Sigma公司;β-1,4-木聚糖(玉米芯)、羧甲基纤维素、琼脂糖,北京索莱宝公司;羟乙基纤维素,上海源叶公司;可得然胶、瓜尔豆胶,上海麦克林公司;甲壳素,上海蓝季公司;微晶纤维素,北京启研公司。
仪器:JY92-IIN超声波细胞粉碎机,宁波新芝公司;3K15高速离心机,美国Sigma公司; KTA Prime Plus蛋白纯化系统,美国GE Healthcare Life Sciences公司;Leica CM1860冷冻切片机,上海徕卡公司;NIKON/Ni-E电动荧光显微镜,日本尼康公司。
KTA Prime Plus蛋白纯化系统,美国GE Healthcare Life Sciences公司;Leica CM1860冷冻切片机,上海徕卡公司;NIKON/Ni-E电动荧光显微镜,日本尼康公司。
1.2 生物信息学分析
利用dbCAN软件预测蛋白结构域组成[19];利用AlphaFold2预测蛋白三级结构[20];使用ExPASy计算重组蛋白的理论分子质量与等电点[21]。
1.3 蛋白克隆表达
目的基因由上海生工公司合成,利用PCR技术对目的基因进行扩增。上游引物为5′-AGGAGATATACCATGGGCATTCCTGATGAGAAGCAATCCC-3′,下游引物为5′-GGTGGTGGTGCTCGAGGCGACGATCAAAGA-CCAGATAATCTATGT-3′。利用限制性内切酶NcoI/XhoI酶切并将目的基因连接至pET-28a(+)载体(保留C端His标签)。利用热击法将重组载体转化至大肠杆菌BL21(DE3)感受态细胞中。将成功转化的大肠杆菌接种至含有30 ng/μL卡那霉素的LB液体培养基中,37 ℃、180 r/min过夜培养。传代后培养至OD600约为0.4时加入异丙基β-D-硫代半乳糖苷(isopropyl β-D-1-thiogalactopyranoside,IPTG,终浓度为0.5 mmol/L),17 ℃、180 r/min诱导培养12 h。离心收集培养后的菌体,使用20 mmol/L、pH 7.5的Na2HPO4-NaH2PO4缓冲液重悬。超声破碎后4 ℃离心取上清液,制得VaCBM6粗蛋白。
1.4 VaCBM6的分离纯化及纯度分析
利用HisTrapTM HP色谱柱和 KTA Prime Plus系统对VaCBM6粗蛋白进行亲和层析纯化,以20 mmol/L,pH 7.5 Tris-HCl缓冲液(含0.3 mol/L NaCl溶液)为流动相进行平衡,使用0~0.5 mol/L咪唑进行梯度洗脱,收集具有高紫外信号的各洗脱管。合并后用HiPrepTM 26/10 Desalting色谱柱脱盐,流动相为20 mmol/L,pH 7.5的Na2HPO4-NaH2PO4缓冲液。利用SDS-PAGE分析蛋白纯度及其分子质量,使用纯化后蛋白进行蛋白结合能力分析。
KTA Prime Plus系统对VaCBM6粗蛋白进行亲和层析纯化,以20 mmol/L,pH 7.5 Tris-HCl缓冲液(含0.3 mol/L NaCl溶液)为流动相进行平衡,使用0~0.5 mol/L咪唑进行梯度洗脱,收集具有高紫外信号的各洗脱管。合并后用HiPrepTM 26/10 Desalting色谱柱脱盐,流动相为20 mmol/L,pH 7.5的Na2HPO4-NaH2PO4缓冲液。利用SDS-PAGE分析蛋白纯度及其分子质量,使用纯化后蛋白进行蛋白结合能力分析。
1.5 不溶性多糖结合能力分析
利用吸附法定性分析[22]VaCBM6与不溶性多糖的结合能力,所用不溶性多糖分别为β-1,3-木聚糖、β-1,4-甘露聚糖、β-1,4-木聚糖(山毛榉木)、微晶纤维素、可得然胶、甲壳素、羧甲基纤维素。具体实验方案如下,将纯化后VaCBM6添加到含有各组不溶性多糖(2 mg)的Na2HPO4-NaH2PO4(20 mmol/L,pH 7.5)缓冲液中,VaCBM6终质量浓度设为50 μg/mL,总体系为0.5 mL。置于25 ℃下振荡孵育3 h,离心10 min(13 000 r/min)后分离上清液与沉淀,沉淀使用Na2HPO4-NaH2PO4(20 mmol/L,pH 7.5)缓冲液洗涤2次。利用SDS-PAGE呈现分析上清液与沉淀中存在的蛋白,以同浓度的无底物组VaCBM6蛋白条带为对照,通过比较不同底物实验组的沉淀泳道是否存在VaCBM6条带,以及上清液泳道中VaCBM6条带浓度变化,定性判断VaCBM6与不溶性多糖的结合能力。
同时,使用紫外吸收法(280 nm)测定无底物对照组与各实验组上清蛋白浓度,以无底物对照组的吸光值为标准进行归一化处理,根据各实验组上清液蛋白浓度变化情况,判断VaCBM6与不溶性多糖的结合能力,验证蛋白结合情况。
1.6 可溶性多糖结合能力分析
利用碳水化合物微阵列和微量滴定板法考察VaCBM6对可溶性多糖的结合能力。所用可溶性多糖分别为紫菜多糖、琼脂糖、β-1,4-木聚糖(玉米芯)、瓜尔豆胶、魔芋葡甘露聚糖、羟乙基纤维素。实验使用缓冲液为PBS(140 mmol/L NaCl溶液,2.7 mmol/L KCl溶液,10 mmol/L Na2HPO4溶液,1.7 mmol/L KH2PO4溶液,pH 7.5)。
碳水化合物微阵列参照FICKO-BLEAN等[23]的方案并稍作修改:取2 μL多糖样品(2 mg/mL)滴加至硝酸纤维素膜上,室温静置干燥30 min;使用含有50 g/L脱脂奶粉的PBS(MPBS,pH 7.5)封闭1 h;然后在硝酸纤维素膜上滴加1 mL VaCBM6(MPBS稀释,终质量浓度为20 μg/mL),孵育2 h后使用超纯水反复清洗硝酸纤维素膜,以去除非特异性结合;滴加连有辣根过氧化物酶(horse radish peroxidase,HRP)的结合6×His标签的单克隆抗体(MPBS稀释,1∶5 000),室温反应2 h;最后加入ECL显色液反应2 min,利用化学发光仪进行检测。
微量滴定板测定参照MCCARTNEY等[24]的方案并稍作修改:分别取100 μL多糖样品(50 μg/mL)滴加在微量滴定板上,4 ℃过夜孵育;超纯水清洗微量滴定板,而后加入200 μL MPBS封闭1 h;清洗后加入200 μL VaCBM6(MPBS稀释,终质量浓度为20 μg/mL)于室温孵育2 h;清洗以去除非特异性结合,随后加入连有HRP的结合6×His标签的单克隆抗体(MPBS稀释,1∶5 000),室温反应2 h;超纯水清洗后加入四甲基联苯胺溶液避光孵育15 min,然后加入1 mol/L HCl溶液终止反应,测定样品在450 nm处的吸光值。
1.7 结合能力定量分析
采用吸附等温线法[25]对VaCBM6与β-1,3-木聚糖的结合能力进行定量分析。具体方案如下,将不同量的VaCBM6添加到含有β-1,3-木聚糖(2 mg)的Na2HPO4-NaH2PO4(20 mmol/L,pH 7.5)缓冲液中,设定VaCBM6终浓度梯度为0~45 μmol/L,总体系为0.5 mL。置于25 ℃下振荡孵育3 h,高速离心分离上清液与沉淀。利用Bradford蛋白浓度测定试剂盒测定上清液中游离蛋白浓度([P],μmol/L),结合蛋白浓度([PC],μmol/g)由原蛋白浓度与上清液中游离蛋白浓度计算得出。使用Origin软件,分别以[P]和[PC]为横纵坐标数据拟合Langmuir吸附等温线,获得相应的结合参数。具体计算如公式(1)所示:
[PC]=[P]×[PC]max×Ka/(Ka×[P]+1)
(1)
计算获得[PC]max和Ka两个常数,其中[PC]max代表β-1,3-木聚糖的最大结合量;Ka是结合常数,Ka值越大代表蛋白与底物结合能力越强。
1.8 荧光探针构建
利用PCR技术对目的基因进行扩增,上下游引物分别为:5′-GGATCGATGGGGATCCATCCCGGATGAAAAACAG-3′和5′-CCATGGTGGCGAATTCACGA-CGATCGAAAACCAGG-3′。重组载体构建与转化方法参照1.3节,选用载体为pRSET EmGFP,限制性内切酶为BamHⅠ/EcoRⅠ,所用宿主为大肠杆菌BL21(DE3)。将成功转化的大肠杆菌接种至含有60 ng/μL氨苄霉素的LB液体培养基培养,蛋白表达与纯化方案参照1.3节及1.4节。将荧光探针命名为EmGFP-VaCBM6,使用荧光分光光度计对EmGFP-VaCBM6进行全波长扫描,测定其最佳激发波长(Ex)和最佳发射波长(Em)。
1.9 紫菜中β-1,3-木聚糖的原位可视化观察
以干紫菜为原料,经超纯水复水后剪取少量紫菜组织,展平后包埋于OCT包埋剂中,置于冷冻切片机中快速冷冻后切片,切片厚度为20 μm。取出紫菜组织切片置于避光染色盒中,加入100 μL纯化后EmGFP-VaCBM6(200 mg/mL)孵育30 min,吸弃染色液后加入100 μL Na2HPO4-NaH2PO4(20 mmol/L,pH 7.5)缓冲液洗涤10 min,吸弃洗涤液后滴加少量人工海水封片观察。利用电动荧光显微镜(NIKON/Ni-E)收集图像,通过NIS-Elements Viewer分析图像。
2 结果与分析
2.1 重组蛋白克隆表达与纯化
VaCBM6在大肠杆菌BL21(DE3)中实现了异源表达。超声破碎制备VaCBM6粗蛋白上清液,于镍柱纯化及凝胶色谱柱脱盐后获得纯蛋白。SDS-PAGE结果显示,纯化后VaCBM6呈现单一条带(图1-c),表明获得电泳纯蛋白。该单一条带的分子质量约为15 kDa,与预测的VaCBM6的分子质量(15.15 kDa)接近。上述结果证明了重组VaCBM6被成功克隆表达及纯化。
2.2 多糖结合能力验证
利用吸附法研究VaCBM6与不溶性多糖的结合能力(图2)。与无底物对照组相比,观察到VaCBM6与β-1,3-木聚糖孵育后的沉淀泳道存在蛋白条带,同时上清液泳道蛋白条带明显变浅,这表明VaCBM6能够结合β-1,3-木聚糖,这是首次证实CBM6家族成员具有β-1,3-木聚糖结合能力。同时VaCBM6与β-1,4-甘露聚糖、β-1,4-木聚糖(山毛榉木)、微晶纤维素、可得然胶、甲壳素、羧甲基纤维素孵育的沉淀泳道未观察到蛋白条带,上清液泳道中蛋白条带也未发生明显变化,表明VaCBM6与上述测试不溶性多糖无结合能力。
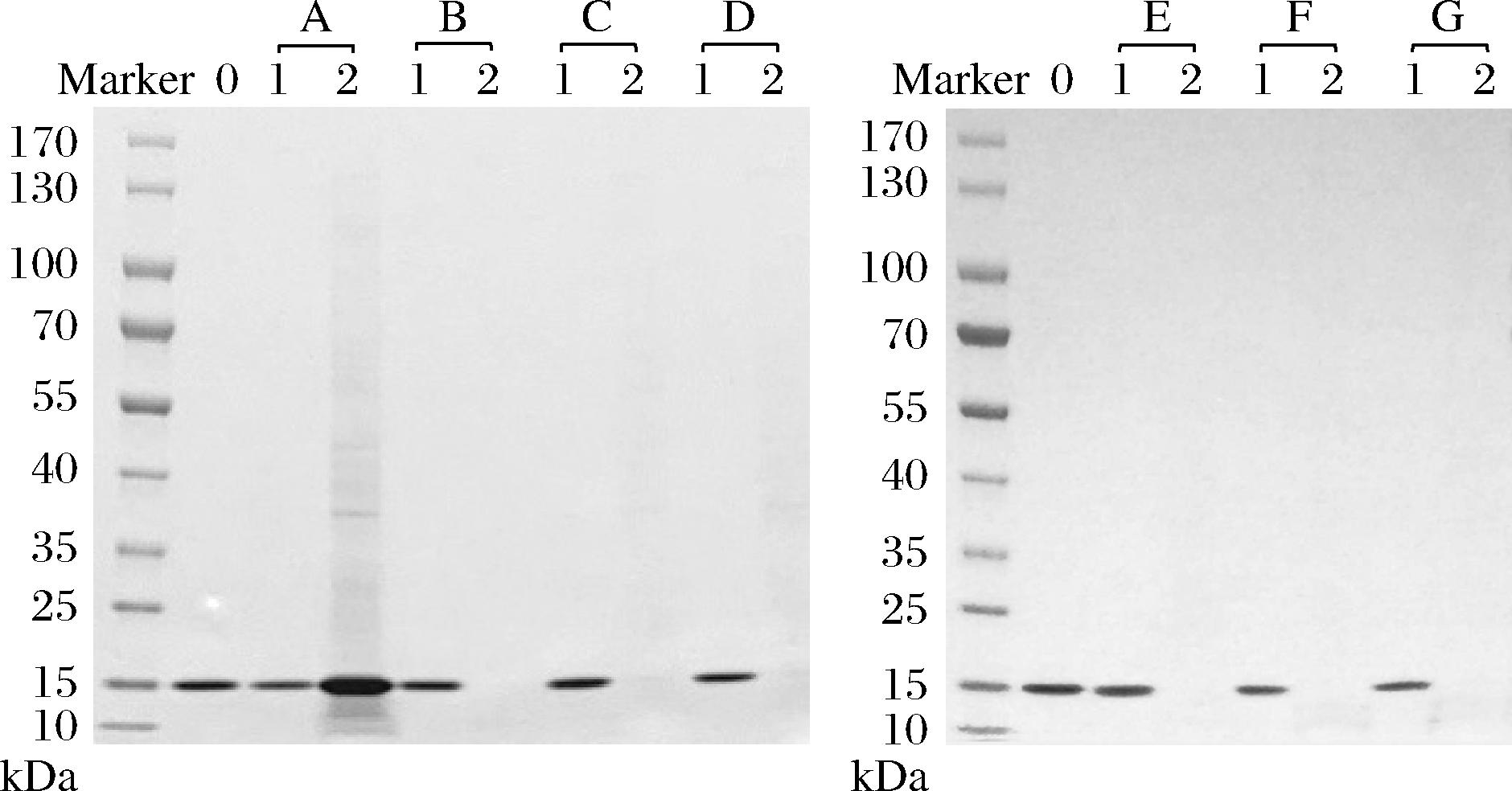
泳道0-纯化VaCBM6(对照);泳道1-上清液中蛋白;泳道2-沉淀中蛋白A-β-1,3-木聚糖;B-β-1,4-甘露聚糖;C-β-1,4-木聚糖;D-微晶纤维素;E-可得然胶;F-甲壳素;G-羧甲基纤维素
图2 VaCBM6对不溶性多糖结合能力分析
Fig.2 Binding ability of VaCBM6 to insoluble polysaccharides
为进一步证实该蛋白的结合特异性,利用紫外吸收法(280 nm)分析了VaCBM6与不溶性多糖的结合能力(图3)。相比于无底物的对照组,添加β-1,3-木聚糖的上清液中的蛋白浓度发生了显著下降,提示VaCBM6能够结合β-1,3-木聚糖;同时,与其他测试不溶性多糖的孵育上清液中的蛋白浓度未发生明显变化,表明VaCBM6对β-1,4-甘露聚糖、β-1,4-木聚糖(山毛榉木)、微晶纤维素、可得然胶、甲壳素、羧甲基纤维素均不具有结合能力。上述结果也与吸附法结果相一致。
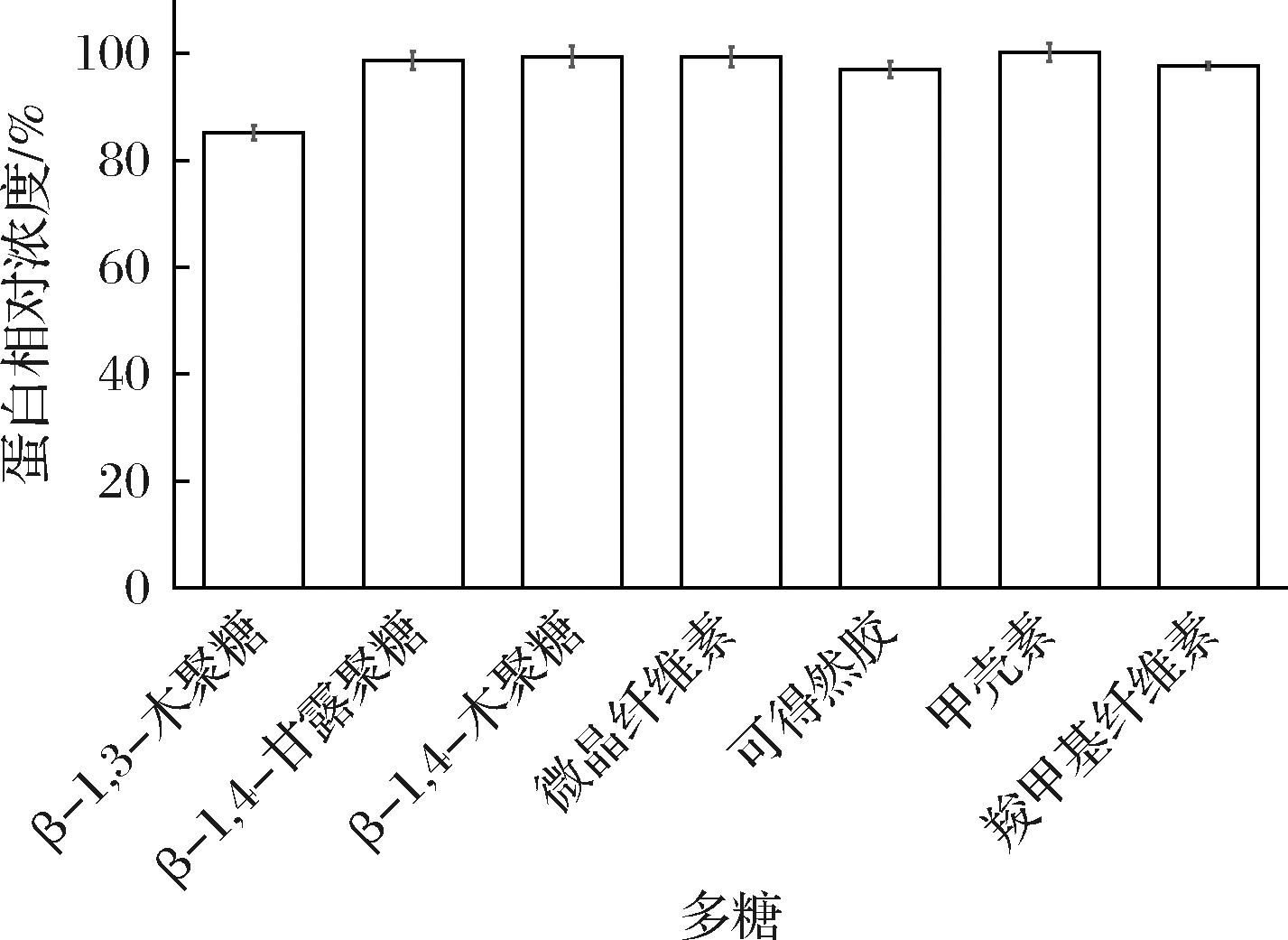
图3 孵育后各实验组上清液蛋白相对于无底物对照组蛋白浓度变化
Fig.3 Concentration of supernatant protein for each experimental group after incubation, in comparison to the control group without substrate
进一步的,利用碳水化合物微阵列和微量滴定板分析研究了VaCBM6对可溶性多糖的结合能力,2种方法一致表明VaCBM6对紫菜多糖、琼脂糖、β-1,4-木聚糖(玉米芯)、瓜尔豆胶、魔芋葡甘露聚糖、羟乙基纤维素均未表现出阳性结合信号(数据未展示),提示VaCBM6对上述测试多糖均不具有结合能力。综上,吸附法、紫外吸收法(280 nm)、碳水化合物微阵列及微量滴定板分析结果表明VaCBM6能够特异性结合β-1,3-木聚糖。
2.3 β-1,3-木聚糖结合能力定量分析
吸附等温线是定量分析不溶性多糖与CBM结合能力的常用方法。通过拟合Langmuir吸附等温线计算得出VaCBM6与β-1,3-木聚糖的结合常数Ka为3.0×105 (mol/L)-1,最大结合量[PC]max为4.44 μmol/g(图4)。典型的碳水化合物与结合蛋白相互作用的Ka值在103~106 (mol/L)-1,这提示VaCBM6对β-1,3-木聚糖具有较强的结合能力,便于后续的实际应用。
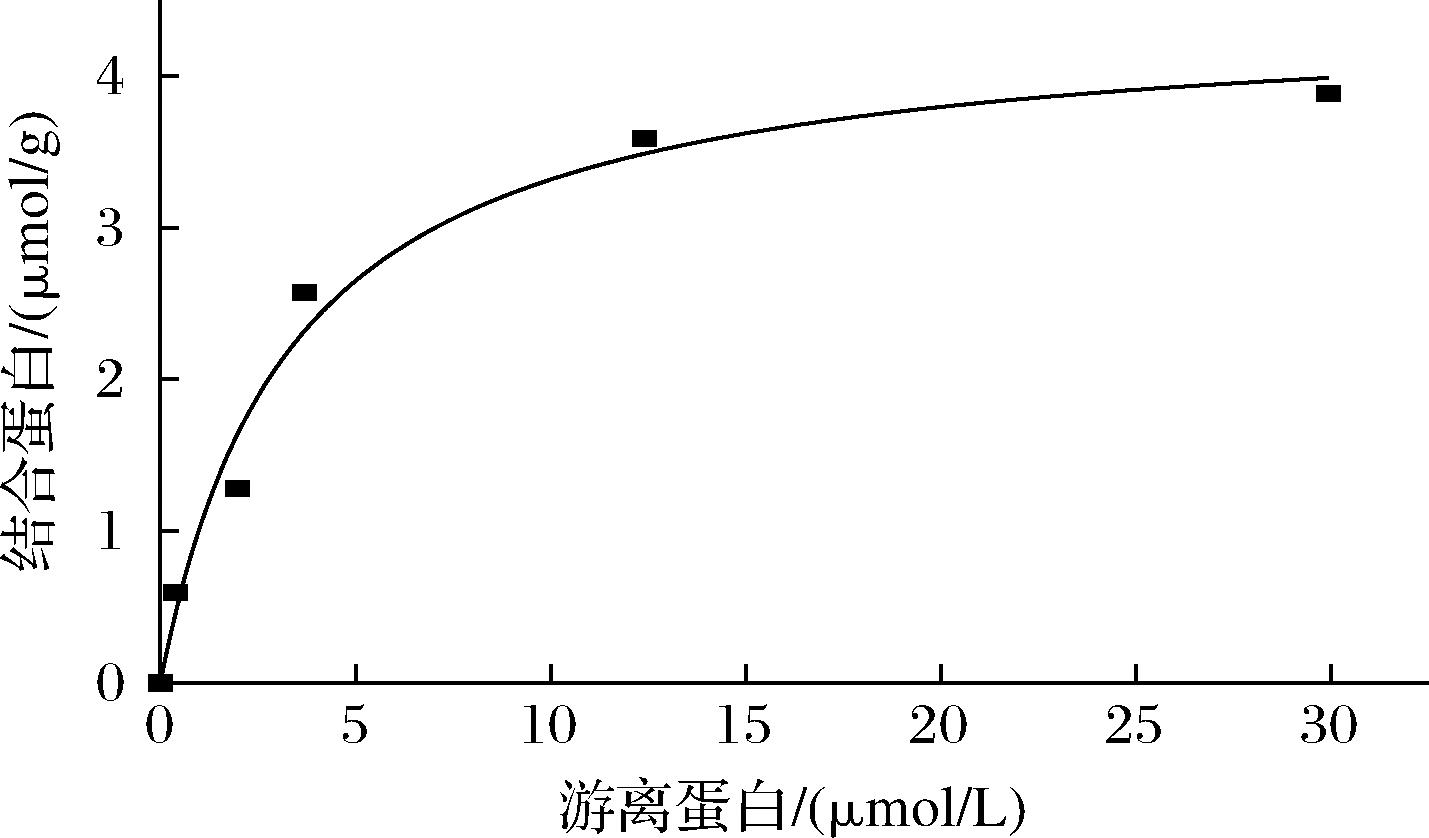
图4 吸附等温线法测定VaCBM6与β-1,3-木聚糖的结合
Fig.4 Adsorption isotherm assay to determine the binding of VaCBM6 with β-1,3-xylan
2.4 基于VaCBM6构建β-1,3-木聚糖荧光探针
原位分析是解决众多多糖相关问题的关键,如多糖在食品中的组成与分布[26]、多糖在食品加工中的变化以及多糖与其他组分的相互作用等[27]。当前多糖原位分析的实现主要依赖于对目标底物具有特异性的分子探针,然而目前尚无β-1,3-木聚糖的特异性探针。多糖结合蛋白常被用作探针服务于多糖的原位分析。为此,将VaCBM6与绿色荧光蛋白EmGFP融合表达,进一步通过亲和纯化获得电泳纯荧光探针EmGFP-VaCBM6。SDS-PAGE结果显示纯化蛋白的分子质量约为45.0 kDa(图5-a),与EmGFP-VaCBM6预测分子质量(45.45 kDa)相一致。3D荧光扫描结果显示所构建探针的最佳激发波长为490 nm,最佳发射波长为510 nm(图5-b)。利用相同方法对该探针与上述不溶性多糖的结合能力进行分析,结果表明EmGFP-VaCBM6仅与β-1,3-木聚糖有结合,这是首次构建得到β-1,3-木聚糖的特异性荧光探针。
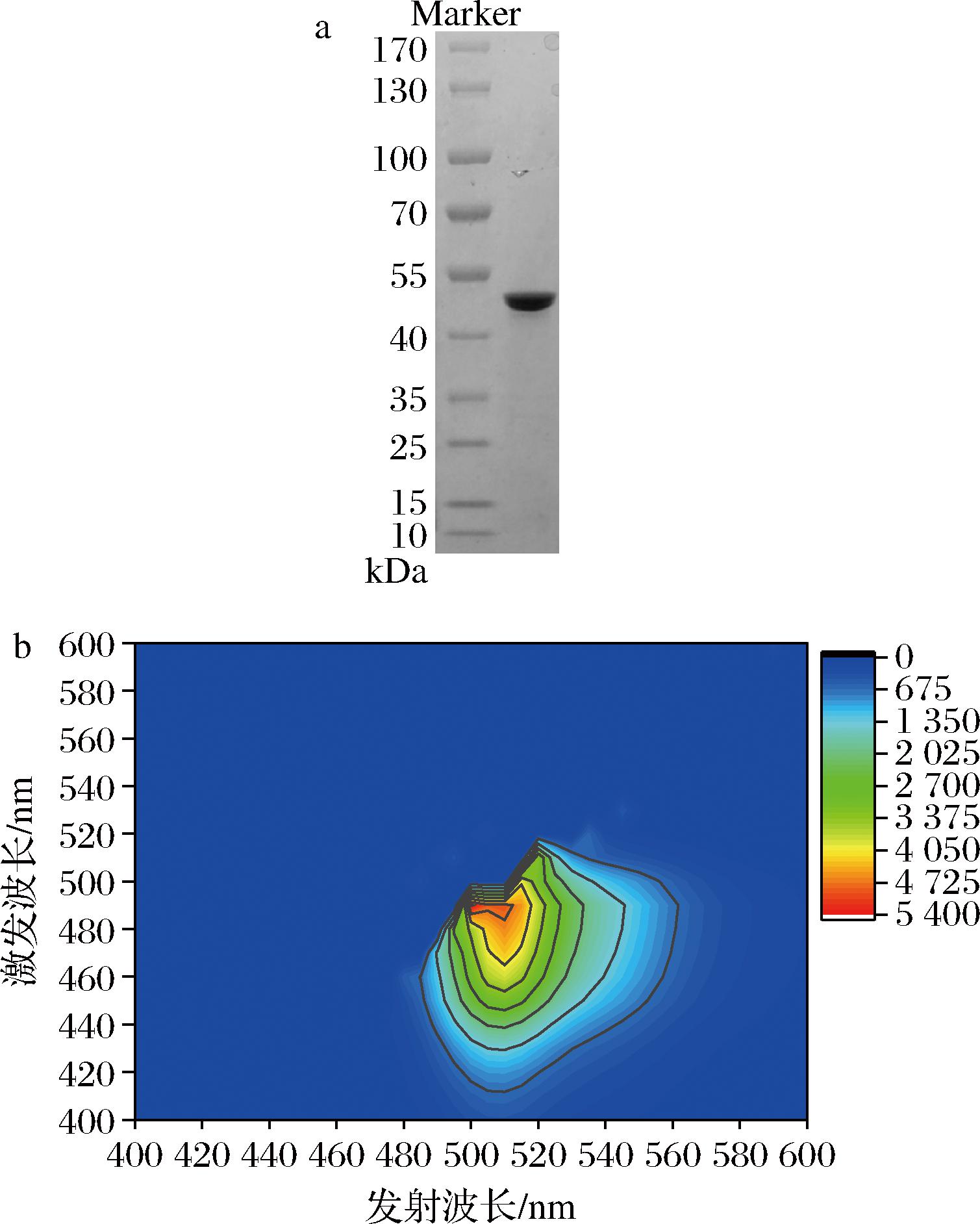
a-SDS-PAGE电泳图;b-3-D全波长扫描图
图5 纯化后EmGFP-VaCBM6分析
Fig.5 Analysis of the purified EmGFP-VaCBM6
2.5 β-1,3-木聚糖在紫菜中的原位可视化观察
为了考察所构建探针在β-1,3-木聚糖原位可视化中的应用潜力,利用EmGFP-VaCBM6对紫菜中的β-1,3-木聚糖进行了染色观察。经EmGFP-VaCBM6染色后,观察到了明显的绿色荧光信号(图6)。比对明场视野下的显微图像,发现这些绿色荧光主要分布在细胞壁上,与先前报道的紫菜中β-1,3-木聚糖的分布模式一致。上述结果证实了EmGFP-VaCBM6在β-1,3-木聚糖原位可视化中的可行性。
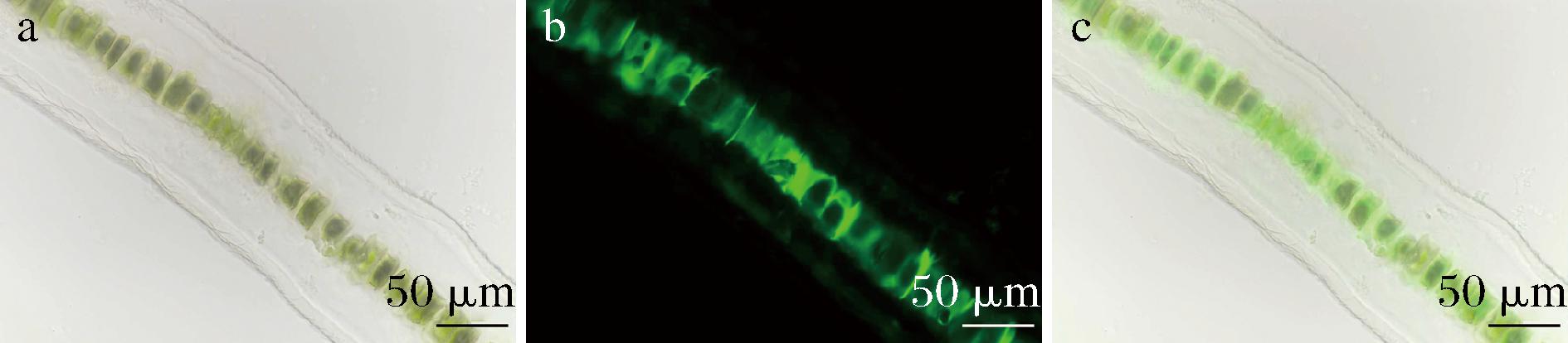
a-明场视野显微图像;b-荧光视野显微图像;c-两通道叠合图
图6 EmGFP-VaCBM6对紫菜染色荧光显微图像
Fig.6 Fluorescence microscopic images of the Porphyrastained seaweed by EmGFP-VaCBM6
3 结论
本研究成功克隆并异源表达了一个新颖的CBM6家族β-1,3-木聚糖结合蛋白VaCBM6,并系统研究了该蛋白的结合能力。VaCBM6能够特异性结合β-1,3-木聚糖,而对β-1,4-甘露聚糖、β-1,4-木聚糖等不溶性多糖,紫菜多糖、琼脂糖等可溶性多糖均无结合能力。吸附等温线结果表明该蛋白对β-1,3-木聚糖的结合常数为3.0×105 (mol/L)-1。将VaCBM6与绿色荧光蛋白EmGFP融合表达构建出首个β-1,3-木聚糖特异性荧光探针,并利用该探针实现了紫菜中β-1,3-木聚糖的荧光显微观察。VaCBM6的发现与表征为β-1,3-木聚糖的研究与开发提供了良好工具。
[1] MAEDA R, IDA T, IHARA H, et al.Induction of apoptosis in MCF-7 cells by β-1,3-xylooligosaccharides prepared from Caulerpa lentillifera[J].Bioscience, Biotechnology, and Biochemistry, 2012, 76(5):1032-1034.
[2] EBRINGEROV A, KARDO
A, KARDO OV
OV A, HROM
A, HROM DKOV
DKOV Z, et al.Immunomodulatory activity of acidic xylans in relation to their structural and molecular properties[J].International Journal of Biological Macromolecules, 2002, 30(1):1-6.
Z, et al.Immunomodulatory activity of acidic xylans in relation to their structural and molecular properties[J].International Journal of Biological Macromolecules, 2002, 30(1):1-6.
[3] ZHANG B J, ZHONG Y D, DONG D J, et al.Gut microbial utilization of xylan and its implication in gut homeostasis and metabolic response[J].Carbohydrate Polymers, 2022, 286:119271.
[4] HROM DKOV
DKOV Z, KO
Z, KO T’
T’ LOV
LOV Z, EBRINGEROV
Z, EBRINGEROV A.Comparison of conventional and ultrasound-assisted extraction of phenolics-rich heteroxylans from wheat bran[J].Ultrasonics Sonochemistry, 2008, 15(6):1062-1068.
A.Comparison of conventional and ultrasound-assisted extraction of phenolics-rich heteroxylans from wheat bran[J].Ultrasonics Sonochemistry, 2008, 15(6):1062-1068.
[5] MANDAL P, PUJOL C A, DAMONTE E B, et al.Xylans from Scinaia hatei:Structural features, sulfation and anti-HSV activity[J].International Journal of Biological Macromolecules, 2010, 46(2):173-178.
[6] DEUTSCHMANN R, DEKKER R F H.From plant biomass to bio-based chemicals:Latest developments in xylan research[J].Biotechnology Advances, 2012, 30(6):1627-1640.
[7] YILMAZ-TURAN S, JIMÉNEZ-QUERO A, MENZEL C, et al.Bio-based films from wheat bran feruloylated Arabinoxylan:Effect of extraction technique, acetylation and feruloylation[J].Carbohydrate Polymers, 2020, 250:116916.
[8] MIKKONEN K S, TENKANEN M.Sustainable food-packaging materials based on future biorefinery products:Xylans and mannans[J].Trends in Food Science &Technology, 2012, 28(2):90-102.
[9] CARTAXO DA COSTA URTIGA S, RODRIGUES MARCELINO H, S CRATES TABOSA DO EGITO E, et al.Xylan in drug delivery:A review of its engineered structures and biomedical applications[J].European Journal of Pharmaceutics and Biopharmaceutics:Official Journal of Arbeitsgemeinschaft Fur Pharmazeutische Verfahrenstechnik e.V, 2020, 151:199-208.
CRATES TABOSA DO EGITO E, et al.Xylan in drug delivery:A review of its engineered structures and biomedical applications[J].European Journal of Pharmaceutics and Biopharmaceutics:Official Journal of Arbeitsgemeinschaft Fur Pharmazeutische Verfahrenstechnik e.V, 2020, 151:199-208.
[10] IRIKI Y, SUZUKI T, NISIZAWA K, et al.Xylan of siphonaceous green algae[J].Nature, 1960, 187:82-83.
[11] MEI X W, CHANG Y G, SHEN J J, et al.Characterization of a novel carrageenan-specific carbohydrate-binding module:A promising tool for the in situ investigation of carrageenan[J].Journal of Agricultural and Food Chemistry, 2022, 70(29):9066-9072.
[12] MCCARTNEY L, BLAKE A W, FLINT J, et al.Differential recognition of plant cell walls by microbial xylan-specific carbohydrate-binding modules[J].Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(12):4765-4770.
[13] AHL L I, GRACE O M, PEDERSEN H L, et al.Analyses of Aloe polysaccharides using carbohydrate microarray profiling[J].Journal of AOAC International, 2018, 101(6):1720-1728.
[14] SHOSEYOV O, SHANI Z, LEVY I.Carbohydrate binding modules:Biochemical properties and novel applications[J].Microbiology and Molecular Biology Reviews:MMBR, 2006, 70(2):283-295.
[15] OKAZAKI F, TAMARU Y, HASHIKAWA S, et al.Novel carbohydrate-binding module of β-1,3-xylanase from a marine bacterium, Alcaligenes sp.strain XY-234[J].Journal of Bacteriology, 2002, 184(9):2399-2403.
[16] KIYOHARA M, SAKAGUCHI K, YAMAGUCHI K, et al.Characterization and application of carbohydrate-binding modules of β-1,3-xylanase XYL4[J].Journal of Biochemistry, 2009, 146(5):633-641.
[17] LIU T, YI Z W, ZENG R Y, et al.The first characterization of a Ca2+-dependent carbohydrate-binding module of β-1, 3-xylanase from Flammeovirga pacifica[J].Enzyme and Microbial Technology, 2019, 131:109418.
[18] YI Z W, CAI Z W, ZENG B, et al.Identification and characterization of a novel thermostable and salt-tolerant β-1,3 xylanase from Flammeovirga pacifica strain WPAGA1[J].Biomolecules, 2020, 10(9):1287.
[19] YIN Y B, MAO X Z, YANG J C, et al.dbCAN:A web resource for automated carbohydrate-active enzyme annotation[J].Nucleic Acids Research, 2012, 40(Web Server issue):W445-W451.
[20] YANG Z Y, ZENG X X, ZHAO Y, et al.AlphaFold2 and its applications in the fields of biology and medicine[J].Signal Transduction and Targeted Therapy, 2023, 8(1):115.
[21] WILKINS M R, GASTEIGER E, BAIROCH A, et al.Protein identification and analysis tools in the ExPASy server[J].Methods in Molecular Biology, 1999, 112:531-552.
[22] ALI E, ZHAO G S, SAKKA M, et al.Functions of family-22 carbohydrate-binding module in Clostridium thermocellum Xyn10C[J].Bioscience, Biotechnology, and Biochemistry, 2005, 69(1):160-165.
[23] FICKO-BLEAN E, BORASTON A B.The interaction of a carbohydrate-binding module from a Clostridium perfringens N-acetyl-β-hexosaminidase with its carbohydrate receptor[J].The Journal of Biological Chemistry, 2006, 281(49):37748-37757.
[24] MCCARTNEY L, GILBERT H J, BOLAM D N, et al.Glycoside hydrolase carbohydrate-binding modules as molecular probes for the analysis of plant cell wall polymers[J].Analytical Biochemistry, 2004, 326(1):49-54.
[25] YIN Q Y, TENG Y G, LI Y H, et al.Expression and characterization of full-length Ampullaria crossean endoglucanase EG65 s and their two functional modules[J].Bioscience, Biotechnology, and Biochemistry, 2011, 75(2):240-246.
[26] LEROUX O, LEROUX F, BAGNIEWSKA-ZADWORNA A, et al.Ultrastructure and composition of cell wall appositions in the roots of Asplenium (Polypodiales)[J].Micron, 2011, 42(8):863-870.
[27] ARLTOFT D, MADSEN F, IPSEN R.Screening of probes for specific localisation of polysaccharides[J].Food Hydrocolloids, 2007, 21(7):1062-1071.