神经酰胺是生物体内一类重要的脂质分子,具有多种生物学功能,广泛参与细胞生长、增殖、分化和凋亡等生命活动[1]。其主要由长链碱基和脂肪酸两部分通过酰胺键共价键合组成,长链碱基主要有二氢鞘氨醇、鞘氨醇和植物鞘氨醇3种类型。神经酰胺具有保湿、屏障、抗肿瘤、调节代谢等多种功能[2-4],在化妆品、食品及药物开发等领域有广阔的应用前景;据调查,2018年全球神经酰胺市场价值约为3.452亿美元,预计在2019年至2029年期间的复合年增长率将超过6%[5],具有较高的商业价值,因而开发神经酰胺的大规模制造技术具有广阔市场前景。
神经酰胺最初从动植物中提取,成本高且神经酰胺组成及含量不稳定;利用微生物细胞工厂制备神经酰胺可克服这些缺陷因而成为目前主要方法。酿酒酵母和威克汉姆西弗酵母(Wickerhamomyces ciferrii)常作为神经酰胺的生产宿主,基于多种代谢改造策略提升神经酰胺的生产。陈国强团队[6-7]在酿酒酵母中通过培养基优化及增强神经酰胺的表达等多种策略,使神经酰胺含量得到了不同比例的提升;KIM等[8]过表达3-酮二氢鞘氨醇还原酶将酿酒酵母神经酰胺含量提升至9.79 mg/g;MURAKAMI等[9]在酿酒酵母中引入人源鞘脂去饱和基因用于生产人源神经酰胺-NS,并通过代谢工程将产量提升了4倍。而W.ciferrii可天然分泌生产四乙酰植物鞘氨醇(Tetraacetyl phytosphingosine, TAPS),工业上通过对其再加工生产神经酰胺。SCHORSCH 等[10]通过代谢工程改造使得TAPS产量达到199 mg/g;而后HAN等[11]在解脂耶式酵母中引入W.ciferrii乙酰转移酶基因进行改造生产,最终TAPS产量达到(650±24) mg/L。但目前尚无利用Pichia pastoris强化神经酰胺合成的报道,对于其神经酰胺生产潜力尚未挖掘。
在酵母中,神经酰胺的从头合成起始于内质网,棕榈酰辅酶A和L-丝氨酸在丝氨酸棕榈酰转移酶(serine palmitoyltransfcrase, SPT)的作用下生成3-酮二氢鞘氨醇;下一步,在3-酮二氢鞘氨醇还原酶的作用下生成二氢鞘氨醇,而后在2种神经酰胺合酶(Lag1和Lac1)的作用下生成神经酰胺[12]。SPT是由Lcb1和Lcb2蛋白形成的二聚体,是生物体内神经酰胺合成的关键限速酶,因而SPT的活性对神经酰胺合成至关重要;传统代谢策略通过增强Lcb1和Lcb2提升SPT活性,研究发现酿酒酵母中,Lcb1和Lcb2受限于orm1和orm2调控[13]。Orm1和Orm2蛋白质可以与SPT相互作用,负反馈调节SPT活性,调控细胞鞘脂稳态[14-15],因而Orm1和Orm2蛋白是神经酰胺合成的重要调控因子。
目前由于商业化的神经酰胺标准品有限,仅限于常见类别如Cer(d18∶0),无法对种类繁多的神经酰胺进行常规鉴别和分析(如高效液相色谱),而随着基于LC-MS/MS的脂质组学技术的发展,可以同时对细胞中所有脂质成分进行鉴定,这为神经酰胺这类脂质分析提供了有力的工具。如JONES等[16]通过脂质组的方法,结合多个标准物质对小鼠肺组织的神经酰胺含量进行了表征,阐释了神经酰胺在相关模型中差异变化。本研究也将采用脂质组学技术分析神经酰胺的组成及相对含量,揭示基因缺失对P.pastoris脂质复杂变化的影响。
P.pastoris具有生长速度快、高密度发酵、易于遗传改造操作等优点,是工业中生产优良底盘细胞,尤其是其高密度发酵特性为降低神经酰胺的成本提供了可能。本研究利用P.pastoris作为宿主菌株,以经过基因改造(抑制非同源修复能力)的G-K菌株为出发菌,通过缺失SPT负反馈调节基因这一策略,探究该策略对神经酰胺合成及其他脂质变化的影响。为工业上利用具有高密度培养优势的P.pastoris生产神经酰胺奠定基础,为神经酰胺的大规模制造提供借鉴思路。
1 材料与方法
1.1 培养基与试剂
LB培养基(g/L):酵母粉5、蛋白胨10、NaCl 10;
酵母浸出粉胨葡萄糖(yeast extract peptone dextrose, YPD)培养基(g/L):酵母粉10、蛋白胨20、葡萄糖20;
以上固体培养基通过添加质量分数为1.5%~2.0%的琼脂粉配制。
含甘油的缓冲性完全培养基(buffered glycerol-complex medium, BMGY)(1L):酵母粉10 g、蛋白胨20 g、甘油10 g、100 mL磷酸钾缓冲液(pH 6.0)、YNB 13.4 g、生物素400 μg。
试剂:酵母粉、蛋白胨,英国Oxoid公司;NaCl、KCl、KH2PO4、K2HPO4、葡萄糖、山梨醇、分析级氯仿、分析级甲醇、甘油,国药集团化学试剂有限公司;GoldView核酸染料、博来霉素、YNB,北京索莱宝生物工程有限公司;质粒小提试剂盒,北京天根生化科技公司;一步克隆、PCR产物回收、胶回收试剂盒,南京诺唯赞生物科技股份有限公司。
1.2 菌株与质粒
菌株:大肠杆菌(Escherichia coli)JM109用于质粒的构建, P.pastoris GS115作为实验宿主菌株;
质粒:pPpT4敲除质粒,pUC57载体质粒。
1.3 实验方法
1.3.1 敲除质粒构建
在https://zlab.bio/guide-design-resources上查询敲除目的基因的sgRNA,选取20 bp作为敲除基因的靶点,在 SnapGene软件进行质粒和引物设计,以pPpT4为模板反向PCR线性化扩增质粒骨架。将PCR产物进行琼脂糖电泳,胶回收后取100 ng化学转化到E.coli JM109中连接成环,验证成功后使用质粒小提试剂盒提取质粒备用。
1.3.2 P.pastoris基因组提取
按文献[17]报道方法进行。
1.3.3 P.pastoris基因敲除
根据目的基因的sgRNA构建敲除质粒,选取目的基因上下游各1 000 bp左右的同源区段分别扩增,通过融合PCR将上下游同源臂进行连接,构建敲除重叠臂。将装有40 μL P.pastoris感受态细胞的EP管置于冰上解冻,加入100 ng敲除质粒和500 ng同源臂片段,吹吸混匀,转移到预冷好的点击杯中,电击后,立刻加入预冷好的0.5 mL YPD培养基和0.5 mL 1 mol/L D-山梨醇,吹吸混匀后转移到新的1.5 mL EP管中,于30 ℃,200 r/min中复苏1~3 h后,涂布于博来霉素的抗性平板中,筛选阳性转化子。
1.3.4 P.pastoris摇瓶发酵
将菌株在YPD平板上划线,于30 ℃恒温培养箱中培养活化,挑取单菌落接种于3 mL YPD试管中,于30 ℃恒温摇床中200 r/min培养过夜。取50 μL接种于5 mL YPD试管中再次活化,过夜培养。以1%种子培养液接种于含有50 mL BMGY培养基进行发酵培养(30 ℃,220 r/min)。
1.3.5 脂质提取
参照HEIN等[18]的方法开展相关实验。
1.3.6 脂质组学分析
脂质的液相色谱质谱分析:色谱柱为Waters CSH C18柱(2.1 mm×100 mm,1.7 μm),Thermo Fisher Ultimate 3000 UPLC系统。A相为含10 mmol/L甲酸铵的乙腈/水(60∶40,体积比),B相为含10 mmol/L甲酸铵的异丙醇/乙腈(90∶10,体积比)。线性梯度进行洗脱:0~3 min,40% B;3~20 min,40%~95% B;20~22.5 min,95% B;22.5~23 min,95%~40% B;23~25 min,40% B,以上均为体积分数;流速0.3 mL/min,进样量0.5 μL。
1.3.7 神经酰胺HPLC测定
HPLC测定通过邻苯二甲醛进行柱前衍生化,具体方法详见文献[19]。
2 结果与分析
2.1 神经酰胺生产菌株的构建
由于P.pastoris存在以非同源修复为主的修复机制,导致基因改造同源重组机制效率较低,降低了转化重组效率。因而抑制P.pastoris中非同源修复是增强菌株代谢工程改造成功率的有效途径。据报道,ku70是非同源修复的关键基因,缺失该基因可以显著提升P.pastoris同源定向修复效率[20]。因此本论文针对P.pastoris GS115基因敲除分两步:首先,通过TBLASTN算法进行同源性搜索,在基因组中检索到与ku70同源的基因PAS_chr3_0329,利用CRISPR/Cas9基因编辑技术对其进行删除,获得G-K菌株,以此为出发菌进行后续改造,对后续一系列代谢工程改造具有显著优势。
酿酒酵母中orm1和orm2基因是鞘脂生物合成途径的关键调控因子[15],通过TBLASTN算法比对orm1、orm2同系物基因检测出一个对应同源基因PAS_chr4_0427。因此第二步以G-K为出发菌,敲除PAS_chr4_0427,获得G-KO菌株。对上述缺失株设计验证引物进行PCR验证,电泳结果显示,PAS_chr3_0329和PAS_chr4_0427均已缺失,菌株构建成功。
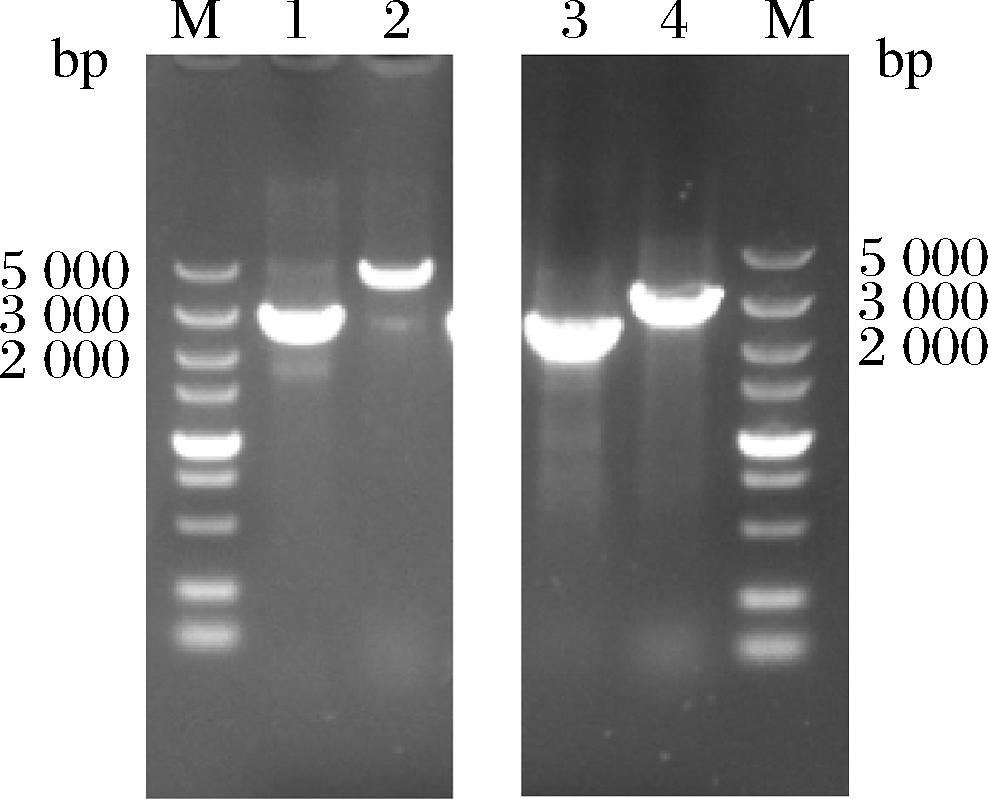
图1 缺失株的PCR验证
Fig.1 Verification of deletion strain by PCR
注:M,Marker;1,ΔPAS_chr3_0329;2,Control;3,ΔPAS_chr4_0427;4,Control。
2.2 生产菌株的生长
为验证基因缺失对菌株的生长是否产生影响,将突变株与野生菌株进行生长曲线的比较。如图2所示,在YPD培养基中,3个菌株在12 h进入对数生长期,野生菌株和G-K在48 h进入稳定生长期,而菌株G-KO在64 h进入稳定期。与GS115相比,G-K的趋势相似,生长速率和生物量略有下降,但差别不大,GS115最大OD600值达24.17,G-K最大OD600达22.93,降低5.13%;与G-K相比,G-KO在对数期生长速率有一定程度下降,但最大OD600仍可达22.15,相比G-K下降了3.52%。G-K缺失PAS_chr4_0427后生长速度下降明显,可能由于细胞SPT的负反馈抑制缺失导致细胞的鞘脂稳态失衡,鞘脂增多导致一定细胞毒性(数据见2.5节);但其最大OD600仍与G-K相差不大,因此从生长特性考虑,G-KO仍是合适生产的菌株。
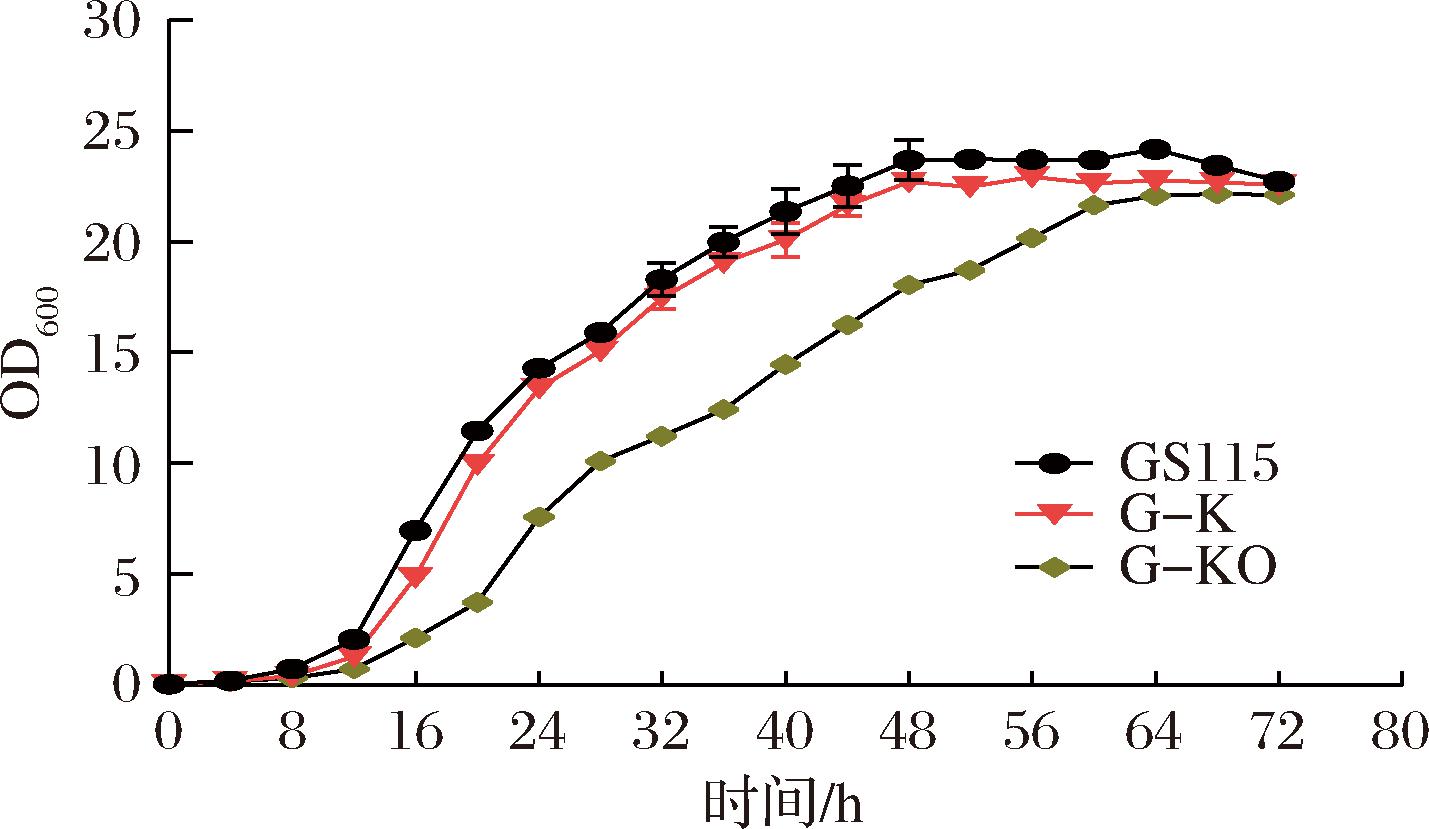
图2 各菌株的生长曲线
Fig.2 Growth curve of each strain
2.3 神经酰胺总量变化
对GS115,G-K和G-KO进行全脂质提取,分析酵母全脂质的组成成分及相对强度,比较不同菌株间神经酰胺的相对含量差异。由图3可知,GS115和G-K中神经酰胺相对含量分别为1.37%和1.28%,两者无统计学差异,说明缺失PAS_chr3_0329基因未对神经酰胺产生明显影响,因此以G-K作为基因改造的出发菌选择是合理的。与G-K相比,G-KO的神经酰胺含量显著增加到9.32%,提升幅度高达628.13%,表明通过缺失SPT负反馈调节因子,可显著促进P.pastoris合成积累神经酰胺。G-KO具备良好的神经酰胺生产潜力,可作为下一步代谢工程改造的出发菌。
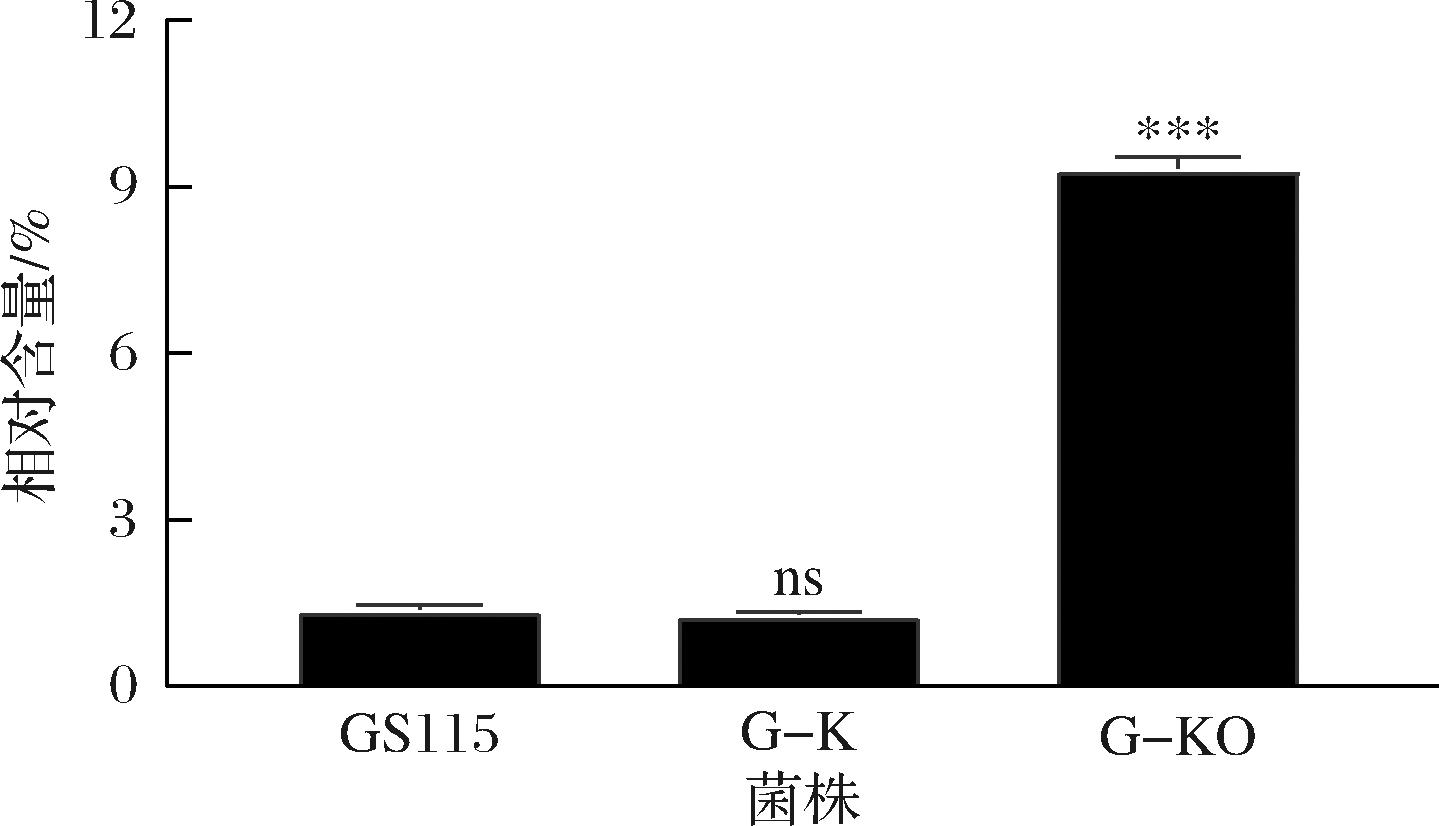
图3 各菌株神经酰胺相对含量
Fig.3 Relative content of ceramide in each strain
2.4 神经酰胺的组成及含量
脂质组学数据鉴定出不同长链碱基的神经酰胺含量占比,因此论文探究P.pastoris合成神经酰胺的不同种类及含量,图4表征了不同离子模式下相对含量。结果显示,在P.pastoris中,神经酰胺的长链碱基部分主要集中在C15~C20,其中以C18长链碱基的神经酰胺最为丰富,这与前人研究相一致[21]。与GS115相比,正离子模式下G-K在(d17∶0)、(d18∶0)、(d20∶0)略有下降,而(d18∶1)有所上升;负离子模式下,G-K在(d18∶1)、(d18∶2)上升,而(d17∶0)、(d18∶0)、(d20∶0)小幅下降,两者神经酰胺相对含量没有显著变化,而在长链碱基的种类中,缺失PAS_chr3_0329基因促进部分神经酰胺种类向不饱和的C18长链碱基神经酰胺转化合成。
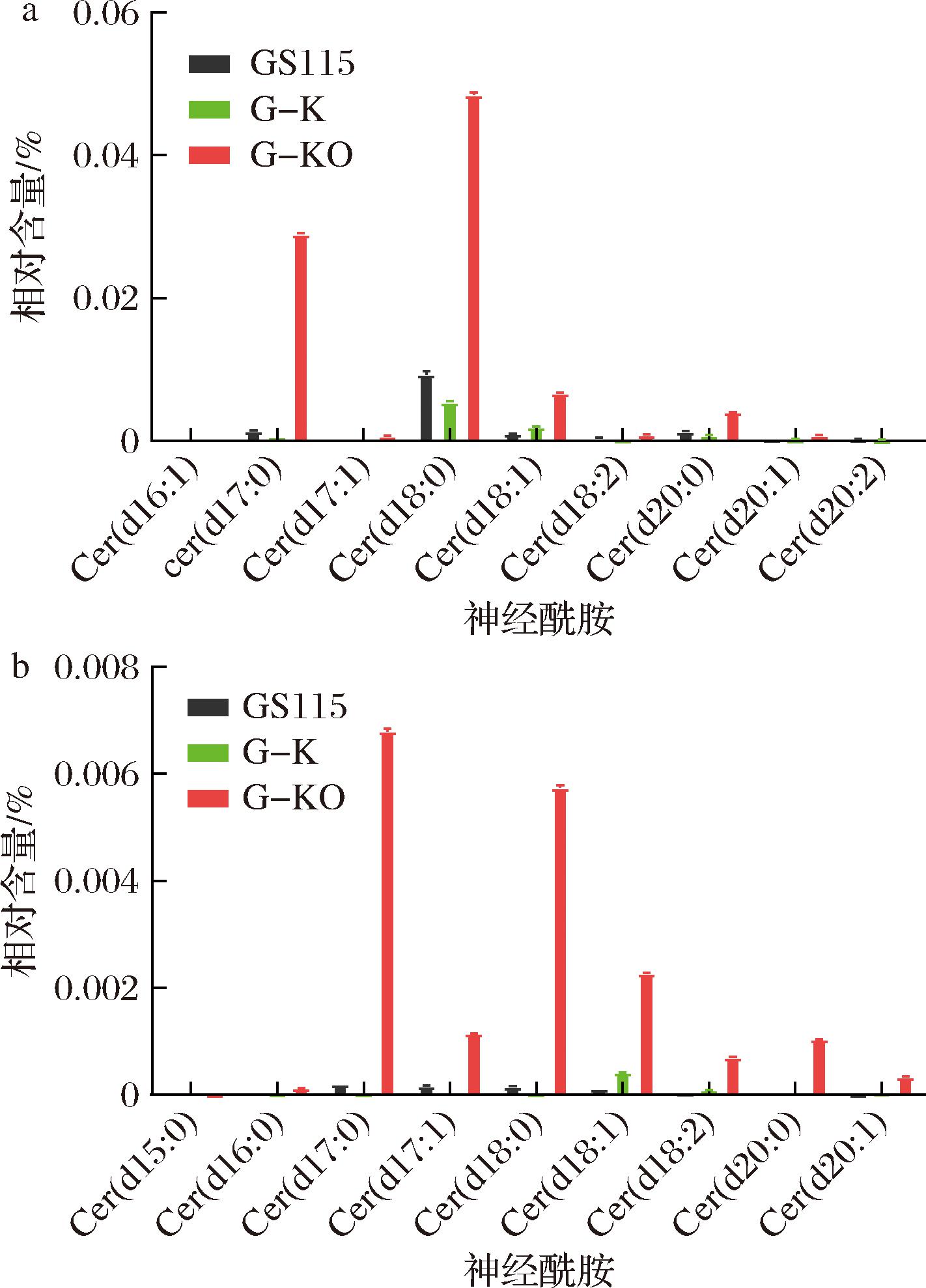
a-正离子模式;b-负离子模式
图4 各菌株中不同种类神经酰胺的相对含量
Fig.4 The relative content of different ceramides in each strain
与G-K相比,G-KO中各长链碱基神经酰胺均显著提升,提升幅度最大的为(d17∶0)、(d18∶0)、(d18∶1)几种神经酰胺,其中(d18∶0)含量最高。缺失PAS_chr4_0427可显著改善P.pastoris的神经酰胺种类分布,以C18长链碱基为主的神经酰胺向具有更少或更多碳原子数长链碱基神经酰胺扩展的趋势,且增加了不饱和C18长链碱基神经酰胺的含量。由于不饱和的神经酰胺与人源的神经酰胺有着更高的相似性[9],因此这将提高神经酰胺的化妆品功效。但另外一方面,不饱和长链碱基的神经酰胺在细胞中可能具备一定细胞毒性,这也可能是其生长速度下降的主要原因,是后续发酵生产中需要考虑的关键因素。
2.5 神经酰胺对其他脂质的影响
脂质组可提供菌株的全脂质信息,通过神经酰胺之外其他脂质的变化,可探究神经酰胺含量增加对酵母细胞脂质的影响。在P.pastoris GS115中,磷脂、鞘脂和甘油脂含量较为丰富,其中磷脂丰度最高,占据细胞全脂质60%以上,而磷脂中又以磷脂酰胆碱(phosphatidylcholine, PC)丰度最高,磷脂酰乙醇胺(phosphatidylethanolamine, PE)丰度次之。图5分别展示了各菌株脂质在正负离子模式下的相对含量变化,图6对鉴定出的相对丰度较高的脂质变化进行直观呈现。对GS115而言,其PC和PE相对含量分别为36.64%和13.41%,缺失PAS_chr3_0329后,G-K中 PC和PE相对含量占比分别达到38.53%和15.81%,呈现小幅增长。
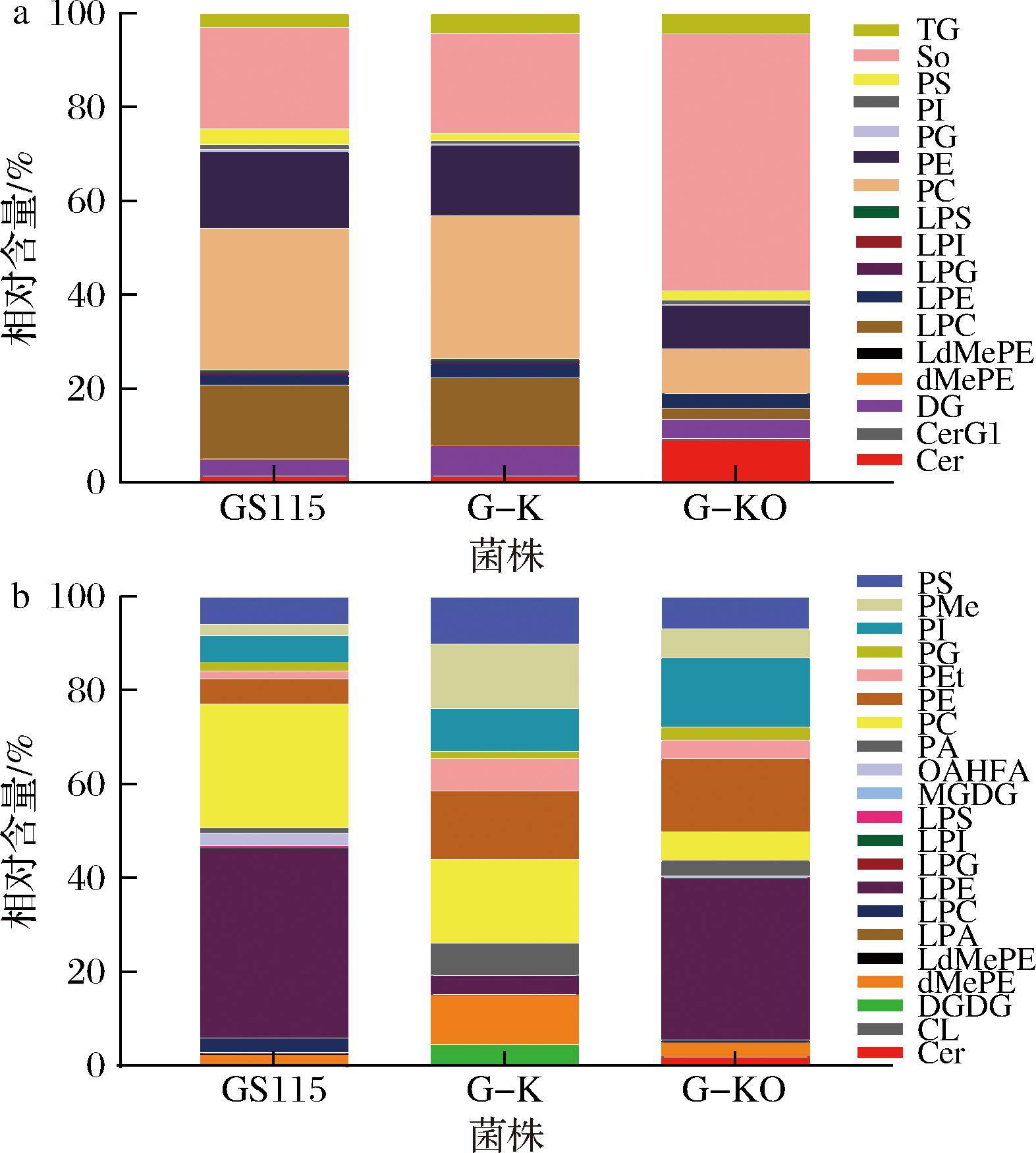
a-正离子模式;b-负离子模式
图5 各菌株的脂质分布
Fig.5 The distribution of different lipids in each strain
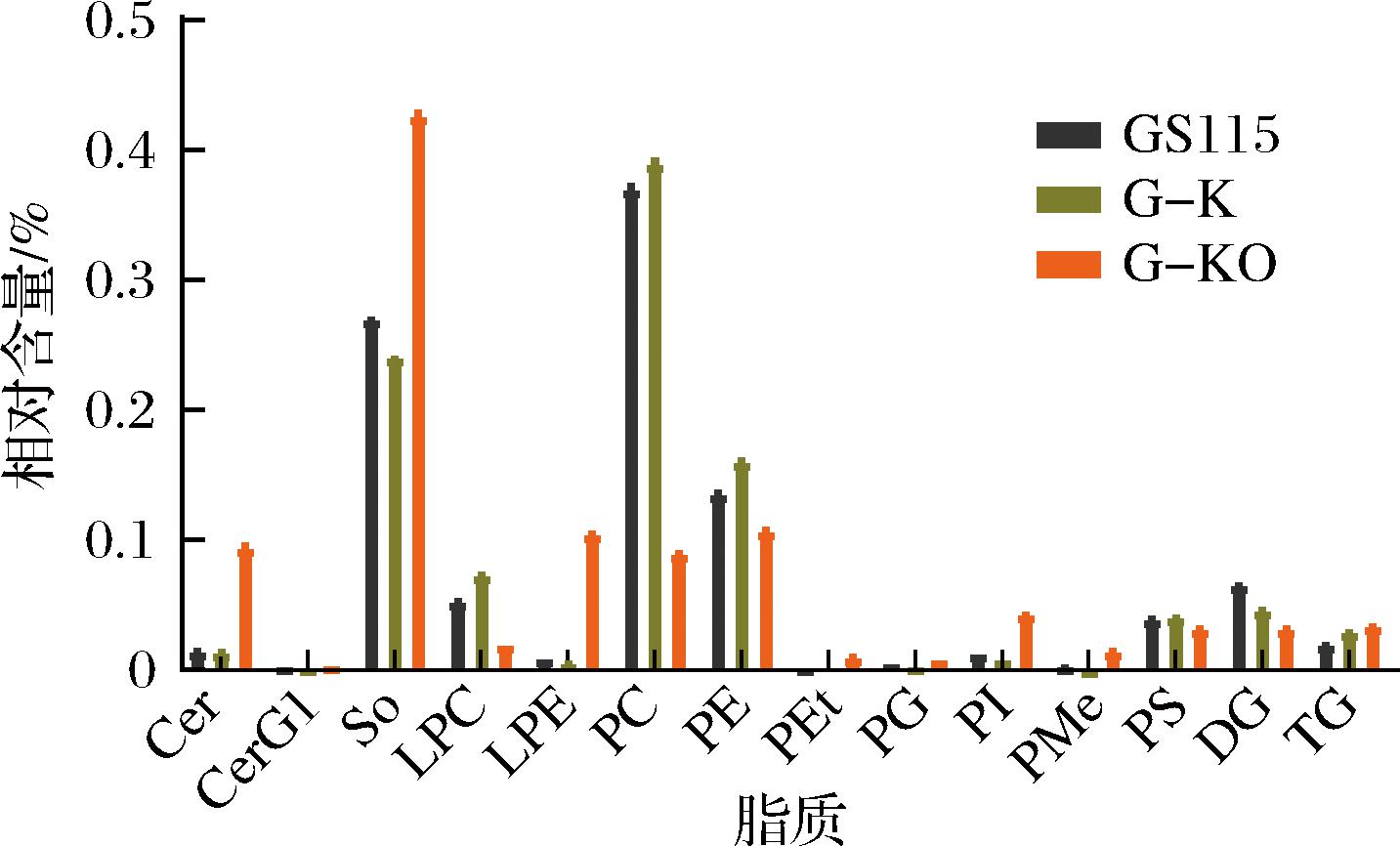
图6 菌株中各脂质相对含量
Fig.6 The relative content of different lipids in each strain
与G-K相比,G-KO中随着神经酰胺的含量提升,磷脂总含量下降,具体为:PC、PE和溶血磷脂酰胆碱(lysophosphatidylcholine, LPC)显著下降,其中PC最为明显,降至8.77%,但与之不同的是,溶血磷脂酰乙醇胺(lysophosphatidylethanolamine, LPE)和磷脂酰肌醇(phosphatidylinositol, PI)则显著上升。
另外一个重要脂质是鞘脂中的鞘氨醇(sphingosine, So),GS115、G-K、G-KO的So相对含量分别为26.73%、23.79%、42.14%,So在G-KO中显著积累。因为在神经酰胺的合成路径中,So是重要的中间物质,缺失SPT负反馈调节因子后,神经酰胺合成效率提升,促进了更多So的合成积累;而鞘脂比例的提升影响了细胞中磷脂的分布,导致PC、PE等磷脂含量下降,先前研究发现在酿酒酵母中△orm1△orm2导致细胞内磷脂的平衡被破坏[22],表明该基因不仅调控细胞中鞘脂的稳态平衡,还可能参与磷脂的调控,改变鞘脂和磷脂的分布。
值得注意的是,PI的含量提升,神经酰胺可以和PI结合生成肌醇磷酸神经酰胺,这是神经酰胺向鞘磷脂转化的方式之一[23],细胞为降低神经酰胺过度积累造成的毒害作用,可能会增加神经酰胺的下游转化,而本研究中PI含量提升机制尚不明确,有待进一步研究。在甘油脂上,对菌株进行改造后二酰基甘油(diacylglycerol,DG)占比小幅下降、而三酰基甘油(triglyceride, TG)呈现小幅上升,DG、TG的含量变化可能是对神经酰胺调控生命活动的响应应激,保护细胞免受培养过程中的氧化胁迫[24]。
2.6 Cer(d18∶0)神经酰胺的产量
由于Cer(d18∶0)是P.pastoris中Cer(d18∶0)中含量最高的神经酰胺类别,而且有商业化标准品,因此通过HPLC测定Cer(d18∶0)神经酰胺的含量。将各菌株进行摇瓶发酵,测定Cer(d18∶0)的神经酰胺合成量。如图7所示,生长曲线与图2相似,在发酵培养基中,GS115和出发菌株G-K的最大生长OD600值分别达到50.19和48.12,未表现出显著差异;与G-K相比,G-KO生长速率和生物量积累有小幅下降,其最终OD600值为44.47,生物量降低了8.20%,但Cer(d18∶0)神经酰胺增幅明显,具体如下:
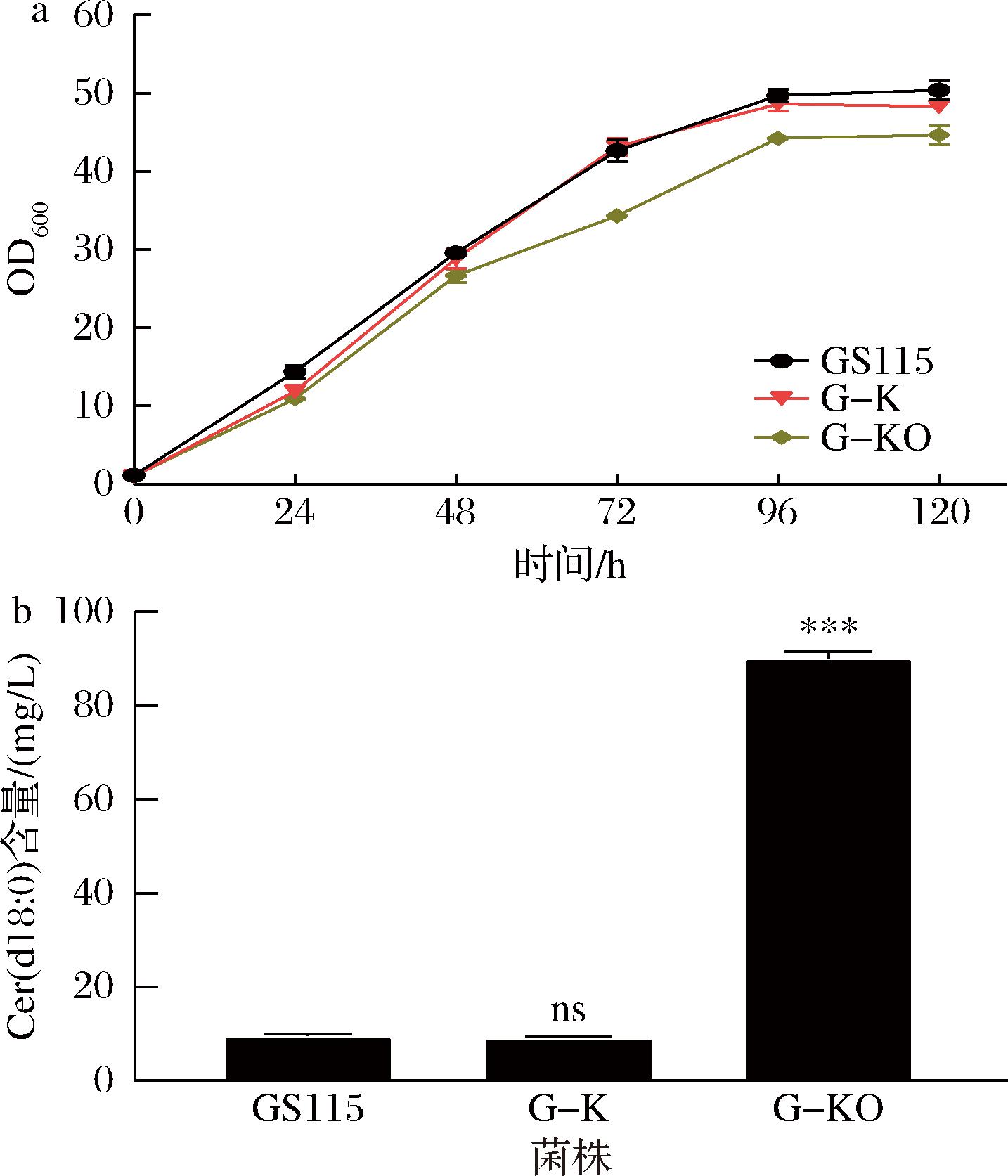
a-OD600;b-Cer(d18∶0)含量
图7 各菌株的生长及Cer(d18∶0)神经酰胺量
Fig.7 The growth curve and Cer(d18∶0) content in each strain
在120 h收集菌体,提取全部脂质,进行洗涤纯化,对Cer(d18∶0)神经酰胺进行HPLC测定。结果显示,GS115和G-K 中Cer(d18∶0)神经酰胺含量分别为9.66 mg/L和9.28 mg/L,两者间无统计学差异。值得关注的是,与G-K相比,G-KO的 Cer(d18∶0)神经酰胺含量显著增加,提升了872.20%,产量达到 90.22 mg/L。与G-K相比,G-KO的神经酰胺总量提升628.13%,低于Cer(d18∶0)的增长水平;通过对Cer(d18∶0)神经酰胺相对含量进行比较,GS115、G-K和G-KO的相对含量分别为0.81%、0.51%、3.83%(图8),相比与G-K,G-KO的Cer(d18∶0)神经酰胺相对含量提升了655.13%,与HPLC测定提升的幅度虽有差距,但两者的整体趋势相近,表明脂质组学方法分析神经酰胺含量变化的可靠性。
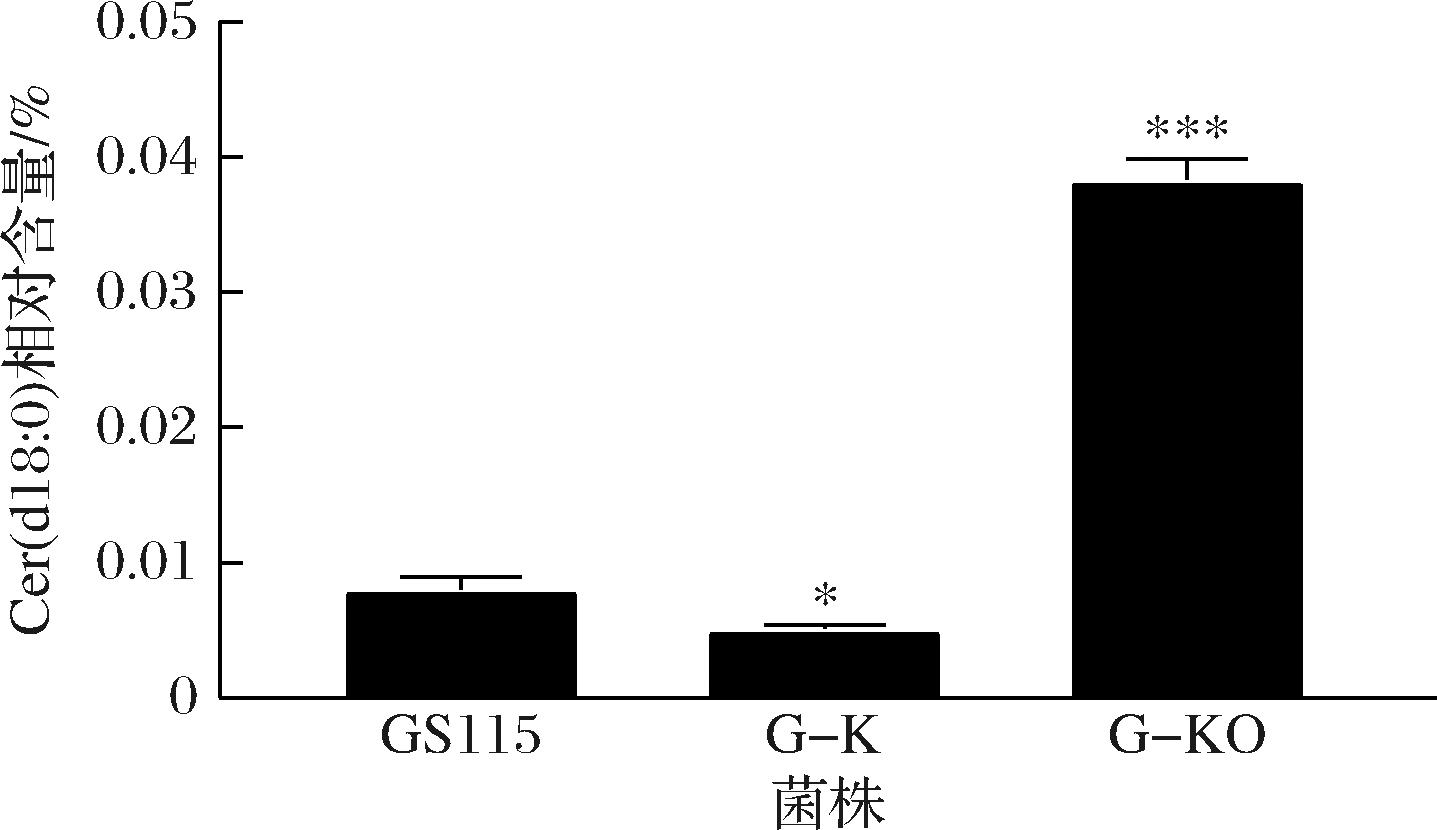
图8 各菌株的Cer(d18∶0)神经酰胺相对含量
Fig.8 The Cer(d18∶0) relative content in each strain
综上,本文首次证实P.pastoris是合成神经酰胺的合适宿主,敲除神经酰胺合成负反馈基因PAS_chr4_0427(G-KO)显著提升了菌株神经酰胺合成量,G-KO具有生产神经酰胺的潜力。
3 结论
本研究针对P.pastoris GS115菌株,通过缺失PAS_chr3_0329得到G-K菌株作为基因改造的优良出发菌,通过敲除G-K中神经酰胺合成负反馈基因PAS_chr4_0427,获得G-KO菌株。结果显示,G-KO菌株在生长上略有下降,但神经酰胺合成水平大幅提高,与G-K相比,G-KO中神经酰胺相对含量提升了628.13%,达到9.32%。此外还改变了神经酰胺组成分布,促进不饱和长链碱基神经酰胺及更少或者更多碳原子数的长链碱基神经酰胺合成;通过HPLC测定神经酰胺中丰度最高的Cer(d18∶0),G-KO中Cer(d18∶0)合成量达到90.22 mg/L,相较于G-K提升了872.20%。
[1] CANALS D, SALAMONE S, HANNUN Y A.Visualizing bioactive ceramides[J].Chemistry and Physics of Lipids, 2018, 216:142-151.
[2] ALIZADEH J, DA SILVA ROSA S C, WENG X H, et al.Ceramides and ceramide synthases in cancer:Focus on apoptosis and autophagy[J].European Journal of Cell Biology, 2023, 102(3):151337.
[3] UCHIDA Y, PARK K.Ceramides in skin health and disease:An update[J].American Journal of Clinical Dermatology, 2021, 22(6):853-866.
[4] VENTURA A E, MESTRE B, SILVA L C.Ceramide domains in health and disease:A biophysical perspective[J].Advances in Experimental Medicine and Biology, 2019, 1159:79-108.
[5] ZHU F F, ZHAO B, HU B, et al.Review of available "extraction+purification" methods of natural ceramides and their feasibility for sewage sludge analysis[J].Environmental Science and Pollution Research, 2023, 30(26):68022-68053.
[6] 林会兰. 酵母神经酰胺的提取、分析和表达[D].北京:清华大学, 2002.LIN H L.Extraction, analysis and expression of yeast ceramide[D].Beijing:Tsinghua University, 2002.
[7] 张琳. 酵母神经酰胺的提取、分析及生产的研究[D].长春:吉林大学, 2002.ZHANG L.Study on extraction, analysis and production of yeast ceramide[D].Changchun:Jilin University, 2002.
[8] KIM S K, NOH Y H, KOO J R, et al.Effect of expression of genes in the sphingolipid synthesis pathway on the biosynthesis of ceramide in Saccharomyces cerevisiae[J].Journal of Microbiology and Biotechnology, 2010, 20(2):356-362.
[9] MURAKAMI S, SHIMAMOTO T, NAGANO H, et al.Producing human ceramide-NS by metabolic engineering using yeast Saccharomyces cerevisiae[J].Scientific Reports, 2015, 5:16319.
[10] SCHORSCH C, KÖHLER T, ANDREA H, et al.High-level production of tetraacetyl phytosphingosine (TAPS) by combined genetic engineering of sphingoid base biosynthesis and L-serine availability in the non-conventional yeast Pichia ciferrii[J].Metabolic Engineering, 2012, 14(2):172-184.
[11] HAN C, JANG M, KIM M J, et al.Engineering Yarrowia lipolytica for de novo production of tetraacetyl phytosphingosine[J].Journal of Applied Microbiology, 2021, 130(6):1981-1992.
[12] DICKSON R C.Thematic review series:Sphingolipids.New insights into sphingolipid metabolism and function in budding yeast[J].Journal of Lipid Research, 2008, 49(5):909-921.
[13] BRESLOW D K, COLLINS S R, BODENMILLER B, et al.Orm family proteins mediate sphingolipid homeostasis[J].Nature, 2010, 463(7284):1048-1053.
[14] LIU M, HUANG C J, POLU S R, et al.Regulation of sphingolipid synthesis through Orm1 and Orm2 in yeast[J].Journal of Cell Science, 2012, 125(Pt 10):2428-2435.
[15] GURURAJ C, FEDERMAN R S, CHANG A.Orm proteins integrate multiple signals to maintain sphingolipid homeostasis[J].The Journal of Biological Chemistry, 2013, 288(28):20453-20463.
[16] JONES J W, CARTER C L, LI F, et al.Ultraperformance convergence chromatography-high resolution tandem mass spectrometry for lipid biomarker profiling and identification[J].Biomedical Chromatography:BMC, 2017, 31(3):10.1002/bmc.3822.
[17] L OKE M, KRISTJUHAN K, KRISTJUHAN A.Extraction of genomic DNA from yeasts for PCR-based applications[J].BioTechniques, 2011, 50(5):325-328.
OKE M, KRISTJUHAN K, KRISTJUHAN A.Extraction of genomic DNA from yeasts for PCR-based applications[J].BioTechniques, 2011, 50(5):325-328.
[18] HEIN E M, HAYEN H.Comparative lipidomic profiling of S.cerevisiae and four other hemiascomycetous yeasts[J].Metabolites, 2012, 2(1):254-267.
[19] SANTANA P, RUIZ DE GALARRETA C M, FANJUL L F.Sphingomyelin and ceramide mass assay[J].Methods in Molecular Biology, 1998, 105:223-231.
[20] WENINGER A, FISCHER J E, RASCHMANOV H, et al.Expanding the CRISPR/Cas9 toolkit for Pichia pastoris with efficient donor integration and alternative resistance markers[J].Journal of Cellular Biochemistry, 2018, 119(4):3183-3198.
H, et al.Expanding the CRISPR/Cas9 toolkit for Pichia pastoris with efficient donor integration and alternative resistance markers[J].Journal of Cellular Biochemistry, 2018, 119(4):3183-3198.
[21] TERNES P, WOBBE T, SCHWARZ M, et al.Two pathways of sphingolipid biosynthesis are separated in the yeast Pichia pastoris[J]. Journal of Biological Chemistry, 2011, 286(13):11401-11414.
[22] HAN S M, LONE M A, SCHNEITER R, et al.Orm1 and Orm2 are conserved endoplasmic reticulum membrane proteins regulating lipid homeostasis and protein quality control[J].Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(13):5851-5856.
[23] WANG W, XIN J X, YANG X, et al.Lipid-gene regulatory network reveals coregulations of triacylglycerol with phosphatidylinositol/lysophosphatidylinositol and with hexosyl-ceramide[J].Biochimica et Biophysica Acta.Molecular and Cell Biology of Lipids, 2019, 1864(2):168-180.
[24] JANI S, DA EIRA D, HADDAY I, et al.Distinct mechanisms involving diacylglycerol, ceramides, and inflammation underlie insulin resistance in oxidative and glycolytic muscles from high fat-fed rats[J].Scientific Reports, 2021, 11(1):19160.