猪肉营养丰富,极易被单增李斯特菌污染。单增李斯特菌因其在自然界中广泛存在且对苛刻环境的耐受力强的特性,对肉及肉制品的食用安全造成严重威胁。应用“栅栏技术”,可以从不同方面控制单增李斯特菌,从而提高猪肉的微生物安全。单增李斯特菌导致宿主患病的能力由该菌的生长特性、抗性和毒性共同决定[1]。课题组前期研究结果表明60% CO2+20% O2+20% N2气调包装结合植物乳杆菌(Lactobacillus plantarum)处理(以下简称CO2-LP)可以通过延长单增李斯特菌的迟滞时间(λ)、降低最大比生长速率(μmax)和最大污染浓度(Nmax)抑制单增李斯特菌的生长[2]。
生长特性决定了猪肉被加工处理前单增李斯特菌的污染浓度,对不利环境的抗性影响了单增李斯特菌进入宿主感染周期的剂量。在猪肉加工阶段,热处理是最为常见的杀菌消毒措施[3]之一。热处理可破坏单增李斯特菌细胞结构,影响胞内蛋白质表达[4],从而导致单增李斯特菌失活或者生长受到抑制,因此单增李斯特菌的热抗性影响了猪肉制品入口前该菌的污染浓度。被消费者摄入后,宿主的胃肠道消化液,包括口腔中的唾液、胃中的胃液(强酸)和肠道中的肠液(高渗透压),是单增李斯特菌进入人体感染周期的首要障碍[5]。经过消化系统后存活的单增李斯特菌才能进入宿主的感染周期。研究食源性致病菌在人工模拟胃肠道环境中的生存能力是探讨细菌对不利环境条件抗性常用的办法之一[6]。
单增李斯特菌对不利环境条件的抗性与菌体本身的特性和抗性基因的表达有关。在温和压力环境下,单增李斯特菌的抗性依赖于细胞内大分子物质的缓冲作用和菌体细胞膜(壁)特有的稳态[7]。在恶劣压力条件下,单增李斯特菌会启动自身的抗性应答机制,包括热抗性、酸抗性和渗透压抗性等抗性基因的表达,以抵抗外界压力[8]。上述这些抗性因子均受到SigmaB(sigB)因子的调控。sigB位于RNA聚合酶全酶中,可以识别基因启动子,并且与特定基因启动子的结合位点结合以开启抗性基因的转录进程,进而积累抗性蛋白,协助单增李斯特菌在恶劣环境下生存[9]。
因此,本研究探索猪肉贮存的整个货架期内,即13 ℃贮存1 d和3 d,25 ℃贮存12 h和24 h时,60% CO2-LP处理猪肉中单增李斯特菌对55 ℃(温和热处理)和65 ℃(中低温处理)热处理、模拟唾液、模拟胃液和模拟肠液的耐受性。用实时荧光定量PCR(quantitative real-time PCR, qPCR)方法检测压力调控基因sigB的相对表达量,探究单增李斯特菌抗性变化的机制。
1 材料与方法
1.1 菌种
试验所用单增李斯特菌(ATCC 13932,血清型4b)由本实验室保藏;植物乳杆菌CICC 6257为益生菌,中国工业微生物菌种保藏管理中心(http://www.china-cicc.org/)。
1.2 化学试剂、耗材与培养基
乳杆菌和单增李斯特菌活化所用培养基为脑心浸液(brain heart infusion, BHI)培养基;乳杆菌的选择性增菌培养基为MRS(de Man, Rogosa and Sharpe)肉汤培养基;单增李斯特菌的选择性增菌培养基为胰酪胨大豆酵母浸膏琼脂培养基(tryptic soya agar with yeast extract, TSA-YE)培养基;用于乳杆菌和单增李斯特菌分离计数的培养基分别为MRS琼脂和PALCAM琼脂,以上培养基购于青岛海博生物技术有限公司。
气调保鲜盒、气调保鲜膜,苏州市凯康机械设备有限公司;RNA反转录试剂盒、qPCR反应相关试剂,南京诺唯赞生物技术有限公司。
9500型普通PCR仪、7500型实时荧光定量PCR仪,美国应用生物仪器公司;NanoDropTM2000微量生化测定仪,美国赛默飞世尔公司。
内参基因16S rRNA的引物序列为ACATCCTTTGACCACTCTGGA及CAACATCTCACGACACGAGC,扩增片段的长度为93 bp;sigB基因的引物序列为TTTGGATTGCCGCTTACC和TCGGGCGATGGACTCTACTA,扩增片段的长度为91 bp。2种引物均由本实验室成员前期设计,经验证特异性良好,已成功运用于多项实证研究中[10-12]。模拟人体消化液的成分参考文献[5,13]的研究,具体组成成分及比例见表1。
表1 模拟人体消化道系统的成分组成
Table 1 Composition of the simulated gastrointestinal fluids
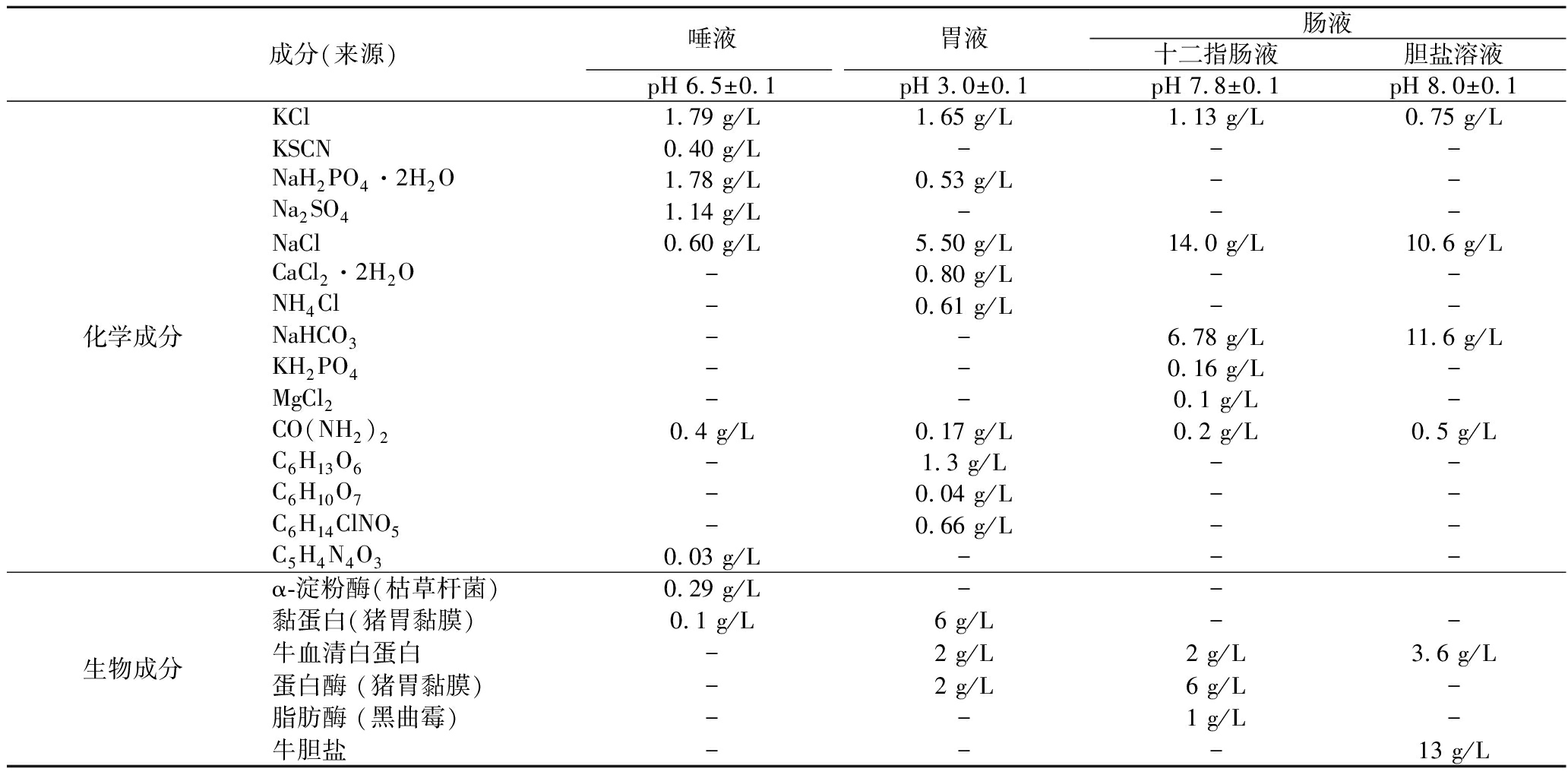
成分(来源)唾液胃液pH 6.5±0.1pH 3.0±0.1肠液十二指肠液胆盐溶液pH 7.8±0.1pH 8.0±0.1化学成分KCl1.79 g/L1.65 g/L1.13 g/L0.75 g/LKSCN0.40 g/L---NaH2PO4·2H2O1.78 g/L0.53 g/L--Na2SO41.14 g/L---NaCl0.60 g/L5.50 g/L14.0 g/L10.6 g/LCaCl2·2H2O-0.80 g/L--NH4Cl-0.61 g/L--NaHCO3--6.78 g/L11.6 g/LKH2PO4--0.16 g/L-MgCl2--0.1 g/L-CO(NH2)20.4 g/L0.17 g/L0.2 g/L0.5 g/LC6H13O6-1.3 g/L--C6H10O7-0.04 g/L--C6H14ClNO5-0.66 g/L--C5H4N4O30.03 g/L---生物成分α-淀粉酶(枯草杆菌)0.29 g/L--黏蛋白(猪胃黏膜)0.1 g/L6 g/L--牛血清白蛋白-2 g/L2 g/L3.6 g/L蛋白酶 (猪胃黏膜)-2 g/L6 g/L-脂肪酶 (黑曲霉)--1 g/L-牛胆盐---13 g/L
1.3 试验方法
1.3.1 细菌培养和接种液制备
试验前一天,用灭菌的接种环分别从TSA-YE和MRS琼脂培养基上挑取一株菌苔,分别接种于100 mL BHI培养基中。单增李斯特菌在37 ℃、110 r/min的摇床上培养18 h,乳杆菌在37 ℃的培养箱中静置培养18 h,使细菌生长到稳定期。然后,分别取10 mL单增李斯特菌和乳杆菌的菌悬液在冷冻离心机中离心收集菌体(4 ℃、8 000 r/min、5 min),菌体分别用10 mL无菌生理盐水重悬,重复两次。最后用无菌生理盐水分别将单增李斯特菌和乳杆菌菌液稀释到105~106 CFU/mL和107~108 CFU/mL左右备用。
1.3.2 猪肉接种、包装及贮藏
猪肉接种步骤:在生物安全柜中,称取已灭菌的猪肉(100 g/份)分装在无菌均质袋中,将猪肉分成两组,一组为对照组(TC),将猪肉接种单增李斯特菌接种液(1 mL)和生理盐水(1 mL)的混合液2 mL;另一组为处理组(T60% CO2-LP),将猪肉接种2 mL乳杆菌(1 mL)和单增李斯特菌(1 mL)的混合液。取两份猪肉接种2 mL无菌生理盐水作为阴性对照。将接种了菌液的猪肉用无菌均质机正反面各拍打2 min,使得菌液在猪肉中均匀分布。单增李斯特菌和乳杆菌的确切初始接种量用平板计数法确认。
在生物安全柜中,将猪肉从无菌均质袋中转移到已经预先紫外辐照杀菌的气调保鲜盒中。TC组和阴性对照组猪肉样品用无菌空气包装,TCO2-LP组猪肉样品进行60% CO2+20% N2+20% O2气调包装。将每组气调包装好的猪肉再随机分成两个亚组,两个亚组中的猪肉分别放置在13 ℃和25 ℃恒温培养箱培养。
1.3.3 单增李斯特菌对温和热处理的耐受性
在猪肉贮存的整个货架期内,即13 ℃贮存1 d和3 d,25 ℃贮存12 h和24 h时取样测定单增李斯特菌的热抗性。在试验前,先对每个取样点猪肉中单增李斯特菌的初始浓度进行计数。
猪肉的热处理在恒温水浴锅中进行,热处理步骤参考JUNEJA等[14]的方法。在试验前,将水浴锅温度调至55 ℃,备用。在每一个取样点,取出猪肉5 g,为使猪肉受热均匀,将猪肉揉成肉丸,放在均质袋底部,将均质袋浸没在热水中加热。预先测得肉丸中心温度升高到55 ℃的时间较短(1 min左右),因此将温度的上升时间记在总加热时间内。热处理完成后,快速的将均质袋拿出来,倒入15 mL冰水中冷却,在30 min内完成单增李斯特菌计数。
1.3.4 单增李斯特菌对中低温热处理的耐受性
为了检测CO2-LP处理后,单增李斯特菌对中低温热处理的耐受性,对猪肉进行65 ℃加热2 min处理。猪肉取样时间与热处理方法同1.3.3节。
1.3.5 单增李斯特菌对模拟人体唾液的耐受性
猪肉取样时间同1.3.3节。模拟人体咀嚼过程如下[5,13]:在试验前将配置好的模拟人类唾液放在水浴锅中保持37 ℃恒温。在每个取样时间,取10 g猪肉放置在无菌均质袋中,加入10 mL模拟唾液,用无菌均质机将猪肉正反面各拍打1 min,使猪肉和模拟唾液混匀。将匀浆的pH值调整到6.5左右,在37 ℃摇床上振荡孵育3 min,然后取出1 mL匀浆,对单增李斯特菌进行计数。计数结果为单增李斯特菌经过模拟唾液之后的存活量,也作为进入模拟人体胃液的单增李斯特菌浓度初始值。
1.3.6 单增李斯特菌对模拟人体胃液的耐受性
人体胃肠道消化是一个连续的过程,因此经过模拟唾液之后的猪肉直接进行模拟胃液消化处理。模拟人体胃液消化过程如下[5,13]:在试验前将配置好的模拟人类胃液放在水浴锅中保持37 ℃恒温。每次模拟咀嚼阶段之后,将19 mL模拟胃液倒入装有猪肉的均质袋中,用均质机将猪肉正反面各拍打1 min,使猪肉和模拟胃液混匀。将匀浆的pH值调整到3.0左右,在37 ℃摇床上振荡孵育2 h,然后取出1 mL匀浆,对单增李斯特菌进行计数。计数结果为单增李斯特菌经过模拟胃液之后的存活量,进入模拟人体肠液的单增李斯特菌浓度初始值。
1.3.7 单增李斯特菌对模拟人体肠液的耐受性
模拟人体肠液消化过程如下[5,13]:在试验前将配置好的模拟人类肠液放在水浴锅中保持37 ℃恒温。每次胃液处理后,将37 mL模拟肠液(29 mL十二指肠溶液和8 mL胆盐溶液)倒入装有猪肉的均质袋中,用无菌均质机将猪肉正反面各拍打1 min,使猪肉和模拟肠液混匀。将匀浆的pH值调整到8.0左右,在37 ℃摇床上振荡孵育2 h,然后取出1 mL匀浆,对单增李斯特菌进行计数。计数结果为单增李斯特菌经过模拟肠液之后的存活量。
1.3.8 单增李斯特菌抗性基因相对表达水平检测
细菌总RNA提取、基因组DNA去除和反转录步骤严格按照RNA反转录试剂盒(HiScript® IIQ RT SuperMix for qPCR(+gDNA wiper)说明书操作。
1.4 数据处理与统计分析
单增李斯特菌的热抗性用公式(1)评估:
(1)
式中:Rh,单增李斯特菌的热抗性;N0,加热前单增李斯特菌的初始浓度,CFU/g;Nh,经过热处理后,单增李斯特菌的浓度,CFU/g;t,加热时间,min。
单增李斯特菌对胃肠道不利环境的抗性用公式(2)评估:
Rg=lg(NI/N0)
(2)
式中:Rg,单增李斯特菌减少量;N0,进入每一个模拟胃肠道消化液前单增李斯特菌的初始浓度,CFU/g;NI,经过每一个胃肠道消化阶段存活下来的单增李斯特菌浓度,CFU/g。
分别计算TCO2-LP和TC组sigB基因与内参基因的差值(ΔCt),然后,比较TCO2-LP和TC组单增李斯特菌sigB基因的检测值差值(ΔΔCt),最后以计算值2-ΔΔCt计算sigB基因在TCO2-LP和TC组单增李斯特菌中的相对表达水平,以表示基因相对表达量。
每个试验重复3次,应用SPSS17.0软件对TCO2-LP和TC组间单增李斯特菌的抗性和及抗性基因表达的差异进行显著性分析。
2 结果与讨论
2.1 单增李斯特菌对温和热处理的抗性
TC和TCO2-LP组单增李斯特菌对55 ℃热处理的抗性见图1。
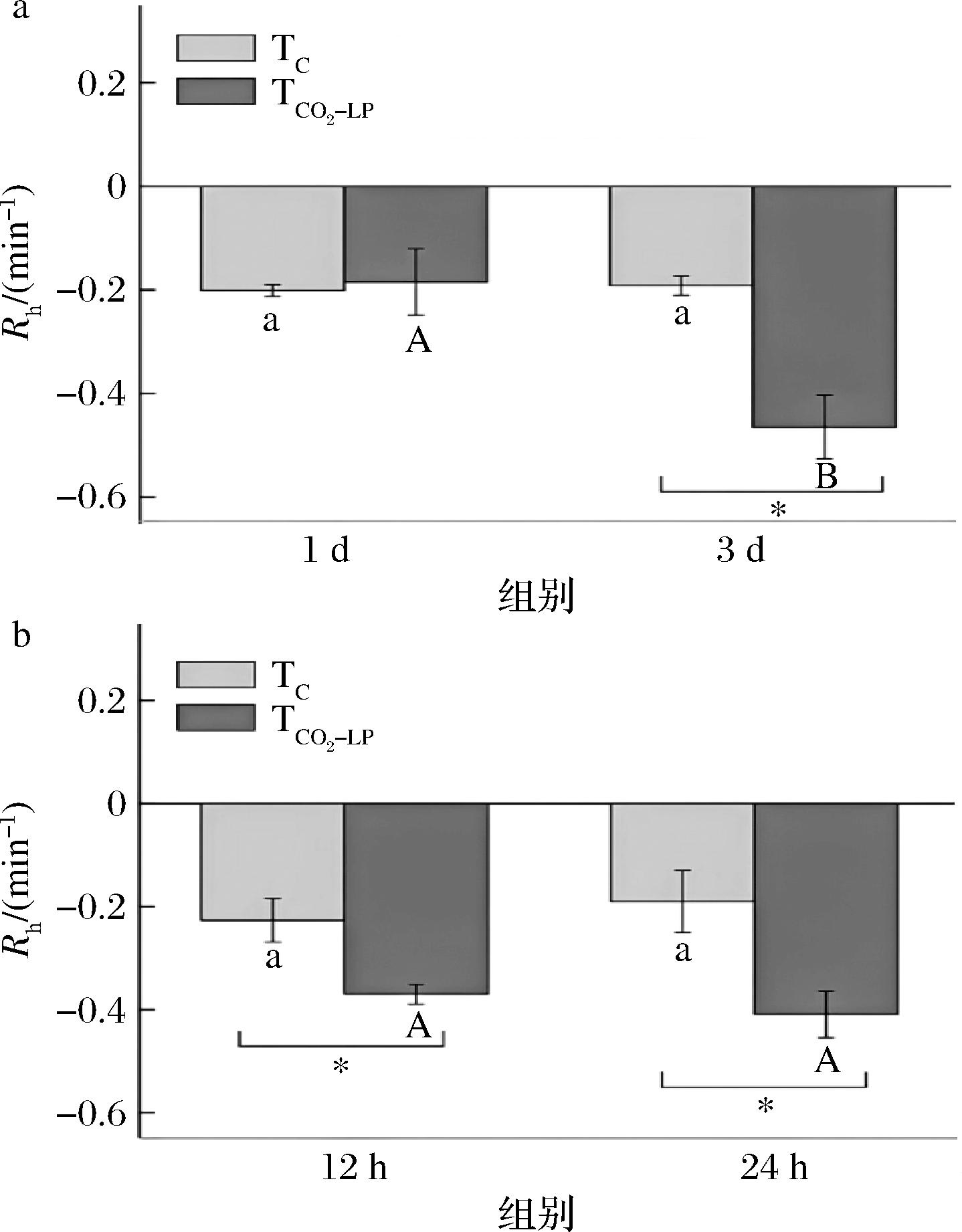
a-13 ℃贮存;b-25 ℃贮存
图1 单增李斯特菌对55 ℃热处理的抗性
Fig.1 The resistance of L.monocytogenes to 55 ℃ heat treatment
注:不同小写和大写字母分别表示TC组和TCO2-LP组单增李斯特菌热抗性有显著差异,*表示TC和TCO2-LP组间单增李斯特菌热抗性有显著差异(P<0.05)(下同)。
由1-a可知,13 ℃贮存1 d时,温和热处理后,TC和TCO2-LP组单增李斯特菌的热抗性无显著差异(P≥0.05);贮存3 d时,TCO2-LP组单增李斯特菌的热抗性显著(P<0.05)低于TC组,TCO2-LP组单增李斯特菌每分钟的减少量是TC组的2.47倍。随着贮存时间的延长,TC组单增李斯特菌对温和热处理的抗性无显著变化(P≥0.05),TCO2-LP组单增李斯特菌的热抗性显著降低(P<0.05)。
如图1-b所示,25 ℃贮存12 h和24 h时,温和热处理后,TCO2-LP组单增李斯特菌每分钟的减少量显著(P<0.05)低于TC组。25 ℃贮存12 h时,温和热处理后,TCO2-LP组单增李斯特菌每分钟的减少量是TC组的1.61倍;贮存24 h时,温和热处理后,TCO2-LP组单增李斯特菌每分钟的减少量是TC组的2.16倍。TC组和TCO2-LP组单增李斯特菌对温和热处理的抗性随着贮存时间的延长均无显著变化(P≥0.05)。
2.2 单增李斯特菌对中低温热处理的抗性
由图2-a可知,13 ℃贮存1 d时,TC和TCO2-LP组单增李斯特菌对中低温热处理的抗性无显著差异(P≥0.05);第3 天,中低温热处理后,TCO2-LP组单增李斯特菌每分钟的减少量是TC组的1.55倍。随着贮存时间延长,TC组单增李斯特菌的热抗性无显著变化(P≥0.05),而TCO2-LP组单增李斯特菌热抗性显著(P<0.05)降低,说明CO2-LP处理可以降低单增李斯特菌对中低温热处理的抗性。
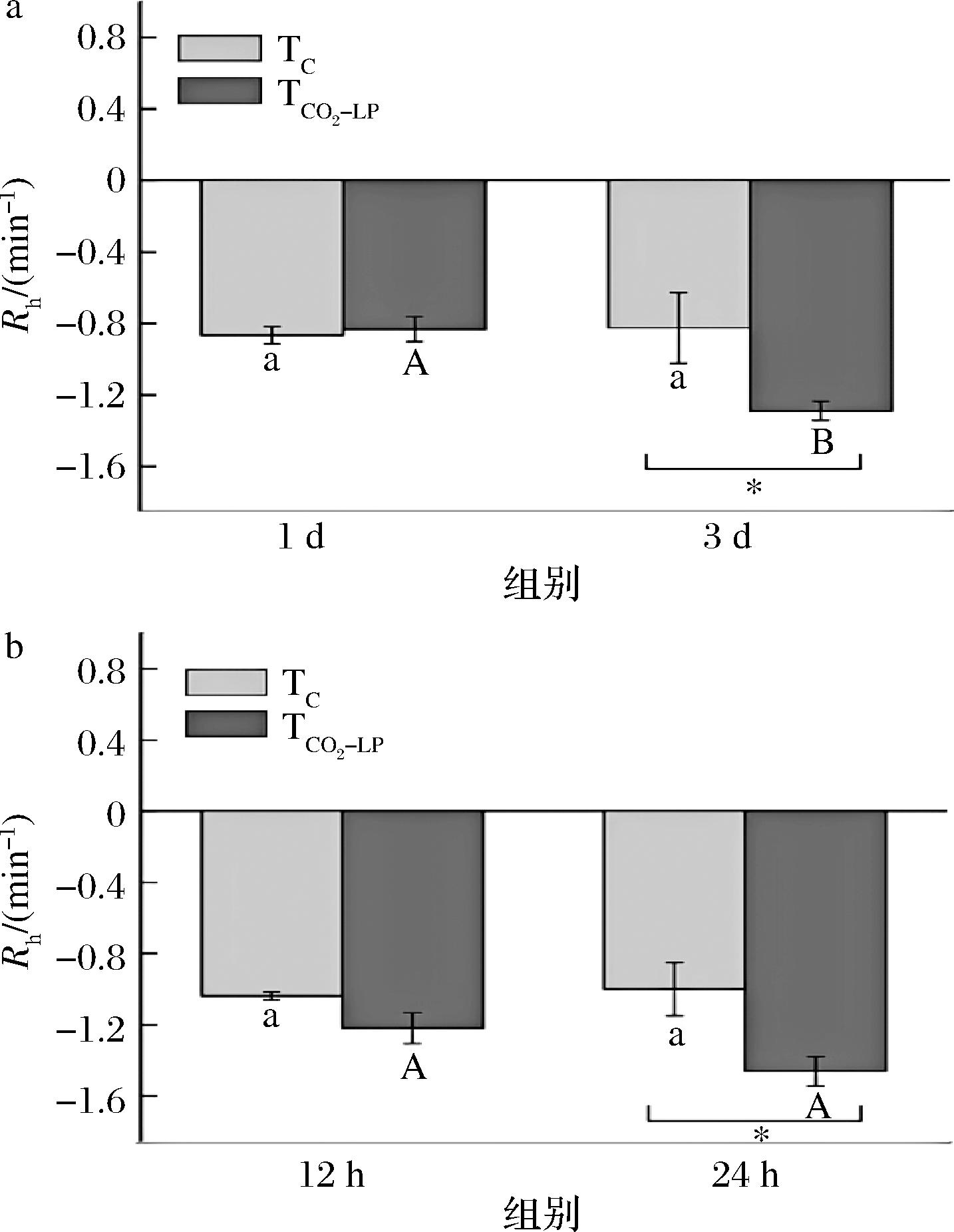
a-13 ℃贮存;b-25 ℃贮存
图2 单增李斯特菌对65 ℃热处理的抗性
Fig.2 The resistance of L.monocytogenes to 65 ℃ heat treatment
由图2-b可知,25 ℃贮存12 h时,尽管热处理后TCO2-LP组单增李斯特菌每分钟的减少量是TC组的1.17倍,TC和TCO2-LP组单增李斯特菌的热抗性无显著差异(P≥0.05)。在贮存24 h时,热处理后TCO2-LP组单增李斯特菌每分钟的减少量是TC组的1.46倍。TC组和TCO2-LP组单增李斯特菌随着贮存时间的延长均无显著性变化(P≥0.05)。值得注意的是,65 ℃加热处理后单增李斯特菌每分钟的减少量远远高于55 ℃热处理,因此较高温度的杀菌处理能够有效降低致病菌的危害。
食品基质中的添加物可能会影响单增李斯特菌的抗性。如IGLESIAS等[15]的研究结果表明生物保护菌株鼠李糖乳杆菌能够降低单增李斯特菌在胃肠道系统中的生存能力。YEHIA等[16]指出,在猪肉和猪肉制品中添加高浓度的NaCl会增加单增李斯特菌的热抗性。本研究结果表明CO2-LP处理猪肉可以降低13 ℃贮存3 d及25 ℃贮存12 h和24 h时单增李斯特菌的热抗性。
2.3 单增李斯特菌对模拟唾液的抗性
由图3可知,13 ℃贮存猪肉的整个货架期内,经过模拟唾液阶段后,TC组单增李斯特菌的存活率几乎为100%。25 ℃贮存12 h和24 h时,经过模拟唾液阶段后,单增李斯特菌的减少量非常低,分别为0.03和0.02 lg CFU/g,这可能是对单增李斯特菌进行计数时不可避免产生的误差。研究结果表明,口腔咀嚼阶段不能杀死单增李斯特菌,不能减少消费者摄入单增李斯特菌的量。另外,和TC组相比,CO2-LP处理对单增李斯特菌的唾液耐受性没有显著(P≥0.05)影响。
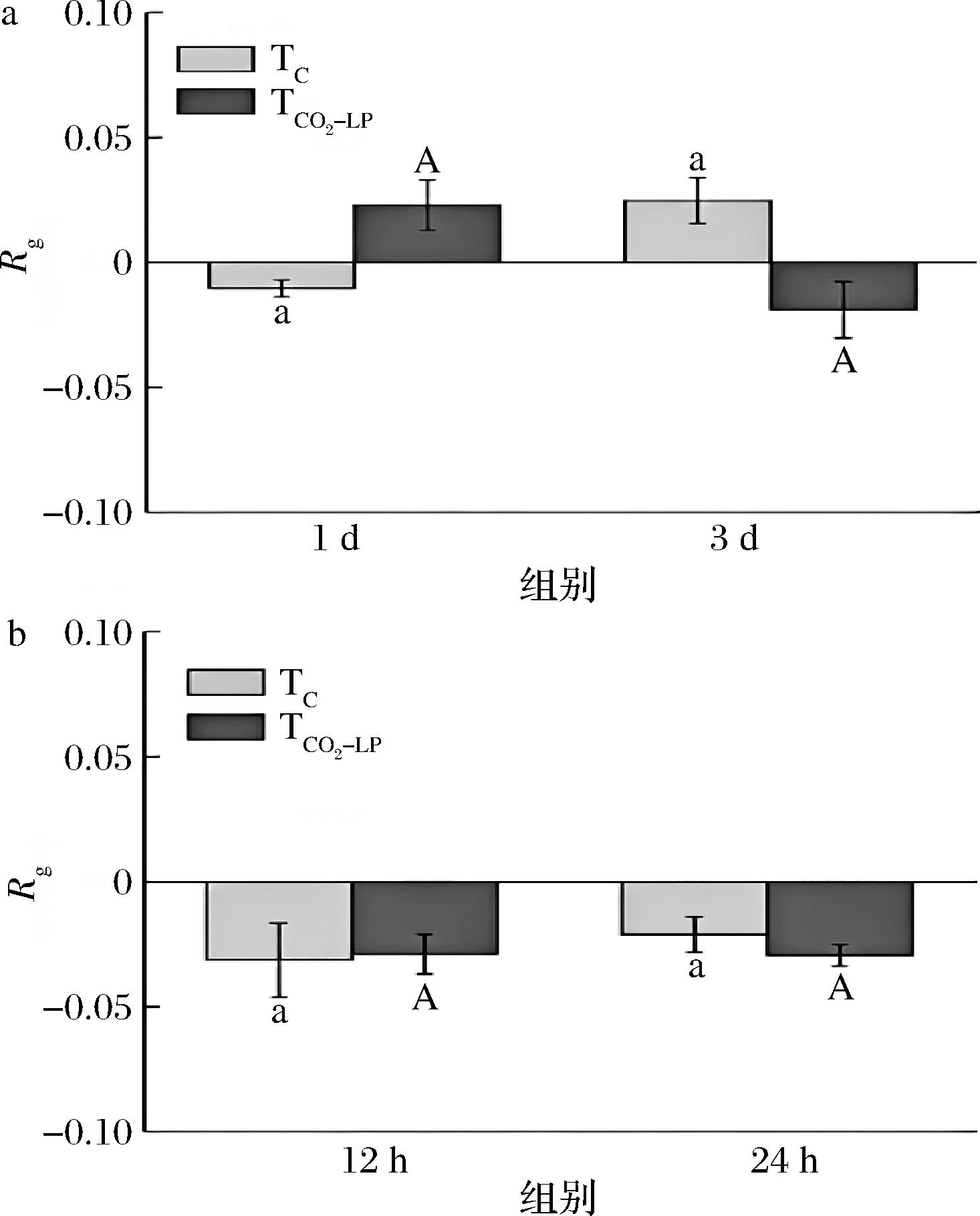
a-13 ℃贮存;b-25 ℃贮存
图3 单增李斯特菌对模拟人体唾液的抗性
Fig.3 The resistance of L.monocytogenes to simulated salivary fluid
研究表明口腔咀嚼阶段不能杀死单增李斯特菌,这和COL S-MED
S-MED 等[5]的研究一致,后者研究了甜瓜和梨中单增李斯特菌对模拟人体消化道中的抗性,结果发现2种水果上单增李斯特菌在口腔咀嚼阶段的浓度几乎没有减少。IGLESIAS等[15]研究了生物防护菌鼠李糖乳杆菌对单增李斯特菌唾液耐受性的影响,也发现鼠李糖乳杆菌对单增李斯特菌的唾液耐受性没有显著影响。这可能和唾液淀粉酶的特性有关,唾液淀粉酶本身是一种水解酶,可作用于直链淀粉和支链淀粉(如猪肉中含的淀粉)产生麦芽糖和少量葡萄糖,而单增李斯特菌可以利用麦芽糖和葡萄糖,提供自身新陈代谢所需能量。因此,咀嚼过程并不能减少人体摄入单增李斯特菌的剂量。
等[5]的研究一致,后者研究了甜瓜和梨中单增李斯特菌对模拟人体消化道中的抗性,结果发现2种水果上单增李斯特菌在口腔咀嚼阶段的浓度几乎没有减少。IGLESIAS等[15]研究了生物防护菌鼠李糖乳杆菌对单增李斯特菌唾液耐受性的影响,也发现鼠李糖乳杆菌对单增李斯特菌的唾液耐受性没有显著影响。这可能和唾液淀粉酶的特性有关,唾液淀粉酶本身是一种水解酶,可作用于直链淀粉和支链淀粉(如猪肉中含的淀粉)产生麦芽糖和少量葡萄糖,而单增李斯特菌可以利用麦芽糖和葡萄糖,提供自身新陈代谢所需能量。因此,咀嚼过程并不能减少人体摄入单增李斯特菌的剂量。
2.4 单增李斯特菌对模拟胃液的抗性
由图4-a可知,13 ℃贮存1 d时,TC和TCO2-LP组单增李斯特菌对胃酸的抗性无显著差异(P≥0.05)。在第3 天,和TC组相比,TCO2-LP组单增李斯特菌对胃酸的抗性显著降低(P<0.05);经过胃酸处理后,TCO2-LP组单增李斯特菌的减少量是TC组的3.31倍。随着贮存时间的延长,TC组单增李斯特菌的胃酸耐受性无显著变化(P≥0.05),而TCO2-LP组单增李斯特菌的胃酸耐受性显著降低(P<0.05),说明CO2-LP处理显著(P<0.05)降低了单增李斯特菌的胃酸耐受性。
a-13 ℃贮存;b-25 ℃贮存
图4 单增李斯特菌对模拟人体胃液的抗性
Fig.4 The resistance of L.monocytogenesto simulated gastric fluid
如图4-b所示,25 ℃贮存时,经胃液处理后,和TC组相比,TCO2-LP组单增李斯特菌在12 h和24 h后对胃酸的抗性都显著降低(P<0.05)。在12 h,TCO2-LP组单增李斯特菌的减少量是TC组的2.85倍。在24 h,TCO2-LP组单增李斯特菌的减少量是TC组的3.14倍。随着贮存时间的延长,TCO2-LP组单增李斯特菌的胃酸耐受性无显著变化(P≥0.05),说明CO2-LP处理在25 ℃贮存的初始阶段就已经起到降低单增李斯特菌胃酸抗性的作用。
TC组单增李斯特菌经过胃液之后浓度减少,说明胃消化阶段能够杀死一部分单增李斯特菌。BARMPALIA-DAVIS等[17]的研究也表明,香肠中的单增李斯特菌在胃液中(pH=2)孵育90 min后,浓度降低了1.3 lg CFU/g。另外,两种温度条件下,在猪肉贮存后期,单增李斯特菌的耐胃酸能力比贮存前期强,这是因为随着时间的延长,单增李斯特菌进入猪肉内部,猪肉中的脂肪、蛋白质和碳水化合物对单增李斯特菌起到保护作用。研究表明,BHI液体培养基中不同单增李斯特菌株经胃液孵育后,浓度均降低5 lg CFU/g左右。而先前的研究表明鸡肉[19]和奶酪[20]的单增李斯特菌在经过胃消化后浓度都有一定降低,但是生存率在90%以上。
研究结果表明CO2-LP处理主要在13 ℃贮存3 d、25 ℃贮存12 h和24 h时,显著降低(P<0.05)单增李斯特菌在模拟胃液中的生存能力。IGLESIAS等[15]的研究也表明将乳酸菌直接涂抹在食品表面,不仅能够抑制单增李斯特菌的生长,还能够降低单增李斯特菌在模拟人体胃液中的生存能力。MIRANDA等[21]发现乳酸乳球菌可通过减少谷氨酸脱氢酶基因的表达,从而降低单增李斯特菌的耐酸性。
2.5 单增李斯特菌对模拟肠液的抗性
由图5可知,在13 ℃和25 ℃贮存的整个货架期内,TC组的单增李斯特菌在经过模拟肠液中都能存活,甚至还缓慢生长。由图5-a可知,在13 ℃贮存第3 天时,CO2-LP处理显著(P<0.05)降低了单增李斯特菌的肠液耐受性,对照组单增李斯特菌增加了0.09 lg CFU/g,TCO2-LP组单增李斯特菌经过肠液处理后浓度降低了0.84 lg CFU/g。由图5-b可知,25 ℃贮存12 h和24 h时,TCO2-LP组的单增李斯特菌经过模拟肠液后的浓度分别降低了0.62 lg CFU/g和0.74 lg CFU/g。CO2-LP处理主要在13 ℃贮存3 d及25 ℃贮存12 h和24 h时,显著降低(P<0.05)单增李斯特菌在模拟肠液中的生存能力。
a-13 ℃贮存;b-25 ℃贮存
图5 单增李斯特菌对模拟人体肠液的抗性
Fig.5 The resistance of L.monocytogenes to simulated intestinal fluid
研究发现,在经过模拟肠液后,TC组的单增李斯特菌都能存活,甚至还缓慢生长,这和鲜切梨[5]和牛肉香肠[17]中的单增李斯特菌对肠液有很好的耐受性的结果一致。而BARBOSA等[18]和CUNHA等[6]的研究则表明营养肉汤中的单增李斯特菌在经过模拟肠液之后,浓度下降到检测限以下。固体基质对细菌有一定的保护作用,液体食品中的单增李斯特菌更容易受到外界环境胁迫的影响。另外,本研究所用菌株为血清型4b,属于致人类患李斯特菌病能力较强的血清型,说明该菌株在人体消化道中的生存能力较强,因此对小肠液的抗性较高。
研究结果表明,CO2-LP在13 ℃贮存1 d时对单增李斯特菌的肠液耐受性没有显著影响(P≥0.05),在13 ℃贮存3 d及25 ℃贮存12 h和24 h时,显著降低单增李斯特菌在模拟肠液中的生存能力(P<0.05)。单增李斯特菌对恶劣环境条件的应答依赖于热抗性、酸抗性和渗透压抗性等抗性基因的表达,这些抗性因子均受到sigB因子的调控,可从分子水平探究单增李斯特菌抗性变化的机制。
2.6 单增李斯特菌抗性基因的相对表达水平
由图6-a可知,13 ℃贮存1 d时,TC和TCO2-LP组单增李斯特菌sigB基因的相对表达量无显著差异(P≥0.05);贮存3 d时,和TC组相比,TCO2-LP组单增李斯特菌sigB基因的相对表达量下降了33%。由图6-b可知,25 ℃贮存12 h和24 h时,和TC组相比,TCO2-LP组单增李斯特菌sigB相对表达量分别下调了41%和48%;这和CO2-LP对单增李斯特菌热抗性、胃液和肠液耐受性的影响趋势一样。YE等[22]的研究表明粪肠球菌产生的细菌素能够降低单增李斯特菌胆盐水解酶基因及sigB基因的表达。MIRANDA等[21]的研究发现,将产乳酸链球菌素的乳酸乳球菌添加到牛奶中能够降低单增李斯特菌sigB基因的表达。刘梅等[23]的研究也指出,绿色魏斯氏菌能够降低单增李斯特菌sigB基因的表达。本研究也表明,60% CO2-LP处理引起单增李斯特菌压力调控基因sigB下调是单增李斯特菌抗性降低的主要原因。
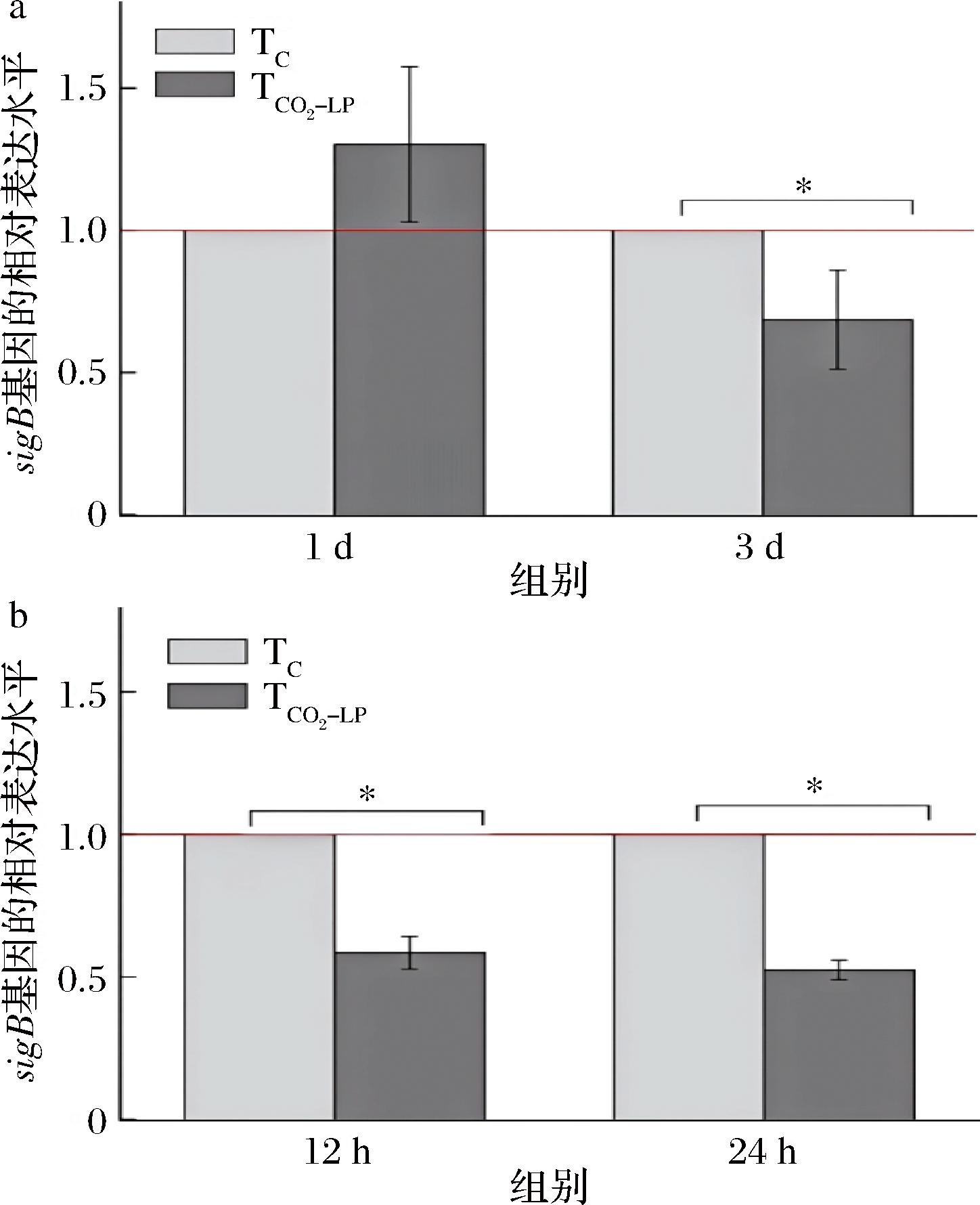
a-13 ℃贮存;b-25 ℃贮存
图6 单增李斯特菌sigB基因的相对表达水平
Fig.6 The relative expression level of sigB in L.monocytogenes
注:*表示TC和TCO2-LP组单增李斯特菌sigB基因的相对表达水有显著差异(P<0.05)
3 结论
CO2-LP处理能够抑制单增李斯特菌的生长,并降低猪肉中单增李斯特菌对不利环境条件的抗性,从而降低单增李斯特菌进入宿主感染周期的剂量。然而进入宿主感染周期后,单增李斯特菌的毒性大小与宿主患病的概率密切相关,因此后续亟待研究60% CO2-LP处理对增李斯特菌毒性的影响,最终为CO2联合益生菌应用到食品中降低单增李斯特菌危害、提高食品的微生物安全提供理论支持和科学依据。
[1] 张文敏, 董庆利, 辛宝, 等.猪肉制品中单增李斯特菌污染现状及致病力控制分析[J].食品与发酵工业, 2023, 49(17):299-307.ZHANG W M, DONG Q L, XIN B, et al.Contamination status and pathogenicity control of Listeria monocytogenes in pork products[J].Food and Fermentation Industries, 2023, 49(17):299-307.
[2] 张文敏, 董庆利, 刘阳泰, 等.二氧化碳联合益生菌对猪肉中单增李斯特菌抑制模型的建立[J].微生物学通报, 2024, 51(3):910-920.ZHANG W M, DONG Q L, LIU Y T, et al.Modeling for the inhibition of CO2 combined with probiotic on Listeria monocytogenes in pork[J].Microbiology China, 2024, 51(3):910-920.
[3] NALBONE L, SORRENTINO G, GIARRATANA F, et al.Effects of osmotic stress on Listeria monocytogenes ATCC 7644:Persistent cells and heat resistance[J].Italian Journal of Food Safety, 2023, 12(1):10880.
[4] OMORI Y, MIAKE K, NAKAMURA H, et al.Influence of lactic acid and post-treatment recovery time on the heat resistance of Listeria monocytogenes[J].International Journal of Food Microbiology, 2017, 257:10-18.
[5] COL S-MED
S-MED P, ABADIAS M, OLIVEIRA M, et al.Influence of fruit matrix and storage temperature on the survival of Listeria monocytogenes in a gastrointestinal simulation[J].Food Control, 2017, 73:1045-1052.
P, ABADIAS M, OLIVEIRA M, et al.Influence of fruit matrix and storage temperature on the survival of Listeria monocytogenes in a gastrointestinal simulation[J].Food Control, 2017, 73:1045-1052.
[6] CUNHA S, KOMORA N, MAGALH ES R, et al.Characterization of clinical and food Listeria monocytogenes isolates with different antibiotic resistance patterns through simulated gastrointestinal tract conditions and environmental stresses[J].Microbial Risk Analysis, 2016, 1:40-46.
ES R, et al.Characterization of clinical and food Listeria monocytogenes isolates with different antibiotic resistance patterns through simulated gastrointestinal tract conditions and environmental stresses[J].Microbial Risk Analysis, 2016, 1:40-46.
[7] EARNSHAW R G, APPLEYARD J, HURST R M.Understanding physical inactivation processes:Combined preservation opportunities using heat, ultrasound and pressure[J].International Journal of Food Microbiology, 1995, 28(2):197-219.
[8] SCHÖFFL F, PR NDL R, REINDL A.Regulation of the heat-shock response[J].Plant Physiology, 1998, 117(4):1135-1141.
NDL R, REINDL A.Regulation of the heat-shock response[J].Plant Physiology, 1998, 117(4):1135-1141.
[9] SA IBUSQUIZA P, NIEROP GROOT M, DEB
IBUSQUIZA P, NIEROP GROOT M, DEB N-VALLES A, et al.Impact of growth conditions and role of sigB on Listeria monocytogenes fitness in single and mixed biofilms cultured with Lactobacillus plantarum[J].Food Research International, 2015, 71:140-145.
N-VALLES A, et al.Impact of growth conditions and role of sigB on Listeria monocytogenes fitness in single and mixed biofilms cultured with Lactobacillus plantarum[J].Food Research International, 2015, 71:140-145.
[10] LIU W K, LI S, CHEN G W, et al.Construction of inlA &inlB genes deletion strain of Listeria monocytogenes[J].Journal of Microbiology, 2017, 37 (1):64-69.
[11] 曾海娟, 刘武康, 谢曼曼, 等.单核细胞性李斯特菌基因dal的敲除及其生物学特性初步分析[J].食品科学, 2017, 38(22):48-53.ZENG H J, LIU W K, XIE M M, et al.Knockout of dal gene and its effect on Listeria monocytogenes[J].Food Science, 2017, 38(22):48-53.
[12] 陈国薇, 刘武康, 丁承超, 等.单增李斯特菌(EGDe)sigB基因缺失株的构建及其生物特性的初步鉴定[J].现代食品科技, 2017, 33(9):102-108.CHEN G W, LIU W K, DING C C, et al.Construction of attenuated Listeria monocytogenes strain EGDe-△sigB and preliminary identification of biological activity of sigB[J].Modern Food Science and Technology, 2017, 33(9):102-108.
[13] OOMEN A G, ROMPELBERG C J M, BRUIL M A, et al.Development of an in vitro digestion model for estimating the bioaccessibility of soil contaminants[J].Archives of Environmental Contamination and Toxicology, 2003, 44(3):281-287.
[14] JUNEJA V K, ALTUNTA E G, AYHAN K, et al.Predictive model for the reduction of heat resistance of Listeria monocytogenes in ground beef by the combined effect of sodium chloride and apple polyphenols[J].International Journal of Food Microbiology, 2013, 164(1):54-59.
E G, AYHAN K, et al.Predictive model for the reduction of heat resistance of Listeria monocytogenes in ground beef by the combined effect of sodium chloride and apple polyphenols[J].International Journal of Food Microbiology, 2013, 164(1):54-59.
[15] IGLESIAS M B, VI AS I, COL
AS I, COL S-MED
S-MED P, et al.Adhesion and invasion of Listeria monocytogenes and interaction with Lactobacillus rhamnosus GG after habituation on fresh-cut pear[J].Journal of Functional Foods, 2017, 34:453-460.
P, et al.Adhesion and invasion of Listeria monocytogenes and interaction with Lactobacillus rhamnosus GG after habituation on fresh-cut pear[J].Journal of Functional Foods, 2017, 34:453-460.
[16] YEHIA H M, ELKHADRAGY M F, ALJAHANI A H, et al.Prevalence and antibiotic resistance of Listeria monocytogenes in camel meat[J].Bioscience Reports, 2020, 40(6):BSR20201062.
[17] BARMPALIA-DAVIS I M, GEORNARAS I, KENDALL P A, et al.Survival of Listeria monocytogenes in a simulated dynamic gastrointestinal model during storage of inoculated Bologna and Salami slices in vacuum packages[J].Journal of Food Protection, 2008, 71(10):2014-2023.
[18] BARBOSA J, BORGES S, MAGALH ES R, et al.Behaviour of Listeria monocytogenes isolates through gastro-intestinal tract passage simulation, before and after two sub-lethal stresses[J].Food Microbiology, 2012, 30(1):24-28.
ES R, et al.Behaviour of Listeria monocytogenes isolates through gastro-intestinal tract passage simulation, before and after two sub-lethal stresses[J].Food Microbiology, 2012, 30(1):24-28.
[19] PETERSON L D, FAITH N G, CZUPRYNSKI C J.Resistance of Listeria monocytogenes F2365 cells to synthetic gastric fluid is greater following growth on Ready-to-Eat deli Turkey meat than in brain heart infusion broth[J].Journal of Food Protection, 2007, 70(11):2589-2595.
[20] MELO J, SCHRAMA D, ANDREW P W, et al.Proteomic analysis shows that individual Listeria monocytogenes strains use different strategies in response to gastric stress[J].Foodborne Pathogens and Disease, 2013, 10(2):107-119.
[21] MIRANDA R O, CAMPOS-GALV O M E M, NERO L A.Expression of genes associated with stress conditions by Listeria monocytogenes in interaction with nisin producer Lactococcus lactis[J].Food Research International, 2018, 105:897-904.
O M E M, NERO L A.Expression of genes associated with stress conditions by Listeria monocytogenes in interaction with nisin producer Lactococcus lactis[J].Food Research International, 2018, 105:897-904.
[22] YE K P, ZHANG X X, HUANG Y, et al.Bacteriocinogenic Enterococcus faecium inhibits the virulence property of Listeria monocytogenes[J].LWT, 2018, 89:87-92.
[23] 刘梅, 黄艳, 刘佳, 等.绿色魏斯氏菌对单增李斯特菌毒力特性的影响[J].南京农业大学学报, 2018, 41(4):736-741.LIU M, HUANG Y, LIU J, et al.Effect of Weissella viridescens on the virulence property of Listeria monocytogenes[J].Journal of Nanjing Agricultural University, 2018, 41(4):736-741.