合成生物学通过重塑生物系统并建立新的遗传回路从而实现对目的产品的精细调控。当前,基于控制遗传回路的启动子工程、信号肽工程、终止子工程、转录因子工程实现了对特定信号(例如糖醇,渗透压,温度)的输出控制,例如ON-OFF、放大、缩小。然而,目前对基于复制原点的遗传回路却鲜有研究,从而限制了地衣芽孢杆菌中生物学元件的应用。质粒是介导微生物遗传改造的关键性工具,在天然状态下,质粒主要分布于古细菌、细菌和真菌等生物领域。它们通过重组或转座的方式,整合并传递基因,推动细菌种群间的遗传交换过程。更重要的是,质粒所携带的基因能赋予宿主菌新的表型,使其更好地适应生存环境[1-2]。环状质粒有θ复制、滚环复制与链替换复制3种复制机制。质粒的复制原点含有重复序列,该序列不仅可以作为质粒编码复制起始蛋白的结合位置,并且具有控制质粒复制起始的功能。在基因表达过程中,质粒编码的复制起始蛋白能特异性地识别并结合复制原点上的特定重复序列,确保了质粒独立于细菌染色体外的自主复制,并且在质粒拷贝数跟稳定性的控制中发挥着至关重要的作用[3]。
在广寄主范围质粒中,已经描述了几种复制原点的重复序列,如P1[4]、F[5]、R6K[6]等。在θ复制的宽宿主范围质粒(pCU1和pSa)以及在条件宽宿主范围的质粒(pPS10)中也发现了重复序列。复制起始蛋白结合的重复序列不限于通过θ机制复制的质粒,因为已经报道了在链置换机制或滚环复制机制的质粒中也存在这些序列[7-8]。地衣芽孢杆菌作为一种高效的异源基因表达系统,具备了发酵周期短、酶系丰富和强蛋白分泌能力等优点。此外,它还被认定为食品安全级菌株。该菌株已用于生产多种物质,如肽类抗生素(杆菌肽)、有机酸(柠檬酸)和聚合物(聚谷氨酸)等[9]。此外,地衣芽孢杆菌在水产养殖、污染防治、生物医学和工业酶制剂生产等领域均有广泛应用[10]。
本研究选取了麦芽寡糖基海藻糖合成酶作为指示产物。2003年,KOBAYASHI等[11]研究者解析了来自Sulfolobus acidocaldarius ATCC 33909的麦芽寡糖基海藻糖合成酶(MTSase)的晶体结构。该酶由3个结构域组成,分别是Domain A、B和C。其中,Domain A和B共同构成了酶的催化中心,1个(β/α)8桶状结构。这个桶状结构的活性位点位于中央β桶的C末端,并包含了一些保守的氨基酸催化残基[11]。这些催化残基可以作用于麦芽寡糖,通过分子内转糖基作用将麦芽寡糖转化为麦芽寡糖基海藻糖,从而作用于海藻糖的高效合成。海藻糖作为细胞膜的稳定剂和生物体的保护剂,具有在极端环境下维持生物大分子稳定性的作用,这一特点使其被广泛应用于医疗、化妆品领域[12]。本研究基于文献调研挖掘天然质粒的关键复制元件-复制原点,然后以拷贝数,稳定性为参考指标,建立了质粒特性在地衣芽孢杆菌中的评价方法。进一步利用筛选得到的高效复制原点pS5,实现了麦芽寡糖基海藻糖合成酶基因在地衣芽孢杆菌中的高水平表达,并且研究了复制原点的茎环结构对于产酶的影响。新的强复制原点的开发与应用为地衣芽孢杆菌表达系统的改进和完善奠定了基础。
1 材料与方法
1.1 菌株和质粒
本研究所用的菌株、复制原点和质粒如表1所示。
表1 本研究使用的菌株、复制原点和质粒
Table 1 Strains,origin of replications and plasmids used in this study

菌株或质粒相关特性来源菌株JM109Escherichiacoli,usedforgenecloning本实验室保藏B.licheniformisCICIMB1522B.licheniformisCICIMB1391knockedoutα-amylasegeneamyLandalkalineproteasegeneaprE本实验室保藏BLK0B.licheniformisCICIMB1522withplasmidpS0本实验室保藏BLK1B.licheniformisCICIMB1522withrecombinantplasmidpS1本研究构建BLK2B.licheniformisCICIMB1522withrecombinantplasmidpS2本研究构建BLK3B.licheniformisCICIMB1522withrecombinantplasmidpS3本研究构建BLK4B.licheniformisCICIMB1522withrecombinantplasmidpS4本研究构建BLK5B.licheniformisCICIMB1522withrecombinantplasmidpS5本研究构建BLK6B.licheniformisCICIMB1522withrecombinantplasmidpS6本研究构建BLT1B.licheniformisCICIMB1522withrecombinantplasmidpT1本研究构建BLT2B.licheniformisCICIMB1522withrecombinantplasmidpT2本研究构建BLT3B.licheniformisCICIMB1522withrecombinantplasmidpT3本研究构建BLT4B.licheniformisCICIMB1522withrecombinantplasmidpT4本研究构建复制原点pAM1fromB.licheniformisCICIMB1522本实验室保藏ORIH1ORIT5withastemringaddedontherightside上海生工合成ORIH2ORIT5withastemringaddedinthemiddle上海生工合成ORIH3ORIT5withastemringaddedontheleftside上海生工合成ORIH4removeORIT5fromonestemring上海生工合成质粒pS0PHY-PLK300,withSulfolobusacidocaldariusderivedMTSasegenesTreYandPP2本实验室保藏pS1ORIT1replacespAM1ofpS0本研究构建pS2ORIT2replacespAM1ofpS0本研究构建pS3ORIT3replacespAM1ofpS0本研究构建pS4ORIT4replacespAM1ofpS0本研究构建pS5ORIT5replacespAM1ofpS0本研究构建pS6ORIT6replacespAM1ofpS0本研究构建pT1PS5withastemringaddedtotherightsideofORIT5本研究构建pT2PS5withastemringaddedinthemiddleofORIT5本研究构建pT3PS5withastemringaddedtotheleftsideofORIT5本研究构建pT4ORIT5RemovepS5fromonestemring本研究构建
1.2 主要试剂
2×Taq PCR Master Mix、2×Pfu PCR Master Mix,杭州宝赛公司;限制性核酸内切酶,美国Thermo公司;2×Phanta Max Master Mix、质粒提取试剂盒、DNA纯化试剂盒、DNA凝胶回收试剂盒,南京诺维赞生物科技股份有限公司;卡那霉素、氨苄青霉素、四环素,美国Sigma-Aldrich公司;麦芽糊精(葡萄糖当量值为12),罗盖特(中国)精细化工有限公司;蛋白胨、酵母粉、琼脂粉,英国OXOID公司;其余试剂购自上海国药集团有限公司。
1.3 培养基
LB培养基(g/L):酵母粉5,NaCl 10,蛋白胨10(固体培养基中加质量分数为1.5%的琼脂粉)。
TB培养基(g/L,pH 7.0);酵母粉24,蛋白胨12,KH2PO4 2.3,K2HPO4 12.54,甘油5。
发酵培养基(g/L,pH 7.5):棉籽蛋白30,蔗糖90,K2HPO4·3H2O 9.12,KH2PO41.36,FeCl30.5,(NH4)2HPO410。
在菌株构建和摇瓶发酵过程中,培养基中添加的氨苄青霉素和四环素终质量浓度分别为100、20 μg/mL。
1.4 引物
引物由上海天霖生物科技有限公司合成(表2)。
表2 本研究中所涉及引物
Table 2 Primers used in this study

引物 引物序列(5′-3′)长度/bpBLK1-FCCGTTTGTTGAACTAAATAAAACCCGCACTATGCCATTAC40BLK1-RAACTAAATAGACGTATCATAGATATAAATGTAATGGCATAGTGCG45BLK2-FAACTACGTTTGTTGAACTACTCTTTAATAAAATAATTTTTCCG43BLK2-RAATAGTGAAGAAAAGAATTTGAAACGTTTGTCTGATTT38BLK3-FTTTGTTGAACTAATCCTCTATCCTCTGATGCTGGAGT37BLK3-RGTTTCTTCTCTTGTTAACAATTTGTTAACAAGATTATTCTC41BLK4-FAACTAAACTTGATTATAGCGACACATTAGAACACG35BLK4-RAACTAAATAGACACATTCGCATTGATTAACCGC33BLK5-FATGGCCCGTTTGTTGAACTAACGCGCCCTGTAGCG35BLK5-RAAATTGTAAACGTTAATATTTTGTTAAAATTCGCGTTAAAT41BLK6-FTGTTGAACTATGTTCTCCCAATTATAGTTCTATTCTTTCTCTATTAAGG49BLK6-RCCAAACAAATAACTAAATAGCGGTAAAACTCCCAAAACAGGCAA44BLT-FTGGCCCGTTTGTTGAACTAACGCGCCCTGTAGCG34BLT-RCAAATAACTAAATAGAAATTGTAAGCGTT29BLA1-FTATGATACGTCTATTTAGTTATTTGTTTGGTCACTTATAACTTTAACAGA50BLA1-RGGCATAGTGCGGGTTTTATTTAGTTCAACAAACGGGCCATATTGT45BLA2-FCTTCACTATTTAGTTATTTGTTTGGTCACTTATAACTTTAACAGATGGG49BLA2-RAGAGTAGTTCAACAAACGTAGTTCAACAAACGGGCCATATTGTTGTA47BLA3-FAGAAACCTATTTAGTTATTTGTTTGGTCACTTATAACTTTAACAGAT45BLA3-RCAGAGGATAGAGGATTAGTTCAACAAACGGGCCATATTGTT42BLA4-FATGTGTCTATTTAGTTATTTGTTTGGTCACTTATAACTTTAACAGATGG49BLA4-RTGTGTCGCTATAATCAAGTTTAGTTCAACAAACGGGCCATAT42BLA5-FTTTACAATTTCTATTTAGTTATTTGTTTGGTCACTTATAACTTTAACAG50BLA5-RATGCGCCGCTACAGGGCGCGTTAGTTCAACAAACGGGCCATATTGT46BLA6-FTTTACCGCTATTTAGTTATTTGTTTGGTCACTTATAACTTTAACAGATG49BLA6-RCTATAATTGGGAGAACATAGTTCAACAAACGGGCCATATTGTTGT45rpsE-FTGGTCGTCGTTTCCGCTTCG20rpsE-RTCGCTTCTGGTACTTCTTGTGCTT24
1.5 重组表达质粒的构建
根据NCBI公布的各基因及载体序列设计引物(表3)。目的基因扩增方法以pS1为例,使用Pfu DNA聚合酶以ORIT1为目的基因,使用引物BLK1-F/BLK1-R扩增并纯化回收得到目的片段,反应条件及反应体系参考Pfu DNA聚合酶使用说明书。通过同源重组将扩增的复制原点片段连接到线性化的载体上,连接产物转化至E.coli JM109,在氨苄青霉素抗性平板上筛选阳性转化子,提取质粒经过测序及酶切验证,具体条件参照此文献[13],最终获得重组质粒pS1,而后在甘油管中保藏菌种。通过PCR扩增得到带有同源臂的ORIT2~ORIT6,复制原点片段通过同源重组连接到线性化的载体上,获得重组质粒pS2~pS6。通过PCR扩增得到ORIH1~ORIH4片段,这些片段经KpnⅠ和SalⅠ双酶切,连接至同样酶切的pS5线性载体上,获得重组质粒pT1~pT4。
表3 六种复制原点
Table 3 Six origin of replication

复制原点来源序列(5′-3′)ORIT1BacillusnattoCGTTTGTTGAACTACTCTTTAATAAAATAATTTTTCCGTTCCCAATTCCACATTGCAATAATAGAAAATCCATCTTCATCGGCTTTTTCGTCATCATCTGTATGAATCAAATCGCCTTCTTCTGTGTCATCAAGGTTTAATTTTTTATGTATTTCTTTTCCCGCTCCTTTCGCTTTCTTCCCTTCCTTTCTCGCCACGTTCGCCGGCTTTCCCCGTCAAGCTCTAAAGGCTGTTGTGTCGGGTGATGTGATTGAAATCTATGAGTATGAGAAGCCTATTCTGTCGGGTAAGGATGCCGAAAATAGAAGGCGTAAGAGTGATGATGAGGATGATTCTCCTGAATGGATTAAGGGGCTTTCTGAGGGCGAAATCGGGGGTTCGCCCTTTGACGTTGGAGTCCACGTTCTTTAATAGTGGACTCTTGTTCCAAACTGGAACAACACTCAACCCTATCTCGGTCTATTCTTTTGATTTATAAGGGATTTTGCCGATTTCGGCCTATTGGTTAACTCCCTTTAGGGTTCCGATTTAGCCAAAAAACTTGATTAGGGTGATGGTTCACGTAGTGGGCAACAAACCACCATAGGAGATTAACCTTTTACGGTGTAAACCTTCCTCCAAATCAGACAAACGTTTCAAATTCTTTTCTTCAORIT2StaphylococcusCCTCTCTTGTATCTTTTTTATTTTGAGTGGTTTTGTCCGTTACACTAGAAAACCGAAAGACAATAAAAATTTTATTCTTGCTGAGGCTCCCTTTAGGGTTCCGATTTAGTGCTTTACGGCACCTCGACCCCAAAAAACTTGATTAGGGTGATGGTTCACGTAGTGGGCCATCGCCCTGATAGACGGTTTTTCGCCCTTTGACGTTGGAGTCCACGTAGTCTGGCTTTCGGTAAGCTAGACAAAACGGACAAAATAAAAATTGGCAAGGGTTTAAAGGTGGAGATTTTTTGAGTGATCTTCTCAAAAAATACTACCTGTCCCTTGCTGATTTTTAAACGAGCACGAGAGCAAAACCCCCCTTTGCTGAGGTGGCAGAGGGCAGGTTTTTTTGTTTCTTTTTTCTCGTAAAATTCTTTCTTATCTTGORIT3BacillusborstelensisAATAAAACCCGCACTATGCCATTACATTTATATCTATGATACGTCCCTTTGACGTTGGAGTCCACGTTCTTTAATAGTGGACTCTTGTTCCAAACTGGAACAACACTCAACCCTATCTCGGTCTATTCTTTTGATTTATAAGGGATTTTGCCGATTTCGGCCTATTGGTTAAAAAATGAGCTGATTTAORIT4BacillussubtilisAACTTGATTATAGCGACACATTAGAACACGCCTAATTTTTTCCAGTTGAAAGGGGGTGGTGAATTGATAGTCAGGTATAAGGCTGTTGTGTCGGGTGATGTGATTGAAATCTATGAGTATGAGAAGCCTATTCTGTCGGGTAAGGATGCCGAAAATAAGGAAACAGCCAAAAACATAGATAAAAACTGGATGGGTCAAAATGAACTTTCCAGTAATGAACGGATAGGTCAGAAGGCGTAAGAGTGATGATGAGGATGATTCTCCTGAATGGATTAAGGGGCTTTCTGAGGGCGATTCTGACGATCTGAGTGGTAAGTATAGGGATGATTCGTTGAGTCGTGCCAGAAAGAAGATCAGGCGGTTAATCAATGCGAATGTGTORIT5BacillussubtilisACGCGCCCTGTAGCGGCGCATTAAGCGCGGCGGGTGTGGTGGTTACGCGCAGCGTGACCGCTACACTTGCCAGCGCCCTAGCGCCCGCTCCTTTCGCTTTCTTCCCTTCCTTTCTCGCCACGTTCGCCGGCTTTCCCCGTCAAGCTCTAAATCGGGGGCTCCCTTTAGGGTTCCGATTTAGTGCTTTACGGCACCTCGACCCCAAAAAACTTGATTAGGGTGATGGTTCACGTAGTGGGCCATCGCCCTGATAGACGGTTTTTCGCCCTTTGACGTTGGAGTCCACGTTCTTTAATAGTGGACTCTTGTTCCAAACTGGAACAACACTCAACCCTATCTCGGTCTATTCTTTTGATTTATAAGGGATTTTGCCGATTTCGGCCTATTGGTTAAAAAATGAGCTGATTTAACAAAAATTTAACGCGAATTTTAACAAAATATTAACGTTTACAATTTORIT6BacillusmegateriumATCCTCTATCCTCTGATGCTGGAGTATACCGAAGAAGATATTTATCGGTTAAACAATGAAGTTGATGACCTTTTAATTAAAGCAGATGTTGTGTCGGGTGATGTGATTGAAATCTATGAGTATGAGAAGCCTATTCTGTCGGGTACCAGTAATGAACGGATAGGTCAAAATGAACTTTCCGGGAAAGTTCAAATTGAACCATCAAGAGAGGTCAAATTGAACCATCCGGATGGTTCAAAAAGAACCACTAATAATAATAACTCTATTAAAAATAACTTTAATAATAATGAGAATAATCTTGTTAACAAATTGTTAACAAGAGAAGAA
表4 五种重组质粒所介导分泌的MTSase蛋白含量
Table 4 Content of MTSase protein secreted by five recombinant plasmids

质粒所占比例/%蛋白含量/(g/L)pT19.80.247pT26.40.151pT310.70.231pT45.60.091pS510.90.221
1.6 重组的转化
参照文献[14]进行地衣芽孢杆菌感受态制备、转化及阳性转化子的筛选。
1.7 重组MTSase的发酵表达
将重组菌于LB平板划线活化,37 ℃培养16 h后挑取单菌落接种于15 mL LB液体培养基中37 ℃、250 r/min培养16 h,取1 mL转接于装液量为30 mL的筛选培养基37 ℃、250 r/min培养。每隔24 h取样,发酵液于4 ℃、12 000 r/min条件下离心30 min,上清液液即为粗酶液用于检测酶活力。
1.8 酶活力检测
用0.2 mol/L,pH 5.5柠檬酸磷酸氢二钠缓冲液配制11.1 g/L麦芽糊精溶液,取900 μL该溶液,加入100 μL MTSase酶液,75 ℃条件下反应10 min,沸水浴20 min终止反应,取500 μL反应液加入1.5 mL DNS溶液沸水浴反应5 min,冰浴冷却5 min定容至25 mL,在OD540 处检测底物酶促反应前后底物吸光值变化,通过麦芽四糖标准曲线计算酶活力。将每小时消耗相当于1 μmoL麦芽四糖所需的酶量定义为1个酶活力单位(U)。
1.9 粗酶液的SDS-PAGE分析
将BLK1~BLK5接种于装液量30 mL的发酵培养基中,42 ℃、250 r/min培养。每隔24 h取样测定发酵过程中酶活力和OD600变化。发酵结束,取发酵液于4 ℃、12 000 r/min条件下离心30 min,上清液在60 ℃条件下热处理1 h后再次离心,取上清液进行SDS-PAGE分析。
1.10 质粒特性评价方法的建立
依照BUSTIN等[15]制定的实时荧光定量PCR信息出版指南(minimum information for publication of quantitative real-time PCR experiments,MIQE)[15],实施实时荧光定量PCR实验并进行分析。首先,采用BioFlux SimplyP总RNA提取试剂盒提取总RNA;然后,将提取好的RNA用反转录试剂gDNA Eraser消除可能残余的基因组DNA后,反转录成cDNA;最后以cDNA为模板,以rpsE为内参基因,ymdA为目的基因,以设计的rpsE-F,rpsE-R,ymdA-F和ymdA-R为引物,加入SYBR Green I预混酶液,在CFX96 Bio-Rad Real-Time PCR Detection System中分别进行rpsE和ymdA定量PCR扩增反应,结束后对获取的RT-qPCR数据进行分析。质粒拷贝数根据2-ΔΔCt计算,计算如公式(1)所示:
质粒拷贝数=2-[(A-B)-(C-D)]
(1)
[:A,待测组目的基因平均Ct值;B,待测组内参基因平均Ct值;C,对照组目的基因平均Ct值;D,对照组内参基因平均Ct值。
RT-qPCR中,根据设计的引物,均选择参考退火温度为60 ℃,具体条件如下:循环1次(95 ℃ 30 s),循环39次(95 ℃ 5 s,60 ℃ 30 s)。
质粒稳定性的测定方法如下:将重组地衣芽孢杆菌BLK1平板活化后接种15 mL LB培养基,37 ℃,200 r/min过夜培养16 h,记为原代(0代),自原代开始,质粒按照1%(150 μL)的接种量接种于不含任何抗生素的15 mL LB液体培养基中,37 ℃,200 r/min,每隔2 d传1代,并确保每代OD600值不超过0.8。第1次在12 h以后就取样,将菌液稀释至10-4~10-6,最后取稀释液各100 μL涂布于空白LB平板上,37 ℃ 过夜培养16 h以上,挑选单菌落100个分别接种至空白LB平板和含抗生素的LB平板,37 ℃过夜培养16 h以上。质粒稳定性计算如公式(2)所示:
质粒稳定性![]()
(2)
2 结果与分析
2.1 复制原点的挖掘及重组质粒的构建
通过文献调研以及NCBI数据库挖掘金黄色葡萄球菌、纳豆芽孢杆菌等来源的质粒,得到6个适用于芽孢杆菌的复制原点,将它们命名为ORIT1~ORIT6(表3)。ORIT1-ORIT6在自由能上存在一定差异,为-1.77~-180.46 kJ/mol。在分子结构层面,自由能的意义在于衡量分子的稳定性和反应力。自由能越低分子越稳定,越难发生反应。反之ΔG越高的话,在动力学上越活跃。ORIT1~ORIT6茎环个数分别是1、4、5、2、8、4,相关研究表明茎环决定了质粒拷贝数和不相容性等,而通过与Rep结合可以增强茎环的曲率。弯曲角度随着其数量的增加而增大。复制起始蛋白诱导的弯曲促进了解旋酶引入复制原点区域。茎环的存在导致DNA结构发生显著扭曲,从而加速了DNA复制过程中的关键步骤,如蛋白质间的信息交流、碱基的配对和双链的解开等。本研究所选复制原点来自质粒最常见的两大类复制方式,其中ORIT1、ORIT3、ORIT4来自θ复制型质粒,ORIT2、ORIT5、ORIT6来自滚环复制型质粒,以此确保复制原点来源的多样性和结果的可靠性。
以带有Sulfolobus acidocaldarius来源MTSase基因TreY和PP2的pHY-PLK300质粒(pS0)为载体,利用同源重组技术,将复制原点片段进行扩增,并将其连接到线性化的载体上,连接产物转化至E.coli JM109。接着将重组质粒pS1、pS2、pS3、pS4、pS5、pS6转入宿主菌地衣芽孢杆菌B1522中。地衣芽孢杆菌重组菌pS1命名为BLK1,同样的,将其余重组菌分别命名为BLK2、BLK3、BLK4、BLK5、BLK6。提取质粒进行菌落PCR验证,结果如图1所示。片段大小与对应表达元件大小接近,表明重组地衣芽孢杆菌构建成功。
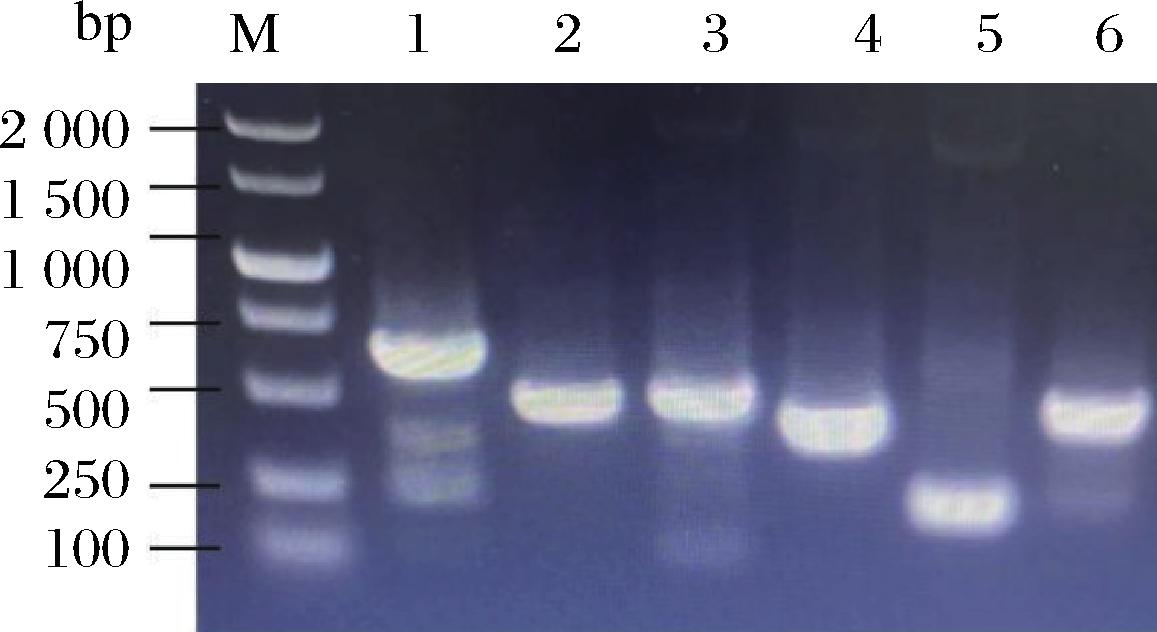
M-marker;1-ORIT1(652 bp);2-ORIT2(425 bp);3-ORIT5(456 bp);4-ORIT6(325 bp);5-ORIT3(188 bp);6-ORIT4(380 bp)
图1 复制原点重组菌菌落PCR验证
Fig.1 PCR validation of the origin of replication recombinant bacterial colony
2.2 人工复制原点的构建
复制原点是天然质粒的关键复制元件,它不仅是支持质粒自主复制的最小顺式作用元件,而且还是启动DNA复制的区域。其中的茎环结构对于质粒复制至关重要,质粒复制起始蛋白(Rep)能够识别茎环上的特定基序,产生局部链的不稳定,从而形成开放复合物以启动质粒复制过程[16]。研究发现茎环上某些短的基序在进化过程中具有很好的保守性,这些区域通常被称为模体(motif),目前模体识别己经成为一种能够从生物序列中提取有用生物信息的方法。它的基本思想是利用存在于DNA序列或结构中的某些特征模式识别相关序列的性质。如果某一部分具有保守性,而这种保守性跟部分生物学功能相关,那么这种特征模式可以用来识别该家族的新成员。MEME模体搜索软件是近几年开发出来的一个较好的模体识别平台,它能够识别出蛋白质或DNA序列中单个或多个模体,而且对于模体在序列中的起始位置并不加限制,识别结果如图2-a所示,pS5(ORIT5)质粒复制原点上的基序E值<<0.05,基序长度为6,符合其碱基序列短小的特点。茎环的位置跟个数对于复制至关重要,环上的任意突变破坏了该区域的螺旋相或固有DNA曲率,阻止了起源的链开放,从而取消了复制活性[17]。如图2-b、图2-c所示,P值均小于0.05,r值在0.7~0.9,结果表明,复制原点的茎环个数与人工表达系统的拷贝数跟稳定性属于强相关且显著性良好。
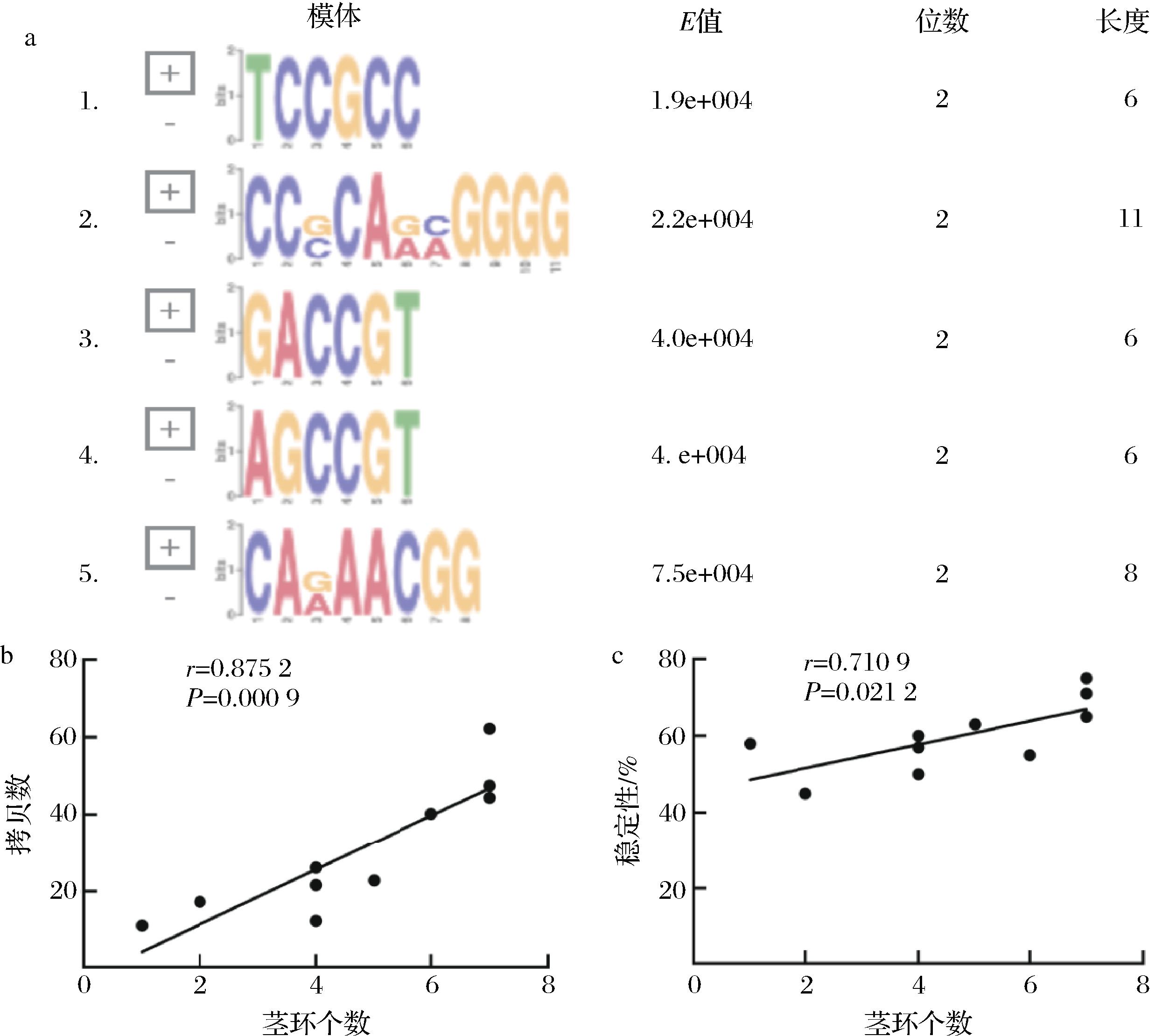
a-模体的筛选;b-茎环个数与拷贝数之间的关系;c-茎环个数与稳定性之间的关系
图2 人工复制原点的构建
Fig.2 Construction of manual origin of replication
2.3 复制原点的特性评价
将地衣芽孢杆菌重组菌在LB平板上进行划线活化,37 ℃培养16 h后,挑选单菌落接种于15 mL LB培养基中,37 ℃、250 r/min培养16~18 h作为种子液,取1 mL种子液转入装液量为30 mL的筛选培养基中,37 ℃、250 r/min培养,每隔一定时间取样,并按照建立的方法评价复制原点特性。结果表明,所选复制原点在地衣芽孢杆菌中均有应用价值。pS0是目前在地衣芽孢杆菌中应用的高效复制原点,在重组质粒稳定性维持在50%左右的前提下(图3-b),BLK5(pS5)的拷贝数是BLK3(pS3)的3.6倍,BLK0(pS0)的2.1倍(图3-a)。不同复制原点介导的目的基因表达水平差异较大,这使得通过改造复制原点来调控目的基因表达能力成为可能,具有一定的应用前景。
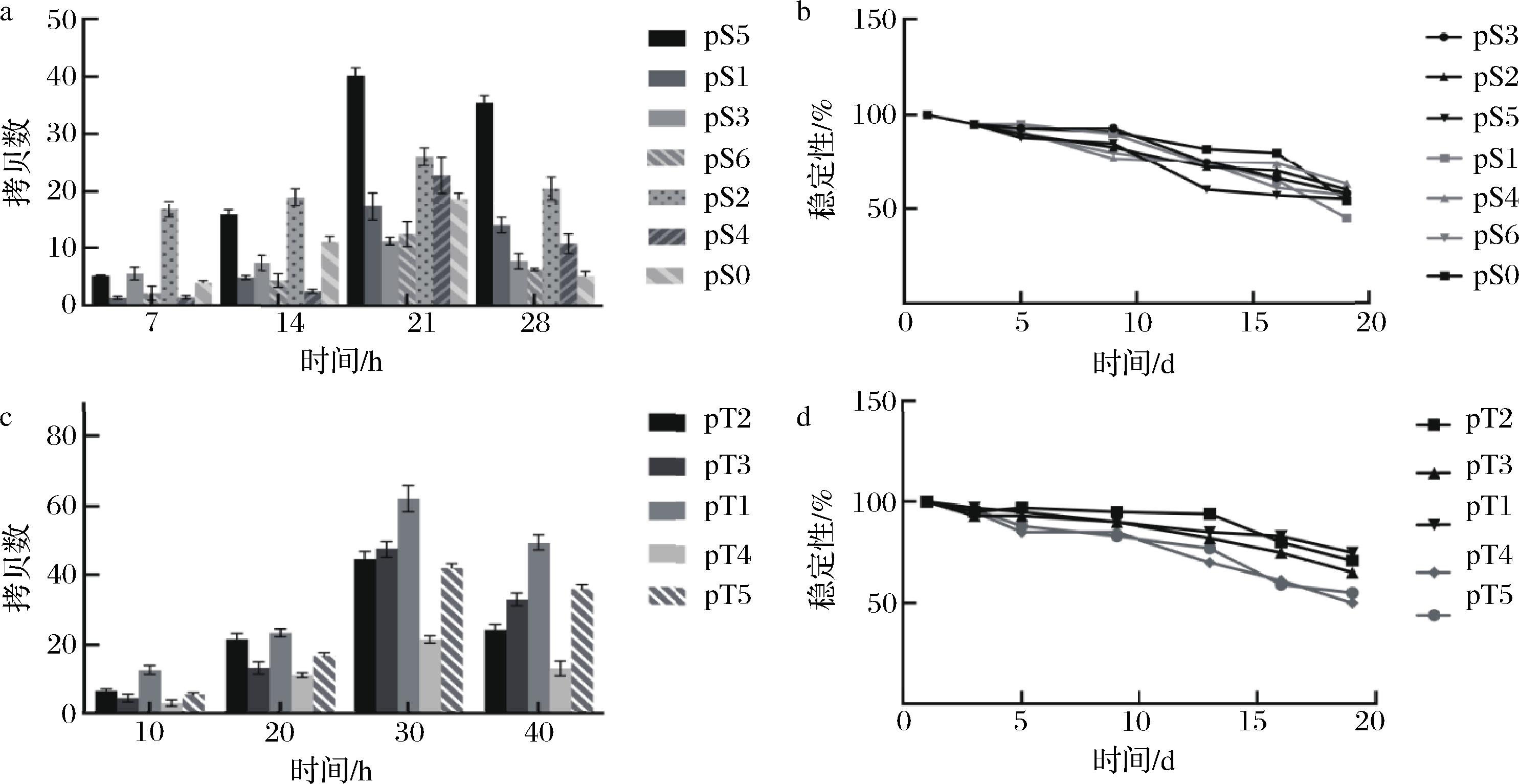
a-天然复制原点的拷贝数比较;b-天然复制原点的稳定性比较;c-人工复制原点的拷贝数比较;d-人工复制原点的稳定性比较
图3 复制原点的拷贝数和稳定性比较
Fig.3 Comparison of copy number and stability of origin of replication
分别构建ORIH1、ORIH2、ORIH3、ORIH4介导下MTSase重组表达质粒pT1、pT2、pT3、pT4、将重组质粒电转至宿主菌B1522,构建重组菌BLT1、BLT2、BLT3、BLT4。其中BLT1(pT1)的稳定性高于BLK5(pS5)36%(图3-d)。BLT1(pT1)的拷贝数最高可以达到62.2,相较于改造前提高了54.7%,是同时期最低值的2.9倍(图3-c)。结果表明相较于天然复制原点,人工复制原点介导表达系统的稳定性和拷贝数都有不同程度的提高。
2.4 天然及人工复制原点介导下MTSase功能表达的发酵验证
本研究选取ORIT1、ORIT2、ORIT3、ORIT4、ORIT5、ORIT6分别构建MTSase重组表达质粒pS1、pS2、pS3、pS4、pS5、pS6,将上述重组质粒电转至宿主菌地衣芽孢杆菌B1522,构建重组菌BLK1、BLK2、BLK3、BLK4、BLK5、BLK6。将重组菌及对照菌(BLK0)接种于发酵培养基中培养,并测定菌浓度及MTSase酶活力(图4)。BLK0、BLK1、BLK2、BLK3、BLK4、BLK5、BLK6酶活力分别可以达到6 065.9、5 817.7、6 855.2、3 900.2、6 449.7、7 603.5、3 993.5 U/mL。其中BLK2、BLK4、BLK5的酶活力相较于对照组均有所提高,比改造前分别提升了13.0%、6.3%、25.3%,其中pS5介导下MTSase酶活力最高。而BLK1、BLK3、BLK6重组菌的酶活力低于对照组,这一结果说明,通过改造复制原点可以调控目的蛋白的表达水平。如图3-b所示,BLK2(60%)、BLK4(63%)、BLK5(55%)的稳定性均高于对照组(54%)。而酶活力低于对照组的3个菌中有2个稳定性要略高于对照组,分别是BLK3(58%)、BLK6(57%),说明在较高酶活力下,重组菌的生长稳定性跟酶活力强相关。BLK0、BLK1、BLK2、BLK3、BLK4、BLK5、BLK6的拷贝数分别是18.7、17.3、26.1、11.2、22.8、40.2、12.4,表明重组菌的拷贝数跟酶活力大小具有较强的一致性且极值比达到了3.6,调控效果明显。通过上述研究,鉴定得到了起始效率较高的复制原点,随后本研究以BLK5(pS5)为研究对象考察基因工程策略对该表达系统介导下MTSase功能表达的影响。
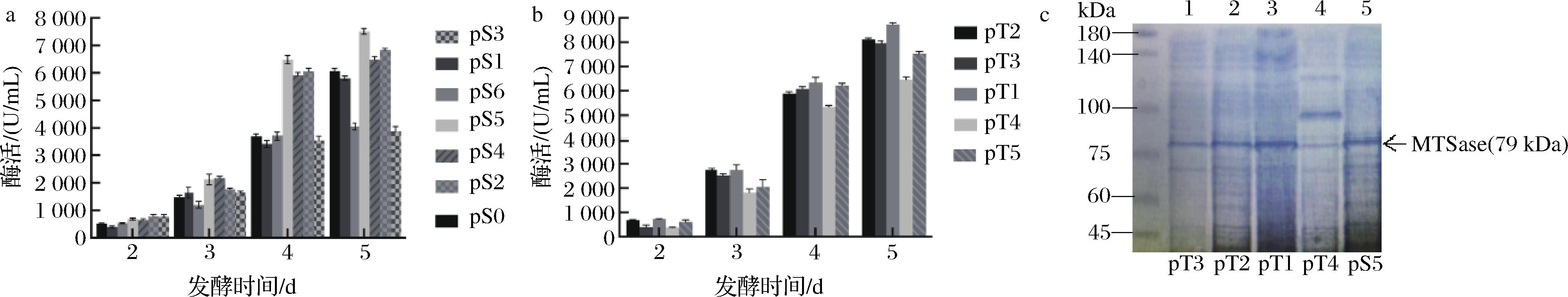
a-天然复制原点对MTSase功能表达的影响;b-人工复制原点对重组菌生长的影响;c-发酵液蛋白SDS-PAGE电泳图
图4 重组菌的酶活力测定
Fig.4 Enzyme activity determination of recombinant bacteria
基于天然复制原点的筛选结果,以pS5为载体,利用基因工程策略构建重组质粒pT1、pT2、pT3、pT4,将上述重组质粒电转至宿主菌B1522,构建重组菌BLT1、BLT2、BLT3、BLT4。将重组菌及对照菌(BLK5)接种于发酵培养基中培养,并测定蛋白浓度和MTSase酶活力(图4)。重组菌BLT1、BLT2、BLT3、BLT4酶活力分别达到8 726.7、8 082.5、7 901.7、6 535.8 U/mL,其中BLT1(pT1)的酶活力相较于BLK5(pS5)提高了14.8%。在同批4个重组菌中只有BLT4酶活力比对照组低14.1%,说明茎环结构对酶活力影响较大。BLT1、BLT2、BLT3的稳定性相较于改造前分别提高了36.4%、29.1%、18.2%,表明茎环结构在一定程度上影响质粒稳定性。在拷贝数方面,BLT2、BLT3提升不明显,只有10%左右,而BLT1提升了51.2%,说明茎环的位置对于重组菌拷贝数的提升至关重要,推测是由于复制起始蛋白对于质粒复制原点上的结合位点有特定取向的需求。相较于天然复制原点,人工复制原点的基因工程策略对于质粒稳定性、拷贝数、目的蛋白的表达量均有不同程度的提高。发酵上清液蛋白SDS-PAGE结果(图4-c)显示,MTSase特异蛋白条带为79 kDa,符合理论条带值大小,利用Image-Lab软件对异性蛋白条带在总条带占比进行分析,1~5号泳道目标条带占比分别是10.7%、6.4%、9.8%、5.6%、10.9%,利用样品浓度及蛋白所占比例计算5种重组质粒所介导分泌的MTSase蛋白含量分别为0.231、0.151、0.247、0.091、0.221 g/L。
3 结论与讨论
地衣芽孢杆菌作为一种食品安全级菌株,在工业酶制剂生产、代谢产物合成等领域展现出广阔的应用前景[18]。尽管地衣芽孢杆菌具有许多应用优势,但目前该菌株在合成生物学标准化遗传工具方面仍存在不足,对外源基因的表达调控研究也相对较少,导致许多外源基因无法在其中稳定表达和遗传。因此,应用高效复制原点来介导目的基因在宿主内的选择性表达,是工业微生物遗传改造的重要基础。
在规模化生产方面,目前可用于地衣芽孢杆菌的复制原点数量有限,且其表达效率尚未进行过全面比较。本研究基于文献调研,以拷贝数,稳定性为参考指标,建立了质粒特性的评价方法。筛选出的来源于枯草芽孢杆菌的复制原点ORIT5介导的表达系统在保持原有质粒稳定性基本不变的前提下,其拷贝数是pHY300内源复制原点介导表达系统的2.15倍,达到了40.2。
目前主要通过定点突变,杂合复制原点等方式获得高效复制原点[19]。定点突变可以通过将复制原点核心区域突变为保守序列的方式提高复制原点的活性,但是该方法不能快速构建特定强度的复制原点。杂合复制原点要求识别和验证较多的核心元素,然而,这一过程可能会妨碍对基因表达的精确调控。
本研究利用新构建的复制原点,成功实现了麦芽寡糖基海藻糖合成酶在地衣芽孢杆菌中的高效表达。在此基础上利用基因工程策略通过改变茎环数量跟位置构建了一系列复制原点,发现改造后复制原点pT1介导表达系统的酶活力在原有pS5基础上提高了54.7%,最高达到8726.7 U/mL。在先前的研究中,相同来源的麦芽寡糖基海藻糖合成酶在地衣芽孢杆菌中的胞外酶活力为6 065.9 U/mL[20]。本研究中地衣芽孢杆菌重组菌酶活力相较之前提高了43.9%,表明地衣芽孢杆菌作为食品酶生产宿主具有一定的工业应用价值,同时本研究为复制原点的改造以及安全表达系统的开发提供了实验数据及理论参考。
[1] WOESE C R,KANDLER O,WHEELIS M L.Towards a natural system of organisms:Proposal for the domains Archaea,Bacteria,and Eucarya[J].Proceedings of the National Academy of Sciences of the United States of America,1990,87(12):4576-4579.
[2] HOLMES M L,PFEIFER F,DYALL-SMITH M L Analysis of the halobacterial plasmid pHK2 minimal replicon[J].Gene,1995,153(1):117-121.
[3] KONIECZNY I,BURY K,WAWRZYCKA A,et al.Iteron plasmids[J].Microbiology Spectrum,2014,2(6).DOI:10.1128/9781555818982.ch2.
[4] ABELES A L,REAVES L D,AUSTIN S J.A single DnaA box is sufficient for initiation from the P1 plasmid origin[J].Journal of Bacteriology,1990,172(8):4386-4391.
[5] MUROTSU T,MATSUBARA K,SUGISAKI H,et al.Nine unique repeating sequences in a region essential for replication and incompatibility of the mini-F plasmid[J].Gene,1981,15(2-3):257-271.
[6] GERMINO J,BASTIA D.The replication initiator protein of plasmid R6K tagged with beta-galactosidase shows sequence-specific DNA-binding[J].Cell,1983,32(1):131-140.
[7] LACKS S A,LOPEZ P,GREENBERG B,et al.Identification and analysis of genes for tetracycline resistance and replication functions in the broad-host-range plasmid pLS1[J].Journal of Molecular Biology,1986,192(4):753-765.
[8] LIN L S,MEYER R J.Directly repeated,20-bp sequence of plasmid R1162 DNA is required for replication,expression of incompatibility,and copy-number control[J].Plasmid,1986,15(1):35-47.
[9] WASCHKAU B,WALDECK J,WIELAND S,et al.Generation of readily transformable Bacillus licheniformis mutants[J].Applied Microbiology and Biotechnology,2008,78(1):181-188.
[10] MURAS A,ROMERO M,MAYER C,et al.Biotechnological applications of Bacillus licheniformis[J].Critical Reviews in Biotechnology,2021,41(4):609-627.
[11] KOBAYASHI M,KUBOTA M,MATSUURA Y.Refined structure and functional implications of trehalose synthase from Sulfolobus acidocaldarius[J].Journal of Applied Glycoscience,2003,50(1):1-8.
[12] 刘宗利,王乃强,刘峰,等.海藻糖在日化领域的应用研究进展[J].精细与专用化学品,2015,23(2):18-20.LIU Z L,WANG N Q,LIU F,et al.Study on the application of trehalose in the daily chemical field[J].Fine and Specialty Chemicals,2015,23(2):18-20.
[13] 赵鑫馨.地衣芽孢杆菌强启动子开发及人工进化[D].无锡:江南大学,2021.ZHAO X X.Development and artificial evolution of strong promoters of Bacillus licheniformis[D].Wuxi:Jiangnan University,2021.
[14] XIAO F X,LI Y R,ZHANG Y P,et al.Construction of a novel sugar alcohol-inducible expression system in Bacillus licheniformis[J].Applied Microbiology and Biotechnology,2020,104(12):5409-5425.
[15] BUSTIN S A,VLADIMIR B,GARSON J A,et al.The MIQE guidelines:minimum information for publication of quantitative real-time PCR experiments[J].Clinical Chemistry,2009,55(4):611-622.
[16] JOSHUA C J,PEREZ L D,KEASLING J D.Functional characterization of the origin of replication of pRN1 from Sulfolobus islandicus REN1H1[J].PloS One,2013,8(12):e84664.
[17] KOWALCZYK L,RAJEWSKA M,KONIECZNY I.Positioning and the specific sequence of each 13-mer motif are critical for activity of the plasmid RK2 replication origin[J].Molecular Microbiology,2005,57(5):1439-1449.
[18] SONG C W,CHELLADURAI R,PARK J M,et al.Engineering a newly isolated Bacillus licheniformis strain for the production of (2R,3R)-butanediol[J].Journal of Industrial Microbiology &Biotechnology,2020,47(1):97-108.
[19] BR ZDA V,LAISTER R C,JAGELSK
ZDA V,LAISTER R C,JAGELSK E B,et al.Cruciform structures are a common DNA feature important for regulating biological processes[J].BMC Molecular Biology,2011,12:33.
E B,et al.Cruciform structures are a common DNA feature important for regulating biological processes[J].BMC Molecular Biology,2011,12:33.
[20] 陆一鸣,李由然,许银彪,等.启动子工程提高海藻糖生产用酶在地衣芽孢杆菌中的表达[J].基因组学与应用生物学,2022,41(8):1703-1712.LU Y M,LI Y R,XU Y B,et al.Promoter engineering improves the expression of trehalose production enzyme in Bacillus licheniformis[J].Genomics and Applied Biology,2022,41(8):1703-1712.