葡萄孢属(核盘菌科)是最古老的真菌类群之一,包含逾35个种,其中灰葡萄孢菌最广为人知且被深入研究[1]。灰葡萄孢菌是引起灰霉病的真菌病原体,侵染方式灵活、传播繁殖快、寄主广泛且能够以分生孢子和/或菌核形式存活较长时间,是一种进化高度成功的植物致病菌[2]。灰葡萄孢菌能够在出苗、开花、挂果、运输/贮存等各阶段侵染园艺作物,每年在全球范围内造成的经济损失达100~1 000亿美元[3],因此防控灰葡萄孢菌侵染对于园艺生产至关重要。当前,施用化学杀菌剂是防治灰葡萄孢菌的主要方式。在我国,常用的杀菌剂主要有苯并咪唑类、琥珀酸脱氢酶抑制剂和苯吡咯等六大类[4]。然而灰葡萄孢菌遗传变异频繁,极易产生抗药性,例如,2016—2022年在湖北、山东等地分离的200余株灰葡萄孢菌对多菌灵和腐霉利的抗药性分别已达95%~100%和85%~100%[4-5]。为实现有效防控,实际生产过程中会增加剂量和施药次数,不仅加速杀菌剂失效,还增加防治成本、加重杀菌剂残留,严重威胁农产品和食品安全。针对灰葡萄孢菌易产生抗药性的问题,研发新型杀菌剂是抗药性治理长期策略的重要方面[6]。挖掘与灰葡萄孢菌生长发育和致病相关的基因、蛋白、毒力因子等进而探究灰葡萄孢菌的侵染机制,可为新型杀菌药剂的研发以及灰葡萄孢孢菌防治提供理论基础。灰葡萄孢菌侵染植物的进程和机制极为复杂,自2005年对灰葡萄孢菌B05.10菌株完成基因组测序以来,国内外诸多课题组构建近160个基因突变体研究其在葡萄孢菌生长、分化及侵染进程中的功能,以期揭示灰葡萄孢菌的侵染机制[7]。然而,灰葡萄孢菌基因组编码近11 000个基因,已明确研究的基因数目所占比例还未到1.5%,我们对灰葡萄孢菌的了解和研究还远远不够[7-8]。因此,继续挖掘与灰葡萄孢菌生长发育和侵染相关的基因并进一步探究其侵染机制是应对灰葡萄孢菌侵染和抗药性产生的长期工作目标。
细胞壁是致病真菌入侵、进攻寄主细胞的最前端,是病原体和宿主建立接触的界面结构,对真菌的生存能力、致病性和毒力至关重要[9]。β-1,3-葡聚糖作为真菌细胞壁的代表性多糖成分,对于维持细胞壁的完整性和形态十分关键;它还是能被病原体识别的病原体相关分子模式,可激活寄主免疫反应[10-11]。β-1,3-葡聚糖是由镶嵌在细胞膜上的β-1,3-葡聚糖合成酶催化转运尿苷二磷酸葡萄糖而生成的长链多糖,编码该酶的基因为β-1,3-葡聚糖合成酶基因(FKS/GLS),该基因对于真菌的存活至关重要[10,12]。在酿酒酵母中已鉴定3个FKS基因(FKS1,FKS2,FKS3),其中FKS1和FKS2联合致死[13]。在粟酒裂殖酵母发现4个β-1,3-葡聚糖合成酶同源基因(BGS1-4),编码BGS1,BGS3,和BGS4蛋白的基因对于维持其生命必不可少[14-16]。与酵母不同的是,大多数丝状子囊菌仅含有1个FKS编码基因且缺失该基因真菌不能存活[17-18]。此外,致病菌的成功侵染依赖于β-1,3-葡聚糖合成酶基因的正确表达。禾生炭疽病菌仅存在一个β-1,3-葡聚糖合成酶基因(GLS1),利用基因沉默技术抑制GLS1的表达致使该病菌的附着胞丧失细胞壁刚性和黑化能力,膨压不足而无法黏附入侵寄主细胞;而GLS1过表达造成其活体营养型菌丝细胞壁β-1,3-葡聚糖外露,激活寄主免疫反应而导致侵染失败[19]。鉴于β-1,3-葡聚糖合成酶及其编码基因在真菌存活和侵染中的重要功能,β-1,3-葡聚糖合成酶作为研发新型抗菌药物的靶点受到极大关注[20]。在致病真菌中,β-1,3-葡聚糖合成酶功能明确,其合成产物β-1,3-葡聚糖是细胞壁中最核心结构组分,这使得β-1,3-葡聚糖合成酶成为最具优势的新型抗菌药物作用靶点[20-21]。例如,在人类抗菌药物研发中,以β-1,3-葡聚糖合成酶为靶点的抑制剂棘白菌素已通过认证并用于人类真菌感染的治疗[21],这为研发防治灰葡萄孢菌的新型抗菌药物提供借鉴。然而在灰葡萄孢菌中,尚未对β-1,3-葡聚糖合成酶进行研究报道。本研究拟利用生物信息学方法对灰葡萄孢菌中编码β-1,3-葡聚糖合成酶的基因进行克隆及生物信息学分析,挖掘β-1,3-葡聚糖合成酶基因的功能;并利用qPCR技术分析该基因在不同非生物胁迫下的表达变化,探究该基因参与应对灰葡萄孢菌生长发育环境的机制,从而为研究β-1,3-葡聚糖合成酶基因功能及其调控机制,进而研发新型抗菌药物防治灰葡萄孢菌侵染提供参考依据。
1 材料与方法
1.1 供试菌株与试剂
本试验用灰葡萄孢菌菌株(Botrytis cinerea S1)分离于感染灰霉病的草莓果实(江苏省镇江市)。基于酿酒酵母β-1,3-葡聚糖合成酶(FKS1)序列(NP_013446.1)从灰葡萄孢菌菌株B05.10基因组中比对得到同源序列XP_024547254.1(Per.Ident为62.23%),据此序列设计引物,克隆灰葡萄孢菌β-1,3-葡聚糖合成酶基因(BcGLS)序列。真菌基因组DNA快速抽提试剂盒、UN1Q-10柱式Trizol总RNA抽提试剂盒、第一链cDNA合成试剂盒,购自上海生工生物工程有限公司;引物合成、基因测序均由上海生工生物工程有限公司完成。
1.2 仪器与设备
iCEN-24R冷冻高速离心机,杭州奥盛仪器有限公司;PCR仪和电泳仪,Bio-Rad公司;ND-1000超微量分光光度计,Nanodrop公司;7500荧光定量PCR仪,美国ABI公司。
1.3 实验方法
1.3.1 灰葡萄孢菌基因组DNA提取及BcGLS克隆
应用上海生工生物工程有限公司的真菌基因组DNA快速抽提试剂盒提取灰葡萄孢菌S1菌株基因组DNA。以灰葡萄孢菌S1菌株基因组DNA为模板,利用附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.037310)中的引物对BcGLS进行多片段扩增测序并拼接,得到BcGLS基因全长序列。
1.3.2 BcGLS基因生物信息学分析
利用在线软件FGENESH(http://www.softberry.com/berry.phtml)分析BcGLS基因结构并预测编码蛋白质氨基酸序列;于NCBI应用Conserved Domain(https://www.ncbi.nlm.nih.gov/cdd)进行保守结构域预测;通过在线网站Expasy-ProtParam tool(https://web.expasy.org/protparam/)分析蛋白质的理化性质以及Expasy-ProtScale(https://web.expasy.org/cgi-bin/protscale/protscale.pl)预测蛋白质的等电点和亲水性;利用THMHM 2.0(https://services.healthtech.dtu.dk/services/TMHMM-2.0/)预测蛋白跨膜结构;利用Signal IP 5.0(https://services.healthtech.dtu.dk/services/SignalP-5.0/)工具预测信号肽;通过PredictProtein(https://predictprotein.org)在线网站预测蛋白序列二级结构及亚细胞定位;利用SWISS-MODEL(https://swissmodel.expasy.org)预测蛋白序列的三级结构;从NCBI中收集已知各物种FKS/GLS氨基酸序列,ClustalX进行序列比对,MEGA11软件设置邻接法用于构建BcGLS与其他物种FKS/GLS系统进化树,并利用在线网站iTOL(https://itol.embl.de)对进化树进行注释和管理。
1.3.3 逆境胁迫条件下灰葡萄孢菌菌丝生长速率及BcGLS基因表达分析
配制含有不同浓度胁迫物质的PDA培养基,渗透胁迫物质氯化钠(NaCl)质量浓度分别为0、10、20、30、40 g/L;细胞壁干扰剂十二烷基硫酸钠(sodium dodecyl sulfate,SDS)质量浓度分别为0、30、60、90、120 mg/L;氧化应激剂过氧化氢(H2O2)浓度分别为0、1、10、100、200 mmol/L。将低温保存的灰葡萄孢菌S1于PDA培养基上活化4~5 d后,取0.5 cm×0.5 cm接种块接种到含有不同浓度胁迫物质的PDA培养基上,置于22~24 ℃黑暗条件下培养,每天记录菌丝生长长度,并计算菌丝生长速率。
将灰葡萄孢菌S1于PDA培养基上培养4~5 d后,取1 cm×1 cm接种块接种到分别含有40 g/L NaCl、120 mg/L SDS、100 mmol/L H2O2的液体培养基中(以未添加任何胁迫物质的液体培养基为对照),置于22~24 ℃黑暗条件下培养4 d后,收取菌丝经液氮速冻后,-80 ℃冰箱保存。每个处理设置3个重复,每个重复接种4个接种块。
取100 mg各处理灰葡萄孢菌菌丝,液氮速冻后快速研磨成粉末并转移至RNAase-Free的离心管中,随后参照UNIQ柱式Trizol总RNA提取试剂盒(B511321;生工生物工(上海)程股份有限公司)说明书进行总RNA提取。应用RNase-Free DNAase Set清除基因组DNA。参照第1链cDNA合成试剂盒(生工生物工(上海)程股份有限公司)说明书进行cDNA第1链的合成。以不同cDNA样品为模板,用灰葡萄孢菌内参基因β-tubulin的特异性引物(正向:ACATGCTCTGCCATTTTCCG;反向:TTGTTAGGGATCCACTCAACGAAG)和BcGLS基因特异性引物(正向:ACACCCCTCGCCCACTAT;反向:TCCTTCCTACGAACTCGAACTAC)通过荧光定量PCR仪进行RT-PCR扩增,每个待测样品设置3个技术重复。反应体系(20 μL):10 μL SybrGreen qPCR Master Mix,引物各0.4 μL、cDNA 2.0 μL、ddH2O 7.2 μL。反应程序:95 ℃ 3 min、45个循环(95 ℃ 15 s、60 ℃ 30 s、72 ℃ 30 s)。采用2-ΔΔCt法计算BcGLS在不同胁迫条件下的相对表达量,并利用SPSS 27软件进行数据处理与分析。
2 结果与分析
2.1 BcGLS基因克隆与基因结构分析
依据酿酒酵母β-1,3-葡聚糖合成酶(FKS1)序列于灰葡萄孢菌模式菌株B05.10基因组中比对得到同源序列,该同源序列为未经鉴定的未知序列(XP_024547254.1)。据此序列设计特异性引物并于本试验分离菌株S1中克隆得到全长7 137 bp的碱基序列。经FGENESH预测,该序列编码1个基因,包含3个外显子和2个内含子;该基因结构预测获得较高评分(559.94),可信度高;其中对外显子2的评分为439.70,远高于外显子1和3(图1)。

CDSf-起始外显子;CDSi-中间外显子;CDSI-末尾外显子;CDSo-唯一外显子;TSS-转录起始位点;PolyA-末端polyA尾区
图1 灰葡萄孢菌β-1,3-葡聚糖合成酶编码基因BcGLS的FGENESH基因结构预测[22]
Fig.1 The gene structure prediction of BcGLS from B.cinerea by FGENESH[22]
2.2 BcGLS基因编码蛋白(BcGLSp)氨基酸结构域分析
经FGENESH预测,BcGLS基因序列编码1 932个氨基酸(氨基酸序列请见附件,https://doi.org/10.13995/j.cnki.11-1802/ts.037310)。在NCBI中应用Conserved Domain在线对该氨基酸序列进行保守结构域及编码蛋白质功能分析(蛋白结构域预测请见附图1,https://doi.org/10.13995/j.cnki.11-1802/ts.037310),结果表明,BcGLSp含有一个β-1,3-葡聚糖合成酶结构域,属于葡聚糖合成酶超家族成员,该家族包含多种β-1,3-葡聚糖合成酶,如酵母中的Gls1,Gls2,和Gls3,能够催化合成β-1,3-葡聚糖。氨基酸序列结构显示其还含有一个β-1,3-葡聚糖合成酶亚基FKS1结构域(FKS1_dom1,pfam14288);另外,该预测蛋白结构域也包含一个麦谷蛋白亚基(Glutenin_hmw,pfam03157),该亚基家族为高分子质量的麦谷蛋白,是谷蛋白弹性的来源,该亚基可能与灰葡萄孢菌细胞壁多糖的高黏度有关。
2.3 BcGLSp的理化性质分析
通过Protparam对BcGLS基因编码的氨基酸序列进行理化性质分析,结果显示,BcGLSp分子式为C10005H15263N2619O2808S90,分子质量220.049 51 kDa,理论等电点7.97。氨基酸组成成分分析表明,BcGLS蛋白Leu含量最高,占总氨基酸的9.4%,Cys含量最低,占比为1.4%,该蛋白序列中不含Pyl和Sec。BcGLSp结构中负电荷残基(Asp+Glu)总数为191个,正电荷残基(Arg+Lys)总数为196个;在酵母中该蛋白半衰期>20 h,其不稳定系数为35.5,因此推测BcGLSp属于稳定蛋白。BcGLS蛋白脂肪系数为84.16,亲水性总平均值为-0.146,推测其为亲水性蛋白。
2.4 BcGLSp的磷酸化位点及亲/疏水性分析
预测蛋白质磷酸化位点以神经网络算法为基础,通过NetPhos 3.1 Server分析发现该蛋白含有176个磷酸化位点,其中包括83个丝氨酸(S),67个苏氨酸(T)和26个酪氨酸(Y),相对应磷酸化势见图2-a,推测BcGLSp被这3种氨基酸激酶磷酸化且激活,最终调控基因表达。
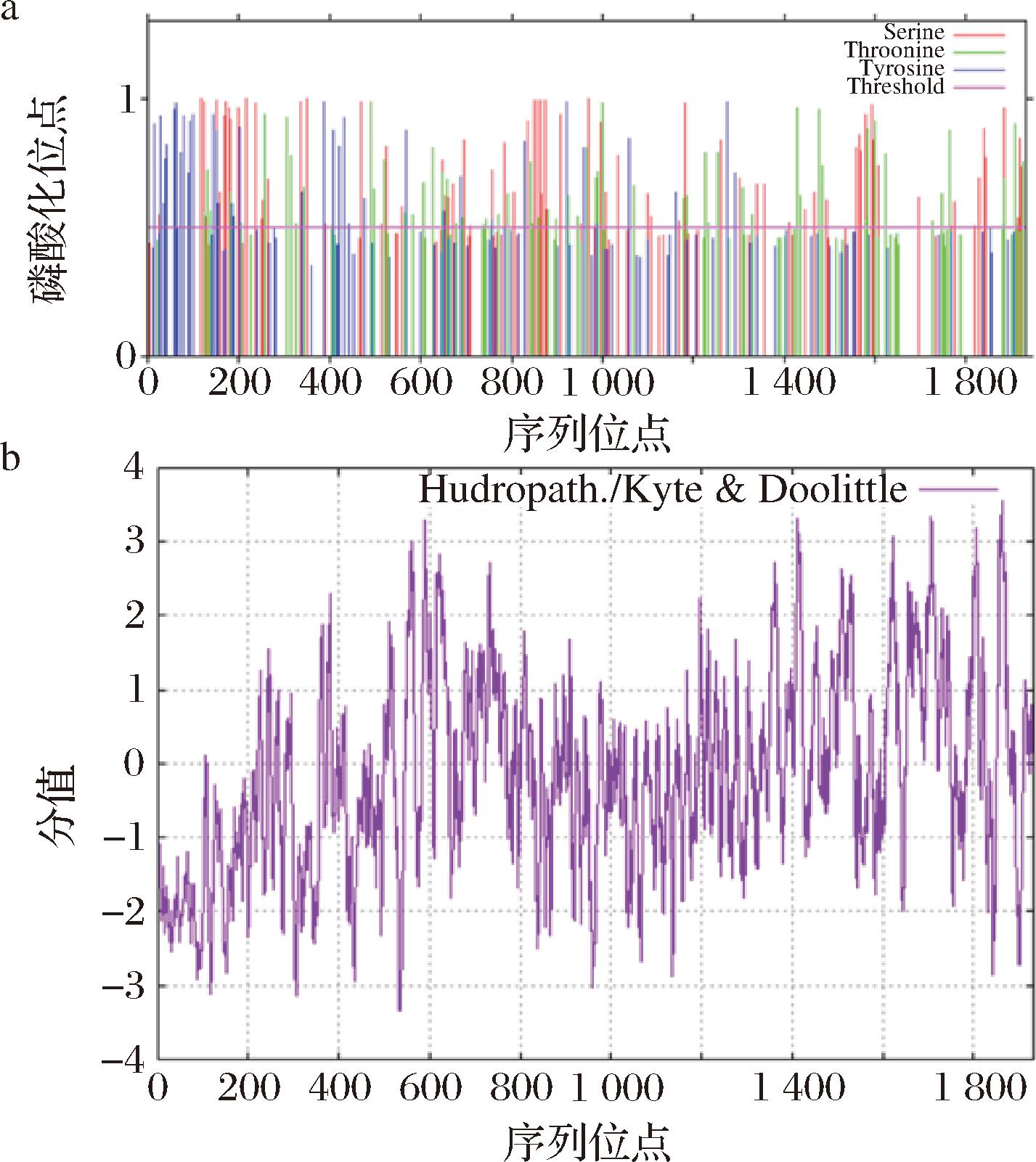
a-BcGLSp磷酸化位点;b-BcGLSp亲水/疏水性
图2 BcGLSp磷酸化位点和亲水/疏水性分析
Fig.2 Analysis of phosphorylation site and hydrophilicity/hydrophobicity of BcGLSp
运用ProtScale工具分析BcGLS基因编码蛋白产物的亲水性和疏水性可知,亲水性分析中最大峰值位于多肽链第533和534位的谷氨酰胺(Q),对应峰数值-3.344,为亲水性最强氨基酸;疏水性分析显示其最大峰值位于第1 864位的亮氨酸(L),对应峰数值3.556,为疏水性最强氨基酸。由图2-b可知,该蛋白序列中亲水性氨基酸多于疏水性氨基酸,疏水区域主要分布在肽链内部,推测BcGLSp为亲水性蛋白,该结果与其理化性质分析结果一致。
2.5 BcGLSp的亚细胞定位,跨膜区结构域和信号肽预测
应用PredictProtein预测BcGLSp亚细胞定位如图3-a所示,该蛋白位于灰葡萄孢菌细胞膜上。利用跨膜预测服务器TMHMM server 2.0分析BcGLSp氨基酸序列,预测结果如图3-b所示,拓扑结构如图3-c所示。结果显示,该酶含有17个跨膜螺旋结构域(transmembrane helix,TMHs),主要位于氨基酸序列第501~743位(N-末端的6个TMHs)和1 348-1 874位(C-末端的11个TMHs)的2个区域,其中FKS1亚基(AA353-462)完全位于细胞膜内侧,属于亲水性区域,而β-1,3-葡聚糖合成酶组件氨基酸序列(AA861-1685)进行多次跨膜折叠;该蛋白的N端位于胞内,C端位于胞外。利用SignalIP 5.0 Server预测信号肽,该蛋白无信号肽,推测其为非分泌型蛋白。
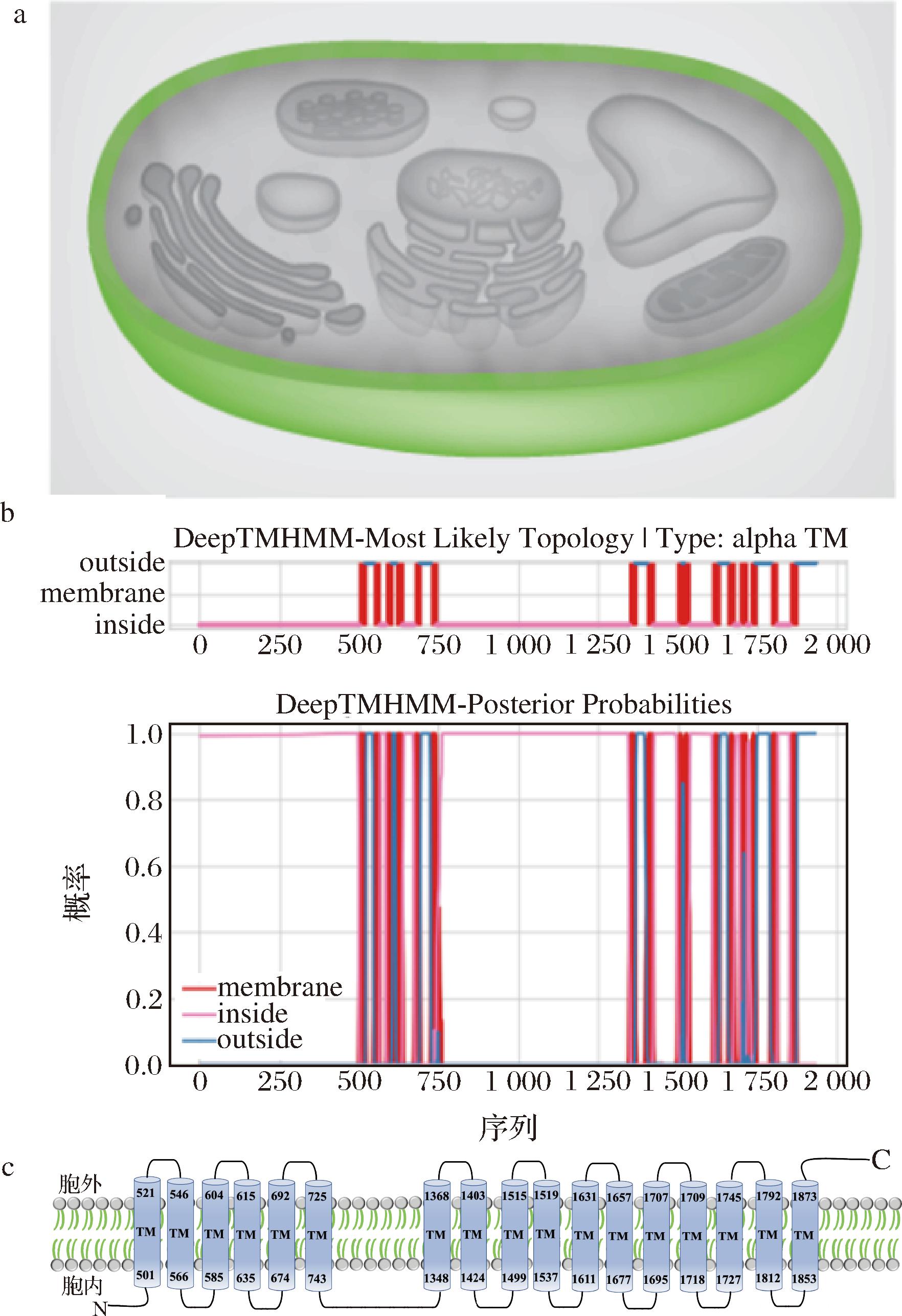
a-BcGLSp亚细胞定位预测;b-BcGLSp跨膜结构域预测;c-BcGLSp拓扑结构
图3 BcGLSp亚细胞定位、跨膜结构域预测和拓扑结构
Fig.3 Prediction of subcellular localization,potential protein transmembrane,and topology of BcGLSp
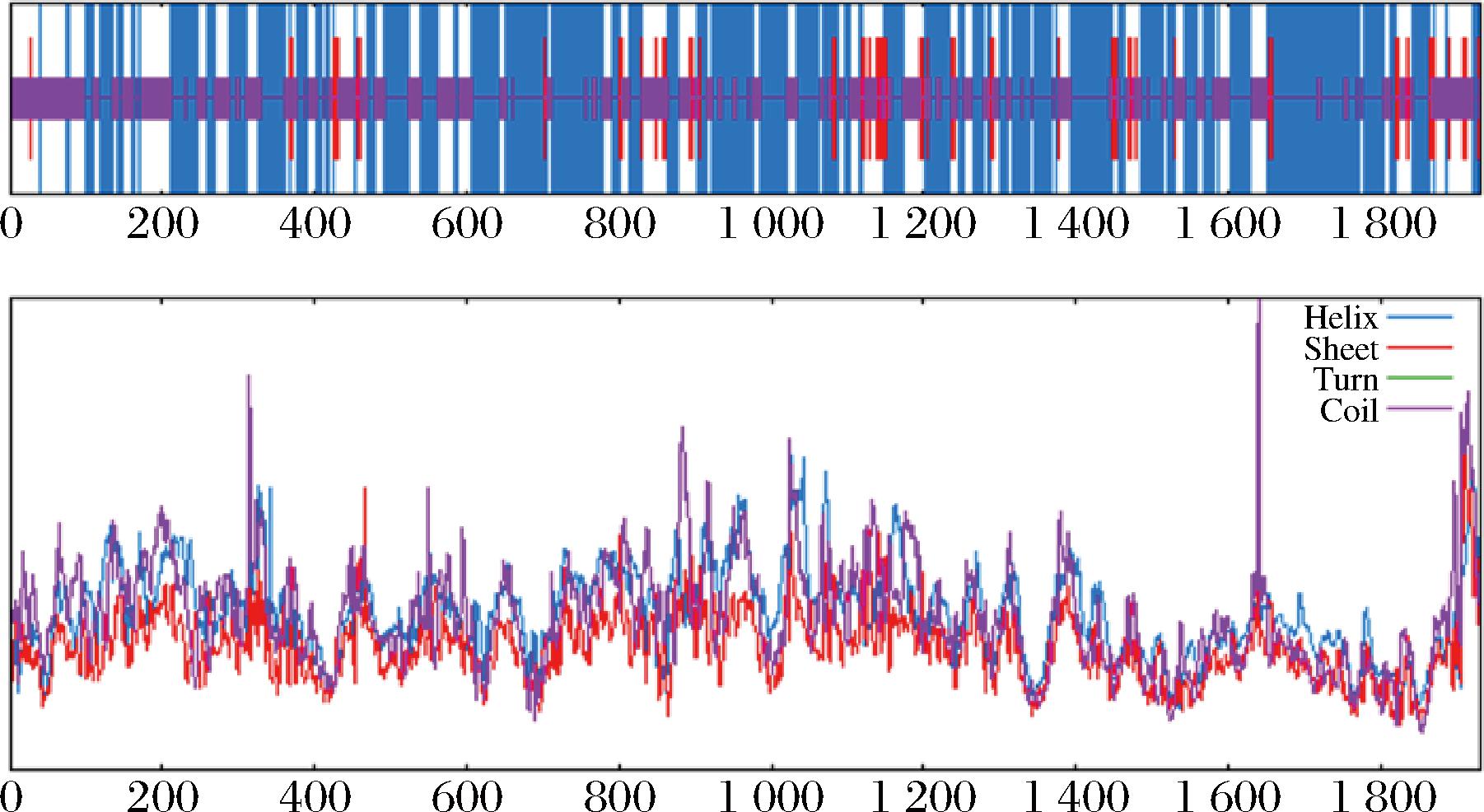
2.6 BcGLSp的二级结构和三级结构预测
通过SOPMA软件分析BcGLSp的二级结构,预测结果表明BcGLSp二级结构由α螺旋、β折叠和不规则卷曲组成,其中α螺旋占比53.88%,不规则卷曲占比41.3%,β折叠占比为4.81%(BcGLSp二级结构预测请见附图2,https://doi.org/10.13995/j.cnki.11-1802/ts.037310)。
通过SWISS-MODEL软件运用同源建模法预测BcGLSp三级结构,得到如图4-a的三维模型,BcGLSp序列与模板蛋白氨基酸序列相似度高达81.26%(U7PL67_SPOS1),因此同源建模法可用于预测BcGLSp三维结构;预测结果表明该模型为跨膜片段与亚细胞定位和跨膜结构域预测相符;拉氏图(图4-b)结果显示BcGLSp 94.04%的氨基酸残基落在允许区和最大允许区,占整个蛋白质的比例高于90%,该模型构象符合立体化学规则,且该预测三级结构全局模型质量评估(global model quality estimation,GMQE)得分为0.76,表明BcGLSp三级结构预测模型质量较高。另外,利用Pymol将该三维模型与已在酿酒酵母中解析得到的β-1,3-葡聚糖合成酶(FKS)三级结构进行比对,如图4-c所示,灰葡萄孢菌BcGLSp的预测模型与酿酒酵母FKS三级结构重合度较高,表明BcGLSp三级结构预测模型可靠度较高。
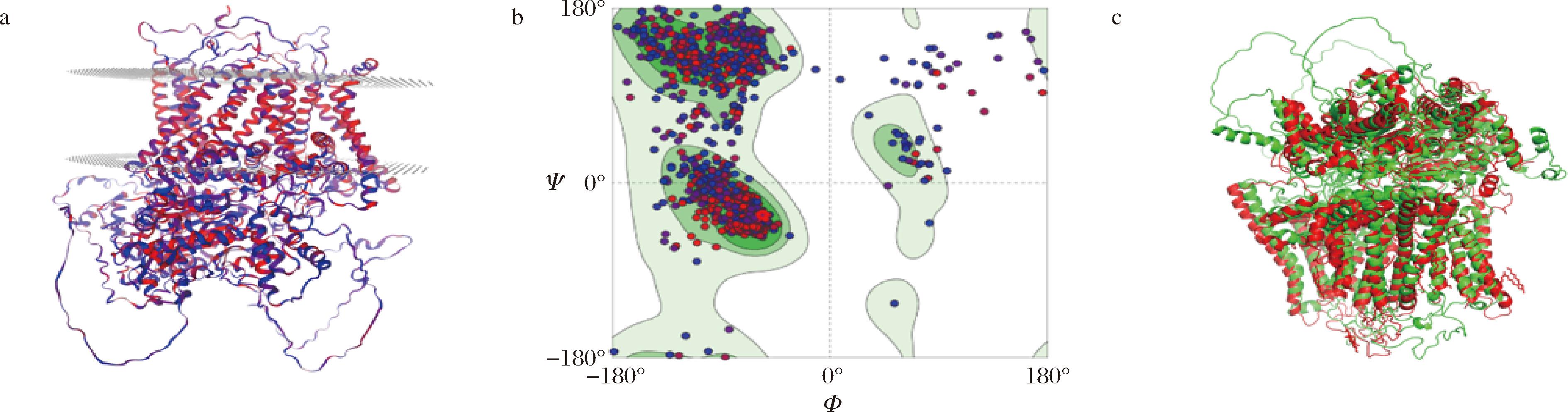
a-BcGLSp三级结构模型;b-BcGLSp三级结构模型拉氏图;c-BcGLSp三级结构与酿酒酵母FKS三级结构比对图
图4 BcGLSp三级结构模型、拉氏图及其与酿酒酵母FKS三级结构比对图
Fig.4 3D model,the Ramachandran plot paragraph of BcGLSp and its comparison with FKS structure from Saccharomyces cerevisiae
注:绿色为酿酒酵母FKS,红色为BcGLS。
2.7 BcGLSp同源性及进化分析
利用NCBI数据库中BLAST对BcGLSp氨基酸序列进行同源比对,下载与之同源性较高且已被鉴定的β-1,3-葡聚糖合成酶蛋白序列,与此同时,下载在其他物种中已被报道发表的β-1,3-葡聚糖合成酶蛋白序列,应用MEGA11软件进行系统进化分析,采用邻接法(neighbor-joining method,NJ)构建系统进化树(图5)。结果表明,灰葡萄孢菌S1菌株与灰葡萄孢菌B05.10以及最新鉴定的草莓葡萄孢的GLS蛋白序列同源性最高,达到97.8%以上,属于同一物种;与山茶叶杯菌β-1,3-葡聚糖合成酶蛋白序列相似性为94.77%,同源性较高,山茶叶杯菌侵染花器,造成山茶花腐病,是山茶属植物最为严重的病害。其次,灰葡萄孢菌S1的BcGLS蛋白序列与狭孢小毛盘菌、二孢白粉菌、景天白粉菌、布氏白粉菌、大豆茎褐腐病菌以及蔷薇双壳菌的GLS蛋白序列同源性在84%~86%,该6种菌均为植物致病菌,与灰葡萄孢菌β-1,3-葡聚糖合成酶蛋白序列同源性较高;与水稻致病真菌稻瘟病菌和禾谷炭疽病菌及人类致病真菌申克孢子丝菌、构巢曲霉、烟曲霉、黄曲霉和米曲霉菌的GLS蛋白序列相似性在74%~78%,同源进化关系较近;而与大型真菌如灰树花、灰盖鬼伞及双色蜡蘑GLS蛋白序列相似性均低于52%,同源性低,进化关系远。

图5 BcGLSp序列同源进化树比对
Fig.5 Comparison of homologous evolutionary trees of BcGLSp
2.8 灰葡萄孢菌在不同胁迫条件下菌丝生长率及BcGLS基因转录表达分析
不同胁迫条件(NaCl、SDS、H2O2)显著抑制灰葡萄孢菌菌丝的生长速率(图6,图7)。如图6-a和图7-a所示,随着NaCl浓度的逐渐升高,灰葡萄孢菌菌丝生长受到严重抑制,其生长速率呈现显著下降的趋势。细胞壁干扰剂SDS在低质量浓度(30 mg/L)时对灰葡萄孢菌菌丝的生长速率抑制率达到64.5%,而后随着SDS浓度的升高,其对灰葡萄孢菌菌丝生长均显著抑制,但不同高浓度SDS对菌丝生长抑制现象无显著变化(图6-b和图7-b)。在氧化应激胁迫下,10 mmol/L H2O2显著抑制菌丝的生长而高浓度如200 mmol/L H2O2下,灰葡萄孢菌菌丝生长完全受到抑制(图7-c)。
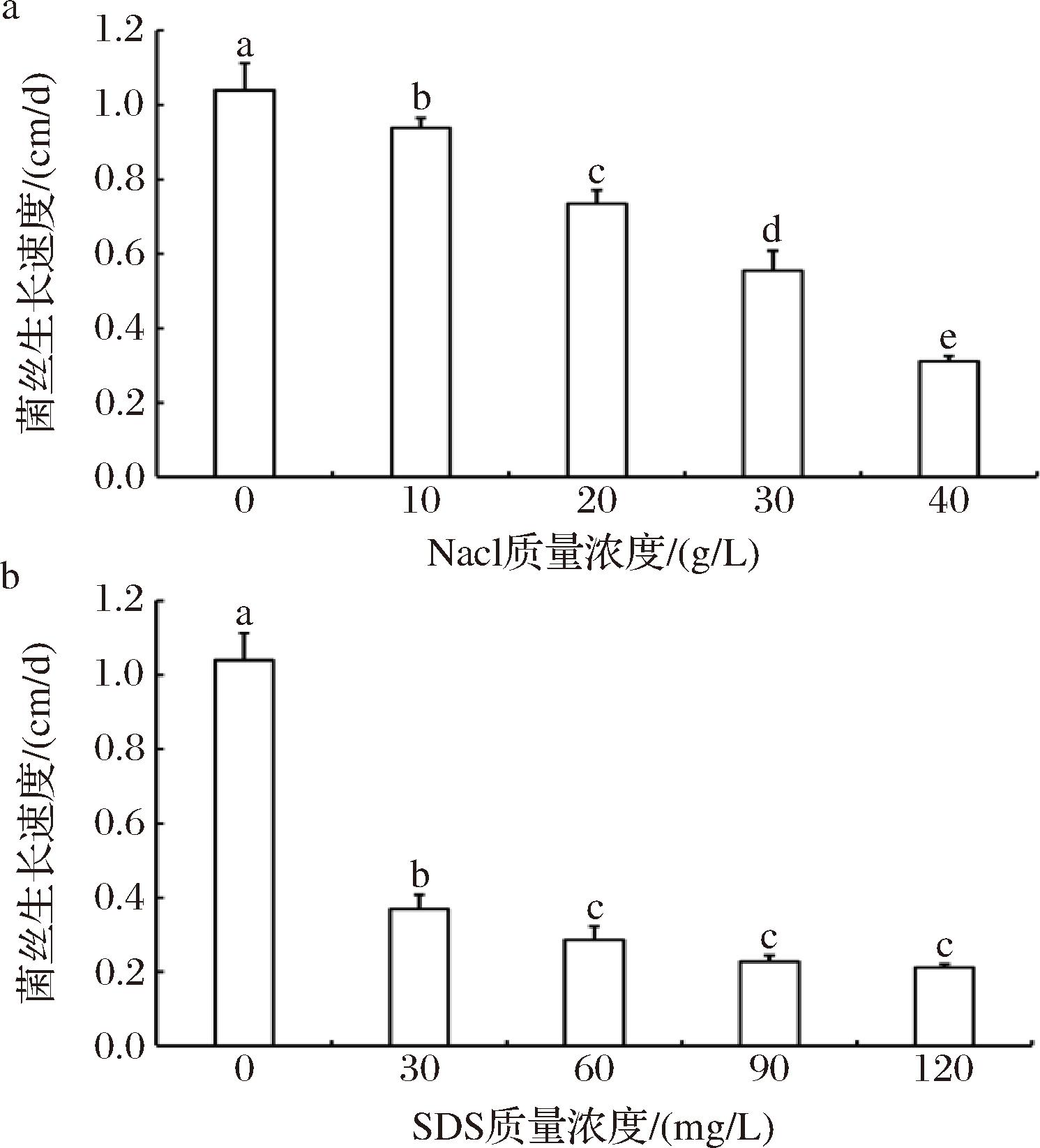
a-不同浓度NaCl下菌丝生长速率;b-不同浓度SDS下菌丝生长速率
图6 不同胁迫条件下灰葡萄孢菌S1菌丝生长速率
Fig.6 Mycelial growth rate of B.cinerea S1 under different abiotic stresses
注:图中不同小写字母表示不同处理间的差异显著(P≤0.05)(下同)。
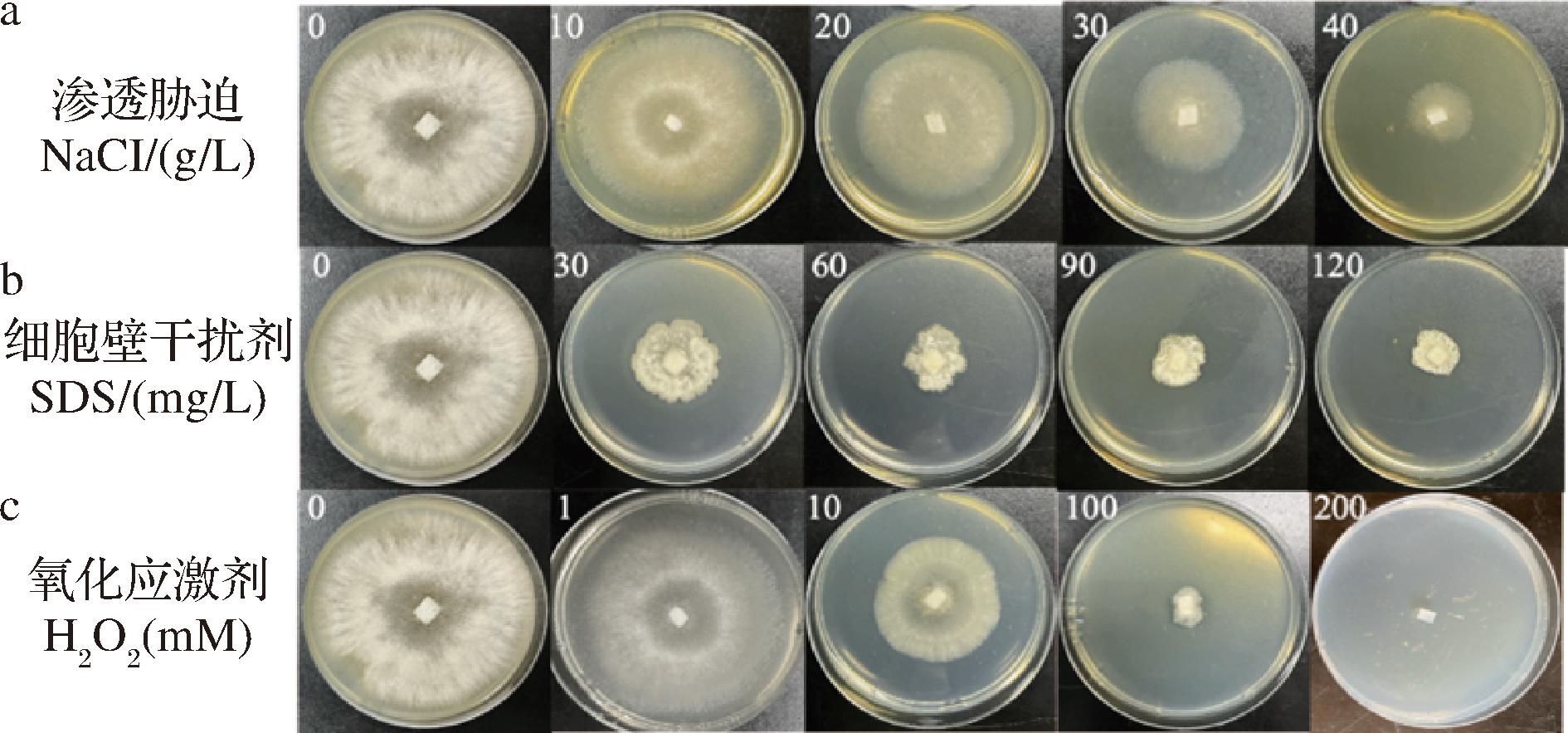
a-不同浓度NaCl下菌丝生长图;b-不同浓度SDS下菌丝生长图;c-不同浓度H2O2下菌丝生长图
图7 不同胁迫条件下灰葡萄孢菌S1菌丝生长图
Fig.7 Mycelial growth images of B.cinerea S1 under different abiotic stresses
灰葡萄孢菌BcGLS基因转录表达水平强烈响应环境胁迫,如NaCl、SDS和H2O2胁迫(图8)。灰葡萄孢菌生长过程中受到NaCl胁迫时,其BcGLS基因相对表达量增加32.43%,与对照相比,基因显著上调表达。SDS为阴离子表面活性剂,可使蛋白质变性,威胁真菌生长与存活;灰葡萄孢菌在含有SDS的液体培养基中生长缓慢,其BcGLS基因相对表达量增加131.3%,可见BcGLS基因强烈响应SDS造成的环境胁迫。高浓度H2O2对微生物有毒害作用,本研究中灰葡萄孢菌生长于含有100 mmol/L H2O2的液体培养基中,其BcGLS基因显著上调表达,上调表达量可达28.07%。综上所述,环境胁迫(NaCl、SDS、H2O2)显著影响灰葡萄孢菌BcGLS基因的转录表达水平。
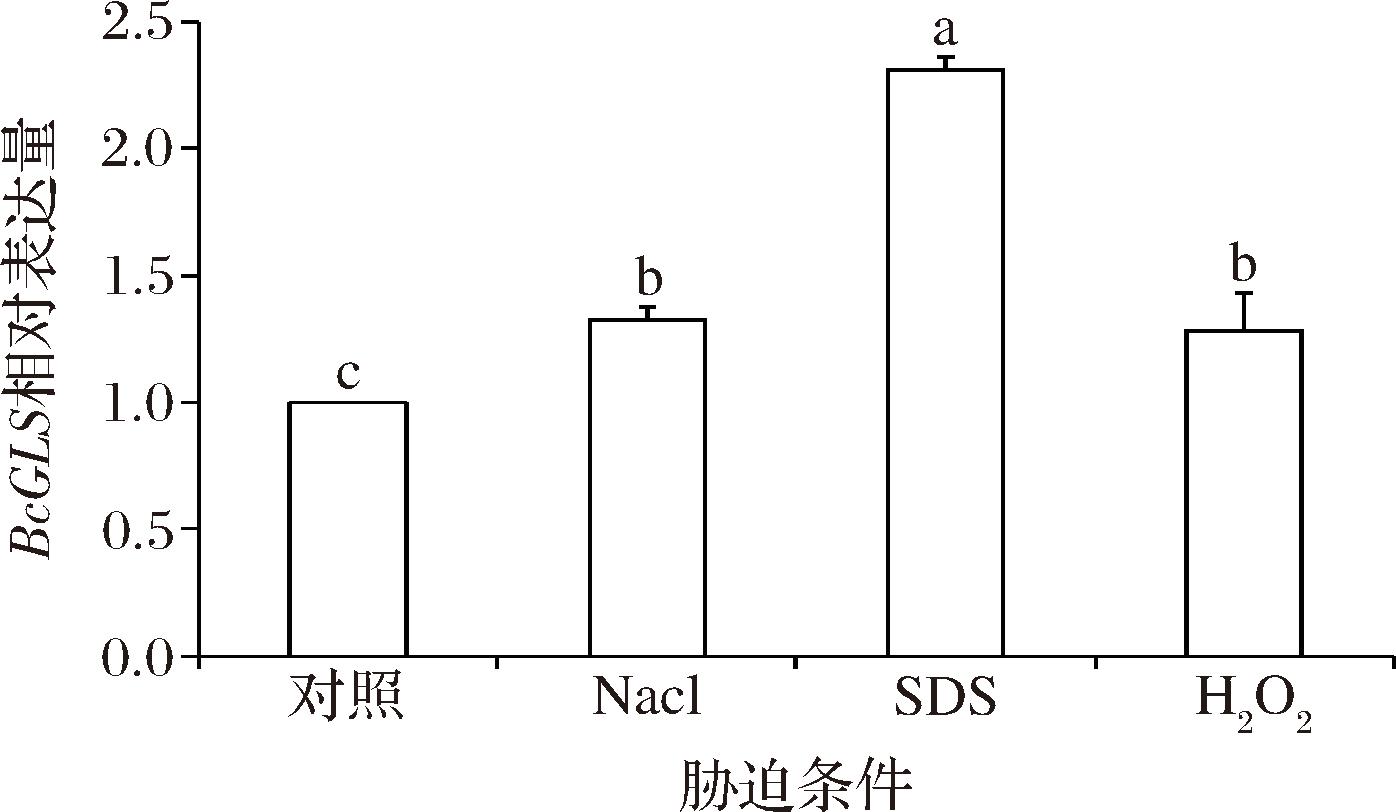
图8 不同胁迫条件下BcGLS基因的转录表达
Fig.8 Transcriptional expression of BcGLS gene under different stress conditions
注:相对表达量采用2-ΔΔCt法计算。
3 结论与讨论
灰霉病严重威胁园艺作物的产量和品质,灰葡萄孢菌作为引起灰霉病的主要病原菌,研究其生命过程中重要蛋白的功能和结构,寻找杀菌剂开发的潜在靶点,有助于新型机制杀菌剂的开发和灰葡萄孢菌的高效防治。真菌细胞壁是包裹在细胞外围的三维网状动态结构,会随着培养条件和环境压力的改变而不断变化[9]。细胞壁通过参与真菌的出芽生长、菌丝形成、交配和产孢等生理过程维持细胞的完整性,同时还作为支架承载、传递其表面的信号分子,直接影响真菌与外界环境的相互作用,因此在真菌致病性和毒力方面起着关键作用[23]。β-1,3-葡聚糖作为核心骨架是真菌细胞壁结构稳定的基础。因此,研究β-1,3-葡聚糖的合成并以此开发β-1,3-葡聚糖合成抑制剂对于阐明灰葡萄孢菌的侵染机制并进行高效防控具有重要意义。
β-1,3-葡聚糖由β-1,3-葡聚糖合成酶催化合成,以β-1,3-葡聚糖合成酶为靶标的杀菌剂/药物已在多个物种中被广泛研究,其中在医药领域以β-1,3-葡聚糖合成酶为靶标蛋白的抑制剂棘白菌素类药剂已被批准上市,但关于灰葡萄孢菌β-1,3-葡聚糖合成酶及其编码基因的研究几乎处于空白。本研究基于灰葡萄孢菌B05.10基因组数据,利用生物信息学手段在实验菌株BcS1中筛选并仅克隆到一个β-1,3-葡聚糖合成酶基因(FKS/GLS)同源的BcGLS基因,对其进行生物信息学分析和不同非生物胁迫下的表达分析。生物信息学分析显示BcGLp编码1 932个氨基酸,相对分子质量约220 kDa,为亲水性蛋白,其位于细胞膜上并存在17个跨膜通道,蛋白三维结构预测模型与已解析得到的酿酒酵母FKS三级结构重合度高,该结果与禾谷炭疽病菌(Colletotrichum graminicola)[19]、灰树花(Grifola frondosa)[24-25]、蛹虫草(Cordyceps militaris)[26]、酿酒酵母(Saccharomyces cerevisiae)[27]等真菌β-1,3-葡聚糖合成酶蛋白生物信息学分析结果一致。灰葡萄孢菌BcGLSp序列与植物致病真菌如山茶叶杯菌、二孢白粉菌、景天白粉菌、稻瘟病菌和禾谷炭疽病菌等的β-1,3-葡聚糖合成酶蛋白序列相似性达74%~95%,进化关系近,因此在稻瘟病菌和禾谷炭疽病菌中已揭示的β-1,3-葡聚糖合成酶基因在侵染进程中的功能作用对于研究灰葡萄孢菌β-1,3-葡聚糖合成酶及其编码基因功能具有可参考性。细胞壁是真菌细胞最外围结构,外界环境的变化直接影响其细胞壁的多糖合成。本研究揭示在NaCl、SDS和H2O2非生物胁迫下,灰葡萄孢菌菌丝生长均受到抑制而基因BcGLS的表达水平显著上升,表明外界胁迫条件通过调控BcGLS的表达影响灰葡萄孢菌细胞壁β-1,3-葡聚糖的合成进而调控其菌丝生长,证实了真菌细胞壁多糖结构随环境压力的改变而不断变化[23]。本研究结果初步确定了灰葡萄孢菌β-1,3-葡聚糖合成酶的结构性质特征以及β-1,3-葡聚糖合成酶基因在胁迫响应中发挥的重要作用,但β-1,3-葡聚糖合成酶基因在灰葡萄孢菌侵染进程中的作用机制以及β-1,3-葡聚糖合成酶的结构性质有待进一步研究。
[1] GARFINKEL A R.The history of Botrytis taxonomy,the rise of phylogenetics,and implications for species recognition[J].Phytopathology,2021,111(3):437-454.
[2] WILLIAMSON B,TUDZYNSKI B,TUDZYNSKI P,et al.Botrytis cinerea:The cause of gray mould disease[J].Molecular Plant Pathology,2007,8(5):561-580.
[3] POVEDA J,BARQUERO M,GONZ LEZ-ANDRÉS F.Insight into the microbiological control strategies against Botrytis cinerea using systemic plant resistance activation[J].Agronomy,2020,10(11):1822.
LEZ-ANDRÉS F.Insight into the microbiological control strategies against Botrytis cinerea using systemic plant resistance activation[J].Agronomy,2020,10(11):1822.
[4] SHAO W Y,ZHAO Y F,MA Z H.Advances in understanding fungicide resistance in Botrytis cinerea in China[J].Phytopathology,2021,111(3):455-463.
[5] ADNAN M,HAMADA M S,HAHN M,et al.Fungicide resistance of Botrytis cinerea from strawberry to procymidone and zoxamide in Hubei,China[J].Phytopathology Research,2019,1:17.
[6] 刘西莉,苗建强,张灿.植物病原菌抗药性及其抗性治理策略[J].农药学学报,2022,24(5):921-936.LIU X L,MIAO J Q,ZHANG C.Fungicide resistance and the management strategies[J].Chinese Journal of Pesticide Science,2022,24(5):921-936.
[7] CHEUNG N,TIAN L,LIU X R,et al.The destructive fungal pathogen Botrytis cinerea-Insights from genes studied with mutant analysis[J].Pathogens,2020,9(11):923.
[8] BI K,LIANG Y,MENGISTE T,et al.Killing softly:A roadmap of Botrytis cinerea pathogenicity[J].Trends in Plant Science,2023,28(2):211-222.
[9] GARCIA-RUBIO R,DE OLIVEIRA H C,RIVERA J,et al.The fungal cell wall:Candida,Cryptococcus,and Aspergillus species[J].Frontiers in Microbiology,2020,10:2993.
[10] LATGÉ J P.The cell wall:A carbohydrate armour for the fungal cell[J].Molecular Microbiology,2007,66(2):279-290.
[11] GEOGHEGAN I,STEINBERG G,GURR S.The role of the fungal cell wall in the infection of plants[J].Trends in Microbiology,2017,25 (12):957-967.
[12] GOW N A R,LATGE J P,MUNRO C A.The fungal cell wall:Structure,biosynthesis,and function[J].Microbiology Spectrum,2017,5(3):FUNK-0035-2016.
[13] ISHIHARA S,HIRATA A,NOGAMI S,et al.Homologous subunits of 1,3-beta-glucan synthase are important for spore wall assembly in Saccharomyces cerevisiae[J].Eukaryotic cell,2007,6(2):143-156.
[14] MART N V,GARC
N V,GARC A B,CARNERO E,et al.Bgs3p,a putative 1,3-beta-glucan synthase subunit,is required for cell wall assembly in Schizosaccharomyces pombe[J].Eukaryotic Cell,2003,2(1):159-169.
A B,CARNERO E,et al.Bgs3p,a putative 1,3-beta-glucan synthase subunit,is required for cell wall assembly in Schizosaccharomyces pombe[J].Eukaryotic Cell,2003,2(1):159-169.
[15] CORTÉS J C G,CARNERO E,ISHIGURO J,et al.The novel fission yeast (1,3)-beta-D-glucan synthase catalytic subunit Bgs4p is essential during both cytokinesis and polarized growth[J].Journal of Cell Science,2005,118(Pt 1):157-174.
[16] CORTÉS J C G,KONOMI M,MARTINS I M,et al.The (1,3)-beta-D-glucan synthase subunit Bgs1p is responsible for the fission yeast primary septum formation[J].Molecular Microbiology,2007,65(1):201-217.
[17] MOUYNA I,HENRY C,DOERING T L,et al.Gene silencing with RNA interference in the human pathogenic fungus Aspergillus fumigatus[J].FEMS Microbiology Letters,2004,237(2):317-324.
[18] RUIZ-HERRERA J,ORTIZ-CASTELLANOS L.Cell wall glucans of fungi.A review[J].The Cell Surface,2019,5:100022.
[19] OLIVEIRA-GARCIA E,DEISING H B.Infection structure-specific expression of β-1,3-glucan synthase is essential for pathogenicity of Colletotrichum graminicola and evasion of β-glucan-triggered immunity in maize[J].The Plant Cell,2013,25(6):2356-2378.
[20] IBE C,MUNRO C A.Fungal cell wall:An underexploited target for antifungal therapies[J].PloS Pathogens,2021,17(4):e1009470.
[21] CORTÉS J C G,CURTO M  ,CARVALHO V S D,et al.The fungal cell wall as a target for the development of new antifungal therapies[J].Biotechnology Advances,2019,37(6):107352.
,CARVALHO V S D,et al.The fungal cell wall as a target for the development of new antifungal therapies[J].Biotechnology Advances,2019,37(6):107352.
[22] THIYAGARAJAN K,LATINI A,CANTALE C,et al.Structural characterization of the DRF1 gene of Aegilops speltoides and comparison of its sequence with those of B and other Triticeae genomes[J].Euphytica,2020,216(10):152.
[23] CABIB E,ARROYO J.How carbohydrates sculpt cells:Chemical control of morphogenesis in the yeast cell wall[J].Nature Reviews Microbiology,2013.11(9):648-655.
[24] CUI F J,WU X H,TAO T L,et al.Functions of a glucan synthase gene GFGLS in mycelium growth and polysaccharide production of Grifola frondose[J].Journal of Agricultural and Food Chemistry,2019,67(32):8875-8883.
[25] JIANG L H,LI X F,ZAN X Y,et al.The β-1,3-glucan synthase gene GFGLS2 plays major roles in mycelial growth and polysaccharide synthesis in Grifola frondosa[J].Applied Microbiology and Biotechnology,2022,106(2):563-578.
[26] FU X,ZAN X Y,SUN L,et al.Functional characterization and structural basis of the β-1,3-glucan synthase CMGLS from mushroom Cordyceps militaris[J].Journal of Agricultural and Food Chemistry,2022,70(28):8725-8737.
[27] HU X L,YANG P,CHAI C D,et al.Structural and mechanistic insights into fungal β-1,3-glucan synthase FKS1[J].Nature,2023,616(7955):190-198.