黄曲霉毒素(aflatoxins,AF)是由黄曲霉、寄生曲霉等真菌毒素代谢产生的化学结构相似的一类化合物[1]。目前,已确定的黄曲霉毒素有20余种,主要为B类、G类和M类。黄曲霉毒素M1(aflatoxins M1,AFM1)来源于哺乳动物摄入受黄曲霉毒素B1(AFB1)污染的饲料或者作物后,在体内通过羟基化作用转换而来,易在乳及乳制品中引起污染[2-4]。长期食用此类乳制品,会对人及动物肝脏组织产生破坏作用,严重的甚至会引发畸变或癌变,对婴幼儿的影响尤为突出,列为I类致癌物[5]。因此,世界各地区对食品中的AFM1限量有着严格的规定,其中我国规定在乳制品中的AFM1最高残留量为0.5 μg/kg,欧盟对乳制品中AFM1的限量在0.05 μg/kg(GB 2761—2017《食品安全国家标准 食品中真菌毒素限量》),因此开发快速、简单、准确性高的AFM1检测方法具有重大意义。
酶联免疫吸附法(enzyme-linked immunosorbent assay,ELISA)由于其高通量、低成本和易于操作,已成为了食品中各种污染物大规模筛选检测中最常用的免疫分析技术,但是采用显色底物产生信号的传统ELISA灵敏度较低,并且检测环境复杂多变时,极易被污染从而产生假阳性影响检测结果[6-8]。因此,提高灵敏度和稳定性是食品真菌毒素快速检测未来研究的热点。目前,为了提高竞争性ELISA的灵敏度,已经研究了许多方法,包括筛选高亲和力抗体、制备低亲和力的包被抗原和使用荧光蛋白偶联抗体后放大检测信号的策略[9]。然而,制备高亲和力的抗体造价昂贵,在动物免疫过程中难以保证高亲和力抗体的制备成功,并由于抗体的结构复杂,不适合体外亲和力成熟。相比之下,通过基因操作手段使纳米抗体进行多聚化,是提高亲和力较为高效的手段[10-13]。
C4结合蛋白(recombinant C4 binding protein alpha,C4BPa)是一种由6~7个相同的α链,且不含有单个β-链组成的血浆糖蛋白[14]。α链的C端部位包含了两个半胱氨酸残基,该部位二级结构构象为α螺旋,该种特殊结构使得C4 bp应用于多肽和大分子蛋白质的多聚化中。因此,在本研究中,将以C4 bpα作为多聚体的基本结构框架,与实验室前期获得的纳米抗体(nanobody-M4,Nb-M4)融合制备一种七聚体融合蛋白,并以此构建icELISA法检测乳制品中的AFM1,为免疫分析技术中灵敏度的提升提供一种可行的方法。
1 材料与方法
1.1 材料与试剂
DH5α、RosettaTM(DE3)带有AFM1纳米抗体基因片段的重组pET25b+载体(pET25b-Nb-M4)由本课题组前期工作完成;限制性内切酶Xho I,苏州Takara公司;载体提取试剂盒,北京天根生物科技;AFM1标准品,美国Sigma公司;AFM1-牛血清白蛋白(bovine albumin,BSA),加拿大Immunechem公司;脱氧雪腐镰刀菌烯醇(deoxynivalenol,DON)、玉米赤霉烯酮(zearalenone,ZEN)、AFB1、AFB2、AFG1、AFG2、AFM2标准品和AFM1免疫亲和柱,青岛普瑞邦公司;氨苄西林(ampicillin,Amp)、卡那霉素(kanamycin,Kana)、硫酸新霉素(neomycin)、琼脂粉和3,3,5,5-四甲基联苯胺(tetramethylbenzidine,TMB),北京索莱宝公司;甲醇和乙腈(色谱纯)、高效液相色谱由C18柱,美国Thermo Fisher Scientific公司;96孔微孔板,亲和色谱柱和镍三乙酸(Ni-nitrilotriacetic acid,Ni-NTA)琼脂糖,上海生工生物科技;HisProbe-HRP,北京Thermo Fisher Scientific;编码C4 bpα的基因序列,苏州GENEWIZ。所有无机化学品和有机溶剂均为分析级。
1.2 仪器与设备
HP 1100型高效液相色谱系统,美国惠普公司;LC 20A高效液相色谱仪、DSC 60A差示扫描量热仪,日本岛津公司;AKTA PURE蛋白纯化仪,Cytiva公司;Multiskan FC酶标仪、PCR扩增仪,美国Thermo公司;Mini-PROTEAN® Tetra电泳仪、Octet分子相互作用仪,ForteBio公司。
1.3 实验方法
1.3.1 重组质粒pET25b-M4的构建
将实验室前期获得的载体pET25b-Nb-M4使用XhoI酶切和琼脂糖凝胶纯化,经柔性氨基酸连接子(GGGGS)同源重组后,将pET25b-Nb-M4载体与编码C4 bpα蛋白的基因序列以热转化(42 ℃,90 s)结合。后将产物电转至新鲜制备的大肠杆菌DH5α化学感受态细胞,并在37 ℃含100 μg/mL氨苄西林的琼脂平板培养过夜[15],采用通用引物:5′-TAATACGACTCACTATAG-3′,对pET25b-M4-C4 bpα进行DNA测序鉴定。
1.3.2 Nb-M4-C4 bpα融合蛋白的表达、纯化和鉴定
Nb-M4-C4 bpα通过自诱导表达产生。首先,通过热转化(42 ℃,90 s)将重组载体pET25b-Nb-M4转化到大肠杆菌Rosetta化学感受态细胞中,在含有10 μg/mL氨苄西林的100 mL自诱导振荡(25 ℃,250 r/min)培养过夜。然后,在4 ℃,8 000×g离心15 min收集细胞,并使用20 mL平衡缓冲液[2 mmol/L KH2PO4,8 mmol/L Na2HPO4,136 mmol/L NaCl,2.6 mmol/L KCl,1 mmol/L苯甲磺酰氟(benzylsulfonyl fluoride,PMSF)和60 mg溶菌酶]重悬。通过冰水浴超声破坏重悬的大肠杆菌细胞,防止靶蛋白降解或变性,之后将其在4 ℃条件下1 000×g离心15 min收集上清液,并通过0.22 μm过滤,将获得的可溶性七聚体融合蛋白命名为Nb-M4-C4 bpα。进一步,使用Ni-NTA亲和层析纯化含有组氨酸标签(histidine-tag,His-tag)的Nb-M4-C4 bpα融合蛋白,用300 mmol/L咪唑洗脱,再用10 mmol/L PBS(pH 7.4)透析。采用12%(体积分数)SDS-PAGE对Nb-M4-C4 bpα进行鉴定。使用Nanodrop2000测定Nb-M4-C4 bpα的浓度,置于-20 ℃保存。最后,利用PyMol软件对Nb-M4-C4 bpα进行结构预测(图1)。
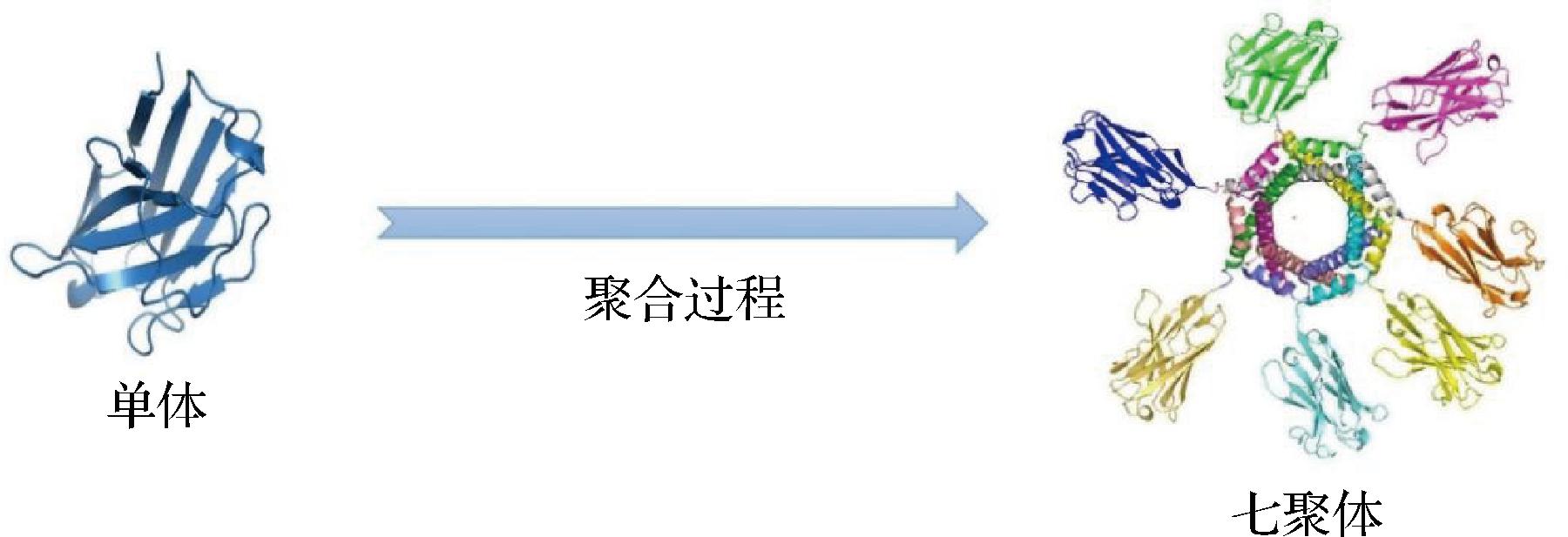
图1 AFM1纳米抗体多聚化示意图
Fig.1 Schematic representation of AFM1 nanobody multimerisation
1.3.3 Nb-M4-C4 bpα的亲和力分析
采用生物膜干涉技术(biolayer interferometry,BLI)测定Nb-M4-C4 bpα与AFM1-BSA的结合动力学曲线。在黑色微孔板中添加200 μL PBST(含0.02%体积分数的吐温-20)活化Ni-NTA传感器探针600 s;将Nb-M4-C4 bpα(1 μg/mL)固化于Ni-NTA传感器探针;探针置于0.01% PBST(体积分数,下同)中平衡90 s;随后在微孔板中分别加入不同浓度的AFM1-BSA(12.5、25、50、100 nmol/L),运行程序Association 120 s使得抗原抗体发生相互作用;运行程序Dissociation 180 s解离;结束运行程序后,将测量数据拟合到1∶1结合模型中[16]。
1.3.4 Nb-M4-C4 bpα的热稳定性分析
将Nb-M4-C4 bpα和Nb-M4稀释至0.1 μg/mL,分别进行如下加热处理:
a)5、15、25、35、45、55、65、75、85、95 ℃加热处理10 min,比较2种抗体在相同加热时间下,随着温度不断升高抗体残留活性的变化趋势。
b)37 ℃加热处理0、10、20、30、40、50、60 min,比较2种抗体在icELISA实验孵育温度下,随着孵育时间不断延长抗体残留活性的变化趋势。
c)85 ℃加热处理0、10、20、30、40、50、60 min,比较2种抗体在持续高温热处理条件下抗体残留活性的变化趋势。
待抗体恢复至室温后,采用icELISA法评估抗体的热稳定性,具体操作:使用0.05 mol/L pH 9.6的碳酸盐缓冲液稀释AFM1-BSA至0.5 μg/mL,每孔100 μL包被于酶标板,4 ℃过夜;弃包被液,洗板后每孔加入300 μL 5%体积分数的脱脂乳,37 ℃封闭1 h;洗板后每孔加入100 μL抗体,37 ℃孵育1 h;洗板后,Nb-M4-C4 bpα孔加入100 μL anti-His tag IgG/HRP,Nb-M4孔加入100 μL anti-rat IgG/HRP,37 ℃孵育1 h;洗板后每孔加入100 μL TMB,37 ℃显色7 min,立即加入1 mol/L HCl终止反应,测定450 nm处的吸光度A。未经热处理的抗体的活性为100%,抗体残留活性的计算如公式(1)所示:
抗体残留活性![]()
(1)
[:A1为经热处理抗体的吸光度;A2为未经热处理抗体的吸光度[17]。
1.3.5 Nb-M4-C4 bpα的最佳工作浓度确定
采用棋盘法将抗原AFM1-BSA稀释为0.25、0.5、1、2、4 μg/mL,100 μL/孔包被于96孔酶标板,4 ℃包被过夜;0.1% PBST洗板后添加300 μL/孔5%质量分数的脱脂乳粉,37 ℃封闭1 h;0.01% PBST洗板后,将纯化好的Nb-M4-C4 bpα用0.01 mol/L PBS pH 7.4梯度稀释(1∶500~1∶32 000),50 μL Nb-M4-C4 bpα与等体积10%(体积分数,下同)甲醇/PBS混合加入96孔酶标板,37 ℃孵育1 h;0.1% PBST洗板后,加入抗His二抗与脱脂乳粉质量比为1∶4 000的稀释液,37 ℃孵育1 h;0.01% PBST洗板后,加入TMB显色液,在37 ℃进行显色反应7 min后,终止反应,测定OD450值,将OD450值为1.0~1.1时Nb-M4-C4 bpα的稀释倍数作为抗体最佳工作浓度。
1.3.6 Nb-M4-C4 bpα icELISA反应体系的优化
免疫分析检测中,其检测能力受到检测体系影响,为了获得更高的信号反应和更好的灵敏度,需要对Nb-M4-C4 bpα构建的icELISA检测方法中的各参数进行优化,以最低IC50值和OD450值为检测标准进行优化。
a)封闭剂优化:采用不同质量分数(下同)的封闭剂[5%脱脂乳粉、5% BSA和5%鸡卵白蛋白(ovalbumin,OVA)]进行Nb-M4-C4 bpα构建的icELISA,选择IC50值最低时的封闭液作为最佳封闭液进行后续实验。
b)pH值的优化:采用不同的pH(5.0、6.0、7.0、7.4、8.0和9.0)的0.01% PBST溶液配制Nb-M4-C4 bpα进行icELISA实验,选择IC50值最低的pH值作为最佳pH值。
c)离子强度优化:用磷酸盐调节0.01%PBST溶液(体积分数),使其离子强度为5、10、20、40、80、100 mmol/L。用不同离子强度的PBST溶液配制Nb-M4-C4bpα进行icELISA,选择IC50值最低时的离子强度作为最佳离子强度值。
d)甲醇含量优化:采用含有不同甲醇体积分数(5%、10%、15%和20%)的PBS溶液配制成系列浓度的AFM1标准品溶液进行icELISA,选择IC50值最低时的甲醇含量作为最佳甲醇含量。
1.3.7 特异性实验
为了评估所建立icELISA的特异性,采用AFM1结构类似物AFB1、AFB2、AFG1、AFG2和其他常见真菌毒素(DON、ZEN)进行Nb-M4-C4 bpα的交叉反应(cross reaction,CR),计算如公式(2)所示:
(2)
[:IC50定义为对AFM1-BSA/Nb-M4-C4 bpα结合达到50%抑制作用的分析物浓度,具体以B/B0表达该数值。
1.3.8 基质评估效应
用于基质效应实验的牛乳购自内蒙古自治区呼和浩特市市场,经过LC-MS/MS鉴定为AFM1阴性。将1 g牛乳与5 mL含有50%甲醇的5 mmol/L PBS pH 7.4混合,经过超声处理15 min,1 000×g离心15 min后,吸取上清液经过0.45 μm过滤器除去杂质。然后,在上清液中加入不同浓度的AFM1标准品,所得加标样品直接用ELISA法检测。
1.3.9 实验的实用性
为了评估该方法在真实样品中的实用性,将1 mL牛乳与不同质量浓度(0.2、0.3、0.4 ng/mL)的AFM1进行混合提取。稀释后的样品溶液用于本实验所构建的icELISA方法检测AFM1。同时,用0.45 μm过滤提取液,然后将其进行HPLC检测进行对照[18-19]。
1.3.10 数据处理
每组实验至少重复3次,实验数值以平均值±标准偏差(mean±SD)表示,应用IBM SPSS 25软件对实验数据进行单因素方差分析(one-way analysis of variance,ANOVA)统计学分析,P<0.05具有统计学意义,采用软件Origin Pro 2022绘图[20]。
2 结果与分析
2.1 Nb-M4-C4 bpα融合蛋白的表达、纯化和鉴定
本实验以实验室先前获得的AFM1纳米抗体Nb-M4作为研究对象,为提高其检测灵敏度,采用多聚策略,选取C4 bpα为连接桥梁,与Nb-M4进行融合,构建了一种七聚体融合蛋白Nb-M4-C4 bpα。C4 bpα的C端寡聚结构域含有2个半胱氨酸,在分子间形成了7个二硫键桥,构建了七聚体中稳定的C4 bpα肽。单体Nb-M4和七聚体Nb-M4-C4 bpα的理论分子质量分别为30 kDa和210 kDa,经SDS-PAGE鉴定发现(图2),七聚体Nb-M4-C4 bpα分子质量约245 kDa,单体Nb-M4的分子质量约30 kDa,条带清晰无杂带,与理论数值基本一致。该结果表明,Nb-M4可以通过C4 bpα中的7个二硫键桥形成稳定的七聚体Nb-M4-C4 bp。
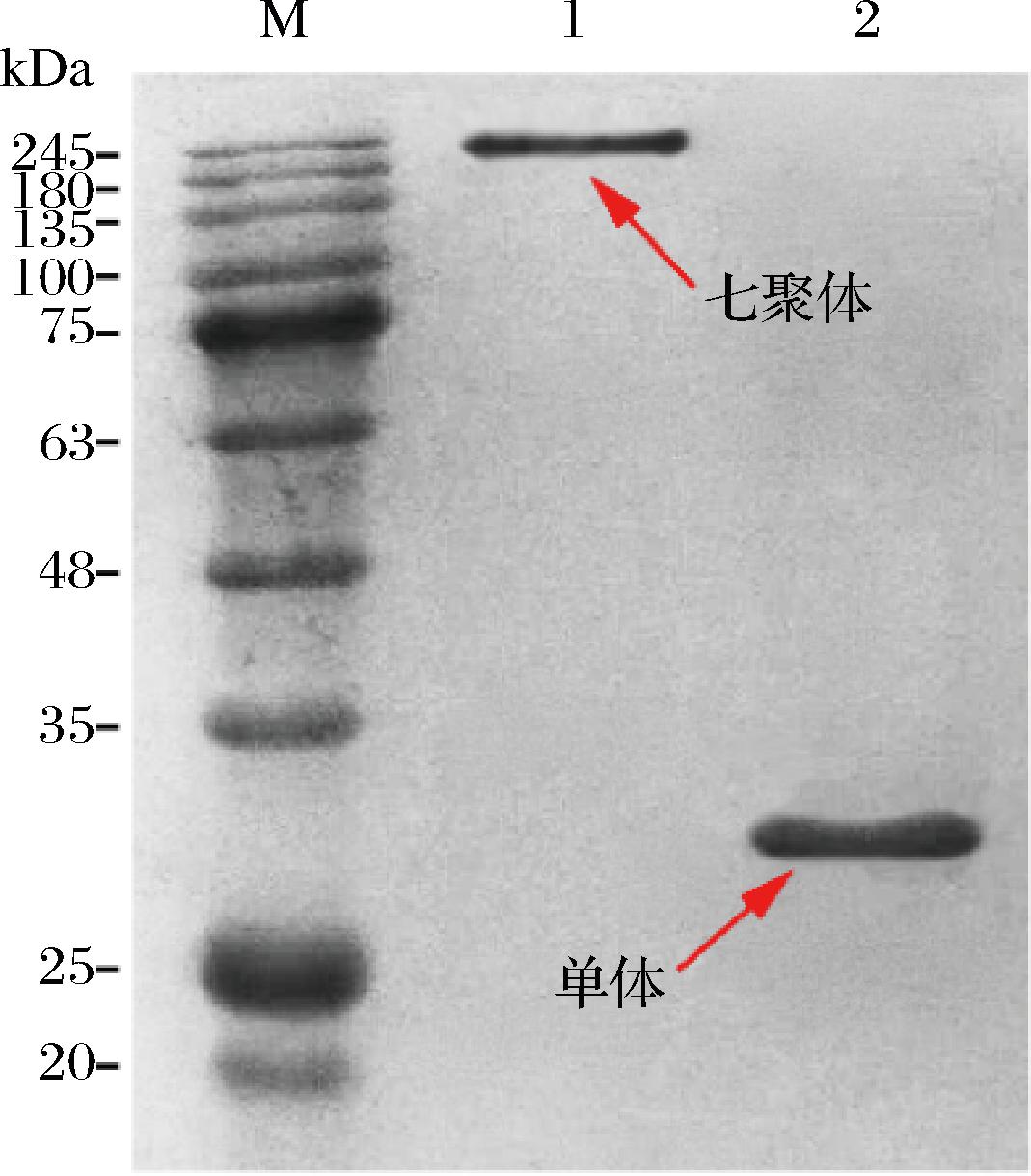
M-DNA MARKER;泳道1-七聚体;泳道2-单体
图2 Nb-M4-C4 bpα蛋白的SDS-PAGE鉴定
Fig.2 SDS-PAGE analysis of the M4-C4 bpα fusion protein
2.2 Nb-M4-C4 bpα的亲和力分析
为评估七聚体Nb-M4-C4 bpα与AFM1-BSA的结合亲和力,本研究使用BLI对抗原抗体的结合过程进行了实时监测,测定了抗原抗体的结合速率常数(association rate constant/on-rate,Kon,M-1·s-1)和解离速率常数解离速率常数(dissociation rate constant,Koff,s-1),并计算得到平衡解离常数(equilibrium binding,KD),公式为KD=Koff/Kon。KD可表征抗体亲和力强弱,是指抗体能够识别和结合的最低抗原浓度,KD值越小抗体的亲和力越强。根据图3可知,七聚体Nb-M4-C4 bpα的KD值为1.12 nmol/L,单体Nb-M4的KD值为2.5 nmol/L[21],该结果表明多聚化方法有效提升了单体Nb-M4的亲和力。
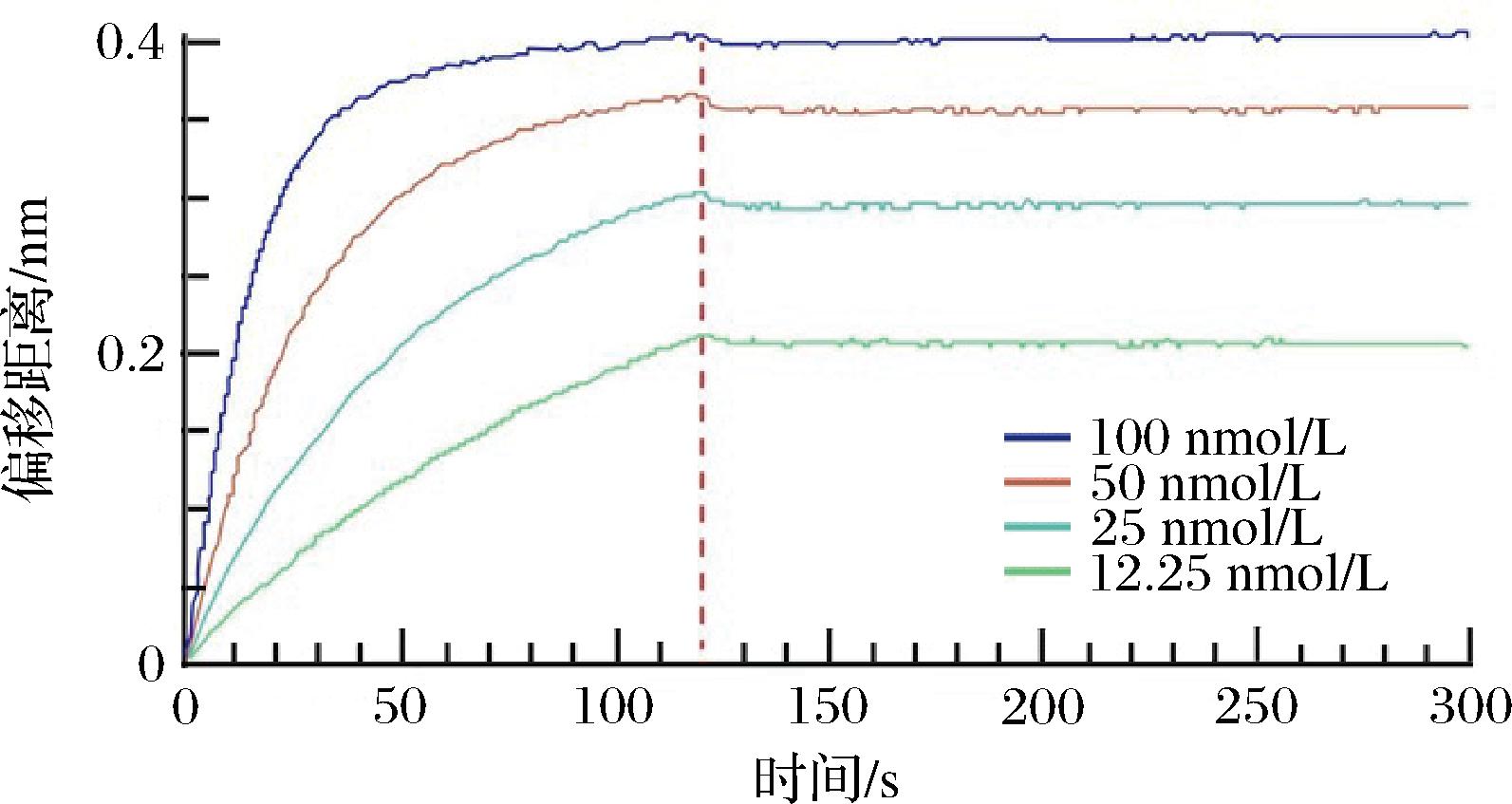
图3 Nb-M4-C4 bpα的动力学结合曲线
Fig.3 Kinetic binding curves of Nb-M4-C4 bpα
2.3 Nb-M4-C4 bpα的热稳定性分析
2.3.1 Nb-M4-C4 bpα耐热性分析
抗体对高温的耐受性是其发挥功能的关键因素,在实际生产应用中,因为环境温度变化较高,如果抗体对于高温耐受性较低,便会受到很大限度的使用场景限制。抗体经过加热处理后,其结构会发生折叠,待冷却后,经加热处理结构会展开,因此抗体的折叠恢复能力成为评价一款抗体的重要特征[22],这种可逆性是纳米抗体区别于常规抗体的主要标志[23]。
为此,本研究对Nb-M4-C4 bpα的热稳定性进行了评估,并与单体Nb-M4进行了比较。根据图4-A可知,在连续加热的温度下孵育10 min后,Nb-M4-C4 bpα和Nb-M4的结合能力均随着温度的升高而下降;经85 ℃热处理10 min后,Nb-M4完全失去活性,而Nb-M4-C4 bpα仍保留约12%的结合活性;当温度持续升高至90 ℃时,Nb-M4-C4 bpα仍然可以与抗原相互作用。在37 ℃连续热处理条件下(图4-C),Nb-M4的活性下降趋势较为明显,直至60 min活性残余50%,而Nb-M4-C4 bpα的活性变化较小,经37 ℃热处理60 min后仍保留70%的抗体活性。为了探究抗体经高温孵育后的可逆性,将2种抗体在85 ℃下进行了不同时间的处理,该结果显示Nb-M4经85 ℃热处理10 min后,抗原结合能力完全丧失,而Nb-M4-C4 bpα仍保留了约35%的结合活性,并且随着热处理时间延长至60 min,仍保留约25%的结合活性(图4-B)。因此,与单体Nb-M4相比,Nb-M4-C4 bpα具有更好的重折叠能力,可作为免疫测定的替代试剂。
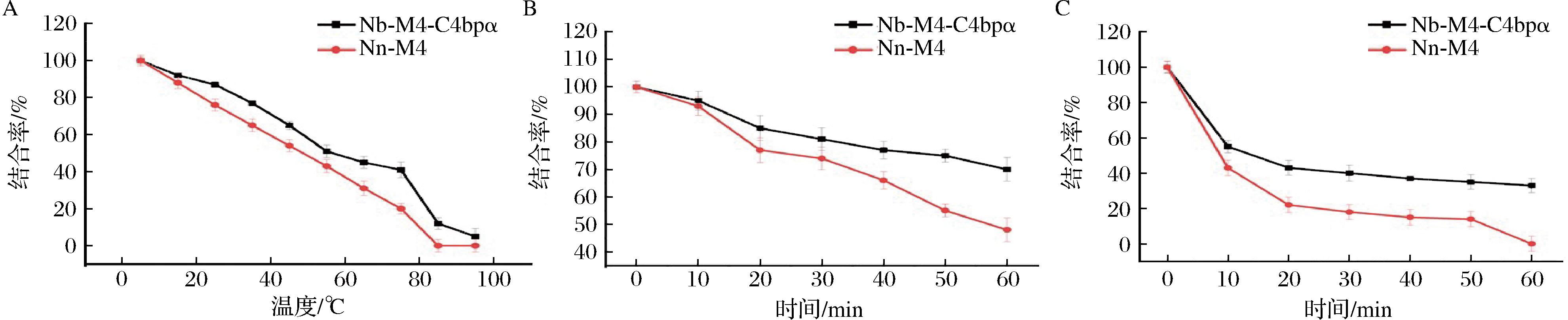
A-连续热处理10 min;B-37 ℃保持10 min;C-85 ℃保持10 min
图4 Nb-M4-C4 bpα的热稳定分析
Fig.4 Thermal stability analysis of Nb-M4-C4 bpα
2.3.2 Nb-M4-C4 bpα的解链温度
解链温度(Tm)值是抗体热稳定性的另一种体现方式,Tm值越高,代表抗体的热稳定性越高,通过DSC测试出的峰值出现的温度越高,代表抗体的耐热性越好。由于纳米抗体分子质量小,仅由一个可变区结构域组成,因此仅有一个Tm温度值。根据图5可知,七聚体Nb-M4-C4 bpα的Tm为71.33 ℃,该结果与此前报道纳米抗体的Tm值在47~85 ℃期间的结果相一致[24]。根据先前研究报道[25],Nb热稳定性较高是因为FR1区的半胱氨酸和FR3区的半胱氨酸会形成一对二硫键,形成稳定构象,增加其结构稳定性,单体Nb-M4进行七聚化后会形成7个稳定的键桥,这一结构会提升纳米抗体的耐热性,从而使得七聚体Nb-M4-C4 bpα能够承受高温和恶劣的极端环境。

图5 Nb-M4-C4 bpα DSC分析
Fig.5 DSC scans of Nb-M4-C4 bpα
2.4 Nb-M4-C4 bpα的最佳工作条件的确定
采用棋盘法确定了包被抗原(AFM1-BSA)和抗体(七聚体Nb-M4-C4 bpα)的最佳工作浓度,结果如表1所示。当七聚体Nb-M4-C4 bpα稀释倍比为1∶8 000 时,OD450值接近1.0,因此选择了1∶8 000作为七聚体Nb-M4-C4 bpα作为最佳工作浓度。同理,当AFM1-BSA质量浓度为0.5 μg/mL时,OD450接近1.0,因此选择0.5 μg/mL作为AFM1-BSA的最佳工作浓度,以该浓度下所建立的竞争性抑制曲线(图6),符合四参数逻辑方程的基本规律,具有良好的线性关系(R2=0.995)。
表1 棋盘滴定法确定最佳抗体包被浓度
Table 1 Checkerboard titration to determine the concentration of antibody encapsulation
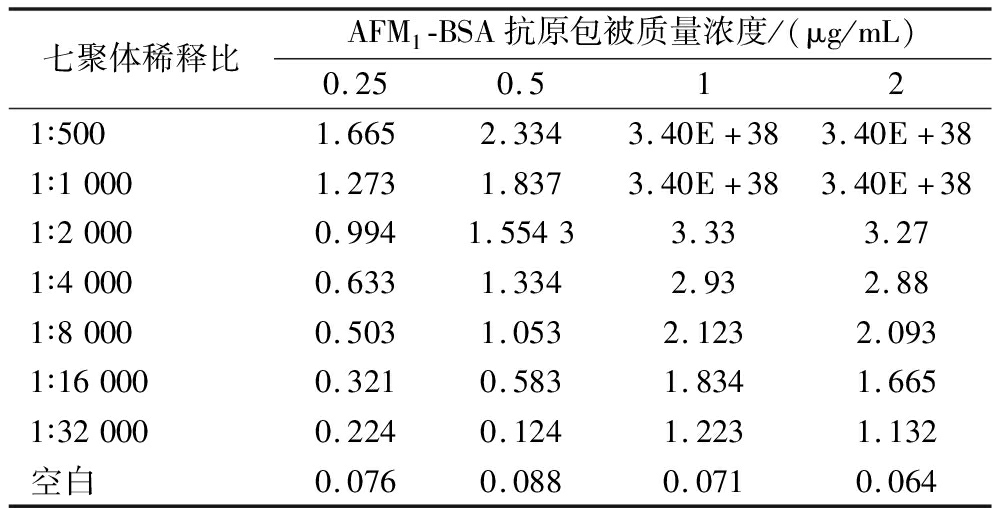
七聚体稀释比AFM1-BSA抗原包被质量浓度/(μg/mL)0.250.5121∶5001.6652.3343.40E+383.40E+381∶10001.2731.8373.40E+383.40E+381∶20000.9941.55433.333.271∶40000.6331.3342.932.881∶80000.5031.0532.1232.0931∶160000.3210.5831.8341.6651∶320000.2240.1241.2231.132空白0.0760.0880.0710.064
注:表身数据为不同组别的OD450,OD450=3.40E+38表示超出酶标仪最大检测限;酶标二抗工作浓度1∶10 000。

图6 Nb-M4-C4 bpα icELISA法竞争抑制曲线
Fig.6 Competitive Inhibition curve for Nb-M4-C4 bpα icELISA
2.5 Nb-M4-C4 bpα icELISA反应体系的优化
进一步,对icELISA反应体系中的封闭剂、pH值、离子强度和甲醇含量等参数进行了优化,以IC50值和OD450值为评价指标。实验选择5%脱脂乳粉、5% BSA、5% OVA封闭96孔酶标板以减少非特异性吸附,结果如图7-A所示,使用3种封闭剂的icELISA的IC50值分别为0.17、0.85、0.21 ng/mL,因此选择5%脱脂乳粉作为封闭剂。实验中分别设置不同的pH条件进行icELISA检测。结果如图7-B所示,当pH=7.4时,具有最低的IC50值为0.54 ng/mL,因此选择pH 7.4作为最适pH值。由于AFM1是高度亲脂性小分子污染物,在进行实际样品检测时,需要进行甲醇溶解AFM1,当甲醇含量过低会造成AFM1溶解不彻底,当甲醇含量过高时会抑制抗原和抗体的结合反应,从而影响检测结果。实验结果如图7-C所示,当甲醇体积分数高于20%时,IC50值较大;当选择甲醇体积分数为5%,IC50值为1.15 ng/mL,效果最好,因此选择5%的甲醇作为最适甲醇含量。实验中分别设置不同的离子强度进行ELISA,结果如图7-D所示,当离子强度为20~80 mmol/L时,IC50变化较小,当离子强度为10 mmol/L时,具有最低的IC50值为1.23 ng/mL,因此选择10 mmol/L作为最适离子强度。
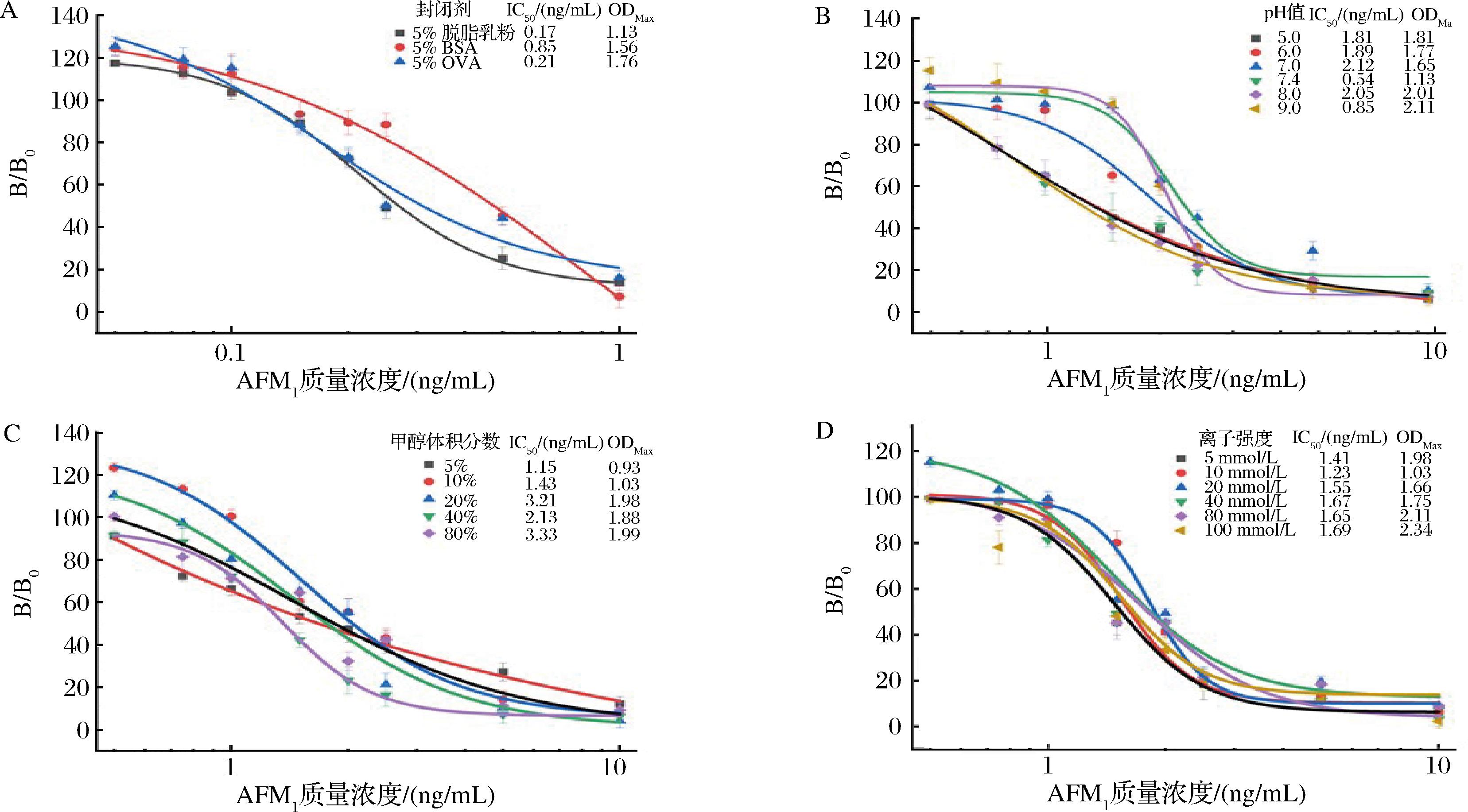
A-封闭剂;B-pH值;C-甲醇体积分数;D-离子强度
图7 AFM1 ic ELISA体系的优化
Fig.7 The optimization of the AFM1 icELISA assay
2.6 Nb-M4(单体)与Nb-M4-C4 bpα(七聚体)的检测性能比较
为了评价单体Nb-M4与七聚体Nb-M4-C4 bpα的检测性能,通过检测不同浓度的AFM1,比较了两者的竞争抑制曲线,如图8所示,七聚体Nb-M4-C4 bpα具有着较高的灵敏度,IC50值为0.13 ng/mL,检测限(IC10)为0.009 ng/mL。灵敏度的提高主要是由于:a)Nb-M4-C4 bpα分子间二硫键结合而成的七聚体,在icELISA检测过程中有着更好的反应接触面积,因此具有较高的亲和力;b)Nb-M4-C4 bpα在反应过程中会提供更多的HisProbe-HRP结合位点,有助于放大检测过程中的信号。然而,当多聚化纳米抗体用于icELISA时,多聚体上多余的纳米抗体会干扰到靶体与包被抗原之间的竞争,从而降低灵敏度。总的来说,icELISA受到以上2个因素影响。
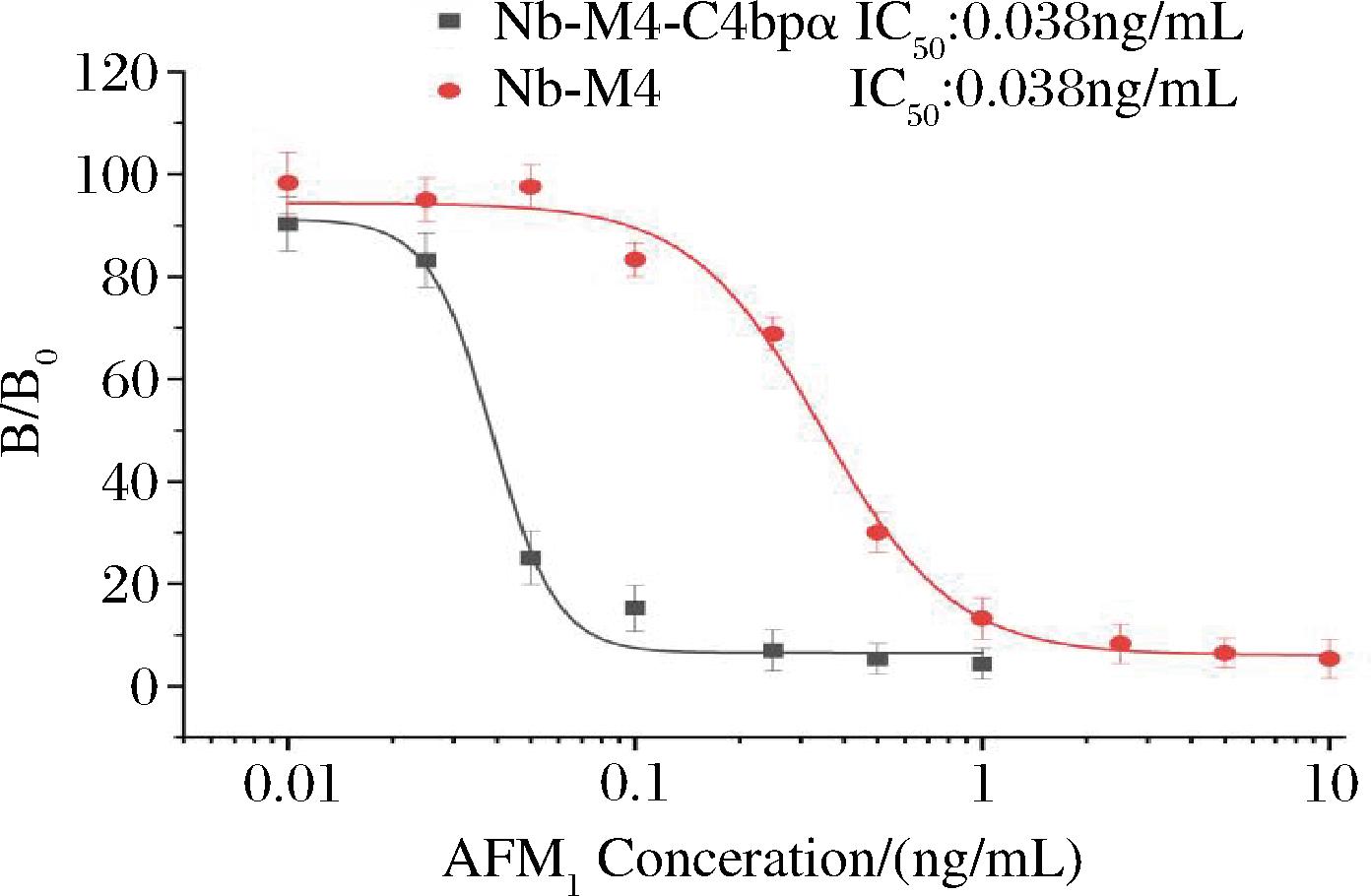
图8 单体(Nb-M4)和七聚体(Nb-M4-C4 bpα)的竞争性抑制曲线
Fig.8 Competitive inhibition curves for monomer (Nb-M4) and heptamer (Nb-M4-C4 bpα)
2.7 icELISA的交叉反应
为了确定所建立的icELISA检测AFM1的特异性,在最佳实验条件下评估对AFB1、AFB2、AFG1、AFG2、DON、ZEN的交叉反应性,其结果以交叉反应率表现如图9所示,该方法对于常见的真菌毒素交叉反应率可以忽略不计(<0.5%),与AFM1结构相似的真菌毒素AFM2交叉反应率为2.32%,与AFB1交叉反应率为1.08%。结果表明,七聚体Nb-M4-C4 bpα保留了纳米抗体Nb-M4的识别特征。此外,该部分结果证明了基于七聚体Nb-M4-C4 bpα的icELISA检测AFM1的高选择性。
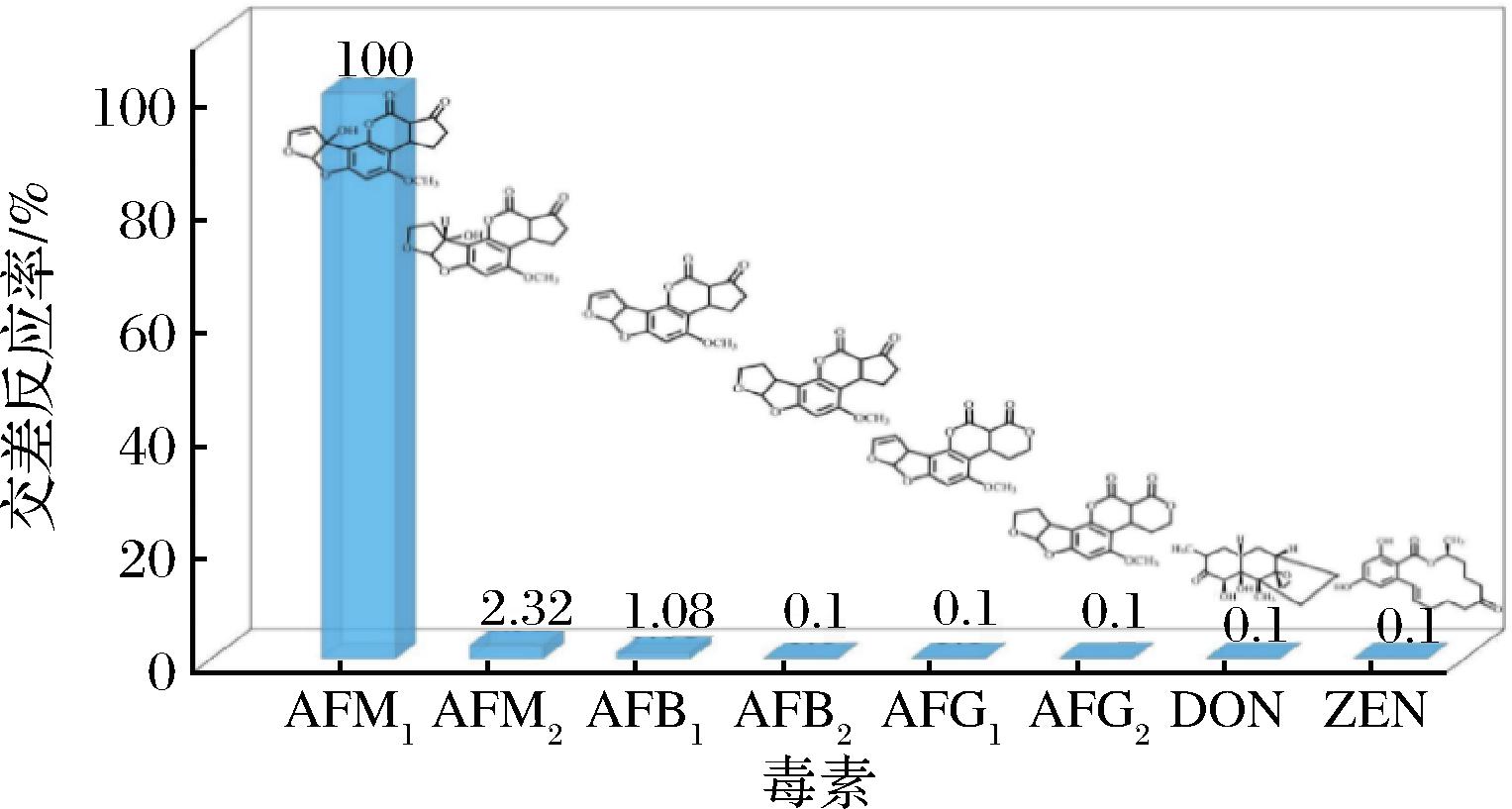
图9 Nb-M4-C4 bpα交叉反应率
Fig.9 Nb-M4-C4 bpα cross-reactivity
2.8 基质效应
基质效应是影响实验准确性的重要指标,为了探究所建立了的检测方法在在实际样品分析中,是否会受到实际样品基质的影响,选取牛乳、酸乳和乳粉三类样品按着前处理方法进行处理后,用于本研究所建立的icELISA方法中探究基质影响。结果如图10所示,使用三类乳品提取液所建立的竞争抑制曲线与10%(体积分数)甲醇/PBS缓冲液所建立的标曲无显著差别,说明样品中的基质对所构建的方法并未造成太大影响,因此证明了Nb-M4-C4 bpα构建的icELISA法在乳制品检测中的应用可行性。
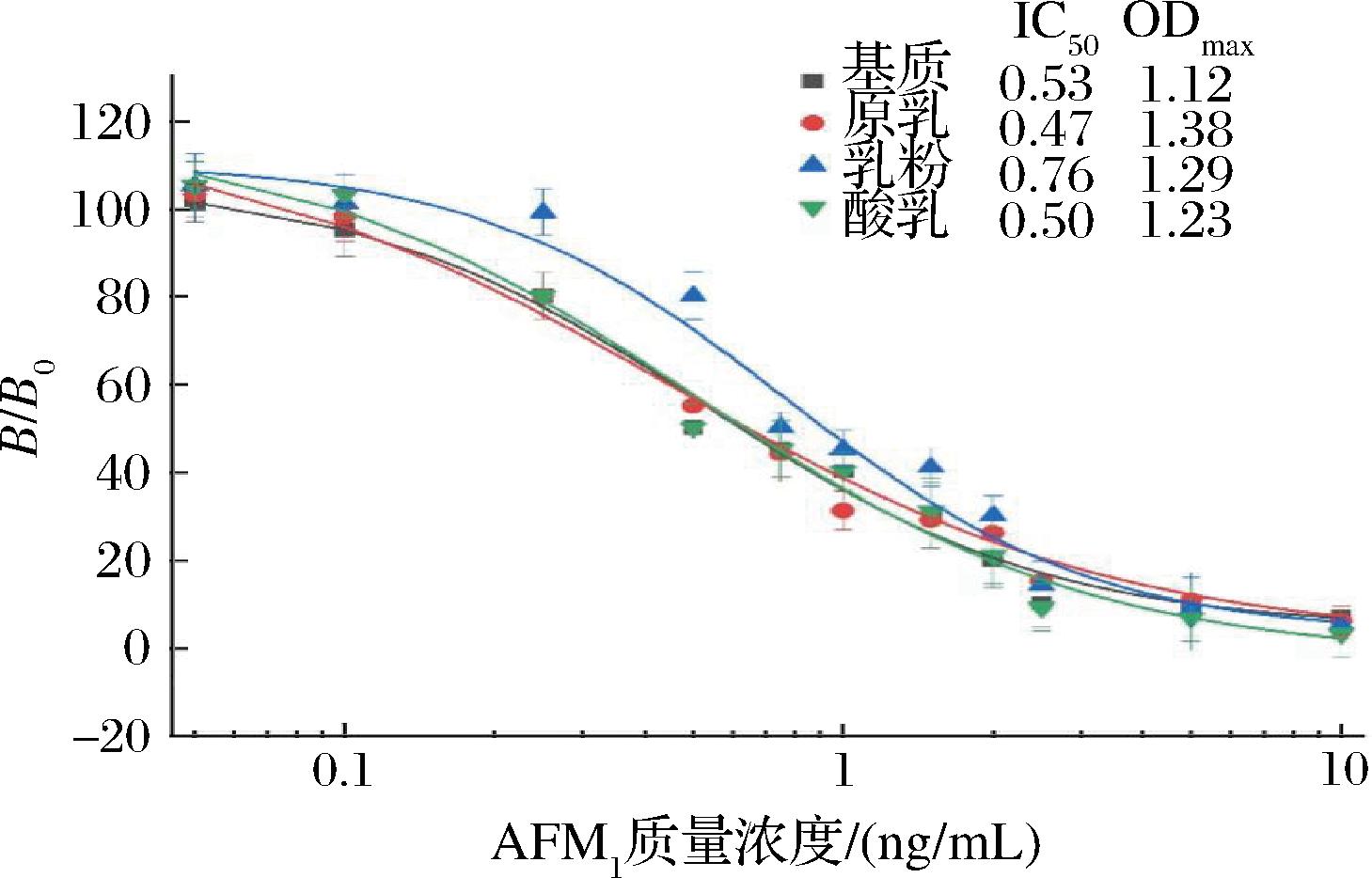
图10 Nb-M4-C4 bpα基质效应影响
Fig.10 Influence of Nb-M4-C4 bpα matrix effects
2.9 icELISA加标回收
为了评测该方法在实际样品分析中的实用性,在不含有AFM1的牛乳样品中加入一系列AFM1标准品(0.2、0.3、0.4 ng/mL)提取后,采用icELISA方法检测。以回收率和相对标准偏差(relative standard deviation,RSD)来评价该方法的准确性和重复性。如表2所示,平均加样回收率为82.32%~99.23%,RSD为2.31%~5.22%;法间加样回收率为77.05%~92.32%,RSD为2.55%~6.02%。为了验证所建立icELISA方法的可靠性,进一步以HPLC对加标样品进行了分析。结果表明,2种方法之间具有良好的相关性(R2=0.995)(图11),所构建的Nb-M4-C4 bpα酶联免疫吸附实验具有良好的实用性。
表2 通过基于Nb-M4-C4 bpα七聚体的ELISA和HPLC测试实际样品中的回收率
Table 2 Recovery in real samples tested by ELISA and HPLC based on M4-C4 bpα heptamer
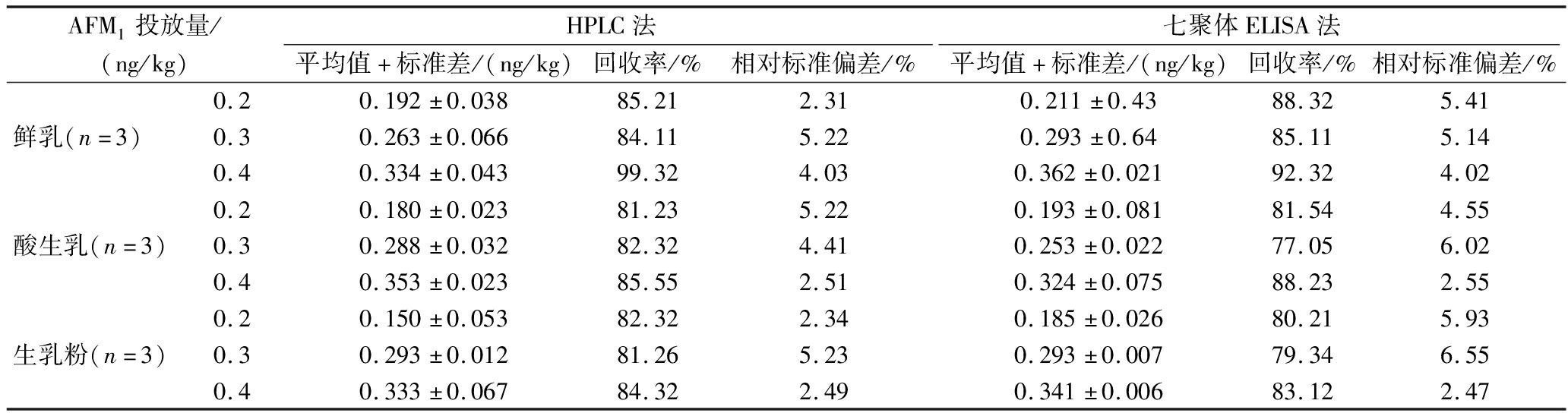
AFM1投放量/(ng/kg)HPLC法七聚体ELISA法平均值+标准差/(ng/kg)回收率/%相对标准偏差/%平均值+标准差/(ng/kg)回收率/%相对标准偏差/%0.20.192±0.03885.212.310.211±0.4388.325.41鲜乳(n=3)0.30.263±0.06684.115.220.293±0.6485.115.140.40.334±0.04399.324.030.362±0.02192.324.020.20.180±0.02381.235.220.193±0.08181.544.55酸生乳(n=3)0.30.288±0.03282.324.410.253±0.02277.056.020.40.353±0.02385.552.510.324±0.07588.232.550.20.150±0.05382.322.340.185±0.02680.215.93生乳粉(n=3)0.30.293±0.01281.265.230.293±0.00779.346.550.40.333±0.06784.322.490.341±0.00683.122.47
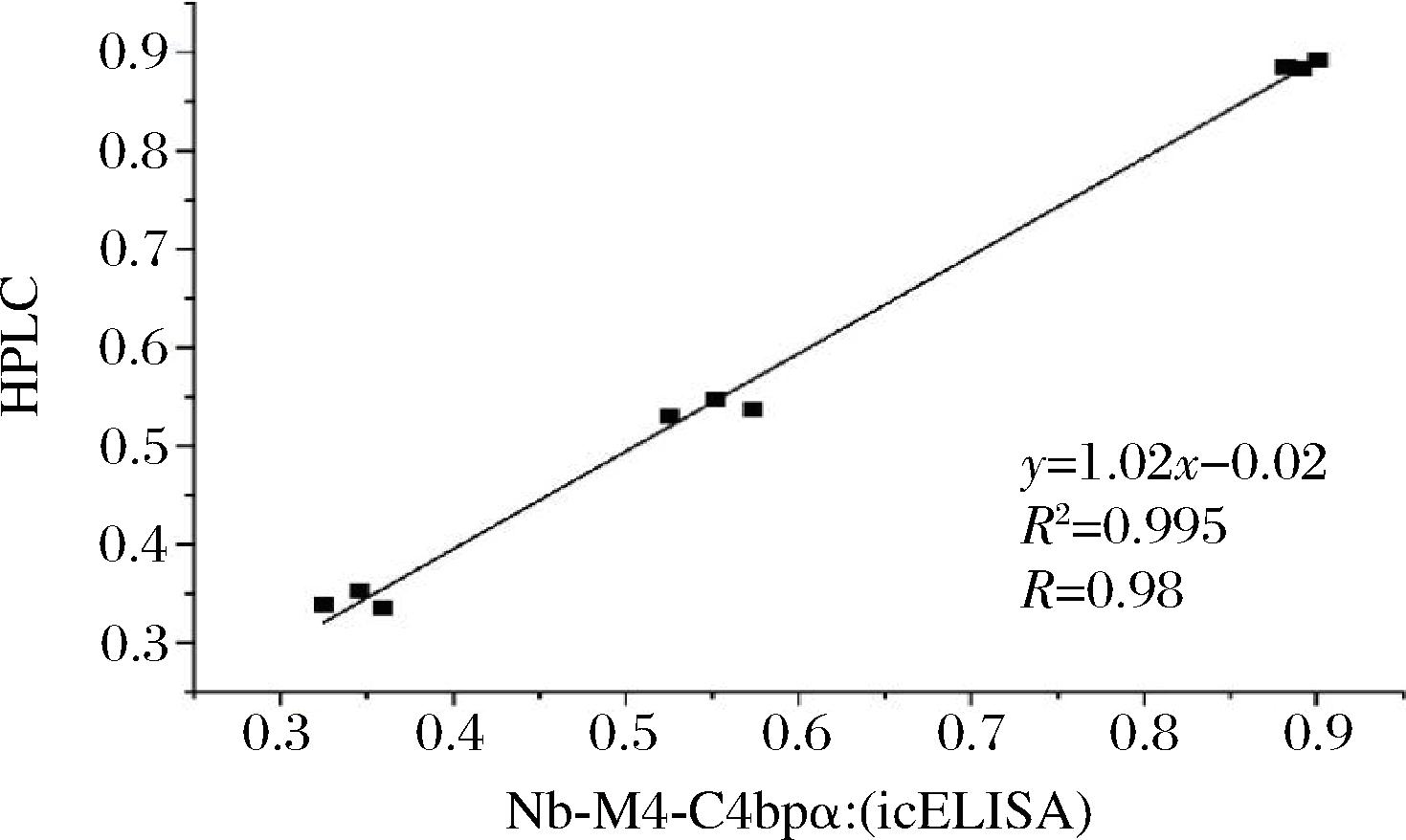
图11 Nb-M4-C4 bpα icELISA与HPLC相关性分析
Fig.11 Correlation of Nb-M4-C4 bpα icELISA with HPLC
3 结论
本研究表明七聚体Nb-M4-C4 bpα既保留了抗原特异识别能力,又提高了单体Nb-M4的亲和力。此外,七聚体Nb-M4-C4 bpα还为分析信号扩增提供了更多的酶结合位点,七聚体Nb-M4-C4 bpα的icELISA灵敏度比单体Nb-M4的灵敏度明显增高。本研究建立的基于七聚体Nb-M4-C4 bpα的icELISA法检测乳制品中的AFM1的检测限为0.009 ng/kg,IC50为0.038 ng/mL满足国家标准中对牛乳中AFM1的最大耐受量的要求。因此,基于七聚体Nb-M4-C4 bpα icELISA方法是一种有效的检测工具。
[1] VAZ A,CABRAL SILVA A C,RODRIGUES P,et al.Detection methods for aflatoxin M1 in dairy products[J].Microorganisms,2020,8(2):246.
[2] HUANG G X,MA J,LI J S,et al.Study on the interaction between aflatoxin M1 and DNA and its application in the removal of aflatoxin M1[J].Journal of Molecular Liquids,2022,355:118938.
[3] FAN J,YUAN X Y,LI W X,et al.Rapid and ultrasensitive method for determination of aflatoxin M1 in milk[J].Food and Agricultural Immunology,2020,31(1):849-858.
[4] MINCU M,STEFAN-VAN STADEN R I,VAN STADEN J F.Molecular recognition of aflatoxin M1 in water and milk samples[J].Electroanalysis,2019,31(6):1034-1039.
[5] GIROLAMI F,BARBAROSSA A,BADINO P,et al.Effects of turmeric powder on aflatoxin M1 and aflatoxicol excretion in milk from dairy cows exposed to aflatoxin B1 at the EU maximum tolerable levels[J].Toxins,2022,14(7):430.
[6] SABATELLI S,GAMBI L,BAIGUERA C,et al.Assessment of aflatoxin M1 enrichment factor in cheese produced with naturally contaminated milk[J].Italian Journal of Food Safety,2023,12(2):11123.
[7] MOLLAKHALILI-MEYBODI N,NEMATOLLAHI A.The occurrence of aflatoxin M1 in milk samples of Iran:A systematic review and meta-analysis[J].Environmental Monitoring and Assessment,2023,195(6):786.
[8] BUZ S H,SZAB
S H,SZAB -S
-S RV
RV RI L C,SZAB
RI L C,SZAB K,et al.Aflatoxin M1 detection in raw milk and drinking milk in Hungary by ELISA-Aone-year survey[J].Journal of Food Composition and Analysis,2023,121:105368.
K,et al.Aflatoxin M1 detection in raw milk and drinking milk in Hungary by ELISA-Aone-year survey[J].Journal of Food Composition and Analysis,2023,121:105368.
[9] ARAN G C,BAYRAÇ C.Simultaneous dual-sensing platform based on aptamer-functionalized DNA hydrogels for visual and fluorescence detection of chloramphenicol and aflatoxin M1[J].Bioconjugate Chemistry,2023,34(5):922-933.
[10] MASSAHI T,KIANI A,SHARAFI K,et al.A systematic literature review for aflatoxin M1 of various milk types in Iran:Human health risk assessment,uncertainty,and sensitivity analysis[J].Food Control,2023,150:109733.
[11] AFSAH-HEJRI L,JINAP S,HAJEB P,et al.A review on mycotoxins in food and feed:Malaysia case study[J].Comprehensive Reviews in Food Science and Food Safety,2013,12(6):629-651.
[12] GIACOMETTI F,TOMASELLO F,SAVINI F,et al.Impact of the implementation of tailored management strategies to reduce the occurrence of aflatoxin M1 in milk-supply chain in Italy[J].Food Control,2023,149:109720.
[13] CHHAYA R S,MEMBRÉ J M,NAG R,et al.Farm-to-fork risk assessment of aflatoxin M1 in milk under climate change scenarios-A comparative study of France and Ireland[J].Food Control,2023,149:109713.
[14] ZHOU C Y,PAN S X,LIU P Y,et al.Polystyrene microsphere-mediated optical sensing strategy for ultrasensitive determination of aflatoxin M1 in milk[J].Talanta,2023,258:124357
[15] GU G Y,BOLTEN S,MOWERY J,et al.Susceptibility of foodborne pathogens to sanitizers in produce rinse water and potential induction of viable but non-culturable state[J].Food Control,2020,112:107138.
[16] SHI Y,XU W,NI D W,et al.Identification and application of a novel deoxynivalenol-degrading enzyme from Youhaiella tibetensis[J].Food Chemistry,2024,435:137609.
[17] CORASSIN C H,BOROWSKY A,ALI S,et al.Occurrence of aflatoxin M1 in milk and dairy products traded in São Paulo,Brazil:An update[J].Dairy,2022,3(4):842-848.
[18] PAVLEK Z,BOSNIR J,KUHARIC Z,et al.The influence of binding of selected mycotoxin deactivators and aflatoxin M1 on the content of selected micronutrients in milk[J].Processes,2022,10(11):2431.
[19] LEBEER S,VERHOEVEN T L A,PEREA VÉLEZ M,et al.Impact of environmental and genetic factors on biofilm formation by the probiotic strain Lactobacillus rhamnosus GG[J].Applied and Environmental Microbiology,2007,73(21):6768-6775.
[20] ASHRAF W,REHMAN A,AHMAD M U D,et al.Assessment of aflatoxin M1 exposure and associated determinants in children from Lahore,Pakistan[J].Food Additives &Contaminants:Part A,2023,40(1):121-133.
[21] RAMALINGAM S,ELSAYED A,SINGH A.An Aflatoxin-M1 biochip using graphene quantum dot-gold hybrid nanoparticles[J].Food Chemistry,2023,403:134302.
[22] 刘颖达,刘海媛,苏娜等.免疫分析技术在检测真菌毒素中的研究进展[J].中国食品卫生杂志,2023(11):35.LIU Y D,LIU H Y,SU N,et al.Research progress of immunoassay technology in the detection of mycotoxins[J]Chinese Journal of Food Hygiene,2023(11):35.
[23] 曹冬梅.抗黄曲霉毒素B1纳米抗体的免疫学性能分析及其体外定点改造[D].南昌:南昌大学,2016.CAO D M.Immunological performance analysis and site-directed modification of anti-aflatoxin B1 nanoantibody in vitro[D].Nanchang:Nanchang University,2016.
[24] 苏娜,明亮,何静,等.免疫检测技术在食源性致病菌中的应用研究进展[J].食品与发酵工业,2024,50(12):343-352.SU N,MING L,HE J,et al.Research progress on application of immunodetection technology in food-borne pathogens[J].Food and Fermentation Industries,2024,50(12):343-352.
[25] KHAN F,JAVAID A,KIM Y M.Functional diversity of quorum sensing receptors in pathogenic bacteria:Interspecies,intraspecies and interKingdom level[J].Current Drug Targets,2019,20(6):655-667.