大曲是白酒酿造生产的糖化剂和发酵剂[1],由小麦、大麦和/或豌豆通过天然开放式固态发酵而制成[2]。大曲中含有细菌、霉菌、酵母等种类丰富的微生物[3],其中高温放线菌(Thermoactinomyces)在大曲中普遍存在[4]且相对含量较高,对白酒酿造生产具有重要作用[5]。高温放线菌于1899年被首次报道,属于厚壁菌门(Firmicutes),芽孢杆菌纲(Bacilli),芽孢杆菌目(Bacillales),高温放线菌科[6](Thermoactinomycetaceae),目前包括5个有效种。该属菌株最适生长温度为45~55 ℃,最适生长pH值为7.0左右,好氧,常见热泉、堆肥、高温大曲等高温环境[7]。根据文献报道[8-9],目前在白酒发酵过程的大曲、酒醅等样品中共检测到3种高温放线菌,包括普通高温放线菌(Thermoactinomyces vulgaris)、中间型高温放线菌(T.intermedius)和大曲高温放线菌(T.daqus)。高温放线菌属菌株不仅能代谢产生苯丙氨酸脱氢酶、α-淀粉酶、多酚氧化酶等多种耐热酶[10],还能产生吡嗪类、酚类、酸类、醇类等风味化合物[11]。因此,在大曲发酵过程中对高温放线菌进行定量分析将有助于生产过程监测和产品质量评价。
传统的纯培养方法检测微生物数量存在耗时长,无法适用于难培养物种等问题。随着分子生物学技术的发展,高通量测序技术被广泛用于酿造微生物多样性的分析[12],但是结果不能代表微生物的真实含量。微生物的绝对定量法包括流式细胞计数法、外标绝对定量法和实时荧光定量PCR(real-time fluorescence quantitative PCR,RT-qPCR)技术等[13]。RT-qPCR通过利用荧光染料或探针与DNA链结合后发出荧光的特性[14],实时检测样品扩增信号,具有特异性强、灵敏度高、定量准确、耗时短[15]等优点,能够快速反应微生物的真实含量。目前,RT-qPCR技术在病毒检测、物种鉴定等方面得到广泛应用,例如CORMAN等[16]利用RT-qPCR技术建立2019-nCoV病毒的快速检测方法;KIM等[17]利用RT-qPCR技术实现了干酪乳杆菌类群的快速鉴定。
本实验下载了9株T.vulgaris、3株T.intermedius、2株T.daqus的全基因组序列,通过序列比对分别筛选出普通高温放线菌、中间型高温放线菌和大曲高温放线菌全基因组中的特异性单拷贝核心基因序列,并其为参考序列设计引物,用标准菌株和大曲样品验证引物的特异性、有效性和准确性并建立了RT-qPCR方法,旨在为大曲发酵过程中3种高温放线菌的快速检测和准确定量提供方法。
1 材料与方法
1.1 材料
1.1.1 菌株
本研究所用菌株均由本实验室从大曲中筛选得到,并按照菌种鉴定程序[18]进行了种水平鉴定,保存于-80℃冰箱。普通高温放线菌(T.vulgaris JN20220704)、中间型高温放线菌(T.intermedius JN20220705)、腾冲莱西氏菌(Laceyella tengchongensis JN20220707)使用TSB培养基55℃培养两天;大曲高温放线菌(T.daqus JN20220706)使用R2A培养基55℃培养两天;象牙色克罗彭斯特氏菌(Kroppenstedtia eburnean)JN20220708、血液克罗彭斯特氏菌(K.pulmonis JN20220709)使用BHI培养基45℃培养两天;枯草芽孢杆菌(Bacillus subtilis JN20220710)、地衣芽孢杆菌(B.licheniformis JN20220711)、戊糖片球菌(Pediococcus pentosaceus JN20220712)、白色链霉菌(Streptomyces albus JN20220713)、大曲岩石芽孢杆菌(Scopulibacillus daqui JN20220714)、海洋芽孢杆菌(Oceanobacillus sojae) JN20220715、腐生葡萄球菌(Staphylococcus saprophyticus JN20220716)使用LB培养基37℃培养2 d。
1.1.2 大曲样品
高温大曲发酵过程样品采集于四川某白酒生产企业,发酵时间为第0、2、5、7、9、12、14、27、42天。
1.2 试剂与仪器
R2A培养基、LB培养基、TSB培养基、BHI培养基,青岛海博生物有限公司;土壤微生物DNA提取试剂盒、细菌基因组DNA提取试剂盒,诺唯赞生物科技公司;Taq qPCR Master Mix、4SGelred核酸染料,生工生物工程(上海)股份有限公司;T100 PCR仪、Image 900凝胶成像分析系统、CFX Opus 96荧光定量PCR仪,美国Bio-Rad公司。
1.3 实验方法
1.3.1 基因组DNA的提取
采用试剂盒提取1.1.1节中所有细菌的基因组DNA,主要用于特异性实验;采用土壤微生物DNA基因组提取试剂盒提取高温大曲基因组DNA,主要用于有效性实验。具体步骤参见试剂盒说明书。
1.3.2 大曲样品扩增子建库及测序
由华大基因科技服务有限公司完成扩增子测序。使用通用引物338F(5′-ACTCCTACGGGAGGCAGCA-3′)/806R(5′-GGACTACHVGGGTWTCTAAT-3′)扩增细菌16S rRNA基因的V3~V4区。使用QIIME 1.9版本进行测序数据分析,包括去除嵌合体和低质量序列,以及使用dada2降噪生成ASV序列处理单元,使用Silval32数据库进行分类器训练,对生成的ASV序列进行物种类别注释。
1.3.3 特异性引物的设计
通过NCBI(www.ncbi.nih.gov)下载9株T.vulgaris的全基因组序列,采用Prokka(v1.12)软件对菌株基因组进行基因预测,之后采用Roary(v3.7.1)软件以编码氨基酸相似性99%分别识别全部T.vulgaris核心基因[19]。采用BLAST(v.2.2.24+)将核心基因代表序列与核酸序列数据库(nucleotide sequence database,nt)进行比对,删除非T.vulgaris相似度>99%的共有核心基因[19],并采用TB tools比对筛选单拷贝共有核心基因,以特异性单拷贝核心基因序列为目的片段,使用Primer 5.0软件设计T.vulgaris特异性定量引物,将设计好的引物序列重新在NCBI中进行比对,确定引物对目的片段具有特异性。同样地,通过NCBI分别下载3株T.intermedius和2株T.daqus的全基因组序列,按此方法设计特异性引物。
1.3.4 PCR、qPCR反应体系
PCR反应体系:上下游引物各1 μL,Tap酶20 μL,模板DNA 1 μL,ddH2O 26 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,63 ℃ 45 s,72 ℃ 60 s,30个循环。qPCR反应体系:上游引物和下游引物各0.5 μL、SYBR Green Master Mix 12.5 μL,模板DNA 1 μL,ddH2O 10.5 μL。qPCR反应条件:95 ℃预变性2.5 min;94 ℃变性15 s,63 ℃退火45 s,40个循环。
1.3.5 引物的特异性和有效性评价
分别以T.vulgaris JN20220704、T.intermedius JN20220705、T.daqus JN20220706基因组为阳性对照,以L.tengchongensis JN20220707、K.eburnean JN20220708、K.pulmonis JN20220709、B.subtilis JN20220710、B.licheniformis JN20220711、P.pentosaceus JN20220712、S.albus JN20220713、S.daqui JN20220714、O.sojae JN20220715、S.saprophyticus JN20220716等大曲中其他细菌为阴性对照,不加样品为空白对照进行PCR和qPCR。通过琼脂糖凝胶电泳和扩增曲线判断引物的特异性。采用设计好的引物,对发酵过程中(第0、2、5、7、9、12、14、27、42天)高温大曲基因组DNA进行PCR扩增并通过琼脂糖凝胶电泳判断引物的有效性。将PCR扩增产物送至生工生物工程(上海)股份有限公司测序,结果与EzBioCloud(https://www.ezbiocloud.net/)数据库比对,根据测序结果判断引物扩增产物的特异性和有效性。
1.3.6 熔解曲线和标准曲线的绘制
使用已知浓度的含有目的片段的单拷贝克隆质粒标准品以10倍稀释,设置10个梯度,以不同浓度标准品作为模板进行RT-qPCR[13]。以每个反应管内荧光值达到阈值时的循环数(Cq值)作为横轴,标准品浓度的对数作为纵轴,绘制定量标准曲线,扩增完成后绘制RT-qPCR熔解曲线。
1.3.7 加标回收率的测定
在空白样品(ddH2O)分别加入不同数量的T.vulgaris(6.55×102、6.55×104、6.55×106 copies/g)、T.intermedius(6.60×102、6.60×104、6.60×106 copies/g)和T.daqus(6.53×102、6.53×104、6.53×106 copies/g),分别计算这3种高温放线菌的空白加标回收率。在高温大曲中加入不同含量的T.vulgaris(6.55×103、6.55×105、6.55×107 copies/g)、T.intermedius(6.60×103、6.60×105、6.60×107 copies/g)和T.daqus(6.53×103、6.53×105、6.53×107 copies/g),每份样品进行3次平行测定,分别计算3种高温放线菌的样品加标回收率。
1.3.8 高温大曲发酵过程中高温放线菌含量变化
以高温大曲发酵过程样品总DNA为模板,进行RT-qPCR反应,对不同发酵时间高温大曲样品中T.vulgaris、T.intermedius、T.daqus的含量分别进行测定,阴性对照为无菌水,根据标准曲线分别计算T.vulgaris、T.intermedius、T.daqus在高温大曲发酵过程中的含量。
1.4 数据分析
使用Microsoft Excel 2010对数据进行处理;使用Graphpad Prism 8软件进行分析绘图。
2 结果与分析
2.1 特异性引物的设计
为构建RT-qPCR快速定性及定量检测方法,通过下载9株T.vulgaris、3株T.intermedius和2株T.daqus的全基因组序列,经过系统的比对分析,筛选得到T.vulgaris特异性核心基因(recA)、T.intermedius特异性核心基因(mdH)、T.daqus特异性核心基因(gyrA),基因序列如图1所示。采用TB tools对比了T.vulgaris中的recA基因,发现在基因组中只出现了1次,是一个单拷贝基因。同样地,T.intermedius中的mdH基因、T.daqus中的gyrA基因均为单拷贝基因。如表1所示,针对单拷贝基因recA、mdH、gyrA分别设计了一对可扩增278、233、291 bp片段的引物,上下游引物均不存在成环、发夹等结构。将特异性引物TV、TI、TD分别在NCBI上进行Blast比对,结果表明引物TV、TI、TD均对扩增目标片段具有特异性,可用于后续qPCR检测方法的构建。
表1 特异性引物TV、TI、TD信息
Table 1 Specific primers TV,TI,TD information
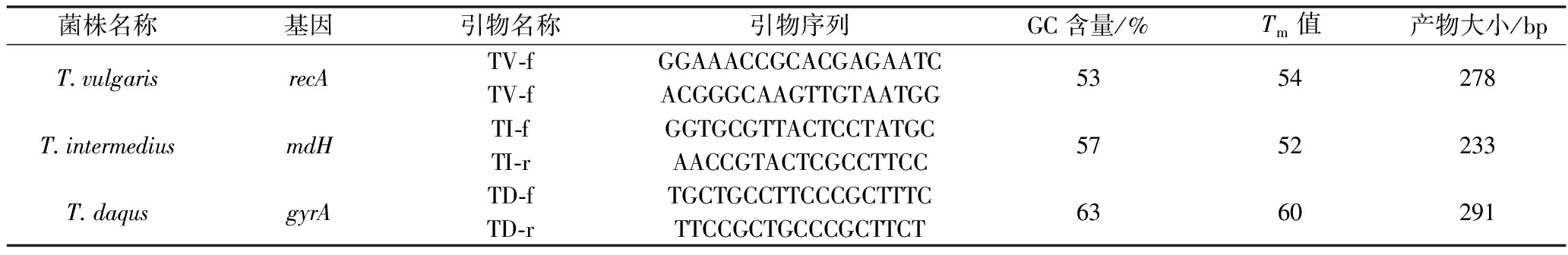
菌株名称基因引物名称引物序列GC含量/%Tm值产物大小/bpT.vulgarisrecATV-fTV-fGGAAACCGCACGAGAATCACGGGCAAGTTGTAATGG5354278T.intermediusmdHTI-fTI-rGGTGCGTTACTCCTATGCAACCGTACTCGCCTTCC5752233T.daqusgyrATD-fTD-rTGCTGCCTTCCCGCTTTCTTCCGCTGCCCGCTTCT6360291
a-T.vulgaris的recA基因序列;b-T.intermedius的mdH基因序列;c-T.daqus的gyrA基因片段
图1 扩增的目标基因片段
Fig.1 Amplification of target fragment gene
2.2 引物扩增的特异性评价
利用引物TV、TI和TD对13种大曲细菌的基因组进行PCR扩增,发现仅T.vulgaris JN20220705(图2-a)、T.intermedius JN20220706(图2-b)和T.daqus JN20220706(图2-c)在琼脂糖凝胶电泳上出现单一条带,表明其特异性较好。经测序,引物TV、TI和TD的PCR产物序列长度分别为278、233、291 bp,与GenBank中登陆序列(NZ_CP039710.1、NZ_JACEIR010000008.1、NZ_JPST01000014.1)同源性为100%,与预期相符。利用引物TV、TI和TD对13种大曲细菌的基因组进行qPCR扩增,发现仅T.vulgaris JN20220705、T.intermedius JN20220706、T.daqus JN20220706出现了扩增曲线,其他菌株均未出现扩增曲线,表明引物TV、TI和TD在qPCR方法上也具有良好的种间特异性,可用于区分T.vulgaris JN20220705、T.intermedius JN20220706、T.daqus JN20220706与其他微生物。
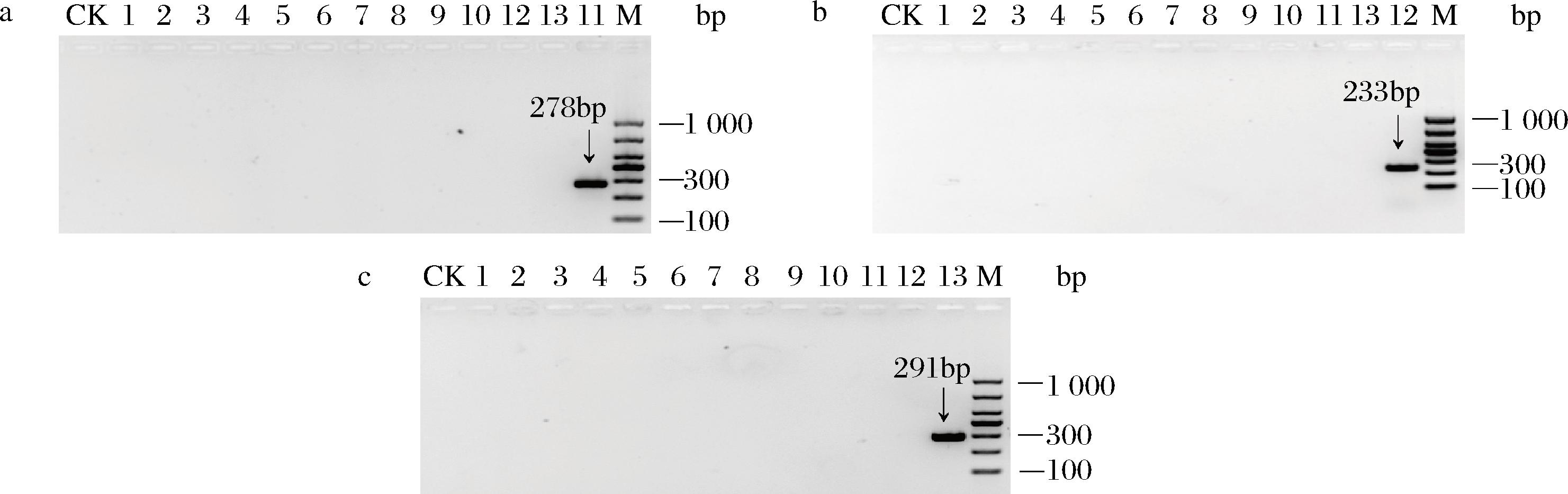
CK-ddH2O;1-L.tengchongensis JN20220707;2-K.eburnean JN20220708;3-K.pulmonis JN20220709;4-B.subtilis JN20220710;5-B.licheniformis JN20220711;6-P.pentosaceus JN20220712:7-S.albus JN20220713;8-S.daqui JN20220714;9-O.sojae JN20220715;10-S.saprophyticus JN20220716;11-T.vulgaris JN20220704;12-T.intermedius JN20220705;13-T.daqus JN20220706;M-1 000 bp marker
a-引物TV特异性评价;b-引物TI特异性评价;c-引物TD特异性评价
图2 引物的特异性评价
Fig.2 Primer specificity evaluation
2.3 引物扩增的有效性评价
通过琼脂糖凝胶电泳分析,发现引物TV、TI和TD均能够对不同发酵时间点(第0、2、5、7、9、12、14、27、42天)采集的高温大曲样品扩增获得单一条带(图3),说明其能够用于高温大曲发酵过程监测。
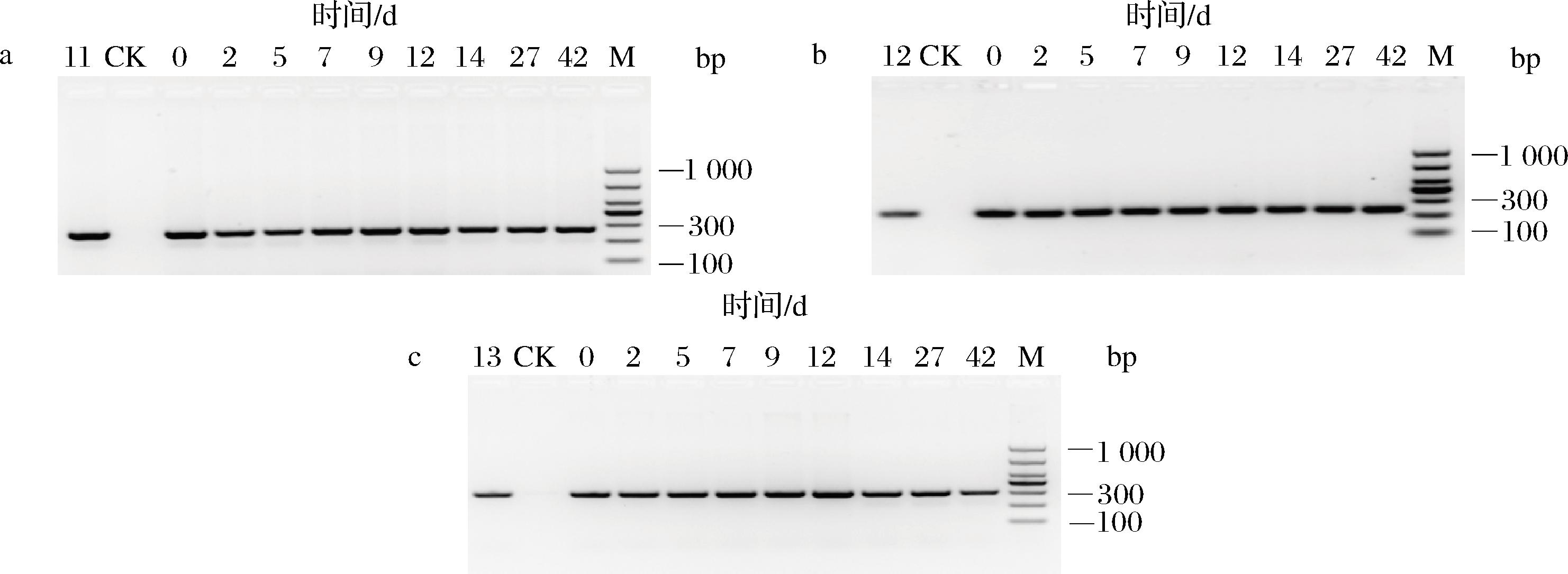
CK-ddH2O;11-T.vulgaris JN20220704;12-T.intermedius JN20220705;13-T.daqus JN20220706;M-1 000 bp marker
a-引物TV有效性评价;b-引物TI有效性评价;c-引物TD有效性评价
图3 引物的有效性评价
Fig.3 Primer Effectiveness evaluation
2.4 熔解曲线和标准曲线的绘制
基因拷贝数在2.82~8.82 lg copies/μL,T.vulgaris、T.intermedius、T.daqus的RT-qPCR扩增曲线基线平整,均呈现典型的倒S曲线。熔解曲线呈现单一熔解峰,说明无引物二聚体及非特异性产物;曲线平稳,说明各浓度质粒的熔解温度均一,扩增产物特异性好。3个菌株的标准曲线线性均较好(R2=0.997 7~0.999 7),扩增效率分别为99.30%、97.79%和97.06%(图4)。

a-T.vulgaris RT-qPCR熔解曲线;b-T.intermedius RT-qPCR熔解曲线;c-T.daqus RT-qPCR熔解曲线;d-T.vulgaris RT-qPCR标准曲线;e-T.intermedius RT-qPCR标准曲线;f-T.daqus RT-qPCR标准曲线
图4 RT-qPCR熔解曲线和标准曲线
Fig.4 RT-qPCR melting curves and standard curves
2.5 qPCR定量方法的准确性评价
以空白样品和已知浓度含有目的片段的单拷贝克隆质粒标准品为样本进行RT-qPCR反应,测定3种高温放线菌的空白加标回收率和样品加标回收率来评价去PCR方法的准确性。结果表明,T.vulgaris、T.intermedius、T.daqus的加标回收率分别在96%~101%、95%~105%、96%~101%(表2),均在正常范围(95%~105%)。这表明基于特异性引物构建的q-PCR 定量方法准确性高,重复性较好,适用于定量含有不同浓度T.vulgaris、T.intermedius、T.daqus的样本。
表2 高温放线菌的加标回收率
Table 2 Recovery of Thermoactinomyces

菌株名称初始浓度/(copies/g)添加浓度/(copies/g)最终浓度/(copies/g)回收率/%06.55×1026.37×102±6.68 97.25±1.0206.55×1046.46×104±7.13×10298.66±1.09T.vulgaris06.55×1066.39×106±8.66×10497.55±1.326.55×1056.55×1036.50×105±6.19×10398.19±0.946.55×1056.55×1051.28×106±1.43×10497.80±1.096.55×1056.55×1076.60×107±7.04×10599.78±1.0606.60×1026.39×102±8.6596.87±1.3106.60×1046.44×104±5.79×10297.63±0.88T.intermedius06.60×1066.49×106±7.76×10498.35±1.186.60×1056.60×1036.55×105±1.01×10498.22±1.516.60×1056.60×1051.35×106±1.90×104102.20±1.446.60×1056.60×1076.48×107±5.05×10597.14±0.7606.53×1026.32×102±3.8696.83±0.5906.53×1046.46×104±4.60×10298.87±0.70T.daqus06.53×1066.48×106±7.76×10499.29±1.196.53×1056.53×1036.52×105±6.14×10398.89±0.936.53×1056.53×1051.27×106±2.04×10497.52±1.666.53×1056.53×1076.50×107±5.95×10598.53±0.90
2.6 高温大曲发酵过程中3种高温放线菌含量的动态变化
采用扩增子测序技术分析高温大曲发酵过程中3种高温放线菌相对丰度变化情况,结果表明,高温大曲发酵过程T.vulgaris的相对丰度呈现先下降后上升再下降的趋势,发酵结束下降到0.02%(图5-a);T.intermedius 仅在发酵前12 d被检出,在发酵14 d到24 d样品中并未检测到,其相对丰度呈现先上升再下降的趋势,发酵第7天时相对丰度最高(3.31%)(图5-b);T.daqus的相对丰度呈现波动的趋势,发酵第9天相对丰度最高(2.65%),发酵结束时相对丰度为0.41%(图5-c)。
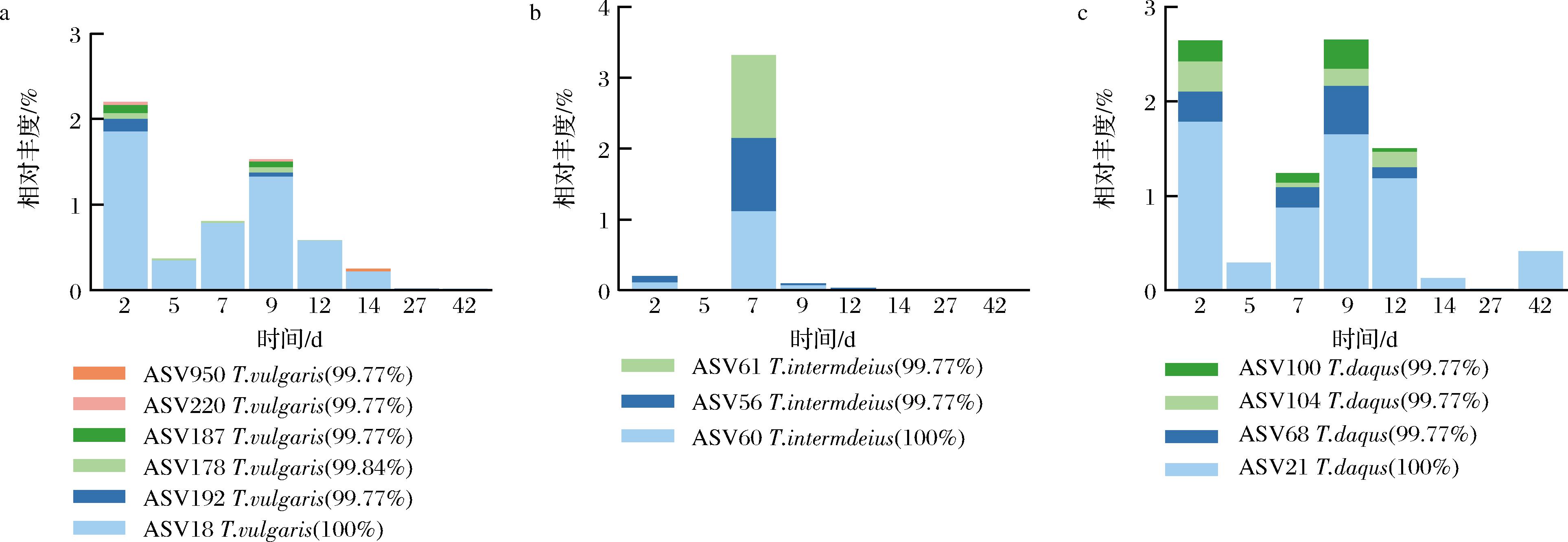
a-T. vulgaris相对丰度变化情况;b-T. intermedius相对丰度变化情况;c-T. daqus相对丰度变化情况
图5 高温大曲发酵过程中T. vulgaris、T. intermedius、T. daqus相对丰度变化情况
Fig.5 Variation of relative abundance of T. vulgaris,T. intermedius and T. daqus during high temperature Daqu fermentation
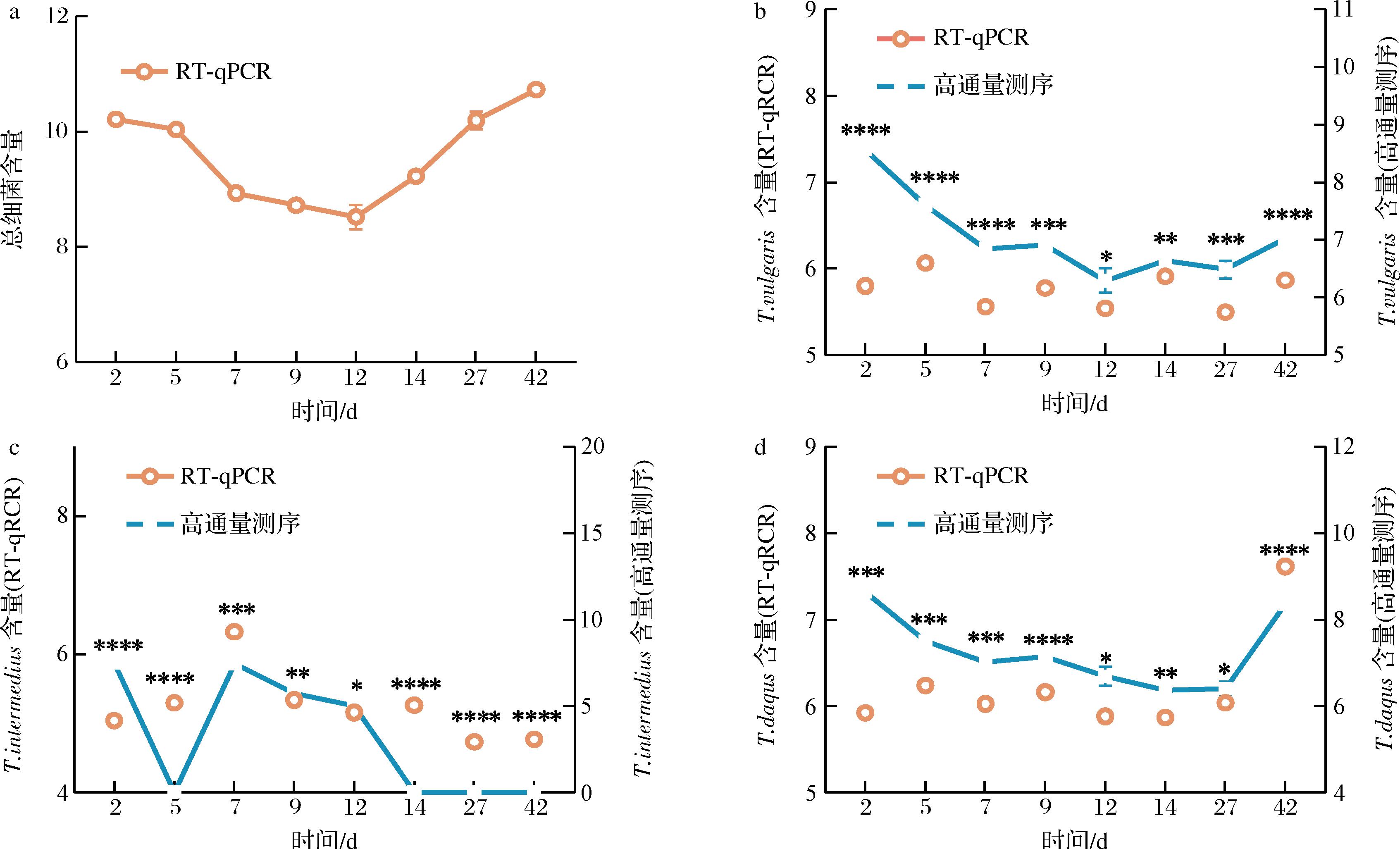
a-总细菌含量变化情况;b-T.vulgaris含量变化情况;c-T.intermedius含量变化情况;d-T.daqus含量变化情况
图6 采用RT-qPCR法定量分析高温大曲发酵过程中总细菌、T.vulgaris、T.intermedius、T.daqus含量
Fig.6 Content of total bacteria,T.vulgaris,T.intermedius and T.daqus during high temperature Daqu fermentation were quantitatively analyzed by RT-qPCR
注:*P<0.05;**P<0.01;***P<0.001;****P<0.000 1。
RT-qPCR结果表明,高温大曲发酵过程中细菌含量呈现先上升后下降再上升的趋势,发酵结束时,细菌总量最高,为(10.32±0.10) lg copies/g(图6-a);T.vulgaris含量在第2天~第5天上升到最高,为(6.06±0.0) lg copies/g,在第5天~第27天呈现先下降后上升再下降的趋势,发酵结束回升到(5.86±0.02) lg copies/g(图6-b);T.intermedius含量在第2天~第7天上升到最高,为(6.33±0.13) lg copies/g,在第7天~第42天不断下降,发酵结束时下降到(4.77±0.14) lg copies/g(图6-c);T.daqus含量与T.vulgaris在第0天~第12天表现同步趋势,在第12天~第14天下降,随后在一直上升,发酵结束时含量最高,达(7.62±0.02) lg copies/g(图6-d)。
将细菌群落的含量乘高温放线菌相对丰度所估算的高温放线菌含量,与RT-qPCR得到的高温放线菌含量进行对比,发现在不同时间的样品中,两者T.vulgaris、T.intermedius、T.daqus的数值均有显著性差异,这与文献的结果一致[20]。3种高温放线菌在大曲发酵过程中相对丰度均低于10%,使用微生物总含量乘各菌种相对丰度得到的菌种含量,适用于高丰度物种(相对丰度>10%),但当物种丰度<10%时,这种计算方法并不准确。这说明本研究建立的RT-qPCR方法适合用于酿造全过程中T.vulgaris、T.intermedius、T.daqus的定量。
3 结论
高度保守、特异性强的基因序列作为模板DNA序列设计特异性引物是对菌种进行检测和定量的关键。目前针对细菌进行种特异性引物的设计大多基于16S rRNA基因序列,T.vulgaris、T.intermedius、T.daqus 及其近缘种16S rRNA基因序列十分接近,应用常规技术鉴定繁琐费时,无法满足菌株快速鉴定和定量的要求。此外,16S rRNA虽然有高度的保守性和特异性,但它为多拷贝基因,在不同物种中拷贝数有所差异,甚至同一菌种中的不同拷贝序列也可能存在差异。定量过程中常以单位样品中的拷贝数计算定量结果,且拷贝数由系统直接读出,默认1个拷贝基因即为1株菌,在定量多拷贝基因时可能会扩大目标菌的数量[19],从而导致定量准确度降低。因此,使用单拷贝核心基因为参考序列设计特异性引物能够使结果更接近真实值。本研究针对T.vulgaris、T.intermedius、T.daqus所设计的引物分别基于recA、mdH、gyrA基因。这3个基因在高温放线菌菌株全基因组中均只出现1次,为单拷贝核心基因,具有高保守性,与蛋白质的编码有关。其中recA基因能够编码调节蛋白,与DNA重组修复有关;mdH基因能够编码苹果酸脱氢酶,gyrA基因能够编码DNA旋转酶。目前,已有许多基于recA、mdH、gyrA基因建立微生物RT-qPCR方法的报道,例如杨小娜等[21]利用recA、glpQ基因合成引物探针,建立了检测蜱传疏螺旋体的双重荧光定量PCR方法;王青龙等[22]利用recA基因特异性引物和探针,建立了RT-PCR法鉴定发酵乳中嗜热链球菌;计若云[23]利用mdH基因建立了鼠伤寒沙门菌及其单相变异体多重PCR检测方法;杨湘黔等[24]以gyrA基因为参考序列,建立了解淀粉芽孢杆菌实时荧光定量PCR测定方法并应用于豆粕发酵。本研究的结果也充分证明,应用recA、mdH、gyrA基因作为模板分别用于T.vulgaris、T.intermedius、T.daqus 的 RT-qPCR检测特异性和准确性都很高。
由于取样量的原因,本研究通过高通量测序技术和RT-qPCR技术在大曲中检测到了3种高温放线菌,是否在大曲中可能还存在其他的高温放线菌物种,需要进一步研究。此外,关于高温放线菌的定性定量检测方法的研究报道较少,因此本研究建立的能够快速检测大曲中3种高温放线菌的方法对研究其在白酒酿造过程中的演替规律、具体功能与价值具有重要意义。
本研究分别以普通高温放线菌recA基因、中间型高温放线菌mdH基因和大曲高温放线菌gyrA基因序列为参考序列,分别设计了一对特异性引物,针对3种高温放线菌分别构建了RT-qPCR方法,3对引物特异性强,有效性好,准确性高,能够方便快速定量检测样品中的T.vulgaris、T.intermedius、T.daqus,可有效用于大曲发酵过程样品中3种高温放线菌的快速鉴定和准确定量,有助于监测与优化酿造生产过程,为进一步解释高温放线菌在白酒酿造过程中的功能提供了思路。
[1] 卢延想.高温大曲中产香微生物的筛选及应用研究[D].天津:天津科技大学,2021.LU Y X.Screening and application of aroma-producing microorganisms in high temperature Daqu[D].Tianjin:Tianjin University of Science and Technology,2021.
[2] SHI W,CHAI L J,FANG G Y,et al.Spatial heterogeneity of the microbiome and metabolome profiles of high-temperature Daqu in the same workshop[J].Food Research International,2022,156:111298.
[3] HOU Q C,WANG Y R,CAI W C,et al.Metagenomic and physicochemical analyses reveal microbial community and functional differences between three types of low-temperature Daqu[J].Food Research International,2022,156:111167.
[4] 束莹.古井贡酒大曲中微生物群落结构的鉴定及分析[D].哈尔滨:哈尔滨工业大学,2013.SHU Y.Identification and analysis of microbial community structure in Gujing disticcery Daqu[D].Harbin:Harbin Institute of Technology,2013.
[5] YANG Y,WANG S T,LU Z M,et al.Metagenomics unveils microbial roles involved in metabolic network of flavor development in medium-temperature Daqu starter[J].Food Research International,2021,140:110037.
[6] 罗小叶,王晓丹,邱树毅.酱香酒生产过程中的微生物研究[J].酿酒科技,2018(10):108-113.LUO X Y,WANG X D,QIU S Y.Research on microbes in the production of Jiangxiang Baijiu[J].Liquor-Making Science &Technology,2018(10):108-113.
[7] 冯慧军,翟磊,程坤,等.高温放线菌属研究进展[J].食品与发酵工业,2017,43(11):257-261.FENG H J,ZHAI L,CHENG K,et al.The research advance of genus Thermoactinomyces[J].Food and Fermentation Industries,2017,43(11):257-261.
[8] LIU S P,ZHOU Y,MA D N,et al.Environment microorganism and mature Daqu powder shaped microbial community formation in mechanically strong-flavor Daqu[J].Food Bioscience,2023,52:102467.
[9] XIAO C,LU Z M,ZHANG X J,et al.Bio-heat is a key environmental driver shaping the microbial community of medium-temperature Daqu[J].Applied and Environmental Microbiology,2017,83(23):e01550-e01517.
[10] 王芙蓉,李靖,赵益梅,等.酱香大曲中高温放线菌的筛选及其产蛋白酶条件优化[J].中国酿造,2022,41(8):132-136.WANG F R,LI J,ZHAO Y M,et al.Screening of high-temperature actinomycetes in sauce-flavor Daqu and optimization of protease-producing conditions[J].China Brewing,2022,41(8):132-136.
[11] 黄武,唐绍培,班世栋,等.高温放线菌FBKL4.010对酱香大曲酶活、理化特性和挥发性成分贡献的研究[J].食品与发酵科技,2022,58(2):15-21;46.HUANG W,TANG S P,BAN S D,et al.Study on the contribution of Thermoactinomycete FBKL4.010 to the enzyme activity,physicochemical properties and volatile components of Moutai-flavor Daqu[J].Food and Fermentation Science &Technology,2022,58(2):15-21;46.
[12] JIANG X Y,PENG Z,ZHU Q,et al.Exploration of seasonal fermentation differences and the possibility of flavor substances as regulatory factors in Daqu[J].Food Research International,2023,168:112686.
[13] 孙佳,陆震鸣,张晓娟,等.实时荧光定量PCR检测金山醋酸乳杆菌的方法与应用[J].微生物学报,2021,61(10):3211-3221.SUN J,LU Z M,ZHANG X J,et al.Establishment and application of real-time fluorescence quantitative PCR for detection of Acetilactobacillus jinshanensis[J].Acta Microbiologica Sinica,2021,61(10):3211-3221.
[14] ZHANG Y,YANG J P,DAI R H,et al.Stability evaluation of candidate reference genes for RT-qPCR normalization in Lasioderma serricorne(F).[J].Journal of Stored Products Research,2021,94:101877.
[15] KOKANE A D,LAWRENCE K,KOKANE S B,et al.Development of a SYBR Green-based RT-qPCR assay for the detection of Indian citrus ringspot virus[J].3 Biotech,2021,11(7):359.
[16] CORMAN V M,LANDT O,KAISER M,et al.Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR[J].Eurosurveillance,2020,25(3):2000045.
[17] KIM E,YANG S M,CHO E J,et al.Novel real-time PCR assay for Lactobacillus casei group species using comparative genomics[J].Food Microbiology,2020,90:103485.
[18] 张锦华,张潇月,白宝清,等.清香型白酒大曲中高产四甲基吡嗪菌株的筛选、鉴定及发酵培养基优化[J].中国酿造,2023,42(3):47-52.ZHANG J H,ZHANG X Y,BAI B Q,et al.Screening identification and fermentation medium optimization of high yield tetramethylpyrazine strains from light-flavor Baijiu Daqu[J].China Brewing,2023,42(3):47-52.
[19] 高旭,白晓晔,郑慧娟,等.双歧杆菌属特异性定量引物的设计及验证[J].微生物学报,2020,60(3):545-555.GAO X,BAI X Y,ZHENG H J,et al.Design and evaluation of Bifidobacterium genus-specific primer for quantification[J].Acta Microbiologica Sinica,2020,60(3):545-555.
[20] TETTAMANTI F A,SRINIVASAN S,LOPEZ A,et al.Complementing 16S rRNA gene amplicon sequencing with total bacterial load to infer absolute species concentrations in the vaginal microbiome[J].Msystems,2020,5(2):e00777-e00719.
[21] 杨小娜,张琳,侯学霞,等.建立双重荧光定量PCR方法检测蜱传疏螺旋体[J].中国人兽共患病学报,2021,37(9):788-794.YANG X N,ZHANG L,HOU X X,et al.Establishing a duplex real-time PCR method for the detection of tick-borne Borrelia[J].Chinese Journal of Zoonoses,2021,37(9):788-794.
[22] 王青龙,貌达,周燕霞,等.实时荧光定量PCR法鉴定发酵乳中嗜热链球菌[J].中国酿造,2022,41(7):149-154.WANG Q L,MAO D,ZHOU Y X,et al.Identification of Streptococcus thermophilus in fermented milk by real-time fluorescent quantitative PCR[J].China Brewing,2022,41(7):149-154.
[23] 计若云.鼠伤寒沙门菌及其单相变异体多重PCR检测方法的建立及流行病学调查[D].扬州:扬州大学,2022.JI R Y.Establishment and epidemiological investigation of multiplex PCR for detection method for detection of Salmonella typhimurium and its monophasic variants[D].Yangzhou:Yangzhou University,2022.
[24] 杨湘黔,崔京春,曾诗娴,等.解淀粉芽孢杆菌实时荧光定量PCR测定方法的建立及其在豆粕发酵中的应用[J].动物营养学报,2021,33(1):494-505.YANG X Q,CUI J C,ZENG S X,et al.Development of real-time fluorescent quantitative PCR detection method for Bacillus amyloliquefaciens and its application in fermented soybean meal[J].Chinese Journal of Animal Nutrition,2021,33(1):494-505.