齐墩果酸是广泛存在于植物中的五环三萜类化合物[1]。齐墩果酸因其显著的保肝作用,已被大力应用于肝脏疾病的治疗[2]。由于其还具有抗氧化、抗菌、抗炎、抗血糖、抗肿瘤等作用,也被应用于其他医药领域[3]。目前,齐墩果酸的获取主要通过植物提取法,但植物中齐墩果酸含量较低,无法满足基本需求。与植物提取法相比,微生物合成法在天然化合物的生产方面有诸多优势,如不依赖于特定植物、生长周期短等[4]。
酿酒酵母是一种安全的模式微生物,已被广泛应用于包括萜类物质在内的多种天然产物的生物合成。齐墩果酸的生物合成途径已被解析。酿酒酵母通过内源的甲羟戊酸(mevalonate acid,MVA)途径生成异戊二烯基焦磷酸(isoprene pyrophosphate,IPP)和二甲基丙烯焦磷酸(dimethyl propenyl pyrophosphate,DMAPP),随后两分子IPP和一分子DMAPP缩合生成法尼基焦磷酸(farnesyl pyrophosphate,FPP),FPP生成角鲨烯后经鲨烯环氧化酶转化为2,3-环氧化角鲨烯。2,3-环氧化角鲨烯通过β-香树脂醇合酶环化生成齐墩果酸前体物质β-香树脂醇后,进一步引入细胞色素P450酶(P450 enzymes,P450s)和为P450s传递电子的细胞色素P450还原酶(cytochrome P450 reductase,CPR)催化β-香树脂醇C28位发生三步连续氧化反应生成齐墩果酸[5](图1)。
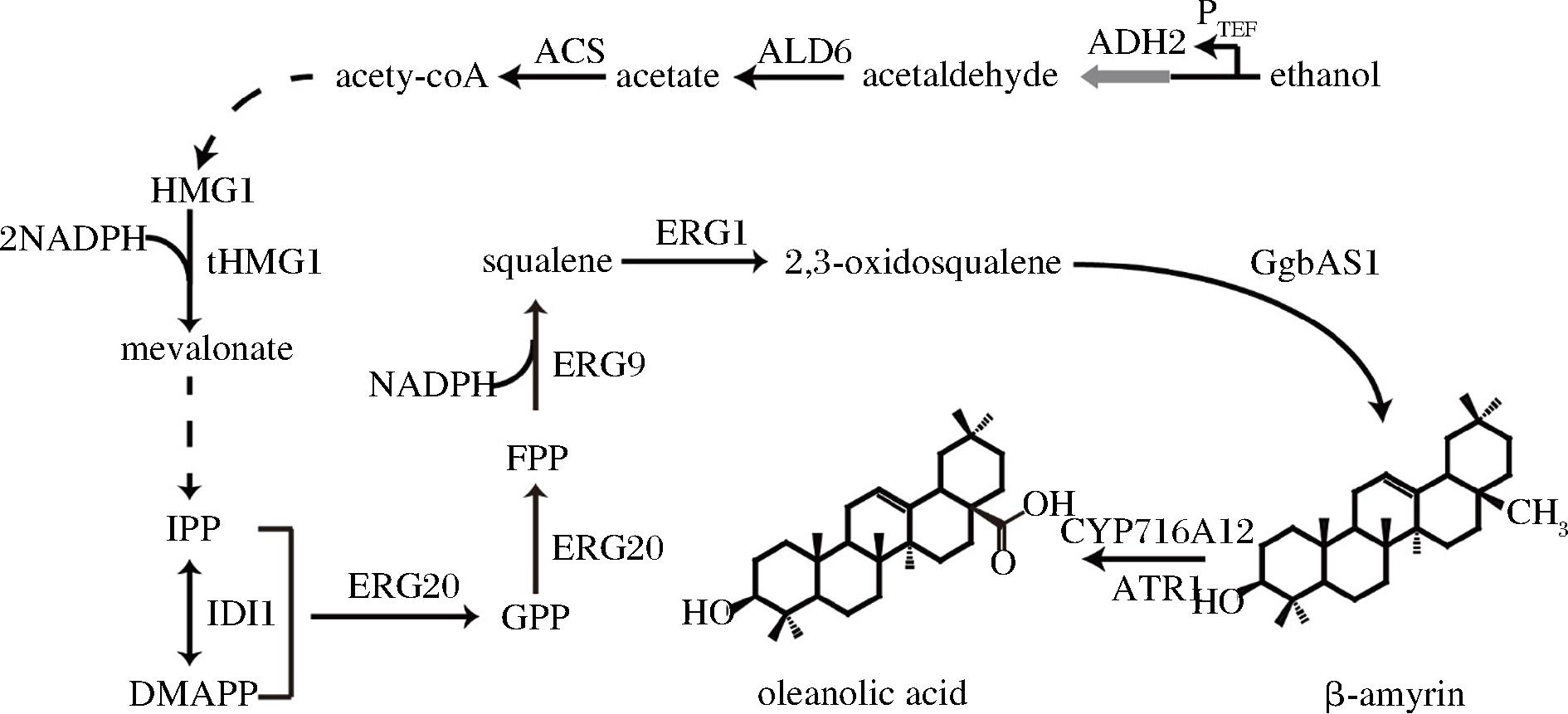
图1 酿酒酵母中齐墩果酸生物合成途径
Fig.1 Biosynthesis pathway of oleanolic acid in Saccharomyces cerevisiae
近年来,关于酿酒酵母从头合成齐墩果酸已取得一些进展。通过强化前体途径关键基因增强代谢通量,如过量表达tHMG1、ERG20、ERG9,以及引入来源于白色念珠菌中活性更高的ERG1等,还通过抑制羊毛甾醇竞争途径的关键基因ERG7等常用策略来提高齐墩果酸的产量[6]。LI等[7]以解脂耶氏酵母为宿主,将适配性最佳的P450s和CPR进行融合表达,将5 L-发酵罐中齐墩果酸产量提高至540.7 mg/L。李春课题组[8]通过筛选适配较高的P450s和CPR,再结合前体途径强化及偶联细胞半乳糖调控网络,将5 L-发酵罐中齐墩果酸产量提高到606.9 mg/L。
虽然齐墩果酸的从头合成已有一定的研究进展,但目前的产量远未达到工业化生产要求,其关键问题还是在于P450s的催化效率低以及异源途径与酿酒酵母底盘细胞之间代谢通量的失衡[9]。酿酒酵母从头合成齐墩果酸需引入植物来源的P450s,而P450s催化活性的激活需要由CPR转移来自NADPH提供的电子[10]。通常认为P450s和CPR偶联反应的最佳比例是1∶1,但是体内P450s的总含量与CPR的实际比例似乎在5∶1~20∶1[11-12]。PETERSON等[13]提出,中心CPR被多个P450分子包围,并且这些P450在团簇内接收电子的速率较低。高表达的CPR可能通过解偶联电子传递产生活性氧,从而对P450s和细胞活性造成抑制[14]。基于以上双酶系统表达的一些原理,有必要对P450s和CPR的表达量和表达比率对P450s的催化活性的影响进行探究。
本研究在酿酒酵母中成功构建齐墩果酸的从头合成途径,通过探究途径中的CYP716A12和ATR1的表达量以及表达比例,发现高的CYP716A12∶ATR1表达比例有利于齐墩果酸的合成。最后,通过增强乙酰辅酶A供应和辅因子NADPH再生,齐墩果酸摇瓶水平和5 L-发酵罐分批补料发酵产量分别达到304.0 mg/L和680.8 mg/L,为目前报道最高产量。本研究为利用酿酒酵母合成齐墩果酸和下游产物相关研究奠定了一定基础。
1 材料与方法
1.1 基因,菌株,质粒及引物
本研究所用菌株JM109用于质粒构建,出发菌株是实验室保存菌株C800,该菌株敲除了GAL80基因,方便后续实验利用GAL系列强启动子。本研究所用基因均由赛索飞公司合成,基因详见表1,所有代谢工程改造菌株详见表2,所有构建的质粒详见表3,所有引物详见表4。
表1 本研究使用的外源基因
Table 1 Heterologous genes used in this study

基因来源登录号GgbAS1GlycyrrhizaglabraAB037203CYP716A12MedicagotruncatulaCBN88268.1ATR1ArabidopsisthalianaBT008426.1
表2 本研究使用的菌株
Table 2 Strains used in this study

菌株特征来源C800CEN.PK2-1D;MATα;ura3-52;his311;trp1-289;leu2-3,112;MAL2-8C;SUC2;gal80:KanMX[15]β01C800;Δ911b::ERG1-PGAL10-GgbAS1本研究β01-1C800;Ty4::ERG1-PGAL10-GgbAS1-TRP-Degradationtag本研究β02β01;Δ911b::PGAL7-tHMG1-PTEF1-IDI1本研究β03β02;ΔDPP1::ERG20-PGAL10-ERG9本研究OA-1β03;ΔDOS2::PPGK1-CYP716A12-PTEF1-ATR1本研究OA-2β03;Ty3::CYP716A12-PGAL10-ATR1-LEU-Degradationtag本研究OA-3OA-1;Ty3::PTEF-CYP716A12-LEU-Degradationtag本研究OA-401OA-3;ΔW10::PTEF-CYP716A12本研究OA-402OA-3;ΔW10::PTEF-ATR1本研究OA-403OA-3;ΔW10::CYP716A12-PGAL10-ATR1本研究OA-501OA-4;ΔW11::CYP716A12-PGAL10-ATR1本研究OA-502OA-4;ΔW11::PTEF-ATR1本研究OA-601OA-5;ΔYJL064W::PPGK1-ALD6-PGAL7-ACS1本研究OA-1-602OA-5;ΔYJL064W::PPGK1-ALD6-PGAL7-ACS2本研究OA-7OA-6;PADH2::HIStag-PTEF本研究OA-8OA-7;Ty2::PTEF-POS5-URA3-Degradationtag本研究
表3 本研究使用的质粒
Table 3 Plasmids used in this study

质粒特征来源pY26-TEFp-GPDpAmpR,URA3,Expressionplasmid本实验pRS426sgRNA,Expressionplasmid本实验室pCT44TY4-TRPdeg本实验室pY26-DPL-1pY26-ERG1-PGAL10-GgbAS1,possessing911bhomologousarm本研究pCT44pCT44-Ty4-ERG1-PGAL10-GgbAS1-TRPDegradationtag本研究pY26-DPL-2pY26-PGAL7-tHMG1-PTEF1-IDI1,possessing911bhomologousarm本研究pY26-DPL-3pY26-ERG20-PGAL10-ERG9,possessingDPP1homologousarm本研究pY26-DPL-4pY26-PPGK1-CYP716A12-PTEF1-ATR1,possessingDOS2homologousarm本研究pCT37-1pCT37-CYP716A12-PGAL10-ATR1-LEU-Degradationtag本研究pCT37-2pCT37-PTEF-CYP716A12-LEU-Degradationtag本研究pY26-DPL-5pY26-PTEF-CYP716A12,possessingW10homologousarm本研究pY26-DPL-6pY26-PTEF-ATR1,possessingW10homologousarm本研究pY26-DPL-7pY26-CYP716A12-PGAL10-ATR1,possessingW10homologousarm本研究pY26-DPL-8pY26-PTEF-ATR1,possessingW11homologousarm本研究pY26-DPL-9pY26-CYP716A12-PGAL10-ATR1,possessing911bhomologousarm本研究pY26-DPL-10pY26-PPGK1-ALD6-PGAL7-ACS1,possessingYJL064Whomologousarm本研究pY26-DPL-11pY26-PPGK1-ALD6-PGAL7-ACS2,possessingYJL064Whomologousarm本研究pY26-DPL-12pY26-PADH2::HIStag-PTEF本研究pCT22pCT22-PTEF-POS5-URA3-Degradationtag本研究pRS426-sgDPP1sgRNAexpressionplasmidforinsertingatDPP1本研究pRS426-sgDOS2sgRNAexpressionplasmidforinsertingatDOS2本研究pRS426-sgYJL064WsgRNAexpressionplasmidforinsertingatYJL064W本研究pRS426-sgW10sgRNAexpressionplasmidforinsertingatW10本研究pRS426-sgW11sgRNAexpressionplasmidforinsertingatW11本研究
表4 本研究使用的引物
Table 4 Primers used in this study

引物名称碱基序列(5′-3′)GgbAS1-FagaacttagtttcgacggatatgtggcgtttgaaaattgccgGgbAS1-RgaatgtaagcgtgacataactcaagtcaaacaaactggagtagatggcERG1-FcaaacctctggcgaagaattttaaccaatcaactcaccaaacaaaaatgggERG1-RatctaagttttctagaactaatgtctgctgttaacgttgcaccGAL10p-FgtaaacgttctgagacattatagttttttctccttgacgttaaagtatagaggtatattaacGAL10p-RttgtaatagctttcccatttatattgaattttcaaaaattcttactttttttttggatggacgTy4-up-FgaaacattttgaagctatatcgcacgcattccatgcTy4-down-RccagtttgtttgacttgaatccgctctaaccgaaaaggaagtHMG1-FcaacatggaagccaacctatttaggatttaatgcaggtgacggacctHMG1-RagttgaatattccctcaaaaatggctgcagaccaattggtgGAL7p-FaccaattggtctgcagccatttttgagggaatattcaactgtttttttttatcatgttgGAL7p-RtcatttttcatttacttctctttgccagcttactatccttcttgaaaatatgcIDI1-FctaagttttctagaactagtatgactgccgacaacaatagtatgccIDI1-RtgaggcttttgacattttatagcattctatgaatttgcctgtcattttccTEFp-FaatgggcataaatataaatggagctcatagcttcaaaatgtttctactcctTEFp-Rctattgttgtcggcagtcatactagttctagaaaacttagattagattgctatgctttc911b-upcattttgaagctatgagctccatttatatttatgcccattcaacatccgatttttttc911b-downaaggatagtaagctggcaaagagaagtaaatgaaaaatgaaatagcatacaaaacagacERG9-FttttgaaaattcaatataaatgggaaagctattacaattggcattgERG9-RaaatgaatggaattgctgtcacgctctgtgtaaagtgtatatataataaaacccaagERG20-FatttacacacaaaacaaaatggcttcagaaaaagaaattaggagagagERG20-RacatggaagccaacctatttaaacattaactctttctagttcttccctaccagTDH1p-FtataaactttagaagtaagaaaccacaccgtggggcTDH1p-Rttctttttctgaagccattttgttttgtgtgtaaatttagtgaagtactgttttttgte22T-Fagaaagagttaatgtttaaataggttggcttccatgttggcte22T-RgtcattgaaaagatatgaaatgtcaaaagcctcaaggtgccDPP1-up-FaacctctggcgaagaattactattattaaggcgcttctgtttttagtcaacDPP1-up-FgccccacggtgtggtttctttggtcgtttgctatgatttaattctgattctttgDPP1-down-FactcaagtgattttagaagaataaaaaagaatatatactccacatgacatacgaaatDPP1-down-RacgacgttgtaaaacgacttatacatagtatgtgttaaggggaacggaaaCYP716A12-FtttacaacaaatataaaacaatggagccaaatttctacttgtctttgttgCYP716A12-RaccttgaggcttttgacatttcaagctttgtgtgggtacaatctaattggATR1-FcaacatggaagccaacctatttaccaaacatctctcaagtatctaccttcagtATR1-RgttttctagaactagtatgactagtgctttgtatgcttctgatttgtttaaacDOS2-up-FcattttgaagctatgagctctttatcctaaaacgtcgaagcaatgDOS2-up-RtcacgacgttgtaaaacgacggtaatgccctttatcaatcagaaatDOS2-down-FgggaacaaaagctggagctcctccaggtgttaatgacgatgaacDOS2-down-RttcagctggcgtaatagcgtgatttttttcatataaaagatatatgtaatgctataaatTy3-up-FattgtacccacacaaagcttgagttaacattacgttaataaataggtatatatgaatatttataccTy3-down-RcaactaaaagcaattacagtcgtcgaacctcactagattatggPGK1p-FtaatctagtgaggttcgacgactgtaattgcttttagttgtgtatttttagPGK1p-RaagtagaaatttggctccattgttttatatttgttgtaaaaagtagataattacttcctTy3-CYP716A12-FtttacaacaaatataaaacaatggagccaaatttctacttgtctttgttgTy3-CYP716A12-RatacctatttattaacgtaatgttaactcaagctttgtgtgggtacaatctaattggYJL064W-up-FgggaacaaaagctggagctctggaactagtcgactctcccgtgatgYJL064W-up-RaggccccacggtgtggtttctgtgttagcctgatcacatcgagcgYJL064W-down-FaattcagctggcgtaatagcctacacaagcacattgcatagaaggatttttcttctYJL064W-down-RcacgacgttgtaaaacgaccaaaaagaggttgaaatcattgaagaacaacccatgagACS1-FtttacaacaaatataaaacaatgtcgccctctgccgtacaatcatcACS1-RtttacaacaaatataaaacaatgtcgccctctgccgtacaatcatcACS2-FagttgaatattccctcaaaaatgacaatcaaggaacataaagtagtttatgaagctACS2-RgaatgtaagcgtgacataacttatttctttttttgagagaaaaattggttctctacagcALD6-FtttacaacaaatataaaacaatgactaagctacactttgacactgctALD6-RcaaacctctggcgaagaattttacaacttaattctgacagcttttacttcagtgtatgADH2-FtttacaacaaatataaaacaatgtctattccagaaactcaaaaagccattatcttcADH2-RcaacatggaagccaacctatttatttagaagtgtcaacaacgtatctaccagcPOS5-FaccttgaggcttttgacattttaatcattatcagtctgtctcttggtcagccPOS5-Rctaagttttctagaactagtatgtttgtcagggttaaattgaataaaccagt
1.2 试剂与培养基
2×Phanta Max Master Mix高保真PCR扩增酶、2×Taq PCR Master Mix PCR扩增酶、DNA纯化试剂盒、质粒提取试剂盒,南京诺唯赞生物科技有限公司;吉布森组装酶,无锡伟沃佰赛生物科技有限公司;鲑鱼精(ssDNA),北京索莱宝科技有限公司;无氨基酵母氮源(no amino yeast nitrogen source,YNB)液体培养基、酵母粉、蛋白胨,生工生物工程(上海)股份有限公司。
大肠杆菌培养基LB(g/L):蛋白胨10、酵母粉5、氯化钠10、固体培养基在此基础上需添加2%(质量分数)琼脂粉,121 ℃,20 min灭菌。
酵母培养基YPD(g/L):蛋白胨20、酵母粉10、葡萄糖20、固体培养基在此基础上需添加2%(质量分数)琼脂粉,115 ℃、20 min灭菌。
筛选培养基YNB(g/L):琼脂粉、葡萄糖、YNB、亮氨酸、色氨酸、组氨酸、尿嘧啶。
发酵罐培养基(g/L):酵母粉10、蛋白胨20、葡萄糖40、七水硫酸镁1.5、碳酸钙4,葡萄糖另外灭菌,其他合并115 ℃,20 min灭菌。
1.3 摇瓶和发酵罐发酵方法
摇瓶发酵方法:将改造后的工程菌株于YPD上划线活化后,挑取单菌落接于50 mL装有5 mL液体YPD的培养基中,30 ℃,220 r/min培养24 h后取250 μL接于250 mL含有25 mL YPD液体培养基中,培养5 d。
发酵罐发酵方法:将于平板活化的菌株,挑单菌落接于50 mL装有5 mL液体YPD的培养基中,30 ℃,220 r/min培养24 h得到一级种子液,再将一级种子液取3 mL接于500 mL装有150 mL YPD培养基中,30 ℃,220 r/min培养24 h得到二级种子液接于5 L-发酵罐中,将温度和pH分别控制在30 ℃和5.5,溶氧维持在30%,待初始葡萄糖消耗尽时流加质量浓度为500 g/L的葡萄糖。
1.4 发酵产物提取及检测
产物提取:将培养5 d后的发酵液,取2 mL于破碎管中,12 000 r/min离心后弃上清。加入与菌体量体积比为1∶1的玻璃珠后再加入1 mL乙酸乙酯。用FastPrep破碎7个循环,离心后上清液过0.22 μL无菌滤膜后待GC-MS及HPLC检测。
前体物质β-香树脂GC-MS检测:色谱柱HP-5 ms;氦气流速1 mL/min;进样温度250 ℃;进样量1 μL;炉温:80 ℃保持2 min,20 ℃/min升高至260 ℃,5 ℃/min升高至290 ℃保持10 min。
齐墩果酸HPLC检测:色谱柱采用安捷伦IX952505-0 Innoval C18 (250 mm×4.6 mm,5 μm),流动相A相为0.01%三氟乙酸水溶液;B相为甲醇,条件为A∶B=10%∶90%。
2 结果与分析
2.1 β-香树脂醇路径构建及前体强化
β-香树脂醇是齐墩果酸合成的前体物质,在酿酒酵母中以2,3-环氧化角鲨烯为前体,外源引入β-香树脂醇合酶合成。本文引入来源于光果甘草(Glycyrrhiza glabra)的β-香树脂醇合酶[16],将编码β-香树脂醇合酶基因GgbAS1和编码鲨烯环氧化酶基因ERG1一起过量表达后导入出发菌株C800中,检测后发现仅产生了6.30 mg/L的β-香树脂醇,而前体物质角鲨烯与对照菌株相比未出现明显降低,这可能是由于单拷贝的GgbAS1,无法实现角鲨烯的高效转化(图2-a)。为了进一步提高β-香树脂醇积累,将GgbAS1和ERG1同时整合于出发菌株的Ty4多拷贝位点增加其表达量,β-香树脂醇产量提高至39.1 mg/L,是单拷贝整合的6倍,同时前体角鲨烯被大量转化仅剩1.5 mg/L(图2-b)。

a-911b位点整合GgbAS1和ERG1 β-香树脂醇产量;b-Ty4多拷贝位点整合GgbAS1和ERG1 β-香树脂醇产量;c、d-前体途径关键基因过量表达β-香树脂醇产量
图2 β-香树脂醇途径构建及前体途径强化
Fig.2 Construction of the β-amyrin pathway and enhancement of the precursor pathways
角鲨烯作为萜类物质合成的关键前体,其充足的供应对产物生产至关重要。HMG1是MVA途径中的限速酶,研究表明将HMG1基因截短可以有效提高MVA的通量。因此过量表达截短的HMG1(tHMG1)是增强萜类产物生产的常用策略;IDI1是平衡IPP和DMAPP的关键基因。将二者同时过量表达,β-香树脂醇产量可达92.7 mg/L,同时角鲨烯积累量明显增加(图2-c)。在此基础上,过量表达角鲨烯合成途径关键基因ERG9、ERG20,得到的菌株b03合成β-香树脂醇产量可达125.8 mg/L(图2-d)。
2.2 齐墩果酸途径构建及优化
2.2.1 齐墩果酸途径构建
在酿酒酵母中以β-香树脂醇为前体,通过引入细胞色素P450酶和细胞色素P450还原酶生成齐墩果酸。本文引入苜蓿(Medicago truncatula)来源的CYP716A12和拟南芥(Arabidopsis thaliana)来源的ATR1合成齐墩果酸。首先将CYP716A12和ATR1一起过量表达后整合于β03菌株中,得到一株产齐墩果酸为12.8 mg/L的菌株,命名为OA-1。该菌株前体β-香树脂醇积累量与对照相比未见明显降低(图3-a),可能是由于CYP716A12和ATR1的表达量不够,无法高效转化前体 β-香树脂醇。为了实现前体物质高效转化生成齐墩果酸,将两基因同时整合于OA-1菌株的Ty3多拷贝位点增加二者表达量,检测发现齐墩果酸产量为79.1 mg/L,产量虽较单拷贝两基因有所提升,但其前体物质β-香树脂醇积累量仍然很高(图3-b),说明CYP716A12和ATR1的表达量并不是影响前体物质高效转化生成齐墩果酸的主要原因。

a-DOS2位点整合CYP716A12和ATR1齐墩果酸产量;b-Ty3多拷贝位点整合CYP716A12和ATR1齐墩果酸产量
图3 齐墩果酸途径构建
Fig.3 Construction of the oleanolic acid pathway
2.2.2 优化P450s和CPR表达比例提高齐墩果酸产量
在2.2.1节中成功构建了齐墩果酸的从头合成途径,但并未实现齐墩果酸的高效生产,同时对关键基因表达量探究发现,等比例增加两关键基因拷贝数不能有效增强CYP716A12高效催化β-香树脂醇生成齐墩果酸。有研究表明P450s∶CPR的表达比率会影响P450s的催化性能,且高表达P450s有利于产物生成。CYP716A12是合成齐墩果酸的关键基因,因此将其整合于OA-1菌株的Ty3多拷贝位点得到OA-3菌株。经检测齐墩果酸产量为148.2 mg/L(图4-a),较OA-1菌株齐墩果酸产量提高了11.5倍,β-香树脂醇和角鲨烯积累量大幅降低,这意味着大量前体物质被有效转化。因此,在合成齐墩果酸的双酶系统中,CYP716A12的高表达有利于齐墩果酸的生成。

a-探究CYP716A12的高表达对齐墩果酸产量的影响;b、c-探究CYP716A12和ATR1具体比例对齐墩果酸的影响
图4 CYP716A12和ATR1表达比例优化提高齐墩果酸产量
Fig.4 Optimizing the expression ratio of CYP716A12 and ATR1 to increase the titer of oleanolic acid
为了继续平衡CYP716A12和ATR1的表达比率,将CYP716A12、ATR1、共表达的ATR1和CYP716A12分别导入OA-3菌株后,发现共表达CYP716A12和ATR1的一个拷贝导入OA-3菌株后可产生189.3 mg/L齐墩果酸,而只增加一个拷贝的CYP716A12,齐墩果酸积累量未明显提升(图4-b)。可见,CYP716A12虽发挥重要作用,但也需要与CPR表达平衡才能发挥其最佳催化活性。接着,又将共表达的CYP716A12和ATR1、ATR1分别整合于OA-4菌株的W11位点,发现单独增加一个ATR1的拷贝数后,齐墩果酸产量可达222.5 mg/L,而增加共表达CYP716A12和ATR1的一个拷贝数后产量为206.3 mg/L(图4-c),证明高比例的CYP716A12∶ATR1表达可以显著提高齐墩果酸的产量。
2.3 增强乙酰辅酶A供给及辅因子再生提高齐墩果酸产量
乙酰辅酶A的调节和再生已被证明可有效改善萜类化合物的合成。在酿酒酵母中,萜类物质的合成是利用胞质中的乙酰辅酶A,其通过丙酮酸脱氢酶(pyruvate dehydrogenase,PDH)旁路或乙醇再氧化途径合成,过量表达两途径中关键酶的编码基因ADH、ALD、ACS是提高乙酰辅酶A含量的常用策略[17]。ALD6和ACS基因编码的酶参与PDH途径,而酿酒酵母存在2个乙酰辅酶A合酶编码基因,即ACS2是组合型表达,ACS1的表达受葡萄糖的抑制[18]。因此,以GAL系列启动子用于ACS1和ACS2的过量表达。首先将ALD6分别与ACS1和ACS2组合,过量表达发现ALD6和ACS1组合产齐墩果酸的量为242.1 mg/L,较过量表达ALD6和ACS2组合产量明显增加(图5-a)。这可能是由于ACS1对前体乙酸的亲和力远高于ACS2。接着,通过增强乙醇的消耗促进乙酰辅酶A的积累。因为ADH2p启动子会受到葡萄糖和乙醇抑制,因此将其替换成强组成型启动子TEFp后,齐墩果酸的产量提升到259.3 mg/L(图5-b)。

a-过量表达ALD6和ACS1/ACS2齐墩果酸产量;b-替换ADH2p启动子齐墩果酸产量;c-CYP716A12利用NADPH示意图;d-过量表达POS5齐墩果酸产量
图5 增强乙酰辅酶A供应和强化NADPH再生系统提高齐墩果酸产量
Fig.5 Enhancing acetyl CoA supply and strengthening the NADPH regeneration system to improve oleanolic acid production
在整个齐墩果酸合成途径中,MVA途径中的许多酶,如tHMG1、ERG9、ERG1都会利用NADPH作为辅因子。而CYP716A12的活性可以与NADPH的再生协同提高。本研究过量表达了MVA途径的关键基因,且高表达了CYP716A12,这需要大量NADPH为其提供电子。POS5是编码NADH激酶的基因,有助于酿酒酵母中NADPH的再生。因此,将POS5整合于多拷贝位点后,得到一株齐墩果酸产量为304.0 mg/L的菌株(图5-d)。
2.4 齐墩果酸高产菌株的发酵优化
稳定适宜的培养条件利于菌株的生长,培养的策略也会影响菌株的生产能力,为了进一步提高菌株产齐墩果酸的能力,对摇瓶水平产304.0 mg/L齐墩果酸的一株菌株进行5 L-发酵罐优化实验。本文在齐墩果酸途径构建中应用了大量GAL系列启动子,该系列启动子在低葡萄糖浓度下被激活,所以需要严格控制发酵过程中的葡萄糖浓度。发酵以初始葡萄糖为40 g/L添加量进行,在发酵12 h后初始葡萄糖耗尽后以15 mL/h的速度补加葡萄糖,但发现菌株在对数生长期及一直到发酵后期OD值都很低,最终产物产量与摇瓶相当(图6-a),分析原因可能是前期补加糖过量,产生过量的乙醇对菌株生长造成胁迫而影响菌株的生产能力。因此,后续实验依据菌株的乙醇积累量来调整葡萄糖的补加量,菌株在12 h后初始葡萄糖耗尽,但此时乙醇积累量较高。因此,以5 mL/h的速度缓慢补加葡萄糖,待乙醇消耗到4~5 g/L时逐步加大葡萄糖的流加速度,但始终维持残糖基本为0的状态。通过实时检测菌株乙醇的积累量来控制葡萄糖的补加速度后,发现菌株长势良好且产量提升至480.2 mg/L(图6-b)。菌株发酵过程中葡萄糖的供应平衡对其生长和生产至关重要,另外发酵过程中的培养条件也非常关键。碳酸钙可以保持发酵系统中pH的相对稳定性,因此在发酵培养基中加入10 g/L 碳酸钙,发现菌株OD最大值得到提升,齐墩果酸的产量提升至550.24 mg/L(图6-c)。另外,在发酵48 h时,开始补加N源为菌株的生长提供充足营养物质,结果表明N源的补加更利于菌株的生长和生产,齐墩果酸产量可达680.8 mg/L(图6-d)。
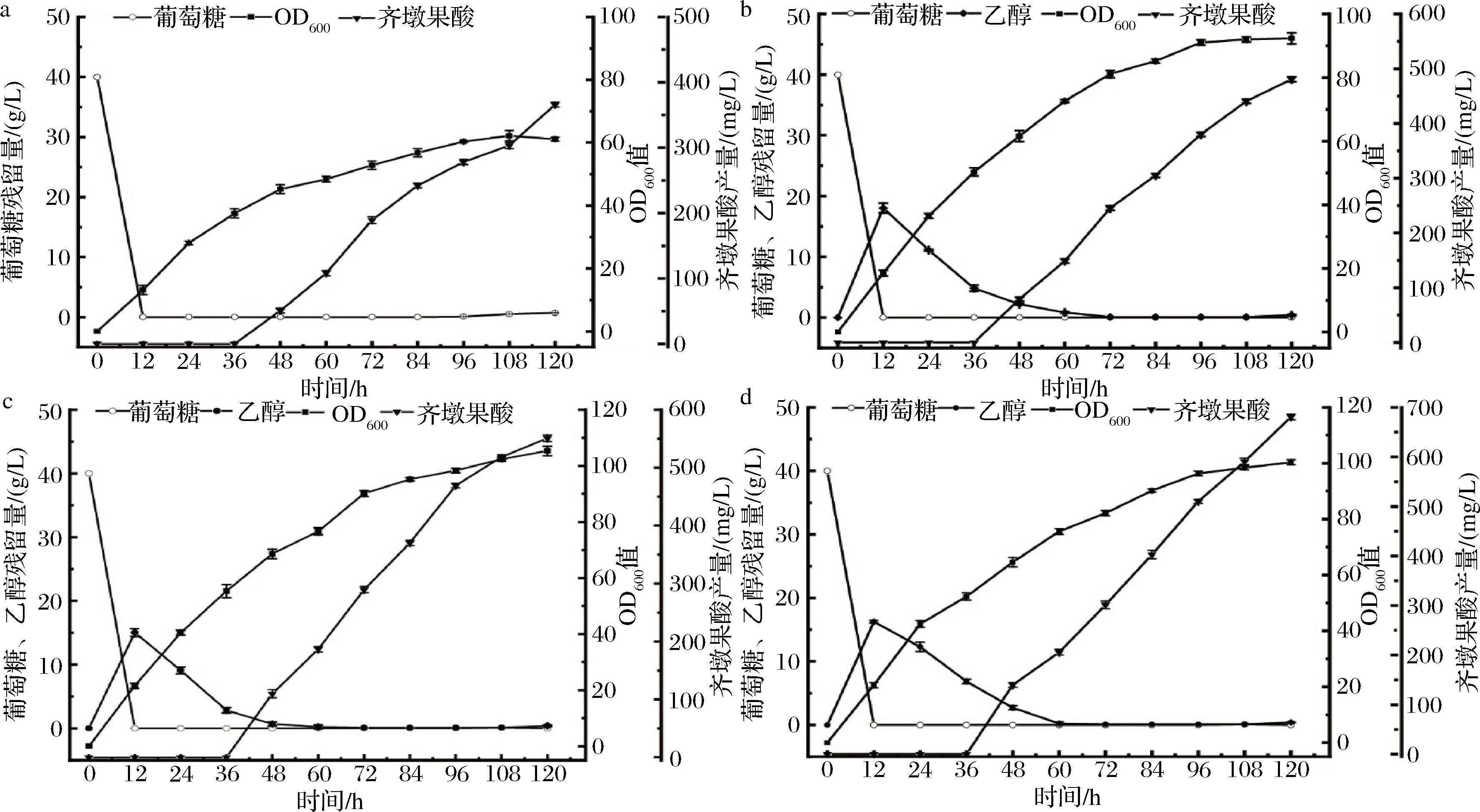
a、b-发酵过程中葡萄糖的补加速度对齐墩果酸产量的影响;c-碳酸钙对齐墩果酸产量影响;d-N源对齐墩果酸产量影响
图6 5 L-发酵罐中高密度发酵齐墩果酸的生产
Fig.6 High-density fermentation for OA production in a 5 L-fermenter
3 讨论
齐墩果酸因其丰富的药理活性在医药领域具有较高利用价值。本文在酿酒酵母中成功构建了齐墩果酸从头合成途径。然而,合成途径中涉及到的细胞色素P450酶的表达一直是制约产物生产的关键问题。针对提高P450s的高效表达的策略,研究人员通过筛选适配性较高的P450s和CPR优化氧化还原体系[19]。通过筛选合适的连接肽构建P450s-CPR融合蛋白,但对连接肽的筛选较严格且并不是所有P450s和CPR都适合融合表达。通过定向进化和理性/半理性设计P450s-CPR相互作用提高电子转移效率[20],但因P450s的蛋白是膜锚定蛋白,难以分离和结晶而缺乏准确的晶体结构。近期SUN等[21]在甘草次酸前体物质11-oxo-β-amyrin的生物合成中,通过平衡CYP88D6与CPR的表达比例提高了CYP88D6的氧化活性,证明高比例的CYP88D6∶CPR更利于前体物质的转化和终产物的生成。XU等[22]通过高表达途径中的P450s调整CYP∶CPR的比率结合其他代谢工程手段实现了薯蓣皂苷从毫克到克级别的合成。本研究主要探究P450s和CPR的具体表达量及表达比例,通过对CYP716A12和ATR1的表达量进行优化以提高P450s的催化活性,证明了CYP716A12的高表达利于齐墩果酸的合成,同时发现高比例的CYP716A12∶ATR1表达不仅利于齐墩果酸的生产,也利于前体物质的转化。另外,CHA等[23]通过筛选不同强度的启动子对HPO/AtCPR的表达进行微调,发现使用强启动子对HPO进行表达,较弱启动子对AtCPR进行表达更利于(+)-诺卡酮生产。此后的研究中,也可以通过筛选不同强度的启动子对CYP716A12/ATR1的表达比率进行调整,可继续探究合适的表达比率具体体现在基因表达量还是在二者表达强度上。
为了继续增强菌株生产齐墩果酸的能力,本研究通过增强乙酰辅酶A继续增加前体通量,以及通过增强辅因子的供应平衡NADPH还原当量提高了齐墩果酸的产量,但增幅没有预想高,关键问题可能还是由于NADPH的失衡,因为在齐墩果酸合成途径中表达了较多拷贝的CYP716A12。在熊果酸的合成中,研究人员通过将可增加熊果酸产量的2个乙酰辅酶A相关基因和参与NADH/NADPH基因组合优化将强化乙酰辅酶A和辅因子工程联合平衡细胞氧化还原的稳态后显著增加熊果酸的产量[24]。后续研究中可以着重研究细胞氧化还原稳态的平衡从而继续提高齐墩果酸的产量。综上,本文利用代谢工程策略实现了目前报道中齐墩果酸的最高产量,为利用酿酒酵母合成齐墩果酸和下游产物相关研究奠定了一定基础。
[1] NISTOR G,TRANDAFIRESCU C,PRODEA A,et al.Semisynthetic derivatives of pentacyclic triterpenes bearing heterocyclic moieties with therapeutic potential[J].Molecules,2022,27(19):6552.
[2] YU Z J,SUN W Z,PENG W B,et al.Pharmacokinetics in vitro and in vivo of two novel prodrugs of oleanolic acid in rats and its hepatoprotective effects against liver injury induced by CCl4[J].Molecular Pharmaceutics,2016,13(5):1699-1710.
[3] WU Y W,YUAN Z B,RAO Y J.Current advances in the biotechnological synthesis of betulinic acid:New findings and practical applications[J].Systems Microbiology and Biomanufacturing,2023,3(2):179-192
[4] LIU X N,DING W T,JIANG H F.Engineering microbial cell factories for the production of plant natural products:From design principles to industrial-scale production[J].Microbial Cell Factories,2017,16(1):125.
[5] DAI Z B,WANG B B,LIU Y,et al.Producing aglycons of ginsenosides in bakers′ yeast[J].Scientific Reports,2014,4:3698.
[6] LU C Z,ZHANG C B,ZHAO F L,et al.Biosynthesis of ursolic acid and oleanolic acid in Saccharomyces cerevisiae[J].AIChE Journal,2018,64(11):3794-3802.
[7] LI D S,WU Y F,WEI P P,et al.Metabolic engineering of Yarrowia lipolytica for heterologous oleanolic acid production[J].Chemical Engineering Science,2020,218:115529.
[8] ZHAO Y J,FAN J J,WANG C,et al.Enhancing oleanolic acid production in engineered Saccharomyces cerevisiae[J].Bioresource Technology,2018,257:339-343.
[9] ZHU M,WANG C X,SUN W T,et al.Boosting 11-oxo-β-amyrin and glycyrrhetinic acid synthesis in Saccharomyces cerevisiae via pairing novel oxidation and reduction system from legume plants[J].Metabolic Engineering,2018,45:43-50.
[10] XU L H,DU Y L.Rational and semi-rational engineering of cytochrome P450s for biotechnological applications[J].Synthetic and Systems Biotechnology,2018,3(4):283-290.
[11] CHENG J,WAN D F,GU J R,et al.Establishment of a yeast system that stably expresses human cytochrome P450 reductase:Application for the study of drug metabolism of cytochrome P450s in vitro[J].Protein Expression and Purification,2006,47(2):467-476.
[12] MURATALIEV M B,FEYEREISEN R,WALKER F A.Electron transfer by diflavin reductases[J].Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics,2004,1698(1):1-26.
[13] PETERSON J A,EBEL R E,O’KEEFFE D H,et al.Temperature dependence of cytochrome P-450 reduction.A model for NADPH-cytochrome P-450 reductase:Cytochrome P-450 interaction[J].The Journal of Biological Chemistry,1976,251(13):4010-4016.
[14] ZANGAR R C,DAVYDOV D R,VERMA S.Mechanisms that regulate production of reactive oxygen species by cytochrome P450[J].Toxicology and Applied Pharmacology,2004,199(3):316-331.
[15] GAO S,ZHOU H R,ZHOU J W,et al.Promoter-library-based pathway optimization for efficient (2 S)-naringenin production from p-coumaric acid in Saccharomyces cerevisiae[J].Journal of Agricultural and Food Chemistry,2020,68(25):6884-6891.
[16] DU M M,ZHU Z T,ZHANG G G,et al.Engineering Saccharomyces cerevisiae for hyperproduction of β-amyrin by mitigating the inhibition effect of squalene on β-amyrin synthase[J].Journal of Agricultural and Food Chemistry,2022,70(1):229-237.
[17] LIU H,FAN J J,WANG C,et al.Enhanced β-amyrin synthesis in Saccharomyces cerevisiae by coupling an optimal acetyl-CoA supply pathway[J].Journal of Agricultural and Food Chemistry,2019,67(13):3723-3732.
[18] JIN K,SHI X,LIU J H,et al.Combinatorial metabolic engineering enables the efficient production of ursolic acid and oleanolic acid in Saccharomyces cerevisiae[J].Bioresource Technology,2023,374:128819.
[19] ZHAO F L,BAI P,LIU T,et al.Optimization of a cytochrome P450 oxidation system for enhancing protopanaxadiol production in Saccharomyces cerevisiae[J].Biotechnology and Bioengineering,2016,113(8):1787-1795.
[20] LI Z,JIANG Y Y,GUENGERICH F P,et al.Engineering cytochrome P450 enzyme systems for biomedical and biotechnological applications[J].Journal of Biological Chemistry,2020,295(3):833-849.
[21] SUN M C,XIN Q,HOU K X,et al.Production of 11-oxo-β-amyrin in Saccharomyces cerevisiae at high efficiency by fine-tuning the expression ratio of CYP450:CPR[J].Journal of Agricultural and Food Chemistry,2023,71(8):3766-3776.
[22] XU L P,WANG D,CHEN J,et al.Metabolic engineering of Saccharomyces cerevisiae for gram-scale diosgenin production[J].Metabolic Engineering,2022,70:115-128.
[23] CHA Y P,LI W,WU T,et al.Probing the synergistic ratio of P450/CPR to improve (+)-nootkatone production in Saccharomyces cerevisiae[J].Journal of Agricultural and Food Chemistry,2022,70(3):815-825.
[24] JIA N,LI J Z,ZANG G W,et al.Engineering Saccharomyces cerevisiae for high-efficient production of ursolic acid via cofactor engineering and acetyl-CoA optimization[J].Biochemical Engineering Journal,2024,203:109189.