胰蛋白酶(EC 3.4.21.4)属于丝氨酸蛋白酶家族,在水解蛋白质肽链中精氨酸或赖氨酸残基的羧基末端的肽或酰胺键方面具有高度选择性和特异性[1-2]。胰蛋白酶被广泛用于制革、食品加工、制药、生化检测和洗涤剂等多种行业[3-4]。在制革工业中,胰蛋白酶用于局部鞣革及皮革脱灰软化,这一步可以起到清除裸皮中的皮垢、纤维基质,疏松胶原调节皮革松紧度的作用,从而使皮革的柔软度更丰满、皮面更光滑富弹性[5-6],也可以利用胰蛋白酶提取具有良好生物相容性和生物降解性的胶原蛋白[7]。在制药工业中,胰蛋白酶可用于水解变性蛋白质,或将蛋白质水解成低分子质量的肽和氨基酸[8],以达到脱敏的目的[5]。在食品工业中,胰蛋白酶的应用集中于水解价值较高的动植物性蛋白、脂肪和淀粉等。
胰蛋白酶最初在牛的胰腺中被发现[9]。如今,胰蛋白酶已从各种来源中被分离出来,比如哺乳动物[7](人、牛、猪和狗)和无脊椎动物。近年来,微生物来源胰蛋白酶也被发现,比如Streptomyces griseus[8]和Bacillus licheniformis[10]。过去胰蛋白酶的商业化生产主要依赖牛或其他哺乳动物的胰腺组织提取[9],然而这种方法存在生产成本高、与结构和功能特性相似的蛋白酶α-糜蛋白酶有污染、稳定性低、纯度低、污染性强等缺点,阻碍了其深入应用[11]。将胰蛋白酶在微生物中进行异源表达可以短时间内产生大量的活性胰蛋白酶[12],并通过改造和发酵优化可以提高表达量和改善催化性质等,为其在医疗、皮革行业中的应用等提供了更多可能性。
如今,人、牛[13]、鱼[14]和虾[15-16]来源的胰蛋白酶已经成功在大肠杆菌中表达。然而,大肠杆菌的成熟胰蛋白酶产量很低,且通常以包涵体的形式出现[17]。与大肠杆菌表达系统相比,法夫驹形氏酵母表达系统更适合重组蛋白的表达,尤其适合表达一些需要进行糖基化或二硫键形成的蛋白[18]。胰蛋白酶原可以在K.phaffii中以可溶性形式表达[19],但其激活依赖于用肠激酶或者胰蛋白酶消化,且产量低、后续没有很多关于酶学性质的报道。目前有研究报道的微生物源胰蛋白酶主要为灰色链霉菌胰蛋白酶(Streptomyces griseus trypsin, SGT),其以胰蛋白酶形式在K.phaffii GS115、大肠杆菌、变铅青链霉菌、枯草芽孢杆菌中均成功实现了异源表达。弗氏链霉菌胰蛋白酶(Streptomyces fradiae trypsin, SFT)和SGT的氨基酸同源性高达85%,但是SFT的异源表达目前只在变铅青链霉菌和大肠杆菌中有过报道,且酶活仅为22~29 mU/mL,具有较大的优化潜力。
本研究通过基因挖掘策略获得了4种不同来源的胰蛋白酶基因,并成功在K.phaffii GS115中异源表达。进分析了重组酶的酶学性质,为后续重组酶的分子改造及应用奠定基础。本文为首次报道弗氏链霉菌来源胰蛋白酶在法夫驹形氏酵母中异源表达。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
Escherichia coli JM109、K.phaffii GS115和质粒pPIC9k均保藏于本实验室。
1.1.2 主要试剂
DNA maker、感受态细胞制备试剂盒、DNA限制性内切酶,大连TaKaRa公司;硫酸卡那链霉素, Sigma公司;质粒提取剂盒、Cycle Pure试剂盒,OMEGA Bio-tek公司;底物BAPNA,上海源叶生物公司;NI-NT纯化树脂预装柱、改良型Bradford法蛋白浓度试剂盒、SDS-PAGE凝胶快速配制试剂盒、肠激酶(100 IU),生物工程(上海)有限公司;NaCl、甘油、胰蛋白胨、琼脂粉,国药集团有限公司。
1.1.3 培养基
LB培养基(g/L)(氯化钠10、蛋白胨10、酵母提取物10)用于培养E.coli JM109,根据需要加入50 μg/mL 硫酸卡那链霉素。YPD培养基(g/L)(酵母提取物10、蛋白胨20、葡萄糖20)用于培养K.phaffii GS115。固体培养基按照15 g/L添加琼脂。缓冲甘油复合培养基(buffered minimal glycerol medium, BMGY)(g/L)(酵母提取物10、胰蛋白胨20、甘油10、磷酸二氢钾100 mmol/L、YNB 13.4,pH6.0)作为培养基用于诱导前的菌体生长。缓冲甲醇复合培养基(buffered methanol-complex medium, BMMY)(g/L)(酵母提取物10、胰蛋白胨20、磷酸二氢钾100 mmol/L、YNB 13.4,pH 6.0)作为发酵培养基用于重组菌产酶。
1.2 仪器与设备
SPX-250生化培养箱,上海跃进医疗器械厂;全自动高压蒸汽灭菌锅,日本HIRAYAMA株式会社;PL2002电子天平,METTLER TOLEDO公司;电子数显pH计,德国Mettler Toledo公司;10 μL-1 mL各种不同型号的移液枪,德国Eppendorf公司;紫外-可见分光光度计,上海MAPADA仪器有限公司;T100 Thermal Cycle PCR仪,美国Bio-Rad公司;GelDoc-ItTM 凝胶成像系统;美国UVP公司;Powerpac Basic蛋白电泳仪,美国Bio-Rad公司;MICROL 17台式高速离心机,美国Thermo Scientific公司;恒温水浴锅,金坛市宏华仪器厂; KTA蛋白纯化系统,美国GE HealthCare公司。
KTA蛋白纯化系统,美国GE HealthCare公司。
1.3 实验方法
1.3.1 基因挖掘和胰蛋白酶基因序列分析
通过NCBI数据库的BLAST对胰蛋白酶基因进行挖掘,使用来自Streptomyces griseus(Genbank No.M64471.1)和Bovine(Genbank No.X54703.1)的胰蛋白酶序列作为模板。选取与模板氨基酸序列同源性大于60%的潜在胰蛋白酶基因,用MEGA6软件搭建胰蛋白酶进化树,同时采用BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)和Clustal W软件对得到的胰蛋白酶氨基酸序列进行比对,以确保相应序列中存在保守的催化区域。
1.3.2 重组菌株的构建
从基因组挖掘中获得的胰蛋白酶编码基因送至金唯智(苏州)经过密码子优化后合成,并连接到质粒pPIC9k。将重组质粒转化到E.coli JM109,于含有50 μg/mL硫酸卡那霉素的LB平板上,在37 ℃培养12 h,从平板上挑出单个菌落,通过菌落PCR进行验证。将阳性转化体转移到含有50 μg/mL硫酸卡那霉素的LB液体培养基中,在37 ℃、180 r/min培养12 h收集菌体提取质粒。提取的质粒用Sal I限制性酶进行线性化处理后通过Cycle Pure Kit(OMEGA Bio-tek,美国)回收。将线性化载体通过电击转化至K.phaffii GS115感受态细胞。使用pPIC9k通用引物(α-factor)进行PCR验证,将验证正确的转化子用于后续诱导培养。
1.3.3 重组胰蛋白酶的表达和纯化
将阳性转化子接种到BMGY培养基中,在30 ℃、180 r/min培养至OD600值达到2.0~6.0时,将溶液转移到50 mL无菌离心管中,以4 000 r/min离心5 min以去除上清液后。将收集的细胞与5 mL无菌生理盐水混合,在4 000 r/min离心5 min洗涤菌液。上述洗涤过程重复2次之后,用BMMY培养基重新悬浮清洗过的酵母细胞,直到OD600值为1.0,并在30 ℃、180 r/min培养。在培养过程中,每24 h加入甲醇,最终质量浓度为10 g/L,以诱导外源蛋白的表达。
重组胰蛋白酶通过Ni-NTA重力柱纯化,使用20~500 mmol/L线性梯度的咪唑溶液。为了防止重力柱的堵塞,粗酶液在纯化前通过0.45 μm的滤膜进行过滤。梯度洗脱缓冲液被设定为20、50、350、500 mmol/L,目标蛋白在350 mmol/L咪唑浓度下被洗脱。用截留分子质量为10 kDa的蛋白质超滤管(PALL,美国)浓缩洗脱液,用PD-10脱盐柱(GE healthcare,美国)进行脱盐。收集样品用于后续SDS-PAGE的分析。蛋白浓度用Bradford蛋白浓度测定试剂盒(上海生工)进行测定。
1.3.4 重组胰蛋白酶的活化
经过诱导表达和纯化,得到的胰蛋白酶以酶原形式存在。为获得活性胰蛋白酶,将90 μL重组胰蛋白酶原与0.4 μL牛肠激酶(100 IU,S10204,中国源叶)混合,在25 ℃孵育16 h。
1.3.5 重组胰蛋白酶的酶活性测定
将43.5 mg N-苯甲酰基-DL-精氨酰-4-硝基苯胺盐酸盐(N-Benzoyl-DL-arginine-4-nitroanilidehydrochloride,BAPNA)溶解于1 mL二甲基亚砜中,之后再将该混合体系溶解于含有10 mmol/L CaCl2的pH 8.0,50 mmol/L Tris-HCL缓冲液。在25 ℃下,测定200 μL粗酶液同3 mL BAPNA底物溶液在光径1 cm的比色皿中,在410 nm下10 min内的吸光值变化。在25 ℃和pH 8.0条件下,ΔA410/min增加0.001被定义为胰蛋白酶的一个酰胺酶水解单位。酶活计算如公式(1)所示:
(1)
式中:BAPNA unit,酰胺酶酶活,U/mL;0.2,加入酶液体积,mL。
1.3.6 酶学性质测定
通过测量不同温度(20、30、40、50、60、70、80 ℃)和不同pH值(4、5、6、7、8、9、10)下胰蛋白酶的活性,绘制胰蛋白酶在不同温度或pH下的酶活曲线。最高的胰蛋白酶活性被记录为100%,而其他酶以酶活性的相对百分比表示。通过将胰蛋白酶在BR缓冲液(pH 4~5.5,pH 8.5~10)和磷酸盐缓冲液(pH 6~8)中孵育1 h后测定酶活性评估胰蛋白酶的pH稳定性。用处理后测定的酶活性除以未处理测得的酶活性,计算出残余活性的相对百分比。在最适条件下,测量胰蛋白酶在不同浓度(0.01~0.1 mmol/L)底物BAPNA下的反应速率,由双倒数法来计算Km和Vmax值。Vmax除以对应的蛋白摩尔数即为转化数kcat值。
1.3.7 生物信息学和统计分析
胰蛋白酶结构的同源模型是通过SWISS-MODEL在线服务器(https://swissmodel.expasy.org/)完成,通过pymol软件实现酶三维结构的可视化分析。由Invitrogen Vector NTI Advance 11.5软件完成序列比对和核苷酸序列比对分析。由ExPASy Prot Param软件分析酶的理论蛋白质分子质量和理论等电点,由SignalP4.0软件预测胰蛋白酶基因的信号肽。由Prism 9软件进行数理统计分析。
2 结果与分析
2.1 胰蛋白酶基因挖掘
基于BLAST分析,以灰色链霉菌胰蛋白酶基因(Genbank No.M64471.1)和牛胰蛋白酶基因(Genbank No.X54703.1)为模板,共获得194个胰蛋白酶基因,并构建了进化树。如图1-A所示,挖掘得到的胰蛋白酶基因可分为真核生物来源酶、细菌来源酶和古细菌来源酶三大类。基于哺乳动物源、甲壳动物源、微生物源胰蛋白酶在法夫驹形氏酵母中表达情况的不同,本文选择了4个不同来源胰蛋白酶基因进行进一步分析,包括来自Homo spanies(GenBank No.AAA61232.1)、Bos taurus(GenBank No.CAA38513.1)、Penaeus vannamei(GenBank No.AEZ67461.1)和S.fradiae ATCC14544(GenBank No.BAA04089.1)的胰蛋白酶基因,其详细信息如表1所示。通过酶氨基酸序列比对,发现4个来源胰蛋白酶基因中均具有丝氨酸蛋白酶保守的组氨酸H57,天门冬氨酸D102 和丝氨酸S195催化三联体(图1-B)[20]。进一步通过在线同源建模4个不同来源胰蛋白酶的三维结构,发现这4种胰蛋白酶具有相似的晶体结构,主要由2个双平行且相互间有一定的角度的b-疏水桶状结构域组成(图1-C)。
表1 通过基因挖掘筛选出的胰蛋白酶基因的信息
Table 1 Information of trypsin genes screened by genome mining
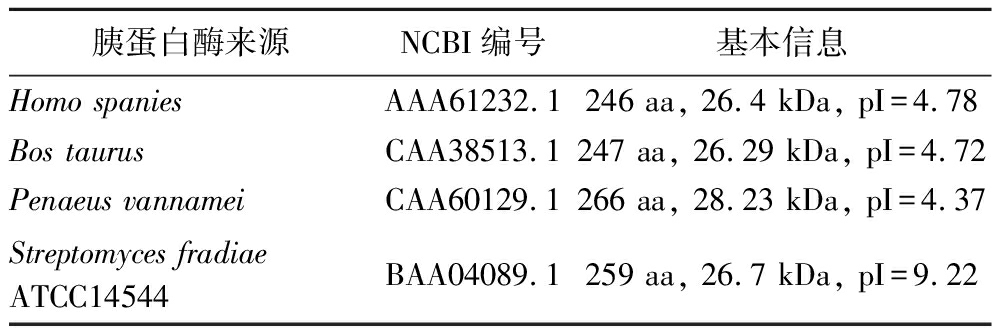
胰蛋白酶来源NCBI编号基本信息Homo spaniesAAA61232.1246 aa, 26.4 kDa, pI=4.78Bos taurusCAA38513.1247 aa, 26.29 kDa, pI=4.72Penaeus vannameiCAA60129.1266 aa, 28.23 kDa, pI=4.37Streptomyces fradiaeATCC14544BAA04089.1259 aa, 26.7 kDa, pI=9.22

A-胰蛋白酶进化树;B-催化保守区域序列比对;C-三维结构
图1 基因挖掘与生物信息学分析
Fig.1 Gene mining and bioinformatics analysis
2.2 胰蛋白酶在K.phaffii GS115中的异源表达和纯化
由于K.phaffii表达系统具有生长速度快、易于操作、表达稳定和产量大等特点,因此是更适于胰蛋白酶的异源表达[21]。质粒pPIC9k是一种带有AOX1启动子的分泌型表达载体,广泛用于K.phaffii表达系统。经密码子优化后,将人源胰蛋白酶(homo spanies trypsin, HST)、牛源胰蛋白酶(bos taurus trypsin, BTT)、虾源胰蛋白酶(penaeus vannamei trypsin, PVT)和弗氏链霉菌源胰蛋白酶基因通过E.coli JM109连接到pPIC9k质粒中,得到pPIC9k-hst、pPIC9k-btt、pPIC9k-pvt和pPIC9k-sft重组质粒,并成功转化到K.phaffii GS115。在BMGY培养基中测定了携带异源胰蛋白酶基因的重组K.phaffii GS115的生长曲线。如图2所示,携带pPIC9k-sft的重组K.phaffii菌株的生长曲线与野生型菌株相似,且其生长速度优于携带其他3种胰蛋白酶基因的重组菌株。携带pPIC9k-sft的重组K.phaffii菌株在稳定期的平均OD600值达到12.4,显著高于携带pPIC9k-hst(8.2)、pPIC9k-btt(3.3)和pPIC9k-pvt(9.4)的重组菌(P<0.05)。此外,携带pPIC9k-sft的重组K.phaffii菌株的迟滞期时间也较短。因此,来自微生物的胰蛋白酶可能具有在K.phaffii中表达的潜在价值。
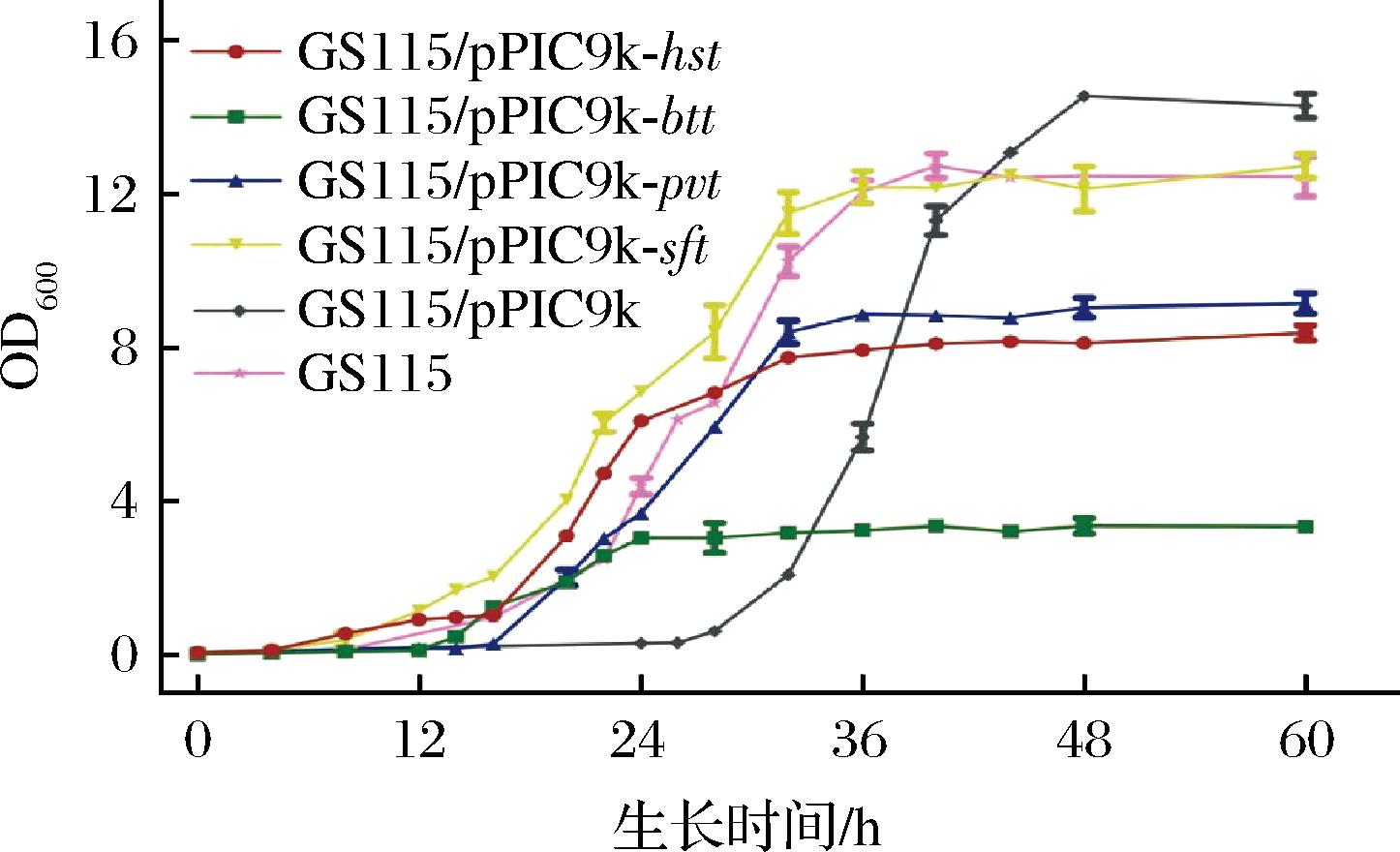
图2 重组K.phaffii菌株生长曲线
Fig.2 Growth curves of recombinant K.phaffii strain
为了进一步评估4种胰蛋白酶的酶学特性,将重组K.phaffii菌株的粗酶液进行浓缩并通过SDS-PAGE进行分析。如图3-A所示,HST、BTT、PVT和SFT的上清粗酶液中均观察到了对应蛋白条带,4种胰蛋白酶的蛋白分子质量分别为40、43、55、27 kDa,与理论分子质量一致。上述结果表明4种来源的胰蛋白酶在K.phaffii中均被成功表达。在此基础上,通过Ni2+亲和色谱法纯化重组胰蛋白酶。如图3-B所示,4种胰蛋白酶被成功纯化。此外,在SDS-PAGE胶上还观察到一些小分子质量的蛋白肽段,这些可能是由胰蛋白酶降解自身序列中的精氨酸和赖氨酸从而产生的自降解条带[22-23]。
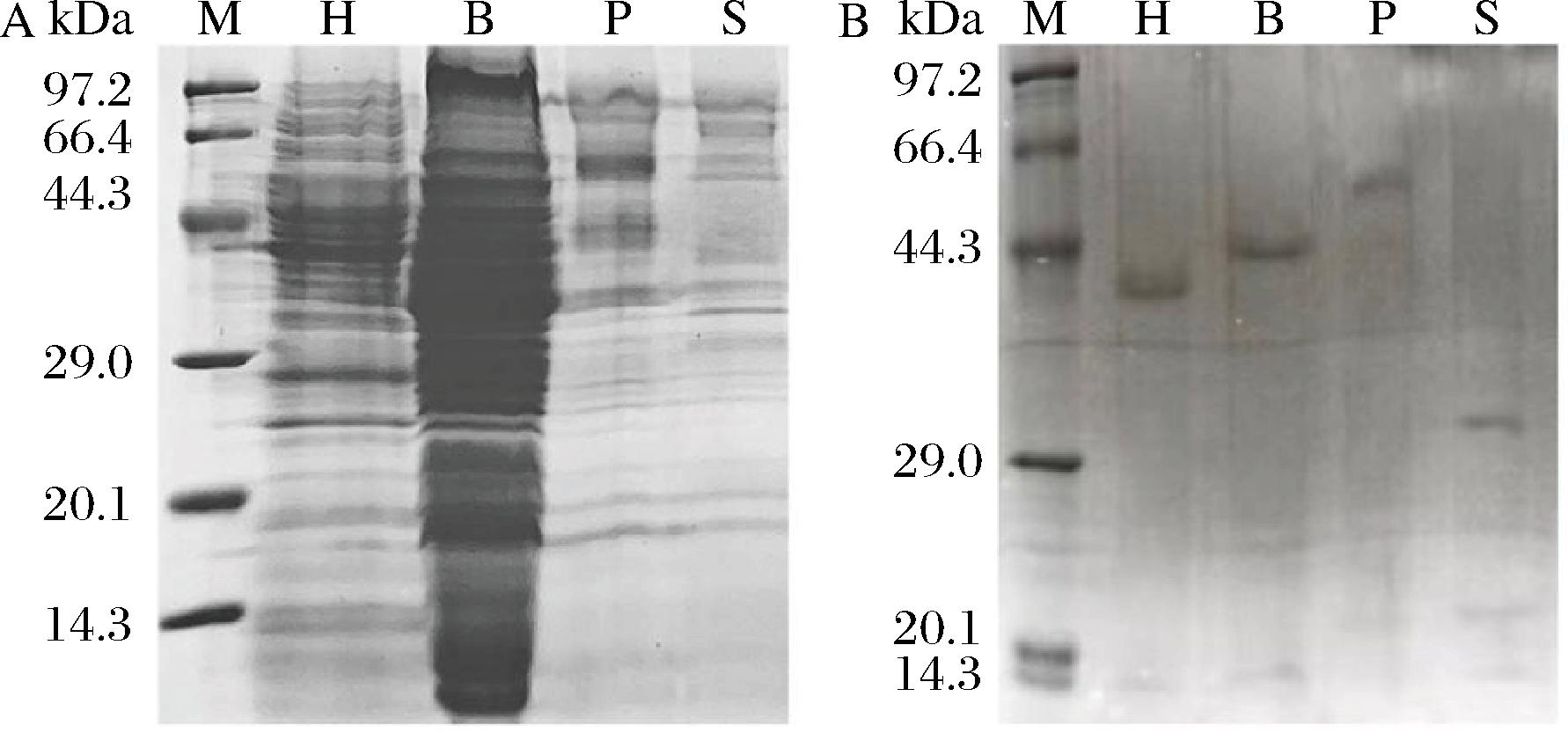
M-Maker;H-Homo spanies trypsin;B-Bos taurus trypsin; P-Penaeus vannamei trypsin;S-S.fradiae trypsin A-异源表达结果;B-蛋白纯化结果
图3 不同来源胰蛋白酶的表达与纯化
Fig.3 Expression and purification of trypsin from different sources
2.3 不同来源胰蛋白酶的酶学特性分析
测定了4种不同来源胰蛋白酶的酶学特性。如图4-A所示,HST、BTT、PVT和SFT的最适温度分别为20、35、40、45 ℃。由于皮革加工的最佳温度为30~45 ℃[24],因此,HST不适合应用于皮革加工。如图4-B所示,HST、BTT、PVT和SFT的最佳pH值分别为9、8.5、8.5、9。其中HST和SFT在pH 8~10能保持至少80%的酶活性,所以他们在碱性环境中具有更强的耐受能力。为了分析胰蛋白酶在碱性环境中的稳定性,将这4种胰蛋白酶在pH 6~11的缓冲液中处理1 h后,在最适条件下测量其剩余活性。如图4-C所示,4个来源的胰蛋白酶在pH 7~9的缓冲液处理后都能保持大于80%的活性。其中,SFT的耐碱性最强,其在pH 9~10缓冲液处理后残留的酶活性至少保持90%以上的酶活,而其他3种胰蛋白酶的剩余酶活均不足70%。进一步测定了重组胰蛋白酶的酶活和比酶活。如图4-D所示,经100 IU肠激酶激活后,HST、BTT、PVT和SFT上清液的酶活性分别为(18.6±1.1)、(12.3±1.7)、(44.7±0.5)、(13.8±2.7)BAPNA U/mL,而这4种胰蛋白酶的比酶活分别为37.2、23.3、43.3、12.3 U/mg。由此可见,SFT在相对较高的温度和碱性条件下具有较高的稳定性,然而其催化性质相对略低。因此,SFT在碱性工业条件下可能具有较好的应用价值,但仍需通过酶工程改造提升其催化性质。

A-最适温度;B-最适pH;C-pH稳定性;D-催化性质
图4 不同来源胰蛋白酶的酶学性质
Fig.4 Enzymatic properties of trypsin from different sources
3 结论
本研究通过基因挖掘从数据库中筛选得到了4种分别来源于Homo spanies、Bos taurus、Penaeus vannamei和S.fradiae ATCC14544的胰蛋白酶基因,并在K.phaffii GS115中成功实现了异源表达。通过对4株K.phaffii重组菌株的生长情况以及酶学性质比较,发现含有S.fradiae来源胰蛋白酶基因的重组K.phaffii具有较好的生长能力,且该酶具有较好的温度和碱性环境耐受能力。然而,由于该酶的催化性质低于其他来源胰蛋白酶,因此后续将进一步通过酶工程改造手段提高其催化活性。
[1] GUDMUNDSD TTIR
TTIR  , P
, P LSD
LSD TTIR H M.Atlantic cod trypsins:From basic research to practical applications[J].Marine Biotechnology, 2005, 7(2):77-88.
TTIR H M.Atlantic cod trypsins:From basic research to practical applications[J].Marine Biotechnology, 2005, 7(2):77-88.
[2] OLSEN J V, ONG S E, MANN M.Trypsin cleaves exclusively c-terminal to arginine and lysine residues[J].Molecular &Cellular Proteomics, 2004, 3(6):608-614.
[3] WANG H, LEI C, ZENG Y H, et al.Reversible inhibition of trypsin activity with soybean flour in hide bating process for leather quality improvement[J].Industrial Crops &Products, 2021, 161:113222.
[4] MUHLIA-ALMAZ N A, S
N A, S NCHEZ-PAZ A, GARC
NCHEZ-PAZ A, GARC A-CARRE
A-CARRE O F L.Invertebrate trypsins:A review[J].Journal of Comparative Physiology B, 2008, 178:655-672.
O F L.Invertebrate trypsins:A review[J].Journal of Comparative Physiology B, 2008, 178:655-672.
[5] KHAMBHATY Y.Applications of enzymes in leather processing[J].Environmental Chemistry Letters, 2020, 18:747-769.
[6] THOMAS A W, SEYMOUR-JONES F L.Action of trypsin upon diverse leathers[J].Industrial &Engineering Chemistry, 1924, 16(2):157-159.
[7] RASCHMANOV H, PAULOV
H, PAULOV L, BRANSK
L, BRANSK B, et al.Production and cleavage of a fusion protein of porcine trypsinogen and enhanced green fluorescent protein (EGFP) in Pichia pastoris[J].Folia Microbiologica, 2018, 63(6):773-787.
B, et al.Production and cleavage of a fusion protein of porcine trypsinogen and enhanced green fluorescent protein (EGFP) in Pichia pastoris[J].Folia Microbiologica, 2018, 63(6):773-787.
[8] ESPEJO-CARPIO F J, PÉREZ-G LVEZ R, GUADIX A, et al.Artificial neuronal networks (ANN) to model the hydrolysis of goat milk protein by subtilisin and trypsin[J].Journal of Dairy Research, 2018, 85(3):339-346.
LVEZ R, GUADIX A, et al.Artificial neuronal networks (ANN) to model the hydrolysis of goat milk protein by subtilisin and trypsin[J].Journal of Dairy Research, 2018, 85(3):339-346.
[9] KAMACI H O, SUZER C, ÇOBAN D, et al.Organogenesis of exocrine pancreas in sharpsnout sea bream (Diplodus puntazzo) larvae:Characterization of trypsin expression[J].Fish Physiology and Biochemistry, 2010, 36:993-1000.
[10] GONG J S, LI W, ZHANG D D,et al.Biochemical characterization of an arginine-specific alkaline trypsin from Bacillus licheniformis[J].International Journal of Molecular Sciences, 2015, 16(12):30061-30074.
[11] WANG Z K, LI X G, TIAN J W, et al.Cloning, heterologous expression and characterization of a novel streptomyces trypsin in Bacillus subtilis SCK6[J].International Journal of Biological Macromolecules, 2020, 147:890-897.
[12] TUBIO G, PIC G A, NERLI B B, et al.Extraction of trypsin from bovine pancreas by applying polyethyleneglycol/sodium citrate aqueous two-phase systems[J].Journal of Chromatography B, 2008, 877(3):115-120.
G A, NERLI B B, et al.Extraction of trypsin from bovine pancreas by applying polyethyleneglycol/sodium citrate aqueous two-phase systems[J].Journal of Chromatography B, 2008, 877(3):115-120.
[13] HANQUIER J, SORLET Y, DESPLANCQ D, et al.A single mutation in the activation site of bovine trypsinogen enhances its accumulation in the fermentation broth of the yeast Pichia pastoris[J].Applied and Environmental Microbiology, 2003, 69(2):1108-1113.
[14] YANG R X, JIN Q, FAN J G.Metabolic dysfunction-associated fatty liver disease:From basic research to clinical application[J].Chinese Medical Journal, 2022, 135(10):1138-1140.
[15] SRIKET P, SRIKET C.A trypsin from royal red prawn (Haliporoides sibogae) and its possible application for collagen hydrolysis[J].Separation Science and Technology, 2015, 50(7):1073-1082.
[16] GUERRERO-OLAZAR N M, CASTILLO-GALV
N M, CASTILLO-GALV N M, GALLEGOS-L
N M, GALLEGOS-L PEZ J A, et al.Biochemical characterization of recombinant Penaeus vannamei trypsinogen[J].Comparative Biochemistry and Physiology, Part B, 2019, 238:110337.
PEZ J A, et al.Biochemical characterization of recombinant Penaeus vannamei trypsinogen[J].Comparative Biochemistry and Physiology, Part B, 2019, 238:110337.
[17] KIM S K, PARK Y C, LEE H H, et al.Simple amino acid tags improve both expression and secretion of Candida antarctica lipase B in recombinant Escherichia coli[J].Biotechnology and Bioengineering, 2015, 112(2):346-355.
[18] YANG N, LING Z M, PENG L, et al.Improvement of enzymatic stability and catalytic efficiency of recombinant Fusariumoxysporum trypsin with different n-terminal residues produced by Pichia pastoris[J].Microbiology and Biotechnology, 2018, 28(9):1482-1492.
[19] SHU M, SHEN W, WANG X J, et al.Expression, activation and characterization of porcine trypsin in Pichia pastoris GS115[J].Protein Expression and Purification, 2015, 114:149-155.
[20] ATACAN K, ![]() B, ÖZACAR M.Improvement of the stability and activity of immobilized trypsin on modified Fe3O4 magnetic nanoparticles for hydrolysis of bovine serum albumin and its application in the bovine milk[J].Food Chemistry, 2016, 212:460-468.
B, ÖZACAR M.Improvement of the stability and activity of immobilized trypsin on modified Fe3O4 magnetic nanoparticles for hydrolysis of bovine serum albumin and its application in the bovine milk[J].Food Chemistry, 2016, 212:460-468.
[21] YANG Z L, ZHANG Z S.Engineering strategies for enhanced production of protein and bio-products in Pichia pastoris:A review[J].Biotechnology Advances, 2018, 36(1):182-195.
[22] BOECHI L, PIERCE L, KOMIVES E A, et al.Trypsinogen activation as observed in accelerated molecular dynamics simulations[J].Protein Science, 2014, 23(11):1550-1558.
[23] OHMURAYA M, YAMAMURA K I.Autophagy and acute pancreatitis:A novel autophagy theory for trypsinogen activation[J].Autophagy, 2008, 4(8):1060-1062.
[24] ZHANG Y F, HUANG H, YAO X H, et al.High-yield secretory production of stable, active trypsin through engineering of the N-terminal peptide and self-degradation sites in Pichia pastoris[J].Bioresource Technology, 2018, 247:81-87.