原油成本的攀升和不稳定、日益增长的环境问题,都促使人们对使用微生物生产生物燃料和生物化学制品的兴趣增加[1]。油化学品是石化产品的替代品,通常从植物油和动物脂肪中提取,其供应量有限[2]。微生物脂肪酸的生物合成已经引起了广泛的关注,因为它提供了一种可再生油化学品的生产方式[3]。游离脂肪酸(free fatty acids,FFAs),即不与其他分子结合的脂肪酸,由于其较低的生产和收获成本以及可以直接地化学转化为广泛的生物燃料和生化产品,包括烷烃、烯烃、脂肪酸甲基酯、脂肪酸乙基酯和脂肪醇,而被人们所需要[4]。
酿酒酵母作为模式真核微生物,基于成熟的遗传操作系统,成熟的表达系统和相对较好的工业条件耐受性,已经被成功开发为可用于多种生物化学品的细胞工厂,并且已经应用于工业生产[5]。野生型酿酒酵母本身具有合成脂肪酸的能力,但其生产效率很低,无法满足实际应用的需求。因而需要对其进行代谢改造,提高其FFAs的生产能力。近年来酿酒酵母已被设计用来生产FFAs,在野生型酿酒酵母内脂肪酸主要由细胞质I型FAS合成的,它是一个大型的、多功能的二聚体复合物,由基因FAS1以及FAS2编码,负责从丙二酰CoA和乙酰CoA合成脂肪酸。首先葡萄糖经糖酵解生成丙酮酸,然后在丙酮酸脱羧酶(pyruvate decarboxylase,PDC)的作用下生成乙醛,随后在乙醛脱氢酶(acetaldehyde dehydrogenase,ALD)的作用下生成乙酸,乙酸在乙酰CoA合成酶(acyl-CoA synthetase,ACS)的作用下生成乙酰CoA,接着在乙酰CoA羧化酶(acetyl CoA carboxylase,ACC1)的催化下生成丙二酰CoA,随后脂肪酸合成酶以乙酰CoA以及丙二酰CoA为前体合成脂肪酸(图1) [6]。
之前的研究表明,通过敲除酰基-CoA合成酶基因FAA1和FAA4,酵母开始分泌FFAs到生长介质中[7],LI等[8]通过破坏FAA1和FAA4能够生产大约80 mg/L的脂肪酸。RUNGUPHAN等[9]通过删除FAA1和FAA4以及过量表达大肠杆菌硫酯酶TesA,生产了207 mg/L的FFAs;CHEN等[10]通过去除FAA1和FAA4以及过量表达一种截短的S.cerevisiae过氧体酰基-CoA硫酯酶5(ACOT5),生产了大约520 mg/L FFAs。LEBER等[4]通过将Δfaa1Δfaa4基因型与过量表达TAGs的积累和水解途径相结合,生产了2.2 g/L的FFAs。冯叨等[11]在多形汉逊酵母中通过敲除FAA1并进行发酵优化实现了1.86 g/L的脂肪酸产量。YU等[12]通过对酿酒酵母代谢途径进行重构,消除了酿酒酵母生长过程中乙醇的产生,使碳流量更多流向脂肪酸生产,重组菌在发酵罐中实现了高达33.4 g/L的游离脂肪酸产量。本文利用CRISPR-Cas9技术连续敲除3个脂酰辅酶A合成酶编码基因、阻断β-氧化途径并上调脂肪酸合成关键酶编码基因ACC1、FAS1、FAS2的表达,酵母工程菌株FFAs的合成能力得到显著提高,为进一步开发工业水平发酵生产FFAs的酵母细胞工厂奠定来基础。
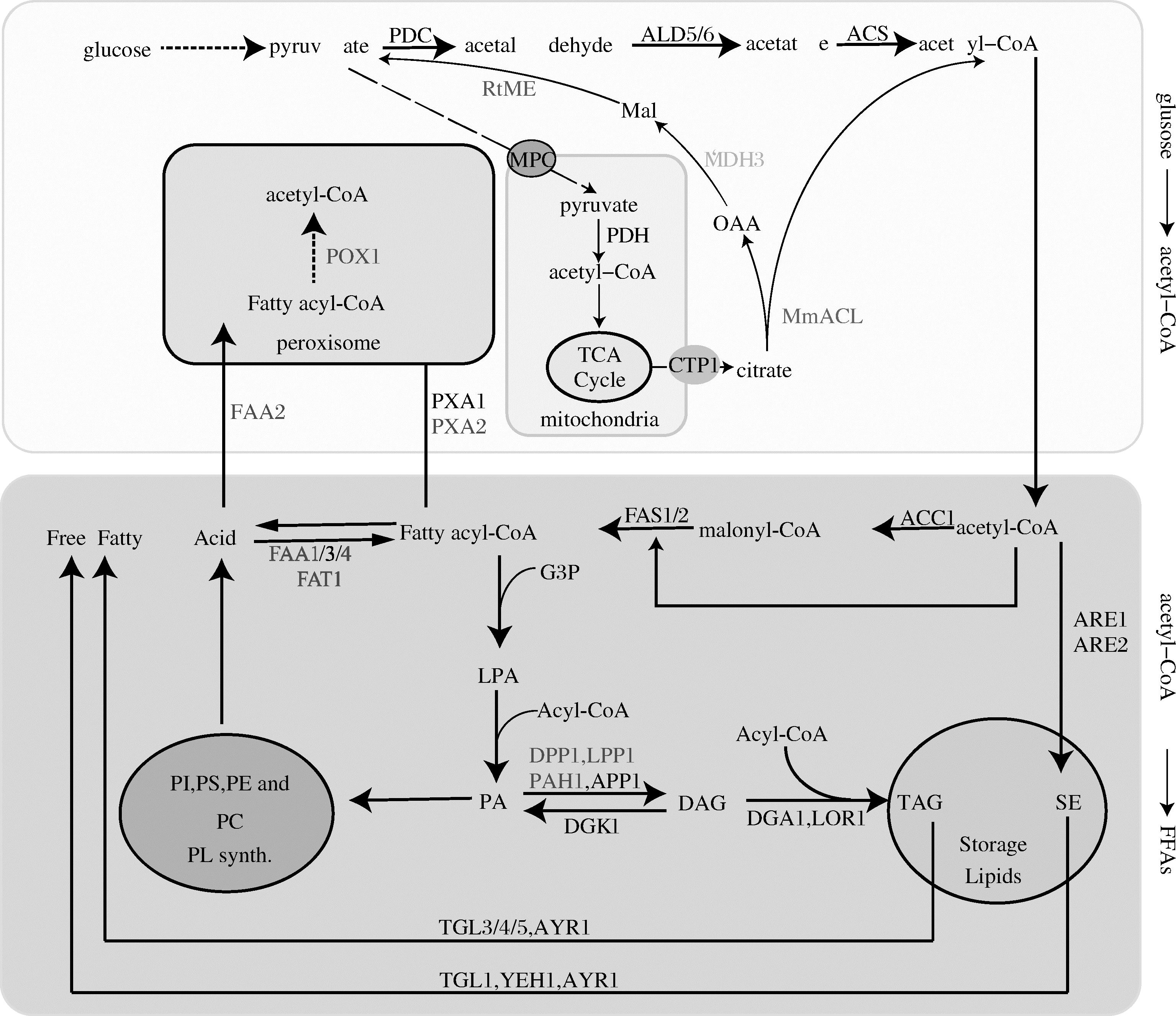
图1 脂肪酸的合成
Fig.1 Synthesis of fatty acids
注:Mal:苹果酸;OAA:草酰乙酸;TAG:三酰基甘油;SE:甾醇酯;DAG:二酰基甘油;LPA:溶血磷脂酸;G3P:三磷酸甘油醛;PA:磷脂酸; PI:磷脂酰肌醇;PS:磷酯酰丝氨酸;PE:磷脂酰乙醇胺;PC:磷脂酰胆碱;PL:磷脂
1 材料与方法
1.1 材料与试剂
质粒B18-pRS426,大肠杆菌菌株JM109,酿酒酵母菌株BY4741,均为本实验室保存;小鼠ATP:柠檬酸裂解酶基因MmACL,圆红冬孢酵母苹果酸酶基因RtME,合成于苏州金唯智生物科技有限公司;Ex Taq和 Prime STAR DNA聚合酶以及限制性核酸内切酶,Takara;重组酶C112、C113,南京诺唯赞生物科技股份有限公司;本研究所用引物(表1)均在苏州金唯智生物科技有限公司合成。
二氯甲烷(dichloromethane,DCM)、十五烷酸、正己烷,上海阿拉丁生化科技有限公司;碘甲烷,梯希爱上海化成工业发展有限公司;12%氯化氢甲醇溶液,上海阿达玛斯试剂有限公司,37种脂肪酸甲酯混标,美国sigma公司,四丁基氢氧化铵,上海麦克林生化股份有限公司。
YPD培养基(g/L):酵母粉10,蛋白胨20,葡萄糖20。
LB培养基(g/L):氯化钠10,蛋白胨10,酵母粉5。
MM培养基(g/L):酵母氮基6.7,硫酸铵10,葡萄糖20。
SM培养基(g/L):酵母氮基6.7,硫酸铵10,葡萄糖20,尿嘧啶0.06。
1.2 仪器与设备
AL104电子分析天平,瑞士梅特勒-托利多公司:HB-100恒温金属浴,杭州博日科技有限公司;TSQ8000三重四极杆气质联用仪,赛默飞世尔科技公司;4K-15台式高速冷冻离心机,美国Sigma公司;UV-2100紫外可见光分光光度计,上海优尼特有限公司;PH-070A干燥箱,杭州汇尔仪器设备有限公司。
1.3 实验方法
1.3.1 质粒构建
质粒构建的方法主要使用一步克隆的组装方法。以酿酒酵母BY4741基因组为模板,以引物UFAA1-F/UFAA1-R,DFAA1-F/DFAA1-R分别克隆基因FAA1上下游同源臂将其与载体pMD19T(simple)连接构建FAA1敲除质粒,以同样的方式构建FAA4,FAT1,POX1,FAA2,PXA2,DPP1,LPP1,PAH1敲除质粒;以质粒B18-pRS426为模板,以引物B18-F1(FAA1)/B18-R1,B18-F2/B18-R2,B18-F3/B18-R3(FAA1)分别扩增B18-1(FAA1),B18-2,B18-3(FAA1)3个片段用一步克隆的方法将其连接构建带有FAA1对应sgRNA的Cas9质粒,同理构建分别带有另外8个基因对应sgRNA的Cas9质粒。
以酿酒酵母BY4741基因组为模板克隆线粒体柠檬酸盐转运蛋白基因CTP1,以及截短的苹果酸脱氢酶基因MDH3′,将CTP1插入质粒PPH01的Not I和Bgl II位点之间获得质粒PPH02,将MDH3′插入质粒PPH02的BamH I和Xho I位点之间获得质粒PPH03;将MmACL插入质粒PTT01的BamH I和Xho I位点之间获得质粒PTT02,将RtME插入质粒PTT02的Not I和Sac I位点之间获得质粒PTT03;以质粒PPH03为模板tADH1-F/tTPI1-R为模板扩增片段,将片段插入PTT03的Xba I位点获得质粒PTTPH01。
1.3.2 菌株构建
本研究酿酒酵母的转化采用改进的LiAc转化法:挑取单菌落过夜培养,次日以OD600=0.1接种于50 mL在250 mL锥形瓶新鲜的YPD培养基中,培养至OD600=0.5~0.8,离心弃上清液,用无菌冰水清洗1次,100 mmol/L的LiAc清洗1次,将菌体重悬在400 μL 100 mmol/L的LiAc中,然后50 μL分装;对分装体系高速离心30 s弃去上清液,向菌体中依次加入240 μL 40% PEG3350,36 μL 1 mol/L LiAc,50 μL片段和质粒,25 μL的单链载体DNA,充分混匀后在30 ℃孵育30 min,42 ℃热激23 min,最后离心后用1 mL无菌水重悬,取50 ~100 μL涂布到MM固体平板上。本研究所涉及的菌株均列于表2中。
表1 本研究所使用的引物
Table 1 Primers used in this research

引物名称 碱基序列UFAA1-FcccgacaggactataaagatATGGTTGCTCAATATACCGTTCCUFAA1-RgcaacatATTTGTCATCATCATTAGGCTTTAATCDFAA1-FgatgatgacaaatATGTTGCTAACATTTGTGTTTATGCCDFAA1-RcacccgctgacgcgccctgaTTAAGACGAACTATAAACGGCGTCUFAA2-FcccgacaggactataaagatCAAGGATCTGGAAGCTGGTCAUFAA2-RcgtcataagtacATTTCGAAACAAGTTTGTTCTTATCCDFAA2-FttcgaaatGTACTTATGACGATTTGGAACACATTCDFAA2-FcacccgctgacgcgccctgaTATCAGGGCTCACTACATGGAAAAUFAA4-FcccgacaggactataaagatATGACCGAACAATATTCCGTTGCUFAA4-RttATCATCTCCTCGTATGTCATGGTTATDFAA4-FgacatacgaggagatgatAAATTAGAATCCATTTACAGATCAAATCCDFAA4-RcacccgctgacgcgccctgaTTAAGTGTTTTCTTTATAAACTCTTTCCACUFAT1-FcccgacaggactataaagatAGGAAAAGCTTGAAAGATGAAATTGUFAT1-RgcagcggtcaagacttcataTCTGAATAATAGCAGGAAAATCCTTGDFAT1-FTATGAAGTCTTGACCGCTGCTGDFAT1-RcacccgctgacgcgccctgaATCGGCTACTCCGGTGTAAAACUPOX1-FcccgacaggactataaagatATGACGAGACGTACTACTATTAATCCCGUPOX1-RtacttcttgCTCTCTCCTGCAACCAATACCGDPOX1-FgcaggagagagCAAGAAGTATTGATAGCGTTTCTAAGCDPOX1-RcacccgctgacgcgccctgaTCACTTGTTTATTTTCGATAAAATTTCCUPXA2-FcccgacaggactataaagatCATACATAGCCAAAGGAGGATCCAUPXA2-RgtctgCATGGAAGCTACGTAACGAAAAGGDPXA2-FtacgtagcttccatgCAGACGGAGTGGTCGTTACATGDPXA2-RcacccgctgacgcgccctgaTATAAAATCTTTCTTCGGTAACTTTTAATTGUDPP1-FcccgacaggactataaagatATGAACAGAGTTTCGTTTATTAAAACGUDPP1-RtaacccaaCATAAGGATGCGATATAGTGAGATCGDDPP1-FcgcatccttatgTTGGGTTATATAATGGCACACTTTTTDDPP1-RcacccgctgacgcgccctgaTTACATACCTTCATCGGACAAAGGULPP1-FcccgacaggactataaagatATGATCTCTGTCATGGCGGATGULPP1-RaataaagggcaccCGAGTTCGTTAGGTACATAACGTTTTDLPP1-FgaactcgGGTGCCCTTTATTAGCTCTAGTAGTAATDLPP1-RcacccgctgacgcgccctgaCTAAACACTAACCGGTGAAGGAAGTUPAH1-FcccgacaggactataaagatATGCAGTACGTAGGCAGAGCTCTUPAH1-RgttgtgatacgtcTACTGGGTATATGGATTTCAGTGAGTT
续表1

引物名称 碱基序列DPAH1-FcccagtaGACGTATCACAACAAAGCAACATTAGDPAH1-RcacccgctgacgcgccctgaTTAATCTTCGAATTCATCTTCGTCGB18-F1(FAA4)TAAGCTTTACAAGGCTGCCAGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTB18-R3(FAA4)TGGCAGCCTTGTAAAGCTTAGATCATTTATCTTTCACTGCGGAGAAGTTTCGB18-F1(PAH1)CTTACTGATTTATCTCCTCCGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTB18-R3(PAH1)GGAGGAGATAAATCAGTAAGGATCATTTATCTTTCACTGCGGAGAAGTTTCGB18-F1(POX1)AAAGTCGAAGGGTCTCTCAGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTB18-R3(POX1)CTGAGAGACCCTTCGACTTTGATCATTTATCTTTCACTGCGGAGAAGTTTCGB18-F1(PXA2)TCTCTATCCACAGTTCTACGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTB18-R3(PXA2)CGTAGAACTGTGGATAGAGAGATCATTTATCTTTCACTGCGGAGAAGTTTCGB18-F1(DPP1)ATTAGTGCAGCTCCTAACAGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTB18-R3(DPP1)CTGTTAGGAGCTGCACTAATGATCATTTATCTTTCACTGCGGAGAAGTTTCGB18-F1(LPP1)GGCTTAAAAAGCACTCCAAGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTB18-R3(LPP1)CTTGGAGTGCTTTTTAAGCCGATCATTTATCTTTCACTGCGGAGAAGTTTCGB18-F1(FAA2)TGGATAAATCGACTGTCCAGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTB18-R3(FAA2)CTGGACAGTCGATTTATCCAGATCATTTATCTTTCACTGCGGAGAAGTTTCGB18-F1(FAA1)GTTCAACTTGGTATTATACGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTB18-R3(FAA1)CGTATAATACCAAGTTGAACGATCATTTATCTTTCACTGCGGAGAAGTTTCGB18-F1(FAT1)TGCGCCATTCTATCTCACGGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTB18-R3(FAT1)CCGTGAGATAGAATGGCGCAGATCATTTATCTTTCACTGCGGAGAAGTTTCGB18-F2CGAAAACGAGCGCTTCCGAAAB18-R1TTTCGGAAGCGCTCGTTTTCGB18-R2ATGCTTCCATTGTCGAAAGTGCGB18-F3CGCACTTTCGACAATGGAAGCATMDH3′-FCGGGATCCATGGTCAAAGTCGCAATMDH3′-RCCGCTCGAGTCAAGAGTCTAGGATGAAACCTP-FATAAGAATGCGGCCGCATGTCCAGTAAAGCTACCAACTP-RCGAGCTCTCAGGCTAGCATAACTAAGAC
表2 本研究构建的质粒与菌株
Table 2 Plasmids and strains constructed in this research

名称特性来源质粒PPH01pHXT7-tTPI1-pPGK1-tADH1本研究构建PPH02pHXT7-CTP1-tTPI1-pPGK1-tADH1本研究构建PPH03pHXT7-CTP1-tTPI1-pPGK1-MDH3′-tADH1本研究构建PTT01pTEF1-tCYC1-pTDH3-tPDC1本研究构建PTT02pTEF1-tCYC1-pTDH3-MmACL-tPDC1本研究构建PTT03pTEF1-RtME-tCYC1-pTDH3-MmACL-tPDC1本研究构建PTTPH01pHXT7-CTP1-tTPI1-pPGK1-MDH3′-tADH1- pTEF1-RtME-tCYC1-pTDH3-MmACL-tPDC1本研究构建B18-pRS426Cas9-sgRNA[18]B18-FAA1Cas9-sgRNA(FAA1)本研究构建B18-FAA2Cas9-sgRNA(FAA2)本研究构建B18-FAA4Cas9-sgRNA(FAA4)本研究构建B18-FAT1Cas9-sgRNA(FAT1)本研究构建B18-POX1Cas9-sgRNA(POX1)本研究构建B18-PAH1Cas9-sgRNA(PAH1)本研究构建B18-DPP1Cas9-sgRNA(DPP1)本研究构建B18-LPP1Cas9-sgRNA(LPP1)本研究构建B18-PXA2Cas9-sgRNA(PXA2)本研究构建菌株BY4741MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0本研究构建BY1BY4741 faa1Δ本研究构建BY2BY4741 faa1Δfaa4Δ本研究构建BY3BY4741 faa1Δfaa4Δfat1Δ本研究构建BY4BY4741 faa1Δfaa4Δfat1Δpox1Δ本研究构建BY5BY4741 faa1Δfaa4Δfat1Δpox1Δfaa2Δ本研究构建BY6BY4741 faa1Δfaa4Δfat1Δpox1Δfaa2ΔΔpxa2Δ本研究构建BY7BY4741 faa1Δfaa4Δfat1Δpox1Δfaa2Δpxa2Δdpp1Δ本研究构建BY8BY4741 faa1Δfaa4Δfat1Δpox1Δfaa2Δpxa2Δdpp1Δlpp1Δ本研究构建BY9BY4741 faa1Δfaa4Δfat1Δpox1Δfaa2Δpxa2Δdpp1Δlpp1Δpah1Δ本研究构建BY10BY4741 faa1Δfaa4Δfat1Δpox1Δfaa2Δpxa2Δdpp1Δlpp1Δpah1Δ, XI-5::pHXT7-CTP1-tTPI1-pPGK1-MDH3′-tADH1- pTEF1-RtME-tCYC1-pTDH3-MmACL-tPDC1本研究构建
1.3.3 产物提取与检测
使用此前描述的方法提取游离脂肪酸并将其衍生为甲酯[13]。将培养72 h的培养物稀释2倍后取200 μL立即加入10 μL 40%四丁基氢氧化铵(碱性催化剂),然后加入200 μL二氯甲烷,其中含有200 mmol/L甲基碘作为甲基供体,含有 100 mg/L十五烷酸作为内标。通过使用涡流混合器将混合物在1 400 r/min下振荡30 min,然后以5 000×g离心以促进相分离。将160 μL二氯甲烷层转移到带有内衬管的GC小瓶中,蒸发4 h至干。将提取的甲酯重悬于160 μL正己烷中。
为了定量细胞内总脂肪酸,用去离子水洗涤沉淀两次。然后通过加入500 μL浓盐酸并在70 ℃下孵育1 h来进行细胞裂解。细胞裂解后,加入500 uL二氯甲烷其中含100 mg/L十五烷酸,涡旋振荡10 min提取脂肪酸,12 000 r/min离心10 min取400 μL下层二氯甲烷层,蒸干后加入500 μL 12%(体积分数)盐酸-甲醇,然后涡旋2 min。在62 ℃下孵育3 h后,将反应混合物冷却至室温,并通过用400 μL正己烷涡旋2 min来提取脂肪酸的甲酯衍生物。在10 000 r/min离心15 min后,收集顶部己烷层。
脂肪酸甲酯通过配备有TG-5 30 m×0.25 mm×0.25 μm色谱柱(ThermoFisher Scientific)和TSQ8000质谱仪(ThermoFisher Scientific)的气相色谱Trace1310(ThermoFisher Science)进行分析。GC程序如下:初始温度为40 ℃,保持2 min;以每分钟30 ℃的速度升至130 ℃,然后以每分钟10 ℃的速率升至280 ℃并保持3 min。入口、传质管线和离子源的温度分别保持在280、300、230 ℃。注入体积为1 μL。载气(氦气)的流速设置为1.0 mL/min,并在全扫描模式(50~650 m/z)下采集数据。使用Xcalibur软件进行最终定量。
2 结果与分析
2.1 构建酰基CoA合成酶缺失突变株
在野生型酿酒酵母中脂肪酸以三酰基甘油(triacylglyceride,TAG)和甾醇酯(sterol ester,SE)的结合态储存在脂滴(neutral lipids,LD)当中,或者以酰基CoA的形式参与细胞代谢。在酿酒酵母中酰基CoA合成酶主要由FAA1,FAA2,FAA3,FAA4,Fat1和Fat2 6个基因编码,其中长链酰基辅酶A合成酶FAA1发挥着主要作用[14]。有文献报道,删除FAA1和FAA4基因能够提高酵母细胞的游离脂肪酸合成能力[7]。本研究以BY4741作为底盘细胞,敲除FAA1和FAA4基因,考察细胞的生长和FFAs的合成。结果表明,突变株的生长速度有一定的下降,但最终的生物量与出发菌株基本一致(图2-a),而胞内的脂肪酸和胞外FFAs均有显著提升,胞外的FFAs浓度达到380.4 mg/L(图2-b)。进一步,累积删除负责长链脂肪酰CoA的合成和脂肪酸转运的Fat1基因,突变菌株BY3的生长和FFAs的合成与BY2突变株相比没有显著区别(图2)。

a-工程菌BY2、BY3和出发菌株BY4741的细胞生长比较; b-工程菌BY2、BY3和出发菌株BY4741的脂肪酸合成能力比较
图2 敲除酰基CoA合成酶的影响
Fig.2 Effect of removal of lipid acyl-CoA synthase
2.2 β-氧化途径的阻断提高FFAs合成
β-氧化是脂肪酸在过氧化物酶体中代谢分解生成乙酰CoA的过程。在酵母中,当脂肪酸不被用作碳源时,β-氧化活性对于酵母细胞的正常生长不是必须的。虽然先前的研究已经通过删除参与脂肪酸降解的初始酶POX1的基因来破坏酿酒酵母中的β-氧化,但由于其他同工酶的作用,这可能不足以完全去除β-氧化活性[15]。为了阻断工程菌株的β-氧化途径,本研究首先敲除了POX1基因获得菌株BY4,其生长状况并未受到显著影响(图3-a),同时FFAs产量提升到了390.1 mg/L。为了进一步提升FFAs的产量,本研究随后敲除了基因PXA2以及FAA2获得一株6重突变体菌株BY6,PXA2编码ABC转运蛋白复合物Pxa1-Pxa2的一个亚基,负责将长链脂肪酸导入过氧化物酶体,FFA2负责在过氧化物酶体中活化一些能够直接穿过过氧化物酶体膜的中链脂肪酸。BY6的生长状况并未受到基因PXA2以及FAA2敲除的影响,与此同时其FFAs产量仅有少量提升,达到了394.9 mg/L,但是其胞内脂肪酸产量得到了较大的提升,达到了622.1 mg/L(图3)。
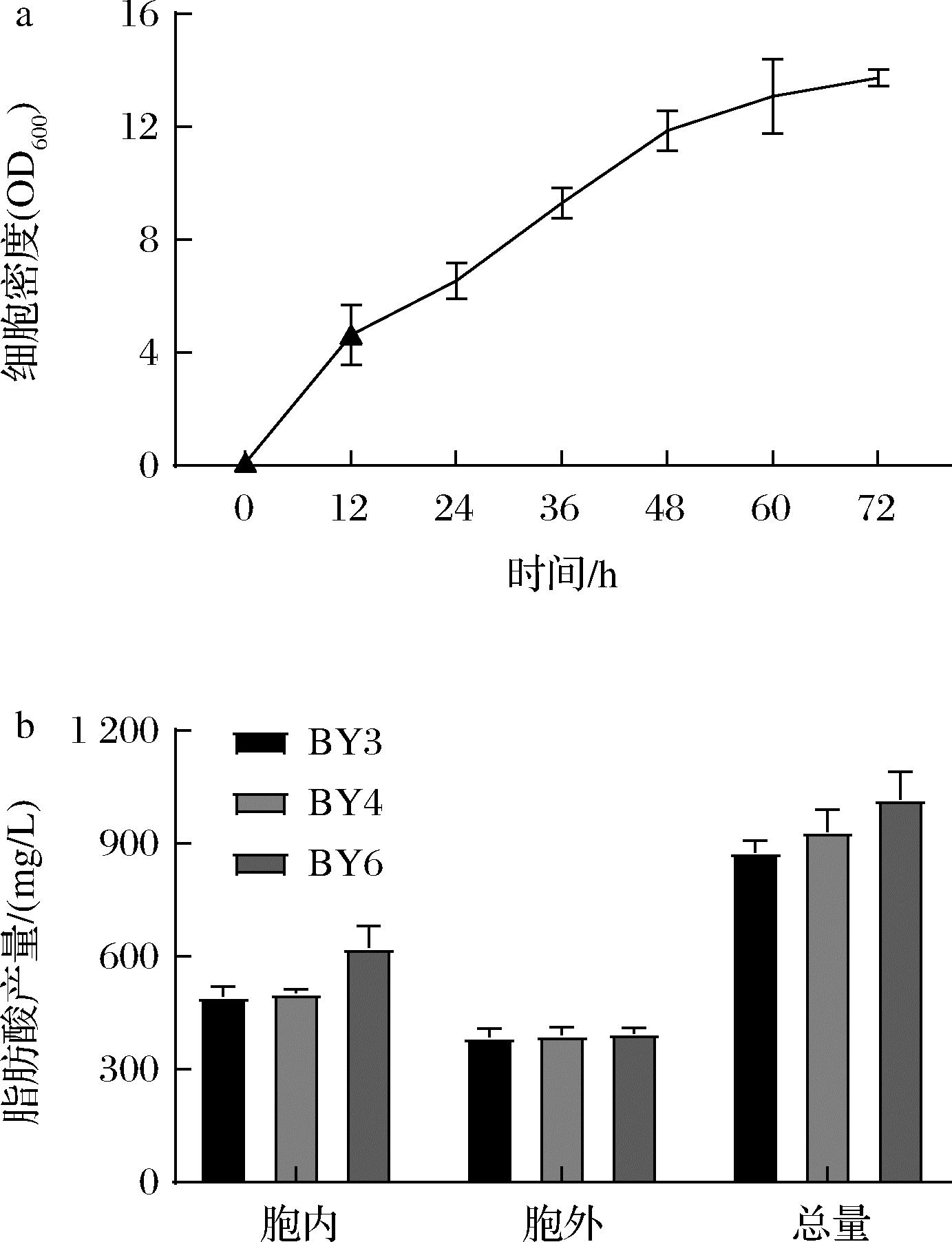
a-工程菌BY3、BY4、BY6细胞生长比较; b-工程菌BY3、BY4、BY6的脂肪酸产量比较
图3 β氧化阻断的影响
Fig.3 Effects of β-oxidative blockade
2.3 提高磷脂酸水平上调脂肪酸的合成
为了进一步提高脂肪酸的合成水平,我们希望能够上调酵母细胞内脂肪酸合成相关的基因的表达。PA被表征为调节脂质代谢的重要信号分子。高水平的PA导致转录调节因子Opi1向细胞核的易位减少[16],阻止其与转录因子Ino2的结合。由于Ino2是许多脂肪酸和磷脂生物合成基因的激活剂,PA水平的增加间接导致脂肪酸生物合成机制的上调[17]。为了提高PA的合成水平,我们旨在去除PA到DAG的去磷酸化,这将允许PA积累,从而干扰Opi1介导的调节(图4)。PA去磷酸化由主要由PAH1、LPP1和DPP1编码的磷脂酸磷酸酶催化,因此,这些基因从菌株中敲除,获得菌株BY9。BY9相对于BY6无论其生长速度还是最终的生物量都有所下降,但是BY9的 FFAs产量达到了497.3 mg/L 相对于BY6提升了26%,同时其胞内脂肪酸产量也有所上升,达到了833.9 mg/L(图5)。
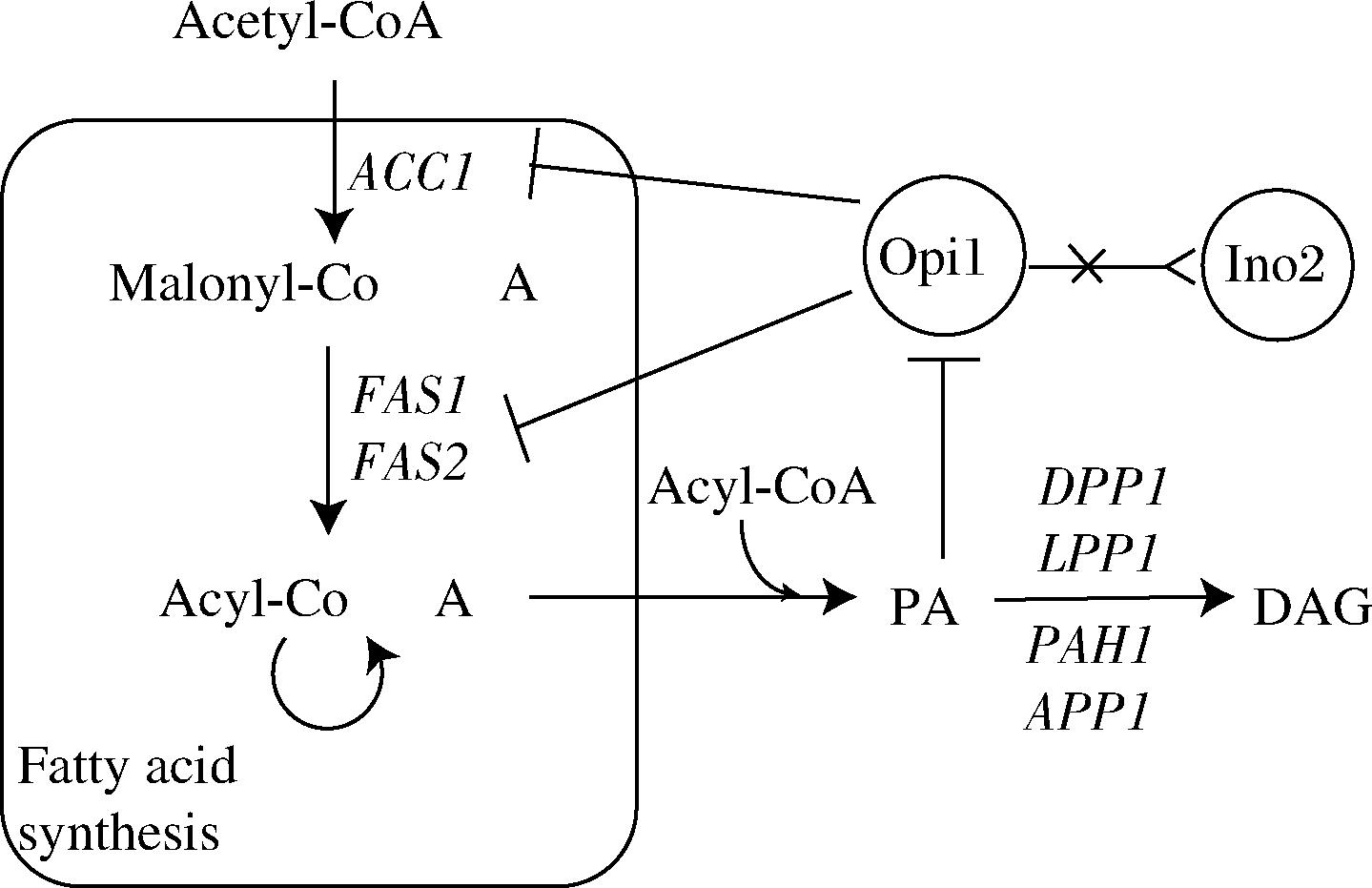
图4 PA对脂肪酸合成的调控
Fig.4 Regulation of fatty acid synthesis by PA

a-工程菌BY6、BY9细胞生长状况评价; b-工程菌BY6、BY9的脂肪酸产量比较
图5 PA上调对脂肪酸合成的影响
Fig.5 PA up-regulation on the yield of fatty acid synthesis
2.4 改造柠檬酸裂解酶途径提高乙酰CoA供应
有文献报道,通过改造柠檬酸裂解酶途径能够增加脂肪酸合成前体胞质乙酰CoA的供应(图1)[18],该途径已被认为在含油酵母的脂质积累中发挥重要作用[19]。本文在菌株BY9内构建了一个由来自小鼠(Mus musculus)的ATP:柠檬酸裂解酶(MmACL),来自圆红冬孢酵母(Rhodospuridium toruloides)的苹果酸酶RtME,去除了过氧化物酶体定位信号肽的内源性苹果酸脱氢酶Mdh3′以及柠檬酸盐转运蛋白Ctp1组成的柠檬酸裂解酶途径。Ctp1负责柠檬酸从线粒体到细胞质的运输;在MmACL作用下以ATP为辅因子胞质中的柠檬酸裂解形成草酰乙酸(oxalacetic acid,OAA)和乙酰CoA,乙酰CoA作为原料合成脂肪酸,而OAA接着参与柠檬酸裂解酶循环;OAA在Mdh3′的催化下形成苹果酸(Mal);RtME催化苹果酸形成丙酮酸,而丙酮酸又会进入到线粒体中参与TCA循环形成柠檬酸,这样我们构建了一个嵌合的柠檬酸裂解酶途径,增加了胞质乙酰CoA的供应。我们将这一途径的4个基因整合在基因组上获得菌株BY10。柠檬酸裂解酶途径的改造未对细胞的生长产生明显的影响(图6-a),工程菌株无论是胞内脂肪酸还是FAAs的产量均有所下降,胞内脂肪酸产量从833.9 mg/L下降到590.7 mg/L,FFAs从497.3 mg/L下降到396.2 mg/L(图6)。
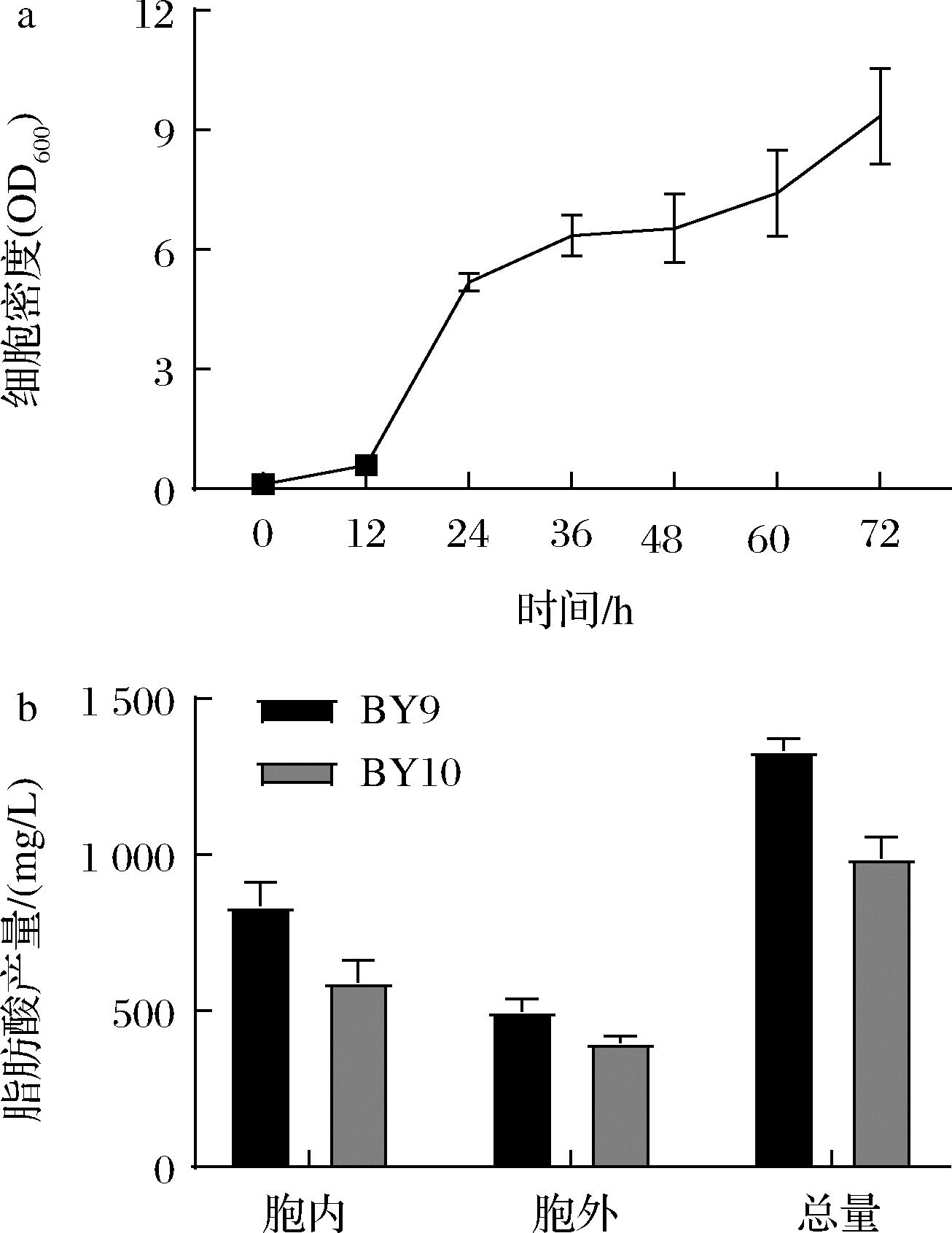
a-工程菌BY9、BY10细胞生长状况评价; b-工程菌BY9、BY10的脂肪酸合成能力评价
图6 柠檬酸裂解酶途径引入对脂肪酸合成的影响
Fig.6 Effect of citrate lyase pathway introduction on fatty acid synthesis
3 结论与讨论
本研究首先通过删除酿酒酵母BY4741中酰基CoA合成酶基因使细胞开始分泌游离脂肪酸到细胞外部,FFAs产量达到384.4 mg/L,而BY4741在胞外未检测到有FFAs;在阻断了β氧化活性后FFAs的滴度进一步提升达到了394.9 mg/L,同时其胞内的脂肪酸浓度也有所提升,从492.5 mg/L提升到了622.1 mg/L;之后DPP1,LPP1,PAH1基因的删除阻断了PA到DAG的去磷酸化,导致PA的积累从而上调了多个与脂肪酸合成相关的基因,FFAs的产量得到了进一步的提升达到了497.3 mg/L;而柠檬酸裂解酶途径的引入导致了FFAs和总脂肪酸产量的下降,这说明单纯的增加乙酰CoA的供应可能并不能提高脂肪酸的合成水平,反而增加了细胞的代谢负担,参与脂肪酸合成的后续步骤的编码基因如乙酰CoA羧化酶ACC1,以及脂肪酸合成酶基因FAS1,FAS2等的上调可能对脂肪酸合成有更重要的影响,这可能也是PA积累能够大幅提升脂肪酸合成能力原因。本研究通过全新的组合策略构建了一株生产FFAs的菌株,为后续进一步提高FFAs的产量或者生产脂肪酸衍生物奠定了基础。
[1] PERALTA-YAHYA P P, ZHANG F Z, DEL CARDAYRE S B, et al.Microbial engineering for the production of advanced biofuels[J].Nature, 2012, 488(7411):320-328.
[2] BIERMANN U, BORNSCHEUER U, MEIER M A R, et al.Oils and fats as renewable raw materials in chemistry[J].Angewandte Chemie International Edition, 2011, 50(17):3854-3871.
[3] LENNEN R M, PFLEGER B F.Microbial production of fatty acid-derived fuels and chemicals[J].Current Opinion in Biotechnology, 2013, 24(6):1044-1053.
[4] LEBER C, POLSON B, FERNANDEZ-MOYA R, et al.Overproduction and secretion of free fatty acids through disrupted neutral lipid recycle in Saccharomyces cerevisiae[J].Metabolic Engineering, 2015, 28:54-62.
[5] FLETCHER E, KRIVORUCHKO A, NIELSEN J.Industrial systems biology and its impact on synthetic biology of yeast cell factories[J].Biotechnology and Bioengineeing, 2016, 113(6):1164-1170.
[6] ZHOU Y J, BUIJS N A, SIEWERS V, et al.Fatty acid-derived biofuels and chemicals production in Saccharomyces cerevisiae[J].Frontiers in Bioengineering and Biotechnology, 2014, 2:32.
[7] SCHARNEWSKI M, PONGDONTRI P, MORA G, et al.Mutants of Saccharomyces cerevisiae deficient in acyl-CoA synthetases secrete fatty acids due to interrupted fatty acid recycling[J].The FEBS Journal, 2008, 275(11):2765-2778.
[8] LI X W, GUO D Y, CHENG Y B, et al.Overproduction of fatty acids in engineered Saccharomyces cerevisiae[J].Biotechnology and Bioengineeing, 2014, 111(9):1841-1852.
[9] RUNGUPHAN W, KEASLING J D.Metabolic engineering of Saccharomyces cerevisiae for production of fatty acid-derived biofuels and chemicals[J].Metabolic Engineering, 2014, 21:103-113.
[10] CHEN L W, ZHANG J H, LEE J, et al.Enhancement of free fatty acid production in Saccharomyces cerevisiae by control of fatty acyl-CoA metabolism[J].Applied Microbiology and Biotechnology, 2014, 98(15):6739- 6750.
[11] 冯叨, 高教琪, 龚志伟, 等.多形汉逊酵母代谢改造生产脂肪酸及发酵条件优化[J].生物工程学报,2022,38(2):760-771. FENG D, GAO J, GONG Z, et al.Production of fatty acids by engineered Ogataea polymorpha[J].Chinese Journal of Biotechnology, 2022, 38(2):760-771.
[12] YU T, ZHOU Y J, HUANG M T, et al.Reprogramming yeast metabolism from alcoholic fermentation to lipogenesis[J].Cell, 2018, 174(6):1549-1558.
[13] HAUSHALTER R W, KIM W, CHAVKIN T A, et al.Production of anteiso-branched fatty acids in Escherichia coli;next generation biofuels with improved cold-flow properties[J].Metabolic Engineering, 2014, 26:111-118.
[14] BLACK P N, DIRUSSO C C.Yeast acyl-CoA synthetases at the crossroads of fatty acid metabolism and regulation[J].Biochimica et Biophysica Acta, 2007, 1771(3):286-298.
[15] KOHLWEIN S D, VEENHUIS M, VAN DER KLEI I J.Lipid droplets and peroxisomes:Key players in cellular lipid homeostasis or a matter of fat-store ‘em up or burn’ em down[J].Genetics, 2013, 193(1):1-50.
[16] CHEN X X, YANG X Y, SHEN Y, et al.Increasing malonyl-CoA derived product through controlling the transcription regulators of phospholipid synthesis in Saccharomyces cerevisiae [J].ACS Synthetic Biology, 2017, 6(5):905-912.
[17] FERREIRA R, TEIXEIRA P G, SIEWERS V, et al.Redirection of lipid flux toward phospholipids in yeast increases fatty acid turnover and secretion[J].Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(6):1262-1267.
[18] ZHOU Y J, BUIJS N A, ZHU Z, et al.Production of fatty acid-derived oleochemicals and biofuels by synthetic yeast cell factories[J].Nature Communications, 2016, 7:11709.
[19] TANG X, FENG H, CHEN W N.Metabolic engineering for enhanced fatty acids synthesis in Saccharomyces cerevisiae[J].Metabolic Engineering, 2013, 16:95-102.