几丁质是自然界含量仅次于纤维素的大分子可再生生物质资源,年产量达到1011 t以上[1]。然而,几丁质具有高度的晶体结构且排列紧密,不溶于水、稀酸或稀碱溶液中,目前仍然是一种难以被开发利用的多糖化合物。与几丁质相比,壳聚糖具有较好的溶解度和生物活性。例如,壳聚糖可用来制备生物膜,应用于食品保鲜和医疗等方面[2]。一般来说,壳聚糖的生物活性主要取决于其理化参数,包括聚合度、脱乙酰度和乙酰化模式。
目前,壳聚糖通常采用化学法制备,即采用浓碱对几丁质进行脱乙酰作用。然而,化学法制备壳聚糖会产生大量的高碱度废水,而且获得的壳聚糖具有随机的乙酰化模式,影响其理化性质。采用几丁质脱乙酰酶(chitin deacetylase, CDA)也可以制备壳聚糖,具有环保的优点,并且生产的壳聚糖是非随机的乙酰化模式[3]。CDA主要在一些真菌中发现,如Saccharomyces cerevisiae[4]、Colletotrichum lindemuthianum[5]、Podospora anserina[6]、Coprinopsis cinerea[7]、Aspergillus nidulan[8]、Mucor rouxii[9]和Pestalotiopsis sp.[10]。迄今为止,已报道的CDA数量较少,且酶活性较低。这是因为,CDA在几丁质上的作用位点仅在N-乙酰基表面,而不是几丁质内部[11]。因此,探索新型高效的CDA,或开发其他应用技术以提高几丁质向壳聚糖的生物转化效率,具有重要的研究意义。
几丁质的高效降解需要多种酶的协同作用。来源于Vibrio harveyi的2种几丁质酶在降解几丁质方面表现出较好的协同作用[12]。裂解性多糖单加氧酶(lytic polysaccharide monooxygenase, LPMO)与几丁质酶协同作用,从而促进几丁质降解[13]。CDA与几丁质酶的协同作用也可能促进几丁质的水解。这是因为,几丁质经过脱乙酰基后,其水溶性提高,这样有利于几丁质的降解[14];当几丁质的糖苷键断裂时,结晶几丁质变得更加疏松,底物可及性增加,从而增强了CDA对几丁质的脱乙酰作用[15]。因此,与单独使用CDA或几丁质酶水解几丁质相比,CDA和几丁质酶的协同水解可能对几丁质的脱乙酰和降解更有效。此外,CDA和几丁质酶对几丁质的协同作用产生壳寡糖和几丁寡糖,其可能比几丁质酶产生的单一几丁寡糖具有更广泛的生物活性和应用。在本研究中,对来源于Rhizopus stolonifer的CDA (RsCDA1)进行了基因克隆、异源表达、酶学特性的研究。另外,还考察了RsCDA1与来源于V.harveyi的几丁质酶(VhChit2)对几丁质脱乙酰和降解的协同作用。
1 材料与方法
1.1 材料与试剂
几丁质和壳聚糖(脱乙酰度:90%),上海源叶生物科技有限公司;N-乙酰氨基葡萄糖GlcNAc和几丁寡糖(GlcNAc)2-6 ,青岛博智汇力生物科技有限公司;表达载体pPIC9K和Pichia pastoris GS115感受态细胞,北京华越洋生物科技有限公司;克隆宿主Escherichia coli DH5α和E.coli BL21(DE3),Novagen公司;限制性内切酶EcoR I、Not I、T4连接酶,ThermoFisher Scientific公司;乙酸检测试剂盒(K-ACET),爱尔兰Megazyme公司;VhChit2为本实验室制备,其对几丁质的活力约为17 U/mg。
MD培养基:3.4 g酵母氮源(无硫酸铵)、10 g硫酸铵、20 g葡萄糖、30 g琼脂粉、1 L蒸馏水,115 ℃灭菌30 min,再加入0.4 mg已过滤除菌的生物素。
BMMG培养基:3.4 g酵母氮源(无硫酸铵)、10 g硫酸铵、10 g酵母粉、20 g蛋白胨、100 mL磷酸钾缓冲液(1 mmol/L, pH 6.0)、10 g甘油、1 L蒸馏水,115 ℃灭菌30 min, 再加入0.4 mg过滤除菌的生物素。
BMMY培养基:3.4 g酵母氮源(无硫酸铵)、10 g硫酸铵、10 g酵母粉、20 g蛋白胨、100 mL磷酸钾缓冲液(1 mmol/L, pH 6.0)、1 L蒸馏水,115 ℃灭菌30 min,再加入0.4 mg过滤除菌的生物素。
胶体几丁质:5 g几丁质粉,加入100 mL浓HCl搅拌均匀, 4 ℃下静置1 d,再加入500 mL的50%预冷乙醇,4 ℃下静置1 d,离心(6 000×g,10 min),沉淀水洗至pH中性。
1.2 仪器与设备
2510电转化仪,德国Eppendorf公司;Ultraflextreme基质辅助激光解析电离串联飞行时间质谱仪,德国Bruker公司;isoprime precisION稳定同位素比质谱仪,德国Elementar公司。
1.3 实验方法
1.3.1 RsCDA1基因的克隆
从R.stolonifer的菌丝体中提取总RNA,然后再合成cDNA。从NCBI搜索RsCDA1的氨基酸序列(GenBank:RCI04983.1),设计引物RsCDA1-F(5′-CCGGAATTCCATGACAAAAAAGAAGCAAAACCAG-3′)和RsCDA1-R(5′-ATAAGAATGCGGCCGCCAGCAATAGCCAACCCGAG-3′)。将限制性内切酶(EcoRI和Not I)酶解后的PCR产物连接至pPIC9K载体并转化至E.coli DH5α。将重组质粒通过电转化(1 500 V,5 ms,2 mm电转化杯)至P.pastoris GS115中,再涂布于MD培养基中,30 ℃倒置培养2 d。
1.3.2 RsCDA1的诱导表达
将重组菌P.pastoris接种至50 mL的BMGY培养基中,28 ℃、250 rpm振荡培养至OD600值达到5.0。离心(3 000×g,5 min),将菌体转移至50 mL的BMMY培养基中,使BMMY培养基的初始OD600值达到2.0,28 ℃、250 r/min振荡培养7 d,每天补充0.5%~2.0%甲醇,对RsCDA1进行诱导表达。每天收集发酵液,4 ℃离心(8 000×g,15 min),上清液通过0.45 μmol/L滤膜过滤,使用组氨酸标签纯化试剂盒对RsCDA1进行纯化,并保存于50 mmol/L、pH 7.0的磷酸钠缓冲液中。采用福林酚法测定RsCDA1的蛋白浓度,采用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳分析RsCDA1的纯度。
1.3.3 RsCDA1的活力及酶学性质
1.3.3.1 CDA活力的测定
在800 μL的胶体几丁质悬浮液(20 mg/mL,pH 6.5)中,加入200 μL的酶液,反应体系在45 ℃下搅拌反应2 h,沸水中加热10 min使酶失活,离心(8 000×g,5 min)。使用乙酸检测试剂盒测定反应上清液中产生的乙酸含量。1单位CDA活力(U)定义为45 ℃、pH 6.5条件下1 h从胶体几丁质中释放1 μmol/L乙酸所需的酶量。
1.3.3.2 最适温度和最适pH
分别考察RsCDA1在30~70 ℃ 和pH 6~9(pH 6~8,50 mmol/L磷酸钠缓冲液;pH 8~9,50 mmol/L Tris-HCl)下的活力,从而确定RsCDA1的最适温度和最适pH。
1.3.3.3 热稳定性和pH稳定性
将RsCDA1分别置于30~50 ℃下保温0.5、1.0、1.5和2.0 h,测定残余活力。将RsCDA1置于pH 6~9 环境中,25 ℃下保温2 h,测定残余活力。
1.3.3.4 金属离子和底物对RsCDA1活力的影响
在酶反应体系中分别添加1 mmol/L的Ca2+(CaCl2)、Mn2+(MnCl2)、Mg2+(MgCl2)、Fe2+(FeSO4)、Zn2+(ZnSO4)、Ni2+(NiSO4)、Cu2+(CuCl2)、Ba2+(BaCl2)和Co2+(CoCl2)等,探究金属离子对RsCDA1活力的影响;分别以20 mg/mL的几丁质酚、虾壳粉、(GlcNAc)1-6代替胶体几丁质,测定RsCDA1的底物特异性。
1.3.4 脱乙酰模式分析
使用5 U/mL的RsCDA1对20 mg/mL的(GlcNAc)4进行酶解。45 ℃、pH 6.5下分别酶解0.5、2、6和24 h,然后沸水中加热10 min,离心(12 000×g,10 min),使用MALDI-TOF-MS分析RsCDA1酶解(GlcNAc)4的脱乙酰化产物。在酶解后的上清液中加入0.5 mg/mL的 NaCl,取1 μL酶解上清液和1 μL 2,5-二羟基苯甲酸溶液(15 mg/mL)先后点样于MALDI 板上进行检测[16]。
1.3.5 RsCDA1与VhChit2的协同作用
将RsCDA1与0.5 U/mL的VhChit2以摩尔浓度比10∶1的比例混合,混合酶或单酶分别对20 mg/mL几丁质粉进行水解,在50 ℃、pH 7.0(50 mmol/L磷酸钠缓冲液)下搅拌反应 2 h。采用3,5-二硝基水杨酸法测定产生的还原糖含量[17],使用乙酸检测试剂盒测定释放的乙酸含量。对几丁质酶的协同作用定义为联合使用RsCDA1与VhChit2产生的还原糖含量与单独使用VhChit2产生的还原糖含量的比值。对CDA的协同作用定义为联合使用RsCDA1与VhChit2产生的乙酸含量与单独使用RsCDA1产生的乙酸含量的比值。
分别考察了反应温度(40~55 ℃)、pH(6~8)、摩尔浓度比(RsCDA1∶VhChit2=1∶1、5∶1、10∶1、20∶1、40∶1)和反应时间(0.5、1.0、1.5、2.0、2.5、3.0 h)对RsCDA1与VhChit2协同作用的影响。
1.3.6 几丁质脱乙酰度的测定
采用元素分析法测定几丁质的脱乙酰度。通过稳定同位素比质谱仪测定几丁质样品的碳氮比(C/N),参照公式(1)计算几丁质样品的脱乙酰度[18]:
脱乙酰度![]()
(1)
2 结果与分析
2.1 RsCDA1的表达与纯化
如图1所示,使用不同添加量的甲醇对RsCDA1进行诱导,CDA活力在前5 d迅速升高,然后趋于稳定水平,但20 g/L甲醇诱导的RsCDA1在5 d后开始缓慢下降。RsCDA1诱导表达的最佳甲醇添加量是10 g/L,过量或少量的甲醇都会抑制该酶的表达(图1-a)。在甲醇质量浓度为10 g/L的条件下,诱导5 d的RsCDA1活力达到13.5 U/mL。通过Ni2+亲和层析,对RsCDA1发酵上清液进行纯化,结果如图1-b所示。纯化后的RsCDA1蛋白电泳上有单条带,分子质量约为35 kDa,与其理论预测值35.1 kDa较符合。RsCDA1的分子质量与此前报道的PaCDA[6]和NaCDA[19]的分子质量(35.5 kDa和36 kDa)较接近。RsCDA1纯酶液的比活力为5.2 U/mg。与PaCDA相比,RsCDA1能够从几丁质中脱除更多的乙酰基,说明RsCDA1具有更高的脱乙酰活力。据报道,CDA活性可以通过测定乙酸盐[6]、4-硝基苯胺[19]和3-甲基-2-苯并噻唑啉腙[20]的含量。其中,从几丁质中释放的乙酸盐含量是CDA活性的最佳反映。

泳道M-标准蛋白Marker;泳道1-RsCDA1发酵上清液; 泳道2-RsCDA1纯酶液 a-不同甲醇浓度和时间诱导的CDA活力;b-RsCDA1的SDS-PAGE图
图1 RsCDA1的表达和纯化
Fig.1 Expression and purification of RsCDA1
2.2 温度与pH对RsCDA1活力和稳定性的影响
RsCDA1在45 ℃时的活力最高,40 ℃和50 ℃时的活力分别为最高活力的78.7%和93.9%,在70 ℃下反应几乎无活性(图2-a)。RsCDA1的最适pH值为6.5,在pH值6.0~7.0时的相对活性超过80%。然而,当pH值为9.0时,RsCDA1的相对活性仅为58.2%(图2-b)。RsCDA1在30 ℃和35 ℃下的酶活性较稳定,保温2 h后仍分别保留了75.4%和65.9%的初始活力,而45 ℃和50 ℃下的酶稳定性较差,保温2 h后的残余活力分别为20.7%和4.1%(图2-c)。RsCDA1在pH 6.5~8.0具有良好的稳定性,此范围pH条件下25 ℃保温2 h的残余活力均保持在70%以上(图2-d)。很多CDAs在pH 7.0~8.0时表现出较高活性,在碱性条件下表现出较高的pH稳定性[21]。然而,以乙二醇几丁质为底物时,来源于C.lindemuthianum的ClCDA在pH 11.5~12.0时仍有较高活力[22],而来源于M.rouxii的MrCDA能够耐受pH 4.5的酸性条件[9]。

a-最适温度;b-最适pH;c-热稳定性;d-pH稳定性
图2 温度和pH对RsCDA1活力和稳定性的影响
Fig.2 Effect of temperatures and pH on the activity and stability of RsCDA1
2.3 金属离子与底物对RsCDA1活力的影响
如表1所示,1 mmol/L的Ca2+、Mn2+、Mg2+、Zn2+、Ba2+和Co2+显著提高了RsCDA1活力,相对活力分别提高到105.3%、109.8%、121.9%、114.0%、111.6%和118.2%(P<0.05);而1 mmol/L的Ni2+和Cu2+则显著抑制RsCDA1活力,相对活力分别下降到90.5%、37.1%和65.8%(P<0.05);Fe2+对RsCDA1活力无显著影响(P>0.05)。有研究表明,部分CDAs如ClCDA和AnCDA,需要Co2+的参与才表现出活性[7, 15]。与胶体几丁质相比,RsCDA1对几丁质粉的相对活力仅为25.6%。此外,该酶不能水解虾壳粉释放乙酸,对底物GlcNAc也没有活力。随着底物几丁寡糖聚合度(n=1~6)的增加,RsCDA1的活力增强。以(GlcNAc)6为底物时,RsCDA1的相对活力达到159.1%。这可能是因为RsCDA1更倾向于去除中间乙酰基而不是末端乙酰基[23]。TOKUYASU等[22]也报道,利用聚合度高的几丁质低聚物作为底物时,可以增强ClCDA的活性。
2.4 RsCDA1的脱乙酰模式
研究RsCDA1对(GlcNAc)4的脱乙酰化产物,揭示其脱乙酰模式,如图3所示。在反应开始后的0.5 h,从一些(GlcNAc)4(A4)中去除一个乙酰基,形成部分脱乙酰化产物(A3D1)。随着反应时间的延长,逐渐产生脱乙酰度较高的A2D2、A1D3、D4。因此,RsCDA1在有足够的酶量和反应时间的前提下,水解几丁寡糖使其完全脱乙酰。有研究表明,使用0.15 μg/μL来源于C.cinerea的CDA3水解2.5 mmol/L的(GlcNAc)4时,无法检测到完全脱乙酰的产物D4,而当酶量增加到0.45 μg/μL时,可以检测到完全脱乙酰的产物D4。此外,CDA3去除几丁质寡糖末端的最后一个乙酰基并产生完全去乙酰化产物需要较长的时间,且随着底物几丁寡糖聚合度的增加,完全去乙酰化产物的形成变慢[24]。RsCDA1对几丁寡糖的完全去乙酰化模式与AnCDA相似[15]。
表1 金属离子与底物对RsCDA1活力的影响
Table 1 Effect of metal ions and substrates on the activity of RsCDA1
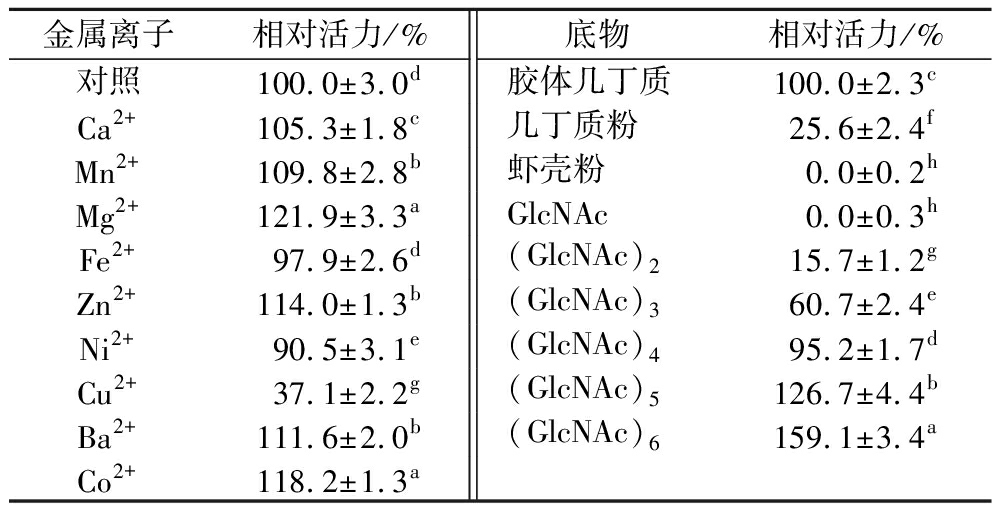
金属离子相对活力/%底物相对活力/%对照100.0±3.0d胶体几丁质100.0±2.3cCa2+105.3±1.8c几丁质粉25.6±2.4fMn2+109.8±2.8b虾壳粉0.0±0.2hMg2+121.9±3.3aGlcNAc0.0±0.3hFe2+97.9±2.6d(GlcNAc)215.7±1.2gZn2+114.0±1.3b(GlcNAc)360.7±2.4eNi2+90.5±3.1e(GlcNAc)495.2±1.7dCu2+37.1±2.2g(GlcNAc)5126.7±4.4bBa2+111.6±2.0b(GlcNAc)6159.1±3.4aCo2+118.2±1.3a
注:同一列中,上标字母完全不同的数值之间有显著性差异(P<0.05)。
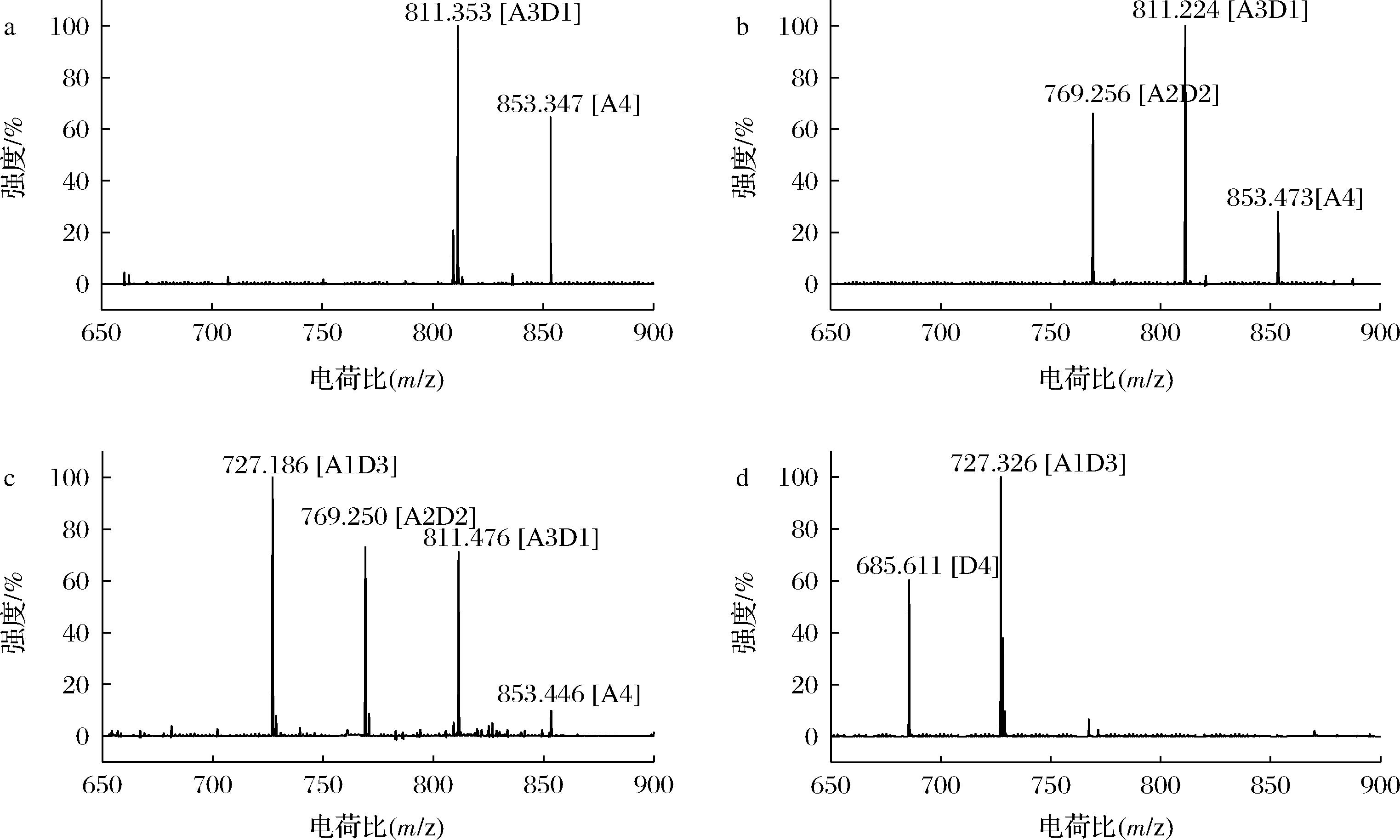
a- 水解0.5 h;b- 水解2 h;c- 水解6 h;d- 水解24 h
图3 RsCDA1水解产物的MALDI-TOF-MS分析
Fig.3 MALDI-TOF-MS analysis of hydrolysis products by RsCDA1
注:A4、A3D1、A2D2、A1D3和D4分别表示乙酰基为4、3、2、1和0的四糖。
2.5 RsCDA1与VhChit2对几丁质的协同水解作用
为了提高VhChit2与RsCDA1对几丁质的协同作用,研究了反应温度、pH、摩尔比和反应时间对协同度的影响,结果如图4所示。RsCDA1在45 ℃、pH 6.5条件下的CDA活力最高(图2);在前期研究中发现,VhChit2在50 ℃、pH 7.0条件下的几丁质酶活力最高[12],RsCDA1与VhChit2最适反应条件的微小差异为它们协同水解几丁质提供了可能。结果表明,RsCDA1与VhChit2协同水解几丁质的最适温度是50 ℃(图4-a),最适pH值是6.5(图4-b)。RsCDA1与VhChit2的摩尔浓度比为10∶1时,几丁质释放出来的还原糖和乙酸含量较高(图4-c)。随着酶解时间的延长,两酶联合使用时对几丁质酶和CDA的协同度均呈现先上升后下降的趋势,最佳的酶解时间是2 h(图4-d)。因此,当联合使用RsCDA1与VhChit2(摩尔浓度比为10∶1)水解几丁质时,在50 ℃、pH 6.5条件下搅拌反应2 h时,对几丁质酶和CDA的协同度分别达到120.3%和175.6%。RsCDA1与VhChit2对几丁质的降解和脱乙酰具有协同作用。

a-不同反应温度的协同度;b-不同反应pH的协同度;c-不同摩尔浓度比的协同度;d-不同反应时间的协同度
图4 反应温度、pH、摩尔浓度比(RsCDA1:VhChit2)和反应时间对RsCDA1与VhChit2协同度的影响
Fig.4 Effect of reaction temperature, pH, molar concentration ratio (RsCDA1:VhChit2), and time on the degree of synergy of RsCDA1 and VhChit2
注:同一指标中字母完全不同的数值之间有显著性差异(P<0.05)。
2.6 RsCDA1与VhChit2协同水解几丁质的脱乙酰度
分别使用VhChit2、RsCDA1及其组合水解几丁质粉,几丁质的脱乙酰度如图5所示。单独使用VhChit2水解,几丁质的脱乙酰度仅为7.2%,而单独使用RsCDA1及联合使用RsCDA1与VhChit2,几丁质的脱乙酰度得到显著提高,分别达到27.5%和35.3%。一般来说,较高的脱乙酰度可以提高几丁质的水溶解性,有助于CHI对几丁质的进一步降解[14]。因此,RsCDA1水解几丁质后脱乙酰度的增加可能是RsCDA1与VhChit2协同降解几丁质的原因之一。
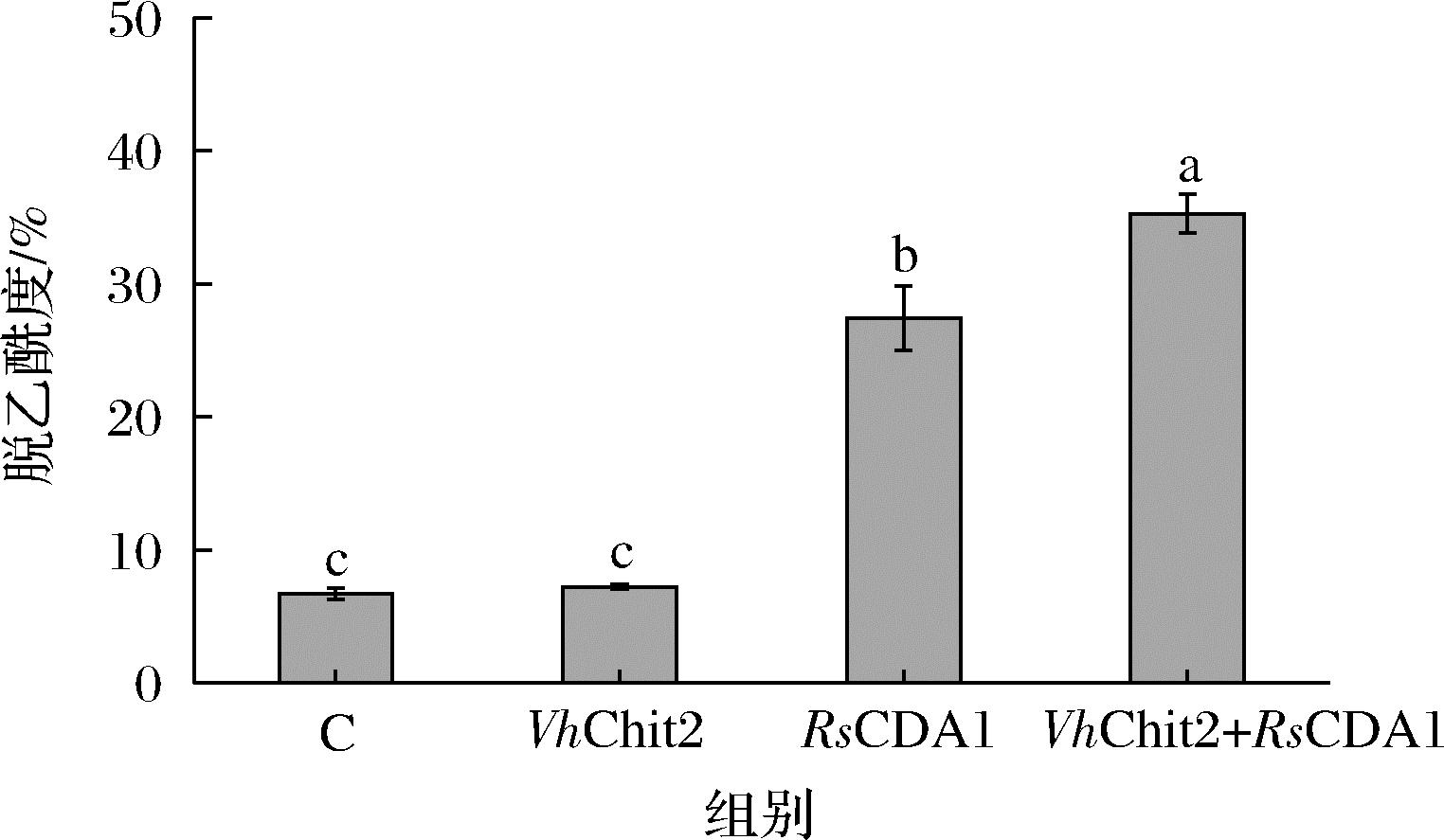
图5 几丁质经VhChit2、RsCDA1及其组合水解后的脱乙酰度
Fig.5 Degree of deacetylation of chitin hydrolyzed by VhChit2, RsCDA1 and their combination
注:C表示几丁质未经酶解。
3 结论
本研究报道了一种来源于R.stolonifer的CDA(RsCDA1),酶活力达到5.2 U/mg。RsCDA1在45 ℃ 和pH 6.5的条件下表现出最高活性。在温度低于40 ℃、pH 6.5~8.0范围内保持相对稳定。1 mmol/L Ca2+、Mn2+、Mg2+、Zn2+、Ba2+和Co2+可以提高RsCDA1的活性,但1 mmol/L的Ni2+和Cu2+对RsCDA1活性有抑制作用。底物特异性结果表明,RsCDA1对(GlcNAc)4-6的脱乙酰活性高于(GlcNAc)1-3。RsCDA1对几丁四糖表现出完全脱乙酰模式。
此外,RsCDA1与几丁质酶VhChit2在几丁质的降解和脱乙酰化方面表现出良好的协同作用。经优化后,两酶协同水解时对几丁质酶和CDA的协同度分别达到120.3%和175.6%。与单独使用RsCDA1水解几丁质(27.5%)相比,RsCDA1与VhChit2的联合使用显著增加了几丁质的脱乙酰度(35.3%)。RsCDA1与VhChit2对几丁质降解的协同作用可能是由于RsCDA1对几丁质的脱乙酰作用。综述所述,这种CDA具有将几丁质绿色、高效转化为壳聚糖的潜力。
[1] MINCEA M, NEGRULESCU A, OSTAFE V.Preparation, modification, and applications of chitin nanowhiskers:A review[J].Reviews on Advanced Materials Science, 2012, 30:225-242.
[2] TOKATLI K, DEMIRDÖVEN A.Effects of chitosan edible film coatings on the physicochemical and microbiological qualities of sweet cherry (Prunus avium L.)[J].Scientia Horticulturae, 2020, 259:108656.
[3] TSIGOS I, MARTINOU A, KAFETZOPOULOS D, et al.Chitin deacetylases:New, versatile tools in biotechnology[J].Trends in Biotechnology, 2000, 18(7):305-312.
[4] MARTINOU A, KOUTSIOULIS D, BOURIOTIS V.Expression, purification, and characterization of a cobalt-activated chitin deacetylase (Cda2p) from Saccharomyces cerevisiae[J].Protein Expression and Purification, 2002, 24(1):111-116.
[5] BLAIR D E, HEKMAT O, SCHÜTTELKOPF A W, et al.Structure and mechanism of chitin deacetylase from the fungal pathogen Colletotrichum lindemuthianum[J].Biochemistry, 2006, 45(31):9416-9426.
[6] HOßBACH J, BUßWINKEL F, KRANZ A, et al.A chitin deacetylase of Podospora anserina has two functional chitin binding domains and a unique mode of action[J].Carbohydrate Polymers, 2018, 183:1-10.
[7] WANG Y X, NIU X, GUO X L, et al.Heterologous expression, characterization and possible functions of the chitin deacetylases, Cda1 and Cda2, from mushroom Coprinopsis cinerea[J].Glycobiology, 2018, 28(5):318-332.
[8] ALFONSO C, NUERO O M, SANTAMAR A F, et al.Purification of a heat-stable chitin deacetylase from Aspergillus nidulans and its role in cell wall degradation[J].Current Microbiology, 1995, 30(1):49-54.
A F, et al.Purification of a heat-stable chitin deacetylase from Aspergillus nidulans and its role in cell wall degradation[J].Current Microbiology, 1995, 30(1):49-54.
[9] KAFETZOPOULOS D, MARTINOU A, BOURIOTIS V.Bioconversion of chitin to chitosan:Purification and characterization of chitin deacetylase from Mucor rouxii[J].Proceedings of the National Academy of Sciences of the United States of America, 1993, 90(7):2564-2568.
[10] CORD-LANDWEHR S, MELCHER R L J, KOLKENBROCK S, et al.A chitin deacetylase from the endophytic fungus Pestalotiopsis sp.efficiently inactivates the elicitor activity of chitin oligomers in rice cells[J].Scientific Reports, 2016, 6:38018.
[11] AYE K N, KARUPPUSWAMY R, AHAMED T, et al.Peripheral enzymatic deacetylation of chitin and reprecipitated chitin particles[J].Bioresource Technology, 2006, 97(4):577-582.
[12] ZHANG Q, ZHANG X Y, HE Y C, et al.The synergistic action of two chitinases from Vibrio harveyi on chitin degradation[J].Carbohydrate Polymers, 2023, 307:120640.
[13] MA L, LIU Z Y, KONG Z J, et al.Functional characterization of a novel copper-dependent lytic polysaccharide monooxygenase TgAA11 from Trichoderma guizhouense NJAU 4742 in the oxidative degradation of chitin[J].Carbohydrate Polymers, 2021, 258:117708.
[14] NGO T H D, NGO D N.Effects of low-frequency ultrasound on heterogenous deacetylation of chitin[J].International Journal of Biological Macromolecules, 2017, 104:1604-1610.
[15] LIU Z L, GAY L M, TUVENG T R, et al.Structure and function of a broad-specificity chitin deacetylase from Aspergillus nidulans FGSC A4[J].Scientific Reports, 2017, 7:1746.
[16] QIN Z, CHEN Q M, LIN S, et al.Expression and characterization of a novel cold-adapted chitosanase suitable for chitooligosaccharides controllable preparation[J].Food Chemistry, 2018, 253:139-147.
[17] YANG S Q, FU X, YAN Q J, et al.Cloning, expression, purification and application of a novel chitinase from a thermophilic marine bacterium Paenibacillus barengoltzii[J].Food Chemistry, 2016, 192:1041-1048.
[18] V ZQUEZ J A, RAMOS P, MIR
ZQUEZ J A, RAMOS P, MIR N J, et al.Production of chitin from Penaeus vannamei by-products to pilot plant scale using a combination of enzymatic and chemical processes and subsequent optimization of the chemical production of chitosan by response surface methodology[J].Marine Drugs, 2017, 15(6):180.
N J, et al.Production of chitin from Penaeus vannamei by-products to pilot plant scale using a combination of enzymatic and chemical processes and subsequent optimization of the chemical production of chitosan by response surface methodology[J].Marine Drugs, 2017, 15(6):180.
[19] CHAI J L, HANG J H, ZHANG C G, et al.Purification and characterization of chitin deacetylase active on insoluble chitin from Nitratireductor aquimarinus MCDA3-3[J].International Journal of Biological Macromolecules, 2020, 152:922-929.
[20] BHAT P, PAWASKAR G M, RAVAL R, et al.Expression of Bacillus licheniformis chitin deacetylase in E.coli pLysS:Sustainable production, purification and characterisation[J].International Journal of Biological Macromolecules, 2019, 131:1008-1013.
[21] ZHAO Y, PARK R, MUZZARELLI R.Chitin deacetylases:Properties and applications[J].Marine Drugs, 2010, 8:24-46.
[22] TOKUYASU K, OHNISHI-KAMEYAMA M, HAYASHI K.Purification and characterization of extracellular chitin deacetylase from Colletotrichum lindemuthianum[J].Bioscience Biotechnology and Biochemistry, 1996, 60(10):1598-1603.
[23] HEKMAT O, TOKUYASU K, WITHERS S G.Subsite structure of the endo-type chitin deacetylase from a deuteromycete, Colletotrichum lindemuthianum:An investigation using steady-state kinetic analysis and MS[J].The Biochemical Journal, 2003, 374:369-380.
[24] BAI Y, WANG Y X, LIU X, et al.Heterologous expression and characterization of a novel chitin deacetylase, CDA3, from the mushroom Coprinopsis cinerea[J].International Journal of Biological Macromolecules, 2020, 150:536-545.