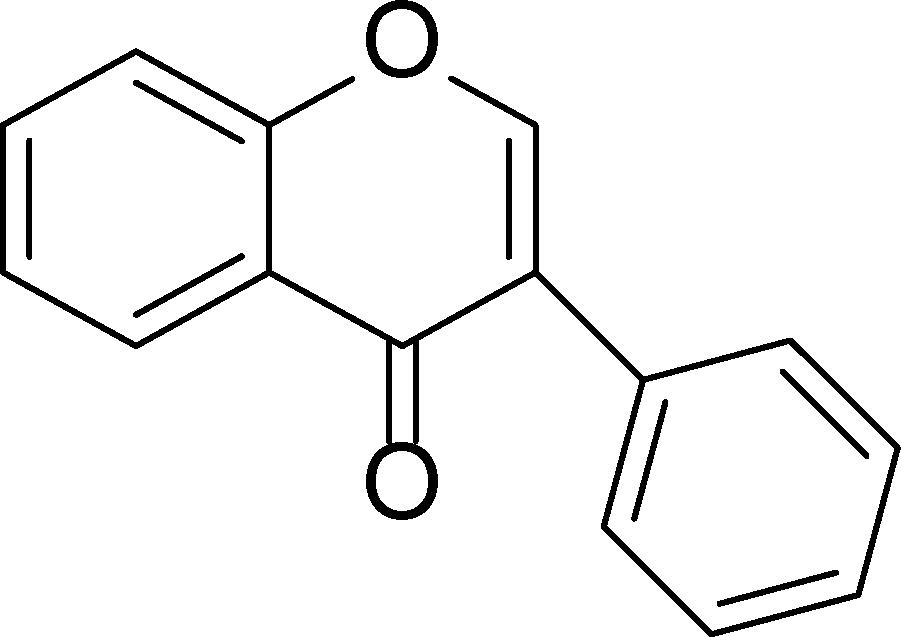
异黄酮属于类黄酮属,由肉桂酸辅酶A等酶合成的酚类化合物,主要活跃于豆科植物中,其中尤以大豆含量最多。根据其结构的不同,可以分为四类,12种。其中3种游离型化合物是异黄酮发挥作用的主要成分。20世纪,有研究学者[1]首次发现异黄酮与哺乳动物雌激素结构相似,并推测它们可能有防癌作用,从而使得异黄酮受到了广大学者的关注。也因为它能和雌激素受体结合,被称为 “植物雌激素”。除此以外,异黄酮还能够预防女性绝经后的骨质疏松症,减少脂质的积累,缓解更年期综合征以及某些激素依赖性癌症,如乳腺癌和前列腺癌等。
异黄酮的药理活性在预防疾病方面具有良好的功效,但是在植物中的含量较少,即使在含量较高的大豆中也仅为1%~3%。因此提高植物体内异黄酮的含量和体外合成异黄酮能够有效的改善异黄酮含量低的现状。基于此,本文总结了目前异黄酮合成途径以及药理活性,重点从生物合成途径以及关键酶和转录因子展开综述,以期为深入研究异黄酮的生物合成机制和植物育种提供参考。
1 异黄酮的结构和存在形式
目前在异黄酮中分离出12种化合物,分为3种游离型苷元和9种结合性苷元,而结合性的苷元是在游离型的苷元的基础上经过酸水解或者β-葡萄糖苷酶催化分解而得到。因此异黄酮主要是以葡萄糖苷的形式存在,其含量占到总异黄酮的97%~98%。异黄酮不同苷元的结构式见附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.034264)。
2 异黄酮的功能研究
异黄酮被证明是有益于人体健康的物质,在抗肿瘤、抗氧化、预防骨质疏松、缓解更年期综合症等方面有着显著的功效。由于异黄酮的结构与β-雌二醇相似,可发挥弱雌激素作用,用于治疗由雌激素缺乏导致的血脂升高[2];也能够与成骨细胞内的受体结合,促进骨质的活性,提高骨密度,预防骨质疏松症[3]。同时,作为类黄酮的一种,它有着黄酮类物质抗氧化的特点,能在抗肿瘤,清除自由基反应等方面发挥作用。以往研究证明染料木素在生理条件下是一种很有效的自由基清除剂[4],可减轻自由基带来的氧化还原反应对机体DNA的损害[5],在保护肝脏防治细胞氧化应激损伤以及改善心血管疾病方面也具有重要作用。因此异黄酮的抗氧化能力可以为机体提供一定的保护。此外,研究也证明异黄酮能够通过调节色氨酸的代谢通路从而缓解由谷氨酸引起的抑郁症状[6]。具体功能见附表2(https://doi.org/10.13995/j.cnki.11-1802/ts.034264)。
3 异黄酮的生物合成
3.1 异黄酮的合成路径
异黄酮的合成过程由苯丙氨酸途径和异黄酮合成途径一起完成。苯丙氨酸代谢途径是异黄酮合成的前提,其中3种主要的异黄酮化合物直接由苯丙氨酸途径合成的,其他异黄酮化合物多数以葡萄糖苷-丙二酰-葡萄糖缀合物的形式贮存于液泡中。具体合成途径为:苯丙氨酸(phenylalanine)作为最初底物,在苯丙氨酸解氨酶(phenylalanine ammonia-lyase,PAL)的作用下生成肉桂酸(cinnamic acid),之后由一种P450 单加氧酶-肉桂酸-4-羟化酶 (cinnamate-4-hydroxylase,C4H)催化形成P-香豆酸,经4-香豆酰-CoA连接酶(4-coumaryl:coa ligase,4CL)连接一个辅酶A进而形成P-香豆酰-CoA。此后一部分的P-香豆酰-CoA在查尔酮合酶 (chalcone synthase,CHS) 的作用下与3 分子的苹果酰 CoA 发生缩合反应, 形成C6-C3-C6的骨架结构。再经过查尔酮还原酶 (chalcone reductase,CHR)催化生成异甘草素(isoliquiritigenin),在此条件下一部分的异甘草素经过查尔酮异构酶(chalcone isomerase,CHI)催化又可生成甘草素(glycyrrhizin),之后经过一系列反应可以生成大豆黄素(daidzein);保留部分的异甘草素经过异黄酮合酶 (isoflavone synthase,IFS)可生成黄豆黄素(glyzein)。另一部分的P-香豆酰-CoA经过CHI能够生成柚皮素。柚皮素是黄酮类和异黄酮类物质合成途径中的共用底物,因此在此步骤中柚皮素的流向将成为影响异黄酮合成的关键因素。之后在CHI和异黄酮合酶(IFS)的共同作用下可催化生成染料木苷(genistein)。
异黄酮合成代谢途径的参与基因数量众多,调控机制复杂,现在公认的异黄酮合成途径是由黄酮类物质合成的前体苯丙氨酸和丙二酰辅酶A经过PAL、C4H、4CL、CHS、CHR、CHI等多种酶的催化、羟基化等过程[9]。具体过程见图1[10]。

图1 异黄酮合成代谢途径及主要化合物
Fig.1 Isoflavone synthesis metabolic pathway and main compounds
3.2 异黄酮合成过程中的关键酶
研究发现,异黄酮的积累是通过复杂的遗传互作网络进行的[11]。在异黄酮合成的每个阶段中都有大量的酶参与,而且酶与酶之间往往是通过相互影响、相互作用参与到合成途径中。例如有研究证明异黄酮的积累由CHS8和IFS2共同调控[12],且与植物的发育阶段有密切关系,呈现出时空表达的特点。在以往异黄酮合成途径的研究中,已挖掘到 PAL、C4H、C3H、CHS 等多个参与异黄酮生物合成的重要酶。本文对异黄酮生物合成通路涉及到的关键酶进行总结,具体信息如表1所示。
表1 异黄酮合成代谢途径中的关键酶及功能
Table 1 Key enzymes and functions in isoflavone synthesis metabolic pathway

相关酶酶功能参考文献苯丙氨酸解氨酶(PAL)苯丙氨酸代谢途径的关键酶和限速酶,影响异黄酮合成途径下游基因的表达[13]肉桂酸-4-羟基化酶(C4H)将反式肉桂酸催化生成香豆酸。在大豆生殖生长后期,促进异黄酮在豆荚和籽粒中的积累[14]黄烷酮-3-羟化酶(F3H)F3H减少了异黄酮的合成与积累。[15]4-香豆酸辅酶 A 连接酶(4CL)催化阿魏酸、香豆酸等底物生成 CoA 酯,也以芥子酸为底物生成芥子醇有助于木质素的积累[16]查尔酮合酶(CHS)CHS可在合成途径中建立基本骨架,是催化反应的关键酶。也影响花色素的合成以及植物抗性。[17]查尔酮还原酶(CHR)控制异黄酮中大豆苷元的生物合成的关键酶,与CHS共同作用催化异甘草素的合成。[18]查尔酮异构酶(CHI)催化分子内的环化反应,即催化二环的查尔酮等形成对应的三环化合物。[19]异黄酮合酶(IFS)两类IFS共同决定了大豆种子中异黄酮的合成,催化genistein的生物合成[20]
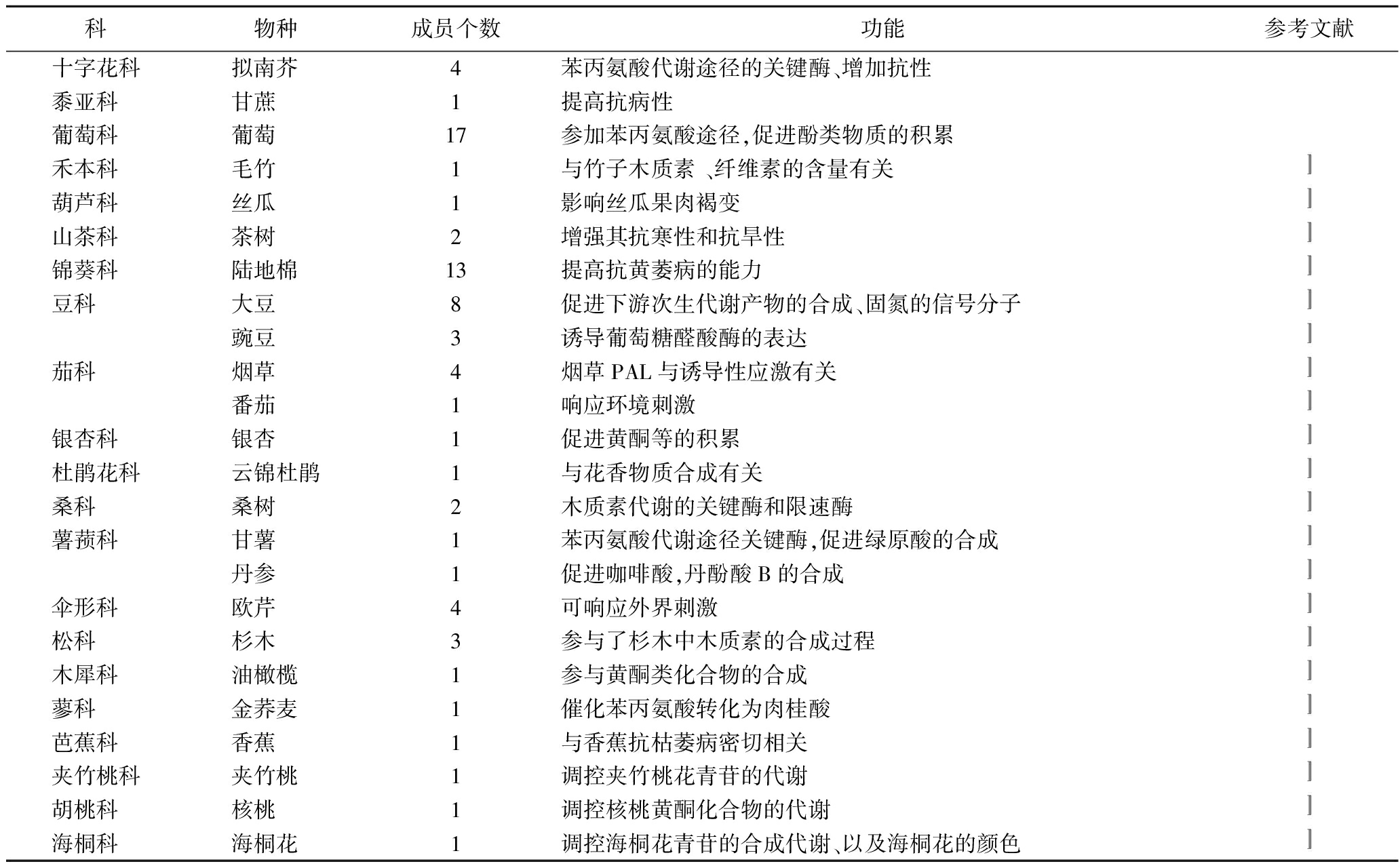
苯丙氨酸解氨酶(PAL)作为植物苯丙氨酸途径的第一个酶,广泛分布于植物和少数微生物中,现已在水稻、玉米、小麦等作物中都已分离纯化得到PAL[21]。在高等植物中PAL家族含有多个家族成员,例如在拟南芥中有4个PAL家族成员,菜豆中有2个、欧芹中存在4个,杨树中至少存在2个,茶树中存在1个PAL家族成员[22]。对已报道的不同物种中PAL的成员个数进行统计(附表3,https://doi.org/10.13995/j.cnki.11-1802/ts.034264),结果表明不同物种中所含PAL的基因家族成员个数不尽相同,但PAL家族成员序列具有较高的同源性,这表明PAL各成员都在异黄酮合成中具有重要意义。作为苯丙氨酸起始关键酶,PAL发挥着重要的作用。研究证实PAL能通过调控植物中苯丙氨酸代谢,影响相关代谢物质的积累,从而影响植物对生物胁迫和非生物胁迫的抗性及植物花色、花香的形成等。PAL多存在于植物表皮下的细胞以及微管组织中,而从亚细胞定位的情况来看PAL多数存在于细胞质中,这是植物苯丙氨酸代谢发生的主要场所。以往研究显示大豆在幼苗时期的异黄酮含量要比成熟期高,说明异黄酮的含量与植物的发育时期有着密切关系,且证实主要由于PAL通过影响4CL、IFS2、CHS2和CHS1等基因的变化来调控异黄酮的合成[23],表明PAL影响着异黄酮合成途径中的下游基因的表达,从而影响着异黄酮的合成。此外,植物不同部位异黄酮的表达也存在差异。异黄酮在水稻的茎、叶、种子中有表达,但是在3个部位随着发育时期的不同,PAL的表达出现差异性[24]。宋修鹏等[25]也发现异黄酮在根中的表达尤其显著,在叶片中的表达量相对而言较少[25]。
肉桂酸-4-羟基化酶(C4H)是植物中第一个被鉴定出来的P450单加氧酶,而且在植物的各组织部位都表达出较高的活性[26]。肉桂酸-4-羟化酶(C4H)以肉桂酸为底物催化形成对-香豆酸[13],参与苯丙氨酸途径的氧化反应[26]。同PAL一样的是在不同植物中C4H的基因个数也不同。C4H在大豆发育前期表达量出现峰值,而且异黄酮的含量也有所升高,因此C4H的表达促进异黄酮的合成[27]。除此以外,C4H和PAL的变化趋势呈现一致,都受到植物的生长发育以及外界刺激因素的调控[26]。
4-香豆酸辅酶 A 连接酶(4CL)是苯丙氨酸途径的最后一个关键酶,催化各种羟基肉桂酸生成对应的硫酯,参与苯丙氨酸和各种末端产物合成途径。其作用机制可以分为2种,一是以香豆素酸、咖啡酸和阿魏酸为底物催化为芥子醇,二是4CL以芥子酸为底物生成芥子酰-辅酶A[28]。4CL在叶片和豆荚中表达,4CL在大豆发育时期的后期出现峰值,促使与C4H共同生成的香豆酰-CoA进入异黄酮生物合成途径[29-30]。
查尔酮合酶(CHS)是异黄酮合成途径中第一个分支酶,是异黄酮表达的关键酶之一[14]。在多数植物中能够催化4-香豆酰-CoA生成查尔酮,参与异黄酮的合成途径,但是在少数植物中也可以将肉桂酰-辅酶A或者咖啡酰-辅酶A作为底物生成黄酮类物质黄烷酮[31]。CHS在异黄酮合成途径中的作用不亚于PAL的作用。在豆科植物中CHS有8个家族成员,在异黄酮合成途径的下游发挥了重要作用。但是8个基因家族成员在植物中具有组织特异性表达的特点,CHS1主要在叶片中表达,CHS2主要在子叶中表达,CHS7和CHS8主要在种皮中表达,而剩余的家族基因在各部位的表达量并不显著。研究表明, CHS能影响花色素的形成,如抑制矮牵牛中CHS基因的表达,矮牵牛出现白色花或花色素分布不同的情况[32]。此外, CHS与大豆抗性形成有关。有研究表明转入CHS基因的番茄中黄酮醇的表达增高,且番茄的抗氧化能力提高[33];而在杨树中过表达的CHS增强了杨树对低温胁迫的耐受性,使其对低温环境的敏感性降低[34]。
查尔酮还原酶(CHR)是大豆中异黄酮合成必不可少的一种酶,有研究表明CHR对于染料木素的合成有重要影响[35]。而且在低大豆苷元含量的品种中CHR的含量也低,这代表着CHR的含量与大豆苷元的合成也有关系。SREEVIDYA等[36]认为CHR发挥作用与CHS有关,CHR将在染料木素合成途径中CHS催化形成的中间物质催化形成异甘草素,有学者推测CHR和CHS的能够结合使得CHS蛋白性质改变,从而使得CHS最终催化产物从查尔酮变为异甘草素[37]。CHR的表达目前仅存于豆科植物中,而且表达量也很低,但CHR的作用机制相对复杂,是否有其他辅助因子的参与也更有待研究。
查尔酮异构酶(CHI)是最早被发现和研究的异黄酮合成的关键酶,这主要因为它能够催化形成类黄酮类色素[33, 37]。在豆科植物中存在2种CHI,即为Ⅰ型 CHI 和Ⅱ型 CHI[38]。Ⅰ型 CHI也存在于一些非豆科植物中,与黄酮类、花青素的合成代谢有关,Ⅱ型 CHI的优势体现在能够催化异甘草素生成甘草素等,进而生成大豆苷元。大豆中异黄酮的合成主要是由Ⅱ型 CHI控制的[39]。有研究表明,两种CHI虽都在大豆中表达,但两者的同源性仅为50%左右[40],而且表达方式也不同,Ⅰ型 CHI主要在花组织与F3H协调表达参与异黄酮的合成途径,Ⅱ型CHI在根中有特异性表达,它通过与IFS协同调节参与异黄酮的合成途径[40]。虽然Ⅰ型CHI和Ⅱ型CHI的表达存在差异,但有趣的是类黄酮和异黄酮的合成过程中Ⅰ型和Ⅱ型CHI共享柚皮素这一重要的中间产物[40]。这代表虽然参与途径各不相同,但各种酶可以通过协同作用参与生物合成机制来控制每种中间产物的流向。
黄烷酮-3-羟化酶 (flavonone 3β-hydroxylase,F3H)的表达与异黄酮的合成呈负相关。这要归咎于F3H与异黄酮合成的相关酶呈现竞争关系[27],花青素也通过类黄酮合成机制来进行合成,F3H会促进花青素的合成,因此它的有效表达将过多底物用于合成花青素。研究证明转入玉米C1和R转录因子的大豆籽粒中能够抑制F3H合成花青素从而使得异黄酮的含量升高[10]。由于合成异黄酮的底物减少,因此作为异黄酮合成的下游基因,抑制F3H或者F3H基因沉默更有利于异黄酮的合成。
异黄酮合酶(IFS)是一种细胞色素P450单加氧酶[35],也是异黄酮类物质合成的第一个催化酶[21]。它能够在NADPH和O2的辅助下催化柚皮素和甘草素完成C2-C3迁移[20]。它首次由HAGMANN团队在处理后的大豆培养基上发现了IFS [41],由于它在植物中的含量较低而且不稳定,因此他发挥作用多同其他酶协作进行。易金鑫等[12]发现在农杆菌中转化CHS8和IFS2的大豆种子异黄酮的含量要高于分别转化两种基因的大豆种子的异黄酮含量。其作用机制目前有两种说法,其一为与CHI合成的柚皮素形成染料木素,其二是在CHR的参与下形成大豆苷元[35]。在豆科植物的合成代谢途径中柚皮素是一种非常重要的前体物质,黄酮、花青素以及染料木素的合成都曾涉及到柚皮素。研究表明柚皮素在F3H的催化下合成花青素,而且也与CHI调节染料木素的合成[42]。
3.3 异黄酮合成过程中的相关转录因子
转录因子通常作用于其目标基因的启动子区域并调节该基因表达[43]。异黄酮合成途径涉及到的多数基因都以基因家族的形式出现,且不同基因家族参与不同的代谢途径。而转录因子能够通过多种机制进行调节,如改变其mRNA/蛋白丰度或通过翻译后修饰[44]。对于异黄酮的合成与累积而言,转录因子的调控必不可少。
MYB转录因子是高等植物中最大的转录因子家族之一[45],在拟南芥有超过100个MYB转录因子[46],参与了包括苯丙氨酸代谢在内的多种植物代谢过程。研究表明MYB转录因子多数为R2R3类型[47],在植物生长发育的多个方面都有着重要作用,如参与次生代谢途径,调控细胞分化,抵御外界的各种胁迫等等[48]。有研究表明GmMYB29和GmMYB133是异黄酮合成的正向调节因子,通过调节IFS2和CHS8的启动子从而激活IFS2和CHS8的表达,从而参与大豆中异黄酮的合成[49]。GmMYB39可以通过抑制CHS的活性,来抑制异黄酮的合成[50]。R1型MYB家族转录因子GmMYB176可以激活CHS8启动子的活性,使其毛状根中的异黄酮的含量增加[51]。2种R2R2型MYB转录因子GmMYB133和GmMYB176能够形成异二聚体,同自身也可形成同二聚体[49],参与异黄酮的合成途径。GmMYB58和GmMYB205也是MYB因子家族的两种关键正向调控物,GmMYB58和GmMYB205能够激活CHS、IFS的启动子活性,促进大豆种子中的异黄酮的合成[29]。GmMYB100是MYB家族R2R3型调控因子,该因子是一种负向调控因子。有研究表明GmMYB100的过表达与大豆毛状根和拟南芥中类黄酮的相关表达基因的水平呈反比, GmMYB100通过调节CHI和IFS的启动子的活性来抑制异黄酮的合成[30]。值得注意的是GmMYB100不光在大豆的根中表达,在花、叶和胚胎中也有表达[30]。GmMYBJ3也是一种典型的R2R3MYB转录因子,事实表明,GmMYBJ3在大豆胚乳中通过激活CHS8和CHI1A的启动子,促进异黄酮的累积[30]。GmMYB12A和GmMYB12B2通过促进PAL、CHS等酶的表达,从而达到促进异黄酮积累的目的[48]。GmMYB183也是MYB转录因子之一,不过与之前的调控机制不同的是该基因能够在盐胁迫下激活GmCYP81E11d的表达促进类黄酮的表达增强了抗盐性[52]。
bHLH转录因子普遍存在于多种生物中,也是植物中最大的转录因子家族之一[53],其中拟南芥中存在162个bHLHs转录因子[53]。bHLH转录因子的结构域可以分为一个HLH区域和一个碱性区域,其中HLH区域含有一个 α螺旋-环-α螺旋的结构部位,碱性区域能够识别作用元件并与之结合[54]。同时,HLH还可以使转录因子之间形成同基因二聚体或者与bHLH其他亚族的转录因子形成异二聚体[53],这是bHLHs转录因子能够发挥作用的原因。同时,多数的bHLHs还可以特异性识别并结合G-Box(5′-CACGTG-3′)序列[55]。以往研究表明MYC含有bHLH结构域,是bHLH家族转录因子的亚族,而在长春花中发现G-Box能够与CrMYC1和CrMYC2结合,促进下游基因表达[56]。其生物学功能涉及到植物生长的多个方面。例如bHLH- MYB结合体可以与WD40形成转录因子复合体参与拟南芥表皮毛的生长[57]。bHLH转录因子也具有调控异黄酮合成的功能。刘德泉等人发现bHLH-MYC类转录因子家族成员GmbHLH3a和GmMYC2在大豆胚乳的发育后期有高表达,促进大豆胚乳中异黄酮的合成[53]。王庆钰等[58]在大豆品种吉林32中得到GmbHLH13A7和GmbHLH3B13两个bHLH转录因子,并且在大豆的花和胚乳中有高表达,并从转基因大豆籽粒中发现异黄酮含量提高了24%~27%,因此两种转录因子都可正向调节异黄酮的合成。王天亮等[54]在吉林32中得到GmbHLH3和GmPIF1两个转录因子,并在大豆发根中有高表达。在过表达的GmbHLH3的大豆株系中,GmPAL1、GmC4H、Gm4CL、GmCHI1B1、GmIFS2和GmF3H基因的表达量也增高。在过表达GmPIF1的株系中,GmCHI1B1、GmPAL1和GmIFS2基因的表达水平提高。这代表GmbHLH3和GmPIF1对大豆发根中异黄酮的合成有正向调节作用。
表2 异黄酮合成过程中相关转录基因
Table 2 Related genes transcribed during isoflavone synthesis
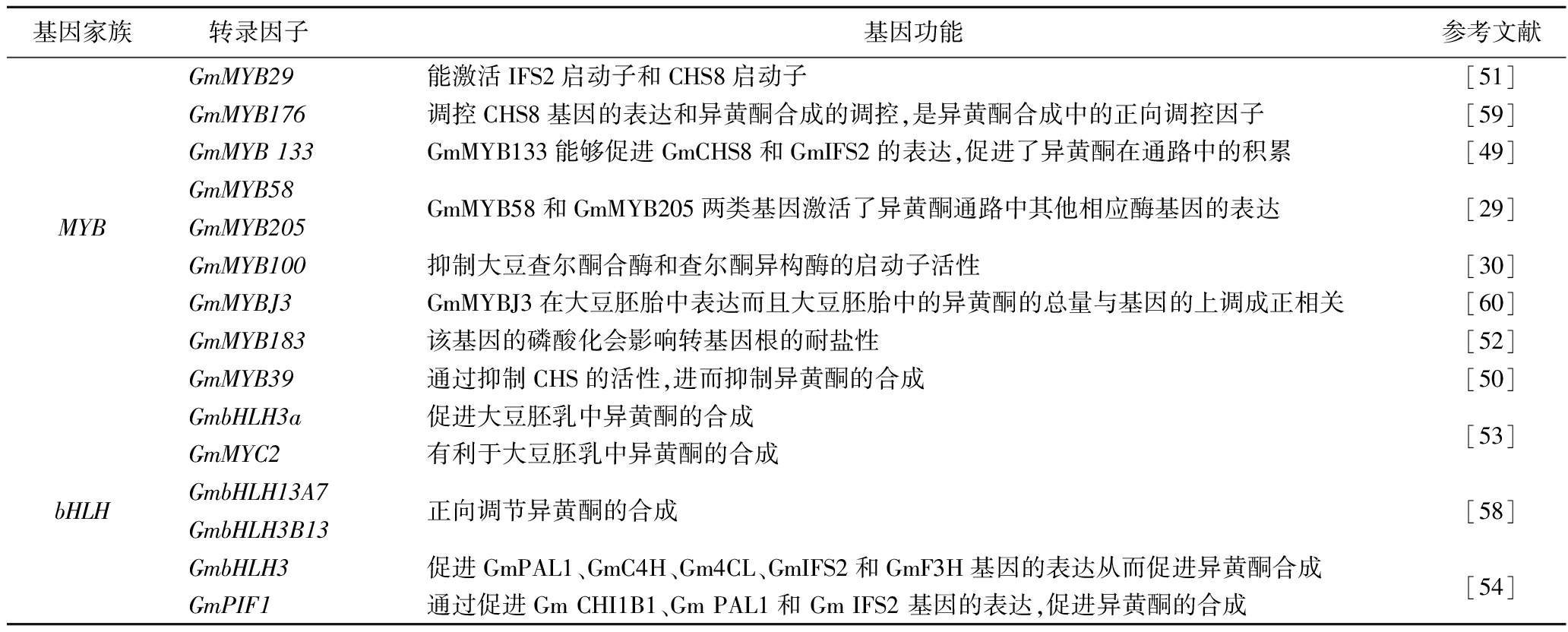
基因家族转录因子 基因功能参考文献MYBGmMYB29GmMYB176GmMYB 133GmMYB58GmMYB205GmMYB100GmMYBJ3GmMYB183GmMYB39能激活IFS2启动子和CHS8启动子调控CHS8基因的表达和异黄酮合成的调控,是异黄酮合成中的正向调控因子GmMYB133能够促进GmCHS8和GmIFS2的表达,促进了异黄酮在通路中的积累GmMYB58和GmMYB205两类基因激活了异黄酮通路中其他相应酶基因的表达抑制大豆查尔酮合酶和查尔酮异构酶的启动子活性GmMYBJ3在大豆胚胎中表达而且大豆胚胎中的异黄酮的总量与基因的上调成正相关该基因的磷酸化会影响转基因根的耐盐性通过抑制CHS的活性,进而抑制异黄酮的合成[51][59][49][29][30][60][52][50]bHLHGmbHLH3aGmMYC2GmbHLH13A7GmbHLH3B13GmbHLH3GmPIF1促进大豆胚乳中异黄酮的合成有利于大豆胚乳中异黄酮的合成正向调节异黄酮的合成促进GmPAL1、GmC4H、Gm4CL、GmIFS2和GmF3H基因的表达从而促进异黄酮合成通过促进Gm CHI1B1、Gm PAL1和 Gm IFS2 基因的表达,促进异黄酮的合成[53][58][54]
4 展望
异黄酮是一种主要活跃于豆科植物中的活性成分,不仅对豆科植物的生长发育具有重要作用,而且对人体具有显著的保健作用,在抗肿瘤、预防心血管疾病和骨质疏松症等方面都有着良好的功效。但由于植物中的含量较少,很难通过从植物中提取异黄酮满足生产需求。因此提高植物体内异黄酮的含量和体外合成异黄酮,研究其合成通路和调控机制是有效途径之一。异黄酮的合成由典型的苯丙氨酸代谢途径和异黄酮合成途径共同完成,其合成过程中涉及到大量的酶。虽然目前研究对涉及异黄酮合成途径的相关酶进行了酶活性分析,并对相应的基因进行克隆、表达分析和功能鉴定,然而异黄酮的生物合成是一个非常复杂的基因互作网络,涉及的转录因子数量十分庞大,因此对于未知的参与异黄酮生物合成途径的相关转录因子还有待挖掘。且异黄酮的存在范围是很小的,是否可以通过遗传转化体系获得更高含量的异黄酮也有待研究。
异黄酮的相关研究工作已经取得了一定的成效,但在以下方面还需要深入研究:a)现阶段在异黄酮的生物合成方面,各阶段的关键基因仍然存在分歧。此外,转录因子是异黄酮合成途径中关键酶发挥作用的前提,就目前的研究来看,还需扩大转录因子的研究范围,挖掘和探索异黄酮合成途径中的相关转录因子,丰富异黄酮合成的调控机制;b)异黄酮在植物中存在范围较窄,需深入研究异黄酮生物合成途径的关键酶和转录因子,通过转基因技术在其他非豆科植物中实现异黄酮的生物合成。
[1] LEPHART E D, WEST T W, WEBER K S, et al. Neurobehavioral effects of dietary soy phytoestrogens[J]. Neurotoxicol Teratol, 2002, 24(1): 5-16.
[2] 逄晓云, 崔岚, 施安国. 大豆蛋白、大豆异黄酮对血脂影响的研究进展[J]. 中国药师, 2004, 7(6):419-421.
PANG X Y, CUI L, SHI A G. Research progress on the effect of soybean protein and soybean isoflavone on blood lipid[J]. China Pharmacist, 2004, 7(6):419-421.
[3] 那晓琳, 崔洪斌. 大豆异黄酮的类雌激素作用与预防骨质疏松研究进展[J]. 中国骨质疏松杂志, 2002, 8(4): 370-371.
NA X L, CUI H B. Research progress on estrogen-like effect of soybean isoflavones and prevention of osteoporosis[J]. Chinese Journal of Osteoporosis, 2002, 8(4): 370-371.
[4] ZIELONKA J, ![]() J, GRYNKIEWICZ G. Radical scavenging properties of genistein[J]. Free Radical Biology and Medicine, 2003, 35(8):958-965.
J, GRYNKIEWICZ G. Radical scavenging properties of genistein[J]. Free Radical Biology and Medicine, 2003, 35(8):958-965.
[5] WEI H C, CAI Q Y, RAHN R O. Inhibition of UV light- and Fenton reaction-induced oxidative DNA damage by the soybcan isoflavone genistein[J]. Carcinogenesis, 1996, 17(1):73-77.
[6] 秦传勇. 大豆苷元对谷氨酸体外诱导海马神经元损伤大鼠的保护作用[J]. 医药导报, 2010, 29(1):25-27.
QIN C Y. The neuroprotective effects of daidzein on hippocampal neurons injury induced by glutamate in vitro on rats[J]. Herald of Medicine, 2010, 29(1):25-27.
[7] GRAHAM T L. Flavonoid and isoflavonoid distribution in developing soybean seedling tissues and in seed and root exudates[J]. Plant Physiology, 1991, 95(2):594-603.
[8] JANSEN R C, NAP J P. Genetical genomics: The added value from segregation[J]. Trends in Genetics, 2001, 17(7):388-391.
[9] 崔艳伟, 李文龙, 常文锁, 等. 大豆异黄酮合成途径相关基因差异表达分析[J]. 植物遗传资源学报, 2016, 17(4):719-725.
CUI Y W, LI W L, CHANG W S, et al. Differential expression analysis of genes associated with isoflavone synthesis in soybean (Glycine max merr.)[J]. Journal of Plant Genetic Resources, 2016, 17(4):719-725.
[10] YU O, SHI J E, HESSION A O, et al. Metabolic engineering to increase isoflavone biosynthesis in soybean seed[J]. Phytochemistry, 2003, 63(7):753-763.
[11] GUTIERREZ-GONZALEZ J J, WU X L, GILLMAN J D, et al. Intricate environment-modulated genetic networks control isoflavone accumulation in soybean seeds[J]. BMC Plant Biology, 2010, 10:105.
[12] 易金鑫, 徐照龙, 王峻峰, 等. GmCHS8和GmIFS2基因共同决定大豆中异黄酮的积累[J]. 作物学报, 2011, 37(4):571-578.
YI J X, XU Z L, WANG J F, et al. GmCHS8 and GmIFS2 gene co-determine accumulation of isoflavonoid in soybean[J]. Acta Agronomica Sinica, 2011, 37(4):571-578.
[13] 王春丽, 王周礼, 杨建利, 等. 大豆异黄酮的合成、调控、生理生态功能及其应用研究进展[J]. 中国农学通报, 2019, 35(19):34-40.
WANG C L, WANG Z L, YANG J L, et al. Biosynthesis, regulation, physiological and ecological functions and application of soybean isoflavones: A review[J]. Chinese Agricultural Science Bulletin, 2019, 35(19):34-40.
[14] 王艳, 武林, 孙梦阳, 等. 不同生育时期大豆异黄酮合成相关酶基因表达的分析[J]. 大豆科学, 2012, 31(6):887-893.
WANG Y, WU L, SUN M Y, et al. Analysis of gene expression underlying soybean isoflavone synthesis relative enzymes at different growth stages[J]. Soybean Science, 2012, 31(6):887-893.
[15] 姜伊娜. 大豆黄烷酮-3-羟化酶基因(f3h)RNA干扰型载体的构建及功能验证[D]. 上海: 上海交通大学, 2009.
JIANG Y N. Construction and valldation of RNA interfereace vector for soybean flavanone 3-hydroxylase gene(F3H) [D].Shanghai: Shanghai Jiao Tong University, 2009.
[16] BAUCHER M, HALPIN C, PETIT-CONIL M, et al. Lignin: Genetic engineering and impact on pulping[J]. Critical Reviews in Biochemistry and Molecular Biology, 2003, 38(4):305-350.
[17] 陈衡, 潘相文, 王飞飞, 等. 大豆异黄酮生物合成与调控的分子机制研究进展[J]. 土壤与作物, 2021, 10(2):126-142.
CHEN H, PAN X W, WANG F F, et al. Molecular mechanisms of isoflavone biosynthesis and regulation in soybean: A review[J]. Soils and Crops, 2021, 10(2):126-142.
[18] 崔慧敏, 曲硕, 万金璐, 等. 大豆CHR2-1基因生物信息学分析与大豆遗传转化[J]. 大豆科学, 2022, 41(3):274-280.
CUI H M, QU S, WAN J L, et al. Bioinformatics analysis of soybean CHR2-1 gene and its genetic transformation to soybean[J]. Soybean Science, 2022, 41(3):274-280.
[19] 田玲. 调控大豆异黄酮合成相关转录因子基因的克隆与表达模式分析[D]. 北京: 中国农业科学院, 2014.
TIAN L. Cloning and expression pattern analysis of transcription factor genes regulating soybean isoflavone synthesis[D].Beijing: Chinese Academy of Agricultural Sciences, 2014.
[20] STEELE C L, GIJZEN M, QUTOB D, et al. Molecular characterization of the enzyme catalyzing the aryl migration reaction of isoflavonoid biosynthesis in soybean[J]. Archives of Biochemistry and Biophysics, 1999, 367(1):146-150.
[21] 侯鹏. 大豆PAL基因家族时空表达及PAL2-1基因对异黄酮含量调控的研究[D]. 哈尔滨: 东北农业大学, 2016.
HOU P. Spatio-temporal expression of PAL gene family in soybean and regulation of PAL2-1 gene on isoflavone content[D].Harbin: Northeast Agricultural University, 2016.
[22] 王婵婵, 王安娜, 吴蕾, 等. 大豆PAL2基因的克隆与分析[J]. 大豆科学, 2010, 29(1):13-17.
WANG C C, WANG A N, WU L, et al. Cloning and analysis of PAL2 gene from soybean[J]. Soybean Science, 2010, 29(1):13-17.
[23] 田玲, 李斌, 张晶莹, 等. 大豆子粒发育过程中异黄酮合成相关酶基因的表达模式与异黄酮积累的相关分析[J]. 植物遗传资源学报, 2014, 15(6):1295-1304.
TIAN L, LI B, ZHANG J Y, et al. Correlation analysis between gene expression of isoflavone synthesis relative enzymes and isoflavone accumulation during the seed development in soybean[J]. Journal of Plant Genetic Resources, 2014, 15(6):1295-1304.
[24] 邵雅芳, 徐非非, 唐富福, 等. 水稻花青素合成相关基因的时空表达研究[J]. 核农学报, 2013, 27(1):9-14.
SHAO Y F, XU F F, TANG F F, et al. The temporal and spatial expression pattern of anthocyanin related genes in rice(Oryza sativa l.)[J]. Journal of Nuclear Agricultural Sciences, 2013, 27(1):9-14.
[25] 宋修鹏, 黄杏, 莫凤连, 等. 甘蔗苯丙氨酸解氨酶基因(PAL)的克隆和表达分析[J]. 中国农业科学, 2013, 46(14):2856-2868.
SONG X P, HUANG X, MO F L, et al. Cloning and expression analysis of sugarcane phenylalanin ammonia-lyase (PAL) gene[J]. Scientia Agricultura Sinica, 2013, 46(14):2856-2868.
[26] 李莉, 赵越, 马君兰. 苯丙氨酸代谢途径关键酶: PAL、C4H、4CL研究新进展[J]. 生物信息学, 2007, 5(4):187-189.
LI L, ZHAO Y, MA J. Recent progress on key enzymes: PAL, C4H, 4CL of phenylalanine metabolism pathway[J]. Chinese Journal of Bioinformatics, 2007, 5(4):187-189.
[27] 崔艳伟. 黄淮海大豆高异黄酮种质筛选与相关基因差异表达分析[D]. 保定: 河北农业大学, 2014.
CUI Y W. Screening of soybean germplasm with high isoflavone content and differential expression analysis of related genes in Huanghuaihai[D].Baoding: Hebei Agricultural University, 2014.
[28] GUI J S, SHEN J H, LI L G. Functional characterization of evolutionarily divergent 4-coumarate: Coenzyme A ligases in rice[J]. Plant Physiology, 2011, 157(2):574-586.
[29] HAN X Y, YIN Q G, LIU J Y, et al. GmMYB58 and GmMYB205 are seed-specific activators for isoflavonoid biosynthesis in Glycine max[J]. Plant Cell Reports, 2017, 36(12):1889-1902.
[30] YAN J H, WANG B, ZHONG Y P, et al. The soybean R2R3 MYB transcription factor GmMYB100 negatively regulates plant flavonoid biosynthesis[J]. Plant Molecular Biology, 2015, 89(1):35-48.
[31] MARTIN C R. Structure, function, and regulation of the Chalcone synthase[J]. International Review of Cytology, 1993, 147:233-284.
[32] GROTEWOLD E, PETERSON T. Isolation and characterization of a maize gene encoding chalcone flavonone isomerase[J]. Molecular and General Genetics MGG, 1994, 242(1):1-8.
[33] AHARONI A, O′CONNELL A P. Gene expression analysis of strawberry achene and receptacle maturation using DNA microarrays[J]. Journal of Experimental Botany, 2002, 53(377):2073-2087.
[34] SHIMADA N, AOKI T, SATO S, et al. A cluster of genes encodes the two types of chalcone isomerase involved in the biosynthesis of general flavonoids and legume-specific 5-deoxy(iso)flavonoids in Lotus japonicus[J]. Plant Physiology, 2003, 131(3):941-951.
[35] 张卓. 大豆查尔酮还原酶基因的克隆和功能分析[D]. 长春: 吉林农业大学, 2014.
ZHANG Z. Cloning and functional analysis of chalcone reductase gene from soybean[D].Changchun: Jilin Agricultural University, 2014.
[36] SREEVIDYA V, SRINIVASA RAO C, SULLIA S, et al. Metabolic engineering of rice with soybean isoflavone synthase for promoting nodulation gene expression in rhizobia[J]. Journal of Experimental Botany, 2006, 57(9):1957-1969.
[37] SPARVOLI F, MARTIN C, SCIENZA A, et al. Cloning and molecular analysis of structural genes involved in flavonoid and stilbene biosynthesis in grape (Vitis vinifera L.)[J]. Plant Molecular Biology, 1994, 24(5):743-755.
[38] MEHDY M C, LAMB C J. Chalcone isomerase cDNA cloning and mRNA induction by fungal elicitor, wounding and infection[J]. The EMBO Journal, 1987, 6(6):1527-1533.
[39] WINKEL-SHIRLEY B. Flavonoid biosynthesis. A colorful model for genetics, biochemistry, cell biology, and biotechnology[J]. Plant Physiology, 2001, 126(2):485-493.
[40] RALSTON L, SUBRAMANIAN S, MATSUNO M, et al. Partial reconstruction of flavonoid and isoflavonoid biosynthesis in yeast using soybean type I and type II Chalcone isomerases[J]. Plant Physiology, 2005, 137(4):1375-1388.
[41] HAGMANN M, GRISEBACH H. Enzymatic rearrangement of flavanone to isoflavone[J]. FEBS Letters, 1984, 175(2):199-202.
[42] 姜伊娜. 大豆黄酮合成酶基因的克隆与RNA干扰调控异黄酮含量的研究[D]. 上海: 上海交通大学, 2013.
JIANG Y N. Cloning of daidzein synthase gene and regulation of isoflavone content by RNA interference[D].Shanghai: Shanghai Jiao Tong University, 2013.
[43] KARIN M. Too many transcription factors: Positive and negative interactions[J]. The New Biologist, 1990, 2(2):126-31.
[44] VOM ENDT D. Transcription factors controlling plant secondary metabolism: What regulates the regulators?[J]. Phytochemistry, 2002, 61(2):107-114.
[45] ZHOU M L, HOU H L, ZHU X M, et al. Soybean transcription factor GmMYBZ2 represses catharanthine biosynthesis in hairy roots of Catharanthus roseus[J]. Applied Microbiology and Biotechnology, 2011, 91(4):1095-1105.
[46] CHEN Y H, YANG X Y, HE K, et al. The MYB transcription factor superfamily of Arabidopsis: Expression analysis and phylogenetic comparison with the rice MYB family[J]. Plant Molecular Biology, 2006, 60(1):107-124.
[47] STRACKE R, WERBER M, WEISSHAAR B. The R2R3-MYB gene family in Arabidopsis thaliana[J]. Current Opinion in Plant Biology, 2001, 4(5):447-456.
[48] 滕巍. 大豆胚尖遗传转化体系的优化及MYB转录因子转基因大豆的鉴定[D]. 长春: 吉林大学, 2015.
TENG W. Optimization of genetic transformation system of soybean embryo tip and identification of MYB transcription factor transgenic soybean[D].Changchun: Jilin University, 2015.
[49] BIAN S M, LI R H, XIA S Q, et al. Soybean CCA1-like MYB transcription factor GmMYB133 modulates isoflavonoid biosynthesis[J]. Biochemical and Biophysical Research Communications, 2018, 507(1-4):324-329.
[50] LIU X Q, YUAN L L, XU L, et al. Over-expression of GmMYB39 leads to an inhibition of the isoflavonoid biosynthesis in soybean (Glycine max. L)[J]. Plant Biotechnology Reports, 2013, 7(4):445-455.
[51] CHU S S, WANG J, ZHU Y, et al. An R2R3-type MYB transcription factor, GmMYB29, regulates isoflavone biosynthesis in soybean[J]. PLoS Genetics, 2017, 13(5): e1006770.
[52] PI E X, XU J, LI H H, et al. Enhanced salt tolerance of rhizobia-inoculated soybean correlates with decreased phosphorylation of the transcription factor GmMYB183 and altered flavonoid biosynthesis[J]. Molecular &Cellular Proteomics: MCP, 2019, 18(11):2225-2243.
[53] 刘德泉. 大豆异黄酮相关基因的表达分析及GmbHLH3a基因的功能鉴定[D]. 长春: 吉林大学, 2015.
LIU D Q. Expression analysis of soybean isoflavone related genes and functional identification of LH 3A gene in GmbH[D].Changchun: Jilin University, 2015.
[54] 王天亮. 大豆bHLH类转录因子GmbHLH3和GmPIF1的功能分析[D]. 长春: 吉林大学, 2020.
WANG T L. Functional analysis of soybean bHLH transcription factors GmbHLH3 and GmPIF1[D].Changchun: Jilin University, 2020.
[55] TOLEDO-ORTIZ G, HUQ E, QUAIL P H. The Arabidopsis basic/helix-loop-helix transcription factor family[J]. The Plant Cell, 2003, 15(8):1749-1770.
[56] CHATEL G, MONTIEL G, PRÉ M, et al. CrMYC1, a Catharanthus roseus elicitor- and jasmonate-responsive bHLH transcription factor that binds the G-box element of the strictosidine synthase gene promoter[J]. Journal of Experimental Botany, 2003, 54(392):2587-2588.
[57] ZHAO M Z, MOROHASHI K, HATLESTAD G, et al. The TTG1-bHLH-MYB complex controls trichome cell fate and patterning through direct targeting of regulatory loci[J]. Development, 2008, 135(11):1991-1999.
[58] 王庆钰,王天亮, 王英, 等. 大豆异黄酮合成相关转录因子GmbHLH13A7和GmbHLH3B13的克隆及其功能鉴定[C].第十届全国大豆学术讨论会论文摘要集. 北京, 2017:97.
WANG Q Y.WANG T L,WANG Y,et al. Isolation and characterization of bHLH transcription factors GmbHLH13A7and GmbHLH3B13 in soybean[C].The 10 th National Soybean Symposium, Soybean Committee of Crop Society of China. Beijing, 2017:97.
[59] YI J X, DERYNCK M R, LI X Y, et al. A single-repeat MYB transcription factor, GmMYB176, regulates CHS8 gene expression and affects isoflavonoid biosynthesis in soybean[J]. The Plant Journal: for Cell and Molecular Biology, 2010, 62(6):1019-1034.
[60] ZHAO M Z, WANG T L, WU P, et al. Isolation and characterization of GmMYBJ3, an R2R3-MYB transcription factor that affects isoflavonoids biosynthesis in soybean[J]. PLoS One, 2017, 12(6): e0179990.