目前,心血管疾病是全球人类死亡的主要原因,据世界卫生组织统计,全球每年约有1 750万人死于心血管疾病,预计在2030年每年将约有2 360万人因心血管疾病而去世,血栓性疾病作为一种严重威胁人类健康的常见心血管疾病,其发病率、致残率和致死率在各类疾病中均排名第一,已成为全球性公共卫生问题[1],但近年不断出现使用抗血栓药物导致副作用的病例报告,如肝素与低分子肝素的诱发大疱性出血皮炎,阿派沙班导致自发性胸髓出血,利伐沙班引发了血小板减少症[2-4],因此安全性高的食源性抗血栓肽逐渐成为研究热点,如TU等[5]通过模拟胃肠道消化乳铁蛋白,在其水解物中鉴定出一种多肽(ENLPEKADRD),该多肽具有体外抗凝血活性;CHENG等[6]从牡蛎蛋白胃酶水解物中分离并鉴定了一种新型食源性抗凝剂七肽,近年来报道的食源性抗血栓肽主要以海洋生物蛋白和动植物蛋白为制备原料[7]。
海参属于药食同源类型的水产品,已有多种海参酶解产物被证实具有提高免疫、抗疲劳等多种生物活性[8-10]。其中玉足海参(Holothuria leucospilota),是热带及亚热带海洋地区的广泛分布的海参种类之一,在中国广东省和海南省等南方沿海地区均有分布[11]。2013年在海南省实现玉足海参池塘养殖并建立规模化的养殖基地[12]。郗亚凡等[13]通过营养成分分析已指出玉足海参体壁富含蛋白质,是开发海参抗血栓活性肽的优质原料。
目前制备食源性抗血栓活性肽主要通过酶解法结合分离纯化及活性鉴定等步骤获得,使用分离、纯化再鉴定的方法需要耗费大量时间,且分离纯化操作繁琐,获得高纯度活性肽难度大,产量低等因素均限制了抗血栓肽研究的发展[7]。近年来,如CAI等[14]采用计算机虚拟筛选获得6种具有潜在降血糖活性的肽。CUI等[15]从通过分子对接复合技术筛选了3种具有抗痛风活性的肽。通过生物信息学和分子对接技术,对多肽进行生物活性预测能够高效筛选出特定活性肽片段,缩短纯化的时间,获得效果优良的目标肽[16-17]。
本研究以玉足海参作为蛋白原料,利用多肽组学表征其酶解产物的完整肽谱,利用生物信息学和分子对接技术,从玉足海参肽谱中快速筛选出抗血栓活性肽,通过合成多肽进行体外抗血栓实验验证,获得具有良好抗血栓活性的海参活性肽,为食源性抗血栓肽的快速筛选提供参考。
1 材料与方法
1.1 材料与试剂
玉足海参,海南省琼海市潭门海鲜市场;凝血酶(1 000 U)、牛纤维蛋白原,北京索莱宝科技有限公司;绵羊血清、Tris-HCl,上海源叶生物科技有限公司;部分凝血活酶时间(activated partial thromboplatin time,APTT)诊断试剂盒、凝血酶原时间(prothrombin time,PT)诊断试剂盒、凝血酶时间(thrombin time,TT)诊断试剂盒,武汉中太生物公司;中性蛋白酶,麦克林试剂公司。
1.2 仪器与设备
JYL-C012型榨汁搅拌机,九阳股份有限公司;DZKW-S-4型电热恒温水浴锅,北京光明医疗仪器有限公司;2-16KL型台式高速冷冻离心机,德国Sigma公司;EYEL4N-1300旋转蒸发仪,埃朗科技国际贸易(上海)有限公司;LGJ-12型真空冷冻干燥机,北京松源冻干发展有限公司;JJ124BC型电子天平,江苏常熟双杰测试仪器厂;超滤装置,Cole-Parmer公司;超高效液相、精确质谱仪、全自动酶标仪酶标仪,Thermo Fisher Scientific公司;半自动4通道凝血分析仪,景川诊断技术有限公司。
1.3 实验方法
1.3.1 玉足海参酶解产物多肽组学分析
1.3.1.1 制备玉足海参酶解产物
鲜活玉足海参低温运输,解剖海参,取出内脏,用蒸馏水清洗,在4 ℃下剥去内脏。海参体壁(50.0 g)悬浮在蒸馏水(1∶5,质量比)中通过榨汁机破碎为匀浆状,适度添加2 mol/L氢氧化钠或2 mol/L盐酸将混合物调节至pH 7.0。放入恒温水浴锅中设置50 ℃,预热后添加中性蛋白酶到海参匀浆中,开启磁力搅拌设置酶解时长为480 min。酶解结束后沸水浴灭活10 min,用冷水快速降温,8 000 r/min,20 min 离心后取上清液,命名为SCEH并储存在-80 ℃冰箱中待进行进一步分析。
1.3.1.2 LC-MS/MS检测
质谱检测之前进行超滤和脱盐前处理,SCEH样品冻干粉加入0.1%三氟乙酸(trifluoroacetic acid,TFA)水溶液复溶,14 000×g离心10 min,取上清液使用10 kDa超滤管(PALL, OD010C35)超滤,13 500×g离心10 min,取滤过液使用C18固相萃取柱(66871, Sigma)脱盐,具体步骤为首先使用乙腈洗涤萃取柱,然后用0.1%TFA平衡萃取柱,超滤后的样品溶液上样脱盐后再次收集。用于液相的溶液A是0.1%甲酸水溶液,溶液B是0.1%甲酸在乙腈(84%乙腈)中的溶液。LC柱(0.15 mm×150 mm,RP-C18,Column Technology Inc.)用95%的液体A平衡,样品通过自动进样器加载到Zorbax 300SB-C18 peptide traps (Agilent Technologies, Wilmington, DE)上。在另一个LC柱分离后,相关的LC梯度设置如下:0~50 min,B液体线性梯度为4%~50%;50~54 min,液体B为50%~100%的线性梯度;54~60 min,其中液体B保持在100%。通过毛细管HPLC分离消化产物,并使用通过质谱分析,分析时间:60 min。检测方式:正离子。根据以下方法收集肽和肽片段的质荷比:在每次扫描(全扫描)后获得10个片段图(MS2扫描)。使用软件Max quant 1.5.5.1使用质谱测试的原始文件(原始文件)检索相应的数据库,Charcot相关参数如下:酶(无)、最大缺失裂解(2)、固定修饰(-)、可变修饰[氧化(M)]、Decoy数据库模式(反向)、数据库(UniProt Aspidochirotida 31557 20221021),肽耐受性(20 mg/L)、MS/MS耐受性(0.1 Da)、FDR(≤0.01)。数据库NCBI (http:// www.ncbi.nlm.nih.gov);BIOPEP- UWM (https://biochemia.uwm.edu.pl/biopep-uwm /);UniProt Aspidochirotida;PDB(https://www.rcsb.org/)。
1.3.2 建立玉足海参抗血栓肽候选库
1.3.2.1 酶解产物初步分离纯化
玉足海参多糖存在于酶解产物中,是影响海参肽体外抗血栓活性测定的主要杂质。本实验选用乙醇沉降法联合超滤法去除SCEH中的多糖,并通过分级超滤进行初步筛选。向SCEH中按照体积比 1∶3 加入无水乙醇搅拌均匀后,静置于4 ℃冰箱12 h。将醇沉后的中性酶酶解液通过旋蒸除去无水乙醇后离心(8 000 r/min, 20 min),取上清液获得SCAS(海参醇沉液)。调整SCAS浓度并使用水系滤膜抽滤后,使用 3 kDa超滤膜对SCAS进行超滤,上述超滤操作获得的截留液与超滤液分别命名为SCUS-1与SCUS-2,并通过真空冻干获得对应的(SECH、SCAC、SCUS-1、SCUS-2)冻干粉,使用GB/T 9695.31—2008《肉制品:总糖含量测定》的分光光度法评估各组分的脱糖效果。此外对SCUS-2使用1 kDa超滤膜进行分级,所获得的截留液和超滤液,分别命名为SCUS-2.1、SCUS-2.2,收集冻干粉,与上述各组分一同保存于-20 ℃冰箱。
1.3.2.2 超滤组分的凝血酶体外抑制效果
配制SCUS-2.1、SCUS-2.2同等浓度溶液,酶标仪设置在405 nm的波长、37 ℃。纤维蛋白原以0.1%(质量分数)的浓度溶于pH值为7.2的Tris-HCl缓冲液中。将140 μL纤维蛋白原溶液和40 μL样品溶液加入板孔中,混合37 ℃孵育5 min测吸光度。然后加入10 μL凝血酶(12 IU/mL)开始反应孵育5 min再次测吸光度。用公式(1)计算多肽的凝血酶抑制率。此外,凝血酶抑制活性均为样品浓度下的凝血酶抑制率,而非测定体系下的终浓度。
凝血酶抑制率![]()
(1)
式中:AS、AC分别为未加凝血酶前的样品组和空白组的吸光度值;ASB、ACB分别为加凝血酶孵育后的样品组和空白组的吸光度值。
1.3.2.3 活性肽候选库的生物信息学分析
通过对比SCUS-2.1、SCUS-2.2的体外活性,确定抗血栓候选肽库的分子质量分布范围,基于1.3.1.2节获得的海参酶解产物多肽组学结果,确定候选肽库中备选的肽序列。使用Expasy(http://web.expasy.org/compute_pi/)计算亲水性和不稳定性;ProtParam(http://web.expasy.org/protparam/)计算相对分子质量;PepDraw(http://www.tulane.edu/~biochem/W W/PepDraw/)计算净电荷和疏水性;TBtools(http://web.expasy.org/protparam/)计算脂肪族指数。
1.3.3 分子对接
将候选肽库中的多肽与重组水蛭素肽段(HIRV2)以Fasta格式文件导入Discovery Studio(2019R2 version,Biovia,SanDiego,CA,USA)软件进行分子建模并最小能量化。受体为凝血酶大分子,从蛋白结构数据库 PDB(https://www.rcsb.org/)下载凝血酶与配体复合物的三维结构。在软件中对表面受体与配体结合位点进行空间区域分析[18]。Discovery Studio 软件搭载的分子对接的算法主要CDOCKER、Gold、LibDock 等,这3种算法被广泛应用于CHARMm 力场中分子相互作用的计算。采用CDOCKER 算法的能量评分来评价多肽与受体作用的结合类型与强弱[18]。
首先在pH值为7.40环境条件下,将受体的三维构型进行质子化。然后除去水分子的空间位阻。通过输入多肽序列建立线性一级结构,采用 Full Minimization 算法优化分子自由能来确定其二级结构,计算步长为15 000步,容差设为0.01。将凝血酶受体外置位点 I 周围球性区域设为对接位点。
1.3.4 合成肽序列及活性验证
1.3.4.1 合成肽序列及纯化处理
多肽合成处理由南京肽谷科技有限公司完成。根据筛选出来的抗血栓活性肽序列C端的第一个氨基酸,按照多肽从C端到N端的序列,依次按照交替进行缩合反应和脱除反应,将多肽剩余的氨基酸全部缩合到树脂上,直至最后一个氨基酸缩合完成。采用茚三酮法检测验证反应完全,使用裂解液将树脂与多肽分离,反复冰乙醚洗涤过滤后得粗品肽。质谱检测粗品的分子质量是否正确,使用高效液相色谱进行纯化分离,冻干,然后得到纯品多肽。
1.3.4.2 体外抗血栓活性验证
合成肽的抗血栓活性通过部分凝血活酶时间,凝血酶原时间和凝血酶时间试剂盒检测进行表征。体外凝血酶抑制率方法与1.3.2.2节相同。
1.4 数据分析
应用 Discovery Studio软件对分子对接结果进行可视化处理。所有试验设置3组平行,数据以平均值±标准差表示,SPSS 26.0统计软件进行Tukey事后检验,P<0.05表示差异有统计学意义。采用Excel 2019和Origin 2023 等软件对数据进行处理和图表的制作。
2 结果与分析
2.1 抗血栓活性肽候选库的建立
2.1.1 确立候选肽库的分子质量分布范围
多肽组学分析作为前期研究,从在SCEH样品中表征出1 795条玉足海参肽序列,为了提高抗血栓活性肽筛选的准确性与筛选效率,缩小候选肽库的规模是必需步骤。为了排除海参多糖对活性测定的影响,通过乙醇沉淀法与超滤法联用,结果见图1。SCEH总糖含量为16.72%,对其加入乙醇沉淀多糖。SCAS的总糖含量下降为9.24%,进而对SCAS进行0.45 nm水系滤膜抽滤后使用3 kDa的超滤膜获得截留液SCUS-1与超滤液SCUS-2。其中SCUS-1与SCUS-2的总糖含量分别为3.76%、2.30%,有效排除海参多糖对凝血酶抑制率测定的影响。

图1 相同浓度下各组分凝血酶抑制率及总糖含量
Fig.1 Thrombin inhibition rate and total sugar content of each component at the same concentration
注:不同小写字母表示差异显著(P<0.05)(下同);各组分质量浓度为1 mg/mL。
已知食源性抗血栓肽的分子质量分布范围为300~3 000 Da[7],所以实验中使用超滤技术将SCUS-2组分按照分子质量分为SCUS-2.1(<1~3 kDa)与SCUS-2.2(1 kDa)2个超滤组分,再对比各个组分的凝血酶抑制率用以确定确立候选肽库的分子质量分布范围。SCUS-2组分通过1 kDa的超滤膜超滤获得SCUS-2.1与SCUS-2.2。由图1可知,在1 mg/mL的质量浓度下SCEH的凝血酶抑制率最高为36.26%,SCUS-2.1仅次于SCEH,为28.63%。可见总糖含量下降一定程度影响了凝血酶抑制率的表现,但是在总糖含量仅有2.30%的SCUS-2.1组分仍表现出较好的凝血酶抑制效果,且显著优于SCUS-2与SCUS-2.2。具有抗血栓活性作用的多肽分子应存在于SCUS-2.1组分中,所以候选肽库的分子质量分布范围设置为1~3 kDa具有可行性。
2.1.2 候选肽库的生物信息特征分析
基于SCEH多肽组学结果,其中分子质量符合1~3 kDa的多肽序列共有116条。凝血酶是一种丝氨酸蛋白酶,是凝血级联反应中一个关键性凝血因子,将可溶性纤维蛋白原转化为不可溶性纤维蛋白链,并催化许多其他凝血相关的反应。生物活性肽通常通过其理化特性与分子结构有效发挥其生物活性,通过生物信息学分析工具从净电荷、亲水性、氨基酸排列特征等多方面分析候选肽库的生物信息学特征是否符合目标活性肽的已知分子特征,有助于提高筛选效率[19]。
如图2-A所示,候选肽库的氨基酸排列特征表现为:在N端首个氨基酸由异亮氨酸(I)、缬氨酸(V)、亮氨酸(L)、苯丙氨酸(F)其中有3种均为脂肪族氨基酸,脂肪族残基有利于帮助多肽与凝血酶S2位点结合。在N2、N3位置主要由谷氨酸(D)和天冬氨酸(E)占据,二者带负电荷。此外天冬氨酸还在C3、C1位置有一定程度的显著表达。根据结构和功能研究,凝血酶对生理促凝/抗凝物质的特异性识别是通过对大分子底物、抑制剂和效应物上2个与其表面位置相反的带正电荷的外部位点进行构象修饰来实现的[20-21]。凝血酶的外置位点I由于存在许多带正电荷的残基而表现出强的正电位,因此它也被称为阴离子结合位点I。这些残基对纤维蛋白原的结合至关重要,带负电荷的活性肽分子可以通过与带正电荷残基结合来抑制凝血酶对纤维蛋白蛋白原的激活。由图2-B可知,肽库中79种肽的不稳定性系数<50,不稳定指数>50则认为该蛋白不稳定易变性,数值越大越不稳定,所以肽库中大部分多肽有稳定性的优势。如图2-C所示,肽库中86.4%多肽均带有负电荷,与高度硫酸化多糖类物质发挥显著抗血栓功效的机理相符[22]。脂肪族指数定义为脂肪族侧链(丙氨酸、缬氨酸、异亮氨酸和亮氨酸)占据的相对体积,由图2-D可知,肽库中71种肽序列脂肪族指数>30,说明脂肪族侧链大量存在。值得注意的是,通常GRAVY (grand average of hydropathicity)数值为2~-2,数值越大疏水性越好,如图2-E所示。肽库中仅有9种肽序列为疏水性肽,其余均表现出优良的亲水性。通过上述讨论,可以得知候选肽库的多种理化特征均有利于筛选抗血栓活性肽,同时具有较好的亲水性与分子稳定性。
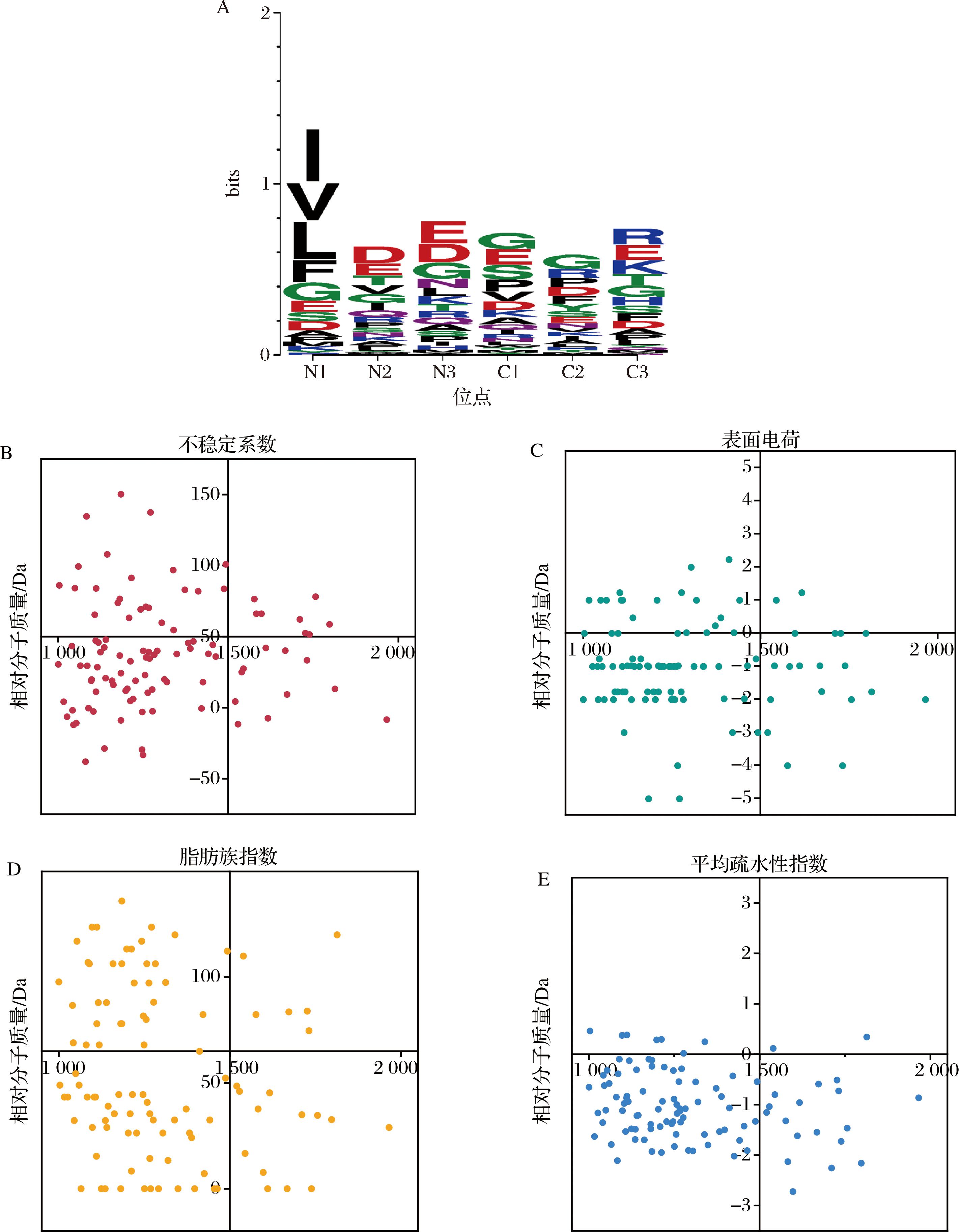
A-端点氨基酸种类特征;B-不稳定系数;C-表面电荷;D-脂肪族指数;E-平均疏水性指数
图2 候选肽库的生物学信息分析
Fig.2 Biological information analysis of candidate peptide libraries
2.2 筛选抗血栓活性肽
2.2.1 分子对接评分与理化性质分析
凝血酶外置位点 I结合可以与纤维蛋白原I和Ⅱ,血栓调节蛋白和肝素辅因子Ⅱ等生理性底物结合发挥其不同的生物学功效,天然存在的水蛭素可通过与上述生理性底物竞争结合外置位点I,从而阻断由凝血酶所带来的后续病理性过程[23-25]。以重组水蛭素碳端12肽(HIRV2)为模板设计的比伐卢定现已在临床中得到广泛应用[26]。本研究以 HIRV2 为模板,制备海参源新型凝血酶抑制肽。将候选肽库中的多肽及 HIRV2 与凝血酶的外置位点 I 进行分子对接,根据分子对接能量值进行排序, 筛选具备凝血酶抑制潜力的生物活性肽。此外,进一步对候选多肽和 HIRV2 的各项理化性质(分子对接能量值、净电荷、 亲水性、亲水性残基比例、脂肪族指数)进行比对分析。由表1可知,在分子对接得分高于HIRV2 的 6 条蛋白片段之中,(LAENEAGEGKPSRSSEP,命名为 SCp-1)与(VFDKEDNGQVNAGE,命名SCp-2)的净电荷和分子对接能力两项重要指标高于其他多肽序列,且GRAVY与亲水残基比例均与HIRV2相近。理论上SCp-1与SCp-2的凝血酶抑制效果优于其余4种多肽,因此选用这2条多肽进行下一步分析。
表1 玉足海参肽分子对接结果与理化性质分析
Table 1 Molecular docking results and physicochemical properties analysis of sea cucumber peptides
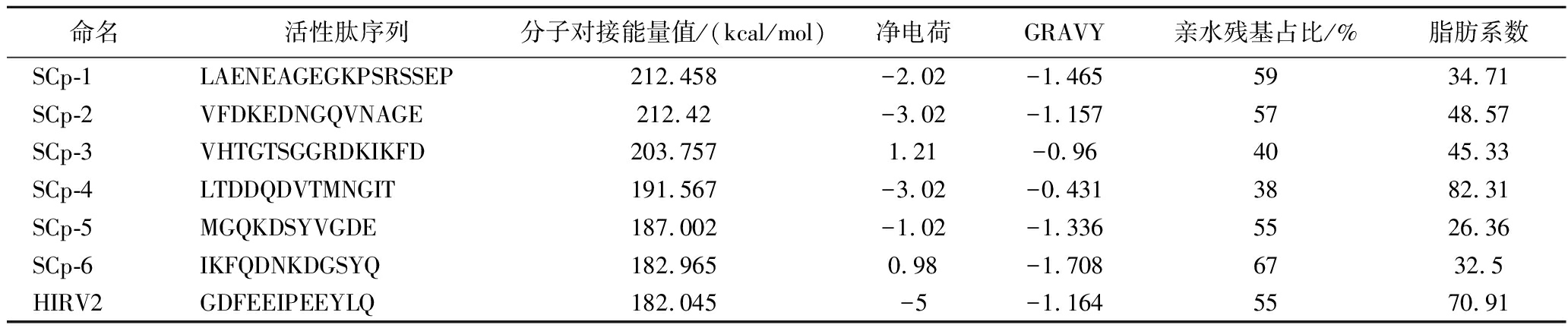
命名活性肽序列分子对接能量值/(kcal/mol)净电荷GRAVY亲水残基占比/%脂肪系数SCp-1LAENEAGEGKPSRSSEP212.458-2.02-1.4655934.71SCp-2VFDKEDNGQVNAGE212.42-3.02-1.1575748.57SCp-3VHTGTSGGRDKIKFD203.7571.21-0.964045.33SCp-4LTDDQDVTMNGIT191.567-3.02-0.4313882.31SCp-5MGQKDSYVGDE187.002-1.02-1.3365526.36SCp-6IKFQDNKDGSYQ182.9650.98-1.7086732.5HIRV2GDFEEIPEEYLQ182.045-5-1.1645570.91
2.2.2 SCp-1和SCp-2与3RM2分子对接作用构象分析
如图3 所示,多肽SCp-1和凝血酶(3RM2)的外置位点 I匹配结合较好,SCp-1与3RM2残基LYS-81、THR-74、ILF-82、GLN-38形成氢键,在ARG-67,ARG-73,LSY-110处通过相互吸引的电荷形成离子键,与VAL-112,LEU-65,ARG-75起疏水作用结合, SCp-1 能够牢牢地锚定在上述位点的空腔内,使其与3RM2形成稳定的复合物。SCp-1的抑制机制与CHEN等[27]报道的凝血酶抑制肽(P13)相似,通常凝血酶分子中的LYS-36、ARG-73、ARG-77、LYS-149是识别纤维蛋白原的结合位点[28],由于SCp-1与凝血酶的ARG-73结合导致识别位点被占据,凝血酶可能不会与纤维蛋白原结合,从而有效抑制凝血酶参与凝血反应。
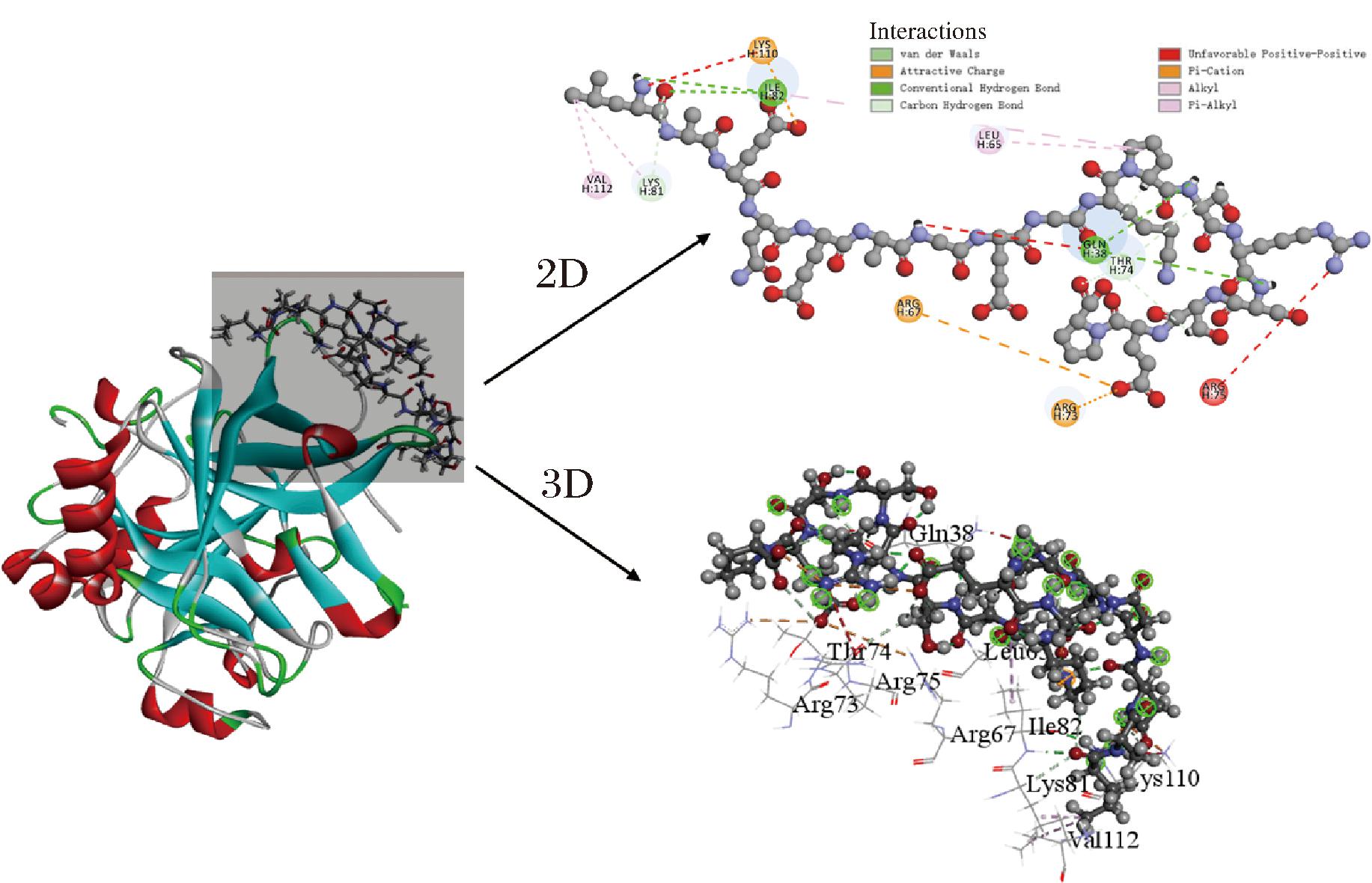
图3 SCp-1与凝血酶分子对接构象
Fig.3 Docking conformation of SCp-1 with thrombin molecule
如图4所示,SCp-2与3RM2中的氨基酸残基形成很强的氢键相互作用,包括典型氢键与非典型氢键2种,典型氢键结合位点有5个,位于LYS-36、GLN-38、THR-74、ILE-82、ASN-78,并在LYS-110、LEU-65、SER-83、MET-84等共5个氨基酸残基位点形成非典型的氢键。上述典型氢键距离短,结合能力强,可以有效促进多肽与凝血酶形成稳定的复合物。该复合物包含了4个由电荷吸引形成的离子键位于ARG-75、ARG-67、LYS-81、TYR-76多种结合键复合作用对锚定凝血蛋白空腔中的活性肽小分子有着重要贡献。值得注意的是,SCp-2能够与LYS-36位点结合,LYS-36是凝血酶识别结合纤维蛋白原的关键位点之一[28],这可能是SCp-2能够抑制凝血酶活性的关键原因。

图4 SCp-2与凝血酶分子对接构象
Fig.4 Docking conformation of SCp-2 with thrombin molecules
2.3 合成肽的体外抗血栓活性验证
2.3.1 体外抗血栓活性验证
为验证2种合成肽的抗血栓活性,本研究测定了4个经典的凝血活性分析指标, 即部分凝血活酶时间、凝血酶时间、凝血酶原时间以及凝血酶抑制率。为了更直观地体现样品对凝血指标的影响效率,以阴性对照组(0.0 mg/mL)为作为基准(100%),计算部分凝血活酶时间、凝血酶时间、凝血酶原时间相对延长比率。此外,为了排除溶剂对凝血指标测量数值的影响,在梯度浓度各组外还设置正常羊血浆组(normal sheep plasma,NSP)用于对照。
部分凝血活酶时间反映内源性通路。单独延长部分凝血活酶时间应考虑内源性因子Ⅷ、Ⅸ、Ⅺ或 Ⅻ缺陷(质量和数量)。如图6-A所示,SCp-1对部分凝血活酶时间的延长效果随梯度浓度增大而显著提升,其中2.0 mg/mL组较阴性对照组的抗凝血效果提升了88.30%。由图7-A可知,SCp-2的部分凝血活酶时间也表现出了显著的剂量依赖趋势,终质量浓度2.0 mg/mL组较阴性对照组凝血时间延长60.07%。2种合成肽在不同浓度下对内源性凝血通路均可产生显著影响。

A-部分凝血活酶时间延长率;B-凝血酶原时间延长率;C-凝血酶时间延长率;D-凝血酶抑制率
图6 SCp-1的体外活性验证
Fig.6 In vitro activity validation of SCp-1

A-部分凝血活酶时间延长率;B-凝血酶原时间延长率;C-凝血酶时间延长率;D-凝血酶抑制率
图7 SCp-2的体外活性验证
Fig.7 In vitro activity validation of SCp-2
凝血酶原时间反映外源性通路。单纯的凝血酶原时间延长通常是由于因子Ⅻ在质量与数量方面的缺陷所导致。由图6-B以及图7-B 可以看出,2种合成肽在不同浓度下对外源性凝血通路均无法产生显著性的影响。
凝血酶时间和凝血酶抑制率二者均反映凝血级联的共同通路。作为共同通路的因子,因子 V、X、Ⅱ和 I 的缺陷(质量和数量)均可延长部分凝血活酶时间和凝血酶时间[6]。这是凝血途径的最后一步,即通过凝血酶的作用将纤维蛋白原转化为纤维蛋白。因此,凝血酶时间对纤维蛋白原和药物如直接和间接凝血酶抑制剂的缺陷十分敏感。二者区别之处在于凝血酶时间测定环境为血浆复杂体系,而凝血酶抑制率的测定环境为单纯的凝血酶,纤维蛋白原和抑制剂存在的相对单一体系。如图6-C与图6-D所示,在0~1.0 mg/mL质量浓度范围内,SCp-1的凝血酶时间与凝血酶抑制率呈现相似的剂量依赖趋势,但质量浓度>1.0 mg/mL的组中凝血酶时间提升趋势并不显著。由图7-C可知,SCp-2的凝血酶时间指标在0~2.0 mg/mL剂量范围内表现为剂量依赖,其中 2.0 mg/mL组对凝血酶时间的提升抗凝血效果达48.36%。不同来源的食源性抗血栓肽有不同的活性表现,XU等[29]报道2.78 mg/mL的大豆蛋白抗凝血肽体外凝血酶抑制率为50 %,与之相比SCp-1 与SCp-2对凝血酶的抑制活性更强。
通过对比分析以上4个关键性凝血指标发现,SCp-1以及SCp-2均体现出可观的内源性抗凝活性以及共同通路的抗凝活性。对不同通路的抗凝活性的强弱比较可知,SCp-1在内源性通路抗凝能力较强,SCp-2则在共同通路表现出更好的抗凝活性。鉴于部分凝血活酶时间,凝血酶时间以及凝血酶抑制率指标仅能反映内源性通路与共同通路的表观结果,所以二者表现出不同的抗凝活性,可能是存在对通路因子特异性反应差异所导致。虽然已知二者均能与凝血酶分子结合,但SCp-1以及SCp-2对凝血级联反应中各凝血因子的分子作用机制仍待进一步深入探究。
3 结论
采用酶解技术从玉足海参中获得其高多肽丰度酶解液,从海参酶解液种鉴定出共计1 796种多肽,凝血酶抑制活性测定结果表明,发现1.0 mg/mL质量浓度下,1~3 kDa组分的凝血酶抑制率最优,选取116条相对分子质量>1 kDa、<3 kDa的多肽建立候选肽库,并从肽库的净电荷、亲疏水性、脂肪族系数、不稳定性指数4种特征与抗血栓活性肽特征高度相似,确定肽库建立的有效性。利用分子对接筛选出 6条未经报道且对接评分优于重组水蛭素片段的多肽,选取其中2条潜力最佳的肽序列进行合成,体外活性验证,实验结果表明,SCp-1与SCp-2在凝血级联反应的内源性途径以及共同途径均具有显著的抗血栓活性,提供了一种快速高效的筛选抗血栓活性肽的方法,为食源性抗血栓活性肽的筛选和开发提供了参考。
[1] World Heart Report 2023:Confronting the World′s Number One Killer[R].World Heart Federation.Geneva, Switzerland.2023.
[2] UCEDA-MARTIN M, LAMBERT A, MIREMONT G, et al.Bullous haemorrhagic dermatitis induced by heparins and other anticoagulants:94 cases from French pharmacovigilance centres and a literature review[J].Annales de Dermatologie et de Vénéréologie, 2022, 149(1):45-50.
[3] VERMA K, REAVEY-CANTWELL J, CAMERON B M.Apixaban-associated spontaneous thoracic intramedullary hemorrhage[J].Clinical Neurology and Neurosurgery, 2021, 202:106512.
[4] TIGLIOGLU M, AKYOL P, SAGLAM B, et al.Thrombocytopenia due to rivaroxaban:A rare adverse effect[J].Transfusion and Apheresis Science, 2020, 59(6):102883.
[5] TU M L, XU S Q, XU Z, et al.Identification of dual-function bovine lactoferrin peptides released using simulated gastrointestinal digestion[J].Food Bioscience, 2021, 39:100806.
[6] CHENG S Z, TU M L, LIU H X, et al.A novel heptapeptide derived from Crassostrea gigas shows anticoagulant activity by targeting for thrombin active domain[J].Food Chemistry, 2021, 334:127507.
[7] 李汉琪, 王治军, 郑清瑶, 等.食源性抗血栓活性肽的研究进展[J].食品科学, 2023, 44(17):268-277.LI H Q, WANG Z J, ZHENG Q Y, et al.Research progress on foodborne antithrombotic peptides[J] Food Science, 2023, 44(17):268-277.
[8] 杨隆恩, 张永平, 张才, 等.海参酶解液对角叉菜胶所致小鼠血栓形成的抑制作用[J].广东海洋大学学报, 2018, 38(4):57-62.YANG L E, ZHANG Y P, ZHANG C, et al.Inhibitory effect of sea cucumber enzymatic hydrolysate on carrageenan-induced thrombus formation in mice[J].Journal of Guangdong Ocean University, 2018, 38(4):57-62.
[9] 李亚娟, 王佳佳, 张才, 等.海参酶解液对斑马鱼糖尿病并发抑郁样行为的改善作用[J].广东海洋大学学报, 2019, 39(1):84-89.LI Y J, WANG J J, ZHANG C, et al.Effect of sea cucumber hydrolysis extract on depression-like behavior of zebrafish induced by diabete[J] Journal of Guangdong Ocean University, 2019, 39(1):84-89.[10] WANG Q Q, SHI J Y, ZHONG H, et al.High-degree hydrolysis sea cucumber peptides improve exercise performance and exert antifatigue effect via activating the NRF2 and AMPK signaling pathways in mice[J].Journal of Functional Foods, 2021, 86:104677.
[11] ZHANG J, LIU X, WANG Y K, et al.Transcriptome and metabolome analyses reveal gender-specific expression genes in sea cucumber (Holothuria leucospilota)[J].Comparative Biochemistry and Physiology Part D:Genomics and Proteomics, 2023, 47:101117.
[12] WEN J P, BO M, YANG L,et al. Developing artificial mixed diets for larval culture of sea cucumber, Holthuria leucospilota, and their effects on the internal microbiota [J]. Aquaculture Reports,2023,33:101868.
[13] 郗亚凡, 林海生, 曹文红, 等.三种南海礁栖海参体壁营养成分分析[J].广东海洋大学学报, 2023, 43(2):113-119.XI Y F, LIN H S, CAO W H,et al.Analysis of nutrient composition in the body wall of three south china sea reef dwelling sea cucumbers[J] Journal of Guangdong Ocean University, 2023, 43(2):113-119.
[14] CAI L, WU S W, JIA C G, et al.Active peptides with hypoglycemic effect obtained from hemp (Cannabis sativa L.) protein through identification, molecular docking, and virtual screening[J].Food Chemistry, 2023, 429:136912.
[15] CUI F C, XI L Q, ZHAO G Q, et al.Screening of xanthine oxidase inhibitory peptides by ligand fishing and molecular docking technology[J].Food Bioscience, 2022, 50:102152.
[16] MAO Z J, JIANG H, SUN J N, et al.Virtual screening and structure optimization of xanthine oxidase inhibitory peptides from whole protein sequences of Pacific white shrimp via molecular docking[J].Food Chemistry, 2023, 429:136837.
[17] ZHU J Y, SUN D, LI X T, et al.Developing new PI3Kγ inhibitors by combining pharmacophore modeling, molecular dynamic simulation, molecular docking, fragment-based drug design, and virtual screening[J].Computational Biology and Chemistry, 2023, 104:107879.
[18] CHENG S Z, WANG Y W, CHEN H, et al.Anticoagulant dodecapeptide suppresses thrombosis in vivo by inhibiting the thrombin exosite-I binding site[J].Journal of Agricultural and Food Chemistry, 2021.63(37):10920-10931
[19] RIVERO-PINO F, MILLAN-LINARES M C, MONTSERRAT-DE-LA-PAZ S.Strengths and limitations of in silico tools to assess physicochemical properties, bioactivity, and bioavailability of food-derived peptides[J].Trends in Food Science &Technology, 2023, 138:433-440.
[20] MATTHEWS J H, KRISHNAN R, COSTANZO M J, et al.Crystal structures of thrombin with thiazole-containing inhibitors:Probes of the S1′ binding site[J].Biophysical Journal, 1996, 71(5):2830-2839.
[21] TABERNO L, CHANG C Y, OHRINGER S L, et al.Structure of a retro-binding peptide inhibitor complexed with human α-thrombin[J].Journal of Molecular Biology, 1995, 246(1):14-20.
[22] DWIVEDI R, POMIN V H.Marine antithrombotics[J].Marine Drugs, 2020, 18(10):514.
[23] JACQUES S L, LEMASURIER M, SHERIDAN P J, et al.Substrate-assisted catalysis of the PAR1 thrombin receptor:Enhancement of macromolecular association and cleavage[J].Journal of Biological Chemistry, 2000, 275(52):40671-40678.
[24] LIU L W, VU T K, ESMON C T, et al.The region of the thrombin receptor resembling hirudin binds to thrombin and alters enzyme specificity[J].Journal of Biological Chemistry, 1991, 266(26):16977-16980.
[25] CHENG S Z, TU M L, LIU H X, et al.Food-derived antithrombotic peptides:Preparation, identification, and interactions with thrombin[J].Critical Reviews in Food Science and Nutrition, 2019, 59:S81-S95.
[26] BOURDON P, JABLONSKI J-A, CHAO B H, et al.Structure-function relationships of hirulog peptide interactions with thrombin[J].FEBS Letters, 1991, 294(3):163-166.
[27] CHEN F Y, HUANG G R.Mechanism and inhibition kinetics of peptide P13 as thrombin inhibitor[J].International Journal of Biological Macromolecules, 2020, 150:1046-1052.
[28] BODE W.Structure and interaction modes of thrombin[J].Blood Cells, Molecules, and Diseases, 2006, 36(2):122-130.
[29] XU R, HUANG Y B, HOU Y, et al.Isolation and identification of thrombin-inhibiting peptides derived from soybean protein[J].Food Biotechnology, 2022, 36(2):154-172.