共轭亚油酸(conjugated linoleic acid, CLA)是具有共轭双键的十八碳二烯酸的总称,是亚油酸(linoleic acid, LA)的位置异构体[1]。CLA最先在牛、羊等反刍动物的脂肪组织中发现[2-4],研究表明具有多种生物活性,如抗癌[5],减肥[6],抗炎[7],抗动脉粥样硬化[8],抗Ⅱ型糖尿病[9],抗过敏[10]等生理功能,因此CLA是被广泛研究的功能性脂肪酸之一,应用于保健品、药物、食品等领域。天然的CLA主要来源于乳制品[11]、反刍动物肉[12]、植物油[13],但含量较低,人们日常饮食摄入量远远低于成人推荐摄入量3.2 g/d[14],需要进行额外的摄入补充。
CLA合成方法主要有化学合成法和微生物合成法,化学法虽产量大,效率较高,但其产物异构体复杂,分离纯化困难,而微生物法具有条件温和,产物单一,无污染等优点。微生物合成法中研究最多的菌种是乳酸菌(lactic acid bacteria, LAB),其不仅生物转化效率高,转化产物与天然食物中CLA异构体组成相似,且是公认安全的微生物[15],良好的益生性使其广泛应用于食品行业。
现研究发现转化生成CLA的乳酸菌所利用的LA形式大多数是游离形式,然而自然界中LA主要以甘油酯形式存在。相较于游离形式,甘油酯型LA具有稳定性更好,生物利用度更高等特点,且单、双甘油酯亚油酸可作为食品添加剂广泛应用于食品中。为提高自然界中亚油酸的利用率以及解决游离亚油酸应用范围较窄等问题,本研究首先筛选了以结合态亚油酸为底物转化生成CLA的乳酸菌,结果显示8株乳酸菌具有较强的转化亚油酸单甘油酯为CLA的能力。在此基础上,为探究菌株可以转化亚油酸单甘油酯的原因,本文通过分析发酵过程中脂肪酸形式变化情况,预测并通过生信分析获得短双歧杆菌CCFM683中编码脂肪酶的候选基因序列,进一步克隆短双歧杆菌CCFM683的脂肪酶候选基因,并在大肠杆菌中进行功能鉴定,证实在短双歧杆菌CCFM683中存在可以特异性将亚油酸单甘油酯水解成游离亚油酸的脂肪酶。本研究筛选获得了能利用结合态亚油酸转化生成CLA的菌株,并在以结合态亚油酸为底物转化CLA的菌株中筛选到编码脂肪酶的基因序列,解释了菌株转化结合态亚油酸为CLA的原因,为后续从基因层面筛选以结合态亚油酸转化CLA的菌株提供新的思路。
1 材料与方法
1.1 材料与试剂
22株乳酸菌、大肠杆菌(Escherichia coli) Top10、E.coli BL21,江南大学食品生物技术中心菌种库;表达载体pET28a(+),Invitrogen公司。
亚油酸单甘油酯标准品、亚油酸甘油二酯标准品、亚油酸甘油三酯标准品、十五烷酸单甘油酯标准品、十七烷酸标准品、十九烷酸甘油二酯标准品、二十一烷酸甘油三酯标准品(纯度均≥99%),NU-CHEK公司;异丙基硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside, IPTG),上海生工生物工程公司;实验中引物和基因测序由苏州金唯智生物科技有限公司完成。
1.2 仪器与设备
SX-300高压蒸汽灭菌锅,日本Tomy Seiko有限公司;厌氧工作站,英国Electrotek 400TG公司;Sorvall ST40R台式冷冻离心机,美国赛默飞世尔科技公司;W-CJ-1FD超净工作台,苏州净化设备有限公司;GCMS-QP2010 Ultra气质联用仪,日本岛津公司,CNWBOND氨基SPE固相萃取柱(500 mg/6 mL),上海安谱实验科技股份有限公司;ND100-2氮气吹扫仪,杭州瑞诚;Tanon化学发光图像分析系统,上海天能科技有限公司。
1.3 试验方法
1.3.1 以结合态亚油酸为底物转化生成CLA乳酸菌筛选
菌株活化与培养:取出保存于-80 ℃的菌株在MRS固体培养基上划线,37 ℃厌氧培养48 h后,挑单菌落于MRS液体培养基中,37 ℃厌氧培养24 h,连续活化3代。将活化的菌体按照2%(体积分数)的接种量分别接种于含有质量浓度为0.3 mg/mL亚油酸甘油酯的MRS培养基中37 ℃厌氧培养48 h。
脂肪酸提取:取1 mL菌液于提脂瓶中,准确加入50 μL内标(十五烷酸单甘油酯标准品,十七烷酸标准品,十九烷酸甘油二酯标准品,二十一烷酸甘油三酯标准品,质量浓度均为2 mg/mL),充分振荡1 min。采用氯仿甲醇法提取总脂肪酸,在菌液中加入2 mL氯仿和1 mL甲醇,充分振荡3~5 min,后2 000 r/min离心10 min,吸取下层至新提脂瓶中,再向菌液中加入1 mL氯仿,重复上述步骤,吸取下层至新提脂瓶中,氮气吹干。
脂肪酸形式分离测定:参考文献[16],略有改动。将所提取脂肪酸氮吹后溶于500 μL的正己烷中,转移至氨基SPE固相萃取柱中,分别用不同的洗脱液冲洗固相萃取柱,用V(正己烷)∶V(氯仿)∶V(乙酸乙酯)=100∶5∶5洗脱上述固相萃取柱后收集液中为甘油三酯,用V(正己烷)∶V(乙酸乙酯)=85∶15洗脱收集液中为甘油二酯,用V(氯仿)∶V(甲醇)=2∶1洗脱收集液中为单甘油酯,用V(乙醚)∶V(冰乙酸)=98∶2洗脱收集液中为游离脂肪酸,将收集液用氮气吹干。
脂肪酸衍生化:采用硫酸甲醇法衍生化,向上述装有洗脱液提脂瓶中加入1 mL 2%(体积分数)硫酸甲醇溶液,充分振荡,70 ℃水浴4 h,后加入饱和NaCl溶液和正己烷各1 mL,充分振荡后2 000 r/min离心10 min,吸取上层正己烷层于气相瓶中进行GC-MS检测。
脂肪酸分析检测:脂肪酸甲酯采用气质联用仪检测。气相色谱柱为Rtx-WAX(30 m×0.25 mm,φ 0.25 μm),程序升温条件:初始温度150 ℃,保持2 min 后以10 ℃/min的速度升温至190 ℃,保持4 min,后以5 ℃/min的速度升温至220 ℃,保持6 min。载气为氦气,采用的进样方式为分流进样。根据文献中共轭亚油酸的质谱碎片进行定性。根据内标采用峰面积归一化法计算脂肪酸含量[17]。
1.3.2 脂肪酶(lipase)异源表达及功能验证
菌株序列比对及筛选:根据NCBI中已鉴定功能的不同来源脂肪酶基因序列(表1)与乳酸菌全基因组序列之间比对。使用Jalview软件分析候选序列与上述模板序列之间的一致性。
表1 不同物种来源的Lipase基因信息
Table 1 Lipase gene information from different species

来源宿主 cDNA编号 Komagataella phaffiiCAH2449117.1Homo sapiensQ99685.2Galemys pyrenaicusKAG8516963.1Mycolicibacterium smegmatisABK74423.1Sus scrofaNP_001137190.1
候选基因Lipase克隆及表达载体构建:利用pET28α(+)作为载体,以乳酸菌基因组为模板,通过PCR扩增目的基因,PCR引物如表2所示,pET28α(+)载体经对应的限制性内切酶酶切2 h后,通过同源重组将扩增产物与pET28α(+)载体连接,后通过化学转化法将连接产物导入E.coli TOP10。通过对转化子菌落PCR验证,测序验证是否为正确转化子,将正确转化子保存于-80 ℃。
表2 PCR引物信息
Table 2 PCR primer information

引物引物序列(5′-3′)pET28α(+)-His-LipaseA FCTCCGTCGACAAGCTATGGCACAGC-CGCAGCpET28α(+)-His-LipaseA RGGTGGTGGTGCTCGATCAGACATG-CAGGAGCGCpET28α(+)-LipaseA-His FAGGAGATATACCATGATGGCACAGC-CGCAGpET28α(+)-LipaseA-His RGACGGAGCTCGAATTGACATGCAG-GAGCGCCACpET28α(+)-His-LipaseB FCTCCGTCGACAAGCTATGCAGGT-GACGATGATCGACGpET28α(+)-His-LipaseB RGGTGGTGGTGCTCGATCAGTACGTG-GCGCTGGpET28α(+)-LipaseB-His FAGGAGATATACCATGATGCAGGT-GACGATGATCGACpET28α(+)-LipaseB-His RGACGGAGCTCGAATTGTACGTG-GCGCTGGCAA
大肠杆菌重组菌诱导表达:将测序正确的重组质粒和空载体pET28α(+)通过化学转化法转入E.coli BL21中,通过PCR验证转化子,将正确的转化子在含Kana抗性的LB板上划线培养出单菌落,活化两代后接入50 mL的LB液体培养基中,在37 ℃培养箱中培养至OD600=0.6~0.8,添加诱导剂IPTG至终浓度为0.1 mmol/L后16 ℃培养18~20 h。诱导结束后6 000 r/min离心10 min,收集菌体。将菌体重悬于PBS溶液,加入适量的玻璃珠,通过组织破碎仪振荡破碎,获得粗蛋白,用于后续Western Blot检测及活性分析。
脂肪酶粗酶液对不同底物催化能力分析[18]:取上述1 mg粗蛋白,加入亚油酸甘油酯至终质量浓度0.3 mg/mL,添加PBS缓冲液定容至1 mL,于37 ℃反应20 min,加入氯仿终止反应。后按照1.3.1节方法进行脂肪酸测定反应体系中游离亚油酸和亚油酸甘油酯浓度。
2 结果与分析
2.1 以结合态亚油酸为底物转化生成CLA的乳酸菌筛选
实验室前期获得了22株转化率相对较高的以游离亚油酸和葵花籽油为底物转化生成CLA的菌株,其中包括短双歧杆菌、植物乳杆菌、戊糖乳植杆菌、干酪乳杆菌、嗜酸乳杆菌、卷曲乳杆菌、乳酸乳球菌乳酸亚种、瘤胃乳杆菌[19]。在此基础上,以这22株菌为研究对象,以亚油酸单甘油酯、亚油酸甘油二酯、亚油酸甘油三酯作为底物,按照1.3.1节方法测定发酵液中CLA含量,如表3所示,当底物为亚油酸单甘油酯时,共有8株菌(短双歧杆菌CCFM683、短双歧杆菌FZJHZD20M12、短双歧杆菌UCC2003、短双歧杆菌FFJND12M6、短双歧杆菌CJ-9-5-1、戊糖乳植杆菌FHuNHHMY67L3、植物乳杆菌DQHXNS9L9-1、植物乳杆菌DXJSLMS1M11-1)CLA产量>65 μg/mL,转化率>20%,其中短双歧杆菌CCFM683的CLA产量最高,达到270 μg/mL,转化率在96%以上。而由表4、表5可知,以亚油酸甘油二酯和亚油酸甘油三酯为底物时,筛选的菌株CLA产量较低,转化率均<10%。综上,乳酸菌中存在可以利用亚油酸单甘油酯为底物转化生成CLA的菌株,且有这8株菌转化生成CLA的能力较强。
表3 乳酸菌以亚油酸单甘油酯为底物发酵培养后发酵液中CLA含量及转化率
Table 3 CLA content and conversion rate in the fermentation broth following fermentation by lactic acid bacteria using linoleic acid monoglyceride as the substrate

菌株c9,t11-CLA/(μg/mL)t10,c12-CLA/(μg/mL)t9,t11-CLA/(μg/mL)总CLA转化率/%短双歧杆菌CCFM683116.15±6.6418.49±0.56106.87±4.7896.68FZJHZD20M12173.81±6.8517.94±0.4575.06±2.4996.52UCC2003139.25±14.2416.21±0.9848.47±3.4767.80FFJND12M6118.55±27.3214.37±1.8955.54±3.1162.45CJ-9-5-153.93±10.8110.86±0.8223.78±3.5428.63戊糖乳植杆菌FHuNHHMY67L329.79±0.758.73±0.0828.47±3.2622.25植物乳杆菌DQHXNS9L9-134.00±6.077.56±0.3462.61±2.9534.59DXJSLMS1M11-141.81±1.248.32±0.2152.98±0.4134.24VSCDJY12L15.64±0.383.17±0.416.99±0.605.25FCQHC25L42.62±0.153.02±0.034.10±0.353.24LABCC IMAU10120 P-81.11±0.091.12±0.034.42±0.082.21DHuNHHMY2L11.34±0.091.97±0.153.22±0.042.17VJLHD11L10.29±0.09ND2.87±0.621.05干酪乳杆菌DNMHLBE5-L104.84±0.405.16±0.205.01±0.274.98DQHXNQ38-35.19±0.364.95±0.474.75±0.444.94DQHXNQ19M204.26±0.104.78±0.114.42±0.084.46HN13-14.68±0.414.13±0.103.39±0.064.05嗜酸乳杆菌FNMGHHHT12L404.30±0.014.70±0.144.08±0.134.34FZJTZ18L254.51±0.274.47±0.393.77±0.114.23卷曲乳杆菌FHNXY64M24.20±0.273.89±0.525.67±0.424.57乳酸乳球菌乳酸亚种DYNDL3434.22±0.442.93±0.456.61±0.744.57瘤胃乳杆菌FJXPY8L14.56±0.024.40±0.134.52±0.304.48
注:ND表示未检出(下同)。
表4 乳酸菌以亚油酸甘油二酯为底物发酵培养后发酵液中CLA含量及转化率
Table 4 CLA content and conversion rate in the fermentation broth following fermentation by lactic acid bacteria using glycerol linoleate as the substrate
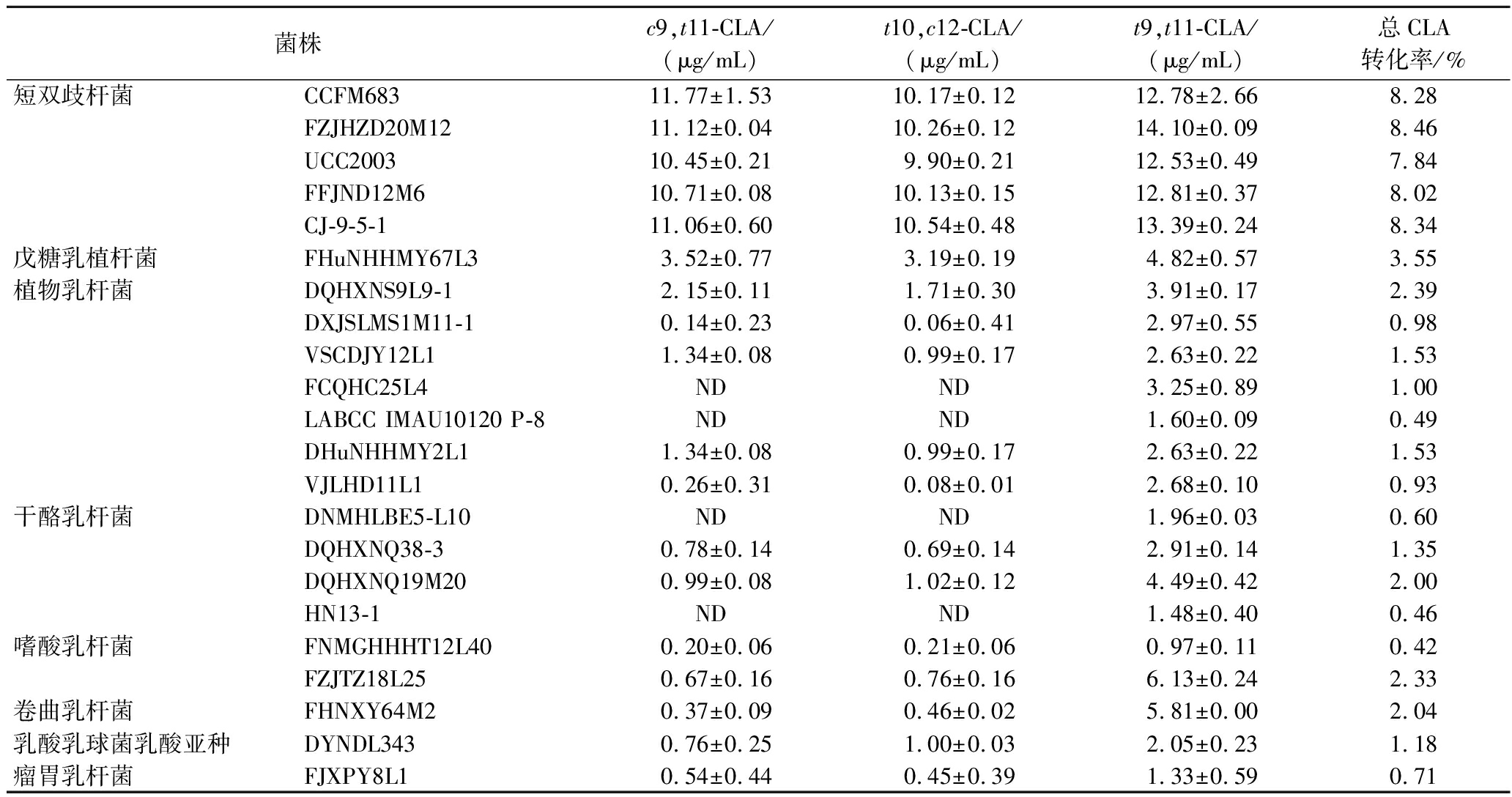
菌株c9,t11-CLA/(μg/mL)t10,c12-CLA/(μg/mL)t9,t11-CLA/(μg/mL)总CLA转化率/%短双歧杆菌CCFM68311.77±1.5310.17±0.1212.78±2.668.28FZJHZD20M1211.12±0.0410.26±0.1214.10±0.098.46UCC200310.45±0.219.90±0.2112.53±0.497.84FFJND12M610.71±0.0810.13±0.1512.81±0.378.02CJ-9-5-111.06±0.6010.54±0.4813.39±0.248.34戊糖乳植杆菌FHuNHHMY67L33.52±0.773.19±0.194.82±0.573.55植物乳杆菌DQHXNS9L9-12.15±0.111.71±0.303.91±0.172.39DXJSLMS1M11-10.14±0.230.06±0.412.97±0.550.98VSCDJY12L11.34±0.080.99±0.172.63±0.221.53FCQHC25L4NDND3.25±0.891.00LABCC IMAU10120 P-8NDND1.60±0.090.49DHuNHHMY2L11.34±0.080.99±0.172.63±0.221.53VJLHD11L10.26±0.310.08±0.012.68±0.100.93干酪乳杆菌DNMHLBE5-L10NDND1.96±0.030.60DQHXNQ38-30.78±0.140.69±0.142.91±0.141.35DQHXNQ19M200.99±0.081.02±0.124.49±0.422.00HN13-1NDND1.48±0.400.46嗜酸乳杆菌FNMGHHHT12L400.20±0.060.21±0.060.97±0.110.42FZJTZ18L250.67±0.160.76±0.166.13±0.242.33卷曲乳杆菌FHNXY64M20.37±0.090.46±0.025.81±0.002.04乳酸乳球菌乳酸亚种DYNDL3430.76±0.251.00±0.032.05±0.231.18瘤胃乳杆菌FJXPY8L10.54±0.440.45±0.391.33±0.590.71
表5 乳酸菌以亚油酸甘油三酯为底物发酵培养后发酵液中CLA含量及转化率
Table 5 CLA content and conversion rate in the fermentation broth following fermentation by lactic acid bacteria using linoleic acid triglyceride as the substrate
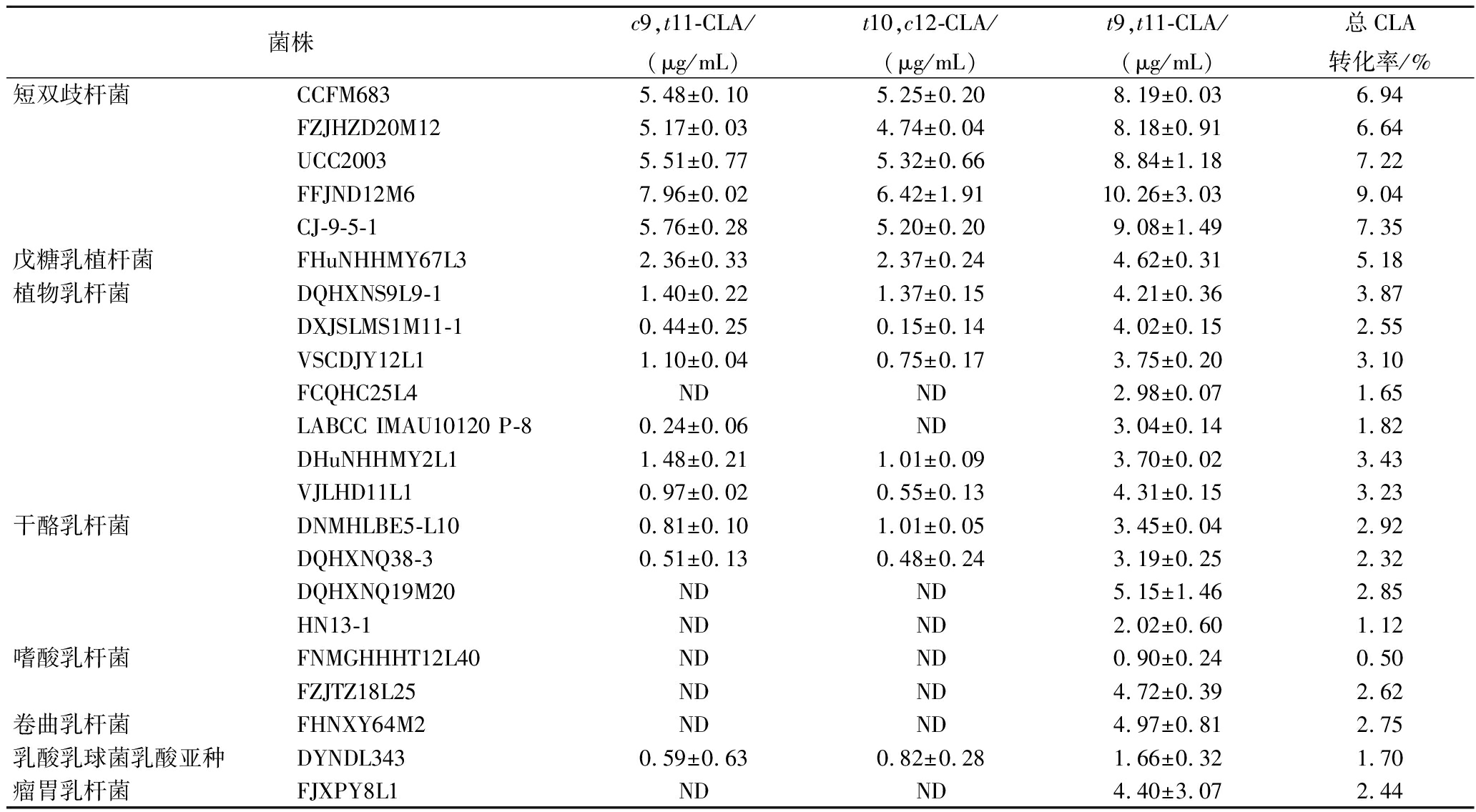
菌株c9,t11-CLA/(μg/mL)t10,c12-CLA/(μg/mL)t9,t11-CLA/(μg/mL)总CLA转化率/%短双歧杆菌CCFM6835.48±0.105.25±0.208.19±0.036.94FZJHZD20M125.17±0.034.74±0.048.18±0.916.64UCC20035.51±0.775.32±0.668.84±1.187.22FFJND12M67.96±0.026.42±1.9110.26±3.039.04CJ-9-5-15.76±0.285.20±0.209.08±1.497.35戊糖乳植杆菌FHuNHHMY67L32.36±0.332.37±0.244.62±0.315.18植物乳杆菌DQHXNS9L9-11.40±0.221.37±0.154.21±0.363.87DXJSLMS1M11-10.44±0.250.15±0.144.02±0.152.55VSCDJY12L11.10±0.040.75±0.173.75±0.203.10FCQHC25L4NDND2.98±0.071.65LABCC IMAU10120 P-80.24±0.06ND3.04±0.141.82DHuNHHMY2L11.48±0.211.01±0.093.70±0.023.43VJLHD11L10.97±0.020.55±0.134.31±0.153.23干酪乳杆菌DNMHLBE5-L100.81±0.101.01±0.053.45±0.042.92DQHXNQ38-30.51±0.130.48±0.243.19±0.252.32DQHXNQ19M20NDND5.15±1.462.85HN13-1NDND2.02±0.601.12嗜酸乳杆菌FNMGHHHT12L40NDND0.90±0.240.50FZJTZ18L25NDND4.72±0.392.62卷曲乳杆菌FHNXY64M2NDND4.97±0.812.75乳酸乳球菌乳酸亚种DYNDL3430.59±0.630.82±0.281.66±0.321.70瘤胃乳杆菌FJXPY8L1NDND4.40±3.072.44
目前研究报道中能转化生成CLA的菌株所利用的LA多为游离形式,而本研究中筛选到能利用亚油酸单甘油酯为底物生物转化CLA的乳酸菌,增添了乳酸菌转化生成CLA的底物形式。亚油酸单甘油酯是一种重要的乳化剂和稳定剂,可以作为食品添加剂在食品工业中应用,使用前景广泛[20]。筛选到以亚油酸单甘油酯为底物转化生成CLA的乳酸菌,为今后乳酸菌发酵生产富含CLA的食品带来新的机遇,并拓宽了亚油酸单甘油酯在食品中的应用范围。
2.2 不同时间点发酵液中脂肪酸形式测定
根据2.1节的结果,短双歧杆菌CCFM683以亚油酸单甘油酯为底物转化CLA能力最强,因此本研究选取短双歧杆菌CCFM683做进一步研究。为了探究菌株能以亚油酸单甘油酯为底物转化生成CLA的原因,确定菌株发酵过程中LA和CLA的形式变化,本文通过固相萃取法和气质联用仪测定发酵液中脂肪酸变化情况,结果如图1所示。在发酵前期0~6 h时,发酵液中亚油酸单甘油酯含量减少,而游离亚油酸含量增多,CLA含量几乎为0,在6~12 h期间,游离亚油酸含量快速减少,此时游离CLA含量增加,而在12 h后,发酵液中几乎没有亚油酸单甘油酯,但存在少量的游离亚油酸。由上述脂肪酸形式随发酵时间变化情况推测,亚油酸单甘油酯首先被水解成游离态LA,后转化生成CLA,本课题组之前研究表明短双歧杆菌CCFM683中存在亚油酸异构酶只能利用游离态LA转化生成CLA[21],因此本研究预测短双歧杆菌CCFM683可能存在脂肪酶,能特异性将亚油酸单甘油酯水解成游离态LA,释放的游离态LA被亚油酸异构酶转化生成CLA。
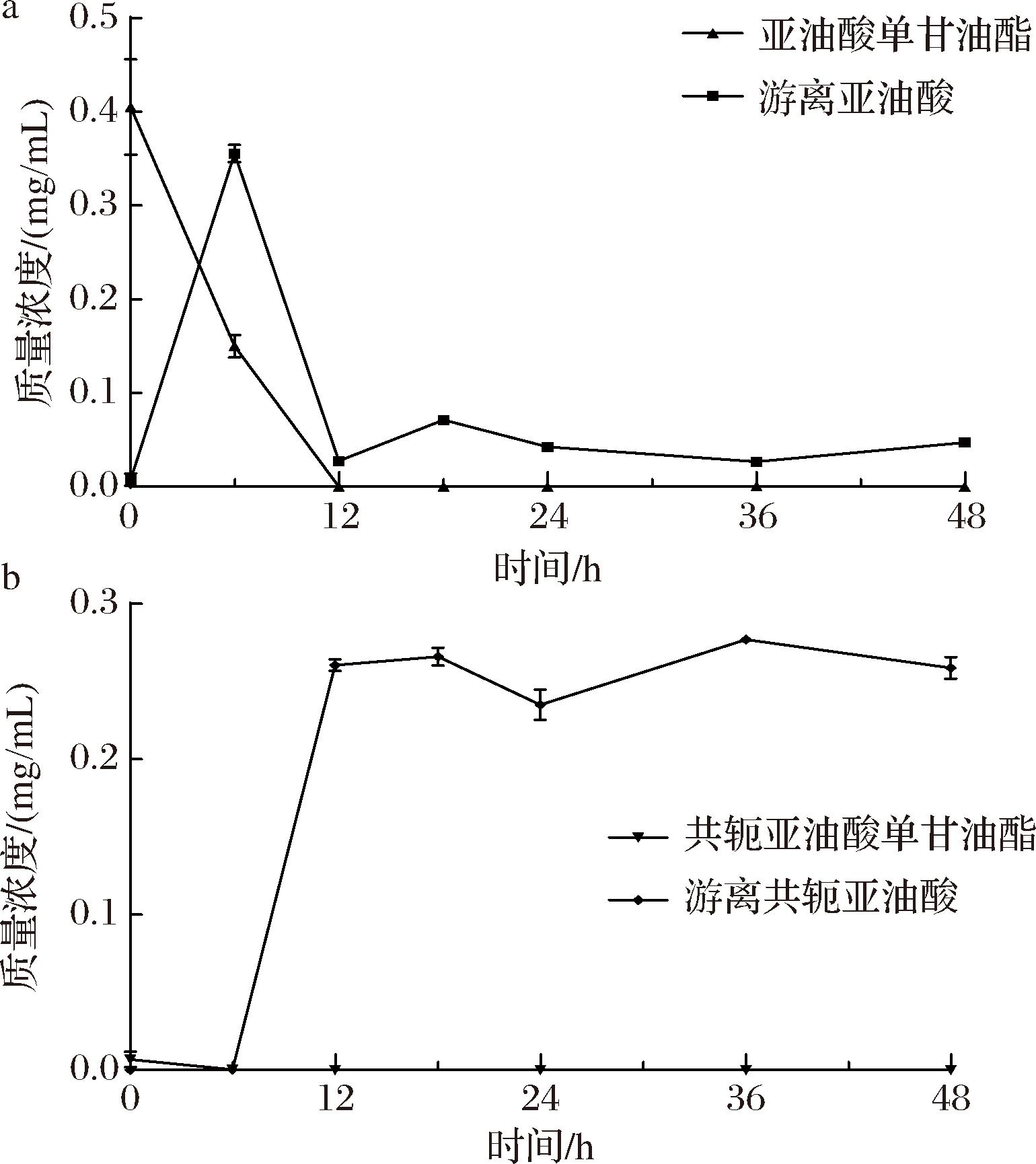
a-亚油酸单甘油酯和游离亚油酸含量;b-共轭亚油酸单甘油酯和游离共轭亚油酸含量
图1 发酵液中单甘油酯脂肪酸和游离脂肪酸随时间变化情况
Fig.1 Fluctuations in monoglyceride fatty acids and free fatty acids over time in the fermentation broth
2.3 短双歧杆菌CCFM683脂肪酶筛选及功能验证
2.3.1 菌株脂肪酶序列比对及筛选
为了证实短双歧杆菌CCFM683中存在能够水解亚油酸单甘油酯的脂肪酶,根据1.3.2节的方法,本研究从已测序的短双歧杆菌CCFM683全基因组中筛选到两段Lipase候选基因序列,并将候选序列分别命名为Lipase A、Lipase B。Lipase A、Lipase B与已鉴定的脂肪酶氨基酸序列比对结果见图2。比对结果显示,Lipase A、Lipase B与不同来源的脂肪酶之间的序列一致性为44.08%、44.90%,其基因序列长度分别为810 bp和1 092 bp,分别编码269和363个氨基酸,图中颜色为深蓝色的氨基酸代表高度相似性的序列。从对比图(图2)中可以看出候选序列与脂肪酶序列之间具有较高的一致性。生物进化过程中,一些重要的基因例如控制结构或功能的部分会保留下来,部分基因会发生突变,因此,如果2组基因序列高度相似,意味着两段序列的功能有相似之处,如果在未知序列中找到与已知序列相似的序列,可根据已知序列对未知序列的功能等进行预测[22]。ALPER等[23]通过基因比对从Rhizopus Oryzae KU45中筛选到脂肪酶高度相似基因序列,经过在大肠杆菌中异源表达,成功验证其具有脂肪酶功能。因此,本研究中对同样和脂肪酶基因序列有相似性的Lipase A/B候选序列进行功能鉴定。

图2 短双歧杆菌CCFM683候选基因Lipase A/B与已鉴定脂肪酶氨基酸序列比对
Fig.2 Comparing the amino acid sequences of the candidate gene Lipase A/B from Bifidobacterium breveCCFM683 with the identified lipases
2.3.2 大肠杆菌重组菌的构建及异源蛋白表达分析
据文献报道,虽然组氨酸标签是常见标签,但是其标签位置可能会对目标蛋白活性有影响,标签若在蛋白表面且远离蛋白催化活性中心,则不会影响目标蛋白的活性,但是若在蛋白催化区域或底物结合区域,则会使目标蛋白活性降低甚至丧失[24],所以本文分别构建了组氨酸标签在N端和C端的pET28α(+)-His-Lipase A/B和pET28α(+)-Lipase A/B-His重组质粒,并导入E.coli Top10中,将测序无误的转化子命名为E.coli Top10-pET28α(+)-His-Lipase A/B和E.coli Top10-pET28α(+)-Lipase A/B-His。从上述保菌的E.coli Top10转化子中提取重组质粒导入E.coli BL21中,将正确的转化子保存并进行诱导表达,并命名为E.coli BL21-pET28α(+)-His-Lipase A/B和E.coli BL21-pET28α(+)-Lipase A/B-His。
本研究使用在线网站Expasy-ProtParam tool预测Lipase A/B加上6×His标签后的分子质量大小分别约为35 kDa和45 kDa。SDS-PAGE和Western-Blot分析结果如图3所示,与预测结果相符,这表明Lipase A/B两者在大肠杆菌E.coli BL21中成功表达。
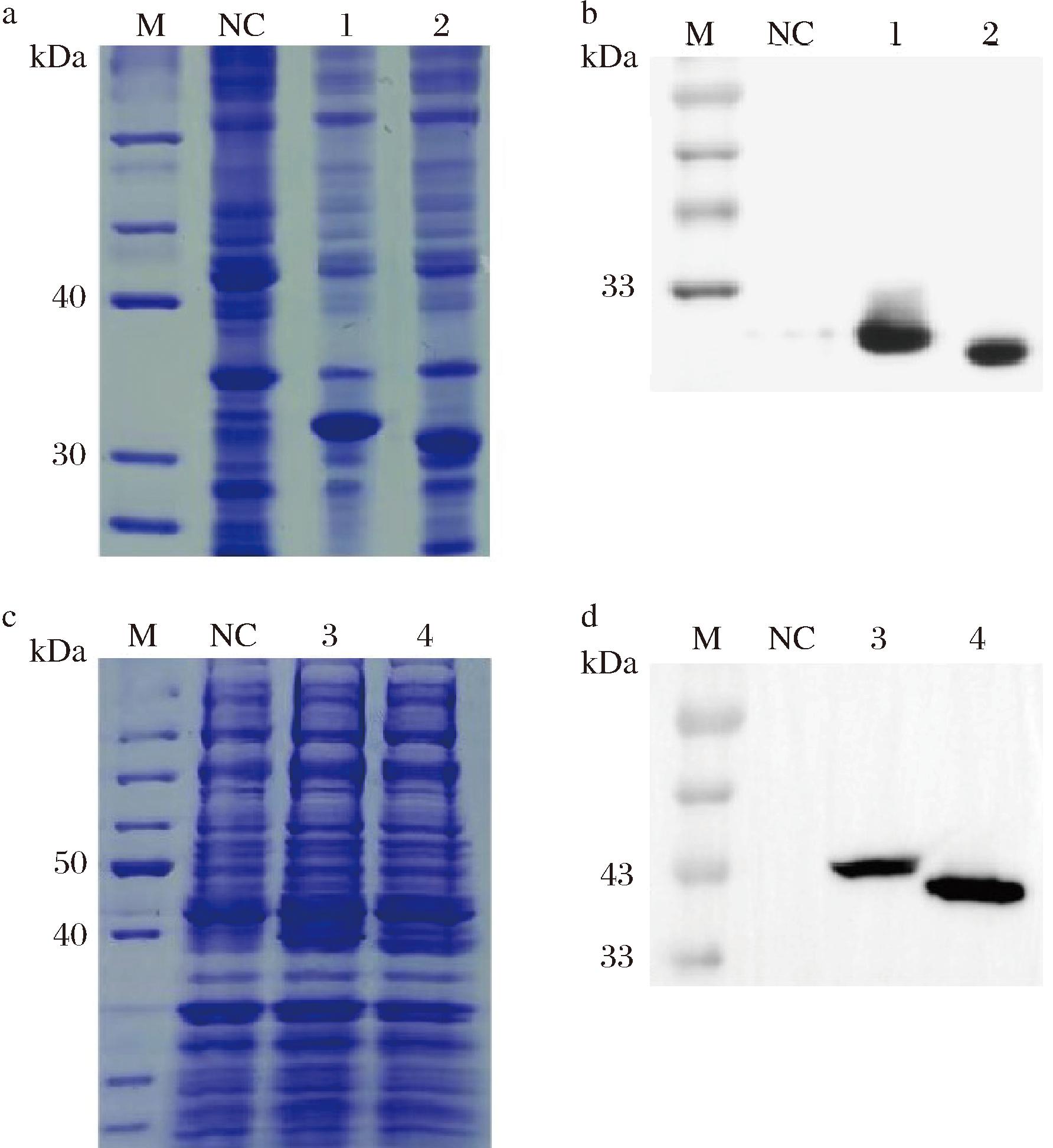
M-Marker;NC-阴性对照菌E.coli BL21-pET28a(+);1-E.coli BL21-pET28α(+)-His-Lipase A;2-E.coli BL21-pET28α(+)-Lipase A-His;3-E.coli BL21-pET28α(+)-His-Lipase B;4-E.coli BL21-pET28α(+)-Lipase B-His a-Lipase A蛋白SDS-PAGE结果;b-Lipase A蛋白Western-Blot结果;c-Lipase B蛋白SDS-PAGE结果;d-Lipase B蛋白Western-Blot结果
图3 短双歧杆菌CCFM683候选基因Lipase A/B目的蛋白诱导表达分析
Fig.3 Analyzing the induced expression of the candidate gene Lipase A/B target protein in Bifidobacterium breve CCFM683
2.3.3 大肠杆菌重组菌对不同形式底物的催化能力分析
为了探究短双歧杆菌CCFM683来源的Lipase在大肠杆菌中异源表达后对各亚油酸甘油酯的水解能力,本研究根据1.3.2节中方法测定各重组菌粗酶液与各亚油酸甘油酯反应后反应体系中亚油酸存在的形式,结果如图4所示。由图4可知,不同组氨酸标签位置对 Lipase 的底物催化能力没有明显的影响。由图4-b、图4-d、 图4-f 结果可知,E.coli BL21-pET28α(+)-His-Lipase B和E.coli BL21-pET28α(+)-Lipase B-His重组菌与导入空载体E.coli BL21-pET28α(+)重组菌相比,其粗酶液与亚油酸单甘油酯反应后游离亚油酸含量均明显增加且亚油酸单甘油酯含量减少,而与亚油酸甘油二酯和亚油酸甘油三酯反应后亚油酸形式未见发生明显变化,由此可见候选基因Lipase B能特异性水解亚油酸单甘油酯。图4-a、图4-c、图4-e显示E.coli BL21-pET28α(+)-His-Lipase A和E.coli BL21-pET28α(+)-Lipase A-His重组菌粗酶液与各亚油酸甘油酯反应后没有明显水解效果。因此,综上结果可以说明Lipase B具有脂肪酶活性,能特异性水解亚油酸单甘油酯。
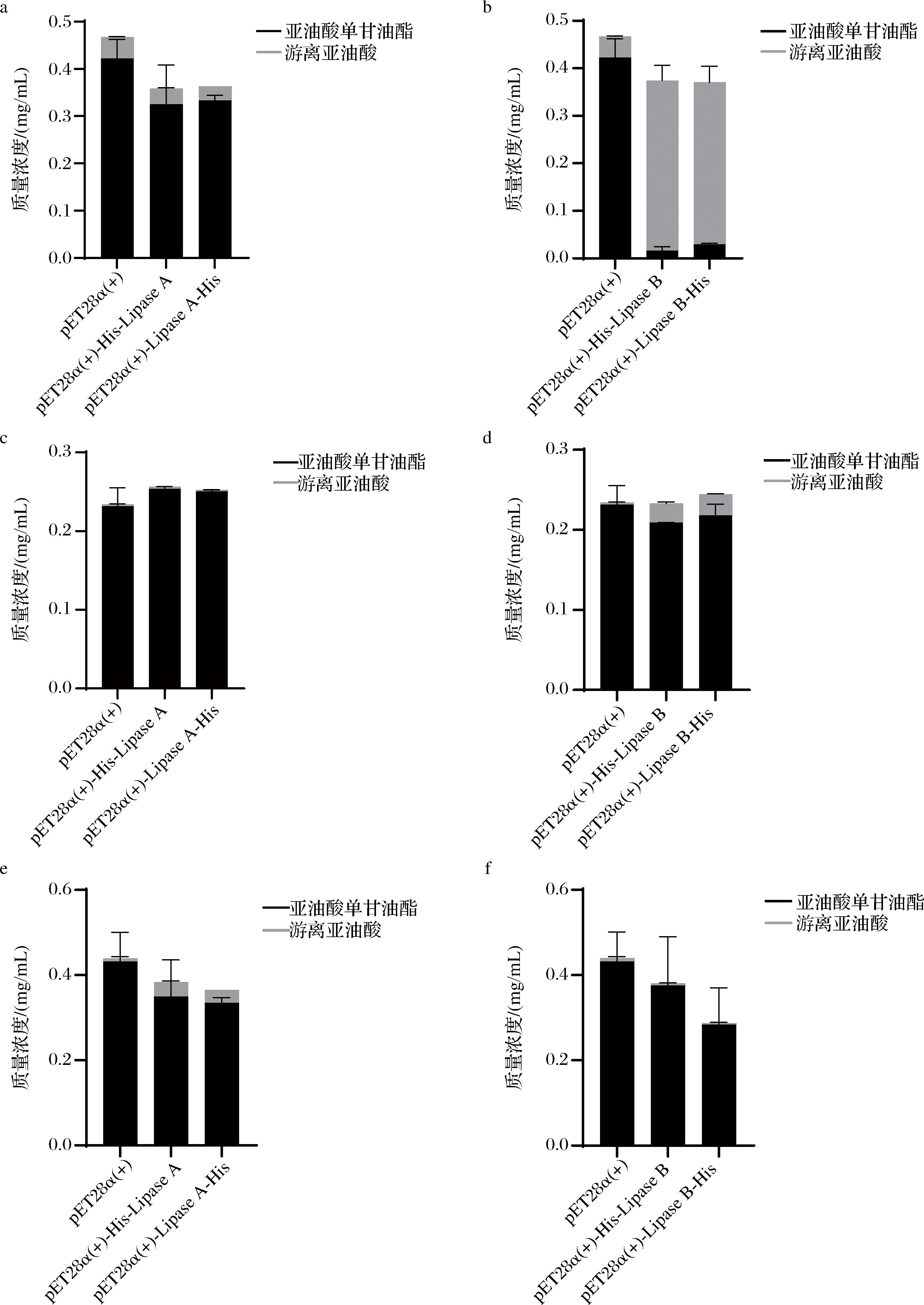
a-Lipase A重组菌与亚油酸单甘油酯反应;b-Lipase B重组菌与亚油酸单甘油酯反应;c-Lipase A重组菌与亚油酸甘油二酯反应;d-Lipase B重组菌与亚油酸甘油二酯反应;e-Lipase A重组菌与亚油酸甘油三酯反应;f-Lipase B重组菌与亚油酸甘油三酯反应
图4 短双歧杆菌CCFM683候选基因Lipase A/B重组菌粗酶液对不同亚油酸甘油酯的水解效果
Fig.4 The hydrolytic effects of the crude enzyme solution from the candidate gene Lipase A/B recombinant bacteria ofBifidobacterium breve CCFM683 on various linoleic glycerides
目前有研究报道从多种微生物中都发现了甘油单酯脂肪酶的存在,该酶能专一作用于甘油单酯,将甘油单酯水解成游离脂肪酸和甘油,但不能水解甘油三酯和甘油二酯。HEIER等[18]鉴定出Yju3p为酵母来源的甘油单酯脂肪酶,酵母中在缺失Yju3p后,甘油单酯水解率明显下降;IMAMURA等[25]从Bacillus sp.H-257鉴定到甘油单酯脂肪酶;TANG等[26]从海洋地衣芽孢杆菌中分离纯化出甘油单酯脂肪酶,在大肠杆菌表达系统中异源表达,并研究了甘油单酯脂肪酶识别及水解底物的过程。
本研究利用基因比对从短双歧杆菌CCFM683全基因组中获得脂肪酶序列具有相似性的基因序列Lipase B,在大肠杆菌中异源表达后,验证具有甘油单酯脂肪酶的功能。2.2节结果显示,短双歧杆菌CCFM683与亚油酸单甘油酯发酵时亚油酸单甘油酯先转变成游离亚油酸后生成CLA,综上结果说明短双歧杆菌CCFM683存在甘油单酯脂肪酶,可以将亚油酸单甘油酯水解成游离亚油酸,随后将游离亚油酸通过亚油酸异构酶转化生成CLA[21]。
目前报道生产甘油单酯脂肪酶的菌株主要为芽孢杆菌、青霉、曲霉和酵母,乳酸菌来源的甘油单酯脂肪酶报道较少,而本研究发现能够以结合态亚油酸为底物生物转化CLA的菌株短双歧杆菌CCFM683中存在甘油单酯脂肪酶,为挖掘新型甘油单酯脂肪酶奠定一定的基础,同时本研究为今后从基因层面筛选以结合态亚油酸为底物转化生成CLA的菌株可以从基因层面筛选提供新的思路。
3 结论
本研究首先分别以不同结合态亚油酸为底物从实验室前期获得的22株乳酸菌中筛选到8株高产CLA的菌株,分别是短双歧杆菌CCFM683、短双歧杆菌FZJHZD20M12、短双歧杆菌UCC2003、短双歧杆菌FFJND12M6、短双歧杆菌CJ-9-5-1、植物乳杆菌DQHXNS9L9-1、植物乳杆菌DXJSLMS1M11-1、戊糖乳植杆菌FHuNHHMY67L3,其中短双歧杆菌CCFM683转化能力最强,以亚油酸单甘油酯为底物生物转化CLA转化率达到96%;进一步研究发现转化能力最强的短双歧杆菌CCFM683发酵过程中亚油酸单甘油酯先被水解成游离亚油酸后转化生成CLA;通过生物信息学分析工具筛选获得短双歧杆菌CCFM683中Lipase相似基因,在大肠杆菌中异源表达进行功能鉴定,其中候选基因Lipase B表现出具有特异性水解亚油酸单甘油酯的功能,证实短双歧杆菌CCFM683中存在甘油单酯脂肪酶。本研究筛选到以亚油酸单甘油酯为底物转化生成CLA的菌株,为提高结合态亚油酸的利用率以及开发富含CLA的发酵食品奠定了基础,通过基因筛选比对以及功能鉴定在高产CLA的菌株中发现甘油单酯脂肪酶的存在为后续实验从基因层面筛选以亚油酸甘油酯转化生成CLA的菌株提供一定参考价值。
[1] YANG B, GAO H, STANTON C, et al.Bacterial conjugated linoleic acid production and their applications[J].Progress in Lipid Research, 2017, 68:26-36.
[2] PARIZA M W, ASHOOR S H, CHU F S.Mutagens in heat-processed meat, bakery and cereal products[J].Food and Cosmetics Toxicology, 1979, 17(4):429-430.
[3] PARIZA M W, LORETZ L J, STORKSON J M, et al.Mutagens and modulator of mutagenesis in fried ground beef[J].Cancer Research, 1983, 43 (5):2444-2446.
[4] HA Y L, GRIMM N K, PARIZA M W.Anticarcinogens from fried ground beef:Heat-altered derivatives of linoleic acid[J].Carcinogenesis, 1987, 8(12):1881-1887.
[5] LAU D S Y, ARCHER M C.The 10t,12c isomer of conjugated linoleic acid inhibits fatty acid synthase expression and enzyme activity in human breast, colon, and prostate cancer cells[J].Nutrition and Cancer, 2010, 62(1):116-121.
[6] MORAES C, OLIVEIRA C A, AMARAL M E, et al.Liver metabolic changes induced by conjugated linoleic acid in calorie-restricted rats[J].Archives of Endocrinology and Metabolism, 2017, 61(1):45-53.
[7] VILADOMIU M, HONTECILLAS R, BASSAGANYA-RIERA J.Modulation of inflammation and immunity by dietary conjugated linoleic acid[J].European Journal of Pharmacology, 2016, 785:87-95.
[8] BRUEN R, FITZSIMONS S, BELTON O.Atheroprotective effects of conjugated linoleic acid[J].British Journal of Clinical Pharmacology, 2017, 83(1):46-53.
[9] BALCI YUCE H, AKBULUT N, OCAKLI S, et al.The effect of commercial conjugated linoleic acid products on experimental periodontitis and diabetes mellitus in Wistar rats[J].Acta Odontologica Scandinavica, 2017, 75(1):21-29.[10] NAKANISHI T, TOKUNAGA Y, YAMASAK M, et al.Orally administered conjugated linoleic acid ameliorates allergic dermatitis induced by repeated applications of oxazolone in mice[J].Animal Science Journal, 2016, 87(12):1554-1561.
[11] CICOGNINI F M, ROSSI F, SIGOLO S, et al.Contents of conjugated linoleic acid isomers cis9, trans11 and trans10, cis12 in ruminant and non-ruminant meats available in the Italian market[J].Italian Journal of Animal Science, 2014, 13(2):3125.
[12] PIMENTEL L L, FONTES A L, SALSINHA A S, et al.Microbiological in vivo production of CLNA as a tool in the regulation of host microbiota in obesity control[J].Studies in Natural Products Chemistry, 2019, 61:369-94.
[13] LIU S L, YU B K, WANG Z G, et al.Highly selective isomerization of cottonseed oil into conjugated linoleic acid catalyzed by multiwalled carbon nanotube supported ruthenium[J].RSC Advances, 2019, 9(36):20698-20705.
[14] KUMARI S, YONG MENG G, EBRAHIMI M.Conjugated linoleic acid as functional food in poultry products:A review[J].International Journal of Food Properties, 2017, 20(3):491-506.
[15] TAGLIAVIA M, NICOSIA A.Advanced Strategies for food-grade protein production:A new E.coli/lactic acid bacteria shuttle vector for improved cloning and food-grade expression[J].Microorganisms, 2019, 7(5):116.
[16] AGREN J J, JULKUNEN A, PENTTIL I.Rapid separation of serum lipids for fatty acid analysis by a single aminopropyl column[J].Journal of Lipid Research, 1992, 33(12):1871-1876.
I.Rapid separation of serum lipids for fatty acid analysis by a single aminopropyl column[J].Journal of Lipid Research, 1992, 33(12):1871-1876.
[17] 杨波. 乳酸菌生物转化共轭亚油酸的研究[D].无锡:江南大学, 2014.YANG B.Study of conjugated linoleic acid bioconversion in lactic acid bacteria[D].Wuxi:Jiangnan University, 2014.
[18] HEIER C, TASCHLER U, RENGACHARI S, et al.Identification of Yju3p as functional orthologue of mammalian monoglyceride lipase in the yeast Saccharomyces cerevisiae[J].Biochimica et Biophysica Acta (BBA), 2010, 1801(9):1063-1071.
[19] 陈科学, 王欣, 夏嘉祎, 等.以葵花籽油为底物生物转化共轭亚油酸双歧杆菌的筛选[J].中国油脂, 2021, 46(1):128-131;145.CHEN K X, WANG X, XIA J Y, et al.Screening of conjugated linoleic acid producing bifidobacteria with sunflower seed oil as substrate[J].China Oils and Fats, 2021, 46(1):128-131;145.
[20] 邹祺. 单脂肪酸甘油酯的合成与应用[D].南京:南京理工大学, 2006.ZOU Q.Synthesizing and applying single fatty acid glycerides[D].Nanjing:Nanjing University of Science &Technology, 2006.
[21] 高鹤. 双歧杆菌生物转化共轭亚油酸的机制研究[D].无锡:江南大学, 2021.GAO H.Mechanism for conjugated linoleic acid transformation by bifidobacteria[D].Wuxi:Jiangnan University, 2021.
[22] 刘欢. 生物多序列比对方法及其关键技术研究[D].合肥:中国科学技术大学, 2023.LIU H.Study on alignment method of biological multiple sequences and key technologies[D].Hefei:University of Science and Technology of China, 2023.
[23] ![]() A,
A, ![]() Ç L.Gene cloning, heterologous expression and biochemical characterization of a novel extracellular lipase from Rhizopus oryzae KU45[J].Iranian Journal of Biotechnology, 2020, 18(2):e2343.
Ç L.Gene cloning, heterologous expression and biochemical characterization of a novel extracellular lipase from Rhizopus oryzae KU45[J].Iranian Journal of Biotechnology, 2020, 18(2):e2343.
[24] 马君燕, 谭海东, 王文霞, 等.组氨酸标签位置对重组鹰嘴豆孢克鲁维酵母外切菊粉酶活性的影响[J].生命科学研究, 2016, 20(3):218-223;277.MA J Y, TAN H D, WANG W X, et al.The effects of His6-tag position in Kluyveromyces cicerisporus exoinulinase on its activity[J].Life Science Research, 2016, 20(3):218-223;277.
[25] IMAMURA S, KITAURA S.Purification and characterization of a monoacylglycerol lipase from the moderately thermophilic Bacillus sp.H-257[J].Journal of Biochemistry, 2000, 127(3):419-425.
[26] TANG W, LAN D M, ZHAO Z X, et al.A thermostable monoacylglycerol lipase from marine Geobacillus sp.12AMOR1:Biochemical characterization and mutagenesis study[J].International Journal of Molecular Sciences, 2019, 20(3):780.