凝结芽胞杆菌(Bacillus coagulans)的分类始于1915年,HAMMER[1]从变质的罐装牛奶中分离并描述了B.coagulans,并在1980年《核准的细菌名录》中被收录[2]。CLERCK等[3]于2004年对B.coagulans的描述进行了修订。GUPTA等[4]于2020年基于全基因组序列分析将B.coagulans分类变迁为凝结魏茨曼氏菌(Weizmannia coagulans)。RAO等[5]于2023年基于全基因组序列分析在属水平将魏茨曼氏菌属(Weizmannia)重新分类为海恩德里克斯氏菌属(Heyndrickxia),将W.coagulans分类变迁为凝结海恩德里克斯氏菌(Heyndrickxia coagulans)。
B.coagulans是一种兼性厌氧、耐酸和耐热的菌种[6],在食品[7]、饲料[8]、医药[9]等行业应用广泛。凝结芽胞杆菌因可产生高浓度的乳酸在工业上得到了很好的开发和利用[10];作为饲料添加剂,对生长育肥猪具有促生长和增强免疫的作用[11];作为益生菌,能够调节消化道微生态平衡,促进蛋白质等营养物质的消化吸收[12-13],通过调节肠道的微生物菌群结构,有助于维护肠道健康[14],此外对宿主具有免疫调节功能,具有治疗肠道疾病的作用[9]。凝结芽胞杆菌在生产有工业价值的产品中发挥着重要作用,因此在经济上具有重要意义。
凝结芽胞杆菌的鉴定及安全性评价近年来备受行业关注,菌种的科学鉴定不仅是安全性评价的基础,也是行业应用的前提。我国对可用于食品的菌种和饲料添加剂菌种实施名单和目录准入管理制度,W.coagulans原用菌种名称:B.coagulans已被列入《可用于食品的菌种名单》和《饲料添加剂品种目录》。同时,为确保菌种的安全性,我国有关部门也发布了相关的指南并制定相关标准中。《直接饲喂微生物和发酵制品生产菌株鉴定及安全性评价指南》由农业农村部于2021年发布,指南中明确要求需通过形态观察、生理生化检测以及分子生物学综合分析将微生物鉴定至少到种或亚种水平。《食品安全国家标准 食品用菌种安全性评价程序》(征求意见稿)由国家卫生健康委员会于2023年发布,其中明确要求对拟评价微生物菌种需提供规范、科学的菌种分类学资料,当菌种分类学地位发生变化时,还应包含其重新分类后的名称及曾用名,并提供基于表型和基因型的菌种鉴定资料。菌种鉴定在食品、饲料等多个领域都具有重要的应用价值,在生物技术和工业领域有广泛的应用。
本文研究了B.coagulans的分类变迁与鉴定,以CICC 20138为研究对象,通过基于表型与基因型的深入鉴定分析,包括菌种宏观和微观形态、生理生化鉴定、16S rRNA基因序列分析,并采用基于全基因组序列的核心基因组多位点序列分型(core genome multilocus sequence typing, cgMLST)和平均核苷酸一致性(average nucleotide identity,ANI)以及数字DNA-DNA杂交(digital DNA-DNA hybridization,dDDH)分析等技术进行菌种鉴定,明确菌种分类学地位,以期为B.coagulans的安全性评价及行业应用奠定基础,为相关菌种的鉴定提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 实验菌株
实验菌株CICC 20138源自中国工业微生物菌种保藏管理中心(China Center of Industrial Culture Collection, CICC)。
1.1.2 试剂与培养基
胰蛋白胨大豆琼脂培养基,北京陆桥技术有限责任公司;VITEK BCL鉴定卡,法国梅里埃公司;细菌基因组DNA提取试剂盒,天根生化科技(北京)有限公司;DNA Marker、2×PCR Mix,北京全式金生物技术有限公司;所有化学试剂均为分析纯。
1.2 仪器与设备
5415D离心机,德国Eppendorf公司;ESCO AC2-6S1生物安全柜,新加坡ESCO公司;BioDrop μLite超微量核酸蛋白分析仪,英国Biochrom公司;ABI 9700 PCR仪,美国ABI公司;Gel Doc EZ凝胶成像分析系统,美国Bio-Rad公司。
1.3 实验方法
1.3.1 菌株转接、活化与保藏
冻干管菌种CICC 20138在生物安全柜中通过平板涂布进行菌种的转接与活化。采用无菌水溶解冻干菌粉,涂布TSA培养基平板,于37 ℃培养48 h,观察结果。挑取单菌落在TSA培养基平板上采用4区划线法进行平板划线培养,待培养好后保藏于4 ℃冰箱备用。
1.3.2 形态学观察
将CICC 20138接种于TSA培养基平板,于37 ℃培养48 h,观察其在TSA培养基上的宏观形态特征,并利用光学显微镜观察菌体微观形态特征[6]。
1.3.3 生理生化特征检测
采用VITEK 2 Compact鉴定系统,将手工鉴定的经验应用于自动化,使用VITEK BCL需氧芽胞杆菌鉴定卡进行CICC 20138的碳氮源利用以及酶活性等生理生化特征鉴定。
1.3.4 16S rRNA基因序列分析
进行菌株CICC 20138的16S rRNA基因序列分析,为保证菌种质量而对菌种进行了重新复核鉴定。按照细菌基因组DNA提取试剂盒(DP302)的说明书操作提取菌株CICC 20138的基因组DNA,为确保所提取的基因组的质量,DNA的浓度和纯度采用超微量核酸蛋白分析仪进行测定。将质控合格的基因组DNA作为模板,进行16S rRNA通用引物27F/1492R的PCR扩增,并进行PCR产物凝胶电泳检测,检测合格进行目的基因的序列测定[3]。将测序获得的16S rRNA基因序列提交至NCBI(https://submit.ncbi.nlm.nih.gov/)数据库获取GenBank登录号,并与模式菌种数据库中已知微生物序列进行序列比对,获得同近缘模式菌种序列相似性。采用CLUSTAL W软件进行近缘菌种多序列比对,使用MEGA 11.0软件基于近缘菌16S rRNA基因序列以邻接法构建CICC 20138和近缘菌种的系统发育树[15],自展值设定为1 000。
1.3.5 基于全基因组序列的cgMLST、ANI、dDDH分析
1.3.5.1 全基因组序列来源及质控
菌株CICC 20138(GCA_009936615.1)的全基因组序列及相关菌种的模式菌株全基因组序列均来源于NCBI(https://www.ncbi.nlm.nih.gov/)数据库,使用CheckM[16]评估下载的全基因组序列质量并进行质控。
1.3.5.2 cgMLST分析
16S rRNA基因序列分析的分辨率相对较低,本研究进一步使用具有更高分辨率的基于核心基因的系统发育分析,使用Glimmer v3.02[17]进行基因预测并获得蛋白质序列。使用CD-HIT[18]对蛋白质序列进行聚类,序列一致性>50%,获得核心基因数据集。使用CLUSTAL W软件对核心基因序列进行多序列比对,使用MEGA 11.0软件基于核心基因组序列邻接法构建CICC 20138和近缘菌种系统发育树[15],自展值设定为1 000,进行菌株CICC 20138核心基因组多位点序列分型分析。
1.3.5.3 ANI、dDDH分析
基于全基因组的分类能够在物种水平上最大限度地提高原核生物种水平分类的分辨率。本研究进一步进行CICC 20138(GCA_009936615.1)基于全基因组序列的ANI和dDDH分析。采用JSpeciesWS在线数据库的ANIb模式(https://jspecies.ribohost.com/jspeciesws/#home)进行CICC 20138同近缘模式菌种的ANI分析,ANI分析的判定阈值为95%~96%[19]。通过ANI值比对,若待测菌种有且仅与1个模式菌种全基因组序列的ANI值>95%~96%,判定与该模式菌种为同种。CICC 20138与近缘模式菌种的dDDH值采用GGDC 3.0(Genome-to-Genome Distance Calculator,http://ggdc.dsmz.de/ggdc.php)进行计算,dDDH分析的判定阈值为70%[20]。基于基因组学的原核物种定义认为,来自同一物种的菌株具有>95%的ANI值和>70%的dDDH值[21]。
2 结果与分析
2.1 菌株CICC 20138的转接活化与形态学观察
菌株CICC 20138经TSA培养基37 ℃培养48 h转接活化,菌落圆形,浅黄色,不透明,表面湿润,边缘不整齐,如图1-a所示。通过光学显微镜进行菌体观察,结果显示菌体大小为(0.4~0.6) μm×(2.2~6.6) μm,呈杆状,成对或单个排列,革兰氏染色呈阳性,如图1-b所示。形态学观察结果显示,CICC 20138的菌落和菌体特征同伯杰氏系统细菌学手册中B.coagulans基本特征的描述相符[6]。
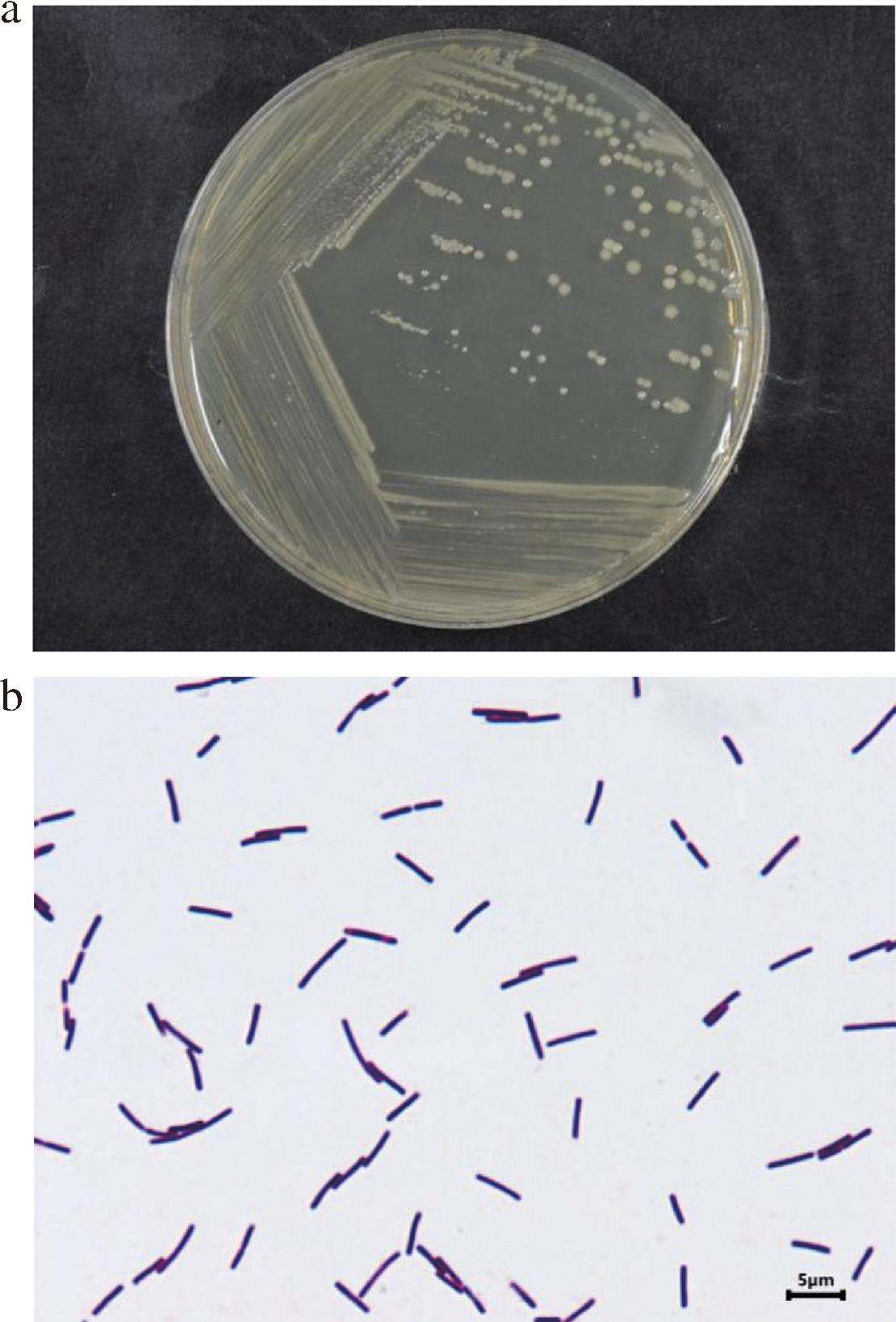
a-菌落;b-菌体
图1 CICC 20138的菌落菌体形态观察结果
Fig.1 Cell morphology observation result of CICC 20138
2.2 菌株CICC 20138生理生化特征
采用VITEK 2 Compact系统中的VITEK BCL需氧芽胞杆菌鉴定卡对CICC 20138进行鉴定。VITEK BCL鉴定卡共有46种检测酶活性和碳氮源利用等的生化试验,可对需氧芽胞杆菌进行生理生化特征鉴定检测[22]。通过VITEK BCL需氧芽胞杆菌鉴定卡检测,检测结果显示CICC 20138能产苯丙氨酸芳胺酶、L-吡咯烷酮芳胺酶、α-半乳糖苷酶、酪氨酸芳胺酶、丙氨酸-苯丙氨酸-脯氨酸芳胺酶、α-葡萄糖苷酶,并能够利用D-半乳糖、麦芽三糖、D-甘露糖、D-葡萄糖,详见表1,如上特征符合B.coagulans在伯杰氏系统细菌学手册中的描述[6],经VITEK BCL鉴定具有97%的鉴定概率百分比[23],可鉴定为B.coagulans。
表1 CICC 20138的VITEK BCL检测结果
Table 1 VITEK BCL test result of CICC 20138

检测项目结果检测项目结果β-木糖苷酶-L-赖氨酸芳胺酶-苯丙氨酸芳胺酶+L-脯氨酸芳胺酶-α-半乳糖苷酶+丙氨酸芳胺酶-丙氨酸-苯丙氨酸-脯氨酸芳胺酶+环糊精-肌醇-甲基-α-D-吡喃葡萄糖苷酸化-α-甘露糖苷酶-麦芽三糖+D-甘露糖+D-松三糖-L-鼠李糖-β-葡糖苷酶-丙酮酸盐-α-葡萄糖苷酶+菊粉-D-葡萄糖+在6.5%氯化钠中生长-卡那霉素耐药-红四氮唑-多粘菌素B耐药-L-天冬氨酸芳胺酶-亮氨酸芳胺酶-β-半乳糖苷酶-L-吡咯烷酮芳胺酶+酪氨酸芳胺酶+β-N-乙酰氨基葡糖苷酶-D-半乳糖+糖原-ELLMAN试剂+甲基-D-木糖苷-甘氨酸芳胺酶-D-甘露醇-N-乙酰-D-氨基葡萄糖-古老糖-β-甘露糖苷酶-磷酰胆碱-D-塔格糖-D-海藻糖+wD-核糖-腐胺同化-竹桃霉素耐药-七叶苷水解+
注:“+”,阳性;“+w”,弱阳性;“-”,阴性。
2.3 16S rRNA基因序列系统发育分析
检测获得CICC 20138的16S rRNA基因序列,提交至NCBI数据库的GenBank登录号为OR690796,将其与模式菌种数据库中已知微生物序列进行序列比对,同近缘模式菌种H.coagulans ATCC 7050T(CP009709)和H.faecalis Marseille-P8953T(GCA_018101145.1_2)的16S rRNA基因序列相似性最高,均为99.20%,同其他模式菌种的16S rRNA基因序列相似性均低于98.65%。以嗜盐喜盐芽胞杆菌(Halobacillus halophilus)DSM 2266T(HE717023)为外群,基于近缘菌16S rRNA基因序列以CLUSTAL W软件进行多序列比对,使用MEGA 11.0软件以邻接法构建CICC 20138和近缘菌种的系统发育树(图2)。基于16S rRNA序列比对的系统发育分析,CICC 20138同H.coagulans和H.faecalis序列相似性最高且在系统发育树中聚在同一分支,可将CICC 20138鉴定至海恩德里克斯氏菌属(Heyndrickxia sp.)。

图2 基于16S rRNA基因序列邻接法构建的CICC 20138和近缘菌种的系统发育树
Fig.2 Neighbor-joining phylogenetic tree of strains CICC 20138 and related species based on 16S rRNA gene sequences
2.4 基于全基因组序列的cgMLST、ANI、dDDH分析
2.4.1 全基因组序列及质控
CICC 20138(GCA_009936615.1)基因组大小为3 386 433 bp,含有216个contigs,基因组DNA G+C含量为46.7 mol%,使用CheckM评估下载的菌株CICC 20138(GCA_009936615.1)及相关模式菌种的全基因组序列的质量,经质控所有菌株的基因组完整度(>98%)和污染百分比(<2%)均符合分析要求。
2.4.2 cgMLST分析
通过cgMLST分析,使用Glimmer v3.02进行基因预测并获得蛋白质序列,获得443个核心基因序列,采用邻接法构建cgMLST系统发育树(图3)。cgMLST系统发育分析显示,CICC 20138与H.coagulans以100%支持率聚在同一分支,且能够与Heyndrickxia内其他的模式菌种明显区分开。
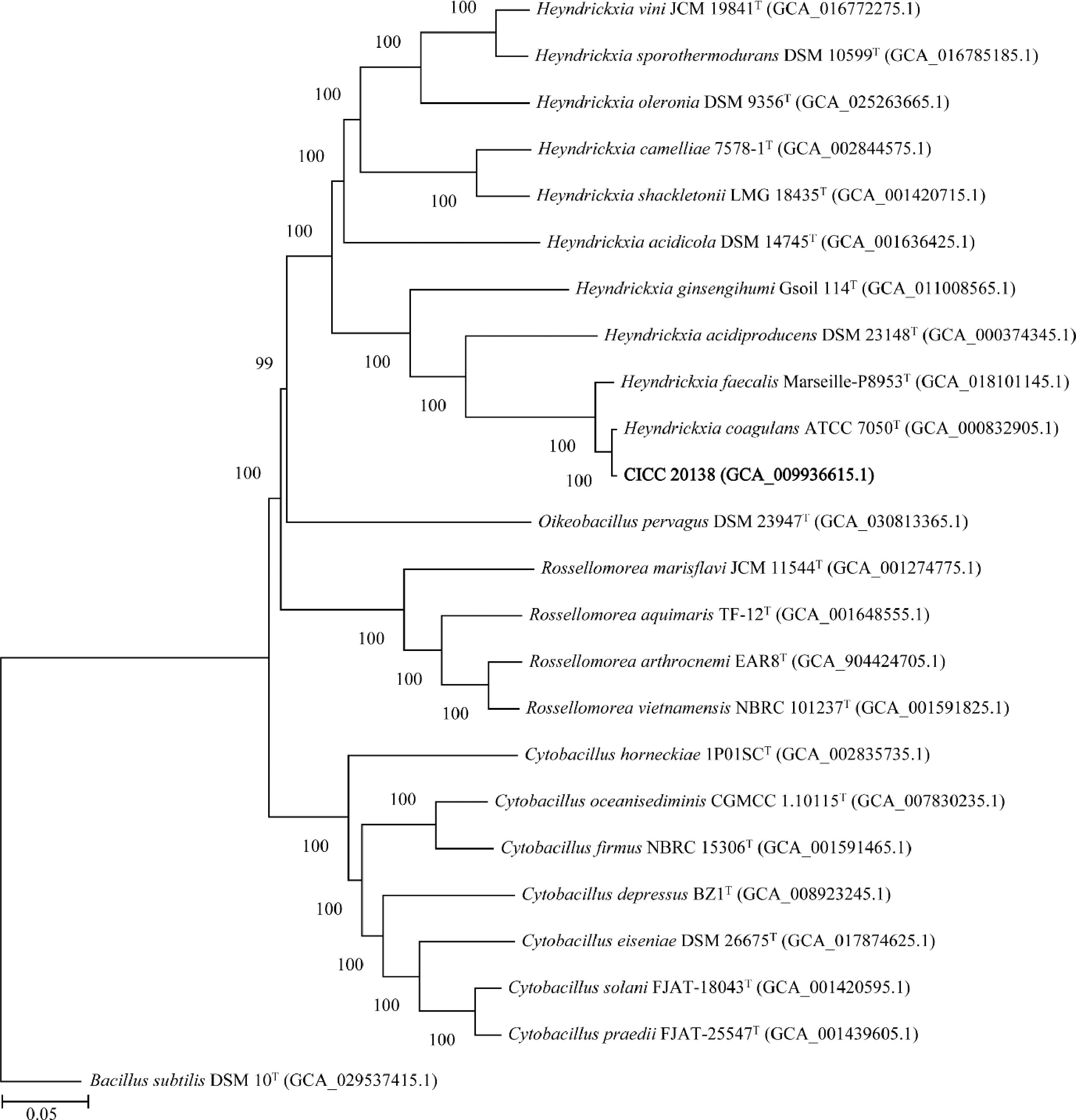
图3 基于核心基因序列邻接法构建的CICC 20138和近缘菌种的系统发育树
Fig.3 Neighbor-joining phylogenetic tree of strains CICC 20138 and related species based on core-genome sequences
2.4.3 ANI、dDDH分析
采用JSpeciesWS在线数据库的ANIb模式进行CICC 20138同近缘模式菌种之间的ANI分析。结果显示CICC 20138同H.coagulans ATCC 7050T(GCA_000832905.1)的ANI值为98.16%,>95%~96%,与其余近缘模式菌种ANI值均<95%(表2),判定CICC 20138与H.coagulans为同种。采用GGDC 3.0公式(2)计算获得CICC 20138同H.coagulans ATCC 7050T(GCA_000832905.1)的dDDH值为87.1%,>70%,且同其余模式菌种的dDDH值均<70%。基于全基因组序列的ANI和dDDH分析结果表明,CICC 20138被鉴定为H.coagulans。B.coagulans当前分类正确名称为H.coagulans,需依据最新分类的正确名称及时进行更名。
表2 CICC 20138和近缘菌种的ANI值 单位:%
Table 2 The ANI values among CICC 20138 and related species

菌株12345678910111-98.1694.4776.5767.7569.7168.0667.0867.4967.5167.51298.01-94.9576.4068.6070.3668.8568.1668.2168.4168.41394.2794.42-75.9668.6170.3868.8968.1768.0068.3568.35476.3475.7975.90-68.9070.8168.5368.3767.9267.5367.53568.1368.1768.1668.89-70.7570.7472.7368.9768.1268.12669.8069.8969.9671.1270.56-69.5170.5667.4867.2767.27768.6068.6768.7368.6270.8169.76-70.2868.5568.3968.39867.8968.0668.1068.3673.0670.8370.58-69.0868.1068.10967.3767.1767.1867.8168.5267.4068.0168.33-67.8467.841067.3567.4267.3967.1967.5266.7667.7867.2967.64-67.311167.3067.4067.3968.1082.2370.2169.8871.7067.4267.18-
注:1-CICC 20138 (GCA_009936615.1);2-Heyndrickxia coagulans ATCC 7050T (GCA_000832905.1);3-Heyndrickxia faecalis Marseille-P8953T (GCA_018101145.1);4-Heyndrickxia acidiproducens DSM 23148T (GCA_000374345.1);5-Heyndrickxia shackletonii LMG 18435T (GCA_001420715.1);6-Heyndrickxia ginsengihumi Gsoil 114T (GCA_011008565.1);7-Heyndrickxia acidicola DSM 14745T (GCA_001636425.1);8-Heyndrickxia oleronia DSM 9356T (GCA_025263665.1);9-Bacillus thermotolerans SGZ-8T (GCA_000812025.2);10-Neobacillus notoginsengisoli JCM 30743T (GCA_003515685.1);11-Heyndrickxia camelliae 7578-1T (GCA_002844575.1)。
3 结论
本研究介绍了B.coagulans的分类变迁,并采用形态学、生理生化、16S rRNA基因序列分析和基于全基因组序列的cgMLST、ANI和dDDH分析对CICC 20138进行鉴定。采用TSA培养基对37 ℃培养的CICC 20138进行转接活化,并进行宏观和微观形态观察、生理生化特征和分子生物学的鉴定。CICC 20138通过16S rRNA基因序列分析鉴定至Heyndrickxia sp.;通过cgMLST分析,CICC 20138与H.coagulans聚在同一分支,且能够与其他近缘模式菌种有效区分开;ANI分析CICC 20138与H.coagulans的ANI值为98.16%,dDDH值为87.1%,可将CICC 20138鉴定至H.coagulans。结果表明,基于全基因组序列的ANI和dDDH分析能够科学准确地将凝结芽胞杆菌鉴定至最新分类的种水平,因B.coagulans当前分类正确名称为H.coagulans,需依据最新分类学正确名称及时更名,确保菌种名称的科学性和有效性。
由于基于16S rRNA基因的系统发育分析确定属和种水平的分类准确性是有限的,已有研究表明来自高通量测序数据的cgMLST分析在乳酸菌分离株中获得了更高的辨别力[24]。为了弥补基于16S rRNA基因序列系统发育分析的低分辨率,本研究进一步使用具有更高分辨率的基于全基因组序列的cgMLST、ANI和dDDH分析。采用全基因组序列分析能够在物种水平上最大限度地提高原核生物种水平分类的分辨率。综上,基于全基因组序列的ANI和dDDH分析可用于菌种的分类鉴定,为菌种的分类学地位确定及安全性评价提供研究基础和技术支持。对于具有潜在重要价值的菌种,全基因组序列分析是了解微生物在分类和鉴定过程中进化的重要工具。
B.coagulans在工业领域应用广泛,曾重新分类为W.coagulans,当前分类正确名称为H.coagulans,目前此3个菌种名称在相关标准和文献中均有使用。本文研究了凝结芽胞杆菌的分类变迁,并采用表型和基因型的分类鉴定技术进行菌种鉴定,明确其最新的分类学地位,为B.coagulans的应用及安全性评价奠定基础。
[1] HAMMER B W.Bacteriological studies on the coagulation of evaporated milk[J].Iowa Agricultural Experimental Station Research Bulletin, 1915, 19:119-131.
[2] SKERMAN V B D, MCGOWAN V,SNEATH P H A. Approved lists of bacterial names[J]. International Journal of Systematic and Evolutionary Microbiology, 1980, 30(1):225-420.
[3] CLERCK E D, RODRIGUEZ-DIAZ M, FORSYTH G, et al.Polyphasic characterization of Bacillus coagulans strains, illustrating heterogeneity within this species, and emended description of the species[J].Systematic &Applied Microbiology, 2004, 27(1):50-60.
[4] GUPTA R S, PATEL S, SAINI N, et al.Robust demarcation of 17 distinct Bacillus species clades, proposed as novel Bacillaceae genera, by phylogenomics and comparative genomic analyses:description of Robertmurraya kyonggiensis sp.nov.and proposal for an emended genus Bacillus limiting it only to the members of the Subtilis and Cereus clades of species[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(11):5753-5798.
[5] RAO M P N, BANERJEE A, LIU G H, et al.Genome-based reclassification of Bacillus acidicola, Bacillus pervagus and the genera Heyndrickxia, Margalitia and Weizmannia[J].International Journal of Systematic and Evolutionary Microbiology, 2023, 73(7):5961.
[6] WHITMAN W B, VOS P D, GARRITY G M, et al.Bergey's Manual of Systematic Bacteriology Second Edition[M].New York:Springer Science, 2009.
[7] KONURAY G, ERGINKAYA Z.Potential use of Bacillus coagulans in the food industry[J].Foods, 2018, 7(6):92.
[8] ZHOU Y H, ZENG Z H, XU Y B, et al.Application of Bacillus coagulans in animal husbandry and its underlying mechanisms[J].Animals (Basel), 2020, 10(3):454.
[9] MU Y, CONG Y.Bacillus coagulans and its applications in medicine[J].Beneficial Microbes, 2019, 10(6):679-688.[10] JUTURU V, WU J C.Microbial production of lactic acid:The latest development[J].Critical Reviews in Biotechnology, 2016, 36(6):967-977.
[11] FU R Q, CHEN D W, TIAN G, et al.Effect of dietary supplementation of Bacillus coagulans or yeast hydrolysates on growth performance, antioxidant activity, cytokines and intestinal microflora of growing-finishing pigs[J].Animal Nutrition, 2019, 5(4):366-372.
[12] 王一帆, 岑丛楠, 刘福奇, 等.凝结芽孢杆菌的益生特性及机制研究进展[J].食品工业科技, 2023, 44(1):458-464.WANG Y F, CEN C N, LIU F Q, et al.Research progress on the probiotic characteristics and mechanism of Bacillus coagulans[J].Science and Technology of Food Industry, 2023, 44(1):458-464.
[13] STECKER R A, MOON J M, RUSSO T J, et al.Bacillus coagulans GBI-30, 6086 improves amino acid absorption from milk protein[J].Nutrition &Metabolism, 2020, 17:93.
[14] MAITY C, GUPTA P A K, SAROJ D B, et al.Impact of a gastrointestinal stable probiotic supplement Bacillus coagulans LBSC on human gut microbiome modulation[J].Journal of Dietary Supplements, 2021, 18(6):577-596.
[15] KUMAR S, STECHER G, LI M, et al.MEGA X:Molecular evolutionary genetics analysis across computing platforms[J].Molecular Biology and Evolution, 2018, 35(6):1547-1549.
[16] PARKS D H, IMELFORT M, SKENNERTON C T, et al.CheckM:Assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes[J].Genome Research, 2015, 25(7):1043-1055.
[17] DELCHER A L, BRATKE K A, POWERS E C, et al.Identifying bacterial genes and endosymbiont DNA with Glimmer[J].Bioinformatics, 2007, 23(6):673-679.
[18] FU L M, NIU B F, ZHU Z W, et al.CD-HIT:Accelerated for clustering the next-generation sequencing data[J].Bioinformatics, 2012, 28(23):3150-3152.
[19] KIM M, OH H S, PARK S C, et al.Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J].International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 2):346-351.
[20] MEIER-KOLTHOFF J P, CARBASSE J S, PEINADO-OLARTE R L, et al.TYGS and LPSN:A database tandem for fast and reliable genome-based classification and nomenclature of prokaryotes[J].Nucleic Acids Research, 2022, 50(D1):D801-D807.
[21] THOMPSON C C, CHIMETTO L, EDWARDS R A, et al.Microbial genomic taxonomy[J].BMC Genomics, 2013, 14:913.
[22] BANOON S R, ALI Z M.Identification the local thermophilic strains of Bacillus licheniformis by VITEK-2 system and Gyrase B gene[J].Al-Kufa University Journal for Biology, 2018, 10(2):47-57.
[23] 杨晶, 王伟欢.VITEK 2 Compact系统的应用及鉴定结果分析[J].中国卫生检验杂志, 2016, 26(18):2643-2645.YANG J, WANG W H.Application of VITEK 2 Compact system and its identification results analysis[J].Chinese Journal of Health Laboratory Technology, 2016, 26(18):2 643-2 645.
[24] HUANG C H, CHEN C C, LIOU J S, et al.Genome-based reclassification of Lactobacillus casei:Emended classification and description of the species Lactobacillus zeae[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(6):3755-3762.