抗生素发现和广泛使用使得数十亿人从细菌感染类的疾病中得以解脱。随着大范围和大批量使用抗生素,其导致的耐药性问题也凸显出来[1]。传统的抗生素逐渐失去了对抗病原微生物的效力,而寻找新的解决方案迫在眉睫。在此背景下,抗菌肽(antimicrobial peptides, AMPs)作为一类天然存在于生物体中的活性分子而备受关注。
AMPs具有广阔的应用前景,由于其独特的抗菌活性被认为是克服抗生素耐药性的候选物质之一。首先抗菌肽在控制动植物疾病方面具有巨大潜力,且病原体产生抗药性的风险较低,研究发现小麦中的两种碱性低分子量蛋白质(Puroindoline a and b),能够控制小麦胚乳的软度,同时还能够抑制多种细菌和真菌的生长并杀死它们[2]。并在畜牧业方面也有着广阔应用前景,SHI等[3]详细介绍了具有强大抗菌活性的猪骨髓抗菌肽的生化参数、结构特征和生物活性等。
AMPs具有快速和非特异性地破坏病原体的能力,它们通过破坏病原体,刺激细胞因子、趋化因子和生长因子的分泌,同时激活树突状细胞,从而参与适应性免疫过程,不同AMPs在免疫细胞(巨噬细胞、单核细胞、淋巴细胞、肥大细胞、树突状细胞、中性粒细胞和嗜酸性粒细胞)中有着复杂的抗炎机制[4]。研究发现,在银屑病中,AMPs不仅作为趋化因子发挥作用,还与ds-cDNA形成复合物作为T细胞的自身抗原,触发自身免疫反应,激活Th17/IL23免疫轴,维持炎症反应[5],是治疗银屑病等疾病的新型治疗方法。同时越来越多的研究表明,AMPs对肿瘤细胞也具有一定的抗增殖和凋亡诱导作用,有望成为肿瘤治疗的新策略之一,通过对之前合成的AMPs进行改造,并进行了生物信息学预测,以选择具有最高抗癌活性的肽段。进一步的体外实验验证发现,筛选出来的抗癌活性片段能够在48 h后抑制乳腺癌细胞系(MDA-MB-231)的生长,并成功阻断了MDA-MB-231的细胞周期,诱导其凋亡[6]。
目前AMPs在食品中有着广泛的应用前景,被视为延长食品保质期和保障食品安全的有效工具;AMPs因其广谱抗菌活性,能够有效抑制多种食品腐败菌和致病菌生长,从而延长食品的保质期,同时AMPs作为天然防腐剂,相较于传统的化学防腐剂,具有更高的安全性和环保性,容易被人体降解,不会在体内积累,从而减少了对人体的潜在风险。AMPs也可以整合到食品包装材料中,形成具有抗菌功能的包装,例如具有抗菌和保鲜功能的可降解保鲜膜。然而,尽管AMPs具有广泛的生物活性和潜在的药用价值,但其研究和应用仍面临着巨大的挑战。
在筛选新型的AMPs候选药物时,必须充分了解其物理化学性质、结构特征、生物活性及特异性分子靶标,而且肽段必须在生物体内具有生物利用度,并能够在血浆中保持稳定[7]。然而通过传统的湿实验对AMPs研究存在诸多弊端,如需要耗费大量时间和人力资源;在AMPs的合成、纯化到活性评价等步骤花费大量的时间和精力;且需要购买昂贵的试剂和设备,进行大量的样品处理和测试,成本较高;同时由于实验条件和设备的限制,传统湿实验方法的样品处理数量十分有限,难以进行高通量的筛选和测试;更重要的是,湿实验通常只能获得AMPs的整体活性信息,难以深入探究AMPs的构效关系,对AMPs优化和设计的效率很低。
深度学习作为人工智能(artificial intelligence,AI)的一个分支,凭借其独特优势在多个领域崭露头角,目前深度学习已经被应用并筛选了几种β-内酰胺酶抑制剂和抗生素替代品,包括AMPs肽、非核糖体肽、细菌素和海洋天然产物;而深度学习强大的数据处理和学习能力为AMPs的研究和设计提供了新的思路和方法。本文在总结AMPs的基本特点和作用机制的基础上,阐述了深度学习模型在AMPs研究中的优势,重点讨论深度学习在AMPs识别和筛选、生成和设计、性质预测3个方面的应用及发展现状。并在总结已有研究的成果的基础上,探讨未来深度学习在AMPs研究中存在的限制和挑战及未来的发展方向。
1 AMPs概述
1.1 AMPs的基本特点
AMPs是一类长度为5~100个氨基酸的小肽分子,分子质量在2 k~7 kDa左右,具有强效和广谱的抗菌特性[8],是所有高等生物免疫系统中天然存在的一部分,对细菌具有很高的选择性,但对宿主细胞的影响较小[9]。AMPs广泛存在于各种生物中[10],两栖动物是AMPs最丰富的来源[11]。根据氨基酸序列、肽的净电荷、蛋白质结构和来源,AMPs被分为几个亚组:第一亚组带负电荷,含5~70个氨基酸残基,其净电荷范围为 -1~-8;第二亚组为α螺旋,长度<40个氨基酸,具有+2~+9的净电荷,大多数氨基酸具有C-末端酰胺化;第三亚组为β-折叠,带正电荷,通常包含2~8个半胱氨酸残基,形成1~4对分子内二硫键;第四亚组是包含特定氨基酸带正电的AMPs,包括精氨酸、脯氨酸、色氨酸、甘氨酸和组氨酸,但缺乏规则的二级结构;第五亚组是来自抗菌蛋白的片段[12],但其整体呈现阳离子肽特性,是其具有高效杀菌机制的原因之一。
1.2 作用机制
AMPs能够发挥抗生素的作用,与抗生素不同,AMPs与细菌的细胞膜相互作用,通过中和电荷,随后通过穿透细胞膜引起细菌死亡,从而降低了细菌耐药性的风险[13],AMPs实现其抗菌活性的作用机制十分复杂,可以直接作用于细菌的细胞壁和细胞膜,或影响不同的细胞内靶标,以及对生物膜形成抑制、调节宿主免疫系统活动等。具体作用机制见图1。

图1 AMPs灭菌作用机制
Fig.1 Mechanism of antimicrobial peptide sterilization action
1.2.1 作用于细菌细胞壁
目前关于AMPs对细菌细胞壁的作用机制研究较少,细菌的细胞壁是其生存所必需的结构,确保了结构完整性并保护细菌免受外部威胁。革兰氏阳性细菌通常具有由多层肽聚糖和磷壁酸组成的厚细胞壁,含有15~50层的肽聚糖和大量的磷壁酸,可以分类为壁磷壁酸或膜磷壁酸。而革兰氏阴性细菌具有较薄的细胞壁且结构复杂,包括单层肽聚糖和包含脂蛋白、磷脂和脂多糖(lipopolysaccharides,LPS)的复杂外膜结构。AMPs与细胞壁组分(如肽聚糖和脂多糖)相互作用,破坏其完整性并导致细菌死亡,AMPs可能与细胞壁的带负电的成分结合,导致细胞膜不稳定,从而细胞内含物泄露,最终导致细菌死亡。
1.2.2 作用于细菌细胞膜
细菌的细胞膜是细菌生存的关键组成部分,起着保护细胞内部结构的作用,细菌细胞膜的重要性在于维持细菌的形态结构和保护细胞内部免受外界环境的不利影响,例如维持细胞内的渗透压和离子平衡,控制物质的进出。然而,微生物细胞膜是大多数AMPs的主要作用靶点;较早的双极化干涉测定法研究了AMPs诱导的膜扰动过程,AMPs与膜的相互作用会导致膜的扭曲和扰动,最终导致膜的破环和细胞死亡[14],VINEETH KUMAR等[15]通过两态模型和界面活性模型解释了膜曲率对肽结合和曲率敏感肽与膜的相互作用的影响。LAU等[16]通过一系列实验,利用电子显微镜、人工膜染料泄露、溶液和固态核磁共振光谱,以及分子动力学模拟技术,验证了一种SMART作用机制,即通过膜适应性和柔软性的改变,破坏细菌的生理功能。YASIR等[17]发现,AMPs可以中和铜绿假单胞菌(革兰氏阴性菌)的脂多糖,在30 s内破坏细胞质膜,细菌在接触的2 h内开始溶解;DASH等[18]研究表明,一种具有单二硫键的β-折叠结构的AMPs Thanatin与负责脂多糖转运的外膜蛋白复合物中LptA和LptD亲和力较高,且Thanatin的N末端β-链与Lpt的N末端β-链形成相互作用界面,来杀灭细菌。JUHL等[19]研究表明,肽和膜上的脂质可以形成多种超分子组装体,其中更疏水的序列倾向于采用跨膜排列,而带电的两性序列则倾向于留在膜界面插层,导致膜的改变和破坏,从而影响细菌细胞活性发挥其抗菌作用。LIZIO等[20]使用圆二色谱、振动圆二色谱、线性二色谱、拉曼光谱、拉曼光学活性、中子反射和分子动力学模拟等方法研究AMPs类似物后发现,所研究的肽类可迅速在膜中分配并定向,同时发现了磷脂与嵌入膜的肽之间存在手性相互作用的证据。JI等[21]研究发现,AMPs能够通过去极化增加外膜和内膜的通透性,并干扰多耐药株金黄色葡萄球菌细胞膜的整合,导致细胞内容物外泄。RIESCO-LLACH等[22]总结了线性十一肽KKLFKKILKYL(BP100)对革兰氏阴性细菌电荷中和以及膜渗透化的作用机制,在与膜的静电相互作用后,BP100会翻转,促进其疏水面插入膜双层,BP100诱导膜变薄和脂质扩散减慢,并促进水的渗透。
AMPs通过物理化学作用与微生物细胞膜结合,在细胞膜表面不断积累并发生结构或构象转变,AMPs还可以与细菌细胞膜上的负电荷分子相互作用,如磷脂和LPS。这些相互作用导致细胞膜的破坏,从而增加了细胞膜的通透性,导致细胞膜溶解并释放细胞内容物,最终使细菌死亡,从而发挥抗菌活性。
1.2.3 作用于细胞内靶标
AMPs不仅可以作用于细胞壁和细胞膜,还可以通过直接穿透或内吞作用进入细胞[23],并靶向作用于细菌细胞内的细胞核、细胞器或蛋白质[24]发挥抗菌作用。AMPs可通过形成瞬时的扭曲缺陷直接穿越细胞膜,与细胞穿膜肽一起进入细胞[25],也可以通过受体介导的转运途径跨膜[26]。BATTISTA等[27]通过凝胶阻滞实验结果表明,LL-III(一种阳离子AMPs)可以与质粒DNA发生相互作用,说明质粒是潜在的胞内靶标。RICILUCA等[28]同样发现,AMPs与核酸结合可能导致抑制真菌生长或其他抗真菌机制的激活,但其与核酸结合的确切机制仍需要进一步的研究来确认。GUO等[29]发现,AMPs能够导致MCF-7癌细胞的线粒体膜电位丧失及细胞色素c的释放。ZHANG等[30]利用PNP(对硝基苯基-β-D-半乳糖苷)和ONPG(酸性酸-对硝基苯基-β-D-半乳糖苷)测定法研究了重组AMPs Bomidin的作用机制,结果表明,Bomidin能渗透到细菌膜中,进一步诱导胞浆内容物的释放,并最终与DNA相互作用形成Bomidin-DNA复合物,影响细菌的基因表达,从而抑制细菌生存。许多AMPs可能同时影响多个靶标,通过复杂的方式产生抗菌作用。此外,不同类型的AMPs可能对不同类型的细菌具有不同的作用机制。
2 深度学习在AMPs研究中的应用
2.1 通过深度学习进行AMPs的识别和筛选
深度学习技术具有强大的模式识别和特征提取能力,能够从大量的数据中学习AMPs的结构与功能之间的关系,为AMPs的筛选和设计提供新的思路和方法。在AMPs识别和筛选中,常用的深度学习模型包括卷积神经网络(convolutional neural network,CNN)、循环神经网络(recurrent neural network,RNN)、长短期记忆网络(long short-term memory, LSTM)、图卷积神经网络(graph convolutional networks, GCN)等。这些模型可以从不同角度对AMPs的序列、结构和功能进行建模和预测(见图2)。
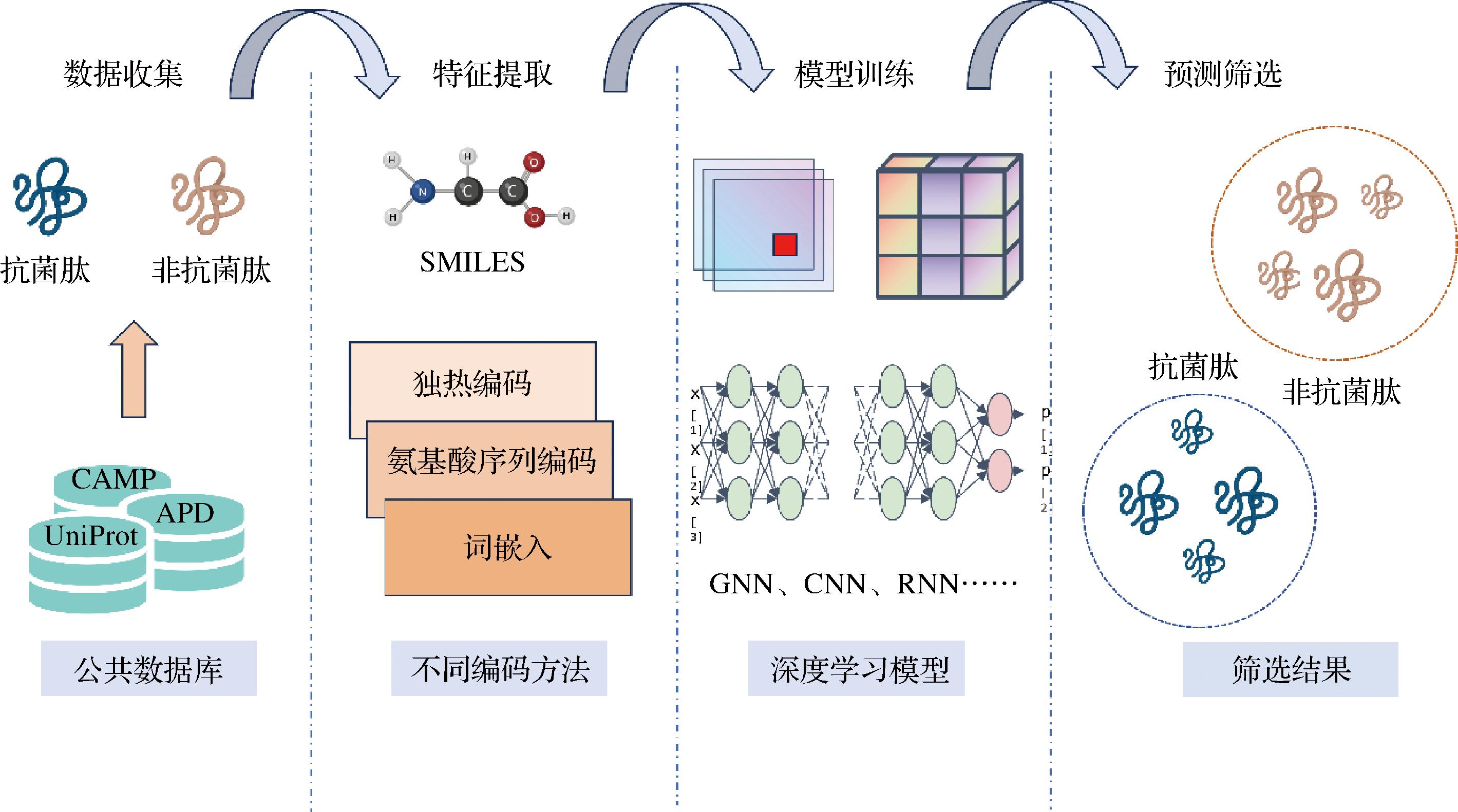
图2 利用深度学习识别和筛选AMPs
Fig.2 Identifying and screening antimicrobial peptides using deep learning
SU等[31]通过设计1个使用嵌入层和多尺度卷积网络的深度学习模型,多尺度卷积网络包含多个不同滤波器长度的卷积层,能够利用多个卷积层捕获的所有潜在特征,并且将额外的信息融合在设计的模型中提出一个融合模型,发现模型在AMPs数据集和AMPs数据库上的准确性要优于先前深度神经网络模型;FU等[32]通过设计计算方法来筛选有前景的AMPs候选物,并对模型中的一些层次中的数据进行可视化,作者克服了深度学习的黑盒特性,并发现了序列中的一些重要模式,该模型可以捕获氨基酸之间的相似性,肽不同序列部分的注意力分数,确定哪些部分在做出预测时更为关键,并自动融合各种异质信息或特征,将来自不同来源或类型的数据整合在一起,以提升分析的准确性和效果;YAN等[33]提出了一种短长度(≤30个氨基酸)AMPs预测方法,称为Deep-AmPEP30,它基于先前模型简化氨基酸组成和CNN的最佳特征集开发而成,准确率为77%,超过了先前的基于机器学习的方法,利用该模型对酵母菌类白色假丝酵母的基因组序列编码的肽进行了筛选,发现了一种20个氨基酸的肽(FWELWKFLKSLWSIFPRRRP),对枯草芽孢杆菌和副溶血弧菌表现出强大的抗菌活性,且该肽的效力与氨苄青霉素相当;PUENTES等[34]为了克服处理大型数据库时计算资源、数据处理复杂性、算法效率等限制,利用细菌基因组内在的多样性,通过结合深度学习模型算法、分子动力学、微生物表面展示和微流控技术设计了一个4阶段的工作流程,并基于SMILES这种用于输入和表示分子反应的线性符号,识别出具有所需溶菌能力的AMPs;DAS等[35]提出了一种用于生成所需属性的AMPs的高效计算方法,该方法通过利用在深度生成自编码器构建的分子潜在空间上训练的分类器进行指导,从而生成新分子,使用深度学习分类器以及高通量分子动力学模拟中导出的物理化学特征对生成的分子进行筛选,测试了20个候选抗微生物肽,其中2个显示出对多种革兰氏阳性和阴性菌具有高效抗菌特性,并具有在大肠杆菌中低耐药性的倾向;LIN等[36]利用最新的AMPs数据集,将蛋白质编码方法与深度学习模型结合起来,发现训练的模型要优于之前的方法,可准确对特定肽序列的抗菌性能进行预测并进行蛋白组筛选;SHARMA等[37]利用支持向量机算法和基于深度学习的特征开发了AniAMPpred模型,识别了动植物基因组中可能的AMPs,结果显示预测具有较高的置信度,并且能够对不同长度肽段进行高精度的AMPs和非AMPs分类,同时,作者使用了HCF和ALF两种编码方法对肽链进行编码,HCF编码是一种基于肽链物理化学性质进行编码的方法,而ALF编码是一种基于氨基酸在肽链中出现频率进行编码的方法,这2种方法均基于氨基酸属性进行编码;SHARMA等[38]还开发了Deep-ABPpred深度学习分类器,该工具利用双向长短期记忆算法(bidirectional long short-term memory, Bi-LSTM),并通过word2vec方法结合氨基酸特征,将蛋白质序列的氨基酸表示为向量,进行序列预测,在蛋白质序列中识别AMPs,该模型在测试集和独立数据集上分别达到了97%和94%的精度,并利用Deep-ABPpred,在链球菌噬菌体的尾蛋白序列中成功识别了AMPs,通过实验室合成以及体外活性测试,发现对所选的革兰氏阳性和革兰氏阴性菌表现出强有力的抗菌活性。
目前,大多数计算方法都可以通过序列本身识别AMPs,但在识别准确性方面仍有提升空间,而预训练策略已经应用于自然语言处理(natural language processing, NLP)的许多任务中,ZHANG等[39]将预训练策略应用于AMPs分类器的模型训练,基于BERT模型(基于Transformer架构的双向编码器模型),在UniProt的蛋白数据上进行了预训练,然后在6个具有较大差异的AMPs数据集上进行了微调和评估,提出了一种新的识别算法模型,该模型有助于捕捉新数据的特定特征和AMPs序列之间的共同特征,改善了识别的准确性;DEE等[40]使用预先训练的语言模型生成氨基酸序列的上下文嵌入来创建模型输入,然后卷积神经网络基于这些上下文嵌入进行训练,作为分类器,最终在两个数据集上进行验证,分别实现了93.33%和88.26%的预测准确率,优于以往的最先进分类模型;人类肠道微生物组编码了大量多样的抗微生物肽,MA等[41]结合多个NLP神经网络模型,包括LSTM、注意力机制和BERT,形成统一的流程,用于从人类肠道微生物组数据中识别候选AMPs,在识别出的2 349个序列中,化学合成了216个候选AMPs,其中181个显示出抗微生物活性,有效克服了AMPs的短序列长度对预测产生的挑战;PANG等[42]提出一种利用Transformer神经网络架构,结合NLP提取肽序列信息的深度学习方法发现,用于计算新型AMPs发现的深度学习方法,实现区分AMPs和其他肽段96.85%的平衡准确率;SINGH等[43]提出名为StaBle-ABPpred的一种基于堆叠集成技术的深度学习分类器,在分类器的第一层使用双向长短期记忆(biLSTM)和注意力机制,并在决策层使用随机森林、梯度回升和逻辑回归的集成来将肽分类为抗菌或其他类型,对结果进行方差分析测试及事后分析,发现比先前现有分类器的预测结果更好,作者应用此模型在链球菌噬菌体T12基因组的所有蛋白质中发现了新的AMPs;SINGH等[44]提出一种基于时间卷积网络的二元分类方法,以在植物和动物的蛋白质组中发现新的抗真菌分子,采用迁移学习技术,在AMPs上对模型进行预训练,克服了缺乏关于抗真菌肽的大型数据集问题,加速了抗真菌药物的开发;SUN等[45]设计一种GCN框架来识别乳酸菌抗菌肽(LABAMPs),LABAMPs是在乳酸菌代谢过程中产生的一类活性多肽,可以抑制或杀灭食品中的病原菌或变质菌,实验基于氨基酸、三肽和它们之间的关系构建异质图,并学习了图卷积神经网络的权重,对2个训练数据集进行了10折交叉验证实验,分别获得了0.916 3和0.937 9的准确率,与其他算法相比都要高;CAO等[46]将几种自然语言处理模型结合起来设计和识别AMPs,包括GAN、BERT、多层感知器(multilayer perceptron,MLP),此模型能够处理和分析蛋白质序列数据,从而从中发现潜在的AMPs;LEE等[47]开发了一个名为AMP-BERT的改进AMPs分类模型,采用经过微调的双向编码器表示来自BERT架构的深度学习模型,旨在从输入肽中提取结构或功能信息,并将每个输入识别为AMPs或非AMPs,并确定了已知AMPs中具有抗微生物功能的特定残基;LI等[48]开发了一种用于预测肽序列数据库中AMPs的注意力深度学习模型,AMPlify,分别在平衡和不平衡数据集上训练出2组模型,这2种模型将用于不同的用例,有助于新型AMPs的发现和开发;SHARMA等[49]开发一种名为ESKAPEE-MICpred的利用迁移学习和集成学习的两输入模型,集成学习的概念通过组合深度学习算法提供的决策实现,而迁移学习的概念则是利用预训练的氨基酸嵌入实现的,旨在解决耐药性病原体对AMPs的最低抑菌浓度值的预测问题;TEUFEL等[50]提出名为DeepPeptide的深度学习模型,可以直接从氨基酸序列预测被切割的肽,相比以前的方法,此模型显示了更高的肽预测精度和召回率,并且能够在为充分注释的蛋白组中识别肽;YANG等[51]提出了一种基于序列衍生和生命语言嵌入的级联计算模型,称为AMPFinder,不仅可以用来识别AMPs还可以用来预测其功能类型,AMPFinder在独立测试数据集上表现出更好的性能,F1分数提高了4.68%,MCC提高了9.94%,AUC提高了3.34%,AP提高了11.87%,通过10倍交叉验证,在公共数据集上AMPFinder的R2偏差降低了0.64%;ZHANG等[52]提出一个跨膜态框架,SenseXAMP,利用肽序列的语义嵌入和蛋白质描述符来提升AMPs的识别效果,SenseXAMP包括一个多输入对齐模块和交叉表示融合模块,以探索2个输入特征之间的隐藏信息,并更好地利用融合特征,更有效的从大量候选肽中识别AMPs;ZHUANG等[53]将2种类型的词嵌入特征与肽序列的统计特征结合起来,开发了名为EnAMP的集成分类器,分别使用Word2vec和Glove词嵌入方法来训练了2个深度神经网络,同时利用肽序列的统计特征训练了随机森林和支持向量机分类器,4个分类器的平均值是最终的预测结果,EnAMP在具有相似计算成本的情况下优于大多数现有模型;SUN等[45]开发了一个GCN框架来识别LABAMPs,作者基于氨基酸、三肽及其之间的关系构建了异构图,并学习了GCN的权重,对2个训练数据集进行了10折交叉验证实验,分别获得了0.916 3和0.937 9的准确率,比其他机器学习和GNN算法均高。
以上总结了近5年来各种深度学习模型的开发和应用,提出了一些新的AMPs预测方法和模型,如表1所示,如基于变压器架构的模型、集成学习模型等,这些模型在分类AMPs方面表现出较高的准确性和效率,例如使用嵌入层和多尺度卷积网络设计的模型,能够在AMPs数据集和AMPs数据库上取得较高的准确性;利用GAN、双向编码器和多层感知器等模型,设计了能够识别AMPs的模型,并发现了其中的重要模式;运用深度学习模型和分子动力学模拟筛选出具有抗微生物活性的肽;结合NLP模型和蛋白质编码方法开发了AMPs分类模型,表现优于先前的方法等。
表1 基于深度学习识别和筛选AMPs方法总结
Table 1 Summary of methods for identifying and screening AMPs based on deep learning

名称类型描述年份ACEP融合模型CNN捕获氨基酸之间的相似性2020Deep-AmPEP30CNN可从基因组序列中识别短长度AMPs2020蛋白质编码与深度学习模型结合CNN预测给定蛋白质序列的抗菌性能并进行蛋白组筛选2021AniAMPpredRNN对多样的肽段长度进行高精度的AMPs和非AMPs分类2021Deep-ABPpredRNN在蛋白质序列中识别新型AMPs2021BERT模型进行预训练NLP有助于捕捉新数据的特定特征和AMPs序列之间的共同特征2021Deep-ABPpredRNN用于在蛋白质序列中识别新的AMPs2022基于时间卷积网络的方法RNN精度预测AMPs2022图卷积神经网络框架GCN比其他机器学习和GNN算法的准确率都要高2022集成分类器NLP几种NLP模型结合起来设计和识别AMPs2023AMP-BERT模型NLPAMP-BERT能够捕捉肽的结构特性进行模型学习2023AMPlify模型NLP有效捕获肽序列中的重要信息2023DeepPeptideRNN能够在未充分注释的蛋白组中识别肽2023AMPFinderNLP准确识别AMPs及其功能类型2023LSTM_PepLSTM生成具有特定潜在治疗效果的新型肽2023SenseXAMP神经网络结构利用蛋白质序列的语义嵌入和蛋白质描述符来提高AMPs的识别性能2023
2.2 通过深度学习进行AMPs的生成和设计
在生物进化过程中,通常会出现新的分子或生物体,它们具有与已知的分子或生物体相似的功能,但在氨基酸序列和结构上变化很小,这种趋势可能会限制新序列的多样性,因为它们可能受到共同祖先的限制,因此有必要生成和设计新的AMPs,以求更多样化的功能。利用深度学习的方法可以更高效的实现抗菌肽的生成与设计(见图3)。
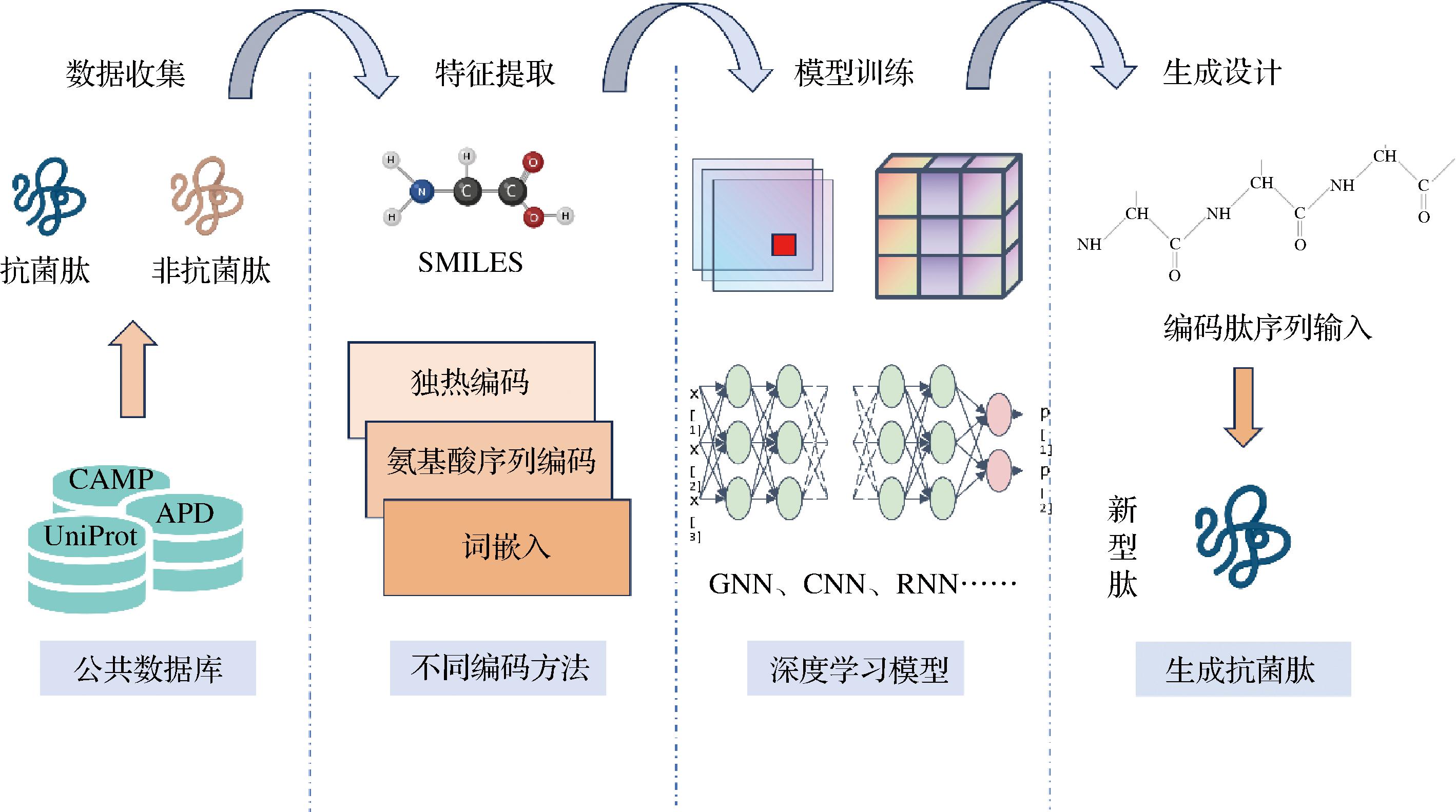
图3 利用深度学习生成设计新型AMPs
Fig.3 Generating and designing novel antimicrobial peptides using deep learning
DEAN等[54]以抗微生物肽作为模型生物分子,在已知抗微生物肽数据库上训练了一个生成式深度学习算法,通过训练一个变分自动编码器(variational auto-encoder, VAE)模型,该模型能够将已知的抗微生物肽数据库中的信息转换为潜在空间中的表示,并且可以在此空间中进行插值以生成新的肽序列,这些新序列可能具有抗微生物活性,并且可以作为潜在的药物候选物;一年后,DEAN等[55]又提出了一种基于VAE和AMPs活性预测模型的肽生成框架PepVAE,用于设计新型AMPs,并且只使用序列和实验最小抑菌浓度数据作为输入,通过对PepVAE生成的序列和抗菌活性预测模型进行广泛的分析,展示了通过预测的抗菌活性进行了实验验证的AMPs的可控生成;WANG等[56]构建了一种LSTM生成模型和一个双向LSTM分类模型,用于设计针对大肠杆菌的潜在抗菌活性的短型新AMPs序列,通过贝叶斯超参数优化,训练和优化了2个版本的生成模型和6个版本的分类模型,这些模型可用于生成1组被分类为抗微生物或非抗微生物的短肽新序列;HASEGAWA等[57]提出了Feedback-AVPGAN,旨在计算生成新型抗病毒肽(anti-virus peptide, AVP)的系统,该系统依赖于对抗网络模型(GAN)和反馈方法的关键前提,GAN是一种使用深度学习方法的生成建模方法,包括一个生成器和一个鉴别器,生成器用于生成肽,生成的肽被反馈给鉴别器以区分AVP和非AVP,使用反馈方法使鉴别器能够从现有的以及生成的合成数据中学习,从而可以解决目前实验获得AVP不多的问题;MAO等[58]提出了一种基于生成网络的合理设计AMPs的方法,称为AMPTrans-LSTM,涉及预训练、迁移学习和模块识别,AMPTrans-LSTM模型有2个子模型,即LSTM和Transformer,它们可以串联连接,充分利用LSTM的稳定性和Transformer模型的新颖性,作者通过分析生成的序列和训练过的AMPs,证明了AMPTrans-LSTM可以拓展训练过的AMPs的设计空间,并产生合理且全新的AMPs序列;PANDI等[59]创新性的将深度学习和无细胞蛋白质合成(cell free protein synthesis, CFPS)技术结合起来,作者使用深度学习设计了数千个AMPs候选肽,并利用计算方法对它们进行了筛选,最终确定了500个优先考虑的候选肽,并将这些候选肽通过CFPS管道进行筛选,最后确定6个AMPs具有光谱活性,可以抵御多重耐药性病原体,并且不会使细菌产生耐药性,作者强调了CFPS技术在生产和测试生物活性肽方面的高通量和低成本的潜力,且该过程仅需要不到24 h;TU S等[60]开发了一种基于离散潜在空间和D-Wave量子退火器的多目标肽设计流水线,通过非支配排序将多个肽特性编码为得分,并使用量子退火器进行优化,该方法有效地增加了抗微生物肽的种类,解决多药耐药细菌病原体的挑战提供了新的思路和方法;ZHANG等[61]建立了一个LSTM模型(LSTM_Pep)来生成新型肽,并对该模型进行了微调,以生成具有特定潜在治疗效果的新型肽;DONG等[62]开发了1个深度学习流水线,其中包括生成器和分类器,使用迁移学习和预训练蛋白质嵌入,对公开可用的数据进行了训练来设计针对痤疮丙酸杆菌(Propionibacterium acnes)具有特异性和抑制活性的肽,合成了一系列42个新的线性肽段,并对其抗微生物选择性和活性进行了评估,其中5种肽表现出对C.acnes的选择性,且最小抑菌浓度为2~4 μg/mL;YU等[63]提出了一种基于深度生成模型的架构Multi-CGAN,它可以从单属性肽数据中学习,并生成我们需要的具有多种属性的抗微生物肽序列,作者验证了Multi-CGAN生成的具有所需属性的肽在生成率方面表现良好。
S等[60]开发了一种基于离散潜在空间和D-Wave量子退火器的多目标肽设计流水线,通过非支配排序将多个肽特性编码为得分,并使用量子退火器进行优化,该方法有效地增加了抗微生物肽的种类,解决多药耐药细菌病原体的挑战提供了新的思路和方法;ZHANG等[61]建立了一个LSTM模型(LSTM_Pep)来生成新型肽,并对该模型进行了微调,以生成具有特定潜在治疗效果的新型肽;DONG等[62]开发了1个深度学习流水线,其中包括生成器和分类器,使用迁移学习和预训练蛋白质嵌入,对公开可用的数据进行了训练来设计针对痤疮丙酸杆菌(Propionibacterium acnes)具有特异性和抑制活性的肽,合成了一系列42个新的线性肽段,并对其抗微生物选择性和活性进行了评估,其中5种肽表现出对C.acnes的选择性,且最小抑菌浓度为2~4 μg/mL;YU等[63]提出了一种基于深度生成模型的架构Multi-CGAN,它可以从单属性肽数据中学习,并生成我们需要的具有多种属性的抗微生物肽序列,作者验证了Multi-CGAN生成的具有所需属性的肽在生成率方面表现良好。
以上总结了近5年来深度学习在AMPs设计领域的应用(见表2),包括几种深度学习方法,如VAE、LSTM、GAN等模型在AMPs设计和生成中的应用,通过结合深度学习模型和生成算法,研究人员能够从已知的抗微生物肽数据库中学习特征,并利用这些特征来生成具有潜在抗菌活性的新肽序列。这些方法不仅提高了AMPs的设计效率,还为克服多药耐药细菌的挑战提供了新的解决方案。深度学习技术的不断创新和发展将为未来抗微生物肽研究提供更多可能性,为生物医药领域的进步做出贡献。
表2 基于深度学习生成和设计AMPs方法总结
Table 2 Summary of methods for generating and designing AMPs based on deep learning

名称方法描述年份VAE模型VAE生成具有抗微生物活性的新肽序列的过程2020LSTM 生成模型LSTM设计针对大肠杆菌的潜在抗菌活性的新型肽序列2021Feedback-AVPGANGAN 高效地生成具有高概率表现出抗病毒活性的肽2022AMPTrans-lstm生成式深度学习算法产生合理且全新的AMPs序列2023深度学习和CFPS技术结合深度学习与其他技术结合设计了数千个 AMPs 候选肽,并利用 CFPS 进行筛选2023多目标肽设计流水线离散潜在空间基于离散潜在空间和 D-Wave 量子退火器的多目标肽设计流水线2023LSTM_PepLSTM生成具有特定潜在治疗效果的新型肽2023Multi-CGANGAN用于生成具有多种属性的抗微生物肽序列2024深度学习流水线深度生成模型包括生成器和分类器,用于设计具有特定特性的肽2024
2.3 通过深度学习进行AMPs结构性质预测
AMPs预测是指利用计算机算法和机器学习技术,通过分析氨基酸序列的特征和结构,来预测蛋白质分子是否具有抗菌活性(见图4)。AMPs是一类具有杀菌或抑菌作用的小分子肽类化合物,它们可以在生物体内或外表现出抗菌和抗真菌活性,预测抗AMPs的功能是一个重要且困难的问题,特别是当AMPs具有多重功能时,即一些AMPs同时具有2种或3种功能类别。
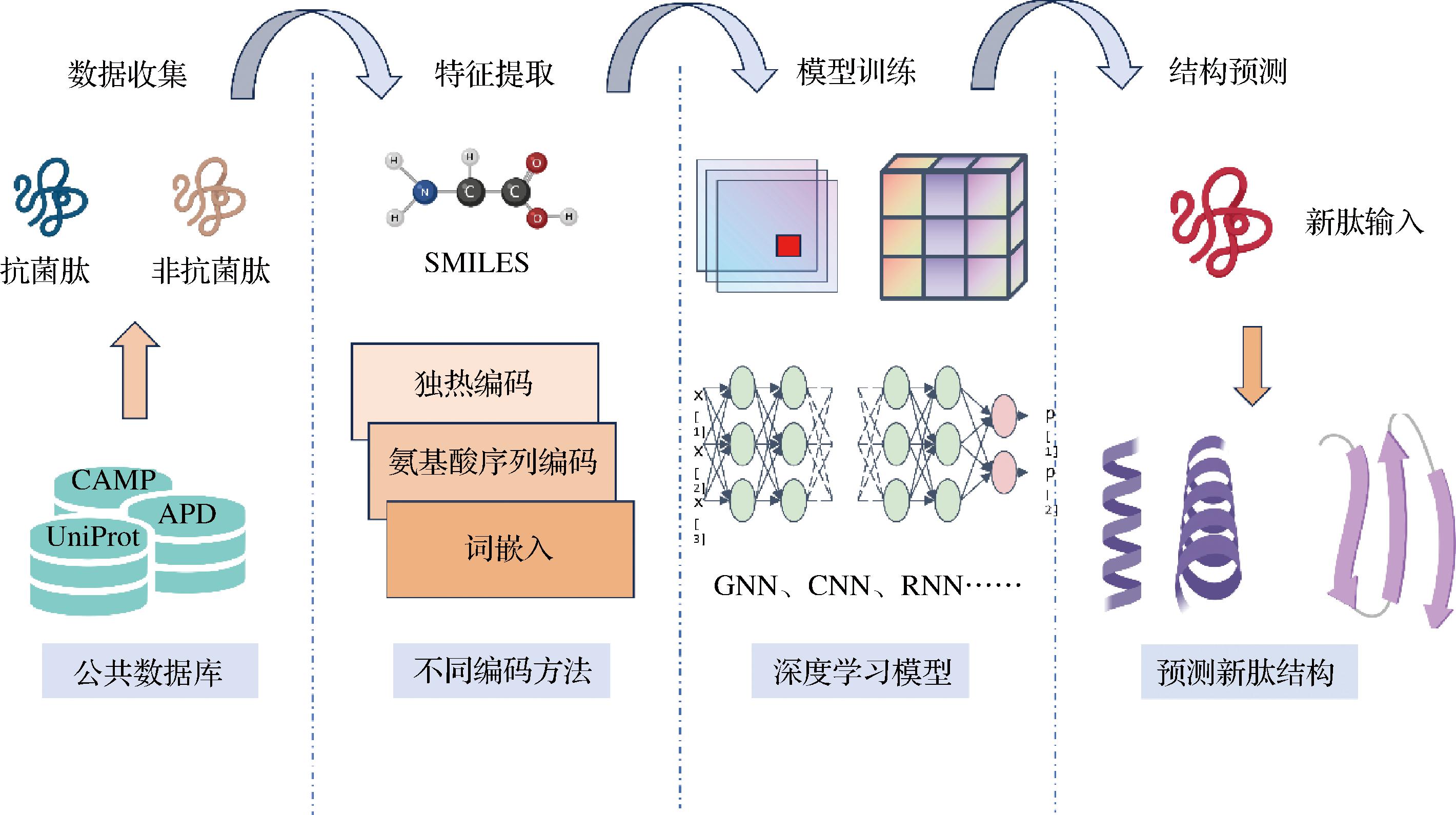
图4 利用深度学习模型预测AMPs结构
Fig.4 Predicting AMPs structures using deep learning models
XIAO等[64]开发了一种名为iAMP-CA2L的新型预测器,通过引入“CNN-BiLSTM-SVM分类器”和“元胞自动机图像”,可以处理同时包含单功能和多功能AMPs的系统,单功能AMPs指的是仅具有1种特定生物功能的AMPs,如抗菌、抗病毒或抗真菌等,多功能AMPs则是指能够同时执行多种生物功能的AMPs,例如既能杀菌又能调节免疫反应,iAMP-CA2L是一个两级的预测器,第一级是确定给定查询肽为AMPs或非AMP,而第二级是预测它是否属于一个或多个功能类型,通过实验证明与原有的预测器相比,iAMP-CA2L的预测性能有了很大的改善;LEE等[65]开发了1个深度学习模型来预测物种特异性的抗微生物活性,采用了多任务学习方法,分别对枯草芽孢杆菌、大肠杆菌、铜绿假单胞菌、金黄色葡萄球菌和表皮葡萄球菌实现了F1分数为0.818、0.696、0.814、0.787和0.719,有效克服了数据不足的问题,同时可以挖掘出具有高效活性的AMPs;LI等[66]开发了一种名为AMPlify的关注深度学习模型,用于预测AMPs的生物活性,该模型在优先考虑来自牛蛙基因组的肽序列的情况下展示了其实用性,并且对一系列细菌物种的生物活性进行了测试,包括对世界卫生组织优先病原体清单中的代表,研究发现,他们预测的4个对多个细菌物种具有活性的新型AMPs,包括对耐碳青霉烯酶产生耐药性的大肠杆菌的活性;RUIZ等[67]提出了1种深度学习模型,称为AMPs-Net,通过将深度学习与分子动力学模拟相结合,预测了大量AMPs的抗菌和抗病毒能力,有效推进了多数AMPs候选物中多数未能显示出实际应用所需的活性和物理化学性质问题的解决方法。
由于深度学习模型通常需要大量数据进行训练,而抗微生物肽的数据量有限,SALEM等[68]为研究抗微生物肽溶血活性预测,收集了3种不同的数据集,并使用了一个名为AMPDeep的模型进行实验,利用迁移学习来克服数据稀缺,并成功采用基于深度学习的模型进行抗微生物肽的溶血活性分类;TANG等[69]开发了名为MLBP(Multi-Label深度学习方法用于确定生物活性肽的多功能性)的方法,此模型以肽序列向量作为输入,以替代其他肽预测器中使用的生物和生理化学特征。使用嵌入层,从序列向量中学习密集的连续特征向量。然后,通过CNN层从特征向量中提取卷积特征,并与双向门控循环单元层相结合,以提高预测性能,它可以同时预测包括抗癌、抗糖尿病、降压、抗炎和抗微生物在内的多种功能;ANSARI等[70]在半监督设置中仅利用有限的已知正样本,通过无标签学习发现可能与某些抗菌特性相关的肽序列,使用了2种学习策略:调整基本分类器和可靠负样本识别,构建了深度学习模型,以预测肽的溶解度、溶血性、与SHP-2的结合以及非污染活性,实验证明其优于传统的正负分类方法;LOBANOV等[71]使用BERT变换器创建序列嵌入,并使用多层感知器和轻注意力机制方法进行分类。其中一个在基准测试中达到了约80%的准确率和特异性,与现有的最佳方法不相上下;RANDALL等[72]介绍了1种深度突变表面定位抗微生物展示(dmSLAY)的方法,以此来揭示抗微生物肽在蛋白质序列中的各个位置的重要性和灵活性,并将此方法应用于一种具有潜力但有毒的抗微生物肽Protegrin-1,鉴定出了数千种序列变体,这些变体对其抗菌活性产生积极或消极影响,作为创新的、高通量的方法,用于阐明抗微生物肽序列-结构-功能关系;THOKKADAM等[73]开发出可以预测Ubonodin变体的RNAP抑制活性的深度学习模型,称为DeepLasso,Ubonodin是一种环肽,对机会性致病菌(Bcc)物种具有良好的活性,通过抑制易感细菌中的RNA聚合酶起作用;WANG等[74]提出了名为AMP-EBiLSTM的深度学习策略,用于准确预测AMPs,并将其性能与集成学习和基准模型进行了比较,实验中利用二进制剖面特征和伪氨基酸组成来进行有效的局部序列捕获和氨基酸信息提取,通过深度学习和集成学习实现了这一目标,有望更准确地预测AMPs;XING等[75]提出了1种新的AMPs预测器,称为iAMP-Attenpred,这是第一次在NLP领域使用流行的BERT模型进行AMPs特征编码,并组合多个模型来发现AMPs,作者将经过预处理的AMPs和非AMPs序列中的每个氨基酸都视为1个单词,然后将其输入到BERT预训练模型中进行特征提取。此外,从BERT方法获取的特征被反馈送到由一维CNN、BiLSTM和注意力机制组成的复合模型中,以更好地区分特征,最后采用扁平化层和各种全连接层进行AMPs的最终分类,此预测器实现了更好的性能指标,如准确性、精确度;XU等[76]构建了全面的AMPs数据集,并提出了一种新的基于深度学习的框架,即iAMPCN(基于CNNs的AMPs识别),来识别AMPs及其相关的22种功能活性,实验证明,iAMPCN在4种类型序列特征基础上显著提高了AMPs及其相关功能活性的预测性能;YAN等[77]提出了MBC-Attention,这是一个多分支CNN架构和注意力机制的组合,用于预测AMPs对大肠杆菌的实验最小抑制浓度,最佳的MBC-Attention模型在3次独立测试中,从数据集中随机抽取的序列中,达到了平均皮尔逊相关系数为0.775,均方根误差为0.533,有利于定量评估AMPs的抗微生物活性;CHEN等[78]构建了名为Deep2Pep(Deep learning to Peptides)新的深度学习方法,它基于序列编码、嵌入和语言标记器,可以通过将序列信息转换为数字向量,结合BiLSTM、注意力-残差算法和BERT编码器,实现对抗菌、降压、抗氧化和降糖肽的预测,其研究使用深度学习算法准确预测了肽的4种活性功能。
以上总结了近5年来深度学习在AMPs结构性质预测上的研究应用和进展(见表3),以介绍不同AMPs预测方法和模型的开发为主线,通过性能评估和实验证明来展示它们的有效性和创新性,并强调了这些方法在解决AMPs预测问题上的重要意义。
表3 基于深度学习预测AMPs结构性质方法总结
Table 3 Summary of methods for predicting AMPs structural and properties based on deep learning

名称类型描述年份iAMP-CA2LCNN引入了CNN-BiLSTM-SVM分类器,处理同时包含单功能和多功能AMPs的系统2021多任务学习方法多任务学习实现对不同菌株的抗微生物活性预测2022AMPlify深度学习模型用于预测AMPs的生物活性,结合了对细菌物种的生物活性测试2022
续表3

名称类型描述年份AMPs-Net深度学习模型高度准确地预测大量AMPs的抗菌和抗病毒能力2022MLBPRNN使用BiLSTM结合卷积神经网络层,实现对多种功能的预测2022AMP-EBiLSTM集成学习利用二进制剖面特征和伪氨基酸组成,预测AMPs2023iAMPCNCNN基于CNNs的AMPs识别,用于识别AMPs及其相关的22种功能活性2023MBC-AttentionTransformer用于预测AMPs对大肠杆菌的实验最小抑制浓度2023Deep2Pep深度生成模型结合BiLSTM、注意力-残差算法和BERT编码器,预测肽的多种活性功能2024
目前预测蛋白质三维结构的深度学习模型—AlphaFold基本解决了从氨基酸序列出发预测蛋白质的三维结构这一问题,人类基因组携带了超过2万个蛋白质的指令,但只有约1/3蛋白质的三维结构通过实验方法得到解析,如蛋白质晶体X射线衍射、冷冻电镜、核磁共振等,而这些蛋白质的结构只确定了其中的一部分,而AlphaFold预测的结构几乎完整的覆盖了人类蛋白质组,目前AlphaFold已有3个版本,最初于2018年发行,采用了经典的CNN架构,虽然与实验室测定的结构仍有差距,但显示了深度学习在蛋白质结构预测方面的巨大潜力,AlphaFold 2于2020年问世,采用了一种名为“attention mechanism”的技术,引入能力更强的Transformer架构,这种技术在处理蛋白质序列时更加准确和高效;而在最近,2024年5月9号,AlphaFold 3登上Nature,据介绍,AlphaFold 3几乎精确地预测了绝大部分生命分子(蛋白质、DNA、RNA、配体等)的结构和相互作用,其模拟预测生命的能力突飞猛进,因此也意味着深度学习模型在预测AMPs结构方面有着更广阔的前景。
2.4 限制和挑战
深度学习在发现和设计AMPs方面的应用可以节省大量人力和时间,表现出突出的优势,然而目前深度学习在AMPs研究的使用仍面临着很多限制与挑战。首先这些深度学习模型大多基于监督学习,需要大量经过验证的AMPs数据集进行训练。与在计算机视觉和NLP等其他领域中广泛使用的方法相比,在AMPs领域可用的数据量非常少[79],因此,如何克服数据限制问题是非常重要的,为了解决这一问题,需要采用适当的正则化技术、数据增强方法以及模型选择和调参等策略来提高模型的泛化能力。其次,AMPs具有多种生物学特性,包括氨基酸序列、结构、亲水性、电荷分布等。这些特性与其抗菌活性密切相关,但也受到其他因素如目标微生物的影响。因此,设计和训练深度学习模型需要考虑这些复杂的生物学特性,并且需要在模型中充分表达这些信息以获得准确的预测结果[80]。再次,多数AMPs预测方法都是二元分类,即只预测AMPs和非AMPs,因此预测AMPs的生物活性是有价值的。在AMPs研究中,通常存在着正负样本不平衡的情况,即活性和非活性AMPs的比例不一致。这导致模型在训练过程中可能会偏向于预测主导类别,而忽略罕见类别。此外,数据的获取和标记可能会受到限制,导致数据集的规模有限,这进一步增加了模型训练的挑战,这种多类或多标签预测问题对数据集提出了更高的要求,其中不平衡和缺失的数据属性对训练深度学习模型构成了重大挑战。同时,深度学习模型通常被认为是黑盒模型,可解释性差,难以解释其预测结果的原因。在AMPs研究中,研究人员需要能够理解模型是如何基于输入数据做出预测的,以便验证模型的可靠性并指导进一步的实验设计。因此,开发可解释性强的深度学习模型成为一项重要的挑战。最后,深度学习模型通常具有大量的超参数和模型结构选择的自由度,如学习率、层数、节点数等。优化这些参数并选择最佳的模型结构对于获得高性能的深度学习模型至关重要。然而,这一过程通常需要耗费大量的计算资源和时间,而且需要对领域知识有深入的理解才能进行有效的调参和优化。
3 总结
深度学习在AMPs设计、发现与预测中的应用是当前研究的热点之一。本文综述了目前深度学习在该领域的现状和未来展望。首先,介绍了由于抗生素滥用导致的细菌耐药性问题,强调了AMPs的广阔应用前景及其独特的作用机制和优势。随后,详细分析了深度学习在AMPs研究中的优势,包括其高效性和广泛适用性。本文分别梳理总结了近年来通过深度学习进行AMPs识别和筛选、生成和设计、结构性质预测等方面的主要研究成果。
尽管取得了显著进展,目前的研究仍面临一些限制和挑战,如数据不足、模型的泛化能力有限等。本文讨论了未来的发展方向,包括引入更多的深度学习技术和模型,以解决当前研究中的挑战。总的来说,深度学习在AMPs设计、发现与预测中的应用展现出了巨大的潜力,未来有望为AMPs研究和药物设计带来更多的创新和突破。
[1] CHENG Q P, ZENG P, CHAN E W C, et al.Development of peptide-based metallo-β-lactamase inhibitors as a new strategy to combat antimicrobial resistance:A mini-review[J].Current Pharmaceutical Design, 2022, 28(44):3538-3545.
[2] MORRIS C F.The antimicrobial properties of the puroindolines, a review[J].World Journal of Microbiology &Biotechnology, 2019, 35(6):86.
[3] SHI S B, SHEN T F, LIU Y Q, et al.Porcine myeloid antimicrobial peptides:A review of the activity and latest advances[J].Frontiers in Veterinary Science, 2021, 8:664139.
[4] LI H X, NIU J H, WANG X L, et al.The contribution of antimicrobial peptides to immune cell function:A review of recent advances[J].Pharmaceutics, 2023, 15(9):2278.
[5] ALECU M, COMAN G, MU ETESCU A, et al.Antimicrobial peptides as an argument for the involvement of innate immunity in psoriasis (Review)[J].Experimental and Therapeutic Medicine, 2020, 20(6):192.
ETESCU A, et al.Antimicrobial peptides as an argument for the involvement of innate immunity in psoriasis (Review)[J].Experimental and Therapeutic Medicine, 2020, 20(6):192.
[6] LAW D, ABDULKAREEM NAJM A, CHONG J X, et al.In silico identification and in vitro assessment of a potential anti-breast cancer activity of antimicrobial peptide retrieved from the ATMP1 Anabas testudineus fish peptide[J].PeerJ, 2023, 11:e15651.
[7] FERNANDES F C, CARDOSO M H, GIL-LEY A, et al.Geometric deep learning as a potential tool for antimicrobial peptide prediction[J].Frontiers in Bioinformatics, 2023, 3:1216362.
[8] TEIXEIRA M C, CARBONE C, SOUSA M C, et al.Nanomedicines for the delivery of antimicrobial peptides (AMPs)[J].Nanomaterials, 2020, 10(3):560.
[9] MAKHLYNETS O V, CAPUTO G A.Characteristics and therapeutic applications of antimicrobial peptides[J].Biophysics Reviews, 2021, 2(1):011301.
[10] LI X, ZUO S Y, WANG B, et al.Antimicrobial mechanisms and clinical application prospects of antimicrobial peptides[J].Molecules, 2022, 27(9):2675.
[11] RIZZETTO G, GAMBINI D, MAURIZI A, et al.The sources of antimicrobial peptides against Gram-positives and Gramnegatives:Our research experience[J].Le Infezioni in Medicina, 2023, 31(3):306-322.
[12] ZHANG Q Y, YAN Z B, MENG Y M, et al.Antimicrobial peptides:Mechanism of action, activity and clinical potential[J].Military Medical Research, 2021, 8(1):48.
[13] ZAINAL BAHARIN N H, KHAIRIL MOKHTAR N F, MOHD DESA M N, et al.The characteristics and roles of antimicrobial peptides as potential treatment for antibiotic-resistant pathogens:A review[J].PeerJ, 2021, 9:e12193.
[14] LEE T H, HALL K N, AGUILAR M I.Antimicrobial peptide structure and mechanism of action:A focus on the role of membrane structure[J].Current Topics in Medicinal Chemistry, 2016, 16(1):25-39.
[15] VINEETH KUMAR T V, SANIL G.A review of the mechanism of action of amphibian antimicrobial peptides focusing on peptide-membrane interaction and membrane curvature[J].Current Protein &Peptide Science, 2017, 18(12):1263-1272.
[16] LAU Q Y, LI J G, SANI M A, et al.Elucidating the bactericidal mechanism of action of the linear antimicrobial tetrapeptide BRBR-NH2[J].Biochimica et Biophysica Acta.Biomembranes, 2018, 1860(8):1517-1527.
[17] YASIR M, DUTTA D, WILLCOX M D P.Comparative mode of action of the antimicrobial peptide melimine and its derivative Mel4 against Pseudomonas aeruginosa[J].Scientific Reports, 2019, 9(1):7063.
[18] DASH R, BHATTACHARJYA S.Thanatin:An emerging host defense antimicrobial peptide with multiple modes of action[J].International Journal of Molecular Sciences, 2021, 22(4):1522.
[19] JUHL D W, GLATTARD E, AISENBREY C, et al.Antimicrobial peptides:Mechanism of action and lipid-mediated synergistic interactions within membranes[J].Faraday Discussions, 2021, 232:419-434.
[20] LIZIO M G, CAMPANA M, DE POLI M, et al.Insight into the mechanism of action and peptide-membrane interactions of aib-rich peptides:Multitechnique experimental and theoretical analysis[J].ChemBioChem, 2021, 22(9):1656-1667.
[21] JI F Y, ZHAO Y, JIANG F Q, et al.Membrane mechanism of temporin-1CEc, an antimicrobial peptide isolated from the skin secretions of Rana chensinensis, and its systemic analogs[J].Bioorganic Chemistry, 2022, 119:105544.
[22] RIESCO-LLACH G, LLANET-FERRER S, PLANAS M, et al.Deciphering the mechanism of action of the antimicrobial peptide BP100[J].International Journal of Molecular Sciences, 2024, 25(6):3456.
[23] BOONE K, WISDOM C, CAMARDA K, et al.Combining genetic algorithm with machine learning strategies for designing potent antimicrobial peptides[J].BMC Bioinformatics, 2021, 22(1):239.
[24] AKBAR S, RAZA A, ZOU Q.Deepstacked-AVPs:Predicting antiviral peptides using tri-segment evolutionary profile and word embedding based multi-perspective features with deep stacking model[J].BMC Bioinformatics, 2024, 25(1):102.
[25] HADJICHARALAMBOUS A, BOURNAKAS N, NEWMAN H, et al.Antimicrobial and cell-penetrating peptides:Understanding penetration for the design of novel conjugate antibiotics[J].Antibiotics, 2022, 11(11):1636.
[26] LUO Y, SONG Y Z.Mechanism of antimicrobial peptides:Antimicrobial, anti-inflammatory and antibiofilm activities[J].International Journal of Molecular Sciences, 2021, 22(21):11401.
[27] BATTISTA F, OLIVA R, DEL VECCHIO P, et al.Insights into the action mechanism of the antimicrobial peptide lasioglossin III[J].International Journal of Molecular Sciences, 2021, 22(6):2857.
[28] RICILUCA K C T, OLIVEIRA U C, MENDONÇA R Z, et al.Rondonin:Antimicrobial properties and mechanism of action[J].FEBS Open Bio, 2021, 11(9):2541-2559.
[29] GUO F L, ZHANG Y, DONG W B, et al.Effect of hydrophobicity on distinct anticancer mechanism of antimicrobial peptide chensinin-1b and its lipoanalog PA-C1b in breast cancer cells[J].The International Journal of Biochemistry &Cell Biology, 2022, 143:106156.
[30] ZHANG M, YU Y L, LIAN L L, et al.Functional mechanism of antimicrobial peptide bomidin and its safety for Macrobrachium rosenbergii[J].Probiotics and Antimicrobial Proteins, 2022, 14(1):169-179.
[31] SU X, XU J, YIN Y B, et al.Antimicrobial peptide identification using multi-scale convolutional network[J].BMC Bioinformatics, 2019, 20(1):730.
[32] FU H Y, CAO Z C, LI M Y, et al.ACEP:Improving antimicrobial peptides recognition through automatic feature fusion and amino acid embedding[J].BMC Genomics, 2020, 21(1):597.
[33] YAN J L, BHADRA P, LI A, et al.Deep-AmPEP30:Improve short antimicrobial peptides prediction with deep learning[J].Molecular Therapy-Nucleic Acids, 2020, 20:882-894.
[34] PUENTES P R, HENAO M C, TORRES C E, et al.Design, screening, and testing of non-rational peptide libraries with antimicrobial activity:In silico and experimental approaches[J].Antibiotics, 2020, 9(12):854.
[35] DAS P, SERCU T, WADHAWAN K, et al.Accelerated antimicrobial discovery via deep generative models and molecular dynamics simulations[J].Nature Biomedical Engineering, 2021, 5(6):613-623.
[36] LIN T T, YANG L Y, LU I H, et al.AI4AMP:An antimicrobial peptide predictor using physicochemical property-based encoding method and deep learning[J].mSystems, 2021, 6(6):e0029921.
[37] SHARMA R, SHRIVASTAVA S, KUMAR SINGH S, et al.AniAMPpred:Artificial intelligence guided discovery of novel antimicrobial peptides in animal Kingdom[J].Briefings in Bioinformatics, 2021, 22(6):bbab242.
[38] SHARMA R, SHRIVASTAVA S, KUMAR SINGH S, et al.Deep-ABPpred:Identifying antibacterial peptides in protein sequences using bidirectional LSTM with word2vec[J].Briefings in Bioinformatics, 2021, 22(5):bbab065.
[39] ZHANG Y, LIN J Y, ZHAO L M, et al.A novel antibacterial peptide recognition algorithm based on BERT[J].Briefings in Bioinformatics, 2021, 22(6):bbab200.
[40] DEE W.LMPred:Predicting antimicrobial peptides using pre-trained language models and deep learning[J].Bioinformatics Advances, 2022, 2(1):vbac021.
[41] MA Y, GUO Z Y, XIA B B, et al.Identification of antimicrobial peptides from the human gut microbiome using deep learning[J].Nature Biotechnology, 2022, 40(6):921-931.
[42] PANG Y X, YAO L T, XU J Y, et al.Integrating transformer and imbalanced multi-label learning to identify antimicrobial peptides and their functional activities[J].Bioinformatics, 2022, 38(24):5368-5374.
[43] SINGH V, SHRIVASTAVA S, KUMAR SINGH S, et al.StaBle-ABPpred:A stacked ensemble predictor based on biLSTM and attention mechanism for accelerated discovery of antibacterial peptides[J].Briefings in Bioinformatics, 2022, 23(1):bbab439.
[44] SINGH V, SHRIVASTAVA S, KUMAR SINGH S, et al.Accelerating the discovery of antifungal peptides using deep temporal convolutional networks[J].Briefings in Bioinformatics, 2022, 23(2):bbac008.
[45] SUN T J, BU H L, YAN X, et al.LABAMPsGCN:A framework for identifying lactic acid bacteria antimicrobial peptides based on graph convolutional neural network[J].Frontiers in Genetics, 2022, 13:1062576.
[46] CAO Q S, GE C, WANG X J, et al.Designing antimicrobial peptides using deep learning and molecular dynamic simulations[J].Briefings in Bioinformatics, 2023, 24(2):bbad058.
[47] LEE H, LEE S, LEE I, et al.AMP-BERT:Prediction of antimicrobial peptide function based on a BERT model[J].Protein Science, 2023, 32(1):e4529.
[48] LI C K, WARREN R L, BIROL I.Models and data of AMPlify:A deep learning tool for antimicrobial peptide prediction[J].BMC Research Notes, 2023, 16(1):11.
[49] SHARMA R, SHRIVASTAVA S, SINGH S K, et al.Artificial intelligence-based model for predicting the minimum inhibitory concentration of antibacterial peptides against ESKAPEE pathogens[J].IEEE Journal of Biomedical and Health Informatics, 2024, 28(4):1949-1958.
[50] TEUFEL F, REFSGAARD J C, MADSEN C T, et al.DeepPeptide predicts cleaved peptides in proteins using conditional random fields[J].Bioinformatics, 2023, 39(10):btad616.
[51] YANG S, YANG Z X, NI X Y.AMPFinder:A computational model to identify antimicrobial peptides and their functions based on sequence-derived information[J].Analytical Biochemistry, 2023, 673:115196.
[52] ZHANG W T, XU Y C, WANG A W, et al.Fuse feeds as one:Cross-modal framework for general identification of AMPs[J].Briefings in Bioinformatics, 2023, 24(6):bbad336.
[53] ZHUANG J J, GAO W Q, SU R.EnAMP:A novel deep learning ensemble antibacterial peptide recognition algorithm based on multi-features[J].Journal of Bioinformatics and Computational Biology, 2024, 22(1):2450001.
[54] DEAN S N, WALPER S A.Variational autoencoder for generation of antimicrobial peptides[J].ACS Omega, 2020, 5(33):20746-20754.
[55] DEAN S N, ALVAREZ J A E, ZABETAKIS D, et al.PepVAE:Variational autoencoder framework for antimicrobial peptide generation and activity prediction[J].Frontiers in Microbiology, 2021, 12:725727.
[56] WANG C, GARLICK S, ZLOH M.Deep learning for novel antimicrobial peptide design[J].Biomolecules, 2021, 11(3):471.
[57] HASEGAWA K, MORIWAKI Y, TERADA T, et al.Feedback-AVPGAN:Feedback-guided generative adversarial network for generating antiviral peptides[J].Journal of Bioinformatics and Computational Biology, 2022, 20(6):2250026.
[58] MAO J S, GUAN S H, CHEN Y Q, et al.Application of a deep generative model produces novel and diverse functional peptides against microbial resistance[J].Computational and Structural Biotechnology Journal, 2023, 21:463-471.
[59] PANDI A, ADAM D, ZARE A, et al.Cell-free biosynthesis combined with deep learning accelerates de novo-development of antimicrobial peptides[J].Nature Communications, 2023, 14(1):7197.
[60] TU S A, BERENGER F, YUMOTO A, et al.Quantum annealing designs nonhemolytic antimicrobial peptides in a discrete latent space[J].ACS Medicinal Chemistry Letters, 2023, 14(5):577-582.
S A, BERENGER F, YUMOTO A, et al.Quantum annealing designs nonhemolytic antimicrobial peptides in a discrete latent space[J].ACS Medicinal Chemistry Letters, 2023, 14(5):577-582.
[61] ZHANG H P, SARAVANAN K M, WEI Y J, et al.Deep learning-based bioactive therapeutic peptide generation and screening[J].Journal of Chemical Information and Modeling, 2023, 63(3):835-845.
[62] DONG Q C, WANG S H, MIAO Y, et al.Novel antimicrobial peptides against Cutibacterium acnes designed by deep learning[J].Scientific Reports, 2024, 14(1):4529.
[63] YU H Q, WANG R H, QIAO J B, et al.Multi-CGAN:Deep generative model-based multiproperty antimicrobial peptide design[J].Journal of Chemical Information and Modeling, 2024, 64(1):316-326.
[64] XIAO X, SHAO Y T, CHENG X, et al.iAMP-CA2L:A new CNN-BiLSTM-SVM classifier based on cellular automata image for identifying antimicrobial peptides and their functional types[J].Briefings in Bioinformatics, 2021, 22(6):bbab209.
[65] LEE B, SHIN M K, YOO J S, et al.Identifying novel antimicrobial peptides from venom gland of spider Pardosa astrigera by deep multi-task learning[J].Frontiers in Microbiology, 2022, 13:971503.
[66] LI C K, SUTHERLAND D, HAMMOND S A, et al.AMPlify:Attentive deep learning model for discovery of novel antimicrobial peptides effective against WHO priority pathogens[J].BMC Genomics, 2022, 23(1):77.
[67] RUIZ PUENTES P, HENAO M C, CIFUENTES J, et al.Rational discovery of antimicrobial peptides by means of artificial intelligence[J].Membranes, 2022, 12(7):708.
[68] SALEM M, KESHAVARZI ARSHADI A, YUAN J S.AMPDeep:Hemolytic activity prediction of antimicrobial peptides using transfer learning[J].BMC Bioinformatics, 2022, 23(1):389.
[69] TANG W D, DAI R Y, YAN W H, et al.Identifying multi-functional bioactive peptide functions using multi-label deep learning[J].Briefings in Bioinformatics, 2022, 23(1):bbab414.
[70] ANSARI M, WHITE A D.Learning peptide properties with positive examples only[J].BioRxiv, 2023:543289.
[71] LOBANOV M Y, SLIZEN M V, DOVIDCHENKO N V, et al.Comparison of deep learning models with simple method to assess the problem of antimicrobial peptides prediction[J].Molecular Informatics, 2024, 43(5):e202200181.
[72] RANDALL J R, VIEIRA L C, WILKE C O, et al.Deep mutational scanning and machine learning uncover antimicrobial peptide features driving membrane selectivity[J].BioRxiv, 2023:551017.
[73] THOKKADAM A, DO T, RAN X C, et al.High-throughput screen reveals the structure-activity relationship of the antimicrobial lasso peptide ubonodin[J].ACS Central Science, 2023, 9(3):540-550.
[74] WANG Y D, WANG L Y, LI C Q, et al.AMP-EBiLSTM:Employing novel deep learning strategies for the accurate prediction of antimicrobial peptides[J].Frontiers in Genetics, 2023, 14:1232117.
[75] XING W X, ZHANG J, LI C, et al.iAMP-Attenpred:A novel antimicrobial peptide predictor based on BERT feature extraction method and CNN-BiLSTM-Attention combination model[J].Briefings in Bioinformatics, 2023, 25(1):bbad443.
[76] XU J, LI F Y, LI C, et al.iAMPCN:A deep-learning approach for identifying antimicrobial peptides and their functional activities[J].Briefings in Bioinformatics, 2023, 24(4):bbad240.
[77] YAN J L, ZHANG B, ZHOU M L, et al.A deep learning method for predicting the minimum inhibitory concentration of antimicrobial peptides against Escherichia coli using Multi-Branch-CNN and Attention[J].mSystems, 2023, 8(4):e0034523.
[78] CHEN L H, HU Z K, RONG Y Z, et al.Deep2Pep:A deep learning method in multi-label classification of bioactive peptide[J].Computational Biology and Chemistry, 2024, 109:108021.
[79] YAN J L, CAI J X, ZHANG B, et al.Recent progress in the discovery and design of antimicrobial peptides using traditional machine learning and deep learning[J].Antibiotics, 2022, 11(10):1451.
[80] LEFIN N, HERRERA-BELÉN L, FARIAS J G, et al.Review and perspective on bioinformatics tools using machine learning and deep learning for predicting antiviral peptides[J].Molecular Diversity, 2024,28(4):2365-2374.