发酵蔬菜起源于我国3 100年前的商代武丁时期,发展至今,已成为中国、韩国、朝鲜、日本、欧洲及东南亚各国重要的发酵食品产业之一[1]。发酵蔬菜因富含有机酸、维生素、膳食纤维等营养物质以及有益微生物(如乳酸菌、片球菌、明串珠菌、汉逊德巴利酵母等)而具有多种生物学功能,且蔬菜发酵过程属于非热加工,相比于其他加工方式,发酵赋予了产品更好的口感、色泽和风味特征,使得发酵蔬菜产品倍受广大消费者喜爱[2]。然而,经过发酵后的蔬菜中亚硝酸盐含量会升高,这是由新鲜蔬菜从土壤中吸收大量的氮(氮化肥残留)转化而来。发酵过程中,蔬菜中的亚硝酸盐可与生物胺发生酰胺反应生成N-亚硝胺,同时亚硝酸盐经人体摄入后在胃酸环境中会与一些蛋白质代谢产生的胺类化合物反应生成N-亚硝胺,N-亚硝胺是强制癌物,可导致鼻咽癌、肝癌、食管癌和膀胱癌等发病率的上升[3]。因此,如何控制或减少发酵蔬菜中的亚硝酸盐含量成为发酵蔬菜工业生产中亟需解决的问题。大量研究表明,蔬菜自然发酵过程中,亚硝酸盐含量在发酵初期增加,出现“亚硝峰”,发酵结束时降到较低水平[4-6],而对亚硝酸盐上升及下降的具体原因和途径的研究较少,因此深入了解蔬菜发酵过程中亚硝酸盐形成及降解机制对于更好地控制发酵蔬菜中的亚硝酸盐含量至关重要。
蔬菜发酵体系中含有丰富的微生物菌系,微生物代谢活动复杂,所形成的代谢产物如有机酸、酶等可能对亚硝酸盐含量产生重要影响[7-8]。因此,对亚硝酸盐代谢功能基因及相关功能微生物的探究不仅有助于揭示发酵蔬菜中亚硝酸盐形成及降解机制,也为微生物手段调控亚硝酸盐含量的应用提供参考。截至目前,对微生物与发酵蔬菜亚硝酸盐转化关系还缺少全面、系统的研究,且对发酵蔬菜的微生物菌群结构研究大多采用的是16S rRNA高通量测序技术,只能确定到微生物的属水平,不能确定具体的菌种种类,从而无法明确地为特定菌株筛选及功能特性研究提供指导。随着现代分子生物学技术的发展,宏基因组学在食品微生物领域探索物种及功能基因多样性分析的应用愈来广泛,不仅可以快速、全面、准确地挖掘出微生物基因组信息,而且能够进行功能及代谢通路的分析,基于这些信息,更便于挖掘菌群的生物多样性、群落结构、功能特性及相互关系[9-11]。本研究通过对取自工厂盐渍发酵1.5年的4种不同发酵蔬菜进行宏基因组测序,分析其微生物群落结构及主要代谢通路,并挖掘与亚硝酸盐代谢有关的关键功能基因和微生物,以期为发酵蔬菜中亚硝酸盐的降解机理解析及亚硝酸盐调控提供理论基础。
1 材料与方法
1.1 材料与试剂
盐渍发酵1.5年的榨菜、萝卜、芥菜、豇豆,采自眉山市某大型盐渍菜生产基地,经测定,4种工业发酵蔬菜中的亚硝酸盐含量均<0.3 mg/kg,远低于国家限量标准20 mg/kg。
PCA(plate count agar)培养基、VRBA(violet red bile agar)培养基、MRS(man rogosa sharpe)培养基、CFC(pseudomonas cfc selective agar)培养基,青岛高科技工业园海博生物技术有限公司;盐酸、亚铁氰化钾、乙酸锌、硼酸钠、对氨基苯磺酸、盐酸萘乙二胺、NaNO2标准品,成都市科隆化学品有限公司。
1.2 仪器与设备
TGL-16K高速冷冻离心机,湖南湘仪科技有限公司;BSP-150生化培养箱,上海博讯实业有限公司医疗设备厂;721G可见分光光度计,上海仪电分析仪器有限公司;Seven Compact S210 pH计,梅特勒-托利多仪器(上海)有限公司。
1.3 实验方法
1.3.1 样品采集与处理
将蔬菜发酵液在4 ℃下8 000 r/min离心10 min,取沉淀送至上海美吉生物医药科技有限公司进行宏基因组测序。
1.3.2 测序流程
样品中的DNA用E.Z.N.A.® Soil DNA Kit试剂盒进行提取,提取的DNA用1%琼脂糖凝胶电泳进行检测。用Covaris M220仪将DNA随机打断成长度约400 bp的片段。使用NEXTFLEXTM Rapid DNA-Seq Kit试剂盒构建PE文库。采用Illumina NovaSeq Reagent Kits试剂盒进行桥式PCR。
1.3.3 生物信息分析流程
将原始测序数据使用Sickle 1.33软件剪切掉数据中的低质量及含N的reads,以获得用于后续分析的高质量序列。将获得的序列使用Megahit 1.1.2软件进行组装,再使用Prodigal 2.6.3(github.com/hyattpd/Prodigal)预测组装结果中的ORF,将核酸长度≥100 bp的基因翻译为氨基酸序列。将预测得的序列用CD-HIT软件(http://www.bioinformatics.org/cd-hit/)进行聚类,取序列相似性≥95%和序列覆盖度为90%的基因构建非冗余基因集。使用DIAMOND软件(http://ab.inf.uni-tuebingen.de/software/diamond/)将非冗余基因集与NR数据库进行比对获得物种注释(比对类型:BLASTP)。使用SOA Paligner软件与非冗余基因集进行比对,并统计基因在对应样品中的丰度信息,并在不同分类学水平(域、界、门、纲、目、科、属、种)上统计物种丰度,从而构建相应分类学水平上的丰度谱。将获得的基因集使用FMAP软件与KEGG数据库(Kyoto Encyclopedia of Genes and Genomes,http://www.genome.jp/kegg/)进行比对获得KEGG注释。基于NR和KEGG注释结果,对不同样品中的物种和功能基因进行可视化分析。
1.4 数据分析
采用R软件进行统计学分析及作图。
2 结果与分析
2.1 不同发酵蔬菜中的微生物群落组成分析
对4个样品的宏基因组测序共获得168.9 Gb原始序列,通过序列组装、基因预测,构建了1 357 213个非冗余基因集。对得到的非冗余基因序列与NR数据库进行比对从而获得物种注释,以确定不同发酵蔬菜中占主导地位的微生物类群。从4种泡菜样品中共鉴定出微生物121门、245纲、526目、977科、2 671个属、11 980种。细菌与真菌门、属和种水平群落组成如图1所示。

a-细菌门水平;b-细菌属水平;c-细菌种水平;d-真菌门水平;e-真菌属水平;f-真菌种水平
图1 工业发酵蔬菜中微生物群落组成结构
Fig.1 The microbial community composition structure in industrially fermented vegetables
细菌群落结构中,发酵豇豆中以厚壁菌门(Firmicutes)为优势菌门,发酵萝卜、芥菜和榨菜中均以Firmicutes和变形菌门(Proteobacteria)为优势菌门(图1-a),这与YU等[12]研究的发酵芥菜中优势细菌门类似。属水平上,乳杆菌属(Lactobacillus)是所有样品的主要优势菌属(图1-b),其中老面乳杆菌(Lactobacillus zymae)和酸面促生乳杆菌(Lactobacillus acidifarinae)在发酵萝卜和榨菜中的相对丰度最高,费斯莫尔德乳杆菌(Lactobacillus versmoldensis)在发酵豇豆中的相对丰度最高(图1-c)。Lactobacillus从属于Proteobacteria,近年来的研究表明,Lactobacillus是泡菜中的优势菌属,与亚硝酸盐含量呈显著负相关,在亚硝酸盐降解过程中起到直接或间接作用[2,13]。XIA等[14]采用短乳杆菌AR123单独发酵或与商业发酵剂混合发酵泡菜均能降低亚硝酸盐含量。XIANG等[15]采用食窦魏斯氏菌和植物乳杆菌共发酵能降低四川泡菜中亚硝酸盐的浓度。乳杆菌既可以通过产细菌素抑制硝酸盐还原菌生长,也可以通过产生亚硝酸盐还原酶以及产酸加快亚硝酸盐的降解,从而抑制亚硝酸盐在发酵蔬菜中的形成[16]。除Lactobacillus外,盐单胞菌属(Halomonas)、枝芽孢杆菌属(Virgibacillus)在发酵芥菜中的相对丰度也较高。有研究表明,大多数盐单胞菌和枝芽孢杆菌可通过产生亚硝酸盐还原酶在亚硝酸盐降解过程中发挥作用[17-18]。
真菌在发酵食品中发挥着重要作用,目前在蔬菜发酵过程中对真菌群落的研究较少。如图1-d所示,子囊菌门(Ascomycota)是4种工业泡菜中的优势菌门。不同发酵蔬菜的真菌属相对丰度不同(图1-e、图1-f),发酵豇豆中的德巴利酵母属(Debaryomyces)、苏吉雅玛酵母(Sugiyamaella)、毕赤酵母属(Pichia)和舍弗氏酵母属(Scheffersomyces)的相对丰度较高;发酵萝卜中以Pichia和米勒酵母属(Millerozyma)的相对丰度最高;发酵芥菜中以Debaryomyces和Pichia的相对丰度最高;榨菜中以Debaryomyces和Millerozyma的相对丰度最高。不同发酵蔬菜的细菌和真菌群落组成及丰度均存在一定差异,这可能是由原料的不同造成的[19]。这些微生物的代谢特征以及功能特征尚未在蔬菜发酵体系中得到广泛研究,因此,需要进一步确定发酵系统中功能基因与微生物群落演替之间的联系。
2.2 KEGG功能注释
将测序得到的基因组序列与KEGG数据库进行比对,以确定基因的功能和途径。得到的KEGG通路包括6大类,分别是代谢通路、环境信息处理、遗传信息处理、细胞进程、人类疾病和生命系统,代谢是表达基因最多的通路(图2-a),其中以氨基酸的生物合成(ko01230)、碳代谢(ko01200)、嘌呤代谢(ko00230)、嘧啶代谢(ko00240)的丰度最高。亚硝酸盐是氮代谢的关键中间体,氮代谢(ko00910)功能基因的相对丰度在发酵豇豆、发酵萝卜、发酵青菜和榨菜中分别达到0.8%、0.9%、1.2%、和0.7%(图2-b)。氮代谢过程主要包括异化硝酸盐还原作用、同化硝酸盐还原作用、反硝化作用、硝化作用、固氮和厌氧氨氧化作用6种途径[20]。除固氮外,异化硝酸盐还原作用、同化硝酸盐还原作用、反硝化作用、硝化作用和厌氧氨氧化作用途径中均存在亚硝酸盐转化过程,通过对氮代谢功能基因进行分析可以为亚硝酸盐形成及降解机制探究提供参考。
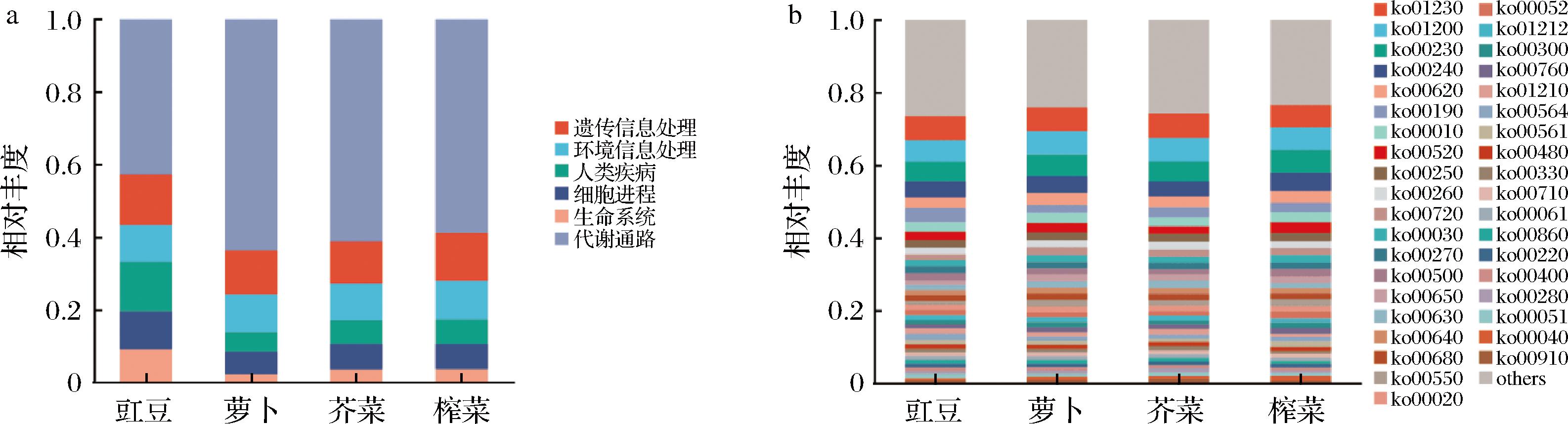
a-一级通路;b-代谢通路
图2 工业发酵蔬菜中KEGG一级通路和代谢通路基因相对丰度统计
Fig.2 Statistics on the relative abundance of genes involved in the KEGG level 1 and metabolism in industrially fermented vegetables
2.3 工业发酵蔬菜中亚硝酸盐形成及降解途径分析
根据宏基因测序所得的氮代谢功能基因丰度,构建了工业发酵蔬菜中的亚硝酸盐代谢反应通路图(图3)。本研究关于工业发酵蔬菜中的亚硝酸盐主要来自硝酸盐和硝基烷烃两种含氮物质的转化,这两种物质可能源自蔬菜原料从土壤中吸收的氮化肥和硝基烷烃土壤熏蒸剂残留化合物[21],发酵过程中分别在硝酸盐还原酶(EC 1.7.7.2)或铁氧还蛋白亚硝酸还原酶(EC 1.7.5.1)、硝基烷烃被硝基单加氧酶(EC 1.13.12.16)的作用下转化成亚硝酸盐。亚硝酸盐形成及降解途径包括反硝化作用、异化硝酸盐还原作用和同化硝酸盐还原作用3种途径。反硝化作用是通过亚硝酸盐还原酶(EC 1.7.2.1)、一氧化氮还原酶[细胞色素c](EC 1.7.2.5)、一氧化氮还原酶[NAD(P)+](EC 1.7.1.14)和一氧化二氮还原酶(EC 1.7.2.4)将![]() 逐步还原为N的过程。同化硝酸盐还原作用和异化硝酸盐还原作用均可以将
逐步还原为N的过程。同化硝酸盐还原作用和异化硝酸盐还原作用均可以将![]() 还原为NH3,两个途径分别由不同的酶主导,亚硝酸盐还原酶[NADH](EC 1.7.1.15)和亚硝酸盐还原酶[细胞色素](EC 1.7.2.2)为异化硝酸盐还原作用途径中的关键酶,铁氧还蛋白亚硝酸盐还原酶(EC 1.7.7.1)和亚硝酸盐还原酶[NAD(P)H](EC 1.7.1.4)为同化硝酸盐还原作用途径中的关键酶。与SONG等[22]的研究结果相似,酸菜发酵过程中亚硝酸盐代谢包括异化硝酸盐还原作用和反硝化作用,其中硝酸盐还原酶(nar)和亚硝酸盐还原酶[NADH](nirB)对硝酸盐还原和亚硝酸盐还原的贡献最大。
还原为NH3,两个途径分别由不同的酶主导,亚硝酸盐还原酶[NADH](EC 1.7.1.15)和亚硝酸盐还原酶[细胞色素](EC 1.7.2.2)为异化硝酸盐还原作用途径中的关键酶,铁氧还蛋白亚硝酸盐还原酶(EC 1.7.7.1)和亚硝酸盐还原酶[NAD(P)H](EC 1.7.1.4)为同化硝酸盐还原作用途径中的关键酶。与SONG等[22]的研究结果相似,酸菜发酵过程中亚硝酸盐代谢包括异化硝酸盐还原作用和反硝化作用,其中硝酸盐还原酶(nar)和亚硝酸盐还原酶[NADH](nirB)对硝酸盐还原和亚硝酸盐还原的贡献最大。

图3 亚硝酸盐形成及降解途径
Fig.3 The formation and degradation pathways of nitrite
注:红色方框中的数字表示酶的EC编号(EC 1.7.5.1:硝酸盐还原酶;EC 1.7.7.2:铁氧还蛋白硝酸盐还原酶;EC 1.13.12.16:硝基单加氧酶;EC 1.7.1.15:亚硝酸盐还原酶[NADH];EC 1.7.2.2:亚硝酸盐还原酶[细胞色素];EC 1.7.7.1:铁氧还蛋白亚硝酸盐还原酶;EC 1.7.1.4:亚硝酸盐还原酶[NAD(P)H];EC 1.7.2.1:亚硝酸盐还原酶;EC 1.7.2.5:一氧化氮还原酶[细胞色素c];EC 1.7.1.14:一氧化氮还原酶[NAD(P)+];EC 1.7.2.4:一氧化二氮还原酶),箭头表示反应方向。该图对应于KEGG数据库的ko00910。
对4种工业发酵蔬菜中亚硝酸盐代谢功能基因丰度进行统计,结果如图4所示。硝酸盐还原酶和硝基单加氧酶在4种发酵蔬菜中的基因数均较高,说明4种发酵蔬菜中的亚硝酸盐形成均与硝酸盐和硝基烷烃有关。反硝化作用的功能基因亚硝酸盐还原酶、一氧化氮还原酶[细胞色素c]、一氧化二氮还原酶和异化硝酸盐还原作用的功能基因亚硝酸盐还原酶[NADH]在4种发酵蔬菜中均呈现较高的基因数(图4-a),同时结合图4-b可以看出,4种发酵蔬菜中亚硝酸盐降解都以反硝化作用和异化硝酸盐还原作用为主,而同化硝酸盐还原作用相对较少。现有研究发现,假单胞菌属(Pseudomonas)[23]、不动杆菌属(Acinetobacter)[24]、芽孢杆菌属(Bacillus)[25]、肠杆菌属(Enterobacter)[26]、海杆菌属(Marinobacter)[27]等均具有参与反硝化、同化和异化硝酸盐还原作用代谢亚硝酸盐的能力,这些微生物存在于蔬菜发酵体系中,在发酵过程中将亚硝酸盐逐步降解,从而使亚硝酸盐含量逐渐下降。
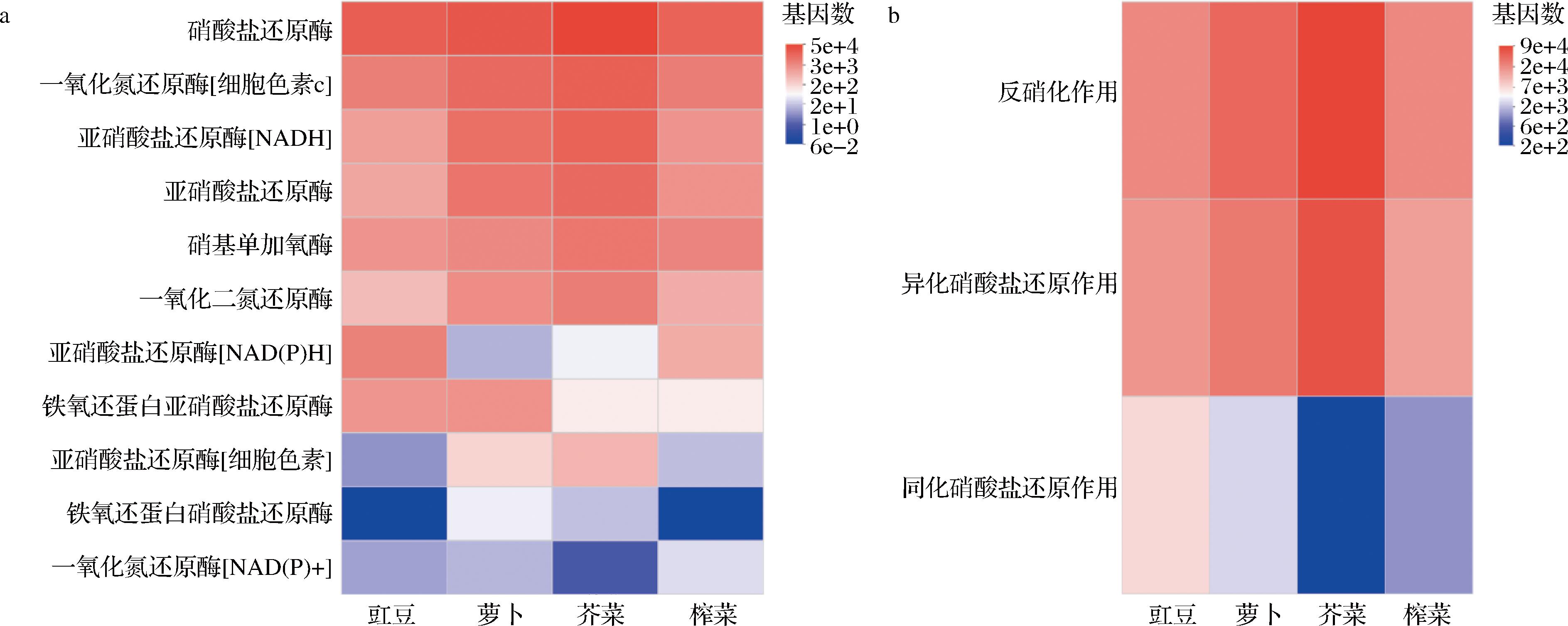
a-代谢关键酶;b-代谢途径
图4 工业发酵蔬菜中亚硝酸盐代谢关键酶和途径基因数
Fig.4 The number of genes for the enzymes and pathways involved in the metabolism of nitrite in industrially fermented vegetables
2.4 亚硝酸盐代谢相关微生物及其与功能基因的贡献度
微生物的反硝化、异化及同化硝酸盐还原作用是发酵蔬菜中亚硝酸盐生成和降解途径中的重要步骤。为确定本研究中与亚硝酸盐代谢有关的微生物,基于样本物种与功能的对应关系,将氮代谢功能基因与微生物进行关联分析,并统计该物种对特定功能的贡献度,结果如图5所示。通过细菌与各发酵蔬菜样品的功能基因的关联分析可以看出(图5-a),各工业发酵蔬菜样品中与亚硝酸盐代谢有关的微生物组存在共性和特性。发酵豇豆中与亚硝酸盐代谢有关的微生物主要包括L. versmoldensis和解淀粉嗜盐碱球菌(Natronococcus amylolyticus);发酵萝卜、芥菜中的相关微生物主要包括吉氏盐单胞菌(Halomonas jeotgali)、橙色盐单胞菌(Halomonas lutea)和反硝化盐单胞菌(Halomonas halodenitrificans)等;榨菜中的相关微生物主要包括H. jeotgali、L. versmoldensis、H. lutea、盐反硝化枝芽孢杆菌(Virgibacillus halodenitrificans)和N. amylolyticus等。其中L. versmoldensis、H. jeotgali、H. lutea和H. halodenitrificans对反硝化作用和异化硝酸盐还原作用的贡献度较高,V. halodenitrificans对异化硝酸盐还原作用的贡献度较高,N. amylolyticus对同化硝酸盐还原作用的贡献度较高。有研究表明,H. jeotgali、N. amylolyticus可以通过产硝酸盐还原酶将硝酸盐还原为亚硝酸盐[28-29]。H. halodenitrificans、V. halodenitrificans在硝酸盐还原酶、亚硝酸盐还原酶、一氧化氮还原酶基因编码的酶催化下实现对亚硝酸盐的转化[30-31],进一步说明这些细菌可能通过自身酶催化在发酵蔬菜亚硝酸盐形成及降解过程中的反应发挥重要作用。
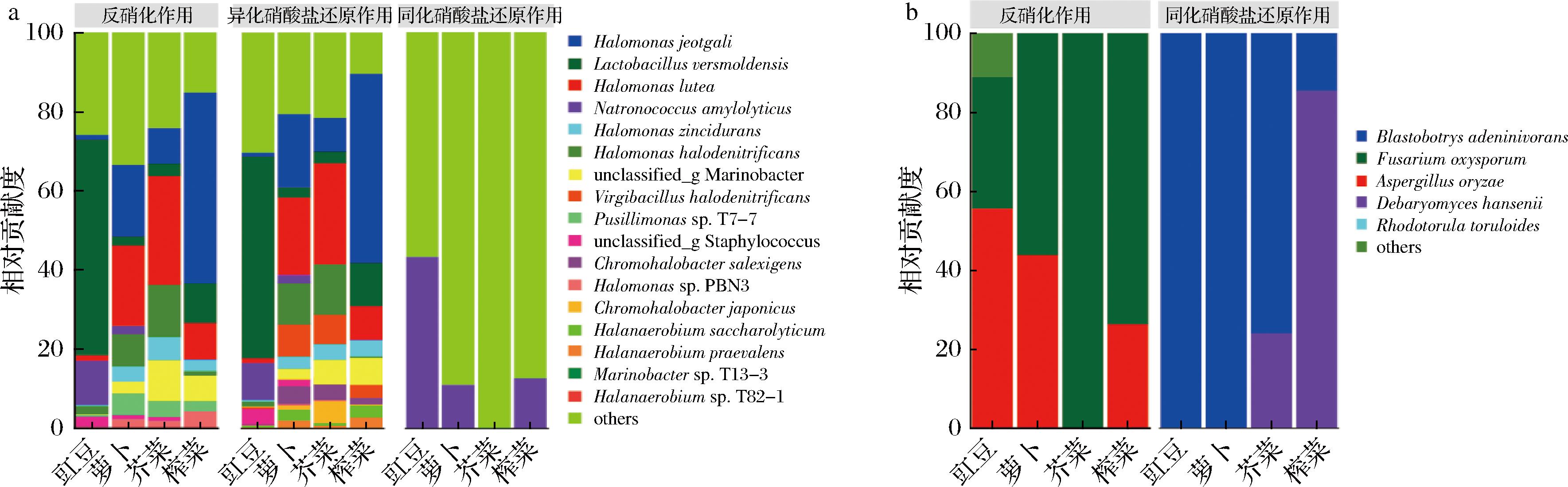
a-细菌;b-真菌
图5 细菌和真菌对工业发酵蔬菜亚硝酸盐代谢功能基因的贡献度
Fig.5 The contribution of bacteria and fungi to nitrite metabolism functional genes in industrially fermented vegetables
与发酵蔬菜亚硝酸代谢有关的真菌种类相对较少(图5-b)。发酵豇豆与发酵萝卜中与亚硝酸盐代谢有关的真菌包括尖孢镰刀菌(Fusarium oxysporum)、米曲霉(Aspergillus oryzae)、食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans);发酵芥菜中与亚硝酸盐代谢有关的真菌包括尖孢镰刀菌(Fusarium oxysporum)、食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans)、汉逊德巴利酵母(Debaryomyces_hansenii);榨菜中与亚硝酸盐代谢有关的微生物包括尖孢镰刀菌(Fusarium oxysporum)、米曲霉(Aspergillus oryzae)、食腺嘌呤芽生葡萄孢酵母(Blastobotrys adeninivorans)、汉逊德巴利酵母(Debaryomyces_hansenii)。以往的研究表明,汉逊德巴利酵母可通过产生亚硝酸盐还原酶(nir)参与同化硝酸盐还原作用从而实现对亚硝酸盐的降解[32],米曲霉与尖孢镰刀菌基因组中编码的亚硝酸盐还原酶[NAD(P)H](nirk)促使该菌参与反硝化作用[33-34],食腺嘌呤芽生葡萄孢酵母中通过硝酸盐还原酶和亚硝酸盐还原酶联合作用主导了同化硝酸盐还原作用的进行[35],也进一步说明上述真菌在发酵蔬菜亚硝酸盐转化过程中发挥了重要作用,促进了亚硝酸盐的形成和降解。
3 结论
本研究利用宏基因组学技术分析了4种不同发酵蔬菜的微生物群落组成,挖掘了基于氮代谢的亚硝酸盐代谢通路、关键功能基因及关联微生物。通过测序获得168.9 Gb原始序列,鉴定出微生物121门、245纲、526目、977科、2671属、11980种。不同原料发酵蔬菜的细菌和真菌群落组成及丰度均存在一定差异,细菌组成上,发酵豇豆、萝卜和榨菜中均以Lactobacillus为优势细菌属;发酵芥菜中以Lactobacillus、Halomonas、Virgibacillus为优势细菌属。真菌组成上,发酵豇豆中以Debaryomyces、Sugiyamaella、Pichia和Scheffersomyces为优势菌属;发酵萝卜中以Pichia和Millerozyma为优势菌属;发酵芥菜中以Debaryomyces和Pichia为优势菌属;榨菜中以Debaryomyces和Millerozyma为优势菌属。KEGG注释结果显示,与亚硝酸盐代谢密切相关的氮代谢功能基因在发酵豇豆、发酵萝卜、发酵芥菜和榨菜中的相对丰度分别达到0.8%、0.9%、1.2%、和0.7%。根据氮代谢功能基因丰度信息构建了发酵蔬菜亚硝酸盐代谢通路,发现亚硝酸盐代谢包括反硝化作用、异化硝酸盐还原作用和同化硝酸盐还原作用3种途径,其中对反硝化作用和异化硝酸盐还原作用贡献较大的微生物为L. versmoldensis、H. jeotgali、H. lutea、H. halodenitrificans、V. halodenitrificans、A. oryzae、F. oxysporum;对同化硝酸盐还原作用贡献较大的微生物为N amylolyticus、D. hansenii、B. adeninivoran。总体而言,蔬菜发酵体系中的微生物可通过酶催化反应在亚硝酸盐形成和降解过程发挥重要作用。本研究在分析发酵蔬菜菌群结构组成的基础上,进一步明确了发酵蔬菜中亚硝酸盐的代谢通路和关键功能基因以及物种与功能基因的关系,后续可进一步对关键微生物进行筛选应用,进一步验证功能基因的表达情况,以期为通过调控微生物(如分离关键功能微生物)从而精准调控发酵蔬菜生产过程亚硝酸盐形成或降解提供研究基础。
[1] 杨姗, 王卫, 赵楠, 等.发酵蔬菜色泽形成机制及影响因素研究进展[J].食品科学, 2022, 43(23):269-276.YANG S, WANG W, ZHAO N, et al.Recent advances in understanding mechanism and influential factors of color formation in fermented vegetables[J].Food Science, 2022, 43(23):269-276.
[2] WANG Z X, SHAO Y Y.Effects of microbial diversity on nitrite concentration in Pao cai, a naturally fermented cabbage product from China[J].Food Microbiology, 2018, 72:185-192.
[3] HUANG Y Y, LIANG M H, ZHAO S, et al.Isolation, expression, and biochemical characterization:Nitrite reductase from Bacillus cereus LJ01[J].RSC Advances, 2020, 10(62):37871-37882.
[4] HUANG T T, WU Z Y, ZHANG W X.Effects of garlic addition on bacterial communities and the conversions of nitrate and nitrite in a simulated pickle fermentation system[J].Food Control, 2020, 113:107215.
[5] HUANG Y Y, JIA X Z, YU J J, et al.Effect of different lactic acid bacteria on nitrite degradation, volatile profiles, and sensory quality in Chinese traditional Paocai[J].LWT, 2021, 147:111597.
[6] YANG X Z, HU W Z, JIANG A L, et al.Effect of salt concentration on quality of Chinese northeast sauerkraut fermented by Leuconostoc mesenteroides and Lactobacillus plantarum[J].Food Bioscience, 2019, 30:100421.
[7] 赵天涛, 陈沛沛, 张晟, 等.异养硝化-好氧反硝化菌氮代谢机理的研究进展[J].重庆理工大学学报(自然科学), 2022(1):194-203.ZHAO T T, CHEN P P, ZHANG S, et al.Research progress on nitrogen metabolism mechanism of heterotrophic nitrification aerobic denitrification bacteria[J].Journal of Chongqing University of Technology (Natural Science), 2022(1):194-203.
[8] 徐柯, 成林林, 袁美, 等.泡豇豆发酵过程中有机酸变化及对亚硝酸盐降解的影响[J].食品与发酵工业, 2019, 45(17):60-65;72.XU K, CHENG L L, YUAN M, et al.Changes in organic acids and effects on nitrite degradation during pickled cowpea (Vigna sinensis) fermentation[J].Food and Fermentation Industries, 2019, 45(17):60-65;72.
[9] 魏雯丽, 宫尾茂雄, 吴正云, 等.基于宏转录组学技术解析工业豇豆泡菜发酵过程中活性微生物群落结构变化[J].食品与发酵工业, 2020, 46(10):60-65.WEI W L, WEI W L, WU Z Y, et al.Analysis of active microbial community structure changes in industrial cowpea pickle fermentation based on meta-transcriptomics technology[J].Food and Fermentation Industries, 2020, 46(10):60-65.
[10] LIU D Q, ZHANG C C, ZHANG J M, et al.Metagenomics reveals the formation mechanism of flavor metabolites during the spontaneous fermentation of potherb mustard (Brassica juncea var.multiceps)[J].Food Research International, 2021, 148:110622.
[11] YU Y Y, LI L, XU Y J, et al.Metagenomics reveals the microbial community responsible for producing biogenic amines during mustard[Brassica juncea (L.)] fermentation[J].Frontiers in Microbiology, 2022, 13:824644.
[12] YU Y Y, LI L, XU Y J, et al.Evaluation of the relationship among biogenic amines, nitrite and microbial diversity in fermented mustard[J].Molecules, 2021, 26(20):6173.
[13] ZHOU Q, ZANG S Z, ZHAO Z N, et al.Dynamic changes of bacterial communities and nitrite character during northeastern Chinese sauerkraut fermentation[J].Food Science and Biotechnology, 2017, 27(1):79-85.
[14] XIA Y J, LIU X F, WANG G Q, et al.Characterization and selection of Lactobacillus brevis starter for nitrite degradation of Chinese pickle[J].Food Control, 2017, 78:126-131.
[15] XIANG W L, ZHANG N D, LU Y, et al.Effect of Weissella cibaria co-inoculation on the quality of Sichuan Pickle fermented by Lactobacillus plantarum[J].LWT, 2020, 121:108975.
[16] 陈功, 唐垚, 赵平, 等.泡菜榨菜亚硝酸盐含量及其N2-NH4+转化机理的研究[J].食品与发酵科技, 2021, 57(2):1-13.CHEN G, TANG Y, ZHAO P, et al.Study on nitrite content and its N2-NH4+ Transforming theory in Paocai and Zhacai[J].Food and Fermentation Sciences &Technology, 2021, 57(2):1-13.
[17] LI W, LI H, LIU Y D, et al.Salinity-aided selection of progressive onset denitrifiers as a means of providing nitrite for anammox[J].Environmental Science &Technology, 2018, 52(18):10665-10672.
[18] HOSSEINI M, AL-RUBAYE M T S, FAKHARI J, et al.Isolation and characterization of denitrifying halophilic bacteria from Bahr Al-Milh Salt Lake, Karbala, Iraq[J].Journal of Applied Biology &Biotechnology, 2018, 6(4):32-36.
[19] 王奇, 王传明, 周雨, 等.泡菜中微生物菌群的研究进展[J].中国调味品, 2021, 46(9):197-200.WANG Q, WANG C M, ZHOU Y, et al.Research progress of microbial community in pickles[J].China Condiment, 2021, 46(9):197-200.
[20] LABS, KANEHISA.Kyoto Encyclopedia of Genes and Genomes (KEGG):ko90010[EB/OL].(2023-10-01)[2023-10-10].https://www.genome.jp/pathway/ko00910.
[21] 陈翠翠, 梁艳辉, 祝杰.土壤熏蒸剂专利技术分析[J].现代农药, 2022, 21(4):26-30;36.CHEN C C, LIANG Y H, ZHU J.Analysis on patent technology of soil fumigation[J].Modern Agrochemicals, 2022, 21(4):26-30;36.
[22] SONG Q Z, ZHAO F K, WANG B B, et al.Metagenomic insights into Chinese northeast Suancai:Predominance and diversity of genes associated with nitrogen metabolism in traditional household Suancai fermentation[J].Food Research International, 2021, 139:109924.
[23] 彭永臻, 钱雯婷, 王琦, 等.基于宏基因组的城市污水处理厂生物脱氮污泥菌群结构分析[J].北京工业大学学报, 2019, 45(1):95-102.PENG Y Z, QIAN W T, WANG Q, et al.Unraveling microbial structure of activated sludge in a full-scale nitrogen removal plant using metagenomic sequencing[J].Journal of Beijing University of Technology, 2019, 45(1):95-102.
[24] 朱婉瑜, 侍浏洋, 赵维, 等.Zn(II)对好氧反硝化菌Acinetobacter sp.JR-142的代谢活性影响[J].微生物学报, 2022, 62(1):275-290.ZHU W Y, SHI L Y, ZHAO W, et al.Effects of Zn(Ⅱ) on metabolic activity of aerobic denitrifier Acinetobacter sp.JR-142[J].Acta Microbiologica Sinica, 2022, 62(1):275-290.
[25] 黄雪芹, 左勇, 张强, 等.芽菜中高效降解亚硝酸盐菌株的分离鉴定[J].中国调味品, 2020, 45(7):8-11.HUANG X Q, ZUO Y, ZHANG Q, et al.Isolation and identification of high-efficiency nitrite-degrading bacteria in sprouts[J].China Condiment, 2020, 45(7):8-11.
[26] 安江波, 刘明健, 刘伟, 等.青贮过程中亚硝酸盐转化途径及影响因素研究进展[J].草地学报, 2023, 31(4):943-951.AN J B, LIU M J, LIU W, et al.Research progress on nitrite transformation pathway and its influencing factors in silage[J].Acta Agrestia Sinica, 2023, 31(4):943-951.
[27] RUAN Y J, KUMAR AWASTHI M, CAI L, et al.Simultaneous aerobic denitrification and antibiotics degradation by strain Marinobacter hydrocarbonoclasticus RAD-2[J].Bioresource Technology, 2020, 313:123609.
[28] VALENZUELA-ENCINAS C, NERIA-GONZ LEZ I, ALC
LEZ I, ALC NTARA-HERN
NTARA-HERN NDEZ R J, et al.Phylogenetic analysis of the archaeal community in an alkaline-saline soil of the former Lake Texcoco (Mexico)[J].Extremophiles:Life Under Extreme Conditions, 2008, 12(2):247-254.
NDEZ R J, et al.Phylogenetic analysis of the archaeal community in an alkaline-saline soil of the former Lake Texcoco (Mexico)[J].Extremophiles:Life Under Extreme Conditions, 2008, 12(2):247-254.
[29] KIM M S, ROH S W, BAE J W.Halomonas jeotgali sp.nov., a new moderate halophilic bacterium isolated from a traditional fermented seafood[J].Journal of Microbiology, 2010, 48(3):404-410.
[30] SAKURAI N, ASADA A, MANO S, et al.Tandem and single genes of three membrane-bound nitrate transporters in the nar gene cluster of the moderately halophilic denitrifier, Halomonas halodenitrificans[J].DNA Sequence, 2006, 17(5):363-369.
[31] 高于涵, 贾亚婷, 吕岳骏, 等.微生物厌氧代谢耦合多环芳烃降解研究进展[J].环境科学与技术, 2023, 46(5):67-75.GAO Y H,JIA Y T,LYU Y J,et al.Research advances in biodegradation of PAHs coupled with microbial anaerobic metabolism[J].Environmental Science &Technology, 2023, 46(5):67-75.
[32] VIGLIOTTA G, DI GIACOMO M, CARATA E, et al.Nitrite metabolism in Debaryomyces hansenii TOB-Y7, a yeast strain involved in tobacco fermentation[J].Applied Microbiology and Biotechnology, 2007, 75(3):633-645.
[33] NAKANISHI Y, ZHOU S M, KIM S W, et al.A eukaryotic copper-containing nitrite reductase derived from a NirK homolog gene of Aspergillus oryzae[J].Bioscience, Biotechnology, and Biochemistry, 2010, 74(5):984-991.
[34] ROHE L, OPPERMANN T, WELL R, et al.Nitrite induced transcription of p450nor during denitrification by Fusarium oxysporum correlates with the production of N2 O with a high 15 N site preference[J].Soil Biology and Biochemistry, 2020, 151:108043.
[35] BÖER E, SCHRÖTER A, BODE R, et al.Characterization and expression analysis of a gene cluster for nitrate assimilation from the yeast Arxula adeninivorans[J].Yeast, 2009, 26(2):83-93.