乳酸菌(lactic acid bacteria,LAB)是一类革兰氏阳性、过氧化氢酶阴性的细菌,它们能够利用碳水化合物产生大量乳酸,被广泛用于各种发酵食品的生产[1]。LAB在食品发酵和应用过程中都会遇到各种环境胁迫,其中酸胁迫是最重要的生存挑战之一。筛选潜在益生菌的首要条件是检测其耐酸性。耐酸可用于保护益生菌在进食过程中及进入胃后免受胃酸的侵害,LAB更高的耐酸性可以使在低pH环境中更好的发挥其益生作用,所以对LAB酸胁迫作用机制的研究,成为乳酸菌研究的热点之一[2-3]。
目前,基因组学、转录组学、蛋白质组学和代谢组学等高通量技术的出现使人们在系统水平上能够很好地理解细菌的耐酸机制,其中代谢组学的优势在于基因和蛋白质表达的微小变化能够在代谢组学水平上被扩大显示出来[4-5]。代谢组学是通过检测细菌中存在的全部相对分子质量<1 000的代谢物并跟踪细菌生长过程与环境之间相互作用的总体结果,更准确地反映细胞的实际生理状态[6]。LC-MS是代谢组学研究中常用的一种分析技术,它具有高峰值分辨率、快速分析和高检测灵敏度等优点,适用于分离和分析各种复杂样品中的微量成分[7]。细胞代谢与胁迫应答密切相关,目前报道植物乳杆菌、路易斯乳杆菌、保加利亚乳杆菌等乳酸菌可以通过调节代谢响应酸胁迫[8]。由于乳酸菌酸胁迫后细胞的生长和代谢状态会改变,因此利用代谢组学更好地分析乳酸菌酸胁迫前后代谢物差异,有利于探究乳酸菌菌株的耐酸机制。
开菲尔慢生乳杆菌(Lentilactobacillus kefiri,L.kefiri)是传统发酵乳制品开菲尔中的优势乳酸菌菌种之一,2020年以前称为开菲尔乳杆菌(Lactobacillus kefiri),2020年乳杆菌属的分类地位变迁才改为现在的名称[9]。有些L.kefiri已被证明具有多种益生作用,主要包括调节肠道菌群、抑制食源性病原体和肠上皮细胞粘附等[10-12]。目前L.kefiri的耐酸特性和耐酸机制研究较少。因此,本研究以课题组前期在传统乳制品中分离出的一株L.kefiri IMAU10804为研究对象,首先评价L.kefiri IMAU10804的耐酸能力,明确L.kefiri IMAU10804在不同酸处理条件下对应的生理状态,以确定后续代谢组学酸胁迫处理的最佳条件,然后使用LC-MS检测菌株酸胁迫前后的代谢物差异。
1 材料与方法
1.1 材料
1.1.1 实验菌株
本实验采用的L.kefiri IMAU10804分离自内蒙古巴林右旗巴彦温都尔苏木传统发酵酸牛奶,L.kefiri IMAU10804由内蒙古农业大学乳酸菌菌种资源库提供。
1.1.2 材料与试剂
乙腈(色谱级)、甲酸(色谱级)、甲醇(色谱级),美国Fisher科技公司;氨水(色谱级),德国Sigma公司;MRS液体培养基、MRS固体培养基,英国Oxoid公司。
1.2 仪器与设备
本实验LC-MS分析的仪器平台为超高效液相色谱串联傅里叶变换质谱UHPLC-Q Exactive HF-X,赛默飞公司;HSS T3色谱柱,美国Waters公司;IQ 7000 Milli-Q纯水仪,Milli-pore公司;Wonbio-96c高通量组织破碎仪,上海万柏生物科技有限公司;JXDC-20氮气吹扫仪,上海净信实业发展有限公司;Bioscreen C全自动生长曲线仪,芬兰Oy Growth Curves Ab有限公司;DHP-9272恒温培养箱,上海一恒科技有限公司;SX-700高压蒸汽灭菌锅,日本Tomy Digital Biology公司;R5810高速冷冻离心机,德国Eppendorf公司;FE 28 pH计,上海梅特勒公司。
1.3 实验方法
1.3.1 L.kefiri IMAU10804的活化
将冻存于-80 ℃的L.kefiri IMAU10804取出,按4%(体积分数)接种量接种到5 mL MRS液体培养基中,在37 ℃恒温培养24 h,连续培养3代,第3代菌液用于后续的实验。
1.3.2 L.kefiri IMAU10804生长曲线的测定
使用全自动生长曲线仪测量L.kefiri IMAU10804菌体密度OD值。每隔1 h,测定波长600 nm处OD值,温度37 ℃,培养时间72 h,振荡时间10 s,然后静止5 s读取OD值,每个时间点进行3次平行测量。以培养时间为横坐标,OD600值为纵坐标,绘制L.kefiri IMAU10804生长曲线,通过观察曲线确定其生长对数期。
1.3.3 L.kefiri IMAU10804不同pH处理下的活菌数的测定
首先制备pH值为4.0、3.5、3.0、2.5的MRS液体培养基,用1 mol/L盐酸缓慢滴加到MRS液体培养基中,搅拌均匀后测定pH值,直至达到目标pH值,将调节好的MRS液体培养基装入试管中,进行121 ℃,15 min灭菌,灭菌后的培养基可以用于后续实验。
取5 mL 1.3.1节活化后的3代菌悬液,吸取0.5 mL菌液进行10倍梯度稀释,平板计数,作为对照组。剩余4.5 mL以4 000×g离心5 min,弃去上清液,将菌体重悬于等体积的pH 4.0、3.5、3.0、2.5的MRS液体培养基中,然后在37 ℃恒温条件下培养3 h,培养结束后,分别取不同pH条件下的菌液0.5 mL,用4.5 mL的灭菌生理盐水(8.5 g/L NaCl溶液)10倍梯度连续稀释,选择10-6、10-7和10-8梯度采用平板倾注法进行平板计数,每个梯度3个平行。平板倒置于培养箱中,37 ℃恒温培养72 h,待菌落长出后进行计数,利用计数结果确定其耐酸情况。
1.3.4 L.kefiri IMAU10804不同处理时间下的活菌数测定
将1.3.1节活化后的3代菌悬液取出5 mL,以4 000×g离心5 min,弃去上清液,将菌体重悬于等体积的pH 3.0的MRS液体培养基中,然后在37 ℃恒温条件下培养,分别在0、15、30、60、90、120 min时取0.5 mL 菌液进行梯度稀释,利用平板倾注法进行计数,每个梯度设3个平行,并计算存活率,如公式(1)所示:
菌株存活率![]()
(1)
1.3.5 LC-MS样品制备和分析
1.3.5.1 样品的制备
参照包秋华等[13]菌液样本前处理方法,即:移取胁迫前后样品各100 μL至灭菌的离心管中,与400 μL含有内标的提取液[V(乙腈)∶V(甲醇)=1∶1]充分混合30 s,在5 ℃,40 kHz的条件下超声提取30 min,将样品放在-20 ℃静置30 min后,在4 ℃条件下,13 000×g离心15 min,吸取上清液经0.22 μm微孔膜过滤后进行上机检测。质量控制(quality control,QC)样品由所有样品等体积制得。首先对QC样品和空白进行色谱分析,以保证色谱系统的稳定性和重复性。样品(正离子和负离子模式)随机注入系统,每6个样品注入后重复QC样品,以控制仪器的稳定性。
1.3.5.2 色谱条件
采用Waters HSS T3(100 mm×2.1 mm i.d., 1.8 m)色谱柱进行色谱分析,正离子流动相为95%(体积分数,下同)超纯水+5%乙腈(含0.1%甲酸),负离子流动相为47.5%乙腈+47.5%异丙醇+5%超纯水(含0.1%甲酸),柱温为40 ℃,进样量为2 μL。梯度洗脱条件见表1。
表1 流动相梯度洗脱条件
Table 1 Gradient elution conditions of mobile phase
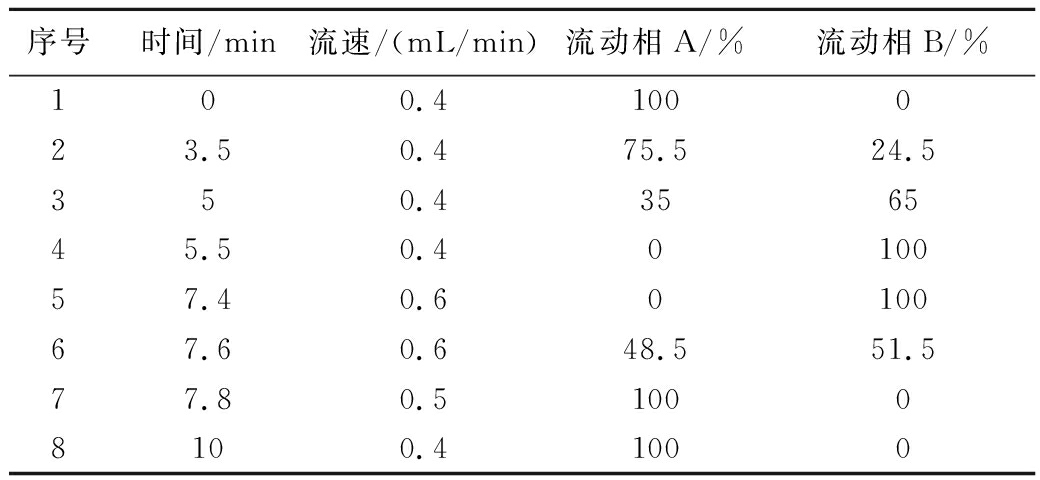
序号时间/min流速/(mL/min)流动相A/%流动相B/%100.4100023.50.475.524.5350.4356545.50.4010057.40.6010067.60.648.551.577.80.510008100.41000
1.3.5.3 质谱条件
质谱参数是根据王道滇等[14]先前研究选择的。使用Q Exactive HF-X系统在正离子和负离子(ESI+/ESI-)扫描模式下进行代谢物检测,质核比扫描范围为70~1 050 m/z,具体参数见表2。
表2 质谱检测参数
Table 2 Mass spectrometry detection parameters

条件正离子模式参数负离子模式参数鞘气流速50 psi50 psi辅助气流速13 psi13 psi辅助气加热温度425 ℃425 ℃喷雾电压3 500 V-3 500 V离子传输管温度325 ℃325 ℃
1.4 数据处理和分析
将LC-MS获得的原始代谢数据导入Proggenesis QI 2.2软件进行处理,将处理后的数据采用正交偏最小二乘法-判别分析(orthogonal partial least-squares discrimination analysis,OPLS-DA)来进行建模分析,并根据2个样本组之间P<0.05,变量投影重要性(variable importance in the projection,VIP)≥1和2组间变化倍数(fold change,FC)≥1.5来确定差异代谢物。图表利用SIMCA 14.1软件同时结合Metaboanalyst 5.0进行制作。根据代谢物数量生成京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)重要通路统计图。
2 结果与分析
2.1 L.kefiri IMAU10804生长曲线的绘制
L.kefiri IMAU10804菌株生长曲线见图1所示。
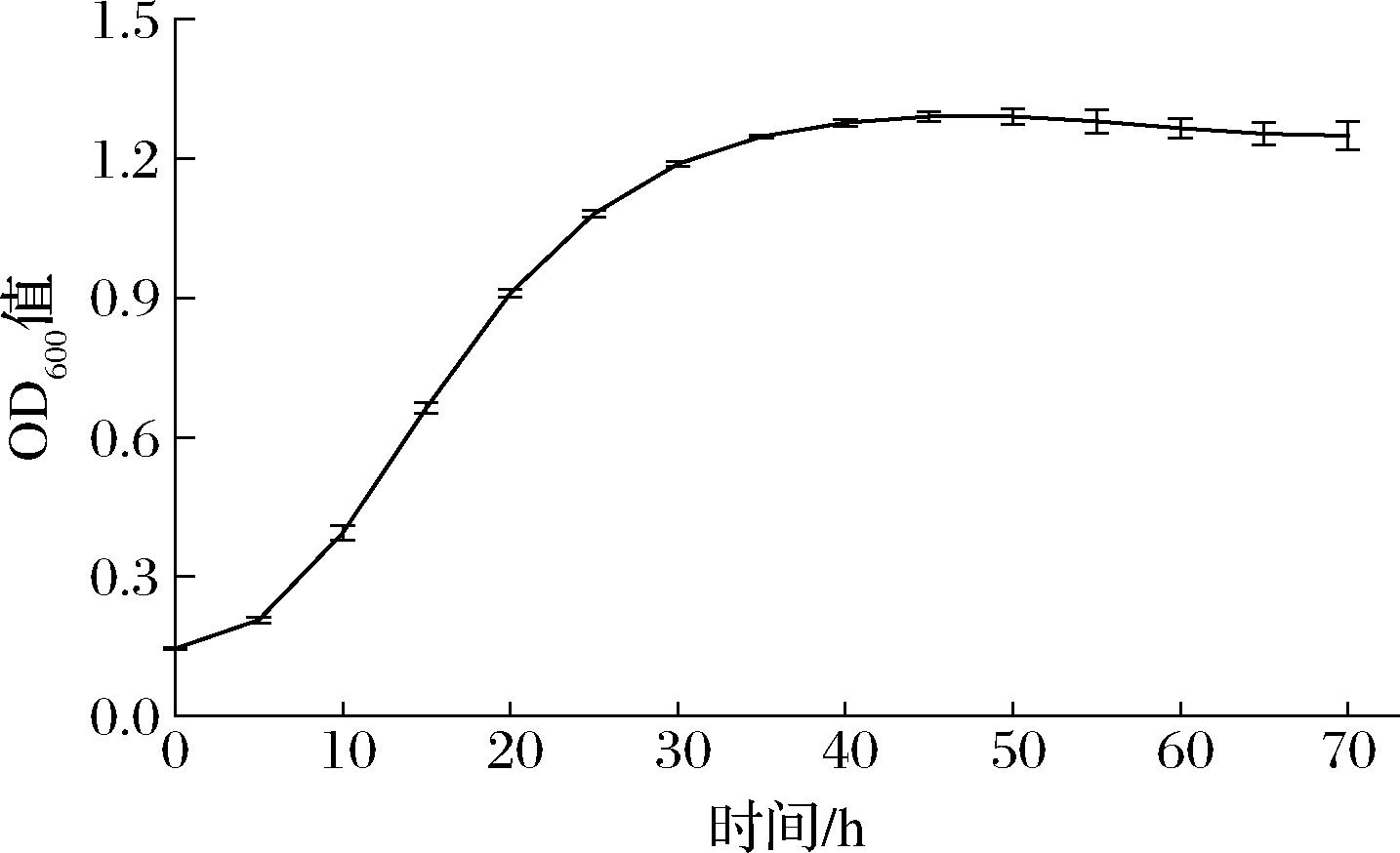
图1 L.kefiri IMAU10804在MRS液体培养基的生长曲线
Fig.1 The growth curve of L.kefiri IMAU10804 in MRS liquid medium
由图1可知L.kefiri IMAU10804的生长曲线呈典型的S型,大致可分为3个阶段,即:延迟期、对数生长期和稳定期。0~6 h时菌体浓度增长缓慢,6~40 h增殖迅速,40 h后浓度趋于平稳,进入稳定生长期。根据已有报道称对数生长期的细菌对于外界环境的变化感知明显[15]。因此,后续实验选取对数生长中期24 h的L.kefiri IMAU10804菌液作为研究对象。
2.2 L.kefiri IMAU10804不同pH处理下的活菌数的测定
为了解L.kefiri IMAU10804的耐酸能力,测定了菌株在不同pH值环境中处理3 h后的活菌数,结果见图2所示。
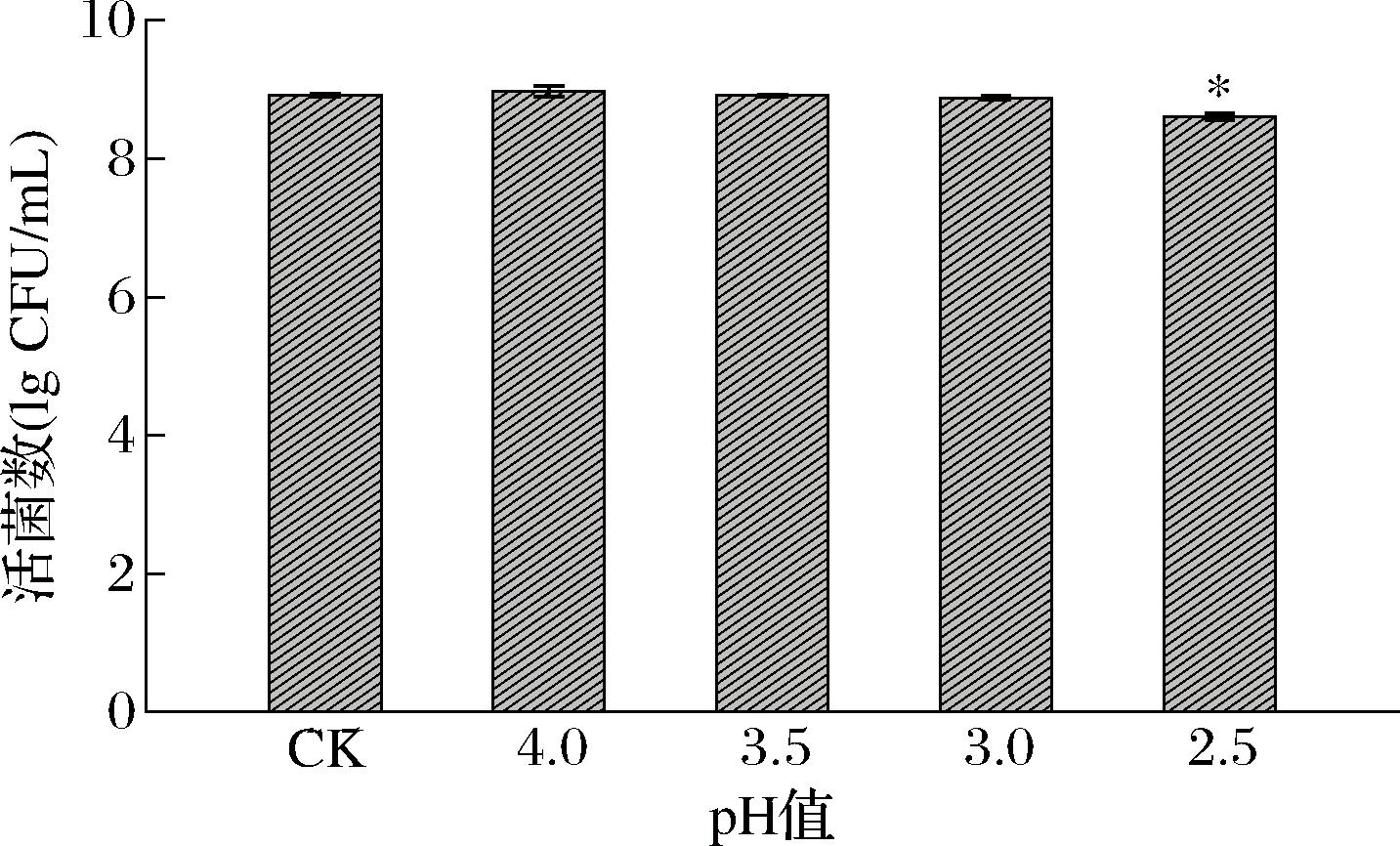
图2 L.kefiri IMAU10804在不同pH环境下处理3 h后的活菌数
Fig.2 The viable count of L.kefiri IMAU10804 after exposed to different pH for 3 hours
注:*代表处理组与对照组之间有显著差异(P<0.05)。
由图2可知,对照组(CK,pH 6.5)即酸胁迫处理前L.kefiri IMAU10804的活菌数为(8.92±0.05) lg CFU/mL;L.kefiri IMAU10804在pH 4.0的环境中处理3 h后,发现活菌数比酸处理前略有升高,说明L.kefiri IMAU10804在pH 4.0的条件下仍能保持生长;pH 3.5和pH 3.0的环境中处理3 h后,活菌数下降,未出现显著性变化(P>0.05);pH 2.5的环境中处理3 h后,活菌数显著低于其他组别,活菌数为(8.61±0.05) lg CFU/mL。上述说明L.kefiri IMAU10804具有一定的耐酸性。
2.3 L.kefiri IMAU10804不同处理时间下的活菌数测定
为了明确L.kefiri IMAU10804在不同酸处理条件下对应的生理状态,为后续代谢组学酸处理条件提供参考,因此测定了pH 3.0条件下不同处理时间对L.kefiri IMAU10804存活率的影响,结果如图3所示。

图3 L.kefiri IMAU10804在pH 3.0环境下处理不同时间的存活率
Fig.3 The survival rate of L.kefiri IMAU10804 exposed to pH 3.0 for different time
注:*代表处理组与对照组之间有显著差异(P<0.05),**代表处理组与对照组之间有极显著差异(P<0.01)。
由图3可知,随着胁迫时间的增长,L.kefiri IMAU10804存活率呈下降的趋势,将L.kefiri IMAU10804在pH 3.0条件下处理0 min作为对照组,此时的存活率为100%。在pH 3.0条件下处理15、30、60 min后L.kefiri IMAU10804的存活率分别为93.8%、93.4%和93%,与对照组相比,没有出现显著的下降(P>0.05),当处理90、120 min后,存活率分别为88%和77.4%,均出现显著的下降(P<0.05)。由上述结果可知,L.kefiri IMAU10804在pH 3.0的MRS液体培养基中胁迫60 min条件下,菌体得到充分的刺激而菌体的存活率无显著下降,菌体活力无显著变化。因此,pH 3.0处理60 min可以作为后续利用代谢组学探究菌株酸胁迫前后代谢组学差异的条件。
2.4 L.kefiri IMAU10804酸胁迫前后代谢物研究
2.4.1 OPLS-DA分析
使用LC-MS分析对L.kefiri IMAU10804酸胁迫前后细胞进行代谢组学分析。利用OPLS-DA来比较来自2种生理状态细胞的代谢组学数据。在OPLS-DA中,R2Y和Q2 2个指标分别表示模型解释率和预测能力[16]。OPLS-DA模型在ESI+模式和ESI-模式下R2Y和Q2值均接近1(图4-A ESI+模式:R2Y=0.998,Q2=0.994;图4-B ESI-模式:R2Y=0.996,Q2=0.995),表明2个模型的解释率和预测能力具有可行性。
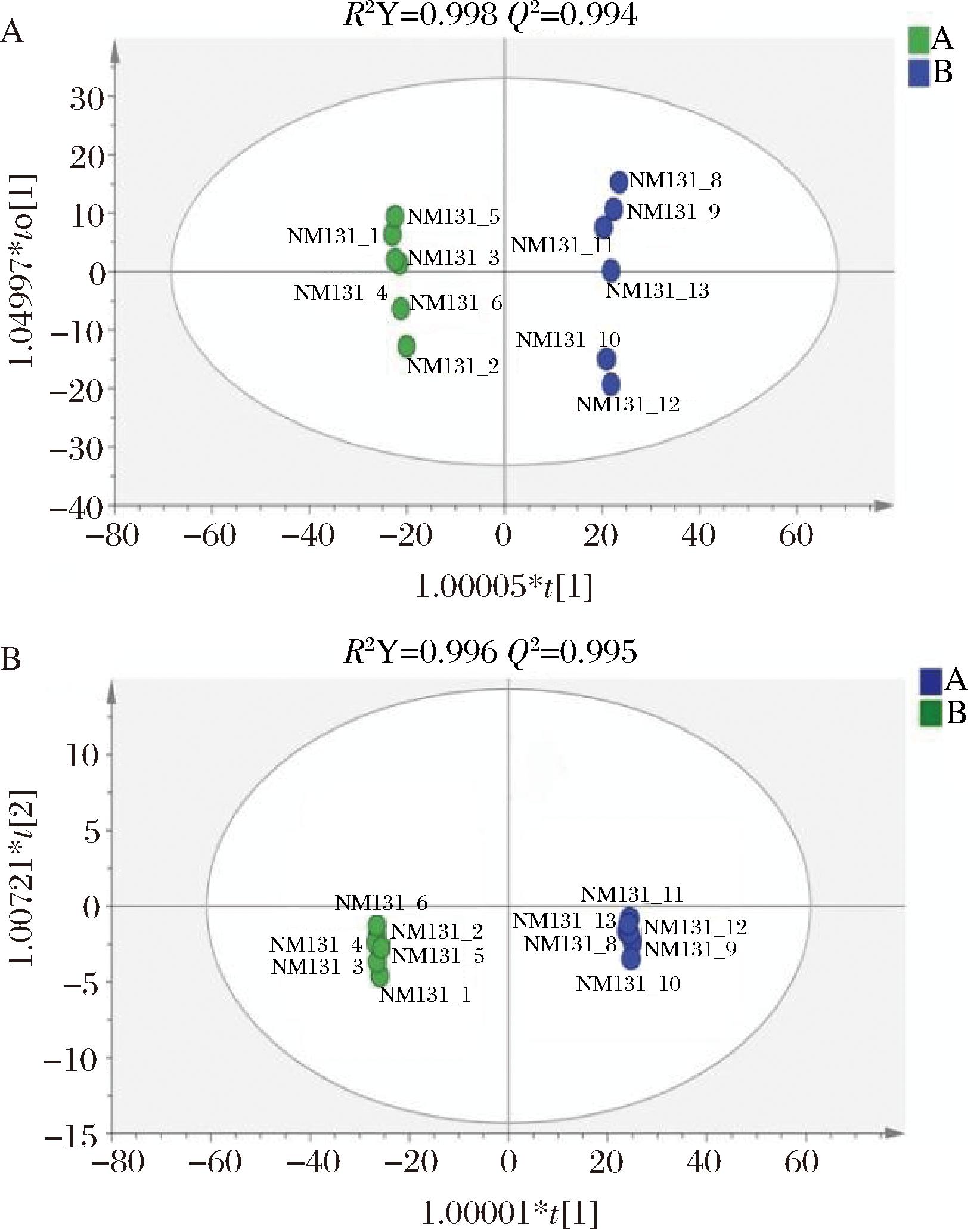
A-正离子模式下的OPLS-DA分析;B-负离子模式下的OPLS-DA分析
图4 L.kefiri IMAU10804酸胁迫前后OPLS-DA模型得分图
Fig.4 Score plot of OPLS-DA before and after L.kefiri IMAU10804 acid stress
注:图中A和B分别表示L.kefiri IMAU10804酸胁迫前后状态。
由于在代谢数据样本量较小的情况下,OPLS-DA分析方法容易出现过拟合现象,为了确认OPLS-DA结果是否为假阳性,置换测试是必要的[17]。因此,ESI+模式和ESI-模式的数据通过排列测试(200个随机排列)进行评估,使用从排列测试获得的回归线和Y轴之间的截距作为标准来确定模型是否过拟合[18]。2个模型中R2和Q2的截距均小于模型变量的解释和预测值(在所有情况下均约等于1;图5-A、图5-B),这表明模型没有过度拟合,生成的模型是可靠的。
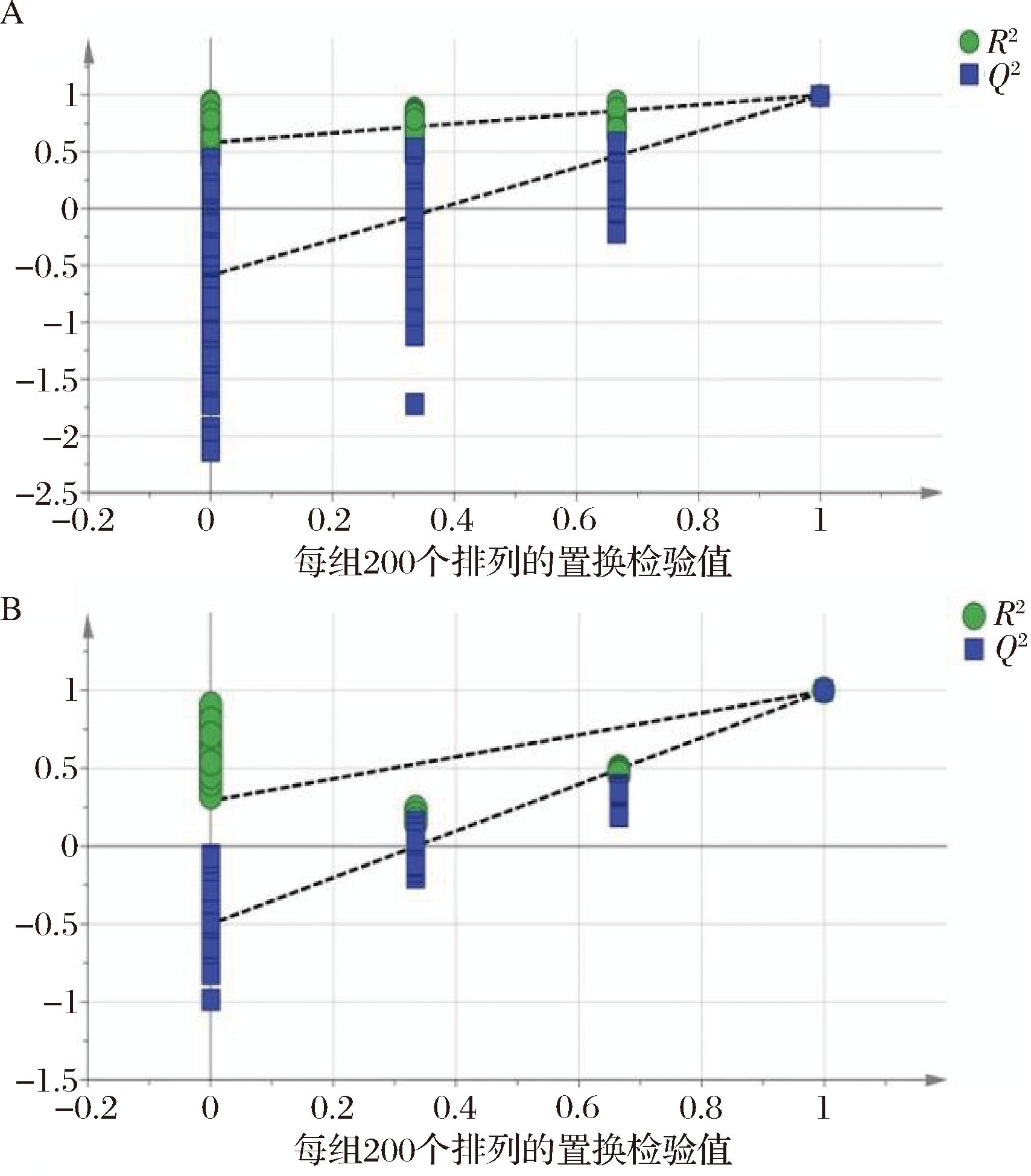
A-正离子模式下的置换检验图;B-负离子模式下的置换检验图
图5 L.kefiri IMAU10804酸胁迫前后置换检验图
Fig.5 Permutation test plots of before and after L.kefiri IMAU10804 acid stress
2.4.2 代谢物物质的种类及重要代谢通路KEGG注释结果
L.kefiri IMAU10804酸胁迫前后样本的代谢组学数据集共鉴定出1 411种代谢物,包括ESI+模式的796种代谢物和ESI-模式的615种代谢物。在HMDB 4.0(Human Metabolome Database)数据库中对获得的代谢产物进行分类汇总,其中,有机酸及其衍生物(含氨基酸)占32.95%,脂类物质和类脂分子占19.39%,其他物质占47.66%。通过代谢物占比可以看出有机酸、氨基酸和脂类物质是主要成分。
根据代谢物数量生成KEGG重要通路统计图,参与某个通路的代谢物越多,说明该生物学通路越活跃。由图6可以看出,重要代谢通路包括ABC转运蛋白、嘌呤代谢、精氨酸和脯氨酸代谢、D-氨基酸代谢、嘧啶代谢、甘油磷脂代谢、赖氨酸降解、氨基糖和核苷酸糖代谢、半胱氨酸和蛋氨酸代谢以及色氨酸代谢。在重要通路分析中,ABC转运蛋白途径是最富集的途径,因为ABC转运蛋白主要是通过介导营养物质和其他分子进入细胞,并利用ATP的结合和水解为各种底物跨膜转运提供动力,维持细胞在酸胁迫下生存所需的平衡条件,保护细胞免受酸胁迫[19]。另外,许多通路涉及氨基酸代谢或与氨基酸代谢有关,这表明可能通过调节氨基酸代谢来保护细胞免受酸性应激以维持细胞生长。例如在精氨酸和脯氨酸代谢途径中,精氨酸转化为瓜氨酸和鸟氨酸,同时产生NH3,并与H+反应使环境碱化,利用这种方式来减少酸性应激给细胞造成的损伤[20]。这些途径的激活对于L.kefiri IMAU10804在酸胁迫过程中实现生理平衡至关重要。
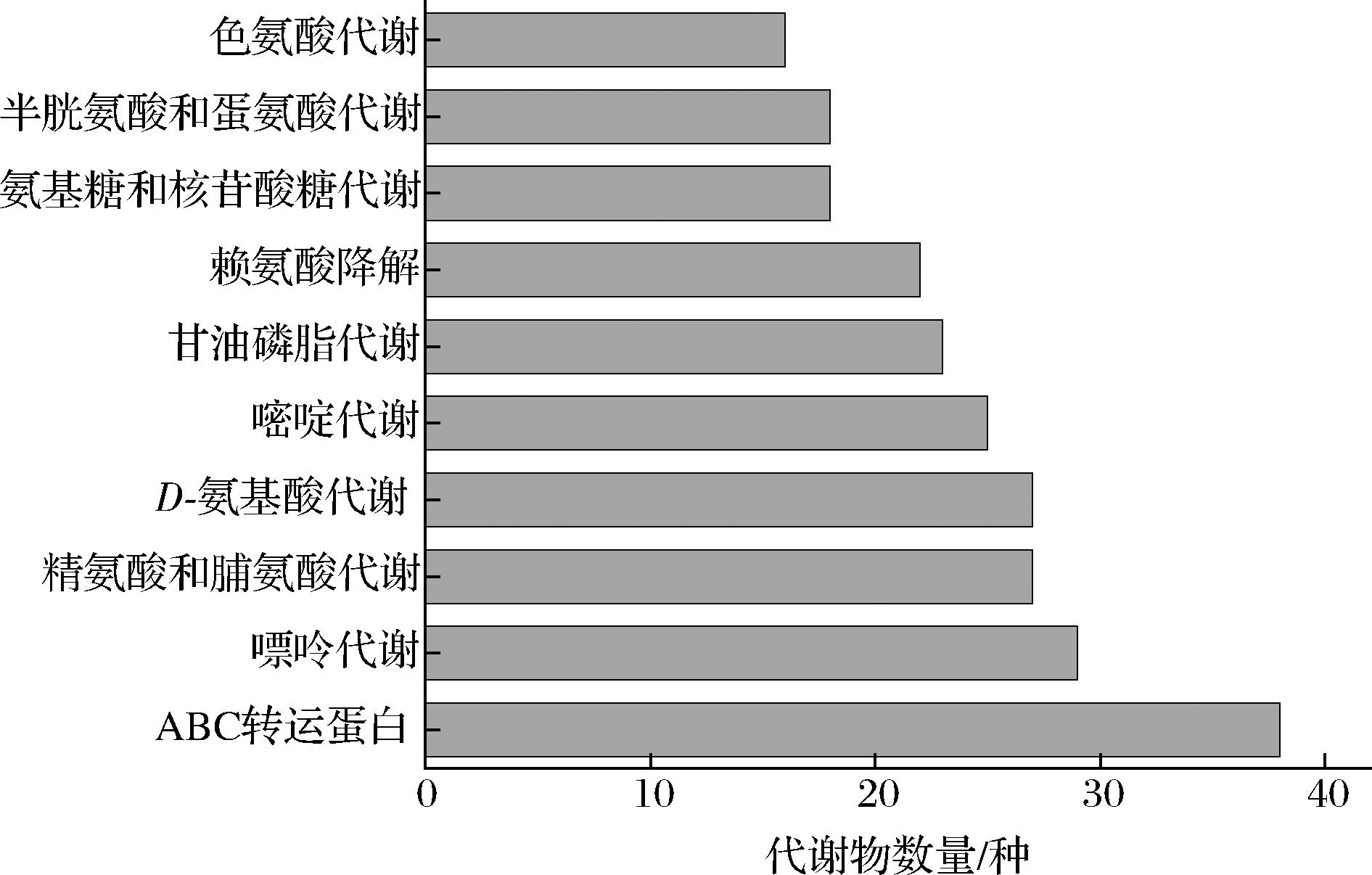
图6 L.kefiri IMAU10804酸胁迫前后KEGG重要通路统计
Fig.6 Statistics of KEGG important pathways before and after acid stress in L.kefiri IMAU10804
2.4.3 差异性代谢物的筛选
对L.kefiri IMAU10804酸胁迫前后样本中检测到的所有代谢产物进行差异性分析,根据P<0.05、VIP≥1和FC≥1.5的筛选原则,在完整的数据集中筛选出了24种差异代谢物。与酸胁迫前菌株相比,酸胁迫后菌株具有17种显著增加和7种显著减少的代谢物,具体差异代谢物见表3。
表3 L.kefiri IMAU10804酸胁迫前后的差异代谢物
Table 3 Differential metabolites of L.kefiri IMAU10804 before and after acid stress

类别序号名称分子式保留时间质荷比(m/z)T/B 氨基酸类1L-丙氨酸C3H7NO20.595 590.055 4↑2L-谷氨酰胺C5H10N2O32.276 6147.076 5↑3天冬氨酸C4H7NO40.593 6132.029 3↑4L-谷氨酸C5H9NO40.740 0256.011 3↑5L-苏氨酸C4H9NO31.335 784.044 9↑6苯丙氨酸C9H11NO25.094 2264.124 1↑7L-2-氨基-6-氧代己酸C6H11NO31.952 2335.146↓84-吡啶氧酸C8H9NO43.476 8182.045 3↑碳水化合物类1D-氨基葡萄糖C12H24N2O101.110 0502.208 2↑2海藻糖C12H22O110.906 1377.085 4↑3低聚木糖C5H12O51.620 0132.064 9↓4乳糖C12H22O110.593 6132.029 3↓5氨基葡萄糖6-磷酸C6H14NO8P1.401 7258.038 4↓维生素类1烟酰胺C6H6N2O3.520 0167.044 9↑2核黄素C17H20N4O60.930 0455.055 5↑324,25,26,27-四聚-23-氧代羟基维生素D3C23H34O32.388 3403.219↓脂类1棕榈油酸(C16:1)C16H32O23.790 0231.076 5↑2氰基丙烯酸异己酯C10H15NO25.012 1180.102 4↑3精对苯二甲酸已二醇酯C20H30O85.834 6433.161 7↑4N-邻氨基苯甲酸酯C12H16NO9P7.840 0534.908 6↑其他1鸟苷C10H13N5O52.126 9282.084 3↓2黄嘌呤C5H4N4O22.429 5283.068 4↑3鸟嘌呤C5H5N5O1.024 1150.041 3↑4辅酶AC21H36N7O16PS2.022 2768.122 1↓总数24
注:T/B表示两种细胞状态之间代谢物水平的显著性上升或显著性下降。↑/↓意味着与酸胁迫前状态的细胞相比,酸胁迫后细胞的代谢物差异性增加或减少。
由表3可知,差异代谢物主要有五大类,包括氨基酸类、碳水化合物类、维生素类、脂类和其他。这些相关代谢物的变化代表了L.kefiri IMAU10804酸胁迫过程中的生理化学变化。
氨基酸是一种有机化合物,对蛋白质构建至关重要,可以支持乳酸菌的生长、繁殖和代谢[21]。L-谷氨酸是细胞内丰富的游离氨基酸,谷氨酸代谢产物是细菌生长的必需物质,是构成细胞壁肽聚糖的主要成分,也在保持细胞的完整性和稳态方面发挥作用[22]。此外,据报道,L-谷氨酰胺主要通过谷氨酰合成酶和谷氨酸脱氢酶转化而成,L-谷氨酰胺有效降低菌在逆境胁迫中受到的氧化损伤程度[23]。本研究中,L.kefiri IMAU10804酸胁迫后L-谷氨酸、L-谷氨酰胺、天冬氨酸、L-丙氨酸等氨基酸含量高于酸胁迫前,推测以上积累的氨基酸在一定程度上可能对逆境条件下L.kefiri IMAU10804起保护作用。
碳水化合物代谢可以产生更多的能量,微生物细胞可以消耗这些能量来对抗酸胁迫,碳水化合物的获取和代谢对乳酸菌在酸胁迫下的生存至关重要[24]。L.kefiri IMAU10804在低pH下细胞内海藻糖、D-氨基葡萄糖含量高于酸胁迫前,海藻糖是生物体对于外部环境变化所形成的一种典型的应激代谢物,对细胞的保护作用是海藻糖中的羟基代替水分子,通过氢键与生物体内的蛋白质、糖类、脂类等大分子物质结合,从而稳定并保持大分子的结构和功能,以保护机体抵抗外部恶劣环境[25]。此外,L.kefiri IMAU10804在低pH下细胞内低聚木糖、乳糖、氨基葡萄糖6-磷酸含量低于酸胁迫前。低聚木糖是由2~9个木糖分子通过β-1,4糖苷键连接而成,近年来的研究表明,低聚木糖除了提供能量和促进有益乳酸菌(如双岐杆菌)的生长外,还具有多种生物学功能[26]。因此,这很好地解释了L.kefiri IMAU10804在低pH环境下消耗能量来抵御酸胁迫。
脂类物质能维持菌体细胞膜的结构和功能,膜脂肪酸组成和含量的改变可以调节细胞膜的通透性和流动性,以此来应对外部酸胁迫[27]。本实验中L.kefiri IMAU10804在低pH下不饱和脂肪酸棕榈油酸(C16:1)含量高于酸胁迫前,吴重德等[27]证明了Lb.casei Zhang适应性进化菌株(pH 4.3 MRS培养基中连续传代70 d)在酸胁迫条件下,细胞膜脂肪酸种类及含量会发生了变化,不饱和脂肪酸/饱和脂肪酸比率(UFA/SFA,不饱和度)增加,来减少由逆境胁迫引起的对细胞造成的伤害。因此推测棕榈油酸(C16:1)含量的增加对L.kefiri IMAU10804在低pH环境过程中应对外部酸胁迫起重要的作用。
除了氨基酸、碳水化合物和脂类物质外,还发现一些来自其他大分子的差异代谢物,例如黄嘌呤和鸟嘌呤。嘌呤不仅是储存能量的主要生物分子,还是核苷酸的组成部分,具有维持细胞生理功能的作用[28-29]。
3 结论
本研究评价了L.kefiri IMAU10804的耐酸性,发现L.kefiri IMAU10804在pH 3.0的MRS液体培养基中胁迫60 min后菌体的存活率为88%,没有出现显著的变化,且在此条件下菌体得到充分的酸刺激;细胞在pH 3.0的MRS液体培养基中胁迫60 min后伴随着大量代谢产物变化,在ESI+模式和ESI-模式下分别鉴定出796种和615种代谢物,其中代谢物主要富集在ABC转运蛋白、精氨酸和脯氨酸代谢、嘌呤代谢等代谢通路,这些途径的激活对于L.kefiri IMAU10804在酸胁迫过程中实现生理平衡是至关重要的;最后根据P<0.05、VIP≥1和FC≥1.5的筛选原则,共筛选出24种主要差异代谢物,与酸胁迫前菌株相比,细胞在胁迫的过程中有17种上调和7种下调的代谢物,推测24种差异代谢物的合成在L.kefiri IMAU10804胁迫过程中起关键作用。本研究提供了关于菌株酸胁迫过程中随之而来的代谢组学变化的有用信息,可为后续乳酸菌产品开发和应用提供参考。
[1] WANG Y Q, WU J T, LV M X, et al. Metabolism characteristics of lactic acid bacteria and the expanding applications in food industry[J]. Frontiers in Bioengineering and Biotechnology, 2021, 9: 612285.
[2] 何进, 徐思杨, 刘波, 等. 乳酸菌在农业和食品加工中的应用研究进展[J]. 微生物学杂志, 2022, 42(4): 1-11.HE J, XU S Y, LIU B, et al. Advance on applied researches of lactic acid bacteria in agriculture and food industry[J]. Journal of Microbiology, 2022, 42(4): 1-11.
[3] GUAN N Z, LIU L. Microbial response to acid stress: mechanisms and applications[J]. Applied Microbiology and Biotechnology, 2020, 104(1): 51-65.
[4] WU C D, HUANG J, ZHOU R Q. Progress in engineering acid stress resistance of lactic acid bacteria[J]. Applied Microbiology and Biotechnology, 2014, 98(3): 1055-1063.
[5] WANG H Y, ZHANG Y H, DAI L X, et al. Metabolomic differences between viable but nonculturable and recovered Lacticaseibacillus paracasei Zhang[J]. Foods, 2023, 12(18): 3472.
[6] 李娜. L. rhamnosus太空诱变株R7970-3发酵牛乳的研制及代谢组学研究[D]. 呼和浩特: 内蒙古农业大学, 2023.LI N. Development of milk fermented by L. rhamnosus space mutant strain R7970-3 and metabolomics study[D]. Hohhot: Inner Mongolia Agricultural University, 2023.
[7] 郑莎莎. 干酪乳杆菌H1发酵红酸汤的品质影响及特征代谢物研究[D]. 贵阳: 贵州大学, 2020.ZHENG S S. Study on quality influence and characteristic metabolites of tomato sour soup fermented by Lactobacillus casei H1[D]. Guiyang: Guizhou University, 2020.
[8] 张筠, 孟祥晨. 乳酸菌的胁迫应答及其对碳水化合物代谢的影响[J]. 中国食品学报, 2017, 17(6): 145-151.ZHANG J, MENG X C. Stress responses and impact of carbonhydrate metabolism in lactic acid bacteria[J]. Journal of Chinese Institute of Food Science and Technology, 2017, 17(6): 145-151.
[9] ZHENG J S, WITTOUCK S, SALVETTI E, et al. A taxonomic note on the genus Lactobacillus: Description of 23 novel genera, emended description of the genus Lactobacillus Beijerinck 1901, and union of Lactobacillaceae and Leuconostocaceae[J]. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(4): 2782-2858.
[10] 张栋, 赵彤, 荀一萍, 等. 一株开菲尔乳杆菌的分离及特性研究[J]. 食品科技, 2018, 43(8): 15-18.ZHANG D, ZHAO T, XUN Y P, et al. Isolation and properties of Lactococcuslactis kefiri[J]. Food Science and Technology, 2018, 43(8): 15-18.
[11] CARASI P, MALAMUD M, SERRADELL M A. Potentiality of food-isolated Lentilactobacillus kefiri strains as probiotics: State-of-art and perspectives[J]. Current Microbiology, 2021, 79(1):21.
[12] MANNA L, RIZZI E, BAFILE E, et al. Lentilactobacillus kefiri SGL 13 and andrographis paniculata alleviate dextran sulfate sodium induced colitis in mice[J]. Frontiers in Nutrition, 2023, 10: 1072334.
[13] 包秋华, 王丽娜, 张雨虹, 等. 益生菌干酪乳杆菌Zhang VBNC态和正常态的代谢组学研究[J]. 中国乳品工业, 2022, 50(6): 10-15.BAO Q H, WANG L N, ZHANG Y H, et al. Metabonomics study of probiotic Lactobacillus casei Zhang VBNC state and normal state[J]. China Dairy Industry, 2022, 50(6): 10-15.
[14] 王道滇, 肖梦林, 魏光强, 等. 基于干酪乳杆菌ZLC01的山羊奶发酵乳及其特征性代谢物研究[J]. 食品与发酵工业, 2024, 50(9):71-80.WANG D D, XIAO M L, WEI G Q, et al. Fermented goat milk with Lactobacillus casei ZLC01 and its characteristic metabolites[J]. Food and Fermentation Industries, 2024, 50(9):71-80.
[15] 林亚男. 亚压力胁迫对Lactobacillus acidophilus LA-5营养消耗模式的影响[D]. 哈尔滨: 东北农业大学, 2022.LIN Y N. The effect of sub-stress on the nutrient consumption pattern of Lactobacillus acidophilus LA-5[D]. Harbin: Northeast Agricultural University, 2022.
[16] SUN Y R, PENG C T, WANG J C, et al. Metabolic footprint analysis of volatile metabolites to discriminate between different key time points in the fermentation and storage of starter cultures and probiotic Lactobacillus casei Zhang milk[J]. Journal of Dairy Science, 2021, 104(3): 2553-2563.
[17] 马立清, 张文羿, 孙天松. 嗜热链球菌S4发酵乳代谢组学的研究[J]. 食品与发酵工业, 2021, 47(19): 71-78.MA L Q, ZHANG W Y, SUN T S. Metabolomics analysis of fermented milk with Streptococcus thermophilus S4[J]. Food and Fermentation Industries, 2021, 47(19): 71-78.
[18] 孙雷. 鼠李糖乳酪杆菌太空诱变株R7970-3在发酵羊乳中的应用研究[D]. 呼和浩特: 内蒙古农业大学, 2023.SUN L. Study on the application of Lacticaseibacillus rhamnosus space mutant strain R7970-3 in fermented goat milk[D]. Hohhot: Inner Mongolia Agricultural University, 2023.
[19] ZHU Z M, YANG J H, YANG P S, et al. Enhanced acid-stress tolerance in Lactococcus lactis NZ9000 by overexpression of ABC transporters[J]. Microbial Cell Factories, 2019, 18(1): 136.
[20] WANG W W, HE J Y, PAN D D, et al. Metabolomics analysis of Lactobacillus plantarum ATCC 14917 adhesion activity under initial acid and alkali stress[J]. PLoS One, 2018, 13(5): e0196231.
[21] MIYAJIMA, M. Amino acids: Key sources for immunometabolites and immunotransmitters[J]. International Immunology, 2020, 32(7): 435-446.
[22] QIAO Y L, LIU G F, LENG C, et al. Metabolic profiles of cysteine, methionine, glutamate, glutamine, arginine, aspartate, asparagine, alanine and glutathione in Streptococcus thermophilus during pH-controlled batch fermentations[J]. Scientific Reports, 2018, 8(1): 12441.
[23] LI H, MA M L, LUO S, et al. Metabolic responses to ethanol in Saccha-romyces cerevisiae using a gas chromatography tandem mass spectrome-try-based metabolomics approach[J]. The International Journal of Biochemistry &Cell Biology, 2012, 44(7): 1087-1096.
[24] ZHANG J, CAIYIN Q, FENG W J, et al. Enhance nisin yield via improving acid-tolerant capability of Lactococcus lactis F44[J]. Scientific Reports, 2016, 6: 27973.
[25] 杨友馨, 王瑞雪, 王俊国. 海藻糖对乳酸菌保护机制的研究进展[J]. 乳业科学与技术, 2021, 44(6): 45-50.YANG Y X, WANG R X, WANG J G. Progress in research on the protective mechanism of trehalose on lactic acid bacteria[J]. Journal of Dairy Science and Technology, 2021, 44(6): 45-50.
[26] SUN Z K, YUE Z H, LIU E, et al. Assessment of the bifidogenic and antibacterial activities of xylooligosaccharide[J]. Frontiers in Nutrition, 2022, 9: 858949.
[27] WU C D, ZHANG J, WANG M, et al. Lactobacillus casei combats acid stress by maintaining cell membrane functionality[J]. Journal of Industrial Microbiology and Biotechnology, 2012, 39(7): 1031-1039.
[28] TANG Z W, YE W R, CHEN H T, et al. Role of purines in regulation of metabolic reprogramming[J]. Purinergic Signalling, 2019, 15(4): 423-438.
[29] MAAN K, BAGHEL R, DHARIWAL S, et al. Metabolomics and transcriptomics based multi-omics integration reveals radiation-induced altered pathway networking and underlying mechanism[J]. NPJ Systems Biology and Applications, 2023, 9(1): 42.