乳酸菌(lactic acid bacteria, LAB)是一类能代谢可发酵碳源产生大量乳酸的细菌总称,通常为革兰氏阳性球菌或杆菌,无内生孢子形成[1]。自然界中已发现至少23个LAB属,其中乳杆菌属、乳球菌属、链球菌属、双歧杆菌属、肠球菌属、片球菌属和明串珠菌属广泛应用于食品和医药等领域,也是人们认识度最高的乳酸菌种类[2]。乳酸菌是发酵食品生产中最常用的发酵剂,不但具有优良的发酵特性,其代谢产物(如抗菌肽、乳酸、醋酸等)对于改善食品品质也至关重要。除在食品发酵领域的应用,乳酸菌在人体健康方面也扮演着重要角色。乳酸菌与肠道微生物群的平衡密切相关,通过竞争性排挤和抑制其他致病菌的生长,有助于维持肠道微生态稳定。此外,乳酸菌能够调节宿主的免疫反应,促进肠道黏膜屏障的完整性,并通过产生有益代谢产物来促进营养吸收[3]。然而,乳酸菌的许多生物学特性和生理功能仍尚未解析。近年来随着高通量测序技术的快速发展,推动乳酸菌基因组和转录组等多组学研究,引发对乳酸菌表观遗传调控的重点关注,其中乳酸菌sRNA(small RNA)研究成为热点。
通常细菌sRNA是一类长度在40~500 nt(核苷酸)之间、不编码蛋白质的RNA分子,主要以转录后调控的形式发挥作用[4]。sRNA与靶标mRNA进行碱基互补配对或与相应蛋白分子相互作用,调节转录和翻译的过程,使细菌快速响应外界环境改变[5]。根据调控靶标的不同机制,sRNA主要分为顺式编码sRNA、反式编码sRNA、调控蛋白活性的sRNA和CRISPR(成簇规律间隔的短回文重复序列)相关sRNA。顺式编码sRNA与靶标mRNA进行严格的碱基互补配对,抑制其表达或使其基因沉默[6]。反式编码sRNA通过与靶标mRNA上7~12个碱基进行不严格的互补配对,抑制或促进靶标mRNA的翻译,加速或减缓靶标mRNA的降解。调控蛋白的sRNA直接与蛋白质相互作用,通过与RNA或DNA靶标竞争来调控RNA结合蛋白或调节酶的活性。而CRISPR RNA(crRNA)是针对外源核酸的短链RNA,其引导核酸酶Cas蛋白与外源核酸结合,从而发挥CRISPR-Cas系统功能清除外源核酸[7]。众所周知,sRNA在调控网络中发挥着关键作用,对理解细菌代谢、环境适应和毒力等生理过程至关重要[8]。
生物信息学预测结合实验验证是研究sRNA的普遍方法。生物信息学预测sRNA需根据已识别的sRNA序列和结构保守性来建立预测模型,如基于比较基因组学模型的QRNA、RNAz、EvoFold和sRNAPredict等软件以及基于机器学习的sRNA预测模型[9]。研究发现大部分sRNA在不同菌种间缺乏高度序列同源性,sRNA序列的差异导致其相互作用的靶标也发生相应变化,所以仅采用生物信息学预测sRNA具有明显的局限性。RNA-Seq(转录组测序)、总RNA标记、DNA微阵列、免疫共沉淀和鸟枪克隆法等已被广泛应用于sRNA实验检测和验证。ZHANG等[10]将全基因组搜索得到的17个sRNA,采用免疫共沉淀和微阵列方法鉴定出与Hfq(RNA分子伴侣蛋白)结合的sRNA。VOGEL等[11]在大肠杆菌中使用鸟枪克隆法构建sRNA的cDNA文库,文库不但包含功能已知的20种sRNA,还发现许多新的sRNA。
鉴于乳酸菌在食品发酵和生物制剂生产中的重要性,展开乳酸菌sRNA研究具有重要的理论和应用价值。了解乳酸菌sRNA的机制和功能可以更好的从分子层面理解其菌株生理特性,为改造菌株代谢途径、提高代谢物产量和应对环境压力胁迫等研究提供新思路。尽管研究表明sRNA在乳酸菌中发挥着重要作用[12],但目前对乳酸菌sRNA的研究非常有限。本文总结乳酸菌sRNA研究的最新进展,重点关注乳酸菌sRNA的发现与鉴定、调控功能及作用机制,并展望乳酸菌sRNA研究的发展方向和挑战,以期为揭示乳酸菌sRNA功能机制及应用提供思路。
1 细菌sRNA作用机制
细菌中研究最广泛的sRNA是反式编码sRNA,其最常见的调控机制是sRNA与靶标mRNA的核糖体结合位点(ribosomebinding site,RBS)进行不完全碱基互补配对,通过阻止核糖体结合来抑制翻译起始(图1-a)。在大肠杆菌中首次发现sRNA MicC通过与核糖体竞争RBS抑制孔蛋白OmpC的翻译[13]。相反的是激活翻译,有些靶标mRNA的SD(Shine-Dalgarno)序列存在二级结构阻止核糖体结合,sRNA作用下RBS被打开,使翻译正常进行(图1-b)。大肠杆菌sRNA DsrA和RyhB就通过这种机制分别激活Rpos[14]和shiA[15]的翻译。目前大多数报道的sRNA通过掩蔽SD序列或AUG起始密码子来抑制靶标mRNA转录和翻译。然而,少数sRNA缺乏明显的SD和AUG互补性,其介导的翻译控制发生在其他mRNA位点。例如,BOUVIER等[16]发现沙门氏菌sRNA RybB通过与靶标mRNA的5′编码区配对来抑制ompN mRNA的翻译。
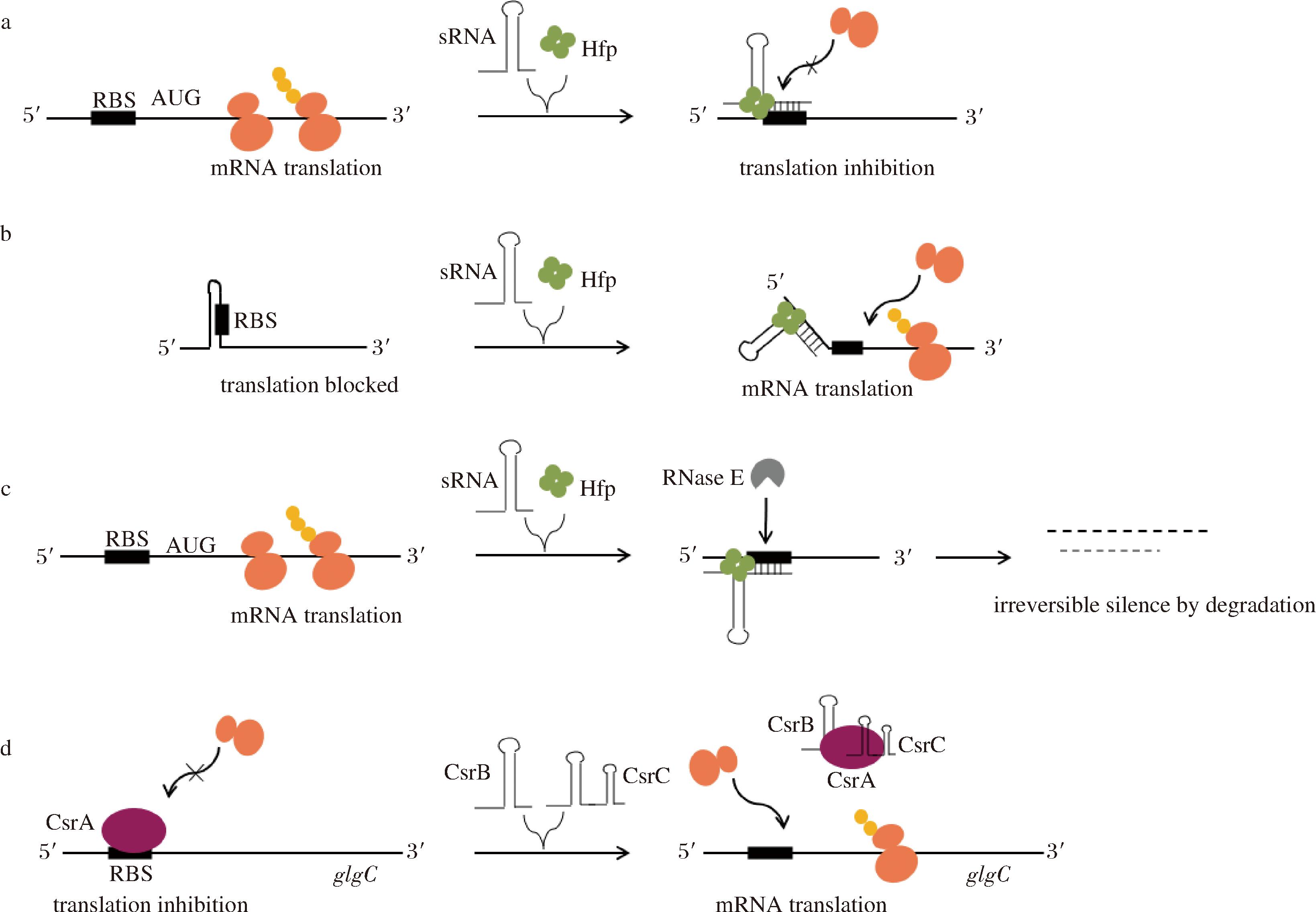
a-sRNA阻断RBS抑制翻译;b-通过改变mRNA结构激活翻译;c-sRNA与靶基因mRNA不完全碱基配对抑制翻译并加速RNase的降解;d-CsrA介导的大肠杆菌glgC调控模型
图1 反式编码sRNA调控靶标的机制
Fig.1 Regulatory mechanisms of trans-encoded sRNAs
另一常见机制是sRNA借助核糖核酸内切酶,使mRNA降解来发挥调节作用,这一过程需要伴侣蛋白的参与(图1-c)。RNase E是细菌中主要的核糖核酸酶,Hfq保护应激诱导的sRNA免受RNase E切割,稳定并促进它们与靶标mRNA的碱基配对[17]。sRNA与靶标mRNA碱基互补配对后,参与靶标mRNA降解的RNase E可以特异性作用于富含AU的单链RNA,将两者同时降解[18]。PRÉVOST等[19]报道RyhB与sodB mRNA碱基配对后诱导RNase E在位于RBS下游>350 nt的远端位点进行切割,完成mRNA的降解。sRNA如何借助核糖核酸酶使mRNA降解的机制还不明确,但猜测一种可能是sRNA与mRNA碱基互补配对后失去核糖体保护,使其更容易受到核糖核酸酶攻击;另一种可能是形成的sRNA/Hfp/RNase E三元复合物对RNA降解有促进作用。
此外,sRNA还遵循RNA结合蛋白螯合机制,使蛋白调节功能受到抑制。例如,大肠杆菌sRNA CrsB和CrsC作为CrsA结合蛋白的拮抗物,调控糖原代谢基因glgC的翻译起始[20]。CsrA蛋白具有与RNA上富含GGA元件结合的特异性亲和力,作为转录后调节因子激活或抑制下游基因的表达,尤其是参与碳代谢途径的基因。CsrA通过与glgC转录物的RBS周围序列结合抑制翻译起始。CsrB和CsrC都含有多个CsrA结合位点,通过结合CsrA使其远离靶标而起作用(图1-d)。因此,sRNA能够通过结合调控蛋白,间接发挥调节基因表达的作用。
2 细菌sRNA生物学功能
细菌sRNA参与调控细胞能量代谢、群体感应、生物膜形成、应激反应、毒力和对生长条件的适应等重要生理过程(表1)。例如,碳氮代谢是细胞生命活动的关键部分,sRNA GlnZ通过调节影响碳和氮通量的基因,在低氮条件下影响大肠杆菌的生长。GlnZ与编码谷氨酰胺合成酶基因glnA mRNA的3′-UTR(非编码区)发生作用,同时抑制参与谷氨酰胺转运和柠檬酸循环的glnP和sucA mRNA的表达[21]。sRNA MalH通过调节大肠杆菌的麦芽孔蛋白表达来促进碳源利用[22]。MalH衍生于malG mRNA 3′端稳定的降解中间体,是麦芽糖摄取操纵子malEFG转录产物的一部分。MalH过表达抑制外膜孔蛋白和高亲和力麦芽糖/麦芽糊精转运蛋白LamB的阻遏物MicA的表达,抑制MalH会显著减少LamB的积累,MalH通过抑制竞争途径,直接或间接促进LamB在外膜中积累。
表1 细菌sRNA的生物学功能
Table 1 Biological functions of sRNAs in bacteria
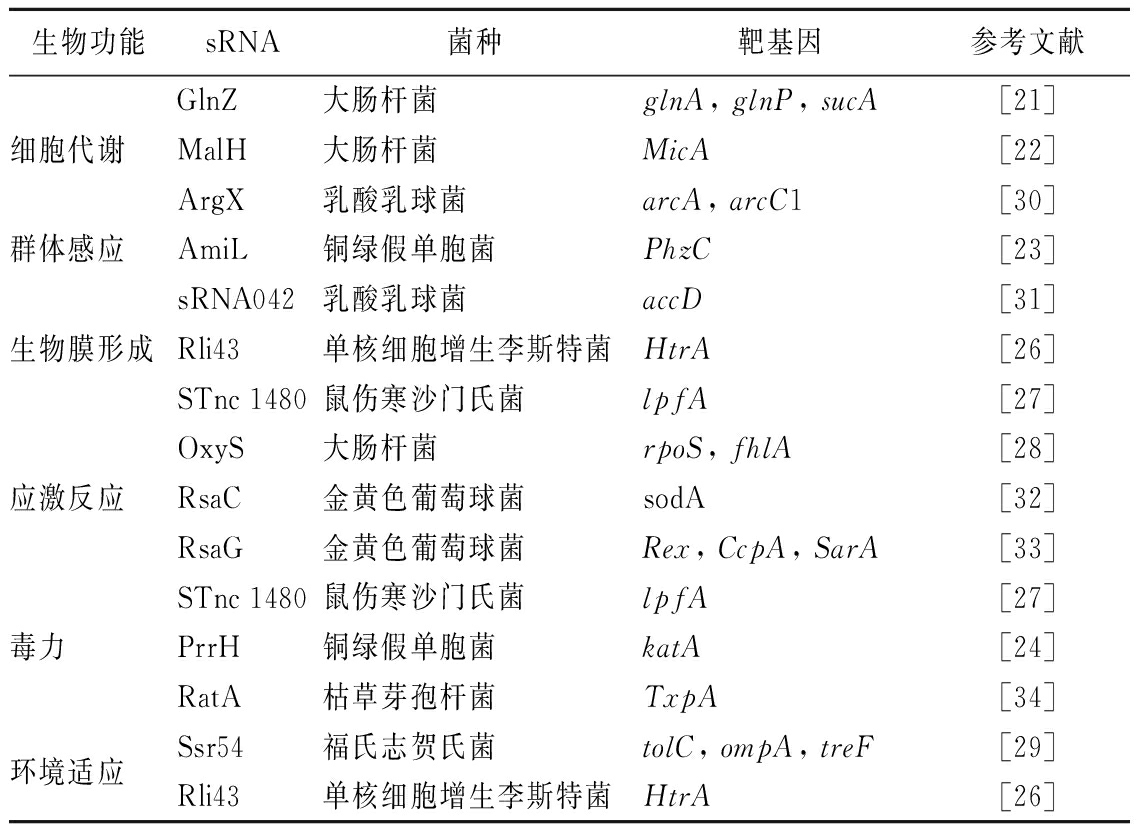
生物功能sRNA菌种靶基因参考文献GlnZ大肠杆菌glnA, glnP, sucA[21]细胞代谢MalH大肠杆菌MicA[22]ArgX乳酸乳球菌arcA, arcC1[30]群体感应AmiL铜绿假单胞菌PhzC[23]sRNA042乳酸乳球菌accD[31]生物膜形成Rli43单核细胞增生李斯特菌HtrA[26]STnc 1480鼠伤寒沙门氏菌lpfA[27]OxyS大肠杆菌rpoS, fhlA[28]应激反应RsaC金黄色葡萄球菌sodA[32]RsaG金黄色葡萄球菌Rex, CcpA, SarA[33]STnc 1480鼠伤寒沙门氏菌lpfA[27]毒力PrrH铜绿假单胞菌katA[24]RatA枯草芽孢杆菌TxpA[34]环境适应Ssr54福氏志贺氏菌tolC, ompA, treF[29]Rli43单核细胞增生李斯特菌HtrA[26]
群体感应(quorum sonsing,QS)是细菌根据细胞密度变化进行基因表达调控的一种生理行为。通过RNA-Seq测序铜绿假单胞菌,AmiL被鉴定为新型QS调节sRNA[23],其表达受las和rhl(QS系统)负调控。AmiL直接靶向PhzC(绿脓素合成关键基因),此外还影响多种QS介导的包括弹性蛋白酶、鼠李糖脂和溶血素在内的毒力因子。sRNA PrrH在铜绿假单胞菌中调节铁稳态和毒力,其表达受铁离子调节限制,PrrH以exsA(Ⅲ型分泌系统主调节基因)依赖性方式促进铜绿假单胞菌的溶血活性,并负调控过氧化氢酶基因katA,从而降低菌体的过氧化氢耐受性[24]。金黄色葡萄球菌Teg41是从编码细胞溶解性α-苯酚可溶性调节蛋白(αPSM)基因座分化转录的sRNA,对αPSM合成起促进作用;Teg41缺失使αPSM含量显著降低,导致金黄色葡萄球菌毒力减弱[25]。
通常细菌sRNA并非发挥单一功能起作用,例如单核细胞增生李斯特菌sRNA rli43介导对环境适应、生物膜形成和毒力的调控[26]。Rli43与htrA mRNA的5′-UTR互补配对,在转录后水平正向调节htrA的表达水平,而HtrA在细菌运动、生物膜形成和毒力基因表达中发挥重要作用。鼠伤寒沙门氏菌sRNA STnc1480直接靶向lpfA mRNA(编码lpf菌毛的主要亚基)正调控基因表达,其有助于在感染肠上皮细胞的生物膜形成,提高菌体肠道定植能力和自身致病性[27]。在大肠杆菌及其他肠杆菌中,sRNA OxyS作为抗突变剂可抑制RpoS(RNA聚合酶的一种σ亚基)翻译,调节fhlA(hyp操纵子激活剂)保护细胞免受DNA氧化损伤[28]。渗透压是细菌生长过程中常见的压力胁迫,福氏志贺氏菌sRNA Ssr54通过调节tolC、ompA和treF(编码胞质海藻糖酶)的表达来影响细胞在高渗条件下的毒力和存活[29];TolC 是一种外膜通道蛋白,在疾病发病机制中有助于细菌黏附、抗生素耐药性和侵袭;OmpA是主要的外膜蛋白,在病原体的毒力中发挥重要作用。
3 乳酸菌sRNA
尽管sRNA广泛存在于原核生物,但在乳酸菌的研究非常有限,以下着重总结四类具有益生功能的乳酸菌sRNA研究进展,如表2所示。
表2 乳酸菌中的sRNA概览
Table 2 Overview of sRNAs in Lactic acid bacteria

菌种sRNA靶基因生物功能参考文献乳酸乳球菌6S RNALLMGnc_147CcpAAraC碳代谢[12]sRNA042accD[31]s015atpG, busAB, ung, ywdA, il-vB酸适应[36]anti41glnP, glnQ, amtB, glnK[37]ArgXarcA, arcC1氨基酸代谢[30]干酪乳酪杆菌SgrSsgrT葡萄糖磷酸应激RatATxpA抗毒素ffsFfh蛋白质运输RNase PtRNAtRNA成熟rli28c环境适应[38]suhB尚未研究生物膜合成RsaA生物膜合成L10 leader酸适应SsrAsmpB核糖体拯救GlmY, GlmZglms生物膜合成保加利亚乳杆菌嗜酸乳杆菌UspSUsp碳代谢[47]嗜热链球菌sts141, sts392, sts318, sts014高渗胁迫适应sts508, sts087, sts372, sts141, sts375, sts119尚未研究饥饿应激调节[51]sts129, sts226, sts166精氨酸合成AsdS生物膜形成[52]
3.1 乳酸乳球菌(Lactococcus lactis)
乳酸乳球菌在食品工业和重组蛋白生产中广泛应用[35]。实际发酵生产中,温度、渗透压和pH等胁迫诱导sRNA对压力快速响应,使细胞克服有害环境的变化。MEULEN等[12]在受环境胁迫的乳酸乳球菌MG1363转录组中发现调控碳代谢的sRNA 6S RNA和LLMGnc_147。WU等[31]通过RNA-seq和RT-PCR技术在乳酸乳球菌F44中筛选到具有良好耐酸性的sRNA042,Northern印迹显示sRNA042在酸应激条件下转录上调。AccD是脂肪酸生物合成的限速酶,脂肪酸含量增加可提高膜的厚度和流动性,进而增强细胞耐酸性能。融合荧光蛋白实验证明sRNA042可以显著增强accD mRNA稳定性。QI等[36]鉴定出sRNA s015可提高F44菌株的耐酸性和乳酸链球菌素(Nisin)产量,点突变实验表明s015在其特定保守区(5′-GAAAAAAAC-3′)与耐酸相关的靶基因atpG、busAB、ung、ywdA和ilvB相互作用,这些基因参与细胞膜形成、氨基酸代谢和蛋白质合成等关键生理过程。MIAO等[37]利用文库构建在F44中筛选出sRNA anti41,通过抑制glnR的表达来提高nisin产量。过表达anti41使glnR的直接抑制靶标(glnP、glnQ、amtB和glnK)的转录水平显著上调,使细胞在酸胁迫下存活率提高3倍,nisin产量也增加29.83%。
精氨酸代谢受到碳分解代谢阻遏物CcpA、转录阻遏物CodY和精氨酸代谢阻遏物ArgR及其伴侣蛋白AhrC的复杂调控,在乳酸乳球菌耐受酸胁迫发挥重要作用。MEULEN等[30]鉴定出sRNA ArgX协同转录调控因子ArgR参与精氨酸代谢调控。ArgX是ArgR mRNA的3′UTR区衍生的sRNA,由自身启动子转录合成,通过高水平精氨酸浓度(25 mmol/L)诱导产生。ArgX启动子上含CRE-box调控元件,使ArgX表达受到碳分解代谢蛋白CcpA的控制。对ArgX缺失突变株与野生型菌株的转录组和蛋白组分析发现,ArgX通过影响精氨酸脱氨酶合成途径arcA操纵子mRNA稳定性和氨基甲酸酯激酶arcC1的翻译来实现转录后调节。TargetRNA软件预测ArgX与arcC1 RBS相互作用,过表达ArgX和荧光蛋白融合实验证明ArgX对arcC1的表达具有抑制作用。由于ArcC1可将氨基甲酰磷酸转化为氨、ATP和CO2,因此推测ArgX引导精氨酸转向ATP、氨、嘧啶代谢或蛋白质合成。
3.2 干酪乳酪杆菌(Lacticaseibacillus casei)
干酪乳酪杆菌作为乳酸菌的一种,常作为酸奶、奶油和干酪等乳制品的发酵剂或辅助发酵剂,尤其在干酪中应用广泛。NAKASHIMA等[38]在干酪乳酪杆菌中发现SgrS、L10 leader、RatA和rli28c等sRNA,分别参与应激反应、蛋白质和生物膜合成及对不同生长阶段的环境适应,但这些sRNA的具体调控机制尚未完全解析。SgrS是一种独特的双功能sRNA,对葡萄糖磷酸应激作出反应,具有调节mRNA靶点的碱基配对功能和编码SgrT蛋白功能[39];L10 leader和RatA在干酪乳酪杆菌中功能不明,但L10 leader在变异链球菌中调节酸适应性[40],RatA在枯草芽孢杆菌中具有抗毒素作用[34];rli28c在干酪乳酪杆菌中抑制乳酸合成、调节对数期细胞生长和维持静止期细胞存活方面发挥重要作用[41]。在指数期检测到sRNA suhB和RsaA在促进生物膜合成中发挥作用[42]。在稳定期检测到sRNA ffs、RNase P和SsrA分别在蛋白质运输[43]、tRNA成熟[44]和核糖体拯救[45]过程发挥作用。此外,还观察到促进细胞壁生物合成的sRNA GlmY和GlmZ[46]。上述研究表明,sRNA在干酪乳酪杆菌中通过促进蛋白质、生物膜和细胞壁的生物合成,从而维持细胞稳定。
3.3 乳杆菌属(Lactobacillus)
保加利亚乳杆菌和嗜酸乳杆菌等乳杆菌广泛存在于发酵食品和动物消化道等环境。保加利亚乳杆菌和嗜酸乳杆菌中鉴定出与应激蛋白(universal stress protein,Usp)相关的sRNA UspS[47],保守结构分析表明乳杆菌属中UspS和6S RNA存在潜在的对应关系。由于大多数乳杆菌缺乏预测的6S RNA,因此UspS功能可能与大肠杆菌6S RNA类似,通过与RNA聚合酶的σ亚基形成复合物并抑制转录。嗜酸乳杆菌和保加利亚乳杆菌UspS下游普遍存在Usp编码基因,表明UspS可能与Usp mRNA相互作用。此外,在保加利亚乳杆菌LBU_0613下游51 bp和LBU_0612上游91 bp的基因间区,发现一种与编码金属β亚基内酰胺酶基因LBU_0667相互作用的sRNA[48]。
3.4 嗜热链球菌(Streptococcus thermophilus)
嗜热链球菌是一种重要的乳酸菌,作为工业乳制品发酵剂常用于酸奶和奶酪的商业生产。尽管链球菌属已有综述分析其sRNA[49],但都缺乏对食品安全级嗜热链球菌的sRNA进行总结概括。嗜热链球菌CRISPR3-Cas系统中Cas9-crRNA复合物充当RNA引导的核酸内切酶,具有crRNA引导的靶序列识别和蛋白介导的DNA切割作用。KARVELIS发现嗜热链球菌LMD-9基因组CRISPR3-Cas的cas9基因上游存在tracrRNA(反式激活crRNA),通过前crRNA成熟形成crRNA,直接调控Cas9介导的DNA干扰[50]。LIU等[51]发现sRNA在嗜热链球菌不同生长阶段通过调节代谢途径发挥作用。例如,sRNAs sts141、sts392、sts318和sts014参与渗透胁迫的调节;sts508、sts087、sts372、sts141、sts375和sts119参与饥饿应激的调节;sts129、sts226、sts166、sts231、sts204、sts145和sts236参与精氨酸合成;sts033、sts341、sts492、sts140、sts230、sts172和sts377参与精氨酸脱亚氨酸酶合成。sts187和sts251的转录水平在指数期显著增强,它们促进生物素、渗透保护剂和ABC转运蛋白相关的靶基因上调,表明sRNA可通过调节转运系统来促进细菌对营养物质的吸收。嗜热链球菌细胞外小泡(extracellular vesicles,EVs)中存在一种参与群体感应的sRNA AsdS,根据生物信息学预测,它可能负责种内通讯、生物膜形成和运输过程[52]。AsdS在其他链球菌中也非常保守,如在人类病原体化脓性链球菌中AsdS同源sRNA MarS参与调节毒力因子的表达。
4 展望
乳酸菌sRNA研究揭示出乳酸菌多样化的遗传调控网络和调控机制,也为乳酸菌在食品工业和健康领域的应用提供新的思路和方法。然而,乳酸菌sRNA研究存在许多亟待解决的问题和挑战。目前乳酸菌sRNA的发现主要依赖高通量测序技术,但乳酸菌sRNA高效快速鉴定仍是一项挑战性任务。因此,需要开发更加高效精准的生物信息学和分子生物学工具,提高对乳酸菌sRNA的识别和注释水平。大量乳酸菌sRNA的功能和调控机制尚未解析清楚,特别是对sRNA与其靶基因的相互作用和对调控机制的理解还非常有限。我们课题组前期建立了嗜热链球菌、乳酸乳球菌和干酪乳酪杆菌等多种乳酸菌的CRISPR/Cas9、nCas9和dCas9基因编辑工具[53-54],可利用CRISPR工具敲除和弱化sRNA等方式深入研究乳酸菌sRNA生物学功能,结合转录组和蛋白组等多组学分析,揭示乳酸菌sRNA参与的具体通路和调控网络,解析其对乳酸菌代谢、生长和适应性的影响。菌群相互作用在乳酸菌发酵和人体健康中起着至关重要的作用,但乳酸菌sRNA在菌群相互作用中的角色不明,未来可重点关注sRNA可能作为菌群之间传递信息和影响相互作用过程的一种调控工具。通过对乳酸菌sRNA的研究,有望开发出更加高效稳定的乳酸菌发酵工艺,提高食品的质量和口感,开发出新型的益生菌产品和肠道微生物调节剂,用于维护和改善人体营养和健康。随着乳酸菌sRNA研究的不断深入,乳酸菌科学研究和产业化应用会带来更多的创新和突破。
[1] GEORGE F, DANIEL C, THOMAS M, et al. Occurrence and dynamism of lactic acid bacteria in distinct ecological niches: A multifaceted functional health perspective[J]. Frontiers in Microbiology, 2018, 9:2899.
[2] MOKOENA M P. Lactic acid bacteria and their bacteriocins: Classification, biosynthesis and applications against uropathogens: A mini-review[J]. Molecules, 2017, 22(8):1255.
[3] BARROSO F A L, DE JESUS L C L, DE CASTRO C P, et al. Intake of Lactobacillus delbrueckii (pExu: hsp65) prevents the inflammation and the disorganization of the intestinal mucosa in a mouse model of mucositis[J]. Microorganisms, 2021, 9(1):107.
[4] FARAZI T A, JURANEK S A, TUSCHL T. The growing catalog of small RNAs and their association with distinct Argonaute/Piwi family members[J]. Development, 2008, 135(7):1201-1214.
[5] STORZ G, VOGEL J, WASSARMAN K M. Regulation by small RNAs in bacteria: Expanding frontiers[J]. Molecular Cell, 2011, 43(6):880-891.
[6] DUTTA T, SRIVASTAVA S. Small RNA-mediated regulation in bacteria: A growing palette of diverse mechanisms[J]. Gene, 2018, 656:60-72.
[7] BEHLER J, HESS W R. Approaches to study CRISPR RNA biogenesis and the key players involved[J]. Methods, 2020, 172:12-26.
[8] NITZAN M, REHANI R, MARGALIT H. Integration of bacterial small RNAs in regulatory networks[J]. Annual Review of Biophysics, 2017, 46:131-148.
[9] LI W J, YING X M, LU Q X, et al. Predicting sRNAs and their targets in bacteria[J]. Genomics, Proteomics &Bioinformatics, 2012, 10(5):276-284.
[10] ZHANG A X, WASSARMAN K M, ROSENOW C, et al. Global analysis of small RNA and mRNA targets of Hfq[J]. Molecular Microbiology, 2003, 50(4):1111-1124.
[11] VOGEL J, BARTELS V, TANG T H, et al. RNomics in Escherichia coli detects new sRNA species and indicates parallel transcriptional output in bacteria[J]. Nucleic Acids Research, 2003, 31(22):6435-6443.
[12] VAN DER MEULEN S B, JONG A D, KOK J. Early transcriptome response of Lactococcus lactis to environmental stresses reveals differentially expressed small regulatory RNAs and tRNAs[J]. Frontiers in Microbiology, 2017, 8:1704.
[13] DAVANLOO P, ROSENBERG A H, DUNN J J, et al. Cloning and expression of the gene for bacteriophage T7 RNA polymerase[J]. Proceedings of the National Academy of Sciences of the United States of America, 1984, 81(7):2035-2039.
[14] MCCULLEN C A, BENHAMMOU J N, MAJDALANI N, et al. Mechanism of positive regulation by DsrA and RprA small noncoding RNAs: Pairing increases translation and protects rpoS mRNA from degradation[J]. Journal of Bacteriology, 2010, 192(21):5559-5571.
[15] SALVAIL H, LANTHIER-BOURBONNAIS P, SOBOTA J M, et al. A small RNA promotes siderophore production through transcriptional and metabolic remodeling[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(34):15223-15228.
[16] BOUVIER M, SHARMA C M, MIKA F, et al. Small RNA binding to 5′ mRNA coding region inhibits translational initiation[J]. Molecular Cell, 2008, 32(6):827-837.
[17] SONNLEITNER E, BL SI U. Regulation of Hfq by the RNA CrcZ in Pseudomonas aeruginosa carbon catabolite repression[J]. PLoS Genetics, 2014, 10(6): e1004440.
SI U. Regulation of Hfq by the RNA CrcZ in Pseudomonas aeruginosa carbon catabolite repression[J]. PLoS Genetics, 2014, 10(6): e1004440.
[18] MACKIE G A. RNase E: at the interface of bacterial RNA processing and decay[J]. Nature Reviews. Microbiology, 2013, 11(1):45-57.
[19] PRÉVOST K, DESNOYERS G, JACQUES J F, et al. Small RNA-induced mRNA degradation achieved through both translation block and activated cleavage[J]. Genes &Development, 2011, 25(4):385-396.
[20] BAKER C S, MOROZOV I, SUZUKI K, et al. CsrA regulates glycogen biosynthesis by preventing translation of glgC in Escherichia coli[J]. Molecular Microbiology, 2002, 44(6):1599-1610.
[21] WALLING L R, KOUSE A B, SHABALINA S A, et al. A 3′ UTR-derived small RNA connecting nitrogen and carbon metabolism in enteric bacteria[J]. Nucleic Acids Research, 2022, 50(17):10093-10109.
[22] IOSUB I A, MARCHIORETTO M, VAN NUES R W, et al. The mRNA derived MalH sRNA contributes to alternative carbon source utilization by tuning maltoporin expression in E. coli[J]. RNA Biology, 2021, 18(6):914-931.
[23] PU J Y, ZHANG S B, HE X, et al. The small RNA AmiL regulates quorum sensing-mediated virulence in Pseudomonas aeruginosa PAO1[J]. Microbiology Spectrum, 2022, 10(2): e0221121.
[24] ZENG S H, SHI Q X, LIU Y Z, et al. The small RNA PrrH of Pseudomonas aeruginosa regulates hemolysis and oxidative resistance in bloodstream infection[J]. Microbial Pathogenesis, 2023, 180:106124.
[25] ZAPF R L, WIEMELS R E, KEOGH R A, et al. The small RNA Teg41 regulates expression of the alpha phenol-soluble modulins and is required for virulence in Staphylococcus aureus[J]. mBio, 2019, 10(1): e02484-18.
[26] WANG L X, JI C H, XIA X Z, et al. A regulatory SRNA Rli43 is involved in the modulation of biofilm formation and virulence in Listeria monocytogenes[J]. Pathogens, 2022, 11(10):1137.
[27] LI J, NING C C, LI N, et al. The small RNA STnc1480 contributes to the regulation of biofilm formation and pathogenicity in Salmonella typhimurium[J]. Archives of Microbiology, 2022, 204(12):716.
[28] ALTUVIA S, WEINSTEIN-FISCHER D, ZHANG A, et al. A small, stable RNA induced by oxidative stress: Role as a pleiotropic regulator and antimutator[J]. Cell, 1997, 90(1):43-53.
[29] YANG G, LI B A, JIA L L, et al. A novel sRNA in Shigella flexneri that regulates tolerance and virulence under hyperosmotic pressure[J]. Frontiers in Cellular and Infection Microbiology, 2020, 10:483.
[30] VAN DER MEULEN S B, HESSELING-MEINDERS A, JONG A D, et al. The protein regulator ArgR and the sRNA derived from the 3′-UTR region of its gene, ArgX, both regulate the arginine deiminase pathway in Lactococcus lactis[J]. PLoS One, 2019, 14(6): e0218508.
[31] WU H, SONG S Y, TIAN K R, et al. A novel small RNA S042 increases acid tolerance in Lactococcus lactis F44[J]. Biochemical and Biophysical Research Communications, 2018, 500(3):544-549.
[32] LALAOUNA D, BAUDE J, WU Z F, et al. RsaC sRNA modulates the oxidative stress response of Staphylococcus aureus during manganese starvation[J]. Nucleic Acids Research, 2019, 47(18):9871-9887.
[33] DESGRANGES E, BARRIENTOS L, HERRGOTT L, et al. The 3′UTR-derived sRNA RsaG coordinates redox homeostasis and metabolism adaptation in response to glucose-6-phosphate uptake in Staphylococcus aureus[J]. Molecular Microbiology, 2022, 117(1):193-214.
[34] SILVAGGI J M, PERKINS J B, LOSICK R. Small untranslated RNA antitoxin in Bacillus subtilis[J]. Journal of Bacteriology, 2005, 187(19):6641-6650.
[35] SONG A A L, IN L L A, LIM S H E, et al. A review on Lactococcus lactis: From food to factory[J]. Microbial Cell Factories, 2017, 16(1):55.
[36] QI J K, CAIYIN Q, WU H, et al. The novel sRNA s015 improves nisin yield by increasing acid tolerance of Lactococcus lactis F44[J]. Applied Microbiology and Biotechnology, 2017, 101(16):6483-6493.
[37] MIAO S, WU H, ZHAO Y, et al. Enhancing nisin yield by engineering a small noncodding RNA anti41 and inhibiting the expression of glnR in Lactococcus lactis F44[J]. Biotechnology Letters, 2018, 40(6):941-948.
[38] NAKASHIMA Y, SHIIYAMA N, URABE T, et al. Functions of small RNAs in Lactobacillus casei-Pediococcus group of lactic acid bacteria using fragment analysis[J]. FEMS Microbiology Letters, 2020, 367(19): fnaa154.
[39] WADLER C S, VANDERPOOL C K. Characterization of homologs of the small RNA SgrS reveals diversity in function[J]. Nucleic Acids Research, 2009, 37(16):5477-5485.
[40] XIA L, XIA W, LI S H, et al. Identification and expression of small non-coding RNA, L10-Leader, in different growth phases of Streptococcus mutans[J]. Nucleic Acid Therapeutics, 2012, 22(3):177-186.
[41] WANG J C, SUN Z T, QIAO J, et al. Metatranscriptome profiling of the dynamic transcription of mRNA and sRNA of a probiotic Lactobacillus strain in human gut [J]. bioRxiv, 2018: 442673.
[42] LI K W, YANG G J, DEBRU A B, et al. SuhB Regulates the Motile-Sessile Switch in Pseudomonas aeruginosa through the Gac/Rsm Pathway and c-di-GMP Signaling[J]. Frontiers in Microbiology, 2017, 8:1045.
[43] PETERSON J M, PHILLIPS G J. Characterization of conserved bases in 4.5S RNA of Escherichia coli by construction of new F′ factors[J]. Journal of Bacteriology, 2008, 190(23):7709-7718.
[44] GÖßRINGER M, LECHNER M, BRILLANTE N, et al. Protein-only RNase P function in Escherichia coli: Viability, processing defects and differences between PRORP isoenzymes[J]. Nucleic Acids Research, 2017, 45(12):7441-7454.
[45] SHIN J H, PRICE C W. The SsrA-SmpB ribosome rescue system is important for growth of Bacillus subtilis at low and high temperatures[J]. Journal of Bacteriology, 2007, 189(10):3729-3737.
[46] SALIM N N, FANER M A, PHILIP J A, et al. Requirement of upstream Hfq-binding (ARN)x elements in glmS and the Hfq C-terminal region for GlmS upregulation by sRNAs GlmZ and GlmY[J]. Nucleic Acids Research, 2012, 40(16):8021-8032.
[47] FOWLER Z M, BRONOVITSKIY S S, ROSE F K, et al. Identification of a small regulatory RNA UspS associated with the universal stress protein in Lactobacillus species[J]. Journal of the South Carolina Academy of Science, 2023, 21(2): 3.
[48] ZHENG H J, LIU E N, SHI T, et al. Strand-specific RNA-seq analysis of the Lactobacillus delbrueckii subsp. bulgaricus transcriptome[J]. Molecular BioSystems, 2016, 12(2):508-519.
[49] XIONG Z Q, LV Z X, SONG X, et al. Recent research advances in small regulatory RNAs in Streptococcus[J]. Current Microbiology, 2021, 78(6):2231-2241.
[50] KARVELIS T, GASIUNAS G, MIKSYS A, et al. crRNA and tracrRNA guide Cas9-mediated DNA interference in Streptococcus thermophilus[J]. RNA Biology, 2013, 10(5):841-851.
[51] LIU G F, CHANG H D, QIAO Y L, et al. Profiles of small regulatory RNAs at different growth phases of Streptococcus thermophilus during pH-Controlled batch fermentation[J]. Frontiers in Microbiology, 2021, 12:765144.
[52] BUSBY L B, HOXHA K, OAKES W C III, et al. Identification of small regulatory RNA transcripts in extracellular vesicles from lactic acid bacteria[J]. The FASEB Journal, 2022, 36(S1): R5232.
[53] SONG X, HUANG H, XIONG Z Q, et al. CRISPR-Cas9D10A Nickase-Assisted Genome Editing in Lactobacillus casei[J]. Applied and Environmental Microbiology, 2017, 83(22): e01259-17.
[54] KONG L H, XIONG Z Q, SONG X, et al. CRISPR/dCas9-based metabolic pathway engineering for the systematic optimization of exopolysaccharide biosynthesis in Streptococcus thermophilus[J]. Journal of Dairy Science, 2022, 105(8):6499-6512.